Chapter 14 簡單線性迴歸分析二部曲
14.1 續
14.1.1 簡單線性迴歸分析首部曲的「續」
繼續往前走之前,作者小編一定要再一次提醒大家,本波士頓數據集案例分析微微書的總目標是
搜尋「1970年代波士頓都會區的最佳『hedonic housing price model』」,某一種迴歸模型。
接下來,我們繼續研究「兩個連續型變數的迴歸模型」,尤其是固定一個變數在「房價中位數」。「房價中位數」是我們「散佈圖」的「y變數」,其他連續型變數則是我們的「x變數」,而且一次只來、只看一個。沿著、順著這樣的「探索之路」,作者小編想在這一章好好解釋、分析、視覺化「簡單線性迴歸模型」。「簡單線性迴歸模型」是最簡單的「兩個連續型變數的迴歸模型」:
\[y = \beta_0 + \beta_1 x + \epsilon\]
接下來幾章,除了介紹「最小平方法(LS)」,也會介紹「LASSO」跟「Ridge」。進而介紹作者小編土耳其同學反對的「機器學習(ML)」。以上不論是
- LS
- LASSO
- Ridge
- ML
- …
都是「挑選模型」的標準,或說是「估計模型」的演算法。「估計」兩個字對讀者諸君而言,或許太遙遠,但「估計」其實骨子裡就是「猜測」,不只是「有憑有據的猜測」,而且這些「猜測」都必須根據「樣本」。現實裡的「樣本」,讓「估計、猜測」的辦法不會只有一種,也就是說,現實裡沒有打遍天下無敵手的猜測辦法,意思是說「沒有贏家演算法」。即便如此,無論如何,接下來R將協助我們全掌握「簡單線性迴歸分析」,並且學習各種演算法的優缺點。
…
作者小編一路寫下來,感覺上,好像我們已經遠離了「分配」。事實上不然,因為「簡單線性迴歸模型」,「x變數」把「y變數的分配」切成許許多多的「小分配」。然後,統計學家企圖把這一些「小分配的平均數」連成「一條直線」:
\[E(y|x = x_0) = \beta_0 + \beta_1 x_0\]
只是不知道,\(\beta_0\)跟\(\beta_1\)到底在哪裡?到底有多大?而已。接下來,我們將好好想辦法
發展
找到\(\beta_0\)跟\(\beta_1\)的辦法、技術、數學、…,任何科學的辦法。
…
14.1.2 繼續的「續」
這一章將繼續談「最小平方法」。上一章,作者小編談過
- 引用知名數據集「
anscombe」示範「散佈圖」的重要性、 - 什麼是「最小平方法」?
- 引用「相關係數」挑「簡單線性迴歸線」的「解釋變數」、
- 變數變換如何影響「簡單線性迴歸線」的「解釋能力」、
- 取得「最小平方法迴歸線」的報表、
- 抓取報表內的各種數字、
接著,我們將討論
- 最小平方法「
lsfit」家族的其他成員,「ls.print」跟「ls.diag」、 - 什麼是「迴歸診斷」?
- 深入探討知名數據集「
anscombe」的第二組、第三組跟第四組子數據集、 - 介紹最簡單的「非線性迴歸線」、
- 示範各種波士頓數據集「房價中位數」與「低社經人口比例」的「非線性迴歸線」、
作者小編持續努力為大家「『示範』『程式能力』的重要性」。
14.3 準備工作
「學習」其實是「找答案之旅」。
如果沒有問題,作者小編認為「不一定要學習」。但是,作者小編認為
為個人增添「光」與「彩」也是一種問題(『How』跟『Why』的問題),
所以嚴格說起來,「學習」是一條「必經之路」。為此,作者小編在這一整套微微書總是「不厭其煩」地出現「昔日在前面章節出現過的程式碼」,還請讀者諸君見諒!
14.3.1 波士頓數據集
require(MASS) # 用「require」不用「library」,是因為如果R已經將套件MASS載入環境,就不需要再載一次。
data(Boston)
boston <- Boston
# 「不動」原始數據集,不論它有「多原始」,是R程式設計的一項絕佳習慣。
# 即便一般使用者是無法任意改變套件MASS的內容物!
### rad (index of accessibility to radial highways)
boston[,"rad"] <- factor(boston[,"rad"], ordered = TRUE)
### chas (= 1 if tract bounds river; 0 otherwise)
boston[,"chas"] <- factor(boston[,"chas"])
head(boston)## crim zn indus chas nox rm age dis rad tax ptratio black lstat
## 1 0.00632 18 2.31 0 0.538 6.575 65.2 4.0900 1 296 15.3 396.90 4.98
## 2 0.02731 0 7.07 0 0.469 6.421 78.9 4.9671 2 242 17.8 396.90 9.14
## 3 0.02729 0 7.07 0 0.469 7.185 61.1 4.9671 2 242 17.8 392.83 4.03
## 4 0.03237 0 2.18 0 0.458 6.998 45.8 6.0622 3 222 18.7 394.63 2.94
## 5 0.06905 0 2.18 0 0.458 7.147 54.2 6.0622 3 222 18.7 396.90 5.33
## 6 0.02985 0 2.18 0 0.458 6.430 58.7 6.0622 3 222 18.7 394.12 5.21
## medv
## 1 24.0
## 2 21.6
## 3 34.7
## 4 33.4
## 5 36.2
## 6 28.7tail(boston)## crim zn indus chas nox rm age dis rad tax ptratio black lstat
## 501 0.22438 0 9.69 0 0.585 6.027 79.7 2.4982 6 391 19.2 396.90 14.33
## 502 0.06263 0 11.93 0 0.573 6.593 69.1 2.4786 1 273 21.0 391.99 9.67
## 503 0.04527 0 11.93 0 0.573 6.120 76.7 2.2875 1 273 21.0 396.90 9.08
## 504 0.06076 0 11.93 0 0.573 6.976 91.0 2.1675 1 273 21.0 396.90 5.64
## 505 0.10959 0 11.93 0 0.573 6.794 89.3 2.3889 1 273 21.0 393.45 6.48
## 506 0.04741 0 11.93 0 0.573 6.030 80.8 2.5050 1 273 21.0 396.90 7.88
## medv
## 501 16.8
## 502 22.4
## 503 20.6
## 504 23.9
## 505 22.0
## 506 11.9### 讀取自製的波士頓數據集中文資訊。
DISvars <- colnames(boston)[which(sapply(boston, class) == "integer")]
CONvars <- colnames(boston)[which(sapply(boston, class) == "numeric")]
colsBostonFull<-readRDS("output/data/colsBostonFull.rds")
colsBostonFull$內容物 <- sapply(boston, class)
colsBostonFull## 變數名
## 1 crim
## 2 zn
## 3 indus
## 4 chas
## 5 nox
## 6 rm
## 7 age
## 8 dis
## 9 rad
## 10 tax
## 11 ptratio
## 12 black
## 13 lstat
## 14 medv
## 說明
## 1 per capita crime rate by town
## 2 proportion of residential land zoned for lots over 25,000 sq.ft.
## 3 proportion of non-retail business acres per town
## 4 Charles River dummy variable (= 1 if tract bounds river; 0 otherwise)
## 5 nitric oxides concentration (parts per 10 million)
## 6 average number of rooms per dwelling
## 7 proportion of owner-occupied units built prior to 1940
## 8 weighted distances to five Boston employment centres
## 9 index of accessibility to radial highways
## 10 full-value property-tax rate per $10,000
## 11 pupil-teacher ratio by town
## 12 1000(Bk - 0.63)^2 where Bk is the proportion of blacks by town
## 13 % lower status of the population
## 14 Median value of owner-occupied homes in $1000's
## 中文翻譯 中文變數名稱 內容物
## 1 每個城鎮人均犯罪率。 犯罪率 numeric
## 2 超過25,000平方呎的住宅用地比例。 住宅用地比例 numeric
## 3 每個城鎮非零售業務英畝的比例。 非商業區比例 numeric
## 4 是否鄰近Charles River。 河邊宅 factor
## 5 氮氧化合物濃度。 空汙指標 numeric
## 6 每個住宅的平均房間數。 平均房間數 numeric
## 7 1940年之前建造的自用住宅比例。 老房子比例 numeric
## 8 距波士頓五大商圈的加權平均距離。 加權平均距離 numeric
## 9 環狀高速公路的可觸指標。 交通便利性 ordered, factor
## 10 財產稅占比(每一萬美元)。 財產稅率 numeric
## 11 生師比。 生師比 numeric
## 12 黑人的比例。 黑人指數 numeric
## 13 (比較)低社經地位人口的比例。 低社經人口比例 numeric
## 14 以千美元計的房價中位數。 房價中位數 numeric14.3.2 anscombe數據集
這一組Tufte教授在「The Visual Display of Quantitative Information」這本書的第十頁用來示範「散佈圖」的數據集,源自於
Anscombe, Francis J. (1973). Graphs in statistical analysis. The American Statistician, 27, 17–21.
anscombe這一個字,在統計界已經是「幾乎一樣的平均數、標準差加相關係數卻完全不一樣的兩維數據集」的「代名詞」了!後來又有兩位學者「Justin Matejka」跟「George Fitzmaurice」循著同樣的思路,利用「Simulated Annealing」演算法找到「比anscombe更anscombe」的數據集,
作者小編企圖在這裡「深入挖掘」這兩大組數據集可能隱藏的「教學契機」。請讀者諸君不要轉台!
require(datasets) # 通常我們是這麼寫「library(datasets)」。
data("anscombe") # 載入。14.4 回顧最小平方法
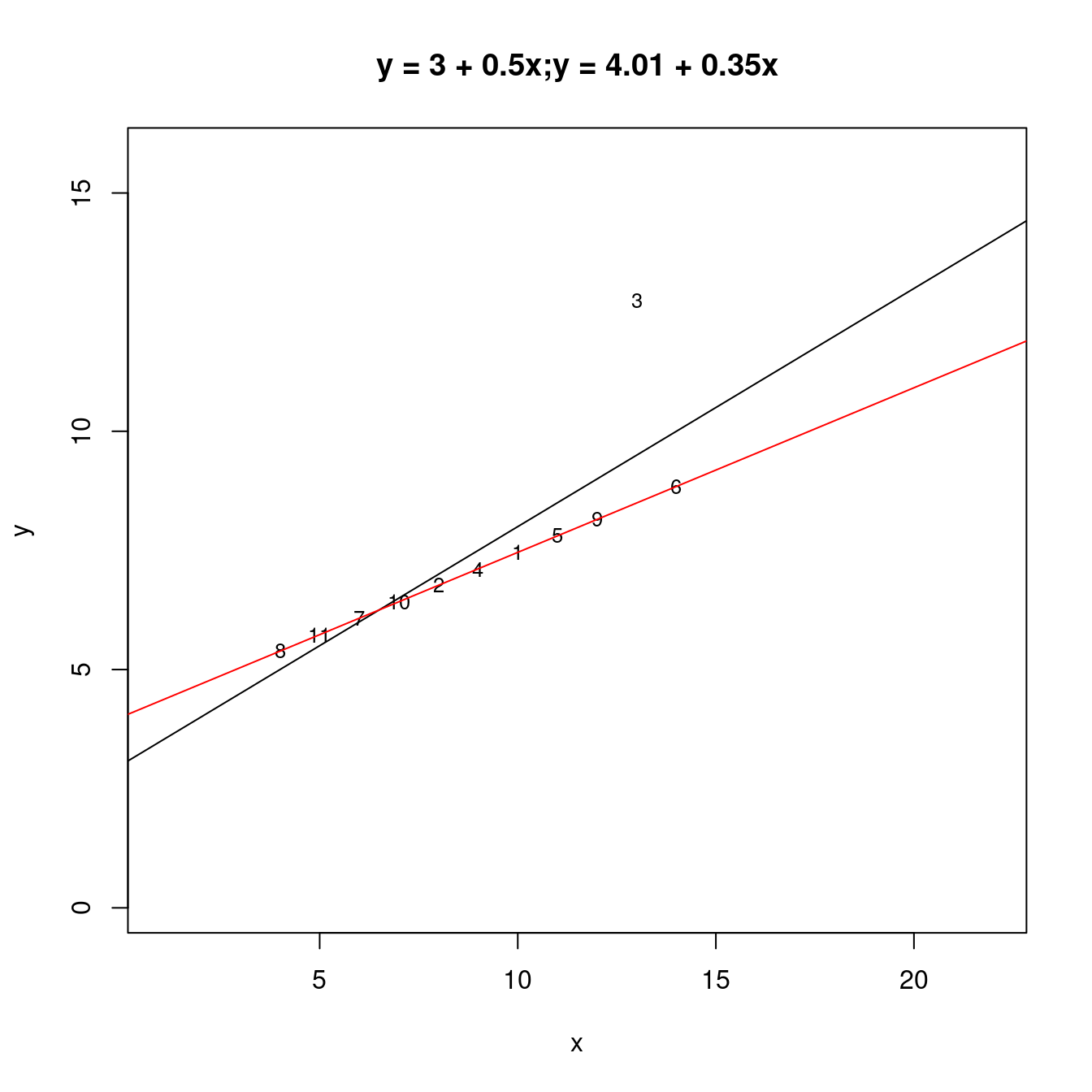
在上一章,作者小編這麼示範「最小平方法」:
「最小平方法迴歸線」在幾何上,就是「按著那顆『黑點』轉動『直線』直到『SSE』出現最小為止」,得到的那一條直線就是答案,就是「最小平方法迴歸線」。
請看作者小編示範這個想法、理論、演算法:
- (動作一:準備動作)準備數據,加上計算單變量與雙變量的摘要統計量。
df <- anscombe[, c("x1", "y1")] # 取出「x1」跟「y1」,其中「1」代表第一組。
colnames(df) <- c("x", "y") # 劃掉其中的「1」。
df # 呼叫出來再一次檢視這一組數據集。## x y
## 1 10 8.04
## 2 8 6.95
## 3 13 7.58
## 4 9 8.81
## 5 11 8.33
## 6 14 9.96
## 7 6 7.24
## 8 4 4.26
## 9 12 10.84
## 10 7 4.82
## 11 5 5.68summary(df) # 單變量的摘要統計量。## x y
## Min. : 4.0 Min. : 4.260
## 1st Qu.: 6.5 1st Qu.: 6.315
## Median : 9.0 Median : 7.580
## Mean : 9.0 Mean : 7.501
## 3rd Qu.:11.5 3rd Qu.: 8.570
## Max. :14.0 Max. :10.840cor(df) # 雙變量的摘要統計量。## x y
## x 1.0000000 0.8164205
## y 0.8164205 1.0000000# 繪製散佈圖。左右上下各加3個單位,方便大家觀察。
plot(df,
xlim = range(df[, "x"]) + c(-3, 3),
ylim = range(df[, "y"]) + c(-3, 3))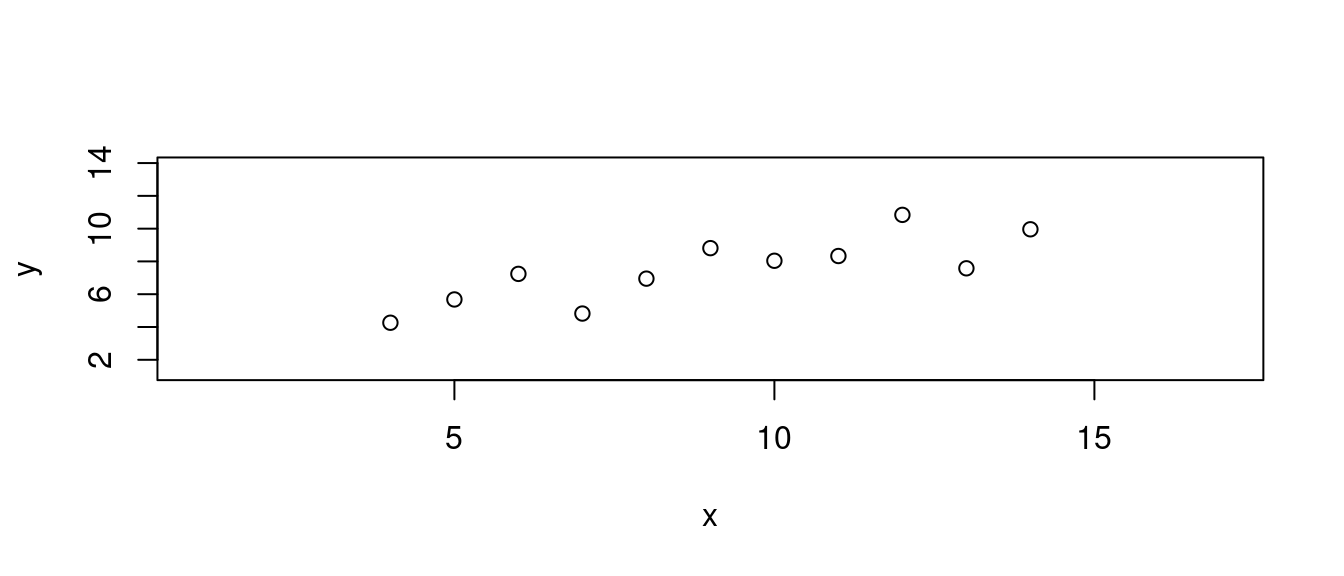
- (動作二:搜尋最小平方法迴歸線)呼叫最小平方法R的函式「
lsfit」。
###
m1 <- lsfit(df[c(1,2,3,4,5,6,7,8,9,10,11), "x"], df[c(1,2,3,4,5,6,7,8,9,10,11), "y"])
m1$coefficients## Intercept X
## 3.0000909 0.5000909- (動作三:再一次搜尋最小平方法迴歸線)現在,只有「一個x」加「一個y」,那「最小平方法迴歸線」在幾何上,就是「按著那顆『黑點』轉動『直線』直到『\(SSE\)』出現最小值為止」,得到的那一條直線就是答案,就是「最小平方法迴歸線」。
###
SSE <- numeric(0) # 準備放置各種可能直線的SSE。
ybar <- mean(df[, "y"]) # 全部y座標的平均數。
xbar <- mean(df[, "x"]) # 全部x座標的平均數。
for (b in seq(-1, 1, 0.1)) {
a <- ybar - b * xbar
predy <- df[, "y"] - (a + b * df[, "x"])
SSE <- c(SSE, sum(predy^2))
}
# 透過觀察散佈圖,或是計算幾組點對點的斜率,定調「斜率」可能的範圍。
# 加上「最小平方法迴歸線」一定通過(xbar,ybar)這一點,就可以取得對應某一個斜率的「截距」。
plot(seq(-1, 1, 0.1), SSE, type = "l") # 顯示SSE的搜尋成果。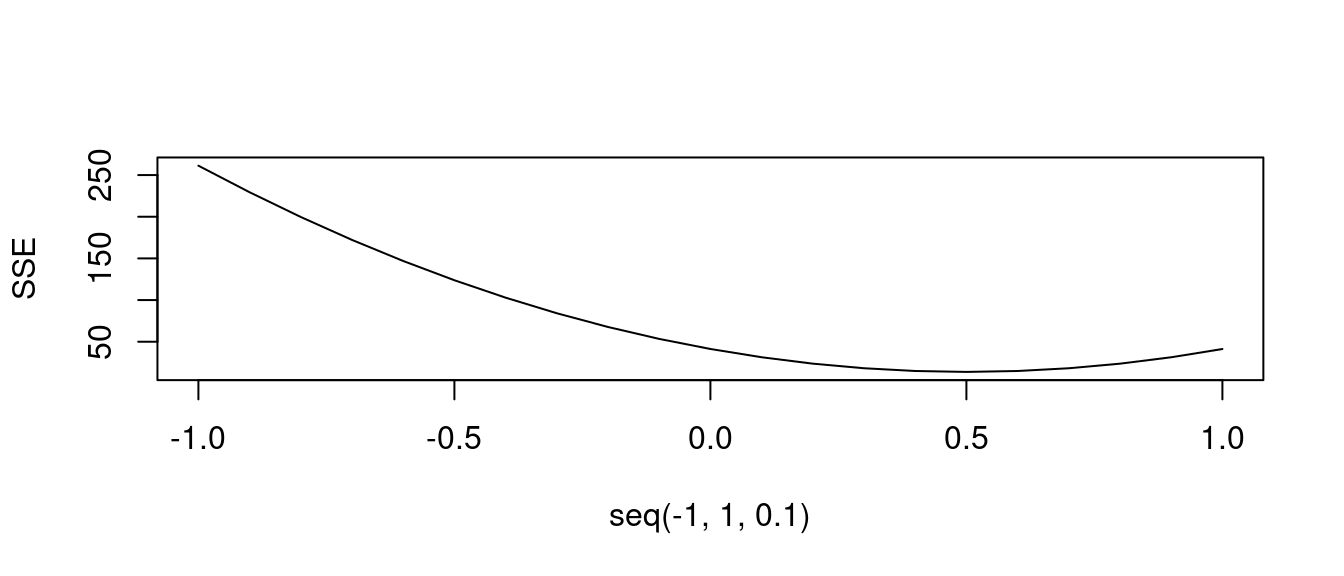
b <- seq(-1, 1, 0.1)[which.min(SSE)] # 最小化SSE的斜率等於多少?
a <- ybar - b * xbar # 對應上述斜率的截距。
LINE <- c(a, b)
names(LINE) <- c("a", "b")
LINE # 報告成果。## a b
## 3.000909 0.500000- (動作四:驗證作者小編的理論)結論:答案是一樣的!也就是說,作者小編的理論得到證實。
plot(df[, c("x", "y")],
xlim = range(df[, "x"]) + c(-3, 3),
ylim = range(df[, "y"]) + c(-3, 3)) # 最簡散佈圖。
abline(a = LINE["a"], b = LINE["b"], col = "red", lwd = 8) # 畫上搜尋成果的直線。
abline(m1, lwd = 3, col = "green") # 畫上R提供的答案直線。
title(paste0("迴圈搜尋成果:", "y = ",
round(LINE["a"], 2),
" + ", round(LINE["b"], 2), "x",
"; R提供的成果:",
"y = ",
round(m1$coefficients[1], 2),
" + ", round(m1$coefficients[2], 2), "x"))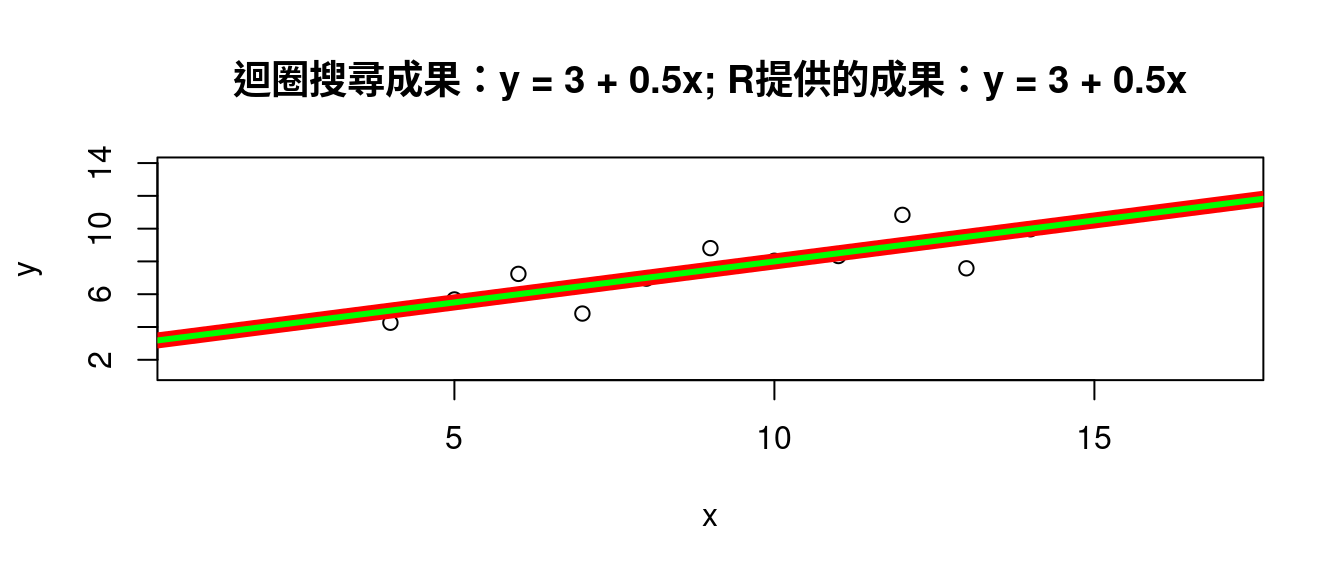
…
14.5 最小平方法的二部曲
整個發展已經進入第六章了,作者小編好像進入了「腸枯思竭」的狀態了。遇上這種「時候」,作者小編會「停下來翻書,找答案、找靈感」。
停在哪裡,就從哪裡找書、翻書?
所以,在回顧作者小編前一章的程式碼之前,讓我們先檢視「好幾下」最小平方法(lsfit)的R官方使用手冊:
knitr::include_url("https://www.rdocumentation.org/packages/stats/versions/3.6.2/topics/lsfit")作者小編到目前為止,未曾討論過
如何『使用』使用手冊?
幾個重點:
- (重點一)先仔細閱讀「簡介」,簡介「只是」一段言簡意賅的英文。比如說,「
lsfit」的簡介就是
Find The Least Squares Fit
The least squares estimate of in the model \(\bold Y = \bold X \beta + \bold \epsilon\) is found.
第一段讓我們知道「lsfit」這個「名字」是怎麼來的?接下來,「lsfit作者介紹『lsfit』的主要功能」。
- (重點二)剪貼範例到自己的環境,並進行測試。比如說,這兩句話就是「
lsfit作者」提供的範例:
lsD9 <- lsfit(x = unclass(gl(2, 10)), y = weight)
ls.print(lsD9)- (重點三)在「See Also」這一段,「手冊作者」會建議「同時參考的手冊」。比如說,「
lsfit作者」建議了「lm」、「ls.print」、「ls.diag」。
…
作者小編原本安排在這一章討論「
LASSO」跟「Ridge」,只根據個人看過「符號」之後對這兩種演算法的想像,但經過測試,發現事實不然,「LASSO」跟「Ridge」無法用在「簡單線性迴歸建模」。作者小編心想「慘了」。好消息是
2021/02/08的早上看過一本談論「深度學習(Deep Learning)」的書,提到「挖掘結構的演算法」;
同一天,驚覺尚未討論「迴歸建模的第三階段:迴歸診斷」,在前一章,作者小編只顯示一張表跟幾張圖,完全沒解釋「什麼是迴歸診斷」。同時也瀏覽了「
ls.diag」的「Value」橋段。…
基於這兩點,作者小編找到了這一章的題材!!!
…
嚴格說起來,作者小編在上一章完全倚重「lm」為大家解析「如何搜尋最小平方法迴歸線」。雖然,官方使用手冊也是這麼建議
lm which usually is preferable;
但是,「lsfit」家族的另外兩位成員:「ls.print」跟「ls.diag」完整呈現「如何搜尋最小平方法迴歸線」的三部曲:
- 找線
- 列印
- 診斷
### 找線
m1 <- lsfit(df[, "x"], df[, "y"])
### 列印
ls.print(m1)## Residual Standard Error=1.2366
## R-Square=0.6665
## F-statistic (df=1, 9)=17.9899
## p-value=0.0022
##
## Estimate Std.Err t-value Pr(>|t|)
## Intercept 3.0001 1.1247 2.6673 0.0257
## X 0.5001 0.1179 4.2415 0.0022### 診斷
ls.diag(m1)## $std.dev
## [1] 1.236603
##
## $hat
## [1] 0.10000000 0.10000000 0.23636364 0.09090909 0.12727273 0.31818182
## [7] 0.17272727 0.31818182 0.17272727 0.12727273 0.23636364
##
## $std.res
## [1] 0.03324397 -0.04331791 -1.77793266 1.11028824 -0.14810075 -0.04050923
## [7] 1.10190458 -0.72515977 1.63487302 -1.45488131 0.16606601
##
## $stud.res
## [1] 0.03134464 -0.04084477 -2.08109891 1.12679993 -0.13980118 -0.03819595
## [7] 1.11695887 -0.70458079 1.83833042 -1.56846043 0.15680897
##
## $cooks
## [1] 0.00006139788 0.00010424672 0.48920927577 0.06163699895 0.00159934188
## [6] 0.00038289951 0.12675648475 0.12269989634 0.27902959338 0.15434122237
## [11] 0.00426801143
##
## $dfits
## [1] 0.01044821 -0.01361492 -1.15781655 0.35632543 -0.05338746 -0.02609280
## [7] 0.51037958 -0.48132031 0.84000076 -0.59896572 0.08724046
##
## $correlation
## Intercept X
## Intercept 1.0000000 -0.9434564
## X -0.9434564 1.0000000
##
## $std.err
## [,1]
## Intercept 1.1247468
## X 0.1179055
##
## $cov.scaled
## Intercept X
## Intercept 1.2650553 -0.12511536
## X -0.1251154 0.01390171
##
## $cov.unscaled
## Intercept X
## Intercept 0.82727273 -0.081818182
## X -0.08181818 0.00909090914.5.1 把ls.print的輸出統計量收錄在一張表
x <- sapply(1, function(w){
ls.print(m1)
})## Residual Standard Error=1.2366
## R-Square=0.6665
## F-statistic (df=1, 9)=17.9899
## p-value=0.0022
##
## Estimate Std.Err t-value Pr(>|t|)
## Intercept 3.0001 1.1247 2.6673 0.0257
## X 0.5001 0.1179 4.2415 0.0022x## [,1]
## summary character,6
## coef.table list,1x["summary",1]## $summary
## Mean Sum Sq R Squared F-value Df 1 Df 2 Pr(>F)
## [1,] "1.2366" "0.6665" "17.9899" "1" "9" "0.0022"x["summary",1]$summary## Mean Sum Sq R Squared F-value Df 1 Df 2 Pr(>F)
## [1,] "1.2366" "0.6665" "17.9899" "1" "9" "0.0022"x["coef.table",1]## $coef.table
## $coef.table[[1]]
## Estimate Std.Err t-value Pr(>|t|)
## Intercept 3.0000909 1.1247468 2.667348 0.025734051
## X 0.5000909 0.1179055 4.241455 0.002169629x["coef.table",1]$coef.table## [[1]]
## Estimate Std.Err t-value Pr(>|t|)
## Intercept 3.0000909 1.1247468 2.667348 0.025734051
## X 0.5000909 0.1179055 4.241455 0.002169629x["coef.table",1]$coef.table[[1]]## Estimate Std.Err t-value Pr(>|t|)
## Intercept 3.0000909 1.1247468 2.667348 0.025734051
## X 0.5000909 0.1179055 4.241455 0.002169629檢查過後,
LSsum <- x["summary",1]$summary
class(LSsum)## [1] "matrix" "array"LScoef <- x["coef.table",1]$coef.table[[1]]
class(LScoef)## [1] "matrix" "array"14.5.2 把ls.diag的輸出統計量收錄在一張表
x <- sapply(1, function(w){
ls.diag(m1)
})
x## [,1]
## std.dev 1.236603
## hat numeric,11
## std.res numeric,11
## stud.res numeric,11
## cooks numeric,11
## dfits numeric,11
## correlation numeric,4
## std.err numeric,2
## cov.scaled numeric,4
## cov.unscaled numeric,414.5.2.1 關於迴歸係數的摘要統計量
x["std.err",1]## $std.err
## [,1]
## Intercept 1.1247468
## X 0.1179055x["std.err",1]$std.err## [,1]
## Intercept 1.1247468
## X 0.1179055class(x["std.err",1]$std.err)## [1] "matrix" "array"x["std.err",1]$std.err["Intercept",1]## Intercept
## 1.124747x["std.err",1]$std.err["X",1]## X
## 0.1179055x["correlation",1]## $correlation
## Intercept X
## Intercept 1.0000000 -0.9434564
## X -0.9434564 1.0000000x["correlation",1]$correlation## Intercept X
## Intercept 1.0000000 -0.9434564
## X -0.9434564 1.0000000class(x["correlation",1]$correlation)## [1] "matrix" "array"x["correlation",1]$correlation["Intercept", "X"]## [1] -0.943456414.5.2.2 關於戴帽子y的摘要統計量
x["hat",1]$hat## [1] 0.10000000 0.10000000 0.23636364 0.09090909 0.12727273 0.31818182
## [7] 0.17272727 0.31818182 0.17272727 0.12727273 0.23636364x["std.res",1]$std.res## [1] 0.03324397 -0.04331791 -1.77793266 1.11028824 -0.14810075 -0.04050923
## [7] 1.10190458 -0.72515977 1.63487302 -1.45488131 0.16606601x["stud.res",1]$stud.res## [1] 0.03134464 -0.04084477 -2.08109891 1.12679993 -0.13980118 -0.03819595
## [7] 1.11695887 -0.70458079 1.83833042 -1.56846043 0.15680897x["cooks",1]$cooks## [1] 0.00006139788 0.00010424672 0.48920927577 0.06163699895 0.00159934188
## [6] 0.00038289951 0.12675648475 0.12269989634 0.27902959338 0.15434122237
## [11] 0.00426801143x["dfits",1]$dfits## [1] 0.01044821 -0.01361492 -1.15781655 0.35632543 -0.05338746 -0.02609280
## [7] 0.51037958 -0.48132031 0.84000076 -0.59896572 0.0872404614.5.3 請專家幫忙
m1 <- lm(y ~ x, data = df)
require(broom)
tidy(m1)## # A tibble: 2 × 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) 3.00 1.12 2.67 0.0257
## 2 x 0.500 0.118 4.24 0.00217augment(m1)## # A tibble: 11 × 8
## y x .fitted .resid .hat .sigma .cooksd .std.resid
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 8.04 10 8.00 0.0390 0.100 1.31 0.0000614 0.0332
## 2 6.95 8 7.00 -0.0508 0.1 1.31 0.000104 -0.0433
## 3 7.58 13 9.50 -1.92 0.236 1.06 0.489 -1.78
## 4 8.81 9 7.50 1.31 0.0909 1.22 0.0616 1.11
## 5 8.33 11 8.50 -0.171 0.127 1.31 0.00160 -0.148
## 6 9.96 14 10.0 -0.0414 0.318 1.31 0.000383 -0.0405
## 7 7.24 6 6.00 1.24 0.173 1.22 0.127 1.10
## 8 4.26 4 5.00 -0.740 0.318 1.27 0.123 -0.725
## 9 10.8 12 9.00 1.84 0.173 1.10 0.279 1.63
## 10 4.82 7 6.50 -1.68 0.127 1.15 0.154 -1.45
## 11 5.68 5 5.50 0.179 0.236 1.31 0.00427 0.166glance(m1)## # A tibble: 1 × 12
## r.squared adj.r.squared sigma statistic p.value df logLik AIC BIC
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 0.667 0.629 1.24 18.0 0.00217 1 -16.8 39.7 40.9
## # ℹ 3 more variables: deviance <dbl>, df.residual <int>, nobs <int>14.7 迴歸診斷當x變數是低社經人口比例時的最小平方法迴歸線
CORmedv <- cor(x = boston[, CONvars[-12]], y = boston[, "medv"])
maxCOR <- rownames(CORmedv)[which.max(abs(CORmedv[,1]))]
plot(boston[, maxCOR], boston[, "medv"],
xlab = colsBostonFull$中文變數名稱[which(colsBostonFull$變數名 == maxCOR)],
ylab = "房價中位數")
title(paste0("最高相關係數", " = ",
cor(boston[, maxCOR],
boston[, "medv"])))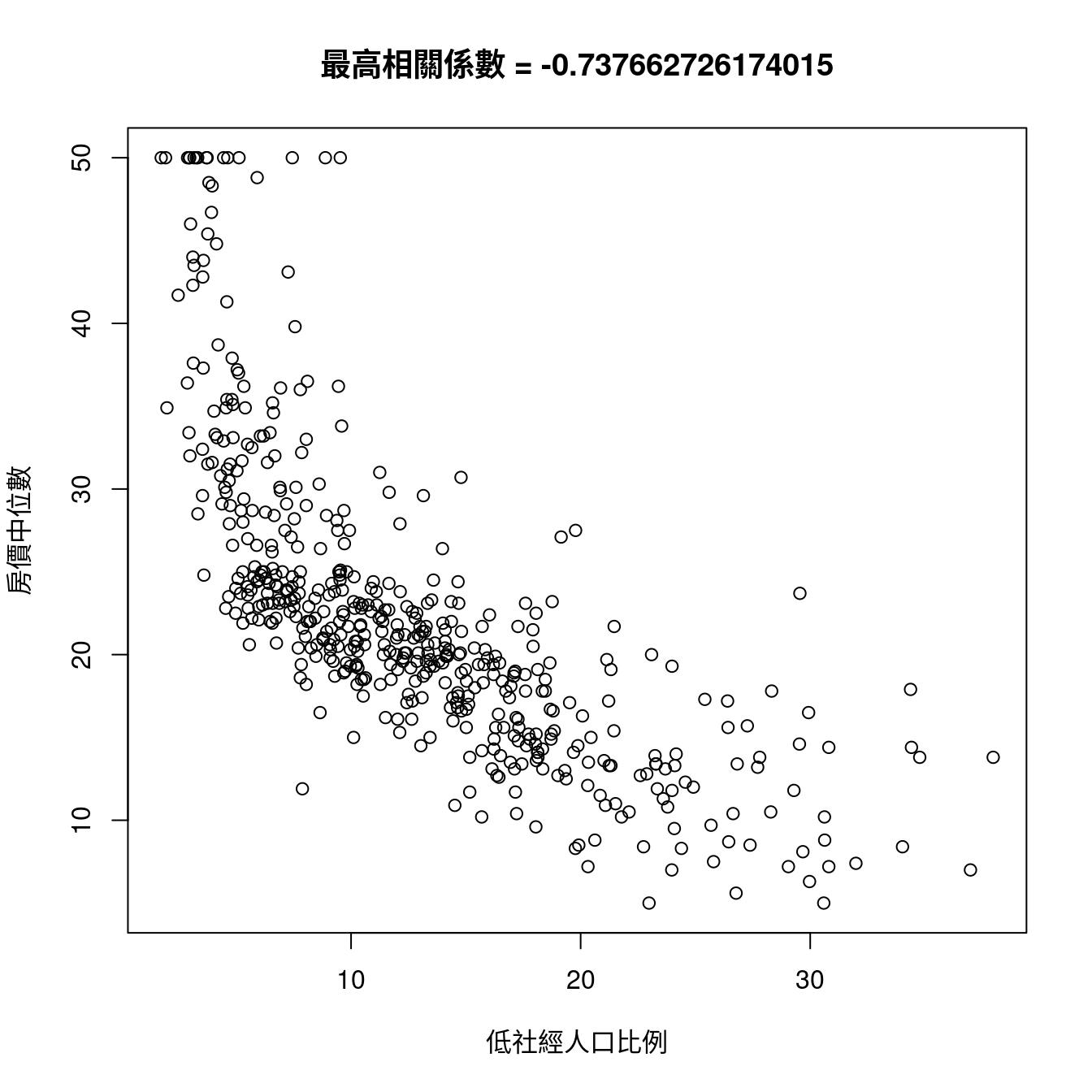
m1 <- lm(medv ~ lstat, data = boston)
par(mfrow = c(2, 2))
plot(m1)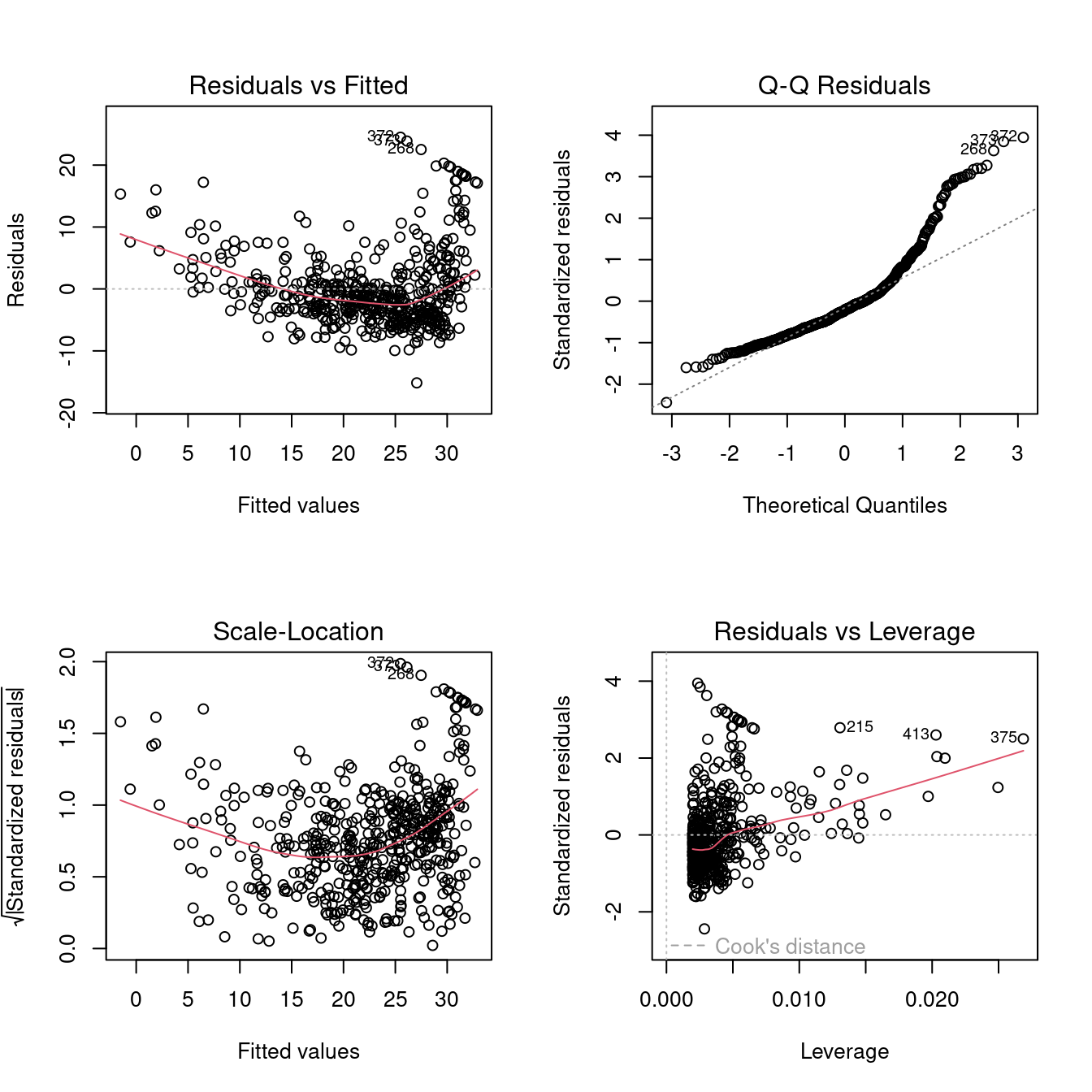
boston$lstat10 <- boston$lstat^(1/10)
m0 <- lm(medv ~ lstat10, data = boston)
par(mfrow = c(2, 2))
plot(m0)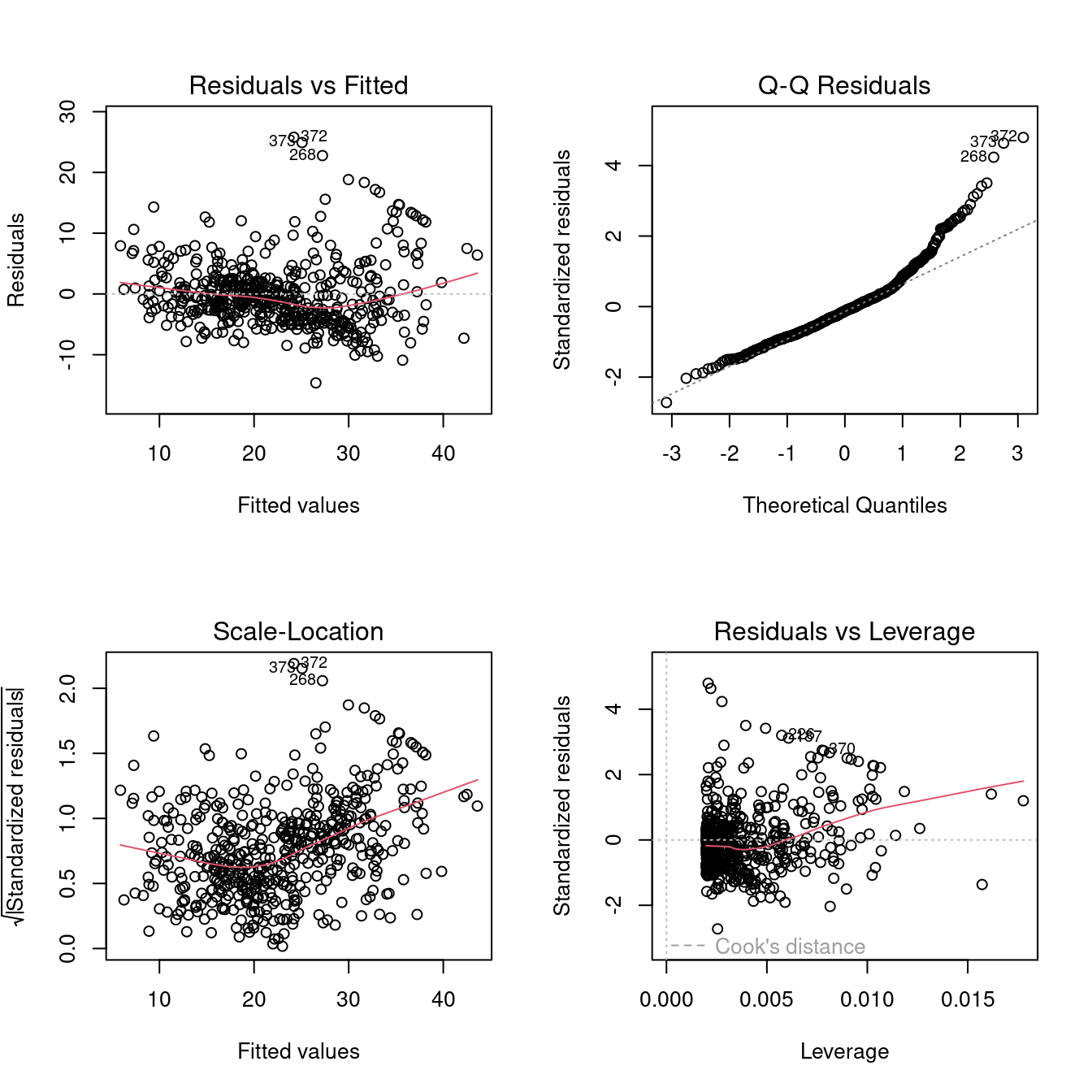
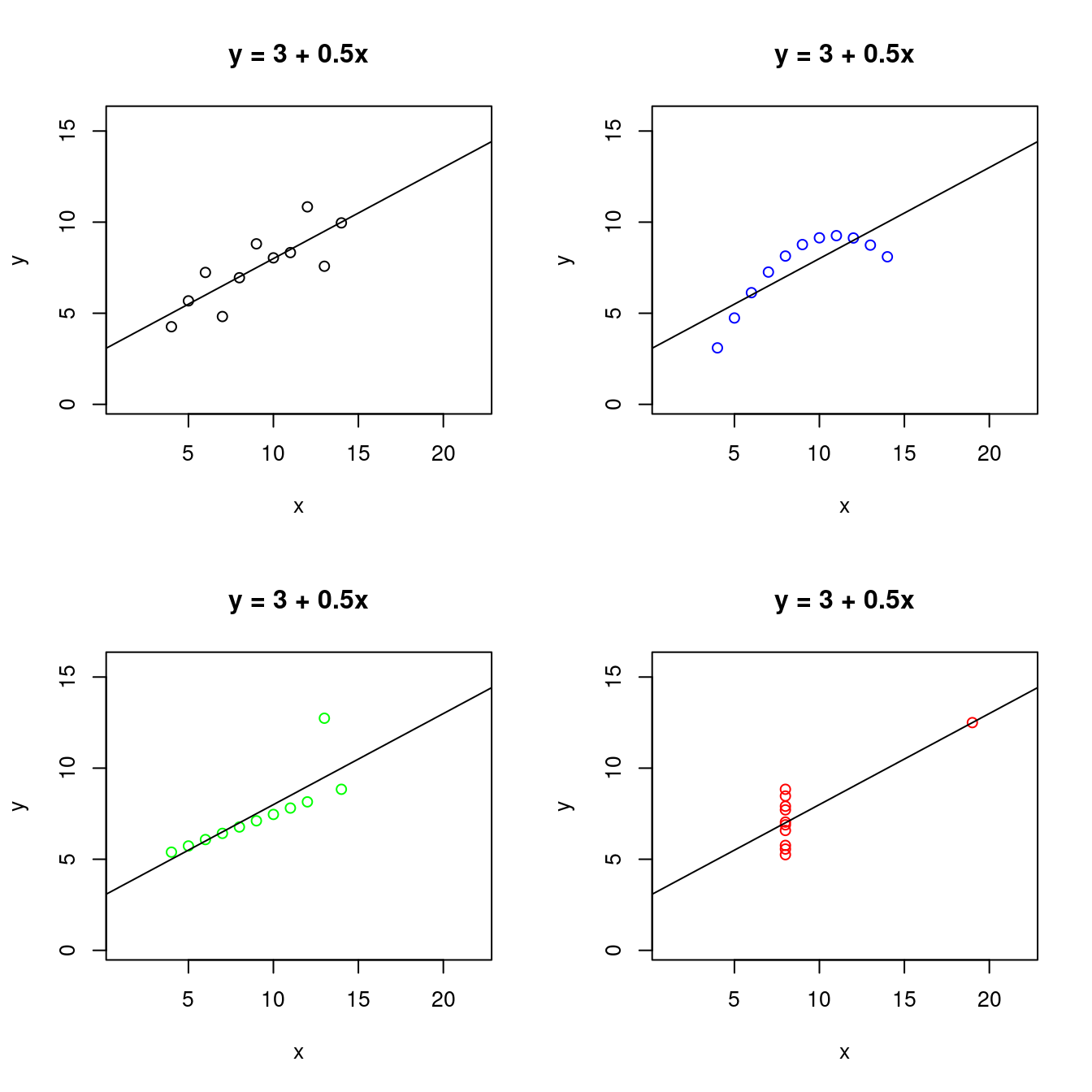
14.8 繼續挖Anscombe數據集
全部子數據集的散佈圖:
14.8.2 繼續挖Anscombe數據集之「y2」
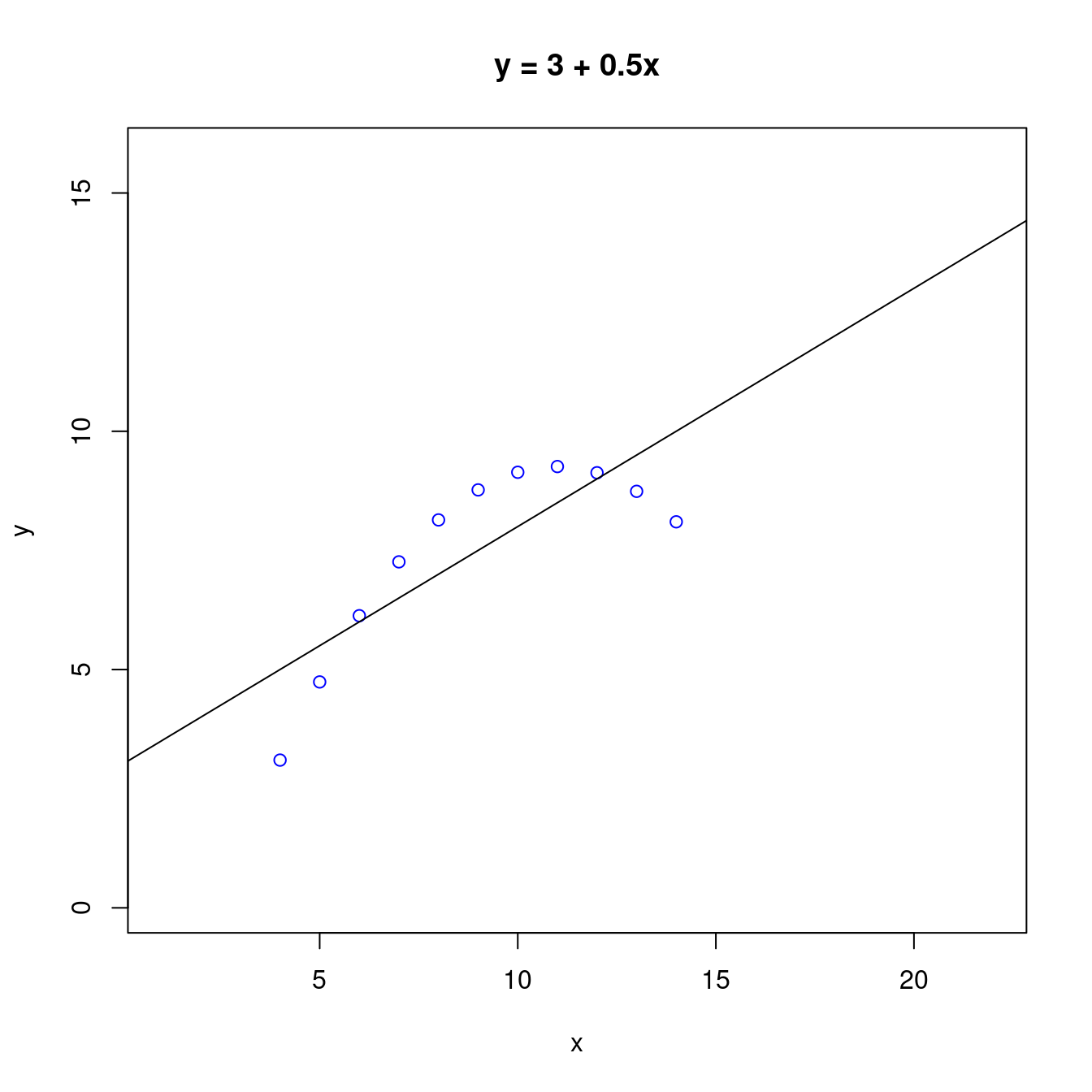
Q1: 找到曲線方程式的估計?
df <- anscombe[, c("x2", "y2")] # 取出「x1」跟「y1」,其中「1」代表第一組。
colnames(df) <- c("x", "y") # 劃掉其中的「1」。
df # 呼叫出來再一次檢視這一組數據集。## x y
## 1 10 9.14
## 2 8 8.14
## 3 13 8.74
## 4 9 8.77
## 5 11 9.26
## 6 14 8.10
## 7 6 6.13
## 8 4 3.10
## 9 12 9.13
## 10 7 7.26
## 11 5 4.74# 繪製散佈圖。左右上下各加3個單位,方便大家觀察。
plot(df,
xlim = range(df[, "x"]) + c(-3, 3),
ylim = range(df[, "y"]) + c(-3, 3))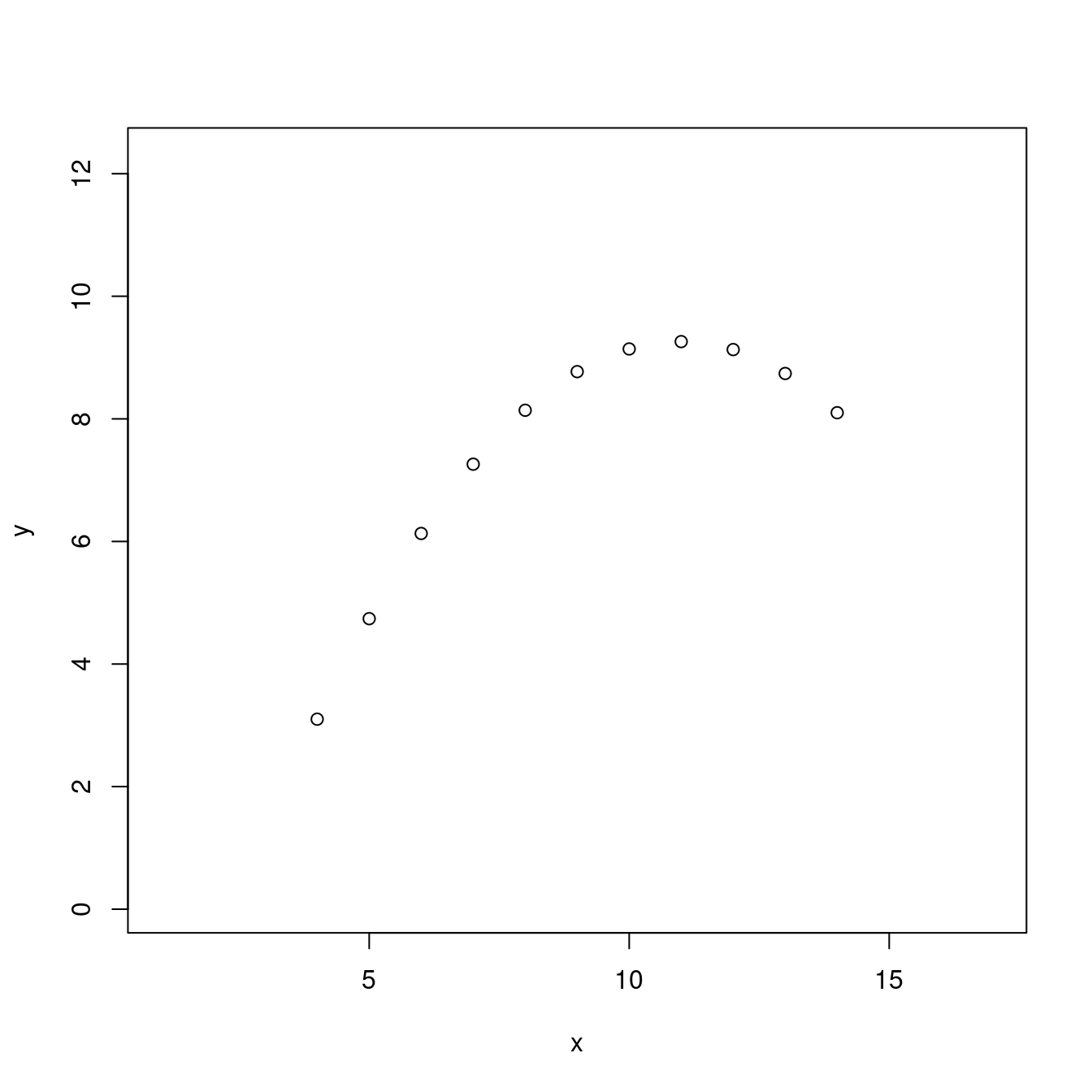
m2poly <- lm(y ~ poly(x, 2, raw = TRUE), data = df)
m2poly##
## Call:
## lm(formula = y ~ poly(x, 2, raw = TRUE), data = df)
##
## Coefficients:
## (Intercept) poly(x, 2, raw = TRUE)1 poly(x, 2, raw = TRUE)2
## -5.9957 2.7808 -0.1267summary(m2poly)##
## Call:
## lm(formula = y ~ poly(x, 2, raw = TRUE), data = df)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.0013287 -0.0011888 -0.0006294 0.0008741 0.0023776
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -5.9957343 0.0043299 -1385 <0.0000000000000002 ***
## poly(x, 2, raw = TRUE)1 2.7808392 0.0010401 2674 <0.0000000000000002 ***
## poly(x, 2, raw = TRUE)2 -0.1267133 0.0000571 -2219 <0.0000000000000002 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.001672 on 8 degrees of freedom
## Multiple R-squared: 1, Adjusted R-squared: 1
## F-statistic: 7.378e+06 on 2 and 8 DF, p-value: < 0.00000000000000022summary(m2poly)$coefficients## Estimate Std. Error t value
## (Intercept) -5.9957343 0.00432994585 -1384.713
## poly(x, 2, raw = TRUE)1 2.7808392 0.00104005552 2673.741
## poly(x, 2, raw = TRUE)2 -0.1267133 0.00005709766 -2219.238
## Pr(>|t|)
## (Intercept) 0.0000000000000000000000828575731
## poly(x, 2, raw = TRUE)1 0.0000000000000000000000004288066
## poly(x, 2, raw = TRUE)2 0.0000000000000000000000019036403fx <- function(x){
A <- summary(m2poly)$coefficients[1]
B <- summary(m2poly)$coefficients[2]
C <- summary(m2poly)$coefficients[3]
return(A + B * x + C * x^2)
}
curve(fx, from = min(df$x), to = max(df$x))
A <- summary(m2poly)$coefficients[1]
B <- summary(m2poly)$coefficients[2]
C <- summary(m2poly)$coefficients[3]
fx <- A + B * df$x + C * df$x^2
points(df$x, df$y)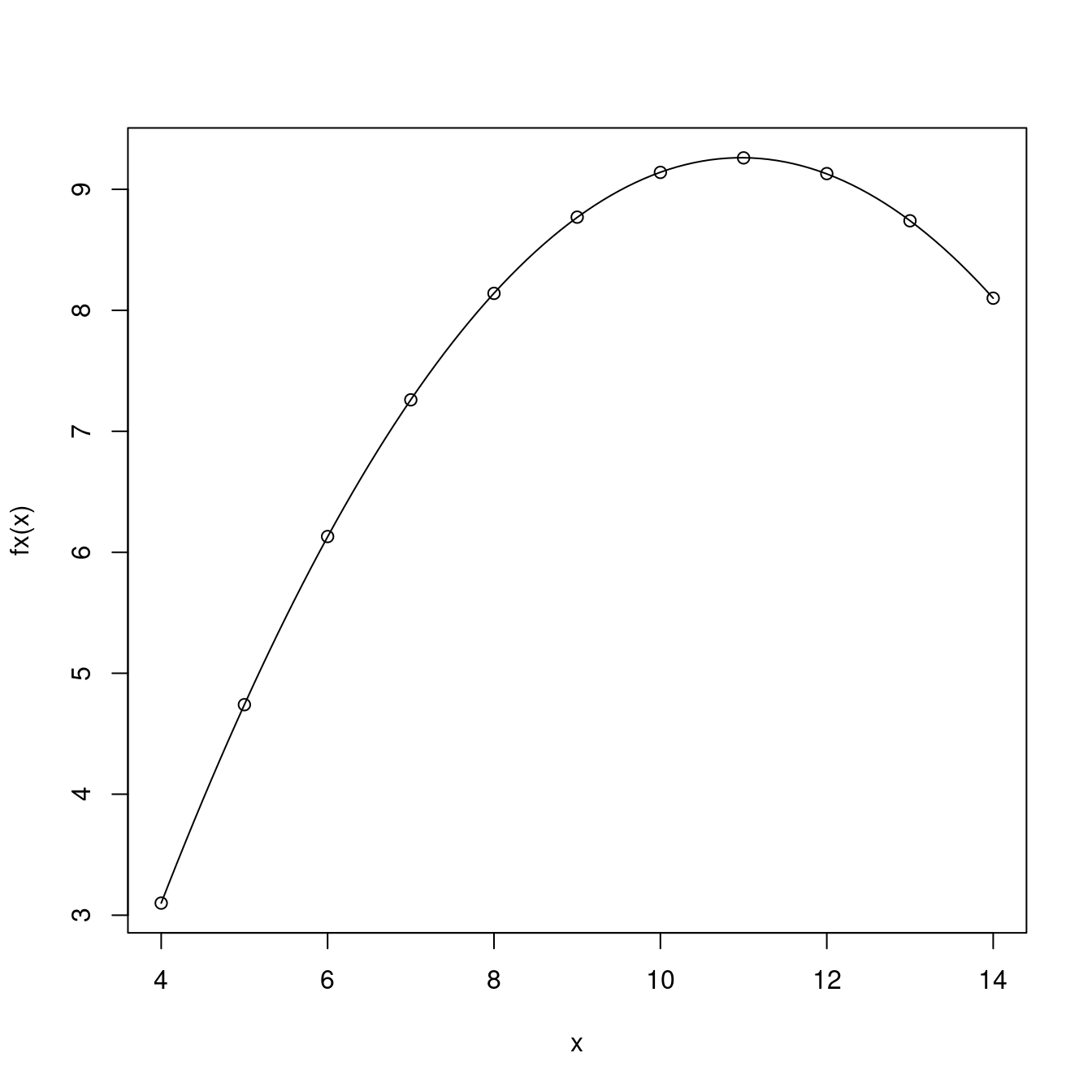
Q2: 找到曲線方程式的斜率方程式的估計?
fx <- expression(A + B * x + C * x^2)
D(fx,'x')## B + C * (2 * x)A <- summary(m2poly)$coefficients[1]
B <- summary(m2poly)$coefficients[2]
C <- summary(m2poly)$coefficients[3]
x <- df$x
eval(D(fx,'x'))## [1] 0.246573427 0.753426573 -0.513706294 0.500000000 -0.006853147
## [6] -0.767132867 1.260279720 1.767132867 -0.260279720 1.006853147
## [11] 1.513706294df$dy <- eval(D(fx,'x'))
m2D <- lm(dy ~ x, data = df)
m2D##
## Call:
## lm(formula = dy ~ x, data = df)
##
## Coefficients:
## (Intercept) x
## 2.7808 -0.2534summary(m2D)##
## Call:
## lm(formula = dy ~ x, data = df)
##
## Residuals:
## Min 1Q Median
## -0.00000000000000024150 -0.00000000000000013165 -0.00000000000000007581
## 3Q Max
## 0.00000000000000006650 0.00000000000000067134
##
## Coefficients:
## Estimate Std. Error t value
## (Intercept) 2.78083916083916182771 0.00000000000000024412 11391458854002518
## x -0.25342657342657343156 0.00000000000000002559 -9903217624018514
## Pr(>|t|)
## (Intercept) <0.0000000000000002 ***
## x <0.0000000000000002 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.0000000000000002684 on 9 degrees of freedom
## Multiple R-squared: 1, Adjusted R-squared: 1
## F-statistic: 9.807e+31 on 1 and 9 DF, p-value: < 0.00000000000000022plot(dy ~ x, data = df)
abline(m2D)
14.8.3 繼續挖Anscombe數據集之「y4」

m48 <- lm(y4[-8] ~ x4[-8], data = anscombe)
m48##
## Call:
## lm(formula = y4[-8] ~ x4[-8], data = anscombe)
##
## Coefficients:
## (Intercept) x4[-8]
## 7.001 NAsummary(m48)##
## Call:
## lm(formula = y4[-8] ~ x4[-8], data = anscombe)
##
## Residuals:
## Min 1Q Median 3Q Max
## -1.751 -1.036 -0.036 0.859 1.839
##
## Coefficients: (1 not defined because of singularities)
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 7.0010 0.3908 17.92 0.0000000239 ***
## x4[-8] NA NA NA NA
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 1.236 on 9 degrees of freedomQ1: 看到「
NA」,怎麼在散佈圖表達這項結果、結局?
14.9 當x變數是低社經人口比例時的「非線性迴歸線」
lm(medv ~ lstat + I(lstat^2), data = boston)##
## Call:
## lm(formula = medv ~ lstat + I(lstat^2), data = boston)
##
## Coefficients:
## (Intercept) lstat I(lstat^2)
## 42.86201 -2.33282 0.04355lm(medv ~ poly(lstat, 2, raw = TRUE), data = boston)##
## Call:
## lm(formula = medv ~ poly(lstat, 2, raw = TRUE), data = boston)
##
## Coefficients:
## (Intercept) poly(lstat, 2, raw = TRUE)1
## 42.86201 -2.33282
## poly(lstat, 2, raw = TRUE)2
## 0.04355require(dplyr)
lm(medv ~ poly(lstat, 2, raw = TRUE), data = boston) %>%
summary()##
## Call:
## lm(formula = medv ~ poly(lstat, 2, raw = TRUE), data = boston)
##
## Residuals:
## Min 1Q Median 3Q Max
## -15.2834 -3.8313 -0.5295 2.3095 25.4148
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 42.862007 0.872084 49.15 <0.0000000000000002
## poly(lstat, 2, raw = TRUE)1 -2.332821 0.123803 -18.84 <0.0000000000000002
## poly(lstat, 2, raw = TRUE)2 0.043547 0.003745 11.63 <0.0000000000000002
##
## (Intercept) ***
## poly(lstat, 2, raw = TRUE)1 ***
## poly(lstat, 2, raw = TRUE)2 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 5.524 on 503 degrees of freedom
## Multiple R-squared: 0.6407, Adjusted R-squared: 0.6393
## F-statistic: 448.5 on 2 and 503 DF, p-value: < 0.00000000000000022lm(medv ~ poly(lstat, 4, raw = TRUE), data = boston) %>%
summary()##
## Call:
## lm(formula = medv ~ poly(lstat, 4, raw = TRUE), data = boston)
##
## Residuals:
## Min 1Q Median 3Q Max
## -13.563 -3.180 -0.632 2.283 27.181
##
## Coefficients:
## Estimate Std. Error t value
## (Intercept) 57.30995522 2.28013871 25.134
## poly(lstat, 4, raw = TRUE)1 -7.02846009 0.73079130 -9.618
## poly(lstat, 4, raw = TRUE)2 0.49548114 0.07489243 6.616
## poly(lstat, 4, raw = TRUE)3 -0.01631017 0.00299359 -5.448
## poly(lstat, 4, raw = TRUE)4 0.00019487 0.00004043 4.820
## Pr(>|t|)
## (Intercept) < 0.0000000000000002 ***
## poly(lstat, 4, raw = TRUE)1 < 0.0000000000000002 ***
## poly(lstat, 4, raw = TRUE)2 0.000000000095 ***
## poly(lstat, 4, raw = TRUE)3 0.000000079843 ***
## poly(lstat, 4, raw = TRUE)4 0.000001903556 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 5.28 on 501 degrees of freedom
## Multiple R-squared: 0.673, Adjusted R-squared: 0.6704
## F-statistic: 257.8 on 4 and 501 DF, p-value: < 0.00000000000000022lm(medv ~ poly(lstat, 6, raw = TRUE), data = boston) %>%
summary()##
## Call:
## lm(formula = medv ~ poly(lstat, 6, raw = TRUE), data = boston)
##
## Residuals:
## Min 1Q Median 3Q Max
## -14.7317 -3.1571 -0.6941 2.0756 26.8994
##
## Coefficients:
## Estimate Std. Error t value
## (Intercept) 73.0433518598 5.5933680650 13.059
## poly(lstat, 6, raw = TRUE)1 -15.1673265415 2.9654385860 -5.115
## poly(lstat, 6, raw = TRUE)2 1.9295909151 0.5712522965 3.378
## poly(lstat, 6, raw = TRUE)3 -0.1307066978 0.0520184925 -2.513
## poly(lstat, 6, raw = TRUE)4 0.0046860540 0.0024065100 1.947
## poly(lstat, 6, raw = TRUE)5 -0.0000841615 0.0000545034 -1.544
## poly(lstat, 6, raw = TRUE)6 0.0000005974 0.0000004783 1.249
## Pr(>|t|)
## (Intercept) < 0.0000000000000002 ***
## poly(lstat, 6, raw = TRUE)1 0.000000449 ***
## poly(lstat, 6, raw = TRUE)2 0.000788 ***
## poly(lstat, 6, raw = TRUE)3 0.012295 *
## poly(lstat, 6, raw = TRUE)4 0.052066 .
## poly(lstat, 6, raw = TRUE)5 0.123186
## poly(lstat, 6, raw = TRUE)6 0.212313
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 5.212 on 499 degrees of freedom
## Multiple R-squared: 0.6827, Adjusted R-squared: 0.6789
## F-statistic: 178.9 on 6 and 499 DF, p-value: < 0.00000000000000022lm(medv ~ poly(lstat, 8, raw = TRUE), data = boston) %>%
summary()##
## Call:
## lm(formula = medv ~ poly(lstat, 8, raw = TRUE), data = boston)
##
## Residuals:
## Min 1Q Median 3Q Max
## -13.7394 -3.1475 -0.7329 2.0959 26.9915
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 58.396095726800 12.726912504630 4.588 0.00000566
## poly(lstat, 8, raw = TRUE)1 -3.035835901367 9.738246653136 -0.312 0.755
## poly(lstat, 8, raw = TRUE)2 -1.827904328094 2.885823586645 -0.633 0.527
## poly(lstat, 8, raw = TRUE)3 0.450872604706 0.436986208173 1.032 0.303
## poly(lstat, 8, raw = TRUE)4 -0.045590109122 0.037496626652 -1.216 0.225
## poly(lstat, 8, raw = TRUE)5 0.002447418535 0.001891937030 1.294 0.196
## poly(lstat, 8, raw = TRUE)6 -0.000072996235 0.000055456649 -1.316 0.189
## poly(lstat, 8, raw = TRUE)7 0.000001142823 0.000000871935 1.311 0.191
## poly(lstat, 8, raw = TRUE)8 -0.000000007328 0.000000005677 -1.291 0.197
##
## (Intercept) ***
## poly(lstat, 8, raw = TRUE)1
## poly(lstat, 8, raw = TRUE)2
## poly(lstat, 8, raw = TRUE)3
## poly(lstat, 8, raw = TRUE)4
## poly(lstat, 8, raw = TRUE)5
## poly(lstat, 8, raw = TRUE)6
## poly(lstat, 8, raw = TRUE)7
## poly(lstat, 8, raw = TRUE)8
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 5.213 on 497 degrees of freedom
## Multiple R-squared: 0.6838, Adjusted R-squared: 0.6787
## F-statistic: 134.4 on 8 and 497 DF, p-value: < 0.00000000000000022lm(medv ~ poly(lstat, 10, raw = TRUE), data = boston) %>%
summary()##
## Call:
## lm(formula = medv ~ poly(lstat, 10, raw = TRUE), data = boston)
##
## Residuals:
## Min 1Q Median 3Q Max
## -14.5340 -3.0286 -0.7507 2.0437 26.4738
##
## Coefficients:
## Estimate Std. Error t value
## (Intercept) 8.71199299049154 26.94421824392828 0.323
## poly(lstat, 10, raw = TRUE)1 50.03781139746272 27.56704578794819 1.815
## poly(lstat, 10, raw = TRUE)2 -23.91309180613620 11.34375335807400 -2.108
## poly(lstat, 10, raw = TRUE)3 5.25470623512324 2.49284220347617 2.108
## poly(lstat, 10, raw = TRUE)4 -0.66142517222178 0.32771148561820 -2.018
## poly(lstat, 10, raw = TRUE)5 0.05183790363705 0.02723590094398 1.903
## poly(lstat, 10, raw = TRUE)6 -0.00261613337024 0.00146400463053 -1.787
## poly(lstat, 10, raw = TRUE)7 0.00008507278434 0.00005069745905 1.678
## poly(lstat, 10, raw = TRUE)8 -0.00000172188563 0.00000109044367 -1.579
## poly(lstat, 10, raw = TRUE)9 0.00000001972541 0.00000001323667 1.490
## poly(lstat, 10, raw = TRUE)10 -0.00000000009766 0.00000000006922 -1.411
## Pr(>|t|)
## (Intercept) 0.7466
## poly(lstat, 10, raw = TRUE)1 0.0701 .
## poly(lstat, 10, raw = TRUE)2 0.0355 *
## poly(lstat, 10, raw = TRUE)3 0.0355 *
## poly(lstat, 10, raw = TRUE)4 0.0441 *
## poly(lstat, 10, raw = TRUE)5 0.0576 .
## poly(lstat, 10, raw = TRUE)6 0.0746 .
## poly(lstat, 10, raw = TRUE)7 0.0940 .
## poly(lstat, 10, raw = TRUE)8 0.1150
## poly(lstat, 10, raw = TRUE)9 0.1368
## poly(lstat, 10, raw = TRUE)10 0.1589
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 5.199 on 495 degrees of freedom
## Multiple R-squared: 0.6867, Adjusted R-squared: 0.6804
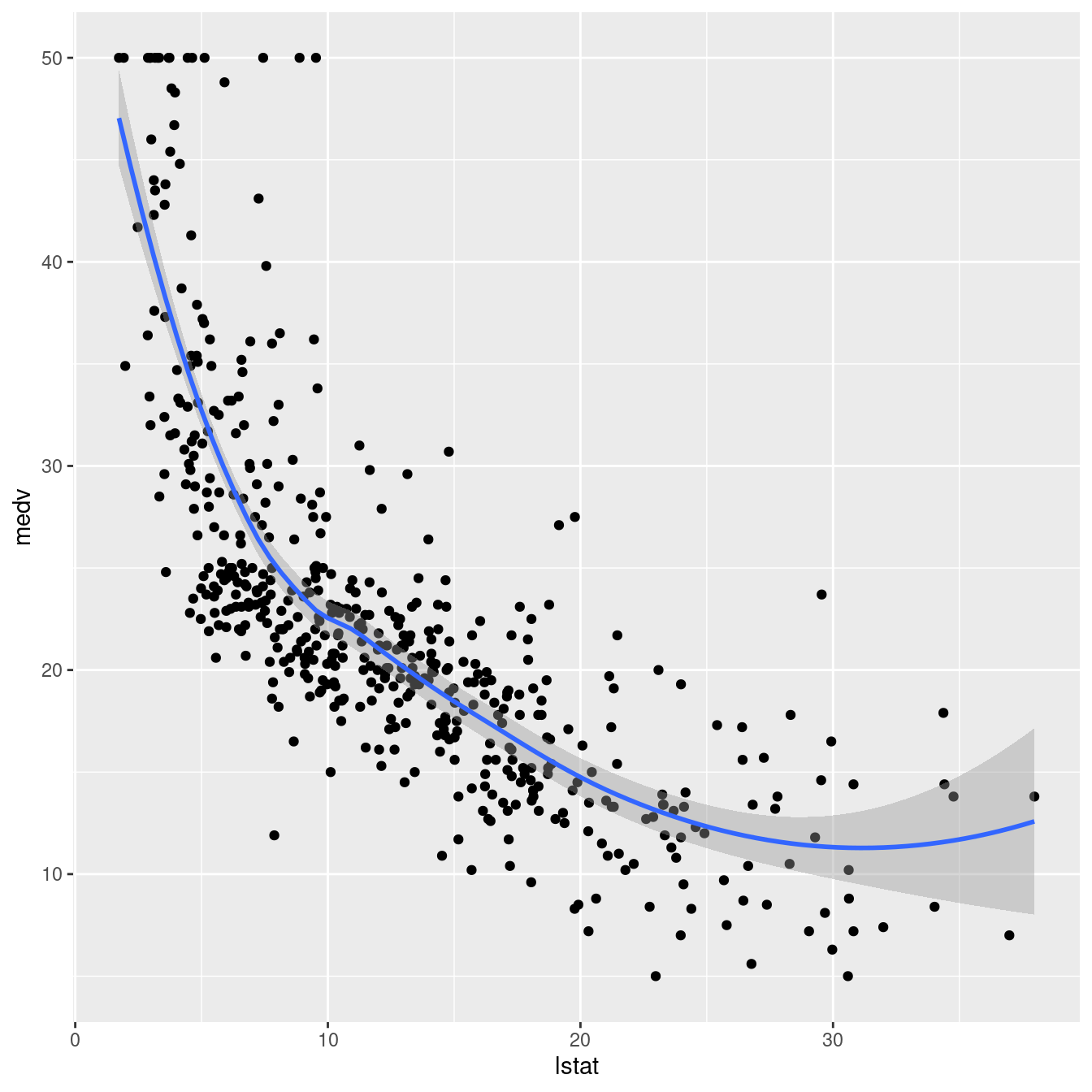
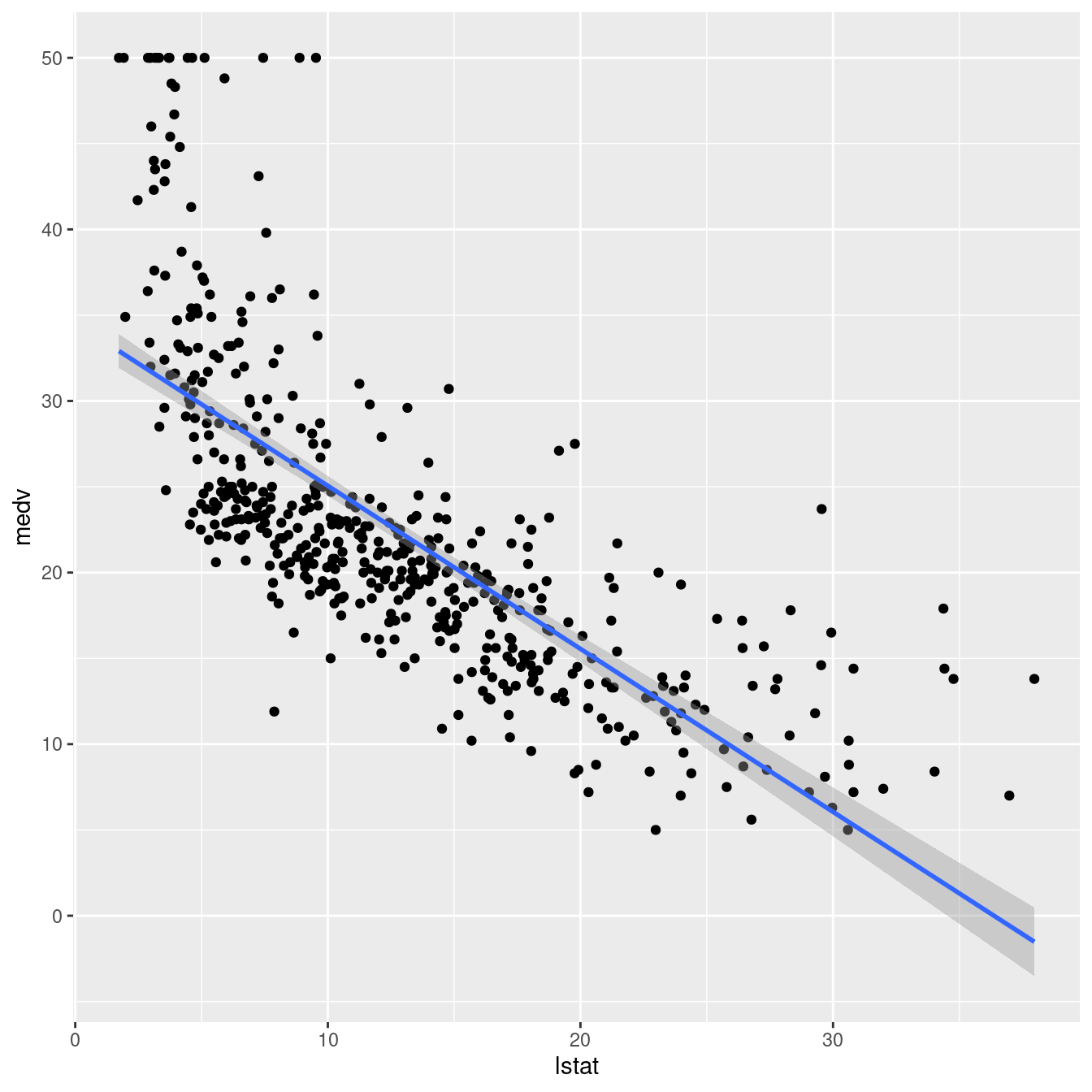
## F-statistic: 108.5 on 10 and 495 DF, p-value: < 0.00000000000000022ggplot(boston, aes(lstat, medv) ) +
geom_point() +
stat_smooth(method = lm, formula = y ~ poly(x, 5, raw = TRUE))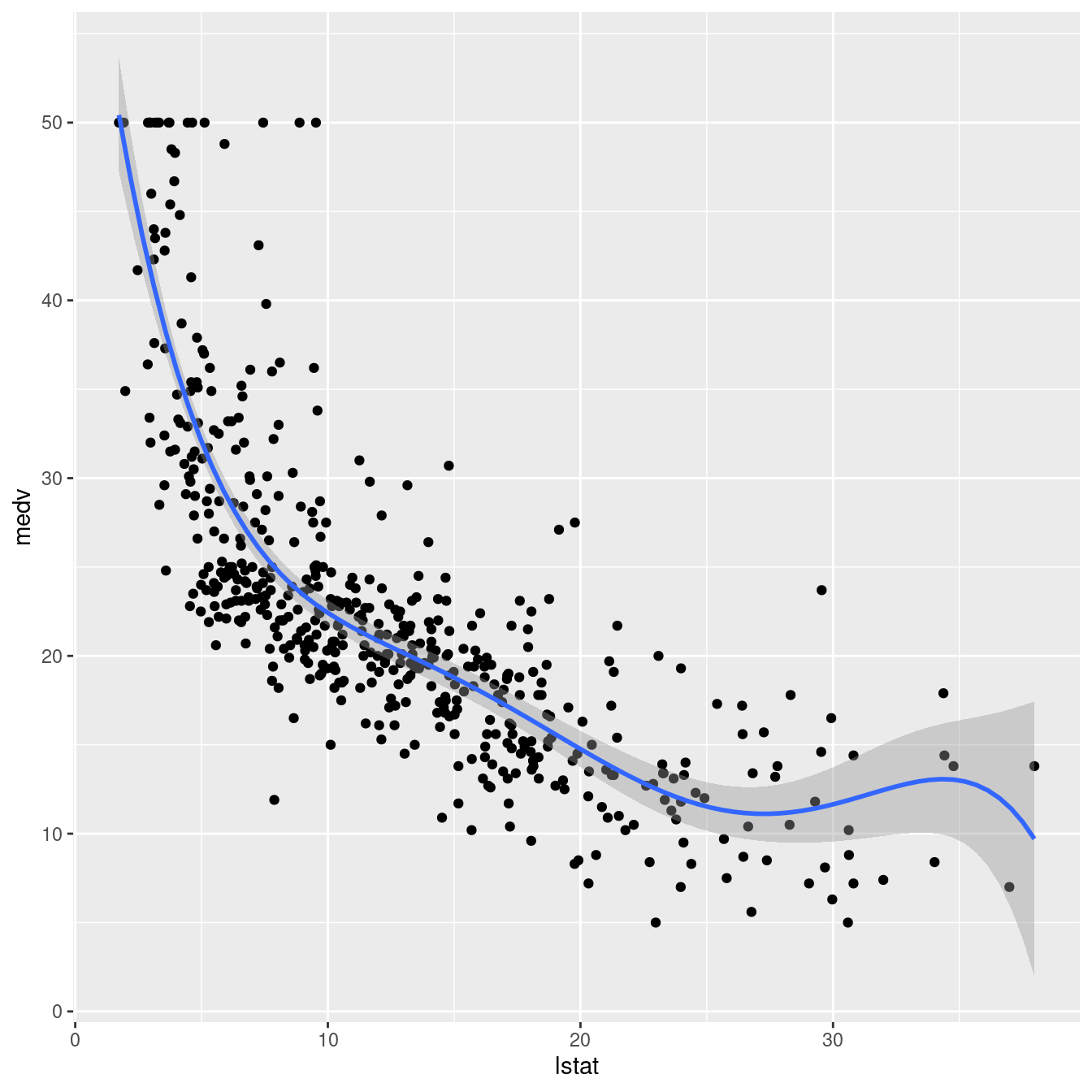
ggplot(boston, aes(lstat, medv) ) +
geom_point() +
stat_smooth(method = lm, formula = y ~ log(x))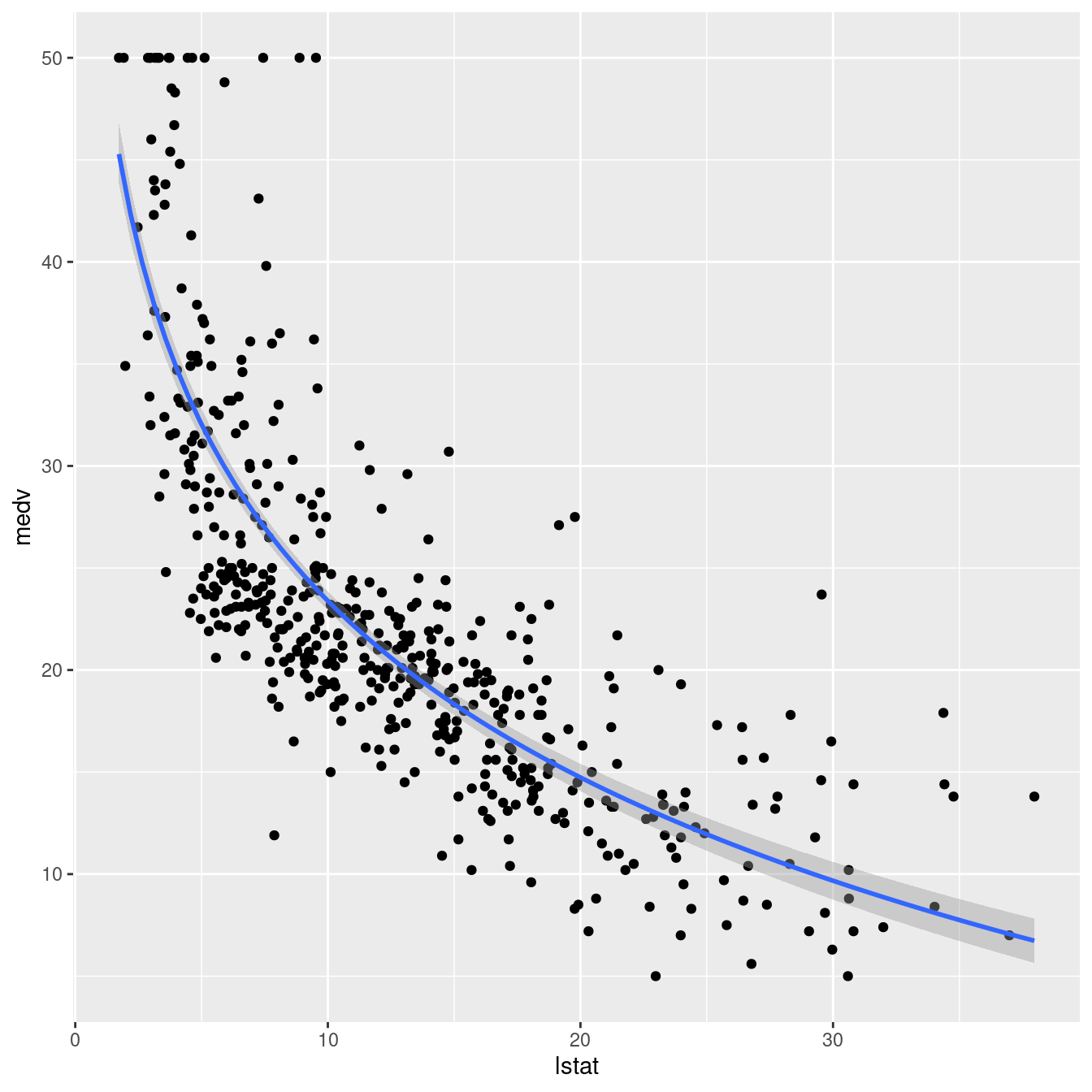
lm(medv ~ log(lstat), data = boston) %>%
summary()##
## Call:
## lm(formula = medv ~ log(lstat), data = boston)
##
## Residuals:
## Min 1Q Median 3Q Max
## -14.4599 -3.5006 -0.6686 2.1688 26.0129
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 52.1248 0.9652 54.00 <0.0000000000000002 ***
## log(lstat) -12.4810 0.3946 -31.63 <0.0000000000000002 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 5.329 on 504 degrees of freedom
## Multiple R-squared: 0.6649, Adjusted R-squared: 0.6643
## F-statistic: 1000 on 1 and 504 DF, p-value: < 0.00000000000000022require(mgcv)
ggplot(boston, aes(lstat, medv) ) +
geom_point() +
stat_smooth(method = gam, formula = y ~ s(x))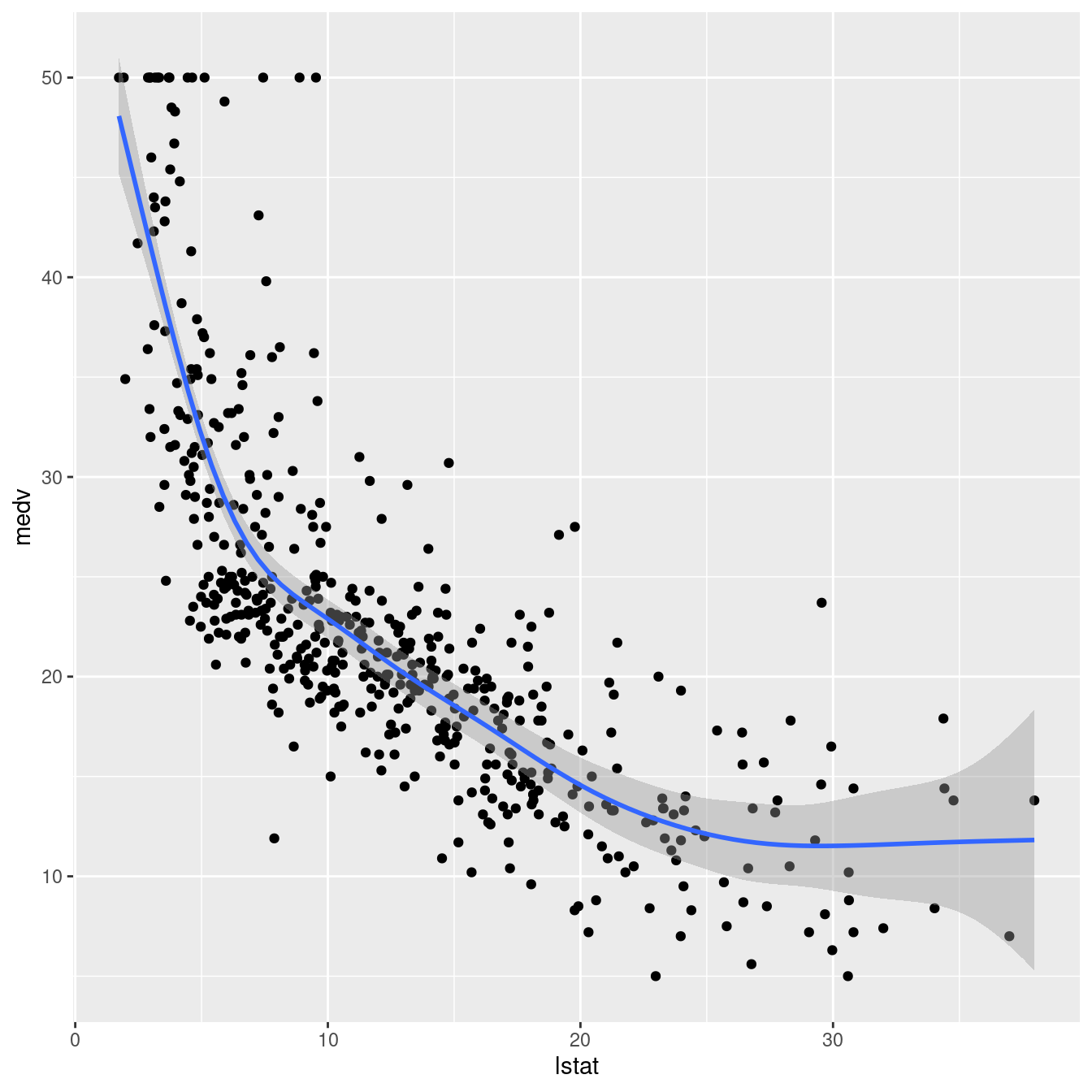
gam(medv ~ s(lstat), data = boston) %>%
summary()##
## Family: gaussian
## Link function: identity
##
## Formula:
## medv ~ s(lstat)
##
## Parametric coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 22.5328 0.2312 97.47 <0.0000000000000002 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Approximate significance of smooth terms:
## edf Ref.df F p-value
## s(lstat) 7.403 8.375 128.4 <0.0000000000000002 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## R-sq.(adj) = 0.68 Deviance explained = 68.5%
## GCV = 27.5 Scale est. = 27.044 n = 506