Capítulo 10 Importación y consolidación de datos
10.1 Importación desde archivos
Para la importación de archivos, ya sean de tipo texto u hojas de cálculo, es necesario que sepamos de dónde vamos a importar los datos.
10.1.1 Directorio de Trabajo
Por default cuando importemos archivos R va a buscar en el directorio de trabajo (Working Directory en inglés). Para conocer cuál es la ruta de nuestro directorio de trabajo utilizaremos la función getwd().
Esta es la ruta de donde podemos colocar nuestros archivos para cargarlos. Si queremos cargar datos de otra carpeta podemos cambiar el directorio de trabajo utilizando setwd().
setwd("c:/Documents/Proyectos/Archivos para R")
getwd()
#> [1] "c:/Documents/Proyectos/Archivos para R"Por temas prácticos vamos a utilizar un archivo ya disponible en uno de los paquetes antes instalados, dslabs, cuando analizamos el nivel de peligrosidad para decidir a qué estado de EEUU viajar. Para ello, podemos usar la función system.file() y determinar cuál es la ruta donde se instaló el paquete dslabs.
Así mismo, podemos listar los archivos y carpetas dentro de esa ruta utilizando la función list.files().
ruta_dslabs <- system.file(package="dslabs")
list.files(ruta_dslabs)
#> [1] "data" "DESCRIPTION" "extdata" "help" "html"
#> [6] "INDEX" "Meta" "NAMESPACE" "R" "script"La carpeta que utilizaremos es extdata. Podemos acceder a la ruta de esta carpeta si modificamos los parámetros de la función system.file().
ruta_dslabs <- system.file("extdata", package="dslabs")
list.files(ruta_dslabs)
#> [1] "2010_bigfive_regents.xls"
#> [2] "calificaciones.csv"
#> [3] "carbon_emissions.csv"
#> [4] "fertility-two-countries-example.csv"
#> [5] "HRlist2.txt"
#> [6] "life-expectancy-and-fertility-two-countries-example.csv"
#> [7] "murders.csv"
#> [8] "olive.csv"
#> [9] "RD-Mortality-Report_2015-18-180531.pdf"
#> [10] "ssa-death-probability.csv"El archivo que utilizaremos es murders.csv. Para construir la ruta completa de este archivo podemos concatenar las cadenas o también podemos utilizar directamente la función file.path(ruta, nombre_archivo).
Finalmente, copiaremos el archivo a nuestro directorio de trabajo con la función file.copy(ruta_origen, ruta_destino).
Podemos validar la copia con la función file.exists(nombre_archivo).
Se recomienda revisar la documentación de las funciones de manipulación de archivos.
10.1.2 Paquetes readr y readxl
Ahora que ya tenemos el archivo en nuestro directorio de trabajo usaremos funciones dentro de los paquetes readr y readxl para importar archivos a R. Ambos están incluidos en el paquete tidyverse que anteriormente instalamos.
Las funciones que más utilizaremos serán read_csv() y read_excel(). Ésta última soporta extensiones .xls y xlsx.
datos <- read_csv("murders.csv")
#> Rows: 51 Columns: 5
#> ── Column specification ────────────────────────────────────────────────────────
#> Delimiter: ","
#> chr (3): state, abb, region
#> dbl (2): population, total
#>
#> ℹ Use `spec()` to retrieve the full column specification for this data.
#> ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.
# Una vez importado pordemos remover el archivo si deseamos
file.remove("murders.csv")
#> [1] TRUEVemos cómo por default detecta las cabeceras en la primera fila y les asigna un tipo de dato por default. Exploremos ahora nuestro objeto datos.
datos
#> # A tibble: 51 × 5
#> state abb region population total
#> <chr> <chr> <chr> <dbl> <dbl>
#> 1 Alabama AL South 4779736 135
#> 2 Alaska AK West 710231 19
#> 3 Arizona AZ West 6392017 232
#> 4 Arkansas AR South 2915918 93
#> 5 California CA West 37253956 1257
#> 6 Colorado CO West 5029196 65
#> 7 Connecticut CT Northeast 3574097 97
#> 8 Delaware DE South 897934 38
#> 9 District of Columbia DC South 601723 99
#> 10 Florida FL South 19687653 669
#> # ℹ 41 more rowsLo primero que nos indica es que el objeto es de tipo tibble. Este objeto es muy parecido a un data frame, pero con características mejoradas como, por ejemplo, la cantidad de filas y columnas en consola, el tipo de dato debajo de la cabecera, el reporte por default solo de los 10 primeros registros en automático, entre muchas otras que iremos descubriendo en este capítulo.
La misma sintáxis y lógica sería para importar un archivo excel. En est caso estamos importando directamente de la ruta del paquete y no de nuestro directorio de trabajo.
ruta_ejempo_excel <- file.path(ruta_dslabs, "2010_bigfive_regents.xls")
datos_de_excel <- read_excel(ruta_ejempo_excel)readr nos brinda 7 diferentes tipos de funciones para importar archivos planos:
| Funcion | Uso |
|---|---|
read_csv() |
Archivos separados por comas |
read_tsv() |
Archivos separados por tabulaciones |
read_delim() |
Archivos delimitados en general |
read_fwf() |
Archivos de ancho fijo |
read_table() |
Archivos tabulares donde las columnas están separadas por espacios en blanco |
read_log() |
Archivos de registros web |
10.1.3 Importar archivos desde internet
Hemos visto cómo podemos ingresar la ruta completa para cargar un archivo directamente desde otra fuente diferente a nuestro directorio de trabajo. De la misma forma, si tenemos un archivo en una ruta de internet podemos pasarlo directamente a R dado que read_csv() y las demás funciones de importación de readr soportan el ingreso de url como parámetro.
Acá vemos la importación de notas de estudiantes del curso Data Science con R.
##### Ejemplo 1:
# Data de histórico de notas
url <- "https://dparedesi.github.io/DS-con-R/notas-estudiantes.csv"
notas <- read_csv(url)notas <- notas %>%
mutate(total = (P1 + P2 + P3 + P4 + P5 + P6)/30*20)
notas %>%
select(P1, P2, P3, P4, P5, total) %>%
summary()
#> P1 P2 P3 P4 P5
#> Min. :1.000 Min. :1.00 Min. :4.000 Min. :1.000 Min. :1.000
#> 1st Qu.:3.000 1st Qu.:4.00 1st Qu.:5.000 1st Qu.:5.000 1st Qu.:5.000
#> Median :4.000 Median :5.00 Median :5.000 Median :5.000 Median :5.000
#> Mean :3.762 Mean :4.19 Mean :4.905 Mean :4.571 Mean :4.429
#> 3rd Qu.:5.000 3rd Qu.:5.00 3rd Qu.:5.000 3rd Qu.:5.000 3rd Qu.:5.000
#> Max. :5.000 Max. :5.00 Max. :5.000 Max. :5.000 Max. :5.000
#> total
#> Min. :10.67
#> 1st Qu.:16.67
#> Median :18.67
#> Mean :17.43
#> 3rd Qu.:19.33
#> Max. :20.00A partir de esto podríamos visualizar un histograma:
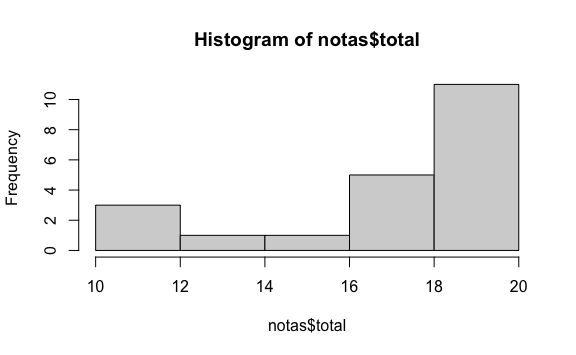
O podríamos comparar entre género cuál es el que tiene la mediana más alta:
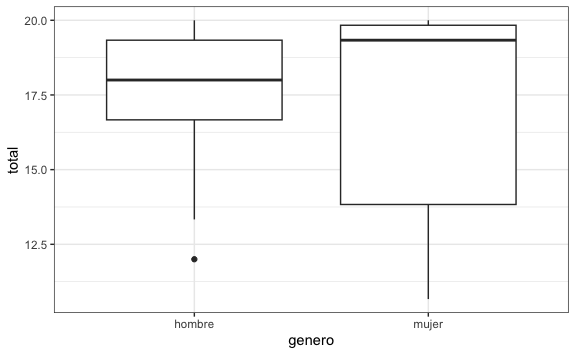
También podríamos extraer información del Covid-19 actualizada.
##### Ejemplo 2:
# Data de Covid-19
url <- "https://covid.ourworldindata.org/data/owid-covid-data.csv"
datos_de_internet <- read_csv(url)
#> Rows: 429435 Columns: 67
#> ── Column specification ────────────────────────────────────────────────────────
#> Delimiter: ","
#> chr (4): iso_code, continent, location, tests_units
#> dbl (62): total_cases, new_cases, new_cases_smoothed, total_deaths, new_dea...
#> date (1): date
#>
#> ℹ Use `spec()` to retrieve the full column specification for this data.
#> ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.
datos_de_internet %>%
arrange(desc(date)) %>%
head(10)
#> # A tibble: 10 × 67
#> iso_code continent location date total_cases new_cases
#> <chr> <chr> <chr> <date> <dbl> <dbl>
#> 1 OWID_ASI <NA> Asia 2024-08-14 NA NA
#> 2 OWID_EUR <NA> Europe 2024-08-14 NA NA
#> 3 OWID_EUN <NA> European Union (27) 2024-08-14 NA NA
#> 4 OWID_HIC <NA> High-income countries 2024-08-14 NA NA
#> 5 LTU Europe Lithuania 2024-08-14 NA NA
#> 6 MYS Asia Malaysia 2024-08-14 NA NA
#> 7 OWID_UMC <NA> Upper-middle-income coun… 2024-08-14 NA NA
#> 8 OWID_WRL <NA> World 2024-08-14 NA NA
#> 9 OWID_ASI <NA> Asia 2024-08-13 NA NA
#> 10 OWID_EUR <NA> Europe 2024-08-13 NA NA
#> # ℹ 61 more variables: new_cases_smoothed <dbl>, total_deaths <dbl>,
#> # new_deaths <dbl>, new_deaths_smoothed <dbl>, total_cases_per_million <dbl>,
#> # new_cases_per_million <dbl>, new_cases_smoothed_per_million <dbl>,
#> # total_deaths_per_million <dbl>, new_deaths_per_million <dbl>,
#> # new_deaths_smoothed_per_million <dbl>, reproduction_rate <dbl>,
#> # icu_patients <dbl>, icu_patients_per_million <dbl>, hosp_patients <dbl>,
#> # hosp_patients_per_million <dbl>, weekly_icu_admissions <dbl>, …10.2 Tidy data
Los datos ordenados o tidy data son los que se obtienen a partir de un proceso llamado data tidying u ordenamiento de datos. Es uno de los procesos de limpieza importantes durante procesamiento de grandes datos o ‘big data’ y es un paso muy utilizado en Data Science. Las principales características son que cada observación diferente de esa variable tiene que ser en una fila diferente y que cada variable que mides tiene que ser en una columna (Leek 2015).
Como nos habremos podido dar cuenta, hemos venido utilizando tidy data desde los primeros capítulos. Sin embargo, no toda nuestra data viene ordenada. La mayoría de ella viene en lo que llamamos datos amplios o wide data.
Por ejemplo, hemos utilizado anteriormente data de Gapminder. Filtremos los datos de Alemania y Corea del Sur para recordar cómo teníamos nuestros datos.
gapminder %>%
filter(country %in% c("South Korea", "Germany")) %>%
head(10)
#> country year infant_mortality life_expectancy fertility population
#> 1 Germany 1960 34.0 69.26 2.41 73179665
#> 2 South Korea 1960 80.2 53.02 6.16 25074028
#> 3 Germany 1961 NA 69.85 2.44 73686490
#> 4 South Korea 1961 76.1 53.75 5.99 25808542
#> 5 Germany 1962 NA 70.01 2.47 74238494
#> 6 South Korea 1962 72.4 54.51 5.79 26495107
#> 7 Germany 1963 NA 70.10 2.49 74820389
#> 8 South Korea 1963 68.8 55.27 5.57 27143075
#> 9 Germany 1964 NA 70.66 2.49 75410766
#> 10 South Korea 1964 65.3 56.04 5.36 27770874
#> gdp continent region
#> 1 NA Europe Western Europe
#> 2 28928298962 Asia Eastern Asia
#> 3 NA Europe Western Europe
#> 4 30356298714 Asia Eastern Asia
#> 5 NA Europe Western Europe
#> 6 31102566019 Asia Eastern Asia
#> 7 NA Europe Western Europe
#> 8 34067175844 Asia Eastern Asia
#> 9 NA Europe Western Europe
#> 10 36643076469 Asia Eastern AsiaNotamos que en cada fila es una observación y cada columna representa una variable. Es data ordenada, tidy data. Por otro lado, podemos ver cómo era la data para estos dos países si accedemos a la fuente en el paquete dslabs.
ruta_fertilidad <- file.path(ruta_dslabs, "fertility-two-countries-example.csv")
wide_data <- read_csv(ruta_fertilidad)
#> Rows: 2 Columns: 57
#> ── Column specification ────────────────────────────────────────────────────────
#> Delimiter: ","
#> chr (1): country
#> dbl (56): 1960, 1961, 1962, 1963, 1964, 1965, 1966, 1967, 1968, 1969, 1970, ...
#>
#> ℹ Use `spec()` to retrieve the full column specification for this data.
#> ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.
wide_data
#> # A tibble: 2 × 57
#> country `1960` `1961` `1962` `1963` `1964` `1965` `1966` `1967` `1968` `1969`
#> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Germany 2.41 2.44 2.47 2.49 2.49 2.48 2.44 2.37 2.28 2.17
#> 2 South K… 6.16 5.99 5.79 5.57 5.36 5.16 4.99 4.85 4.73 4.62
#> # ℹ 46 more variables: `1970` <dbl>, `1971` <dbl>, `1972` <dbl>, `1973` <dbl>,
#> # `1974` <dbl>, `1975` <dbl>, `1976` <dbl>, `1977` <dbl>, `1978` <dbl>,
#> # `1979` <dbl>, `1980` <dbl>, `1981` <dbl>, `1982` <dbl>, `1983` <dbl>,
#> # `1984` <dbl>, `1985` <dbl>, `1986` <dbl>, `1987` <dbl>, `1988` <dbl>,
#> # `1989` <dbl>, `1990` <dbl>, `1991` <dbl>, `1992` <dbl>, `1993` <dbl>,
#> # `1994` <dbl>, `1995` <dbl>, `1996` <dbl>, `1997` <dbl>, `1998` <dbl>,
#> # `1999` <dbl>, `2000` <dbl>, `2001` <dbl>, `2002` <dbl>, `2003` <dbl>, …Vemos que la data original tenía dos filas, una por país y luego cada columna representaba un año. Esto es lo que llamamos data amplia o wide data. Normalmente tendremos wide data que primero tenemos que convertir a tidy data para poder posteriormente realizar nuestros análisis.
10.2.1 Transformando a tidy data
La librería tidyverse nos facilita dos funciones para poder pasar de wide data a tidy data y viceversa. Para ello usaremos la función gather() para convertir de wide data a tidy data y la función spread() para convertir de tidy data a wide data.
10.2.1.1 Función gather
Veamos la utilidad con el objeto wide_data que hemos creado en la sección anterior como resultado de importar datos del csv. Primero apliquemos la función gather() para explorar la conversión que se realiza por default.
tidy_data <- wide_data %>%
gather()
tidy_data
#> # A tibble: 114 × 2
#> key value
#> <chr> <chr>
#> 1 country Germany
#> 2 country South Korea
#> 3 1960 2.41
#> 4 1960 6.16
#> 5 1961 2.44
#> 6 1961 5.99
#> 7 1962 2.47
#> 8 1962 5.79
#> 9 1963 2.49
#> 10 1963 5.57
#> # ℹ 104 more rowsVemos cómo la función gather() ha recopilado las columnas en dos, la clave (key en inglés) y el valor (value en inglés). Podemos cambiar el título de estas nuevas columnas, por ejemplo “año” y “fertilidad”.
tidy_data <- wide_data %>%
gather(año, fertilidad)
tidy_data
#> # A tibble: 114 × 2
#> año fertilidad
#> <chr> <chr>
#> 1 country Germany
#> 2 country South Korea
#> 3 1960 2.41
#> 4 1960 6.16
#> 5 1961 2.44
#> 6 1961 5.99
#> 7 1962 2.47
#> 8 1962 5.79
#> 9 1963 2.49
#> 10 1963 5.57
#> # ℹ 104 more rowsSin embargo, hemos visto cómo nos ha recopilado todas las columnas incluyendo la columna inicial del país. Para excluir alguna columna utlizaremos el signo -.
tidy_data <- wide_data %>%
gather(año, fertilidad, -country)
tidy_data
#> # A tibble: 112 × 3
#> country año fertilidad
#> <chr> <chr> <dbl>
#> 1 Germany 1960 2.41
#> 2 South Korea 1960 6.16
#> 3 Germany 1961 2.44
#> 4 South Korea 1961 5.99
#> 5 Germany 1962 2.47
#> 6 South Korea 1962 5.79
#> 7 Germany 1963 2.49
#> 8 South Korea 1963 5.57
#> 9 Germany 1964 2.49
#> 10 South Korea 1964 5.36
#> # ℹ 102 more rowsAhora ya tenemos la data ordenada. Por default los datos de las columnas del wide data, en este caso año, son recopilados como texto. Para prevenir ello utilizaremos el atributo convert=TRUE.
tidy_data <- wide_data %>%
gather(año, fertilidad, -country, convert=TRUE)
tidy_data
#> # A tibble: 112 × 3
#> country año fertilidad
#> <chr> <int> <dbl>
#> 1 Germany 1960 2.41
#> 2 South Korea 1960 6.16
#> 3 Germany 1961 2.44
#> 4 South Korea 1961 5.99
#> 5 Germany 1962 2.47
#> 6 South Korea 1962 5.79
#> 7 Germany 1963 2.49
#> 8 South Korea 1963 5.57
#> 9 Germany 1964 2.49
#> 10 South Korea 1964 5.36
#> # ℹ 102 more rowsEsta data ya estaría lista para crear gráficos usando ggplot().
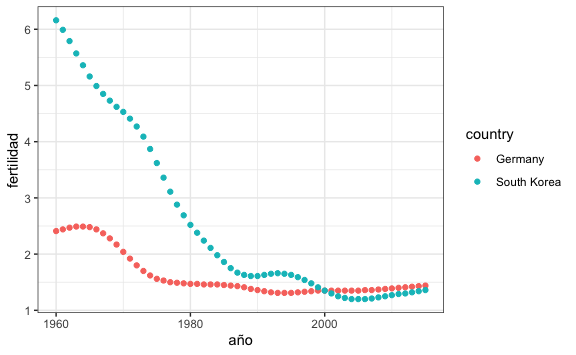
10.2.1.2 Función spread
Algunas veces, como veremos en la sección siguiente, nos será de utilidad regresar de filas a columnas. Para ello utilizaremos la función spread(), en donde ingresaremos como argumentos el nombre de la columna que queremos separar (key) y el valor que colocaremos en cada campo. Además, podemos utilizar el operador : para indicar desde qué columna hasta qué columna queremos seleccionar.
10.2.2 Función separate
En los casos descritos anteriormente teníamos un situación con data relativamente ordenada. Solo tuvimos que hacer una transformación de recopilación y convertimos a tidy data. Sin embargo, la data no siempre es almacenada de una forma tan fácil de interpretar. Algunas veces tenemos data como esta:
ruta <- file.path(ruta_dslabs, "life-expectancy-and-fertility-two-countries-example.csv")
data <- read_csv(ruta)
#> Rows: 2 Columns: 113
#> ── Column specification ────────────────────────────────────────────────────────
#> Delimiter: ","
#> chr (1): country
#> dbl (112): 1960_fertility, 1960_life_expectancy, 1961_fertility, 1961_life_e...
#>
#> ℹ Use `spec()` to retrieve the full column specification for this data.
#> ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.
data %>%
select(1:5) #Reportamos las primeras 5 columnas
#> # A tibble: 2 × 5
#> country `1960_fertility` `1960_life_expectancy` `1961_fertility`
#> <chr> <dbl> <dbl> <dbl>
#> 1 Germany 2.41 69.3 2.44
#> 2 South Korea 6.16 53.0 5.99
#> # ℹ 1 more variable: `1961_life_expectancy` <dbl>Si aplicamos diretamente gather() no tendríamos aun nuestra data ordenada. Veamos:
data %>%
gather(clave, valor, -country)
#> # A tibble: 224 × 3
#> country clave valor
#> <chr> <chr> <dbl>
#> 1 Germany 1960_fertility 2.41
#> 2 South Korea 1960_fertility 6.16
#> 3 Germany 1960_life_expectancy 69.3
#> 4 South Korea 1960_life_expectancy 53.0
#> 5 Germany 1961_fertility 2.44
#> 6 South Korea 1961_fertility 5.99
#> 7 Germany 1961_life_expectancy 69.8
#> 8 South Korea 1961_life_expectancy 53.8
#> 9 Germany 1962_fertility 2.47
#> 10 South Korea 1962_fertility 5.79
#> # ℹ 214 more rowsUtilizaremos la función separate() para separar una columna en múltiples columnas utilizando un separador determinado. En este caso nuestro separados sería el caracter _. Así mismo, agregaremos el atributo extra="merge" para indicar que si hay más de un caracter separador que no los separe y los mantenga unidos.
data %>%
gather(clave, valor, -country) %>%
separate(clave, c("año", "otra_variable"), sep="_", extra = "merge")
#> # A tibble: 224 × 4
#> country año otra_variable valor
#> <chr> <chr> <chr> <dbl>
#> 1 Germany 1960 fertility 2.41
#> 2 South Korea 1960 fertility 6.16
#> 3 Germany 1960 life_expectancy 69.3
#> 4 South Korea 1960 life_expectancy 53.0
#> 5 Germany 1961 fertility 2.44
#> 6 South Korea 1961 fertility 5.99
#> 7 Germany 1961 life_expectancy 69.8
#> 8 South Korea 1961 life_expectancy 53.8
#> 9 Germany 1962 fertility 2.47
#> 10 South Korea 1962 fertility 5.79
#> # ℹ 214 more rowsYa tenemos separado el año, pero aun esta data no es tidy data dado que hay una fila para fertilidad y una fila para esperanza de vida para cada país. Tenemos que pasar estos valores de fila a columnas. Y para ello ya aprendimos que podemos usar la función spread()
data %>%
gather(clave, valor, -country) %>%
separate(clave, c("año", "otra_variable"), sep="_", extra = "merge") %>%
spread(otra_variable, valor)
#> # A tibble: 112 × 4
#> country año fertility life_expectancy
#> <chr> <chr> <dbl> <dbl>
#> 1 Germany 1960 2.41 69.3
#> 2 Germany 1961 2.44 69.8
#> 3 Germany 1962 2.47 70.0
#> 4 Germany 1963 2.49 70.1
#> 5 Germany 1964 2.49 70.7
#> 6 Germany 1965 2.48 70.6
#> 7 Germany 1966 2.44 70.8
#> 8 Germany 1967 2.37 71.0
#> 9 Germany 1968 2.28 70.6
#> 10 Germany 1969 2.17 70.5
#> # ℹ 102 more rowsEn otros casos, en vez de separar una columna vamos a querer unirlas. En futuros casos veremos cómo también puede ser útil la función unite(columna_1, columna2).
10.3 Ejercicios
- Explora el siguiente archivo: https://dparedesi.github.io/DS-con-R/uber_peru_2010.csv e importalo en el objeto
uber_peru_2010.
Solución
url <- "https://dparedesi.github.io/DS-con-R/uber_peru_2010.csv"
# Usaremos read_csv dado que está separado por comas
uber_peru_2010 <- read_csv(url)
# Al importarlo nos damos cuenta que está separado por ";"
uber_peru_2010 %>%
head()
# Por lo tanto importamos nuevamente usando read_dlim
uber_peru_2010 <- read_delim("external/uber_peru_2010.csv",
delim = ";",
col_types = cols(.default = "c")
)
uber_peru_2010 %>%
head()- Explora el siguiente archivo: https://www.datosabiertos.gob.pe/sites/default/files/DATASET_SINADEF.csv e importalo en el objeto
defunciones.
Solución
url <- "https://www.datosabiertos.gob.pe/sites/default/files/DATASET_SINADEF.csv"
url <- "https://dparedesi.github.io/DS-con-R/DATASET_SINADEF.csv"
# Usaremos read_delim porque está delimitado por ";" y no por ","
# Además cambiamos el encoding para evitar error en carga
defunciones <- read_delim(url, ";",
local = locale(encoding = "latin1"))- Descarga el archivo “https://dparedesi.github.io/DS-con-R/resources-other-idd.xlsx” y carga la pestaña “Deflators” al objeto
data.
Solución
# Almacenamos el url
url <- "https://dparedesi.github.io/DS-con-R/resources-other-idd.xlsx"
# Creamos un nombre & ruta temporal para nuestro archivo. Ver: ?tempfile
archivo_temporal <- tempfile()
# Descargamos el archivo en nuestro temporal
download.file(url, archivo_temporal)
# Importamos el excel
dat <- read_excel(archivo_temporal, sheet = "Deflators")
# Eliminamos el archivo temporal
file.remove(archivo_temporal)Para los siguientes archivos ejecuta el siguiente código para que tengas acceso a los objetos referidos en los problemas:
# PBI por países
url <- "https://dparedesi.github.io/DS-con-R/pbi.csv"
pbi <- read_csv(url)
# Enfermedades por años por países
url <- "https://dparedesi.github.io/DS-con-R/enfermedades-evolutivo.csv"
enfermedades_wide <- read_csv(url)
# Cantidad de alcaldes mujeres
url <- "https://dparedesi.github.io/DS-con-R/alcaldes-mujeres.csv"
alcaldes_mujeres <- read_csv(url)
# Evolutivo de una universidad
url <- "https://dparedesi.github.io/DS-con-R/universidad.csv"
universidad <- read_csv(url)- Explora el objeto
pbiy conviértelo a tidy data. Luego muestra un gráfico de líneas donde se diferencie la evolución del PBI de cada país.
Solución
- Explora el objeto
enfermedades_widey conviértelo a tidy data.
Solución
# Solución
enfermedades_1 <- enfermedades_wide %>%
gather(enfermedad, cantidad, -pais, -año, -poblacion)
enfermedades_1
# Solución alternativa. En vez de indicar qué obviar, indicamos qué tomar en cuenta
enfermedades_2 <- enfermedades_wide %>%
gather(enfermedad, cantidad, HepatitisA:Rubeola)
enfermedades_2- Explora el objeto
alcaldes_mujeresy luego conviértelo a tidy data
- Explora el objeto
universidady luego conviértelo a tidy data
10.4 Uniendo tablas
Regularmente tendremos datos de diferentes fuentes que luego tendremos que combinarlos para poder realizar nuestros análisis. Para ello aprenderemos diferentes grupos de funciones que nos permitirán combinar múltiples objetos.
10.4.1 Funciones join
Las funciones join son las más utilizadas en el cruce de tablas. Para utilizarlas tenemos que asegurarnos que tenemos instalado la librería dplyr.
Esta librería nos incluye una variedad de funciones para combinar tablas.
| Funcion | Uso |
|---|---|
left_join() |
Solo mantiene filas que tienen información en la primera tabla. |
right_join() |
Solo mantiene filas que tienen información en la segunda tabla. |
inner_join() |
Solo mantiene filas que tienen información en ambas tablas. |
full_join() |
mantiene todas las filas de ambas tablas. |
semi_join() |
Mantiene la parte de la primera tabla para la que tenemos información en la segunda. |
anti_join() |
Mantiene los elementos de la primera tabla para los que no hay información en la segunda. |
Para ver con ejemplos las funciones join utilizaremos los siguiente archivos:
url_1 <- "https://dparedesi.github.io/DS-con-R/j_tarjeta.csv"
url_2 <- "https://dparedesi.github.io/DS-con-R/j_cliente.csv"
data_1_tarjetas <- read_csv(url_1, col_types = cols(dni = col_character()))
data_2_clientes <- read_csv(url_2, col_types = cols(dni = col_character()))
data_1_tarjetas
#> # A tibble: 6 × 3
#> dni tipo_cliente tarjeta
#> <chr> <chr> <chr>
#> 1 45860518 premium VISA oro
#> 2 46534312 bronce Mastercard Black
#> 3 47564535 plata VISA plantinum
#> 4 48987654 bronce American Express
#> 5 78765434 oro VISA Signature
#> 6 41346556 premium Diners Club
data_2_clientes
#> # A tibble: 8 × 4
#> dni nombres apellido_paterno apellido_materno
#> <chr> <chr> <chr> <chr>
#> 1 49321442 Iver Castro Rivera
#> 2 47564535 Enrique Gutierrez Rivasplata
#> 3 48987654 Alexandra Cupe Gaspar
#> 4 47542345 Christiam Olortegui Roca
#> 5 41346556 Karen Jara Mory
#> 6 45860518 Hebert Lopez Chavez
#> 7 71234321 Jesus Valle Mariños
#> 8 73231243 Jenny Sosa Sosa10.4.1.1 Left join
Dadas dos tablas con un mismo identificador (en nuestro caso nuestro identificador consta solo de una sola columna: DNI), la función left join mantiene la información de la primera tabla y la completa con los datos que cruce en la segunda tabla
left_join(data_1_tarjetas, data_2_clientes, by = c("dni"))
#> # A tibble: 6 × 6
#> dni tipo_cliente tarjeta nombres apellido_paterno apellido_materno
#> <chr> <chr> <chr> <chr> <chr> <chr>
#> 1 45860518 premium VISA oro Hebert Lopez Chavez
#> 2 46534312 bronce Mastercard Bl… <NA> <NA> <NA>
#> 3 47564535 plata VISA plantinum Enrique Gutierrez Rivasplata
#> 4 48987654 bronce American Expr… Alexan… Cupe Gaspar
#> 5 78765434 oro VISA Signature <NA> <NA> <NA>
#> 6 41346556 premium Diners Club Karen Jara MoryComo vemos, las tres primeras columnas son exactamente las mismas que teníamos inicialmente y a la derecha de esas columnas vemos las columna de la otra tabla para los valores que sí llegó a cruzar los datos. En este caso estamos ante una inconsistencia de datos puesto que todos los clientes de la data_1_tarjetas deberían de estar en data_2_clientes. Esta inconsistencia nos podría llevar a tener que mapear el proceso de pérdida de datos, etc.
10.4.1.2 Right join
Dadas dos tablas con un mismo identificador, la función right join mantiene la información de la segunda tabla y la completa con los datos que cruce en la primera tabla
right_join(data_1_tarjetas, data_2_clientes, by = "dni")
#> # A tibble: 8 × 6
#> dni tipo_cliente tarjeta nombres apellido_paterno apellido_materno
#> <chr> <chr> <chr> <chr> <chr> <chr>
#> 1 45860518 premium VISA oro Hebert Lopez Chavez
#> 2 47564535 plata VISA plantinum Enrique Gutierrez Rivasplata
#> 3 48987654 bronce American Expr… Alexan… Cupe Gaspar
#> 4 41346556 premium Diners Club Karen Jara Mory
#> 5 49321442 <NA> <NA> Iver Castro Rivera
#> 6 47542345 <NA> <NA> Christ… Olortegui Roca
#> 7 71234321 <NA> <NA> Jesus Valle Mariños
#> 8 73231243 <NA> <NA> Jenny Sosa SosaLa idea es la misma que en left_join, solo que esta vez los NA están en las primeras dos columnas.
10.4.1.3 Inner join
En este caso solo tendremos la intersección de las tablas. Solo se mostrará el resultado de los datos que estén en ambas tablas.
inner_join(data_1_tarjetas, data_2_clientes, by = "dni")
#> # A tibble: 4 × 6
#> dni tipo_cliente tarjeta nombres apellido_paterno apellido_materno
#> <chr> <chr> <chr> <chr> <chr> <chr>
#> 1 45860518 premium VISA oro Hebert Lopez Chavez
#> 2 47564535 plata VISA plantinum Enrique Gutierrez Rivasplata
#> 3 48987654 bronce American Expr… Alexan… Cupe Gaspar
#> 4 41346556 premium Diners Club Karen Jara Mory10.4.1.4 Full join
Full join es un cruce total de ambas. Nos muestra todos los datos que están tanto en la primera como en la segunda tabla.
full_join(data_1_tarjetas, data_2_clientes, by = "dni")
#> # A tibble: 10 × 6
#> dni tipo_cliente tarjeta nombres apellido_paterno apellido_materno
#> <chr> <chr> <chr> <chr> <chr> <chr>
#> 1 45860518 premium VISA oro Hebert Lopez Chavez
#> 2 46534312 bronce Mastercard B… <NA> <NA> <NA>
#> 3 47564535 plata VISA plantin… Enrique Gutierrez Rivasplata
#> 4 48987654 bronce American Exp… Alexan… Cupe Gaspar
#> 5 78765434 oro VISA Signatu… <NA> <NA> <NA>
#> 6 41346556 premium Diners Club Karen Jara Mory
#> 7 49321442 <NA> <NA> Iver Castro Rivera
#> 8 47542345 <NA> <NA> Christ… Olortegui Roca
#> 9 71234321 <NA> <NA> Jesus Valle Mariños
#> 10 73231243 <NA> <NA> Jenny Sosa Sosa10.4.1.5 Semi join
El caso del semi join es muy parecido al left_join con la diferencia que solo nos muestra las columnas de la primera tabla y nos elimina los datos que no llegaron a cruzar (lo que en left_join salen como NA). Además, no figura ninguna de las columnas de la tabla 2. Esto es como hacer un filtro solicitando lo siguiente: muéstrame solo los datos de la tabla 1 que estén también en la tabla 2.
10.4.2 Uniendo sin un mismo identificador
Así mismo tendremos algunos momentos en los que necesitamos solo combinar dos objetos, sin utilizar la intersección de ningún tipo. Para ello usaremos las funciones bind. Estas funciones nos permiten poner juntas dos vectores o tablas ya sea en filas o columnas.
10.4.2.1 Unión de vectores
Si tenemos dos o más vectores del mismo tamaño podemos crear la unión de las columnas para crear una tabla utilizando la función bind_cols(). Veamos con un ejemplo:
10.4.2.2 Unión de tablas
En el caso de las tablas el uso es el mismo. Así mismo, también podemos unir las filas de dos o más tablas. Para ver su aplicación primero creemos algunas tablas de ejemplo:
tabla_1 <- data.frame(
nombre = c("Jhasury", "Thomas", "Andres", "Josep"),
apellido = c("Campos", "Gonzales", "Santiago", "Villaverde"),
direccion = c("Jr. los campos 471", "Av. Casuarinas 142", NA, "Av. Tupac Amaru 164"),
telefono = c("976567325", "956732587", "961445664", "987786453")
)
tabla_2 <- data.frame(
edad = c(21, 24, 19, 12),
signo = c("Aries", "Capricornio", "Sagitario", "Libra")
)
# Crea una tabla de la fila 2 a la 3 de la tabla 1
tabla_3 <- tabla_1[2:3, ]Una vez tenemos nuestras tablas procedamos a unirlas. Vemos que no tienen un identificador en común.
resultado <- bind_cols(tabla_1, tabla_2)
resultado
#> nombre apellido direccion telefono edad signo
#> 1 Jhasury Campos Jr. los campos 471 976567325 21 Aries
#> 2 Thomas Gonzales Av. Casuarinas 142 956732587 24 Capricornio
#> 3 Andres Santiago <NA> 961445664 19 Sagitario
#> 4 Josep Villaverde Av. Tupac Amaru 164 987786453 12 LibraAsí mismo, podemos unir por filas de esta forma:
resultado <- bind_rows(tabla_1, tabla_3)
resultado
#> nombre apellido direccion telefono
#> 1 Jhasury Campos Jr. los campos 471 976567325
#> 2 Thomas Gonzales Av. Casuarinas 142 956732587
#> 3 Andres Santiago <NA> 961445664
#> 4 Josep Villaverde Av. Tupac Amaru 164 987786453
#> 5 Thomas Gonzales Av. Casuarinas 142 956732587
#> 6 Andres Santiago <NA> 96144566410.5 Web Scrapping
El Web Scrapping es el proceso de extracción de datos de un sitio web. Lo usaremos cuando necesitamos extraer directamente datos de tablas que son presentadas en sitios web. Para ello utilizaremos la librería rvest, incluida en la librería tidyverse.
La función que más utilizaremos será read_html() y como argumento colocaremos el url de la web de donde queremos extraer los datos. No estamos hablando de un url que nos descargar un archivo de texto sino de una página web como esta:

Así, usaremos read_html() para almacenar todo el html de la web y luego poco a poco ir accediendo a los datos de la tabla en R.
data_en_html <- read_html("https://es.wikipedia.org/wiki/Anexo:Pa%C3%ADses_hispanos_por_poblaci%C3%B3n")
data_en_html
#> {html_document}
#> <html class="client-nojs vector-feature-language-in-header-enabled vector-feature-language-in-main-page-header-disabled vector-feature-sticky-header-disabled vector-feature-page-tools-pinned-disabled vector-feature-toc-pinned-clientpref-1 vector-feature-main-menu-pinned-disabled vector-feature-limited-width-clientpref-1 vector-feature-limited-width-content-enabled vector-feature-custom-font-size-clientpref-1 vector-feature-appearance-pinned-clientpref-1 vector-feature-night-mode-enabled skin-theme-clientpref-day vector-toc-available" lang="es" dir="ltr">
#> [1] <head>\n<meta http-equiv="Content-Type" content="text/html; charset=UTF-8 ...
#> [2] <body class="skin--responsive skin-vector skin-vector-search-vue mediawik ...Ahora que tenemos la data almacenada en el objeto tenemos que ir buscando la data, hacer scrapping. Para ello usaremos la función html_nodes("table") para acceder al nodo “table”.
Finalmente, tenemos que ir índice por índice buscando la tabla que nos interesa. Para darle formato de tabla usaremos html_table. En este caso usaremos doble corchete porque se trata de una lista de listas y la función setNames() para cambiarle el nombre a las columnas.
# Damos formato de tabla y almacenamos en wide_tabla
tabla_en_bruto <- tablas_web[[1]] %>%
html_table
# Cambiamos los nombres de las cabeceras
tabla_en_bruto <- tabla_en_bruto %>%
setNames(
c("N", "pais", "poblacion", "prop_poblacion", "cambio_medio", "link")
)
# Convertimos a tibble
tabla_en_bruto <- tabla_en_bruto %>%
as_tibble()
# Reportamos primeras filas
tabla_en_bruto %>%
head(5)
#> # A tibble: 1 × 2
#> N pais
#> <lgl> <chr>
#> 1 NA Este artículo o sección se encuentra desactualizado.La información sumi…Ya tenemos nuestra data importada y ya podríamos empezar a explorar el contenido a detalle de la misma.
10.6 Ejercicios
Para los siguientes ejercicios utilizaremos objetos de la librería Lahman, la cual contiene datos de jugadores de béisbol de EEUU. Ejecuta el siguiente Script antes de empezar a resolver los ejercicios.
install.packages("Lahman")
library(Lahman)
# Top 10 jugadores del año 2016
top_jugadores <- Batting %>%
filter(yearID == 2016) %>%
arrange(desc(HR)) %>% # ordenado por número de "Home run"
slice(1:10) # Toma de la fila a la 10
top_jugadores <- top_jugadores %>% as_tibble()
# Listado de todos los jugadores de béisbol de los últimos años
maestra <- Master %>% as_tibble()
# Premios ganados por jugadores
premios <- AwardsPlayers %>%
filter(yearID == 2016) %>%
as_tibble()- Usando el objeto top_jugadores y el objeto Maestra, reporta los siguientes campos
playerID,nameFirst,nameLast,HRde los top 10 jugadores del 2016.
Solución
- Reporta el ID y los nombres de los top 10 jugadores que han ganado al menos un premio, objeto
premios, en el 2016.
- Reporta el ID y los nombres de los jugadores que ganaron al menos un premio en el 2016, pero no son parte de los top 10.
Solución
# Primero calculamos todos los premios de los que no son top 10:
ID_de_premiados_no_top <- anti_join(premios, top_10, by = "playerID") %>%
select(playerID)
# Como un jugador pudo haber obtenido varios premios obtenemos valores únicos
ID_de_premiados_no_top <- unique(ID_de_premiados_no_top)
# Luego cruzamos con la maestra para obtener los nombres
nombres_de_otros <- left_join(ID_de_premiados_no_top, maestra, by = "playerID") %>%
select(playerID, nameFirst, nameLast)
nombres_de_otros- Almacena las tablas de la siguiente página web: http://www.stevetheump.com/Payrolls.htm que contiene la planilla que paga cada equipo de béisbol de EEUU a Febrero del 2020 en el objeto
html. Luego, accede a los nodos utilizandohtml_nodes("table")y almacénalo en el objetonodos. Finalmente, reporta el nodo 4 en formatohtml_table.
Solución
- Del objeto
nodoscreado en el ejercicio anterior, almacena el nodo 4 en la el objetoplanilla_2019y el nodo 5 en el objetoplanilla_2018. Ahora dale formato a ambas tabla de tal forma que las cabeceras sean: equipo, planilla_2018 o planilla_2019 de acuerdo a la tabla. Finalmente, haz un cruce de ambas tablas por nombre de equipo utilizandofull_join().
Solución
planilla_2019 <- nodos[[4]] %>%
html_table()
planilla_2018 <- nodos[[5]] %>%
html_table()
####### Planilla 2019: ################
#Eliminamos la fila 15 que es el promedio de la liga:
planilla_2019 <- planilla_2019[-15, ]
#Filtramos las columnas solicitadas:
planilla_2019 <- planilla_2019 %>%
select(X2, X4) %>%
rename(equipo = X2, planilla_2019 = X4)
# Eliminamos fila 1 dado que es la cabecera de origen
planilla_2019 <- planilla_2019[-1,]
####### Planilla 2018: ################
# Seleccionamos las dos columnas que nos interesan y
#cambiamos de nombre a cabeceras
planilla_2018 <- planilla_2018 %>%
select(Team, Payroll) %>%
rename(equipo = Team, planilla_2018 = Payroll)
####### Full join: ################
full_join(planilla_2018, planilla_2019, by = "equipo")