G
get_pwc_label() {rstatix}¡Atención!
Instalar el paquete rstatix para hacer uso de la función get_pwc_label.
Para ello utilizar el siguiente código:
install.packages("rstatix")| Descripción | Argumentos |
|---|---|
| Extrae información de etiquetas de pruebas estadísticas. Útil para etiquetar gráficos con resultados de pruebas |
|
Ejemplo práctico disponible en letra G, función ggboxplot() {ggpubr}get_test_label() {rstatix}¡Atención!
Instalar el paquete rstatix para hacer uso de la función get_test_label.
Para ello utilizar el siguiente código:
install.packages("rstatix")| Descripción | Argumentos |
|---|---|
| Extrae información de etiquetas de pruebas estadísticas. Útil para etiquetar gráficos con resultados de pruebas |
|
Ejemplo práctico disponible en letra G, función ggboxplot() {ggpubr}ggboxplot() {ggpubr}¡Atención!
Instalar el paquete ggpubr para hacer uso de la función ggboxplot.
Para ello utilizar el siguiente código:
install.packages("ggpubr")| Descripción | Argumentos |
|---|---|
| Crear un gráfico de caja con puntos |
|
palette
"Dark2", "Set1"
add
"jitter" : agrega los valores de un vector numérico de forma dispersa
"mean" : agrega la media en el boxplotafluentes = factor(rep(c("Archibarca","Cerro Overo","Rosario"), each = 12))
MOT = c(1.40,2.16,0.74,0.86,1.13,1.75,1.86,1.16,2.36,1.46,0.98,1.14,
4.14,2.25,2.16,2.45,2.56,2.36,2.78,2.69,3.16,2.48,2.63,2.59,
1.10,1.05,1.15,0.60,1.11,0.63,0.89,0.75,1.03,1.06,1.09,0.55)
data.kruskal = data.frame(afluentes, MOT)
library(ggpubr)
ggboxplot(data = data.kruskal,
x = "afluentes",
y = "MOT",
color = "afluentes",
palette = "Dark2",
add = c("jitter","mean"))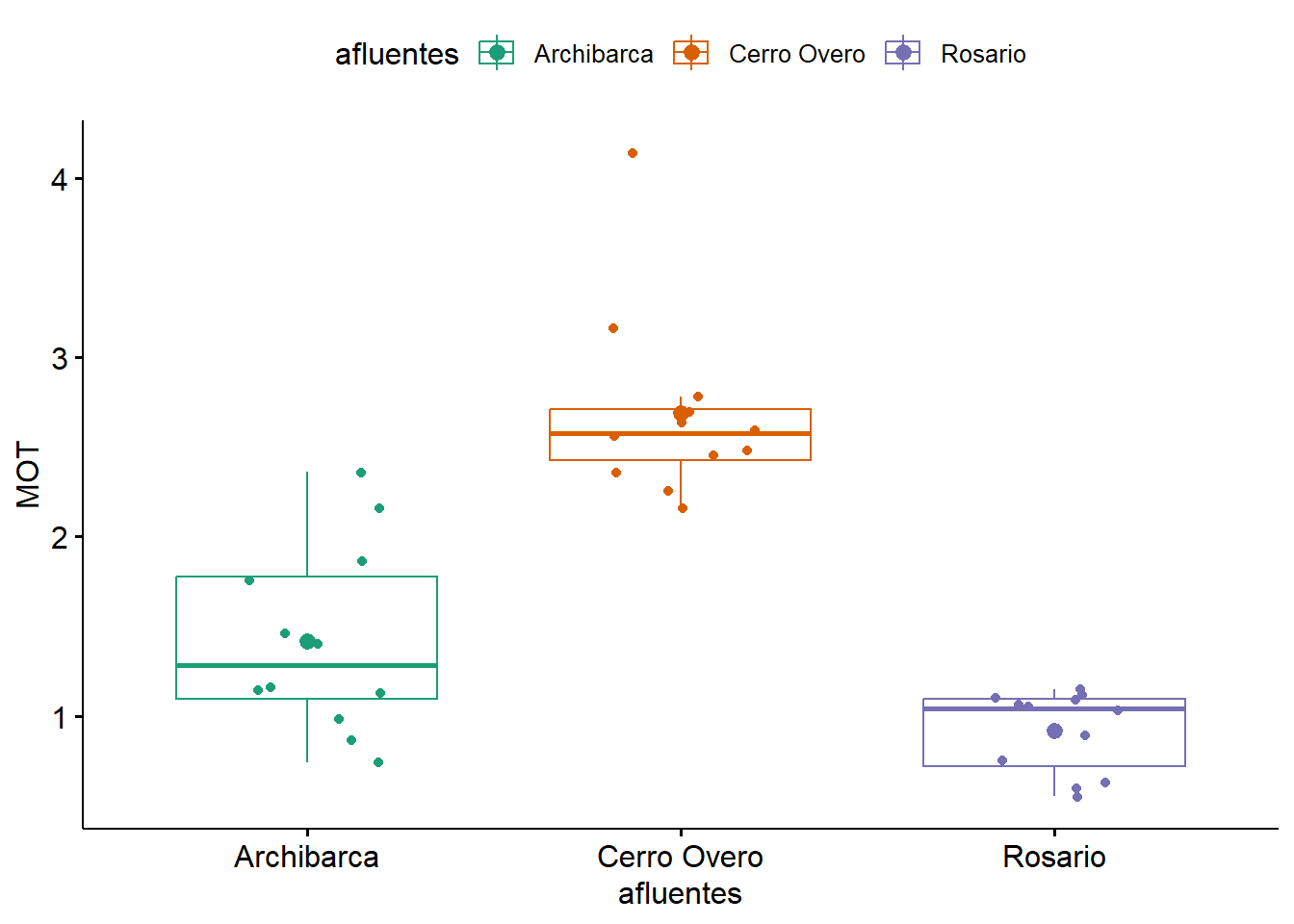
ggboxplot(data = data.kruskal,
x = "afluentes",
y = "MOT",
color = "afluentes",
palette = "Set1",
add = "mean")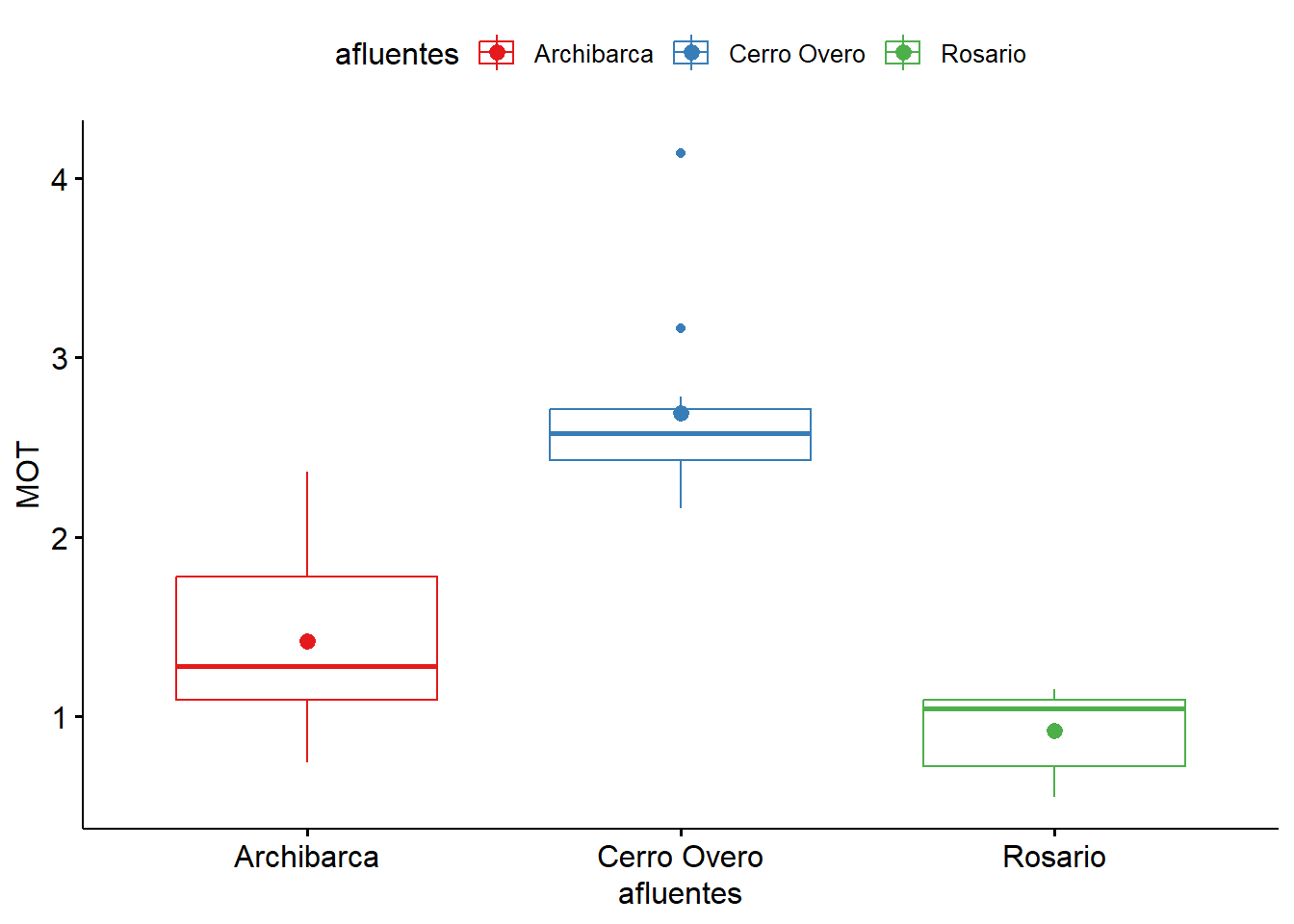
Con el símbolo + se añaden nuevas capas al gráfico construido.
#theme()
ggboxplot(data = data.kruskal,
x = "afluentes",
y = "MOT",
color = "afluentes",
palette = "Set1",
add = "mean") +
theme(legend.position = "none")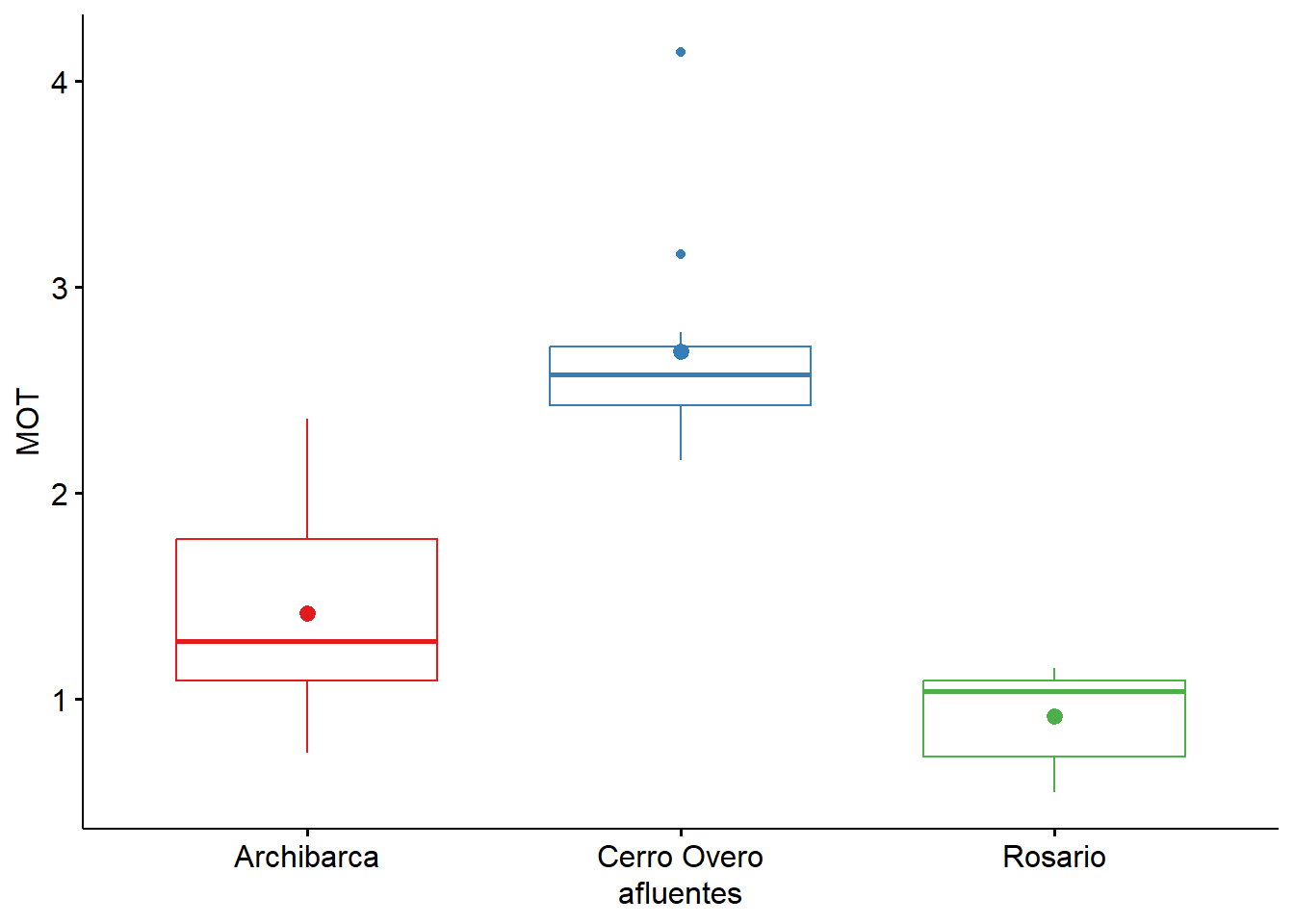
#add_xy_position()
library(rstatix)
pwc = data.kruskal %>%
wilcox_test(MOT ~ afluentes,
p.adjust.method = "bonferroni",
paired = F)
library(tidyverse) #para utilizar %>%
pwc = pwc %>%
add_xy_position(x = "afluentes")
#stat_pvalue_manual()
ggboxplot(data = data.kruskal,
x = "afluentes",
y = "MOT",
color = "afluentes",
palette = "Set1",
add = "mean") +
stat_pvalue_manual(data = pwc,
hide.ns = TRUE)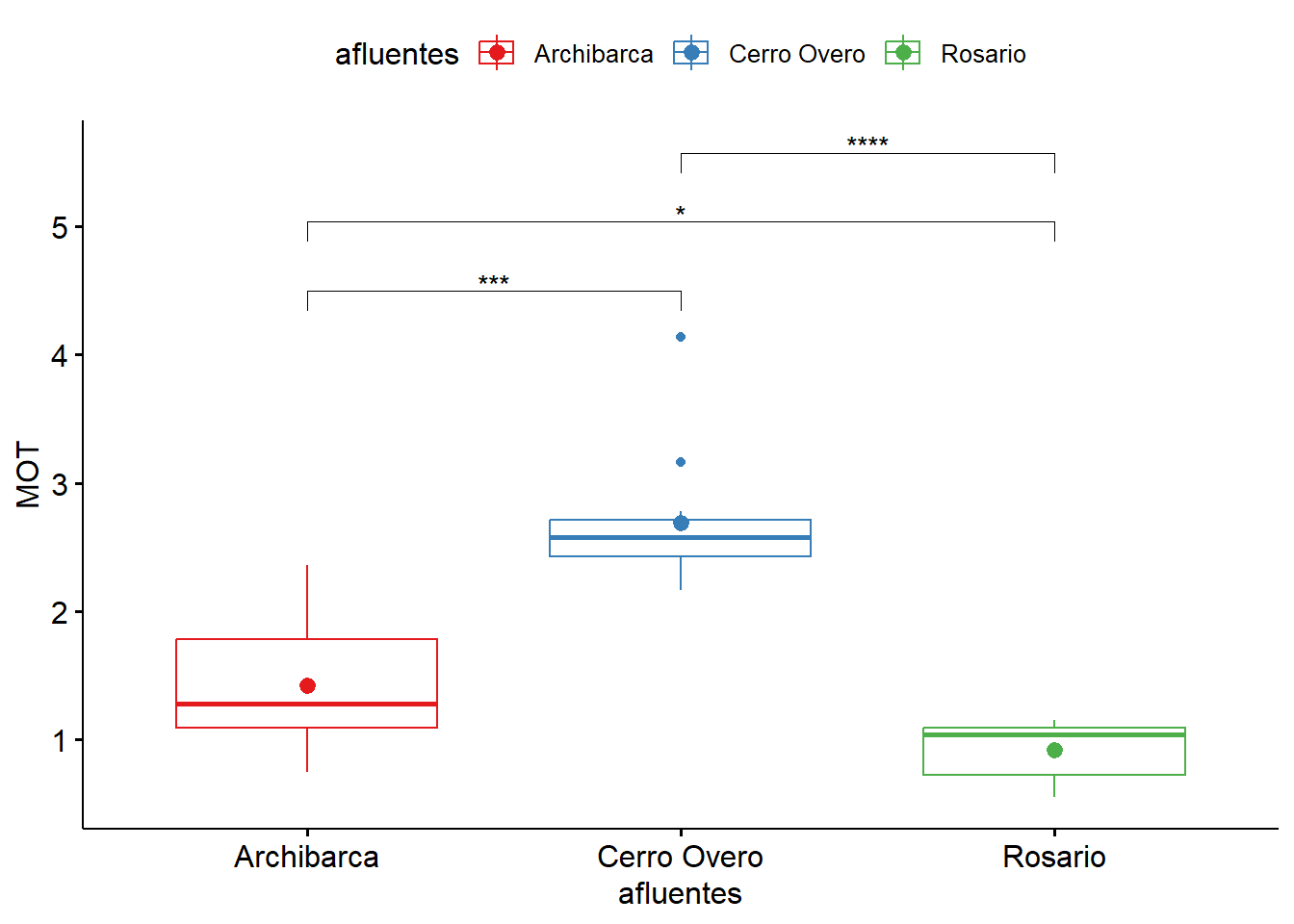
# labs() get_test_label() y get_pwc_label()
res.kruskal = data.kruskal %>%
kruskal_test(MOT ~ afluentes)
data.kruskal %>%
ggboxplot(x = "afluentes",
y = "MOT",
color = "afluentes",
palette = "Dark2",
add = c("jitter","mean")) +
theme(legend.position = "none") +
stat_pvalue_manual(pwc,
hide.ns = TRUE) +
labs(subtitle = "Subtítulo",
caption = "Título en parte inferior")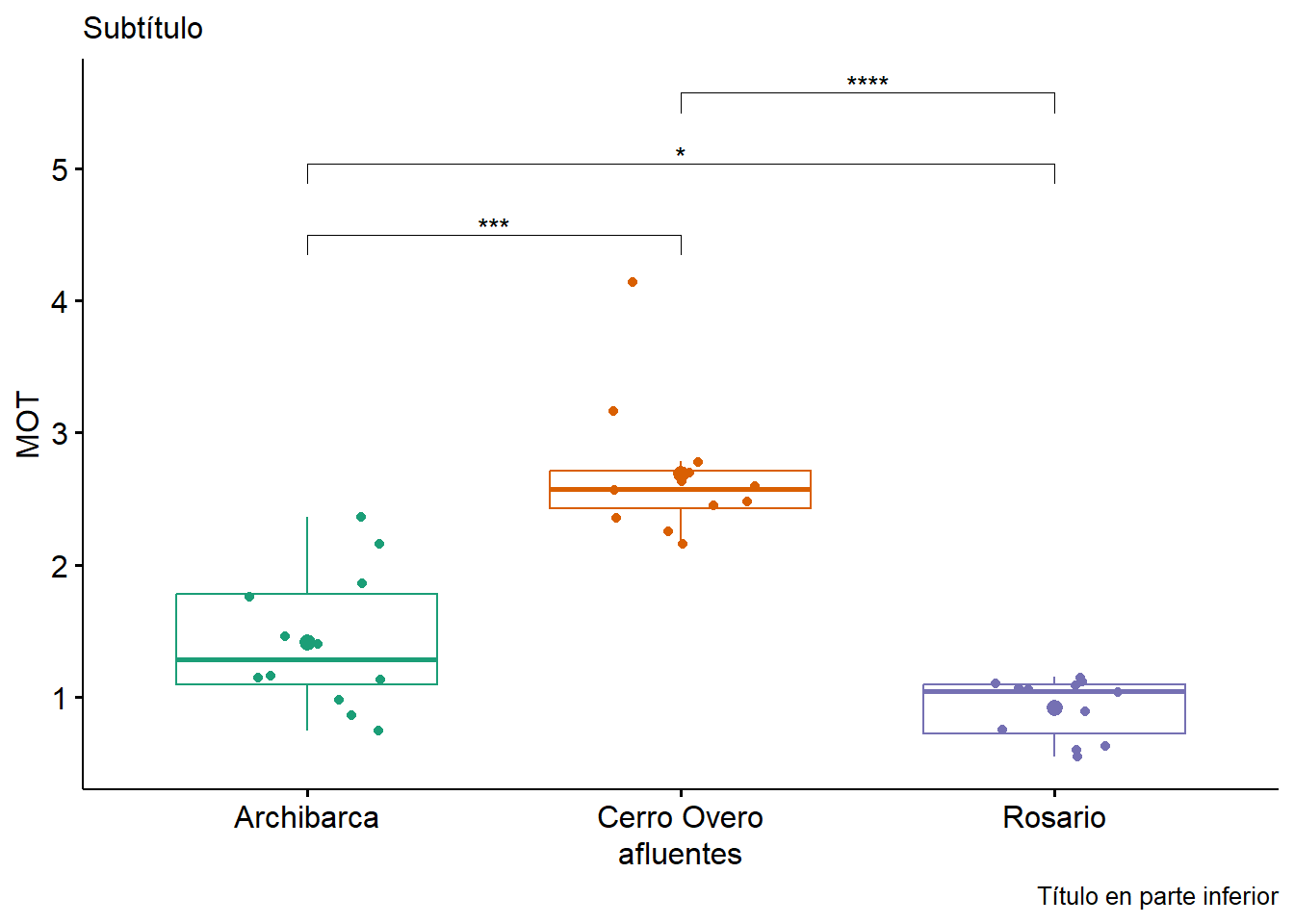
data.kruskal %>%
ggboxplot(x = "afluentes",
y = "MOT",
color = "afluentes",
palette = "Dark2",
add = c("jitter","mean")) +
stat_pvalue_manual(pwc,
hide.ns = TRUE) +
labs(title = "tidyverse, rstatix, ggpubr",
subtitle = get_test_label(res.kruskal,
type = "expression",
detailed = TRUE),
caption = get_pwc_label(stat.test = pwc,
type = "expression"))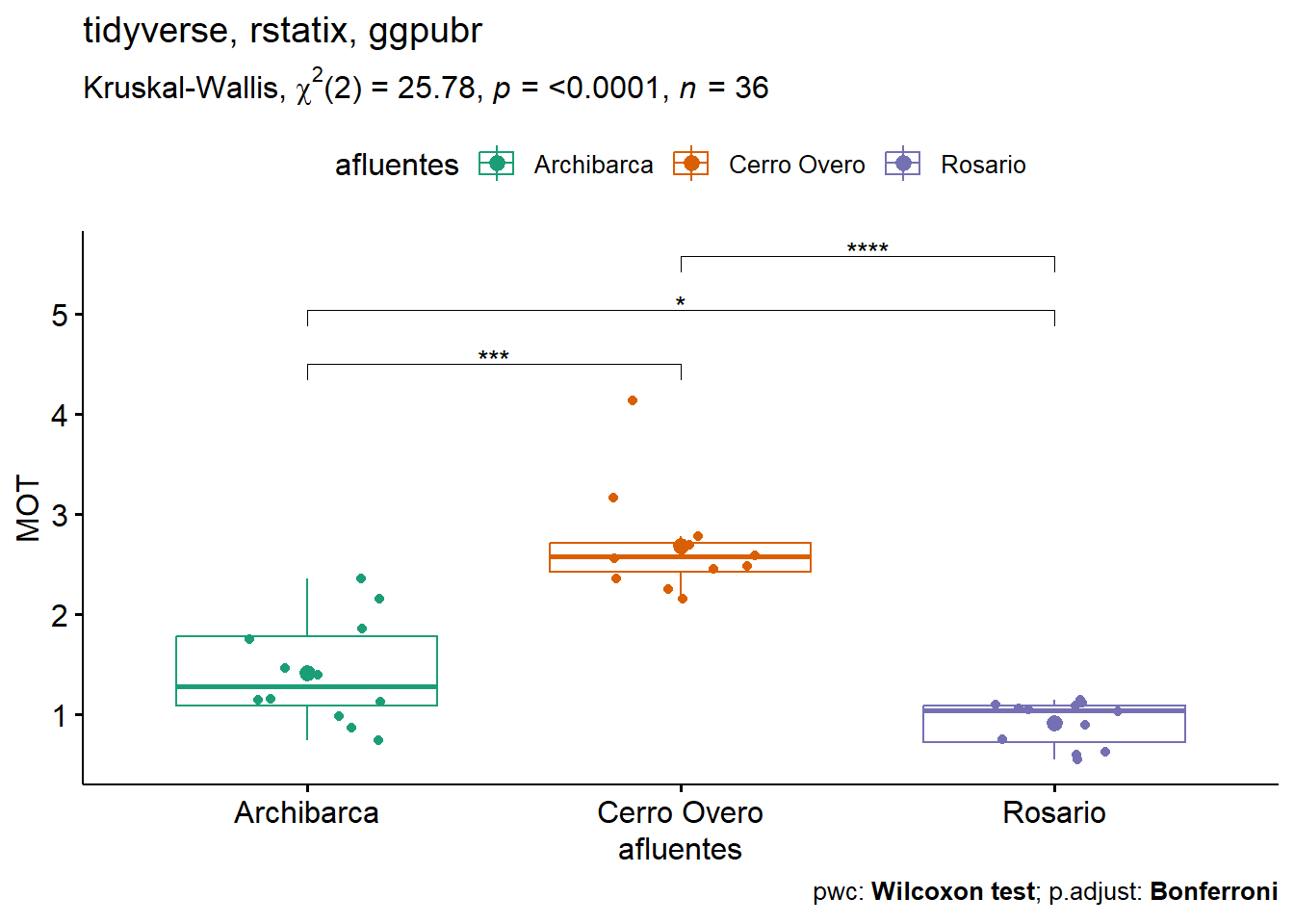
glht() {multcomp}¡Atención!
Instalar el paquete multcomp para hacer uso de la función glht.
Para ello utilizar el siguiente código:
install.packages("multcomp")| Descripción | Argumentos |
|---|---|
| Realiza comparaciones múltiples |
|
#Importación de datos
datos.glht = data.frame(
CO2 = factor(
rep(
c("0.0","0.083","0.29","0.50","0.86"),
each = 10
)
),
respuesta = c(
62.6,59.6,64.5,59.3,58.6,64.6,50.9,56.2,52.3,62.8,
50.9,44.3,47.5,49.5,48.5,50.4,35.2,49.9,42.6,41.6,
45.5,41.1,29.8,38.3,40.2,38.5,30.2,27.0,40.0,33.9,
29.5,22.8,19.2,20.6,29.2,24.1,22.6,32.7,24.4,29.6,
24.9,17.2,7.8,10.5,17.8,22.1,22.6,16.8,15.9,8.8
)
)
#Construcción del modelo
modelo.glht = lm(respuesta ~ CO2, data = datos.glht)library(multcomp)
dunnett = glht(model = modelo.glht,
linfct = mcp(CO2 = "Dunnett"))grepl() {base}| Descripción | Argumentos |
|---|---|
| Se utiliza para buscar patrones de texto en un vector de caracteres y devuelve un vector lógico que indica si cada elemento del vector contiene una coincidencia con el patrón especificado |
|
## [1] "Acidulado" "Dulce" "Dulce" "Dulce"
## [5] "Acidulado" "Acido" "Dulce" "Amargo"
## [9] "Acidulado" "Dulce-amargo" "Dulce" "Acido-amargo"
## [13] "Acido" "Acido" "Dulce" "Dulce"
## [17] "Amargo-acidulado"## [1] FALSE TRUE TRUE TRUE FALSE FALSE TRUE FALSE FALSE TRUE TRUE FALSE
## [13] FALSE FALSE TRUE TRUE FALSE# No ignora las diferencias entre mayuscúlas y minúsculas
grepl(pattern = "d",
x = datos$Caracterización,
ignore.case = T)## [1] TRUE TRUE TRUE TRUE TRUE TRUE TRUE FALSE TRUE TRUE TRUE TRUE
## [13] TRUE TRUE TRUE TRUE TRUEEn este caso se está realizando la búsqueda de patrones que contengan la letra “D” (en el primer código) y la letra “d” (en el segundo código) en la columna Caracterización de la Tabla 1: datos.
group_by() {dplyr}¡Atención!
Instalar el paquete dplyr para hacer uso de la función group_by.
Para ello utilizar el siguiente código:
install.packages("dplyr")| Descripción |
|---|
| La mayoría de las operaciones de datos se realizan en grupos definidos por variables. group_by() toma un tabla existente y lo convierte en una tabla agrupada donde las operaciones se realizan “por grupo” |
## # A tibble: 2 × 2
## Departamento Mediana
## <chr> <dbl>
## 1 Humahuaca 3.74
## 2 Tumbaya 3.64