10 Daten umstrukturieren und zusammenfügen
Datenhandling umfasst oft auch etwas komplexere Schritte, als lediglich relevante Variablen zu selektieren und nach bestimmten Bedingungen zu filtern. In diesem Kapitel sehen wir uns daher an, wie wir Daten umstrukturieren und mehrere Datensätze zusammenfügen können.
10.1 Daten umstrukturieren
Wir haben in Kapitel 7.1.1 gesehen, dass dieselben Daten ganz unterschiedlich tabellarisch abgebildet werden können. Oftmals ist es jedoch so, dass wir unsere Daten in einem ganz bestimmten Format benötigen, um mit diesen weiterarbeiten zu können – beispielsweise um diese an eine Funktion, die ein statistisches Verfahren implementiert, zu übergeben oder diese grafisch darzustellen. Hierfür bietet das Tidyverse einige Funktionen, um Datensätze schnell umzustrukturieren.
10.1.1 Wide vs. Long Data
Einer der häufigsten Fälle der Umstrukturierung von Datensätzen betrifft das Konvertieren von Wide Data in Long Data und umgekehrt.
Nehmen wir als Beispiel einen Datensatz, der die Auflagezahlen verschiedener politischer Wochenmagazine beinhält:
## # A tibble: 3 x 4
## medium auflage_2018 auflage_2019 auflage_2020
## <chr> <dbl> <dbl> <dbl>
## 1 Der Spiegel 708077 701337 685799
## 2 Stern 539191 476097 422156
## 3 Focus 425737 373847 328587Man spricht hierbei von Wide Data: Mehrere Beobachtungen desselben Wertetyps stehen in unterschiedlichen Spalten – in diesem Fall haben wir drei Spalten, in denen jeweils Auflagenzahlen stehen. Unsere Fallebene ist das einzelne Medium: jeweils eine Zeile für Spiegel, Stern und Focus.
Wir könnten dieselben Daten aber auch so darstellen:
## # A tibble: 9 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Der Spiegel 2018 708077
## 2 Der Spiegel 2019 701337
## 3 Der Spiegel 2020 685799
## 4 Stern 2018 539191
## 5 Stern 2019 476097
## 6 Stern 2020 422156
## 7 Focus 2018 425737
## 8 Focus 2019 373847
## 9 Focus 2020 328587Hierbei handelt es sich um Long Data: alle Beobachtungen desselben Wertetyps, also z. B. die Auflagenzahlen, stehen in einer Spalte, die weiteren Spalten dienen zur Identifikation dieser Beobachtungen, hier also, für welches Jahr und für welches Medium diese gelten. Unsere Fallebene wäre also Medium und Jahr: jeweils eine Zeile pro Medium und Jahr.
Wide Data ist häufig intuitiver zu lesen, insbesondere wenn es sich um bivariate Verteilungen handelt, da die Darstellung einer Kreuztabelle gleicht. Tatsächlich würden wir diese erste tabellarische Darstellung aber nicht als tidy bezeichnen, da derselbe Werte- bzw. Beobachtungstyp in unterschiedlichen Spalten steht.
Viele Funktionen in R sind jedoch auf Long Data ausgelegt. Häufig müssen wir daher Wide Data in Long Data transformieren:
10.1.2 Wide Data in Long Data transformieren mit pivot_longer()
Mittels pivot_longer() können wir Wide Data in Long Data umwandeln. Als erstes Argument nutzen wir hierbei wie immer den Datensatz, den wir transformieren möchten, gefolgt von einem Vektor, der alle Spalten umfasst, die “länger” gemacht werden sollen.
Als Beispiel nutzen wir den ersten Auflagen-Datensatz aus dem vorherigen Unterkapitel, der über folgenden Code erstellt wird:
auflagen_wide <- tibble(
medium = c("Der Spiegel", "Stern", "Focus"),
auflage_2018 = c(708077, 539191, 425737),
auflage_2019 = c(701337, 476097, 373847),
auflage_2020 = c(685799, 422156, 328587)
)Wir möchten nun alle Auflagenspalten (auflage_2018, auflage_2019, auflage_2020) transformieren, sodass alle Auflagenwerte in einer Spalte stehen. Wir übergeben diese Spalten daher als zweites Argument als pivot_longer():
## # A tibble: 9 x 3
## medium name value
## <chr> <chr> <dbl>
## 1 Der Spiegel auflage_2018 708077
## 2 Der Spiegel auflage_2019 701337
## 3 Der Spiegel auflage_2020 685799
## 4 Stern auflage_2018 539191
## 5 Stern auflage_2019 476097
## 6 Stern auflage_2020 422156
## 7 Focus auflage_2018 425737
## 8 Focus auflage_2019 373847
## 9 Focus auflage_2020 328587Das führt uns schon (fast) zum gewünschten Ergebnis. Wie wir sehen, stehen nun alle Auflagenzahlen in der Spalte value, die Information aus den Spaltennamen stehen in der Spalte name. Wir können den Aufruf aber noch etwas anpassen, um die Ausgabe zu optimieren:
- Mit nur drei Variablen war es kein großer Aufwand, alle Variablen zum umtransformieren einzeln anzugeben. Wenn wir jedoch Auflagenzahlen der vergangenen 50 Jahre hätten, wäre dies sehr viel Tipparbeit. Wir können jedoch alle Hilfsfunktionen, die auch bei
select()(siehe Kapitel 8.2.1) zur Verfügung stehen verwenden. In unserem Fall könnten wir uns die Arbeit beispielsweise mitstarts_with("auflage")abkürzen.19 - Die neuen Variablen haben recht generische Namen. Hier können wir mit den Argumenten
names_to(Name(n) der neuen Spalte(n) basierend auf den alten Spaltennamen) undvalues_to(Name der neuen Wertespalte) Text-Vektoren übergeben, die die neuen Spaltennamen beinhalten. In unserem Fall bieten sich dahernames_to = "jahr"undvalues_to = "auflage"an. - Der Bestandteil
"auflage_"ist redundant; schöner wäre es wenn lediglich die Jahreszahl angeben wird. Hierfür können wir das Argumentnames_prefixnutzen und einen Textbestandteil der Spaltennamen angeben, der abgeschnitten werden soll – in unserem Fall alsonames_prefix = "auflage_".
auflagen_wide %>%
pivot_longer(starts_with("auflage"), names_to = "jahr", values_to = "auflage", names_prefix = "auflage_")## # A tibble: 9 x 3
## medium jahr auflage
## <chr> <chr> <dbl>
## 1 Der Spiegel 2018 708077
## 2 Der Spiegel 2019 701337
## 3 Der Spiegel 2020 685799
## 4 Stern 2018 539191
## 5 Stern 2019 476097
## 6 Stern 2020 422156
## 7 Focus 2018 425737
## 8 Focus 2019 373847
## 9 Focus 2020 328587Schon fast perfekt! Das einzige Schönheitsfehler ist, dass die Variable jahr nun vom Typ character ist – pivot_longer() weist diesen Typ allen Informationen zu, die aus den ursprünglichen Spaltennamen stammen. Sinnvoller wäre ein numerischer Objekttyp. Hier können wir dem Argument names_transform eine Liste übergeben (auch wenn dies nur eine Variable betrifft), die für alle aus den ursprünglichen Spaltennamen generierten Variablen jeweils den gewünschten Objekttyp angibt:
# Da nun alles passt, weisen wir unseren neuen Long-Datensatz auch einem Objekt zu
auflagen_long <- auflagen_wide %>%
pivot_longer(starts_with("auflage"), names_to = "jahr", values_to = "auflage",
names_prefix = "auflage_", names_transform = list(jahr = as.integer))
auflagen_long## # A tibble: 9 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Der Spiegel 2018 708077
## 2 Der Spiegel 2019 701337
## 3 Der Spiegel 2020 685799
## 4 Stern 2018 539191
## 5 Stern 2019 476097
## 6 Stern 2020 422156
## 7 Focus 2018 425737
## 8 Focus 2019 373847
## 9 Focus 2020 328587Betrachten wir ein zweites, noch etwas komplexeres Beispiel:
auflagen_wide2 <- tibble(
medium = c("Der Spiegel", "Stern", "Focus"),
auflage_2018_q1 = c(708077, 539191, 425737),
auflage_2018_q2 = c(704656, 528860, 417759),
auflage_2018_q3 = c(716663, 514889, 412165),
auflage_2018_q4 = c(712268, 480739, 413276),
auflage_2019_q1 = c(701337, 476097, 373847),
auflage_2019_q2 = c(707459, 464489, 367101),
auflage_2019_q3 = c(719326, 466019, 364254),
auflage_2019_q4 = c(691451, 440284, 349944),
auflage_2020_q1 = c(685799, 422156, 328587)
)
auflagen_wide2## # A tibble: 3 x 10
## medium auflage_2018_q1 auflage_2018_q2 auflage_2018_q3 auflage_2018_q4 auflage_2019_q1 auflage_2019_q2 auflage_2019_q3 auflage_2019_q4 auflage_2020_q1
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Der Spiegel 708077 704656 716663 712268 701337 707459 719326 691451 685799
## 2 Stern 539191 528860 514889 480739 476097 464489 466019 440284 422156
## 3 Focus 425737 417759 412165 413276 373847 367101 364254 349944 328587Nun haben in den Spaltennamen gleich zwei Informationen: das Jahr und das jeweilige Quartal. Es ist daher sinnvoll, diese Informationen beim Transformieren in getrennten Spalten zu speichern. Hierzu können wir zusätzlich das Argument names_sep heranziehen, mit dem wir ein(e) Zeichen(kette) übergeben – in unserem Fall den Unterstrich _, an der wir die ursprünglichen Spaltennamen weiter aufteilen möchten. Entsprechend müssen wir in names_to dann mehrere neuen Spaltennamen angeben – in unserem Fall 2, da wir jahr und quartal trennen möchten:20
auflagen_long2 <- auflagen_wide2 %>%
pivot_longer(starts_with("auflage"), names_to = c("jahr", "quartal"), values_to = "auflage",
names_prefix = "auflage_", names_sep = "_", names_transform = list(jahr = as.integer))
auflagen_long2## # A tibble: 27 x 4
## medium jahr quartal auflage
## <chr> <int> <chr> <dbl>
## 1 Der Spiegel 2018 q1 708077
## 2 Der Spiegel 2018 q2 704656
## 3 Der Spiegel 2018 q3 716663
## 4 Der Spiegel 2018 q4 712268
## 5 Der Spiegel 2019 q1 701337
## 6 Der Spiegel 2019 q2 707459
## 7 Der Spiegel 2019 q3 719326
## 8 Der Spiegel 2019 q4 691451
## 9 Der Spiegel 2020 q1 685799
## 10 Stern 2018 q1 539191
## # ... with 17 more rowspivot_longer() bietet also eine mächtige, wenn auch anfangs nicht immer intuitive Syntax zum umformen von Datensätzen – aber auch ohne die Kenntnis aller Zusatzargumente hätten wir einen tranformierten Datensatz, den wir mit wenigen zusätzlichen Funktionen wie select() oder rename() sowie mutate() in unsere gewünschte Form bringen können.
10.1.3 Long Data in Wide Data transformieren mit pivot_wider()
Umgekehrt können wir natürlich auch Wide Data in Long Data transformieren. Die passende Funktion heißt pivot_wider(). Hier geben wir mittels names_from an, die Werte welcher Variablen in Spalten überführt werden sollen, und definieren mit values_from, welche Variable die Werte für die neu erzeugten Spalten liefert.
Nehmen wir Long-Variante unseres jährlichen Auflagen-Datensatzes:
## # A tibble: 9 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Der Spiegel 2018 708077
## 2 Der Spiegel 2019 701337
## 3 Der Spiegel 2020 685799
## 4 Stern 2018 539191
## 5 Stern 2019 476097
## 6 Stern 2020 422156
## 7 Focus 2018 425737
## 8 Focus 2019 373847
## 9 Focus 2020 328587Um diesen in den ursprünglichen Wide-Datensatz zu transformieren, wollen wir also die Werte in jahr in Spaltennamen umwandeln und die Werte in auflage auf diese neuen Spalten verteilen. Wir geben also names_from = "jahr" und values_from = "auflage" an:
## # A tibble: 3 x 4
## medium `2018` `2019` `2020`
## <chr> <dbl> <dbl> <dbl>
## 1 Der Spiegel 708077 701337 685799
## 2 Stern 539191 476097 422156
## 3 Focus 425737 373847 328587Da Spaltennamen, die nicht mit Buchstaben beginnen, suboptimal sind und wir nun nicht mehr direkt sehen können, dass es sich um Auflagen-Daten handelt, können und sollten wir mittels names_prefix auch wieder die ursprünglichen Spaltennamen herstellen21:
## # A tibble: 3 x 4
## medium auflage_2018 auflage_2019 auflage_2020
## <chr> <dbl> <dbl> <dbl>
## 1 Der Spiegel 708077 701337 685799
## 2 Stern 539191 476097 422156
## 3 Focus 425737 373847 328587Im zweiten Fall stehen in mehreren Spalten Informationen, die wir in Spaltennamen überführen wollen, nämlich jahr und quartal:
## # A tibble: 27 x 4
## medium jahr quartal auflage
## <chr> <int> <chr> <dbl>
## 1 Der Spiegel 2018 q1 708077
## 2 Der Spiegel 2018 q2 704656
## 3 Der Spiegel 2018 q3 716663
## 4 Der Spiegel 2018 q4 712268
## 5 Der Spiegel 2019 q1 701337
## 6 Der Spiegel 2019 q2 707459
## 7 Der Spiegel 2019 q3 719326
## 8 Der Spiegel 2019 q4 691451
## 9 Der Spiegel 2020 q1 685799
## 10 Stern 2018 q1 539191
## # ... with 17 more rowsZum Transformieren nutzen wir erneut names_from, nur geben dieses Mal beide Variablen an, die für die Spaltennamen kombiniert werden sollen, und übergeben mit names_sep wieder ein Trennzeichen22:
auflagen_long2 %>%
pivot_wider(names_from = c(jahr, quartal), values_from = auflage, names_prefix = "auflage_", names_sep = "_")## # A tibble: 3 x 10
## medium auflage_2018_q1 auflage_2018_q2 auflage_2018_q3 auflage_2018_q4 auflage_2019_q1 auflage_2019_q2 auflage_2019_q3 auflage_2019_q4 auflage_2020_q1
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Der Spiegel 708077 704656 716663 712268 701337 707459 719326 691451 685799
## 2 Stern 539191 528860 514889 480739 476097 464489 466019 440284 422156
## 3 Focus 425737 417759 412165 413276 373847 367101 364254 349944 32858710.1.4 Spalten aufteilen mit separate()
Schauen wir uns dieselben Daten nochmals in einer anderen Darstellung an. Mehrere Werte in einer Spalte – gruselig, aber kommt vor:
auflagen_gruselig <- tibble(
medium = c("Der Spiegel", "Stern", "Focus"),
auflagen_18_bis_20 = c("708077;701337;685799",
"539191;476097;422156",
"425737;373847;328587"),
)
auflagen_gruselig## # A tibble: 3 x 2
## medium auflagen_18_bis_20
## <chr> <chr>
## 1 Der Spiegel 708077;701337;685799
## 2 Stern 539191;476097;422156
## 3 Focus 425737;373847;328587Hier hilft uns die Funktion separate(), mit der wir mit dem Argument col eine Spalte zum Trennen, mit dem Argument into einen Vektor mit neuen Spaltennamen und dem Argument sep ein Trennzeichen, an dem die bisherigen Werte getrennt werden sollen, mitteilen.
In unserem Fall möchten wir die Spalte auflagen_18_bis_20 am Semikolon ; aufteilen:
auflagen_gruselig %>%
separate(col = auflagen_18_bis_20, into = c("auflage_2018", "auflage_2019", "auflage_2020"), sep = ";")## # A tibble: 3 x 4
## medium auflage_2018 auflage_2019 auflage_2020
## <chr> <chr> <chr> <chr>
## 1 Der Spiegel 708077 701337 685799
## 2 Stern 539191 476097 422156
## 3 Focus 425737 373847 328587Das hat schon gut funktioniert, nur ist der Objekttyp nun jeweils character. Mit dem zusätzlichen Argument convert = TRUE versucht separate(), den neuen Spalten direkt einen passenderen numerischen Objekttyp zu geben:
auflagen_gruselig %>%
separate(col = auflagen_18_bis_20, into = c("auflage_2018", "auflage_2019", "auflage_2020"), sep = ";",
convert = TRUE)## # A tibble: 3 x 4
## medium auflage_2018 auflage_2019 auflage_2020
## <chr> <int> <int> <int>
## 1 Der Spiegel 708077 701337 685799
## 2 Stern 539191 476097 422156
## 3 Focus 425737 373847 32858710.2 Daten zusammenfügen
In größeren Forschungsprojekten haben wir oftmals nicht nur einen Datensatz, sondern mehrere Datensätze – etwa weil verschiedene Teildatensätze getrennt erhoben wurden oder weil diese auf unterschiedlichen Ebenen liegen. Für weitere Analysen sollen diese nun zusammengefügt werden.
10.2.1 Teildatensätze zusammenfügen mit bind_rows() und bind_cols()
Beginnen wir mit dem einfachsten Fall, dem Zusammenfügen von gleichförmigen Datensätzen: wir wollen einem bestehenden Datensatz also entweder neue Zeilen (= neue Fälle) oder neue Spalten (=neue Variablen hinzufügen). Hierfür bietet das Tidyverse die Funktionen bind_rows() und bind_cols(). Bei beiden werden als Argumente einfach die zu verbindenden Datensätze angegeben.
Nehmen wir einmal an, wir hätten die Auflagendaten aus den vorherigen Beispielen getrennt für die Nachrichtenmedien erhoben:
# Aufteilen des Datensatzes
auflagen_spiegel <- auflagen_long %>%
filter(medium == "Der Spiegel")
auflagen_stern <- auflagen_long %>%
filter(medium == "Stern")
auflagen_focus <- auflagen_long %>%
filter(medium == "Focus")Vor uns liegen nun also drei Datensätze, die jeweils Daten eines Nachrichtenmediums enthalten:
## # A tibble: 3 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Der Spiegel 2018 708077
## 2 Der Spiegel 2019 701337
## 3 Der Spiegel 2020 685799## # A tibble: 3 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Stern 2018 539191
## 2 Stern 2019 476097
## 3 Stern 2020 422156## # A tibble: 3 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Focus 2018 425737
## 2 Focus 2019 373847
## 3 Focus 2020 328587Praktischerweise sind die Datensätze gleichförmig: alle drei haben dieselben Variablen. Wir können diese also einfach mit bind_rows() untereinander kleben, um einen gemeinsamen Datensatz zu erstellen:
## # A tibble: 9 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Der Spiegel 2018 708077
## 2 Der Spiegel 2019 701337
## 3 Der Spiegel 2020 685799
## 4 Stern 2018 539191
## 5 Stern 2019 476097
## 6 Stern 2020 422156
## 7 Focus 2018 425737
## 8 Focus 2019 373847
## 9 Focus 2020 328587Fehlende Variablen in einem Teildatensatz werden automatisch durch fehlende Werte ersetzt:
auflagen_spiegel %>%
select(-auflage) %>% # Wir entfernen zu Demonstrationszwecken die Auflage-Spalte
bind_rows(auflagen_stern, auflagen_focus)## # A tibble: 9 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Der Spiegel 2018 NA
## 2 Der Spiegel 2019 NA
## 3 Der Spiegel 2020 NA
## 4 Stern 2018 539191
## 5 Stern 2019 476097
## 6 Stern 2020 422156
## 7 Focus 2018 425737
## 8 Focus 2019 373847
## 9 Focus 2020 328587Hier haben wir den Vorteil, dass die Identifikationsvariable medium bereits vorhanden ist, sodass wir die Auflagendaten auch im vollständigen Datensatz problemlos zuordnen können. Was, wenn das nicht der Fall ist?
# Wir entfernen zu Demonstrationszwecken die Medium-Variable
auflagen_spiegel <- select(auflagen_spiegel, -medium)
auflagen_stern <- select(auflagen_stern, -medium)
auflagen_focus <- select(auflagen_focus, -medium)
auflagen_spiegel## # A tibble: 3 x 2
## jahr auflage
## <int> <dbl>
## 1 2018 708077
## 2 2019 701337
## 3 2020 685799Zwar können wir die Teildatensätze nun anhand des Namens gut identifizieren, bekommen aber bei einem Gesamtdatensatz Probleme, die Auflagenzahlen zuzuordnen:
## # A tibble: 9 x 2
## jahr auflage
## <int> <dbl>
## 1 2018 708077
## 2 2019 701337
## 3 2020 685799
## 4 2018 539191
## 5 2019 476097
## 6 2020 422156
## 7 2018 425737
## 8 2019 373847
## 9 2020 328587In solchen Fällen können wir die Datensätze beim “zusammenkleben” benennen und über das .id-Argument einen Spaltennamen für die Identifikationsvariable festlegen:
bind_rows("Der Spiegel" = auflagen_spiegel,
"Stern" = auflagen_stern,
"Focus" = auflagen_focus,
.id = "medium")## # A tibble: 9 x 3
## medium jahr auflage
## <chr> <int> <dbl>
## 1 Der Spiegel 2018 708077
## 2 Der Spiegel 2019 701337
## 3 Der Spiegel 2020 685799
## 4 Stern 2018 539191
## 5 Stern 2019 476097
## 6 Stern 2020 422156
## 7 Focus 2018 425737
## 8 Focus 2019 373847
## 9 Focus 2020 328587Ganz ähnlich funktioniert auch bind_cols(), nur dass wir dieses Mal Variablen, also Spalten hinzufügen. Erinnern wir uns an den Wide-Datensatz der Auflagendaten:
## # A tibble: 3 x 4
## medium auflage_2018 auflage_2019 auflage_2020
## <chr> <dbl> <dbl> <dbl>
## 1 Der Spiegel 708077 701337 685799
## 2 Stern 539191 476097 422156
## 3 Focus 425737 373847 328587Nun haben wir auch noch ein paar ältere Auflagendaten erhoben:
auflagen_alt <- tibble(
medium = c("Der Spiegel", "Stern", "Focus"),
auflage_2016 = c(793087, 719290, 474285),
auflage_2017 = c(771066, 595729, 456020)
)
auflagen_alt## # A tibble: 3 x 3
## medium auflage_2016 auflage_2017
## <chr> <dbl> <dbl>
## 1 Der Spiegel 793087 771066
## 2 Stern 719290 595729
## 3 Focus 474285 456020Wir ergänzen diese Variablen mittels bind_cols():
## New names:
## * medium -> medium...1
## * medium -> medium...5## # A tibble: 3 x 7
## medium...1 auflage_2018 auflage_2019 auflage_2020 medium...5 auflage_2016 auflage_2017
## <chr> <dbl> <dbl> <dbl> <chr> <dbl> <dbl>
## 1 Der Spiegel 708077 701337 685799 Der Spiegel 793087 771066
## 2 Stern 539191 476097 422156 Stern 719290 595729
## 3 Focus 425737 373847 328587 Focus 474285 456020Wie wir sehen, hat das ganz gut funktioniert – die Mediumspalte ist nun doppelt vorhanden, aber mittels select() könnten wir diese schnell ausschließen. Generell ist aber bis auf in Ausnahmefällen vom Hinzufügen von Variablen mittel bind_cols() abzuraten, da die Prozedur sehr fehleranfällig ist – sind beide Datensätze nicht gleich sortiert, haben Sie schnell Daten miteinander verbunden, die gar nicht zusammengehören. Ist eine Identifikations-Variable vorhanden, so gibt es deutlich sinnvollere und sicherere Funktionen:
10.2.2 Relationale Daten zusammenführen
Nehmen wir an, dass wir ein Forschungsprojekt zu politischen Wochenmagazinen durchführen. Wir haben dazu neben den bereits bekannten Auflagenzahlen auch noch Daten zu den Magazinen selbst und einige Artikel erhoben und diese in den Datensätzen auflagen, info und artikel abgelegt.
Der Datensatz auflagen enthält die uns bereits bekannten Auflagenzahlen auf Ebene Medium pro Quartal und Jahr:
# Falls Sie direkt mitarbeiten möchten:
# Kopieren Sie bei den folgenden drei Code-Blöcken den Code
# um die Beispieldatensätze zu erstellen
auflagen <- auflagen_long2
auflagen## # A tibble: 27 x 4
## medium jahr quartal auflage
## <chr> <int> <chr> <dbl>
## 1 Der Spiegel 2018 q1 708077
## 2 Der Spiegel 2018 q2 704656
## 3 Der Spiegel 2018 q3 716663
## 4 Der Spiegel 2018 q4 712268
## 5 Der Spiegel 2019 q1 701337
## 6 Der Spiegel 2019 q2 707459
## 7 Der Spiegel 2019 q3 719326
## 8 Der Spiegel 2019 q4 691451
## 9 Der Spiegel 2020 q1 685799
## 10 Stern 2018 q1 539191
## # ... with 17 more rowsDer Datensatz info enthält allgemeine Informationen zu den Magazinen auf Mediums-Ebene:
info <- tibble(
medium = c("Der Spiegel", "Stern", "Focus"),
sitz = c("Hamburg", "Hamburg", "Berlin"),
erscheinungstag = c("Samstag", "Donnerstag", "Samstag"),
erstausgabe = lubridate::dmy(c("04-01-1947", "01-08-1948", "18-01-1993"))
)
info## # A tibble: 3 x 4
## medium sitz erscheinungstag erstausgabe
## <chr> <chr> <chr> <date>
## 1 Der Spiegel Hamburg Samstag 1947-01-04
## 2 Stern Hamburg Donnerstag 1948-08-01
## 3 Focus Berlin Samstag 1993-01-18Der Datensatz artikel schließlich enthält alle für das Forschungsprojekt relevanten Artikel auf Artikel-Ebene:
artikel <- tibble(
titel = c("Ein spannender Artikel", "Noch ein Artikel", "Und noch ein Artikel", "Und noch einer"),
autor_innen = c("Max Mustermann", "Erika Musterfrau", "John Doe; Jane Doe", "Mario Rossi"),
medium = c("Stern", "Stern", "Der Spiegel", "Focus"),
ausgabe = c(1L, 1L, 1L, 1L),
jahr = c(2020L, 2020L, 2020L, 2020L),
seiten = c("20-22", "11", "17-25", "104-106")
)
artikel## # A tibble: 4 x 6
## titel autor_innen medium ausgabe jahr seiten
## <chr> <chr> <chr> <int> <int> <chr>
## 1 Ein spannender Artikel Max Mustermann Stern 1 2020 20-22
## 2 Noch ein Artikel Erika Musterfrau Stern 1 2020 11
## 3 Und noch ein Artikel John Doe; Jane Doe Der Spiegel 1 2020 17-25
## 4 Und noch einer Mario Rossi Focus 1 2020 104-106Insgesamt haben wir also drei verschiedene Datensätze, deren Fälle allesamt auf unterschiedlichen Ebenen liegen. Allerdings hängen die Datensätze auch implizit zusammen: die Variable medium findet sich in allen drei Datensätzen; über sie können wir zwei oder alle drei Datensätze zusammenführen. Datensätze, die über Variablen miteinander verbunden werden können, nennt man relationale Daten, sie stehen also in einer Beziehung zueinander.
10.2.2.1 Schlüsselvariablen (Keys)
Variablen, über die Datensätze zusammengefügt werden, bezeichnet man als Schlüsselvariablen, im Englischen auch Keys. Schlüsselvariablen identifzieren Fälle in einem Datensatz eindeutig. Je nach Datenaufbereitung kann hierzu eine einzige Variable ausreichen; manchmal sind aber auch Kombinationen aus mehreren Variablen nötig, um Fälle eindeutig zuzuordnen.
Zudem werden zwei Typen von Schlüsselvariablen unterschieden:
- als primary key wird eine Variable bzw. eine Kombination von Variablen bezeichnet, die im aktuellen Datensatz einen Fall eindeutig identifizieren.
- als foreign key wird eine Variable bzw. eine Kombination von Variablen bezeichnet, die in einem anderen Datensatz Fälle eindeutig identifizieren.
Im Beispieldatensatz info ist medium der primary key. Jeder Wert von medium kommt exakt einmal vor und identifiziert einen Fall (= eine Zeile) somit eindeutig:
## # A tibble: 3 x 4
## medium sitz erscheinungstag erstausgabe
## <chr> <chr> <chr> <date>
## 1 Der Spiegel Hamburg Samstag 1947-01-04
## 2 Stern Hamburg Donnerstag 1948-08-01
## 3 Focus Berlin Samstag 1993-01-18Wie sieht die Sache im Datensatz artikel aus:
## # A tibble: 4 x 6
## titel autor_innen medium ausgabe jahr seiten
## <chr> <chr> <chr> <int> <int> <chr>
## 1 Ein spannender Artikel Max Mustermann Stern 1 2020 20-22
## 2 Noch ein Artikel Erika Musterfrau Stern 1 2020 11
## 3 Und noch ein Artikel John Doe; Jane Doe Der Spiegel 1 2020 17-25
## 4 Und noch einer Mario Rossi Focus 1 2020 104-106Hier wäre titel der naheliegendste primary key, da darüber alle Fälle eindeutig identifiziert werden können. medium taugt hier hingegen nicht als primary key, da der Wert "Stern" nicht eindeutig ist – es gibt zwei Artikel vom Stern im Datensatz, entsprechend reicht die bloße Angabe "Stern" nicht aus, um einen Fall in artikel eindeutig
zu identifizieren.
medium kann hier aber als foreign key für den Datensatz info dienen, da die Werte aus medium im Datensatz artikel sich eindeutig Fällen im Datensatz info zuordnen lassen – jeder der drei einzigartigen Werte von medium im Datensatz artikel ("Stern", "Der Spiegel" & "Focus") kommt nur einmal in der Variable medium im Datensatz info vor.
Da medium in info primary key und in artikel foreign key ist, bilden diese beiden Datensätze über die Schlüsselvariablen medium eine Relation. Wir können nun Daten aus dem Datensatz info dem Datensatz artikel zuordnen und beispielsweise für jeden Artikel in artikel die Variable erscheinungstag hinzufügen; für alle Spiegel-Artikel erhalten wir dann den Wert "Samstag", für alle Stern-Artikel den Wert "Donnerstag", usw.
Bevor wir nun die Datensätze tatsächlich miteinander verbinden, schauen wir uns noch einen komplizierteren Fall an. Was ist der primary key im Datensatz auflagen:
## # A tibble: 27 x 4
## medium jahr quartal auflage
## <chr> <int> <chr> <dbl>
## 1 Der Spiegel 2018 q1 708077
## 2 Der Spiegel 2018 q2 704656
## 3 Der Spiegel 2018 q3 716663
## 4 Der Spiegel 2018 q4 712268
## 5 Der Spiegel 2019 q1 701337
## 6 Der Spiegel 2019 q2 707459
## 7 Der Spiegel 2019 q3 719326
## 8 Der Spiegel 2019 q4 691451
## 9 Der Spiegel 2020 q1 685799
## 10 Stern 2018 q1 539191
## # ... with 17 more rowsAktuell existiert keine einzelne Identifikationsvariable, mit der wir einen Eintrag in auflagen eindeutig identifzieren können – sowohl medium als auch jahr und quartal enthalten doppelte Werte.23 Der primary key ist in diesem Fall eine Kombination aus den drei Variablen medium, jahr und quartal, da jede der Merkmalskombinationen dieser drei Variablen nur ein einziges Mal auftritt.
In solchen Fällen ist es sinnvoll, über eine laufende Nummer eine Variable zu erstellen, die als primary key fungieren kann. Dies können wir schnell mit den uns bereits bekannten Funktionen umsetzen:
## # A tibble: 27 x 5
## medium jahr quartal auflage id
## <chr> <int> <chr> <dbl> <int>
## 1 Der Spiegel 2018 q1 708077 1
## 2 Der Spiegel 2018 q2 704656 2
## 3 Der Spiegel 2018 q3 716663 3
## 4 Der Spiegel 2018 q4 712268 4
## 5 Der Spiegel 2019 q1 701337 5
## 6 Der Spiegel 2019 q2 707459 6
## 7 Der Spiegel 2019 q3 719326 7
## 8 Der Spiegel 2019 q4 691451 8
## 9 Der Spiegel 2020 q1 685799 9
## 10 Stern 2018 q1 539191 10
## # ... with 17 more rows10.2.2.2 Join-Operationen
Um Datensätze nun miteinander zu verbinden, greifen wir auf _join-Funktionen zurück. Das Tidyverse orientiert sich dabei an Konzepten und Bezeichnungen, die auch in der Datenbanksprache SQL verwendet werden. Dabei werden zum Zusammenfügen vier Arten von Joins unterschieden (im Folgenden gehen wir davon aus, dass die beiden Datensätze, die wir verbinden möchten, x und y heißen):
inner_join(): Alle Zeilen, die sowohl inxals auch inyvorkommen, und alle Spalten ausxundyleft_join(): Alle Zeilen, die inxvorkommen und alle Spalten ausxundyright_join(): Alle Zeilen, die inyvorkommen und alle Spalten ausxundyfull_join(): Alle Zeilen ausxundyund alle Spalten ausxundy
Man kann diese Unterschiede auch durch ein Venn-Diagramm verdeutlichen:
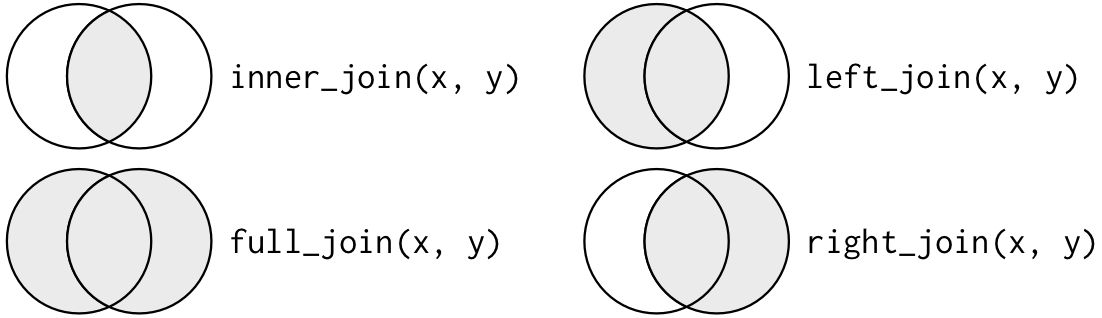
Join-Operationen. Quelle: R for Data Science
Daneben gibt es noch Join-Operationen, die weniger zum Zusammenfügen als zum Filtern von Datensätzen auf Basis von anderen Datensätzen zu gebrauchen sind, da dabei keine Spalten aus einem Datensatz in den anderen kopiert werden:
anti_join(): Alle Zeilen inx, die nicht auch inyvorkommen, und alle Spalten ausx(nicht aber ausy)semi_join(): Alle Zeilen inx, die auch inyvorkommen, und alle Spalten ausx(nicht aber ausy)
In der Praxis benötigen wir vor allem left_join() und inner_join(), anti_join() wird uns aber bei der automatisierten Inhaltsanalyse wieder begegnen.
Alle _join()-Funktionen benötigen als Argumente die beiden Datensätze, die zusammengefügt (bzw. gefiltert) werden sollen. Wird kein weiteres Argument angegeben, werden automatisch alle gleichnamigen Variablen in beiden Datensätzen als Schlüsselvariablen verwendet.
## Joining, by = "medium"## # A tibble: 4 x 9
## titel autor_innen medium ausgabe jahr seiten sitz erscheinungstag erstausgabe
## <chr> <chr> <chr> <int> <int> <chr> <chr> <chr> <date>
## 1 Ein spannender Artikel Max Mustermann Stern 1 2020 20-22 Hamburg Donnerstag 1948-08-01
## 2 Noch ein Artikel Erika Musterfrau Stern 1 2020 11 Hamburg Donnerstag 1948-08-01
## 3 Und noch ein Artikel John Doe; Jane Doe Der Spiegel 1 2020 17-25 Hamburg Samstag 1947-01-04
## 4 Und noch einer Mario Rossi Focus 1 2020 104-106 Berlin Samstag 1993-01-18Wir sehen zunächst in der Konsole, dass die Join-Operation auf der Basis der Schlüsselvariable medium erfolgte, da diese als einzige in beiden Datensätzen zu finden ist. Das Ergebnis ist ein Datensatz, der alle Variablen aus beiden Datensätzen enthält und diese auf Basis der Schlüsselvariablen alle Spaltenwerte den richtigen Zeilen zugeordnet hat.
Mit dem Argument by können wir die Schlüsselvariable(n) auch explizit angeben; dies ist vor allem dann praktisch, wenn die Schlüsselvariablen in beiden Datensätzen unterschiedlich heißen. Auch bei gleichnamigen Variablen ist aber dennoch sinnvoll, die Schlüsselvariablen explizit zu nennen, um etwaigen Problemen vorzubeugen:
## # A tibble: 4 x 9
## titel autor_innen medium ausgabe jahr seiten sitz erscheinungstag erstausgabe
## <chr> <chr> <chr> <int> <int> <chr> <chr> <chr> <date>
## 1 Ein spannender Artikel Max Mustermann Stern 1 2020 20-22 Hamburg Donnerstag 1948-08-01
## 2 Noch ein Artikel Erika Musterfrau Stern 1 2020 11 Hamburg Donnerstag 1948-08-01
## 3 Und noch ein Artikel John Doe; Jane Doe Der Spiegel 1 2020 17-25 Hamburg Samstag 1947-01-04
## 4 Und noch einer Mario Rossi Focus 1 2020 104-106 Berlin Samstag 1993-01-18Zu beachten ist, dass left_join() auch Ergebnisse liefert, wenn die Schlüsselvariablen in y nicht eindeutig ist bzw. sind. Dies ist der Fall, wenn wir beide Datensätze im obigen Beispiel vertauschen. Zur Erinnerung, medium im artikel-Datensatz enthält Mehrfachwerte:
## # A tibble: 4 x 6
## titel autor_innen medium ausgabe jahr seiten
## <chr> <chr> <chr> <int> <int> <chr>
## 1 Ein spannender Artikel Max Mustermann Stern 1 2020 20-22
## 2 Noch ein Artikel Erika Musterfrau Stern 1 2020 11
## 3 Und noch ein Artikel John Doe; Jane Doe Der Spiegel 1 2020 17-25
## 4 Und noch einer Mario Rossi Focus 1 2020 104-106Welche der beiden "Stern"-Zeilen ordnet left_join() nun info zu?
## # A tibble: 4 x 9
## medium sitz erscheinungstag erstausgabe titel autor_innen ausgabe jahr seiten
## <chr> <chr> <chr> <date> <chr> <chr> <int> <int> <chr>
## 1 Der Spiegel Hamburg Samstag 1947-01-04 Und noch ein Artikel John Doe; Jane Doe 1 2020 17-25
## 2 Stern Hamburg Donnerstag 1948-08-01 Ein spannender Artikel Max Mustermann 1 2020 20-22
## 3 Stern Hamburg Donnerstag 1948-08-01 Noch ein Artikel Erika Musterfrau 1 2020 11
## 4 Focus Berlin Samstag 1993-01-18 Und noch einer Mario Rossi 1 2020 104-106Die Antwort: beide Zeilen – left_join() dupliziert also Zeilen in x, wenn es auf Basis der Schlüsselvariablen mehrere Entsprechungen in y gibt. Daher ist es nach einer Join-Operation immer sinnvoll zu prüfen, ob der resultierende Datensatz auch den gewünschten Umfang hat.
10.3 Übungsaufgaben
Erstellen Sie für die folgenden Übungsaufgaben eine eigene Skriptdatei oder eine R-Markdown-Datei und speichern diese als ue10_nachname.R bzw. ue10_nachname.Rmd ab.
Laden Sie den Datensatz facebook_europawahl.csv, der schon aus den vorigen Übungen bekannt ist. Wählen Sie zunächst die Variablen id, party, timestamp sowie comments_count, shares_count und reactions_count aus.
Wir möchten diesen Datensatz nun auf eine “Medium-pro-Tag-und-Facebook-Metrik”-Ebene umstrukturieren, sodass für jeden Post (zu erkennen an der Variablen id) drei Zeilen existieren, in der jeweils einmal die Anzahl der Kommentare, die Anzahl der Shares sowie die Anzahl der Reactions steht.
Der resultierende Datensatz sollte fünf Variablen haben: die Identifikationsvariablen id, party, timestamp (aus dem alten Datensatz) und metric (gibt an, um welche der drei Facebook-Metriken es sich handelt) sowie die Wertvariable value (die Anzahl der jeweiligen Facebook-Metrik).
Auf Moodle finden Sie den Datensatz facebook_codings.csv. Dieser enthält für die Facebook-Posts aus facebook_europawahl.csv manuelle Codierungen, ob bestimmte Themen in diesen Posts vorkommen oder nicht. Zudem ist die id-Variable der Facebook-Posts angegeben.
Laden Sie auch diesen Datensatz in R und fügen ihn mit facebook_europawahl zusammen, sodass für jeden Post neben den API-Informationen (timestamp, message, Facebook-Metriken etc.) auch die manuellen Codierungen ersichtlich sind.
Da alle Spalten bis auf
mediumtransformiert werden sollen, könnten wir alternativ auch mit-mediumangeben, um alle Spalten außer eben dieser zu nutzen.↩︎Noch schöner wäre es, wenn wir automatisch auch noch das
qausq1,q2etc. entfernen und das Quartal auch alsintegerspeichern könnten, aber die nötigen Funktionen zum Umgang mit Textvariablen lernen wir erst in zwei Wochen. Möglich ist es aber.↩︎Im Gegensatz zu
pivot_longerentferntnames_prefixbeipivot_widerdas Prefix nicht, sondern fügt dieses hinzu.↩︎Tatsächlich wäre der Unterstrich
_aber auch bereits als Default-Wert vonnames_sepeingestellt.↩︎Tatsächlich sind die Werte in
auflageaktuell noch eindeutig, aber Wertvariablen sind nicht gut als Schlüsselvariablen geeignet; sammeln wir noch mehr Auflagendaten aus früheren Jahren und von mehr Magazinen, steigt die Wahrscheinlichkeit, dass irgendwann auch mal ein doppelter Wert auftaucht.↩︎