Documento 10 Iris dataset
1 - Cargamos los paquetes necesarios, además del dataset Iris.
A continuación, mostramos las 10 primeras filas del dataset a modo de ejemplo de visualización inicial:
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|
| 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| 5.4 | 3.9 | 1.7 | 0.4 | setosa |
| 4.6 | 3.4 | 1.4 | 0.3 | setosa |
| 5.0 | 3.4 | 1.5 | 0.2 | setosa |
| 4.4 | 2.9 | 1.4 | 0.2 | setosa |
| 4.9 | 3.1 | 1.5 | 0.1 | setosa |
2 - Utilizar las funciones que se han visto para analizar el dataset, ver número de transacciones, items del dataset, información estadística del dataset. NOTA: dataset debe ser preprocesado.
- Con el comando str vemos la estructura de nuestro dataset, además del número de variables, tipo de dichas variables y sus primeros valores.
NOTA: Información antes del preprocesado.
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...Aquí vamos a comprobar la dimensión de nuestro dataset y, posteriormente, ver si se encuentra algún NA para eliminarlo del dataset.
[1] 150 5[1] 0Con el comando summary obtenemos la información estadística pedida: mínimos, máximos, medias, etc de las variables de nuestro dataset.
Sepal.Length Sepal.Width Petal.Length Petal.Width
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
Median :5.800 Median :3.000 Median :4.350 Median :1.300
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
Species
setosa :50
versicolor:50
virginica :50
Discretizar de tres formas, guardando los resultados en tres datasets.
2.a) Transformando el tipo de las variables (convirtiendo a factor).
# Como vemos en str, sólo la variable "Species" es de tipo factor,
# convertimos las demás:
dsIris1 <- iris
dsIris1$Sepal.Length <- ordered(cut(dsIris1$Sepal.Length, c(0, 5.4, 6.3, Inf)), labels = c("SL_Short", "SL_Medium", "SL_Large"))
#b1 <- c(4.3, 5.4, 6.3, 7.9)
#dsIris1$Sepal.Length <- cut(iris$Sepal.Length, breaks = b1, include.lowest = TRUE)
dsIris1$Sepal.Width <- ordered(cut(dsIris1$Sepal.Width, c(0, 2.9, 3.2, Inf)), labels = c("SW_Short", "SW_Medium", "SW_Large"))
#b2 <- c(2, 2.9, 3.2, 4.4)
#dsIris1$Sepal.Width <- cut(iris$Sepal.Width, breaks = b2, include.lowest = TRUE)
dsIris1$Petal.Length <- ordered(cut(dsIris1$Petal.Length, c(0, 2.63, 4.9, Inf)), labels = c("PL_Short", "PL_Medium", "PL_Large"))
#b3 <- c(1, 2.63, 4.9, 6.9)
#dsIris1$Petal.Length <- cut(iris$Petal.Length, breaks = b3, include.lowest = TRUE)
dsIris1$Petal.Width <- ordered(cut(dsIris1$Petal.Width, c(0, 0.867, 1.6, Inf)), labels = c("PW_Short", "PW_Medium", "PW_Large"))
#b4 <- c(0.1, 0.867, 1.6, 2.5)
#dsIris1$Petal.Width <- cut(iris$Petal.Width, breaks = b4, include.lowest = TRUE)A continuación, mostramos cómo ha cambiado el dataset actual (dsIris1) del original mostrando de nuevo las 10 primeras filas:
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|
| SL_Short | SW_Large | PL_Short | PW_Short | setosa |
| SL_Short | SW_Medium | PL_Short | PW_Short | setosa |
| SL_Short | SW_Medium | PL_Short | PW_Short | setosa |
| SL_Short | SW_Medium | PL_Short | PW_Short | setosa |
| SL_Short | SW_Large | PL_Short | PW_Short | setosa |
| SL_Short | SW_Large | PL_Short | PW_Short | setosa |
| SL_Short | SW_Large | PL_Short | PW_Short | setosa |
| SL_Short | SW_Large | PL_Short | PW_Short | setosa |
| SL_Short | SW_Short | PL_Short | PW_Short | setosa |
| SL_Short | SW_Medium | PL_Short | PW_Short | setosa |
2.b) Discretizando los datos (convirtiendo en categóricos)
Creamos la variable en la que guardaremos el nuevo dataset
Cambiamos el valor de los campos del nuevo dataset:
# Sepal.Length
dsIris2$Sepal.Length[dsIris2$Sepal.Length <= 5.4] <- "[4.3,5.4]"
dsIris2$Sepal.Length[dsIris2$Sepal.Length > 5.4
& dsIris2$Sepal.Length <= 6.3] <- "(5.4, 6.3]"
dsIris2$Sepal.Length[dsIris2$Sepal.Length > 5.4] <- "(6.3,7.9]"
# Sepal.Width
dsIris2$Sepal.Width[dsIris2$Sepal.Width <= 2.9] <- "[2, 2.9]"
dsIris2$Sepal.Width[dsIris2$Sepal.Width > 2.9
& dsIris2$Sepal.Width <= 3.2] <- "(2.9, 3.2]"
dsIris2$Sepal.Width[dsIris2$Sepal.Width > 3.2] <- "(3.2, 4.4]"#Petal.Length
dsIris2$Petal.Length[dsIris2$Petal.Length <= 2.63] <- "[1, 2.63]"
dsIris2$Petal.Length[dsIris2$Petal.Length > 2.63
& dsIris2$Petal.Length <= 4.9] <- "(2.63, 4.9]"
dsIris2$Petal.Length[dsIris2$Petal.Length > 4.9] <- "(4.9, 6.9]"
dsIris2$Petal.Width[dsIris2$Petal.Width <= 0.867] <- "[0.1, 0.867]"
dsIris2$Petal.Width[dsIris2$Petal.Width > 0.867
& dsIris2$Petal.Width <= 1.6] <- "(0.867, 1.6]"
dsIris2$Petal.Width[dsIris2$Petal.Width > 1.6] <- "(1.6, 2.5]"Convertimos los valores de tipo character en factor, creando 3 posibles valores (levels) de cada una de las variables.
dsIris2$Sepal.Length <- as.factor(dsIris2$Sepal.Length)
dsIris2$Sepal.Width <- as.factor(dsIris2$Sepal.Width)
dsIris2$Petal.Length <- as.factor(dsIris2$Petal.Length)
dsIris2$Petal.Width <- as.factor(dsIris2$Petal.Width)A continuación, mostramos cómo ha cambiado el dataset actual (dsIris2) del original mostrando de nuevo las 10 primeras filas:
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|
| [4.3,5.4] | (3.2, 4.4] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | (2.9, 3.2] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | (2.9, 3.2] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | (2.9, 3.2] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | (3.2, 4.4] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | (3.2, 4.4] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | (3.2, 4.4] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | (3.2, 4.4] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | [2, 2.9] | [1, 2.63] | [0.1, 0.867] | setosa |
| [4.3,5.4] | (2.9, 3.2] | [1, 2.63] | [0.1, 0.867] | setosa |
2.c) Usando el comando discretize.
Para cada uno de los valores que no son de tipo factor, aplicamos el comando discretize para discretizar dichos valores.
dsIris3 <- iris
dsIris3$Sepal.Length <- discretize(iris$Sepal.Length,
labels = c("SL_short", "SL_medium", "SL_long"),
method = "frequency", breaks = 3)
dsIris3$Sepal.Width <- discretize(iris$Sepal.Width,
labels = c("SW_short", "SW_medium", "SW_long"),
method = "frequency", breaks = 3)
dsIris3$Petal.Length <- discretize(iris$Petal.Length,
labels = c("PL_short", "PL_medium", "PL_long"),
method = "frequency", breaks = 3)
dsIris3$Petal.Width <- discretize(iris$Petal.Width,
labels = c("PW_short", "PW_medium", "PW_long"),
method = "frequency", breaks = 3)A continuación, mostramos cómo ha cambiado el dataset actual (dsIris3) del original mostrando de nuevo las 10 primeras filas:
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|
| SL_short | SW_long | PL_short | PW_short | setosa |
| SL_short | SW_medium | PL_short | PW_short | setosa |
| SL_short | SW_long | PL_short | PW_short | setosa |
| SL_short | SW_medium | PL_short | PW_short | setosa |
| SL_short | SW_long | PL_short | PW_short | setosa |
| SL_medium | SW_long | PL_short | PW_short | setosa |
| SL_short | SW_long | PL_short | PW_short | setosa |
| SL_short | SW_long | PL_short | PW_short | setosa |
| SL_short | SW_medium | PL_short | PW_short | setosa |
| SL_short | SW_medium | PL_short | PW_short | setosa |
Por último, convertimos los datasets a transacciones:
dsIris1 <- as(dsIris1, "transactions")
dsIris2 <- as(dsIris2, "transactions")
dsIris3 <- as(dsIris3, "transactions")
dsIris3## transactions in sparse format with
## 150 transactions (rows) and
## 15 items (columns)Vemos su estructura:
Formal class 'transactions' [package "arules"] with 3 slots
..@ data :Formal class 'ngCMatrix' [package "Matrix"] with 5 slots
.. .. ..@ i : int [1:750] 0 5 6 9 12 0 4 6 9 12 ...
.. .. ..@ p : int [1:151] 0 5 10 15 20 25 30 35 40 45 ...
.. .. ..@ Dim : int [1:2] 15 150
.. .. ..@ Dimnames:List of 2
.. .. .. ..$ : NULL
.. .. .. ..$ : NULL
.. .. ..@ factors : list()
..@ itemInfo :'data.frame': 15 obs. of 3 variables:
.. ..$ labels : chr [1:15] "Sepal.Length=SL_short" "Sepal.Length=SL_medium" "Sepal.Length=SL_long" "Sepal.Width=SW_short" ...
.. ..$ variables: Factor w/ 5 levels "Petal.Length",..: 3 3 3 4 4 4 1 1 1 2 ...
.. ..$ levels : Factor w/ 15 levels "PL_long","PL_medium",..: 10 9 8 13 12 11 3 2 1 6 ...
..@ itemsetInfo:'data.frame': 150 obs. of 1 variable:
.. ..$ transactionID: chr [1:150] "1" "2" "3" "4" ...Información estadística:
transactions as itemMatrix in sparse format with
150 rows (elements/itemsets/transactions) and
15 columns (items) and a density of 0.3333333
most frequent items:
Sepal.Width=SW_long Sepal.Length=SL_medium Petal.Width=PW_long
56 53 52
Sepal.Length=SL_long Petal.Length=PL_long (Other)
51 51 487
element (itemset/transaction) length distribution:
sizes
5
150
Min. 1st Qu. Median Mean 3rd Qu. Max.
5 5 5 5 5 5
includes extended item information - examples:
labels variables levels
1 Sepal.Length=SL_short Sepal.Length SL_short
2 Sepal.Length=SL_medium Sepal.Length SL_medium
3 Sepal.Length=SL_long Sepal.Length SL_long
includes extended transaction information - examples:
transactionID
1 1
2 2
3 3Por último, inspeccionamos las primeras filas (a modo de ejemplo):
items transactionID
[1] {Sepal.Length=SL_short,
Sepal.Width=SW_long,
Petal.Length=PL_short,
Petal.Width=PW_short,
Species=setosa} 1
[2] {Sepal.Length=SL_short,
Sepal.Width=SW_medium,
Petal.Length=PL_short,
Petal.Width=PW_short,
Species=setosa} 23 - Utilizar algoritmo apriori para obtener las reglas de asociación con confianza 0.5 y soporte 0.01. LLamar estas reglas r1.
Apriori
Parameter specification:
confidence minval smax arem aval originalSupport maxtime support minlen
0.5 0.1 1 none FALSE TRUE 5 0.01 1
maxlen target ext
10 rules FALSE
Algorithmic control:
filter tree heap memopt load sort verbose
0.1 TRUE TRUE FALSE TRUE 2 TRUE
Absolute minimum support count: 1
set item appearances ...[0 item(s)] done [0.00s].
set transactions ...[15 item(s), 150 transaction(s)] done [0.00s].
sorting and recoding items ... [15 item(s)] done [0.00s].
creating transaction tree ... done [0.00s].
checking subsets of size 1 2 3 4 5 done [0.00s].
writing ... [532 rule(s)] done [0.00s].
creating S4 object ... done [0.00s].set of 532 rules 4 - Encontrar reglas redundantes en r1 de dos formas distintas. Eliminarlas. Guardar en una variable las redundantes. Buscar en el paquete arules una función que te calcula las reglas redundantes.
Para empezar, definimos reglas redudantes como:
\(X => Y\) es redundante si existe un subset \(X’\) tal que: \(conf(X’ -> Y) >= conf(X -> Y)\)
Apunte: Una regla será redundante si existen reglas mas generales con la misma o mayor confianza, es decir, una regla más especifica es redundante si es igual o incluso menos predictiva que una regla más general.
FORMA 1:
# Guardando las reglas redundantes
idxRed <- which(is.redundant(r1))
vRed <- r1[idxRed]
# Guardando en r1 las reglas sin redundancia
idxNoRed <- which(!is.redundant(r1))
r1 <- r1[idxNoRed]
r1## set of 196 rulesSi no queremos usar índices:
## set of 0 rules## set of 196 rulesFORMA 2:
subset.matrix <- is.subset(r1, r1, sparse = FALSE)
subset.matrix[lower.tri(subset.matrix, diag = TRUE)] <- NA
reglas_redundantes2 <- colSums(subset.matrix, na.rm = TRUE) >= 1
length(which(reglas_redundantes2))## [1] 163## set of 33 rulesCon esta segunda forma obtenemos muchas menos reglas, obteniendo sólo un total de 33. Esta no es una versión muy eficiente de cómo sacar reglas redundantes y no se usa hoy en día.
5 - Generar 3 conjuntos de reglas que cumplan que ciertos valores estén a la izquierda y/o derecha. Llamarlas r2,r3,r4.
r2 <- apriori(dsIris3,
parameter = list(supp = 0.01, conf = 0.50),
appearance = list(default="rhs",lhs="Sepal.Length=SL_medium"))## Apriori
##
## Parameter specification:
## confidence minval smax arem aval originalSupport maxtime support minlen
## 0.5 0.1 1 none FALSE TRUE 5 0.01 1
## maxlen target ext
## 10 rules FALSE
##
## Algorithmic control:
## filter tree heap memopt load sort verbose
## 0.1 TRUE TRUE FALSE TRUE 2 TRUE
##
## Absolute minimum support count: 1
##
## set item appearances ...[1 item(s)] done [0.00s].
## set transactions ...[15 item(s), 150 transaction(s)] done [0.00s].
## sorting and recoding items ... [15 item(s)] done [0.00s].
## creating transaction tree ... done [0.00s].
## checking subsets of size 1 2 done [0.00s].
## writing ... [3 rule(s)] done [0.00s].
## creating S4 object ... done [0.00s].r3 <- apriori(dsIris3,
parameter = list(supp = 0.01, conf = 0.50),
appearance = list(default = "lhs", rhs="Species=versicolor"))## Apriori
##
## Parameter specification:
## confidence minval smax arem aval originalSupport maxtime support minlen
## 0.5 0.1 1 none FALSE TRUE 5 0.01 1
## maxlen target ext
## 10 rules FALSE
##
## Algorithmic control:
## filter tree heap memopt load sort verbose
## 0.1 TRUE TRUE FALSE TRUE 2 TRUE
##
## Absolute minimum support count: 1
##
## set item appearances ...[1 item(s)] done [0.00s].
## set transactions ...[15 item(s), 150 transaction(s)] done [0.00s].
## sorting and recoding items ... [15 item(s)] done [0.00s].
## creating transaction tree ... done [0.00s].
## checking subsets of size 1 2 3 4 5 done [0.00s].
## writing ... [51 rule(s)] done [0.00s].
## creating S4 object ... done [0.00s].r4 <- apriori(dsIris3,
parameter = list(supp = 0.01, conf = 0.50),
appearance = list(rhs="Species=versicolor", lhs="Petal.Width=PW_medium"))## Apriori
##
## Parameter specification:
## confidence minval smax arem aval originalSupport maxtime support minlen
## 0.5 0.1 1 none FALSE TRUE 5 0.01 1
## maxlen target ext
## 10 rules FALSE
##
## Algorithmic control:
## filter tree heap memopt load sort verbose
## 0.1 TRUE TRUE FALSE TRUE 2 TRUE
##
## Absolute minimum support count: 1
##
## set item appearances ...[2 item(s)] done [0.00s].
## set transactions ...[2 item(s), 150 transaction(s)] done [0.00s].
## sorting and recoding items ... [2 item(s)] done [0.00s].
## creating transaction tree ... done [0.00s].
## checking subsets of size 1 2 done [0.00s].
## writing ... [1 rule(s)] done [0.00s].
## creating S4 object ... done [0.00s].6 - Unir r2 y r3 y buscar reglas duplicadas y reglas redundantes.
## set of 54 rules## set of 53 rulesHay una regla repetida, la quitamos:
## set of 18 rules- Otra forma (lapply):
rUnion <- lapply(c(r2,r3), unique)
- Otra forma
rUnion <- union(r2,r3) # Quita las duplicadas directamente rUnion
7 - Hacer la intersección de r2 y r3.
Si queremos hallar la intersección de dos conjuntos de reglas basta con obtener los duplicados.
# Array de bool donde los TRUE son elementos duplicados
regs = c(r2,r3)
duplicados <- duplicated(regs)
# Obtenemos los índices de dichos elementos
elems <- which(duplicados)
# Guardamos en rInterseccion los elementos duplicados
rInterseccion <- regs[elems]
rInterseccion## set of 1 rules8 - Usa subset para inspeccionar las reglas con distintas condiciones.
- 1 - Selecciona todas las reglas con items “Species=setosa” o “Petal.Width=PW_medium” en la parte derecha y lift > 2
rules.sub_2 <- subset(r1, subset = rhs %in% c("Petal.Width=PW_medium","Species=setosa")&lift > 2)
inspect(rules.sub_2)## lhs rhs support confidence lift count
## [1] {Sepal.Length=SL_short} => {Species=setosa} 0.26666667 0.8695652 2.608696 40
## [2] {Petal.Length=PL_medium} => {Petal.Width=PW_medium} 0.28666667 0.8775510 2.742347 43
## [3] {Species=versicolor} => {Petal.Width=PW_medium} 0.30000000 0.9000000 2.812500 45
## [4] {Petal.Length=PL_short} => {Species=setosa} 0.33333333 1.0000000 3.000000 50
## [5] {Petal.Width=PW_short} => {Species=setosa} 0.33333333 1.0000000 3.000000 50
## [6] {Sepal.Width=SW_long} => {Species=setosa} 0.25333333 0.6785714 2.035714 38
## [7] {Sepal.Length=SL_short,
## Sepal.Width=SW_medium} => {Species=setosa} 0.07333333 1.0000000 3.000000 11
## [8] {Sepal.Length=SL_short,
## Sepal.Width=SW_short} => {Petal.Width=PW_medium} 0.03333333 0.7142857 2.232143 5
## [9] {Sepal.Length=SL_short,
## Species=versicolor} => {Petal.Width=PW_medium} 0.03333333 1.0000000 3.125000 5
## [10] {Sepal.Length=SL_short,
## Sepal.Width=SW_long} => {Species=setosa} 0.18666667 1.0000000 3.000000 28
## [11] {Sepal.Width=SW_medium,
## Petal.Length=PL_medium} => {Petal.Width=PW_medium} 0.10666667 0.9411765 2.941176 16
## [12] {Sepal.Width=SW_medium,
## Species=versicolor} => {Petal.Width=PW_medium} 0.11333333 0.9444444 2.951389 17
## [13] {Sepal.Length=SL_medium,
## Sepal.Width=SW_medium} => {Petal.Width=PW_medium} 0.07333333 0.7857143 2.455357 11
## [14] {Sepal.Width=SW_short,
## Petal.Length=PL_medium} => {Petal.Width=PW_medium} 0.16666667 0.9259259 2.893519 25
## [15] {Sepal.Width=SW_short,
## Species=versicolor} => {Petal.Width=PW_medium} 0.17333333 0.9629630 3.009259 26
## [16] {Sepal.Length=SL_medium,
## Sepal.Width=SW_short} => {Petal.Width=PW_medium} 0.12666667 0.7307692 2.283654 19
## [17] {Petal.Length=PL_medium,
## Species=versicolor} => {Petal.Width=PW_medium} 0.28666667 0.9347826 2.921196 43
## [18] {Sepal.Length=SL_long,
## Petal.Length=PL_medium} => {Petal.Width=PW_medium} 0.06666667 0.9090909 2.840909 10
## [19] {Sepal.Length=SL_medium,
## Species=versicolor} => {Petal.Width=PW_medium} 0.18666667 0.9032258 2.822581 28
## [20] {Sepal.Length=SL_medium,
## Sepal.Width=SW_long} => {Species=setosa} 0.06666667 0.7692308 2.307692 10
## [21] {Sepal.Width=SW_medium,
## Petal.Length=PL_medium,
## Species=versicolor} => {Petal.Width=PW_medium} 0.10666667 1.0000000 3.125000 16
## [22] {Sepal.Length=SL_long,
## Sepal.Width=SW_medium,
## Petal.Length=PL_medium} => {Petal.Width=PW_medium} 0.03333333 1.0000000 3.125000 5
## [23] {Sepal.Length=SL_medium,
## Sepal.Width=SW_medium,
## Species=versicolor} => {Petal.Width=PW_medium} 0.07333333 1.0000000 3.125000 11
## [24] {Sepal.Width=SW_short,
## Petal.Length=PL_medium,
## Species=versicolor} => {Petal.Width=PW_medium} 0.16666667 1.0000000 3.125000 25
## [25] {Sepal.Length=SL_long,
## Sepal.Width=SW_short,
## Petal.Length=PL_medium} => {Petal.Width=PW_medium} 0.02000000 1.0000000 3.125000 3
## [26] {Sepal.Length=SL_medium,
## Sepal.Width=SW_short,
## Petal.Length=PL_medium} => {Petal.Width=PW_medium} 0.11333333 0.9444444 2.951389 17
## [27] {Sepal.Length=SL_long,
## Sepal.Width=SW_short,
## Species=versicolor} => {Petal.Width=PW_medium} 0.02666667 1.0000000 3.125000 4- 2 - Selecciona solo las reglas con items “Species=setosa” y “Petal.Width=PW_medium” en la parte izquierda
rules.sub <- subset(r1, subset=lhs %ain% c("Species=versicolor", "Petal.Width=PW_medium"))
inspect(rules.sub)## lhs rhs support confidence lift count
## [1] {Petal.Width=PW_medium,
## Species=versicolor} => {Petal.Length=PL_medium} 0.28666667 0.9555556 2.925170 43
## [2] {Sepal.Width=SW_short,
## Petal.Width=PW_medium,
## Species=versicolor} => {Petal.Length=PL_medium} 0.16666667 0.9615385 2.943485 25
## [3] {Sepal.Length=SL_medium,
## Petal.Width=PW_medium,
## Species=versicolor} => {Petal.Length=PL_medium} 0.18666667 1.0000000 3.061224 28
## [4] {Petal.Length=PL_long,
## Petal.Width=PW_medium,
## Species=versicolor} => {Sepal.Length=SL_long} 0.01333333 1.0000000 2.941176 2- 3 - Usa coincidencia parcial para todos los item que se correspondan con la variable Species
## lhs rhs support confidence lift count
## [1] {Sepal.Length=SL_short} => {Species=setosa} 0.26666667 0.8695652 2.608696 40
## [2] {Sepal.Width=SW_short} => {Species=versicolor} 0.18000000 0.5744681 1.723404 27
## [3] {Petal.Width=PW_medium} => {Species=versicolor} 0.30000000 0.9375000 2.812500 45
## [4] {Petal.Length=PL_medium} => {Species=versicolor} 0.30666667 0.9387755 2.816327 46
## [5] {Petal.Length=PL_short} => {Species=setosa} 0.33333333 1.0000000 3.000000 50
## [6] {Petal.Width=PW_short} => {Species=setosa} 0.33333333 1.0000000 3.000000 50
## [7] {Sepal.Width=SW_long} => {Species=setosa} 0.25333333 0.6785714 2.035714 38
## [8] {Sepal.Length=SL_medium} => {Species=versicolor} 0.20666667 0.5849057 1.754717 31
## [9] {Sepal.Length=SL_long} => {Species=virginica} 0.24666667 0.7254902 2.176471 37
## [10] {Petal.Length=PL_long} => {Species=virginica} 0.31333333 0.9215686 2.764706 47
## [11] {Petal.Width=PW_long} => {Species=virginica} 0.31333333 0.9038462 2.711538 47
## [12] {Sepal.Length=SL_short,
## Sepal.Width=SW_medium} => {Species=setosa} 0.07333333 1.0000000 3.000000 11
## [13] {Sepal.Length=SL_short,
## Sepal.Width=SW_short} => {Species=versicolor} 0.03333333 0.7142857 2.142857 5
## [14] {Sepal.Length=SL_short,
## Petal.Width=PW_medium} => {Species=versicolor} 0.03333333 1.0000000 3.000000 5
## [15] {Sepal.Length=SL_short,
## Sepal.Width=SW_long} => {Species=setosa} 0.18666667 1.0000000 3.000000 28
## [16] {Sepal.Width=SW_medium,
## Petal.Width=PW_medium} => {Species=versicolor} 0.11333333 1.0000000 3.000000 17
## [17] {Sepal.Width=SW_medium,
## Petal.Length=PL_medium} => {Species=versicolor} 0.10666667 0.9411765 2.823529 16
## [18] {Sepal.Length=SL_medium,
## Sepal.Width=SW_medium} => {Species=versicolor} 0.07333333 0.7857143 2.357143 11
## [19] {Sepal.Width=SW_medium,
## Petal.Width=PW_long} => {Species=virginica} 0.12000000 0.9473684 2.842105 18
## [20] {Sepal.Length=SL_medium,
## Sepal.Width=SW_short} => {Species=versicolor} 0.12000000 0.6923077 2.076923 18
## [21] {Sepal.Width=SW_short,
## Petal.Width=PW_long} => {Species=virginica} 0.10666667 0.9411765 2.823529 16
## [22] {Petal.Length=PL_medium,
## Petal.Width=PW_medium} => {Species=versicolor} 0.28666667 1.0000000 3.000000 43
## [23] {Sepal.Width=SW_long,
## Petal.Width=PW_medium} => {Species=versicolor} 0.01333333 1.0000000 3.000000 2
## [24] {Sepal.Length=SL_long,
## Petal.Length=PL_medium} => {Species=versicolor} 0.07333333 1.0000000 3.000000 11
## [25] {Sepal.Width=SW_long,
## Petal.Length=PL_medium} => {Species=versicolor} 0.03333333 1.0000000 3.000000 5
## [26] {Sepal.Length=SL_medium,
## Sepal.Width=SW_long} => {Species=setosa} 0.06666667 0.7692308 2.307692 10
## [27] {Sepal.Length=SL_long,
## Petal.Length=PL_long} => {Species=virginica} 0.24666667 0.9250000 2.775000 37
## [28] {Sepal.Length=SL_long,
## Petal.Width=PW_long} => {Species=virginica} 0.24000000 0.9473684 2.842105 36
## [29] {Sepal.Length=SL_long,
## Sepal.Width=SW_long} => {Species=virginica} 0.08000000 0.8000000 2.400000 12
## [30] {Petal.Length=PL_long,
## Petal.Width=PW_long} => {Species=virginica} 0.29333333 0.9565217 2.869565 44
## [31] {Sepal.Width=SW_long,
## Petal.Length=PL_long} => {Species=virginica} 0.08666667 1.0000000 3.000000 13
## [32] {Sepal.Length=SL_medium,
## Sepal.Width=SW_medium,
## Petal.Length=PL_long} => {Species=virginica} 0.01333333 1.0000000 3.000000 2
## [33] {Sepal.Length=SL_medium,
## Sepal.Width=SW_medium,
## Petal.Width=PW_long} => {Species=virginica} 0.02000000 1.0000000 3.000000 3
## [34] {Sepal.Length=SL_medium,
## Sepal.Width=SW_short,
## Petal.Length=PL_medium} => {Species=versicolor} 0.11333333 0.9444444 2.833333 17
## [35] {Sepal.Width=SW_short,
## Petal.Length=PL_medium,
## Petal.Width=PW_long} => {Species=virginica} 0.01333333 1.0000000 3.000000 2
## [36] {Sepal.Length=SL_long,
## Sepal.Width=SW_short,
## Petal.Width=PW_long} => {Species=virginica} 0.06000000 1.0000000 3.000000 9
## [37] {Sepal.Length=SL_medium,
## Petal.Length=PL_long,
## Petal.Width=PW_medium} => {Species=virginica} 0.01333333 1.0000000 3.000000 2
## [38] {Sepal.Length=SL_medium,
## Sepal.Width=SW_long,
## Petal.Width=PW_long} => {Species=versicolor} 0.01333333 0.6666667 2.000000 2
## [39] {Sepal.Length=SL_long,
## Petal.Length=PL_long,
## Petal.Width=PW_long} => {Species=virginica} 0.24000000 0.9729730 2.918919 36Siendo:
- %in%: Selecciona conjuntos de elementos que coincidan con cualquier elemento dado.
- %ain%: Selecciona solo conjuntos de elementos que coincidan con todos los elementos dados.
- %oin%: Selecciona conjuntos de elementos que solo coincidan con el elemento dado.
- %pin%: %in% con ‘partial matching’.
9 - Usa summary, sort.
Summary: Devuelve una visión general del conjunto de reglas de asociación.
## set of 196 rules
##
## rule length distribution (lhs + rhs):sizes
## 2 3 4
## 51 109 36
##
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 2.000 2.000 3.000 2.923 3.000 4.000
##
## summary of quality measures:
## support confidence lift count
## Min. :0.01333 Min. :0.5000 Min. :1.339 Min. : 2.00
## 1st Qu.:0.04000 1st Qu.:0.7025 1st Qu.:2.109 1st Qu.: 6.00
## Median :0.11333 Median :0.9208 Median :2.713 Median :17.00
## Mean :0.13480 Mean :0.8470 Mean :2.542 Mean :20.22
## 3rd Qu.:0.24000 3rd Qu.:1.0000 3rd Qu.:2.951 3rd Qu.:36.00
## Max. :0.33333 Max. :1.0000 Max. :3.261 Max. :50.00
##
## mining info:
## data ntransactions support confidence
## dsIris3 150 0.01 0.5Sort: Ordena los elementos dependiendo del valor de las variables pasadas por parámetro:
## set of 196 rules10 - Explicar y usar los comandos de paquete arules siguientes: dissimilarity, image, is.redundant, is.significant, itemFrequency.
- Dissimilarity: Utiliza la función de distancia para calcular estadísticas de disimilitud agrupando variables.
Disimilitud: Se corresponde con el cálculo de falta de semejanza entre dos objetos principalmente.
## 1 2
## 2 0.6666667
## 3 0.6666667 0.6666667- image: Genera gráficos “de nivel” para inspeccionar visualmente las matrices de incidencia binarias.
# Cogemos los primeros 50 elementos para que la imagen no
# se vea tan sobrecargada
image(dsIris3[1:50])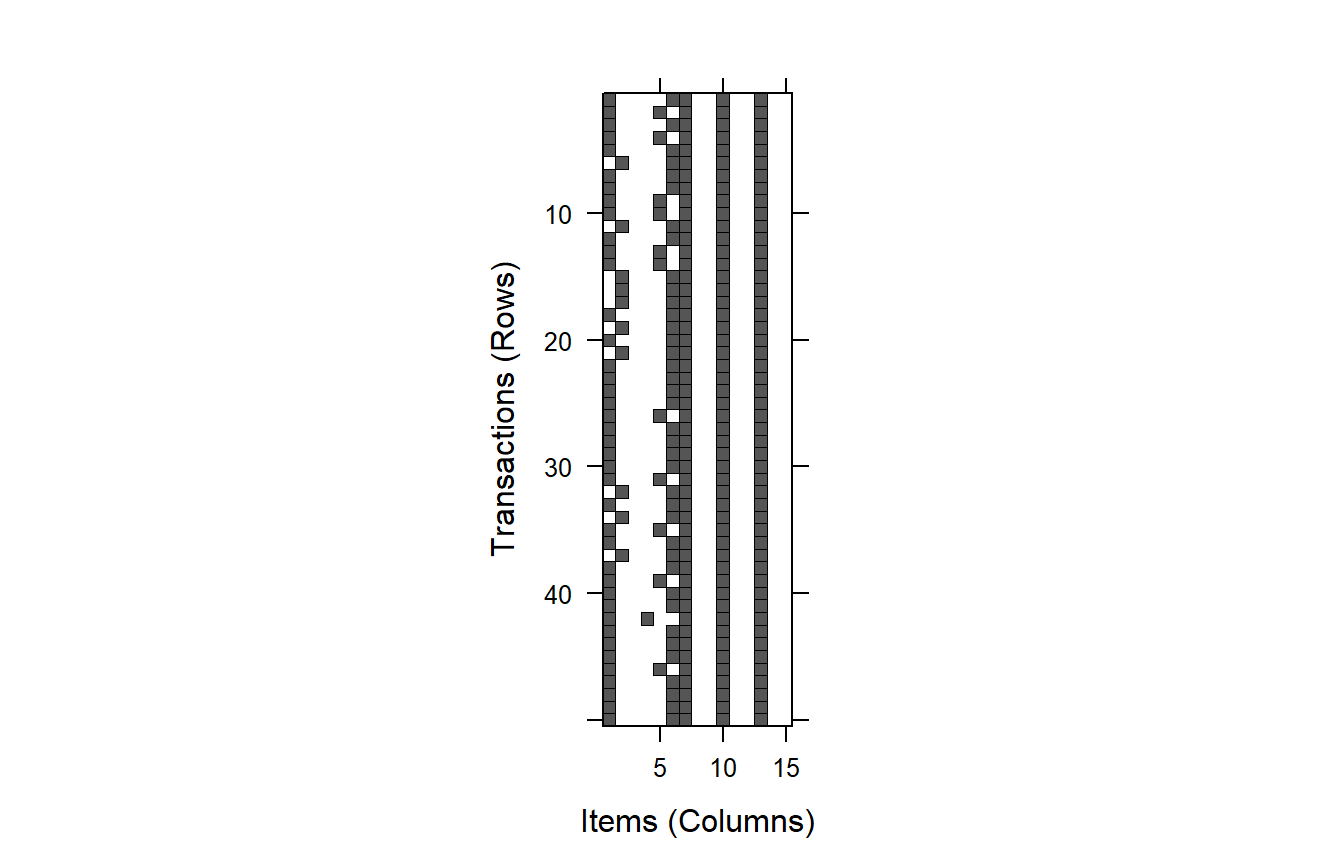
- itemFrequency: devuelve la frecuencia de cada item.
## Sepal.Length=SL_short Sepal.Length=SL_medium Sepal.Length=SL_long
## 0.13775510 0.28061224 0.21938776
## Sepal.Width=SW_short Sepal.Width=SW_medium Sepal.Width=SW_long
## 0.22959184 0.19387755 0.22959184
## Petal.Length=PL_short Petal.Length=PL_medium Petal.Length=PL_long
## 0.06632653 0.28571429 0.19387755
## Petal.Width=PW_short Petal.Width=PW_medium Petal.Width=PW_long
## 0.06632653 0.26530612 0.22959184
## Species=setosa Species=versicolor Species=virginica
## 0.06632653 0.27040816 0.18877551## Sepal.Length=SL_short Sepal.Length=SL_medium Sepal.Length=SL_long
## 0.3066667 0.3533333 0.3400000
## Sepal.Width=SW_short Sepal.Width=SW_medium Sepal.Width=SW_long
## 0.3133333 0.3133333 0.3733333
## Petal.Length=PL_short Petal.Length=PL_medium Petal.Length=PL_long
## 0.3333333 0.3266667 0.3400000
## Petal.Width=PW_short Petal.Width=PW_medium Petal.Width=PW_long
## 0.3333333 0.3200000 0.3466667
## Species=setosa Species=versicolor Species=virginica
## 0.3333333 0.3333333 0.3333333- is.redundant: Encuentra los elementos redudantes de un conjunto de reglas
Ejemplo ya visto en varias ocasiones a lo largo de la presentación.
- is.significant: Encuentra reglas donde el LHS y el RHS dependen el uno del otro.
## [1] FALSE FALSE FALSEEn este caso vemos que no depende ninguna regla entre r2 y r3