Capítulo 11 Pesquisa bibliográfica e Bibliometria
Uma etapa muito importante em qualquer pesquisa científica é fazer o levantamento bibliográfico. Temos diversas bases de dados nas quais podemos fazer essa busca, sendo que o PubMed é uma das principais.
O número de publicações acadêmicas está aumentando em ritmo acelerado e está se tornando cada vez mais inviável manter-se atualizado com tudo o que está sendo publicado.
Os pesquisadores usam diferentes abordagens qualitativas e quantitativas de revisão de literatura para entender e organizar descobertas anteriores. Entre eles, a bibliometria tem o potencial de introduzir um processo de revisão sistemática, transparente e reprodutível baseado na medição estatística da ciência, dos cientistas ou da atividade científica.
11.1 Pesquisa bibliográfica
Antes de fazer uma análise bibliométrica, é preciso fazer uma pesquisa bibliográfica. Para isso temos que estabelecer um pergunta a ser respondida. Com base no que queremos saber, definimos os descritores com os quais montaremos nosso termo de nossa busca.
Para auxiliar na escolha dos descritores, podemos usar o DeCS/MeSH (https://decs.bvsalud.org/). Aqui poderemos ver, entre outras coisas: - a grafia em diferentes idiomas - os sinônimos
11.1.1 Termos da busca
Uma vez definidos os descritores, usamos os operadores boleanos AND, OR, NOT para compor o termo de busca.
Exemplos:
- butyrylcholinesterase OR pseudocholinesterase
- butyrylcholinesterase AND acetylcholinesterase
- butyrylcholinesterase NOT alzheimer
- ((butyrylcholinesterase OR pseudocholinesterase) AND acetylcholinesterase) NOT alzheimer
Uma vez montado o termo de busca, podemos usá-lo em diferentes bases de dados para fazer nossa pesquisa bibliográfica.
11.1.2 Bases de dados
Temos diversas bases de dados. No Brasil temos o Portal de Periódicos CAPES, ao qual temos acesso completo apenas a partir das Universidades.
Portal de Periódicos CAPES (https://www-periodicos-capes-gov-br.ezl.periodicos.capes.gov.br/index.php?)
Também a partir do Portal de Periódicos CAPES, temos acesso a outras bases como a Web of Science e Scopus.
Além dessas, temos várias outras, como:
PubMed (https://pubmed.ncbi.nlm.nih.gov/)
Dimensions (https://app.dimensions.ai/discover/publication
)
11.2 PubMed
No caso do PubMed, é possível fazer a busca diretamente pelo R, utilizando o pacote pubmedR. Podem ver dicas de utilização do pacote em: https://cran.r-project.org/web/packages/pubmedR/vignettes/A_Brief_Example.html
Vamos ver um exemplo de utilização, em query montamos o termo de busca e colocamos alguns filtros. Com a função pmQueryTotalCount podemos ver quantos registros a busca retorna:
library(pubmedR)
query <- "PON1[Title/Abstract] AND english[LA]
AND Journal Article[PT] AND 2018:2023[DP]"
res <- pmQueryTotalCount(query = query, api_key = NULL)
res$total_count## [1] 965Com a função pmApiRequest vamos fazer o download dos dados e com a função pmApi2df transformamos em um dataframe:
## Documents 200 of 200## ================================================================================São baixados os metadados de cada referência, como título, autores, resumo, revista, ano, etc.
## [1] "AU" "AF" "TI" "SO" "SO_CO" "LA"
## [7] "DT" "DE" "ID" "MESH" "AB" "C1"
## [13] "CR" "TC" "SN" "J9" "JI" "PY"
## [19] "PY_IS" "VL" "DI" "PG" "GRANT_ID" "GRANT_ORG"
## [25] "UT" "PMID" "DB" "AU_UN" "AU_CO" "AU1_CO"## [1] "METHYLPREDNISOLONE DOES NOT ENHANCE PARAOXONASE 1 ACTIVITY DURING CARDIOPULMONARY BYPASS SURGERY-A RANDOMIZED, CONTROLLED CLINICAL TRIAL."Esses dados, em sua maioria, são caracteres, mas também podemos trabalhar com dados não estruturados no R. O pacote tm tem funções para podermos trabalhar com texto. Vamos pegar os títulos dos artigos:
library(tm)
library(dplyr)
texto <- iconv(df$TI, to = "UTF-8")
docs <- Corpus(VectorSource(texto))
docs <- docs %>%
tm_map(removePunctuation) %>%
tm_map(stripWhitespace)
docs <- tm_map(docs, content_transformer(tolower))
docs <- tm_map(docs, removeWords, stopwords("english"))A seguir criamos um dataframe com as palavras em ordem decrescente de frequência:
dtm <- TermDocumentMatrix(docs)
matrix <- as.matrix(dtm)
words <- sort(rowSums(matrix),decreasing=TRUE)
df2 <- data.frame(word = names(words),freq=words)
head(df2)## word freq
## paraoxonase paraoxonase 45
## patients patients 38
## activity activity 37
## disease disease 30
## study study 27
## paraoxonase1 paraoxonase1 24E com esse dataframe podemos fazer uma nuvem de palavras:
11.3 Bibliometria
Para fazer uma análise bibliométrica usamos o pacote bibliometrix O manual pode ser acessado em https://www.bibliometrix.org/vignettes/Introduction_to_bibliometrix.html (Aria and Cuccurullo 2017)
Você pode fazer sua busca no Web of Science, depois clique em exportar, arquivo de texto sem formatação, escolha a opção de quais quer salvar e selecione a opção registro completo, referências citadas e clique em exportar. Note que com essa opção é possível salvar 500 referências de cada vez, então se tiver mais de 500 referências, você terá vários arquivos, mas poderá importá-los de uma só vez no R.
Para fazer a bibliometria usaremos dados baixados, em formato texto, do Web of Science. Para fazer a bibliometria, podemos usar dados de várias bases de dados, mas a Web of Science é a que oferece o maior número de metadados, permitindo utilizar todas as opções de análise.
## Warning: package 'bibliometrix' was built under R version 4.3.1## Please note that our software is open source and available for use, distributed under the MIT license.
## When it is used in a publication, we ask that authors properly cite the following reference:
##
## Aria, M. & Cuccurullo, C. (2017) bibliometrix: An R-tool for comprehensive science mapping analysis,
## Journal of Informetrics, 11(4), pp 959-975, Elsevier.
##
## Failure to properly cite the software is considered a violation of the license.
##
## For information and bug reports:
## - Take a look at https://www.bibliometrix.org
## - Send an email to info@bibliometrix.org
## - Write a post on https://github.com/massimoaria/bibliometrix/issues
##
## Help us to keep Bibliometrix and Biblioshiny free to download and use by contributing with a small donation to support our research team (https://bibliometrix.org/donate.html)
##
##
## To start with the Biblioshiny app, please digit:
## biblioshiny()##
## Attaching package: 'bibliometrix'## The following object is masked _by_ '.GlobalEnv':
##
## logo## The following object is masked from 'package:magick':
##
## logo## The following object is masked from 'package:IRanges':
##
## trim## The following object is masked from 'package:gdata':
##
## trim##
## Converting your wos collection into a bibliographic dataframe
##
## Done!
##
##
## Generating affiliation field tag AU_UN from C1: Done!Podemos ver um resumo da análise:
##
##
## MAIN INFORMATION ABOUT DATA
##
## Timespan 2013 : 2023
## Sources (Journals, Books, etc) 261
## Documents 500
## Annual Growth Rate % -18.6
## Document Average Age 6.31
## Average citations per doc 16.2
## Average citations per year per doc 2.076
## References 14641
##
## DOCUMENT TYPES
## article 452
## article; book chapter 1
## article; early access 5
## article; proceedings paper 21
## meeting abstract 3
## proceedings paper 1
## review 17
##
## DOCUMENT CONTENTS
## Keywords Plus (ID) 1476
## Author's Keywords (DE) 1260
##
## AUTHORS
## Authors 2331
## Author Appearances 3225
## Authors of single-authored docs 12
##
## AUTHORS COLLABORATION
## Single-authored docs 19
## Documents per Author 0.215
## Co-Authors per Doc 6.45
## International co-authorships % 29
##
##
## Annual Scientific Production
##
## Year Articles
## 2013 47
## 2014 41
## 2015 38
## 2016 51
## 2017 52
## 2018 54
## 2019 66
## 2020 44
## 2021 52
## 2022 44
## 2023 6
##
## Annual Percentage Growth Rate -18.6
##
##
## Most Productive Authors
##
## Authors Articles Authors Articles Fractionalized
## 1 GULCIN I 38 POHANKA M 8.17
## 2 TASLIMI P 32 GULCIN I 6.58
## 3 LOCKRIDGE O 21 TASLIMI P 5.23
## 4 MASSON P 19 LOCKRIDGE O 4.52
## 5 KUCA K 14 TURKAN F 3.43
## 6 MUSILEK K 14 MASSON P 3.10
## 7 SCHOPFER LM 14 SCHOPFER LM 3.08
## 8 STEPANKOVA S 13 SAXENA A 2.64
## 9 JUN D 11 STEPANKOVA S 2.06
## 10 KOVARIK Z 11 KUCA K 2.02
##
##
## Top manuscripts per citations
##
## Paper DOI TC TCperYear NTC
## 1 GULCIN I, 2016, J ENZYM INHIB MED CH 10.3109/14756366.2015.1135914 144 16.0 4.81
## 2 TURKAN F, 2019, BIOORG CHEM 10.1016/j.bioorg.2019.02.013 135 22.5 8.56
## 3 AKSU K, 2016, ARCH PHARM 10.1002/ardp.201600183 119 13.2 3.97
## 4 OZTASKIN N, 2017, BIOORG CHEM 10.1016/j.bioorg.2017.07.010 117 14.6 5.04
## 5 BAYRAK C, 2017, BIOORG CHEM 10.1016/j.bioorg.2017.03.001 114 14.2 4.91
## 6 ERDEMIR F, 2018, J MOL STRUCT 10.1016/j.molstruc.2017.11.079 113 16.1 6.29
## 7 GARIBOV E, 2016, J ENZYM INHIB MED CH 10.1080/14756366.2016.1198901 112 12.4 3.74
## 8 GULCIN I, 2016, J ENZYM INHIB MED CH-a 10.3109/14756366.2015.1094470 110 12.2 3.67
## 9 OZGUN DO, 2016, J ENZYM INHIB MED CH 10.3109/14756366.2016.1149479 109 12.1 3.64
## 10 OZBEY F, 2016, J ENZYM INHIB MED CH 10.1080/14756366.2016.1189422 107 11.9 3.57
##
##
## Corresponding Author's Countries
##
## Country Articles Freq SCP MCP MCP_Ratio
## 1 TURKEY 71 0.1429 54 17 0.239
## 2 USA 68 0.1368 46 22 0.324
## 3 CZECH REPUBLIC 42 0.0845 32 10 0.238
## 4 CHINA 41 0.0825 35 6 0.146
## 5 BRAZIL 32 0.0644 24 8 0.250
## 6 PAKISTAN 28 0.0563 16 12 0.429
## 7 RUSSIA 26 0.0523 16 10 0.385
## 8 INDIA 20 0.0402 17 3 0.150
## 9 GERMANY 19 0.0382 15 4 0.211
## 10 KOREA 15 0.0302 12 3 0.200
##
##
## SCP: Single Country Publications
##
## MCP: Multiple Country Publications
##
##
## Total Citations per Country
##
## Country Total Citations Average Article Citations
## 1 TURKEY 2581 36.35
## 2 USA 980 14.41
## 3 ITALY 477 34.07
## 4 CZECH REPUBLIC 470 11.19
## 5 CHINA 404 9.85
## 6 BRAZIL 319 9.97
## 7 PAKISTAN 318 11.36
## 8 INDIA 273 13.65
## 9 POLAND 265 18.93
## 10 KOREA 229 15.27
##
##
## Most Relevant Sources
##
## Sources Articles
## 1 CHEMICO-BIOLOGICAL INTERACTIONS 31
## 2 MOLECULES 22
## 3 JOURNAL OF ENZYME INHIBITION AND MEDICINAL CHEMISTRY 18
## 4 BIOORGANIC CHEMISTRY 17
## 5 JOURNAL OF BIOCHEMICAL AND MOLECULAR TOXICOLOGY 11
## 6 EUROPEAN JOURNAL OF MEDICINAL CHEMISTRY 10
## 7 ARCHIV DER PHARMAZIE 9
## 8 BIOORGANIC & MEDICINAL CHEMISTRY 7
## 9 ARCHIVES OF TOXICOLOGY 6
## 10 JOURNAL OF MOLECULAR STRUCTURE 6
##
##
## Most Relevant Keywords
##
## Author Keywords (DE) Articles Keywords-Plus (ID) Articles
## 1 BUTYRYLCHOLINESTERASE 296 ACETYLCHOLINESTERASE 152
## 2 ACETYLCHOLINESTERASE 262 BUTYRYLCHOLINESTERASE 88
## 3 MOLECULAR DOCKING 46 ALZHEIMERS-DISEASE 86
## 4 CARBONIC ANHYDRASE 34 DERIVATIVES 65
## 5 ENZYME INHIBITION 32 CHOLINESTERASE 42
## 6 ORGANOPHOSPHATE 17 DESIGN 42
## 7 MOLECULAR MODELING 13 CHOLINESTERASES 37
## 8 ANTIOXIDANT 12 INHIBITION 35
## 9 ANTIOXIDANT ACTIVITY 12 IN-VITRO 34
## 10 BIOSCAVENGER 12 BIOLOGICAL EVALUATION 33Podemos ver os principais autores sobre o tema e sua produção ao longo do tempo

## $dfAU
## Author year freq TC TCpY
## 1 GULCIN I 2016 7 787 87.4444444
## 2 GULCIN I 2017 10 732 91.5000000
## 3 GULCIN I 2018 9 544 77.7142857
## 4 GULCIN I 2019 6 362 60.3333333
## 5 GULCIN I 2020 3 31 6.2000000
## 6 GULCIN I 2021 1 29 7.2500000
## 7 GULCIN I 2022 1 0 0.0000000
## 8 JUN D 2013 2 48 4.0000000
## 9 JUN D 2014 1 9 0.8181818
## 10 JUN D 2017 2 36 4.5000000
## 11 JUN D 2018 1 6 0.8571429
## 12 JUN D 2019 2 2 0.3333333
## 13 JUN D 2020 1 14 2.8000000
## 14 JUN D 2021 1 2 0.5000000
## 15 JUN D 2022 1 0 0.0000000
## 16 KOVARIK Z 2013 2 77 6.4166667
## 17 KOVARIK Z 2016 2 45 5.0000000
## 18 KOVARIK Z 2017 2 34 4.2500000
## 19 KOVARIK Z 2018 1 21 3.0000000
## 20 KOVARIK Z 2019 1 21 3.5000000
## 21 KOVARIK Z 2020 2 27 5.4000000
## 22 KOVARIK Z 2021 1 9 2.2500000
## 23 KUCA K 2013 3 62 5.1666667
## 24 KUCA K 2015 2 27 2.7000000
## 25 KUCA K 2017 2 36 4.5000000
## 26 KUCA K 2018 1 6 0.8571429
## 27 KUCA K 2019 5 38 6.3333333
## 28 KUCA K 2020 1 14 2.8000000
## 29 LOCKRIDGE O 2013 1 13 1.0833333
## 30 LOCKRIDGE O 2014 2 43 3.9090909
## 31 LOCKRIDGE O 2015 3 89 8.9000000
## 32 LOCKRIDGE O 2016 5 95 10.5555556
## 33 LOCKRIDGE O 2017 3 41 5.1250000
## 34 LOCKRIDGE O 2018 4 30 4.2857143
## 35 LOCKRIDGE O 2019 1 5 0.8333333
## 36 LOCKRIDGE O 2020 1 1 0.2000000
## 37 LOCKRIDGE O 2022 1 2 0.6666667
## 38 MASSON P 2013 1 17 1.4166667
## 39 MASSON P 2014 2 28 2.5454545
## 40 MASSON P 2015 1 17 1.7000000
## 41 MASSON P 2016 3 73 8.1111111
## 42 MASSON P 2017 2 29 3.6250000
## 43 MASSON P 2018 2 5 0.7142857
## 44 MASSON P 2019 4 18 3.0000000
## 45 MASSON P 2020 2 12 2.4000000
## 46 MASSON P 2021 1 1 0.2500000
## 47 MASSON P 2022 1 1 0.3333333
## 48 MUSILEK K 2013 1 19 1.5833333
## 49 MUSILEK K 2015 2 27 2.7000000
## 50 MUSILEK K 2018 1 6 0.8571429
## 51 MUSILEK K 2019 5 27 4.5000000
## 52 MUSILEK K 2020 1 14 2.8000000
## 53 MUSILEK K 2021 4 13 3.2500000
## 54 SCHOPFER LM 2013 1 13 1.0833333
## 55 SCHOPFER LM 2014 1 20 1.8181818
## 56 SCHOPFER LM 2015 1 40 4.0000000
## 57 SCHOPFER LM 2016 2 28 3.1111111
## 58 SCHOPFER LM 2017 3 41 5.1250000
## 59 SCHOPFER LM 2018 3 28 4.0000000
## 60 SCHOPFER LM 2019 1 5 0.8333333
## 61 SCHOPFER LM 2020 1 1 0.2000000
## 62 SCHOPFER LM 2022 1 2 0.6666667
## 63 STEPANKOVA S 2013 1 37 3.0833333
## 64 STEPANKOVA S 2014 2 20 1.8181818
## 65 STEPANKOVA S 2015 1 19 1.9000000
## 66 STEPANKOVA S 2016 2 33 3.6666667
## 67 STEPANKOVA S 2018 1 12 1.7142857
## 68 STEPANKOVA S 2020 3 13 2.6000000
## 69 STEPANKOVA S 2021 2 11 2.7500000
## 70 STEPANKOVA S 2022 1 1 0.3333333
## 71 TASLIMI P 2016 5 533 59.2222222
## 72 TASLIMI P 2017 8 636 79.5000000
## 73 TASLIMI P 2018 9 544 77.7142857
## 74 TASLIMI P 2019 5 353 58.8333333
## 75 TASLIMI P 2021 1 29 7.2500000
## 76 TASLIMI P 2022 4 5 1.6666667
##
## $dfPapersAU
## # A tibble: 186 × 7
## Author year TI SO DOI TC TCpY
## <chr> <dbl> <chr> <chr> <chr> <dbl> <dbl>
## 1 GULCIN I 2022 EVALUATION OF ANTICHOLINERGIC, ANTIDIABETIC AND ANTIOXIDANT ACTIVITY OF ASTRA… KSU … 10.1… 0 0
## 2 GULCIN I 2021 DETERMINATION OF ANTICANCER PROPERTIES AND INHIBITORY EFFECTS OF SOME METABOL… JOUR… 10.1… 29 7.25
## 3 GULCIN I 2020 ANTICHOLINERGIC AND ANTIOXIDANT ACTIVITIES OF AVOCADO (FOLIUM PERSEAE) LEAVES… INTE… 10.1… 24 4.8
## 4 GULCIN I 2020 SYNTHESIS AND PHARMACOLOGICAL EFFECTS OF NOVEL BENZENESULFONAMIDES CARRYING B… TURK… 10.3… 4 0.8
## 5 GULCIN I 2020 SCREENING OF NON-ALKALOID ACETYLCHOLINESTERASE AND CARBONIC ANHYDRASE ISOENZY… JOUR… 10.1… 3 0.6
## 6 GULCIN I 2019 THE IMPACTS OF SOME SEDATIVE DRUGS ON ALPHA -GLYCOSIDASE, ACETYLCHOLINESTERAS… LETT… 10.2… 18 3
## 7 GULCIN I 2019 THE EFFECTS OF SOME ANTIBIOTICS FROM CEPHALOSPORIN GROUPS ON THE ACETYLCHOLIN… ARCH… 10.1… 47 7.83
## 8 GULCIN I 2019 PHYTOCHEMICAL CONTENT, ANTIOXIDANT ACTIVITY, AND ENZYME INHIBITION EFFECT OF … JOUR… 10.1… 93 15.5
## 9 GULCIN I 2019 SYNTHESIS, CHARACTERIZATION, CRYSTAL STRUCTURES, THEORETICAL CALCULATIONS AND… JOUR… 10.1… 60 10
## 10 GULCIN I 2019 SAR EVALUATION OF DISUBSTITUTED TACRINE ANALOGUES AS PROMISING CHOLINESTERASE… INDI… 10.5… 9 1.5
## # ℹ 176 more rows
##
## $graph
Também podemos ver a co-ocorrência de palavras-chave:
NetMatrix <- biblioNetwork(M1, analysis = "co-occurrences", network = "keywords", sep = ";")
net=networkPlot(NetMatrix, normalize="association", weighted=T, n = 30, Title = "Keyword Co-occurrences", type = "fruchterman", size=T,edgesize = 5,labelsize=0.7)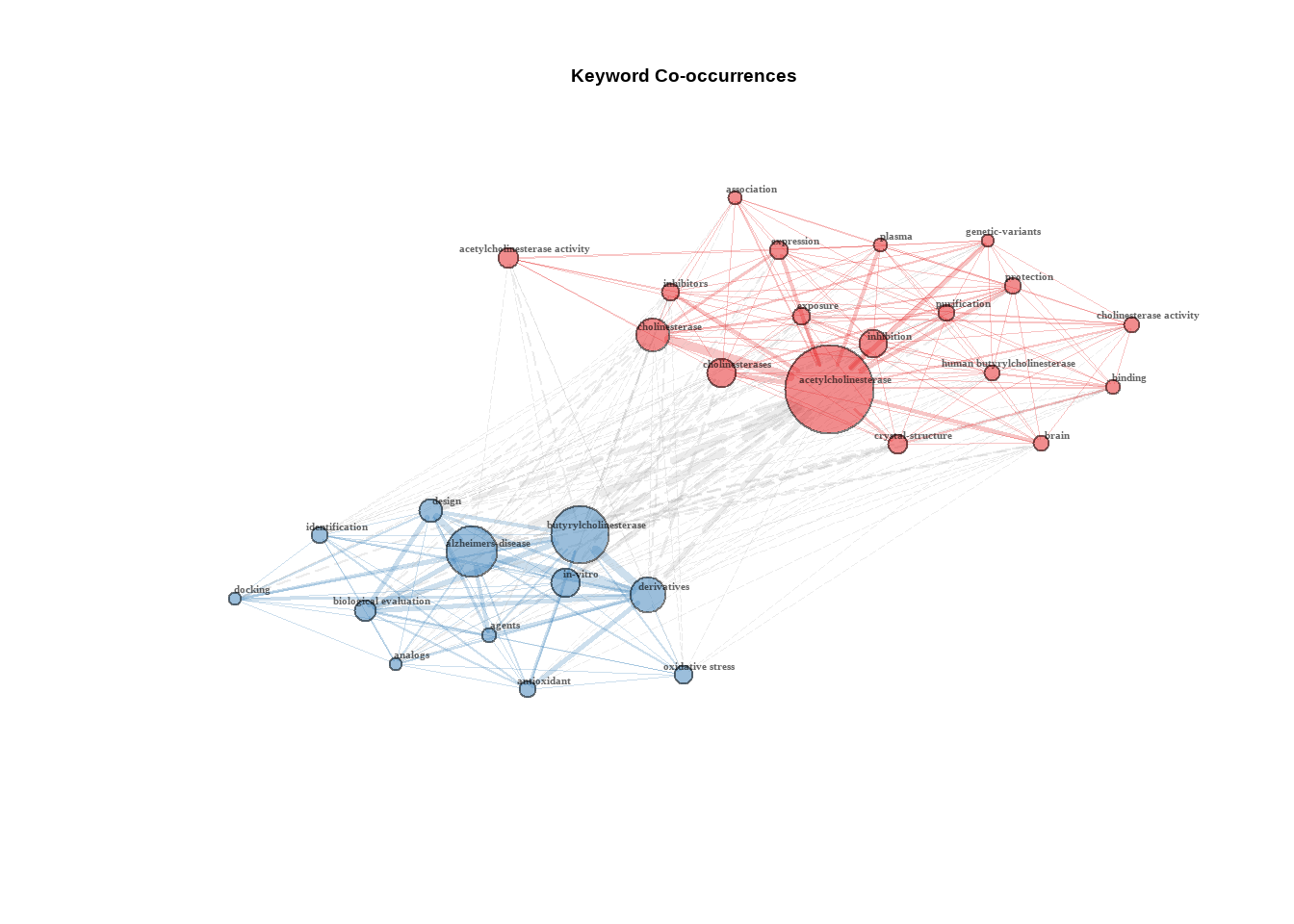
E estruturas conceituais usando as palavras-chave:
CS <- conceptualStructure(M1,field="ID", method="CA", minDegree=4, clust=5, stemming=FALSE, labelsize=10, documents=10)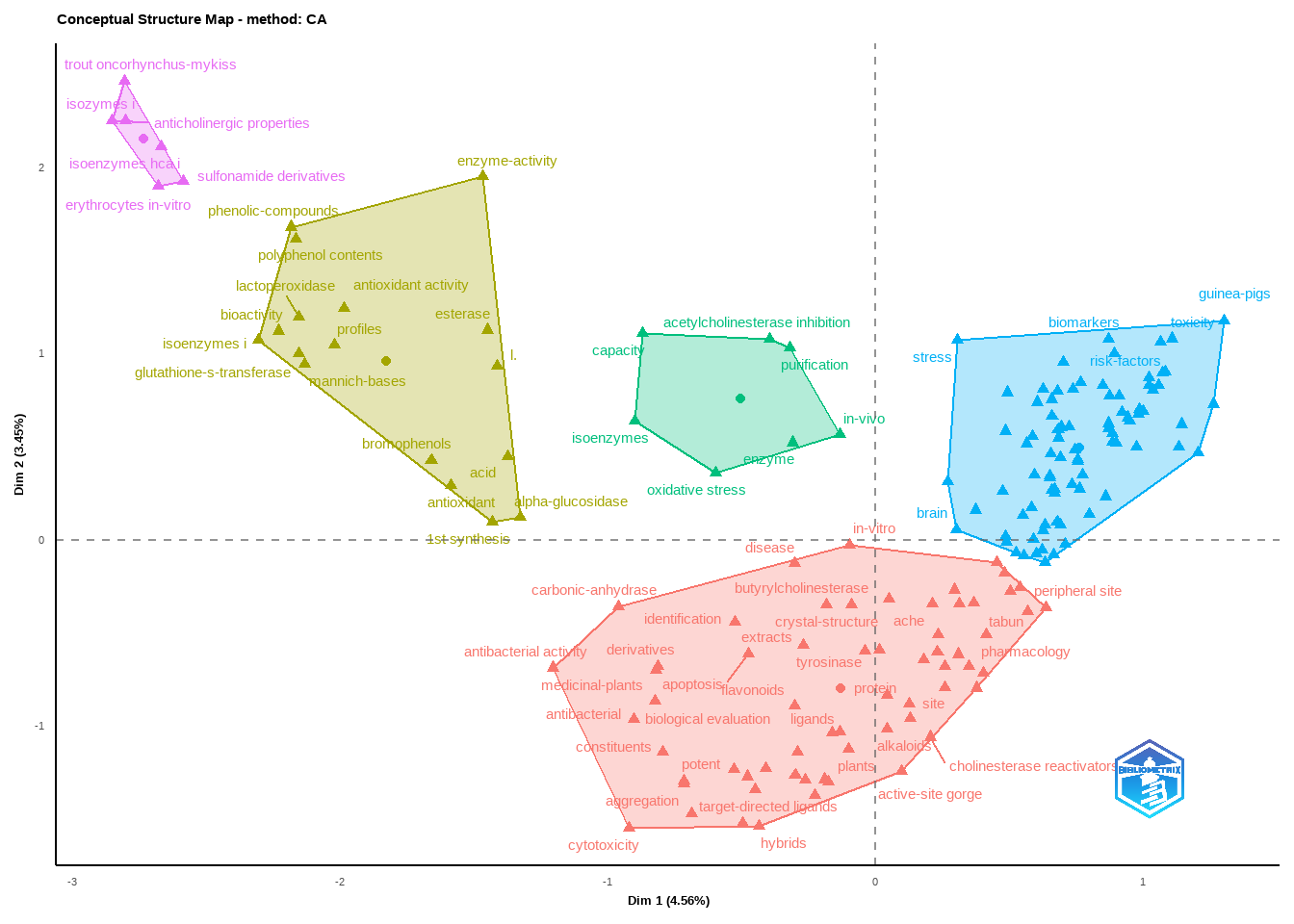

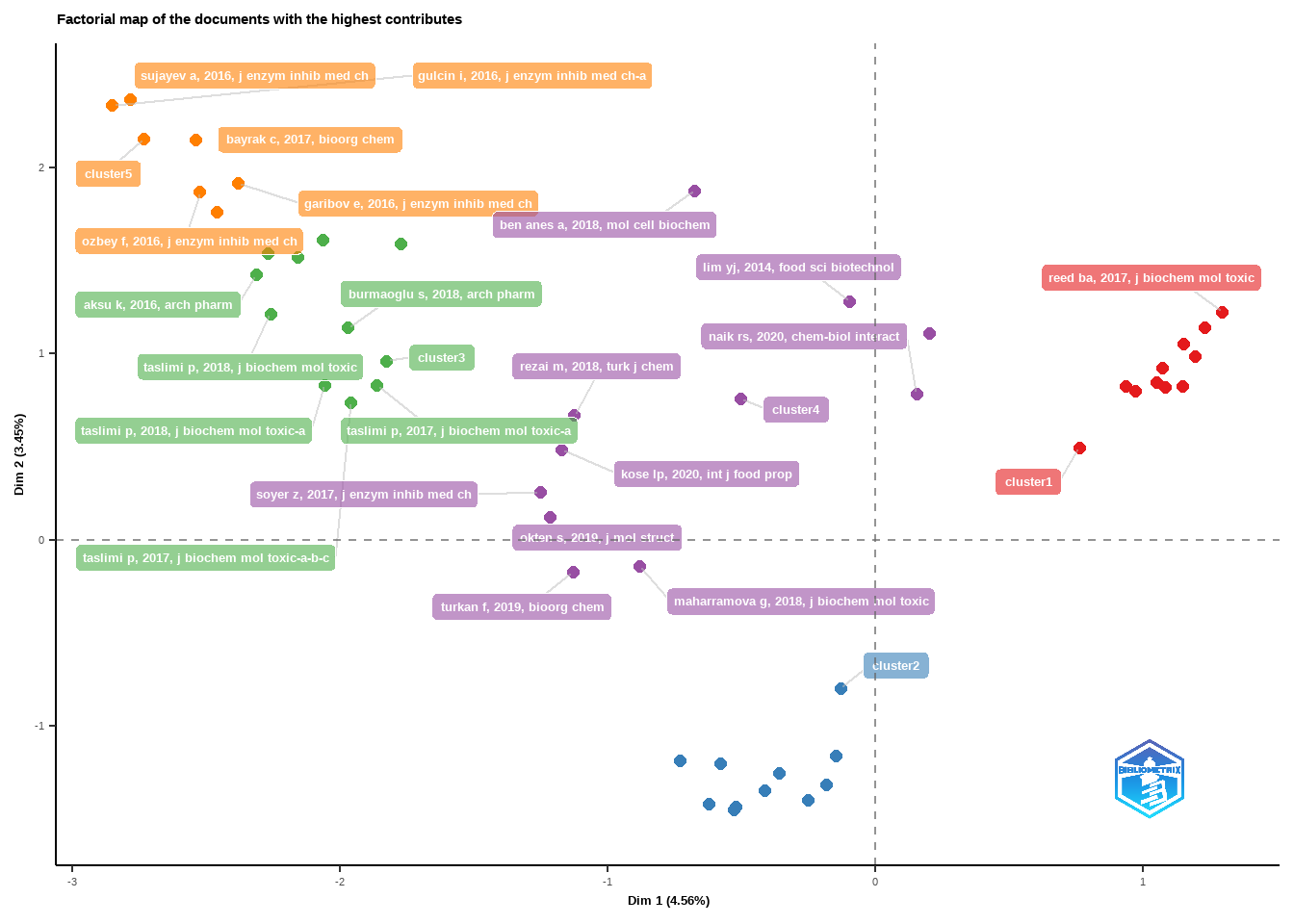
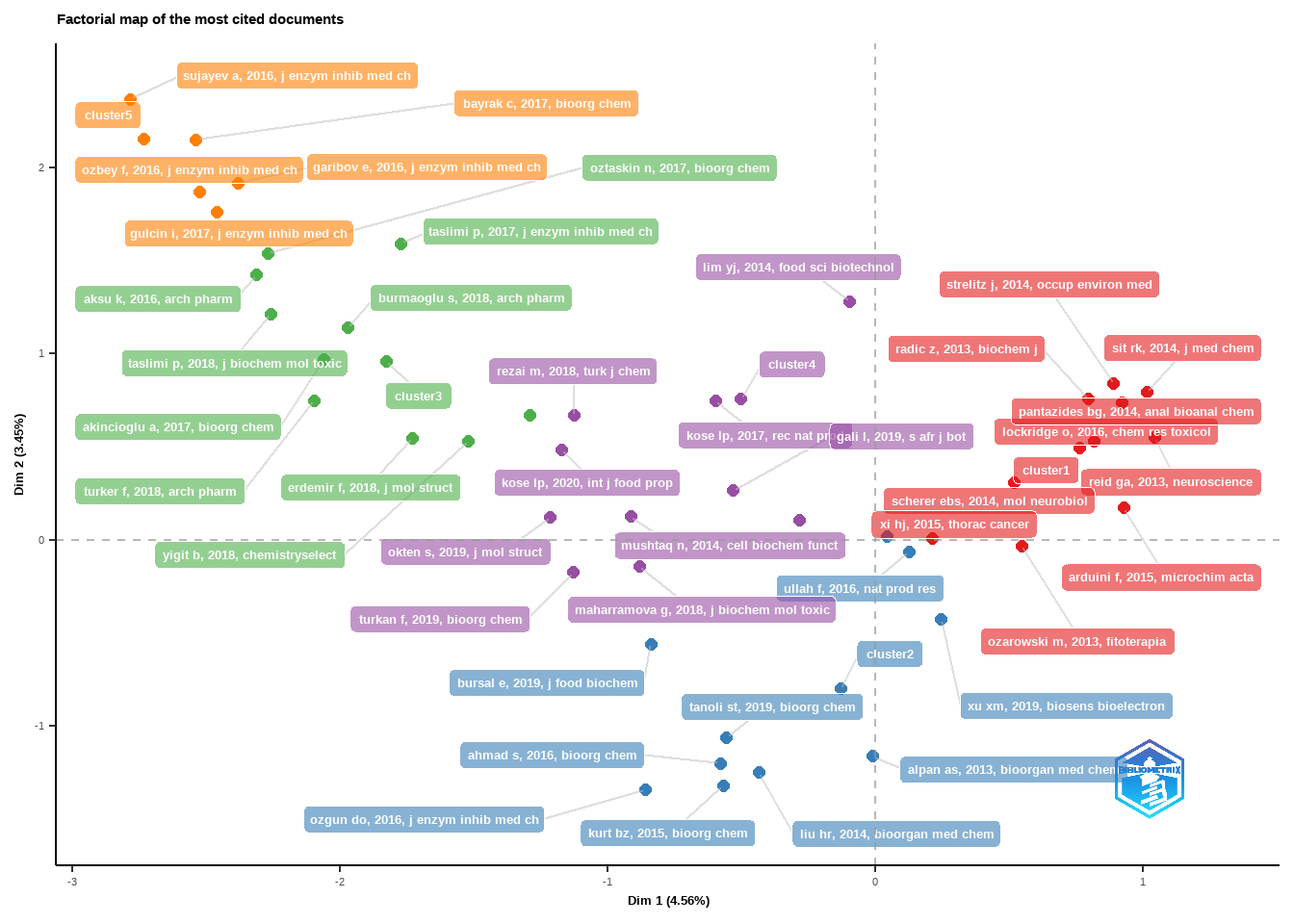
Toda a análise bibliométrica também pode ser feita utilizando shiny, para isso usamos a seguinte função: biblioshiny(), com a qual será aberta uma janela interativa, onde carregamos o arquivo e podemos fazer as análises e gerar os gráficos.
Para maiores detalhes, consulte a documentação no site https://www.bibliometrix.org/