Capítulo 4 Gráficos
O R permite fazer qualquer tipo de gráfico, os quais podem ser personalizados e salvos em diversos formatos e resoluções.
Existem muitos pacotes gráficos no R e nesse capítulo vamos ver alguns deles.
4.1 Gráficos com o pacote graphics (R base)
4.1.1 Introdução aos gráficos do R
Quando instalamos o R, já temos um pacote para gráficos (graphics) e com ele podemos fazer e personalizar os principais tipos de gráficos.
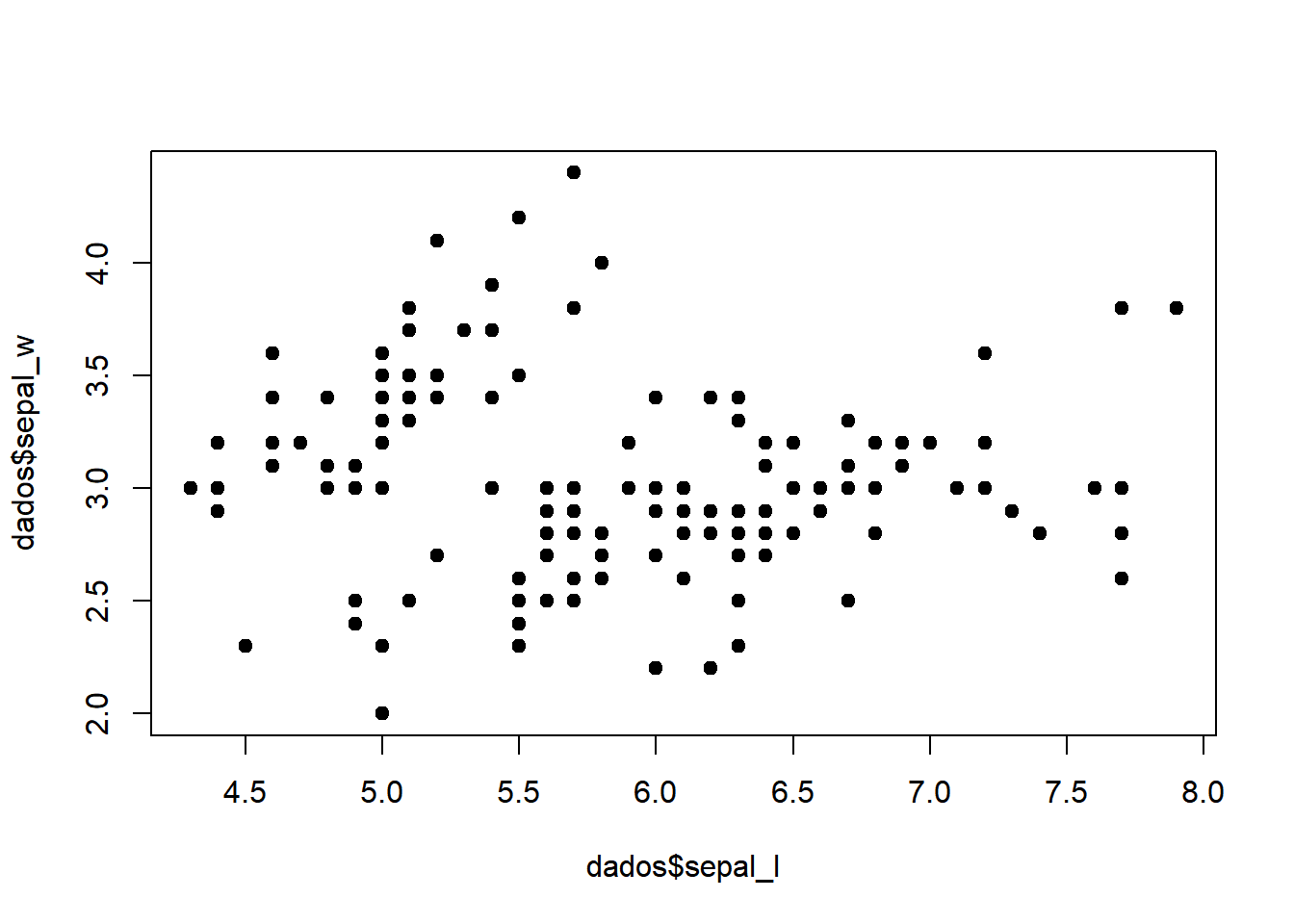
Vamos começar carregando os dados. Usaremos o arquivo iris e vamos criar um gráfico de dispersão com as variáveis comprimento e largura das sépalas.
## sepal_l sepal_w petal_l petal_w class
## 1 5.1 3.5 1.4 0.2 Iris-setosa
## 2 4.9 3.0 1.4 0.2 Iris-setosa
## 3 4.7 3.2 1.3 0.2 Iris-setosa
## 4 4.6 3.1 1.5 0.2 Iris-setosa
## 5 5.0 3.6 1.4 0.2 Iris-setosa
## 6 5.4 3.9 1.7 0.4 Iris-setosa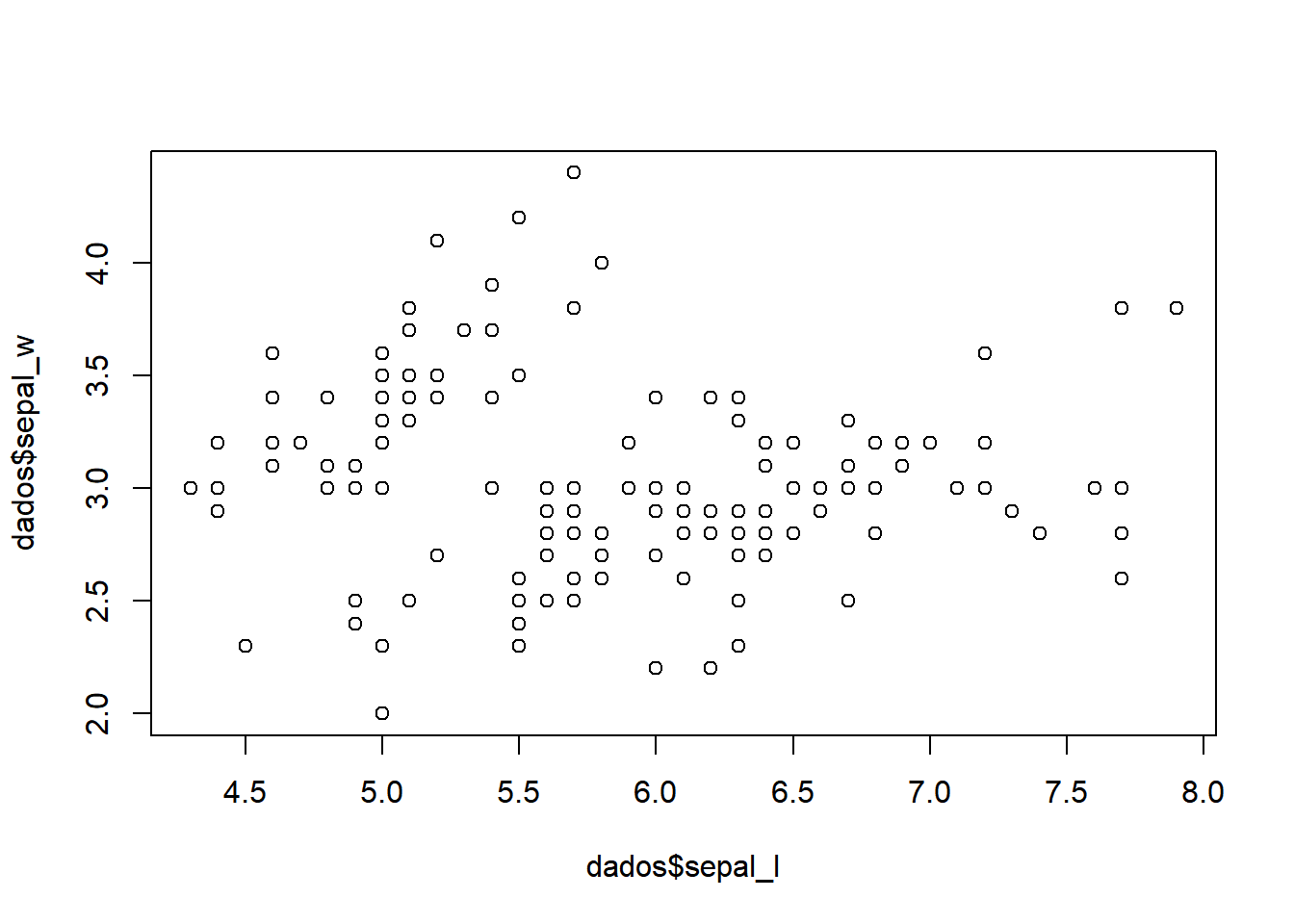
Podemos usar mais parâmetros, como type.
- p: Points.
- l: Lines.
- b: Both.
- c: The lines part alone of b
- o: Both “overplotted”
- h: Histogram like (or high-density) vertical lines.
- n: No plotting.
Podemos mudar o padrão dos pontos com pch Os valores vão de 0 a 25:
4.1.2 Salvar
Para salvar um gráfico podemos clicar na opção export, na janela Plots do RStudio ou podemos fazer assim:
png(file="figura1.png", units="in", res = 300, width = 5,height = 5)
plot(dados$sepal_l,dados$sepal_w)
dev.off()## png
## 2As opções de units são: px, in, cm ou mm. Também podemos salvar em outros formatos como jpg ou tif.
Para entender as questões de tamanho e resolução de imagem, consulte: https://pixelcalculator.com/en
É possível fazer vários gráficos na mesma janela usando par() e mfrow().
Para um arranjo 2 por 2:
par(mfrow=c(2,2))
plot(dados$sepal_l,dados$sepal_w)
plot(dados$petal_l,dados$petal_w)
plot(dados$sepal_l,dados$petal_w)
plot(dados$sepal_w,dados$petal_l)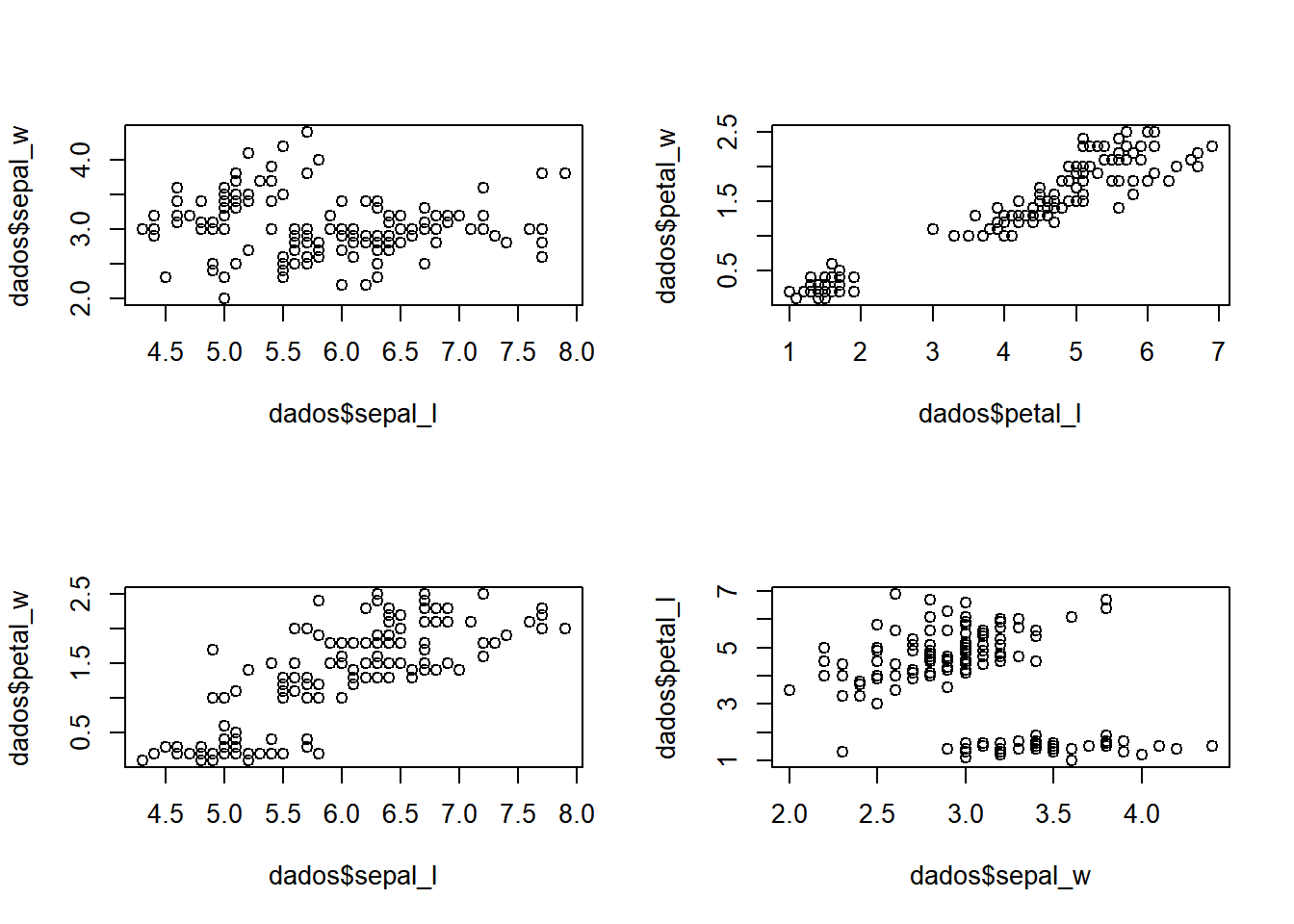
Para voltar ao normal:
Personalizando o gráfico
Podemos usar vários parâmetros para dar nome ao gráficos e aos eixos, especificar as cores, etc.
plot(dados$sepal_l,dados$sepal_w,
xlab="Nome eixo X", ylab="Nome eixo Y",
main="Título do gráfico",
xlim=c(3,9),#limites do eixo x
ylim=c(1,6),#limites do eixo y
col="darkmagenta",#cor dos pontos
pch=22,#formato dos pontos
bg="darkolivegreen",#cor de preenchimento
tcl=0.4,#tamanho dos traços dos eixos
las=1,#orientação dos valores nos eixos: 0,1,2,3
cex=1.5,#tamanho do ponto
bty="n")#altera as bordas: "o" (default), 
O gráfico pode ficar mais informativo:
Colocando legenda das cores:
dados$class<-as.factor(dados$class)
plot(dados$sepal_l,dados$sepal_w, col = dados$class)
legend('topright', col=unique(dados$class), legend=levels(dados$class), pch =1)
Se quiser especificar as cores:
Brincando com as cores:
val <- dados$sepal_l + dados$sepal_w
maxnovo<-1
minnovo<-0
valcol<-((val-min(val))/(max(val) - min(val)))*(maxnovo - minnovo) + minnovo
plot(dados$sepal_l,dados$sepal_w, pch = 15, col = gray(valcol))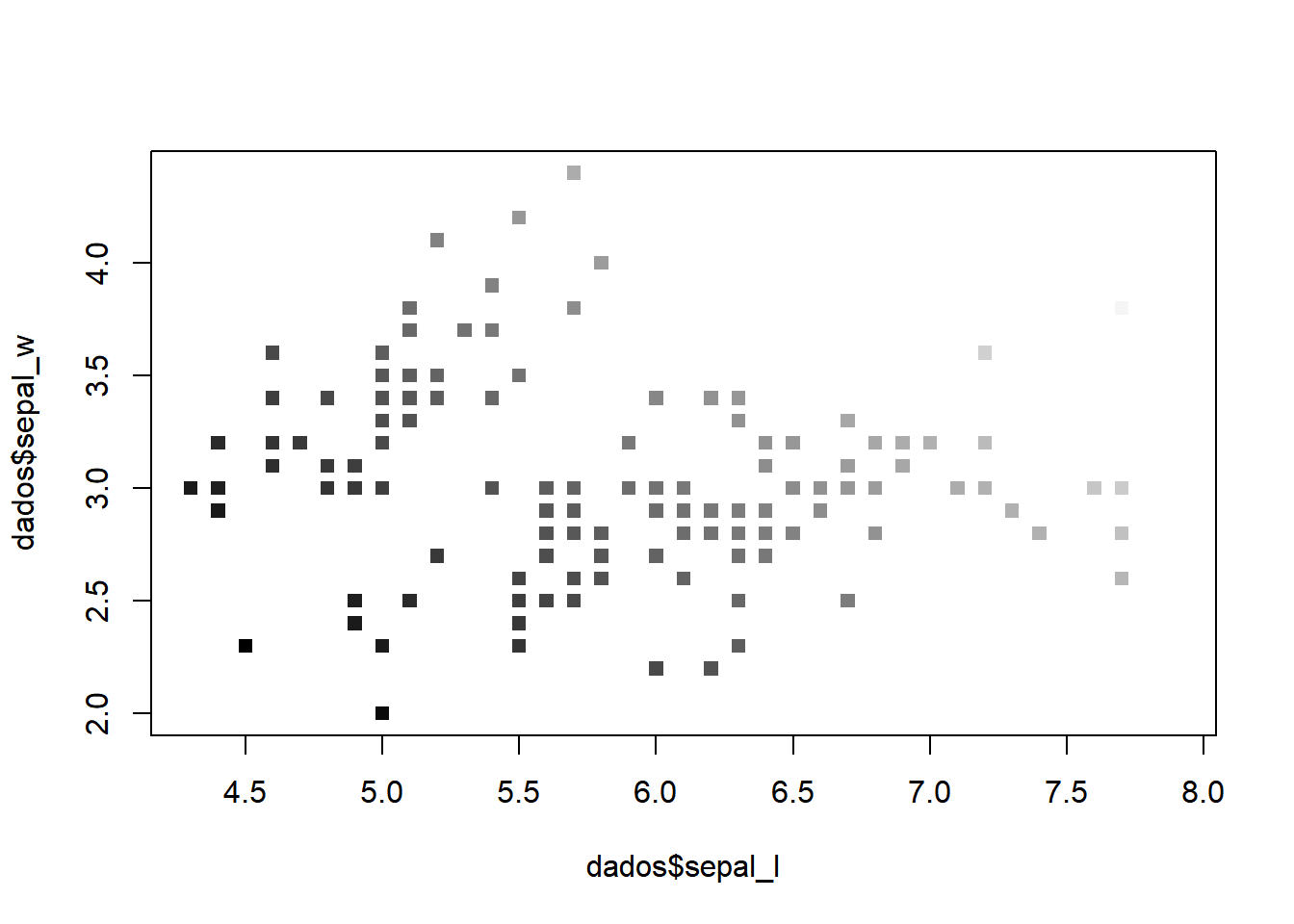
Podemos explorar os dados:

Acrescentando cor por espécie:
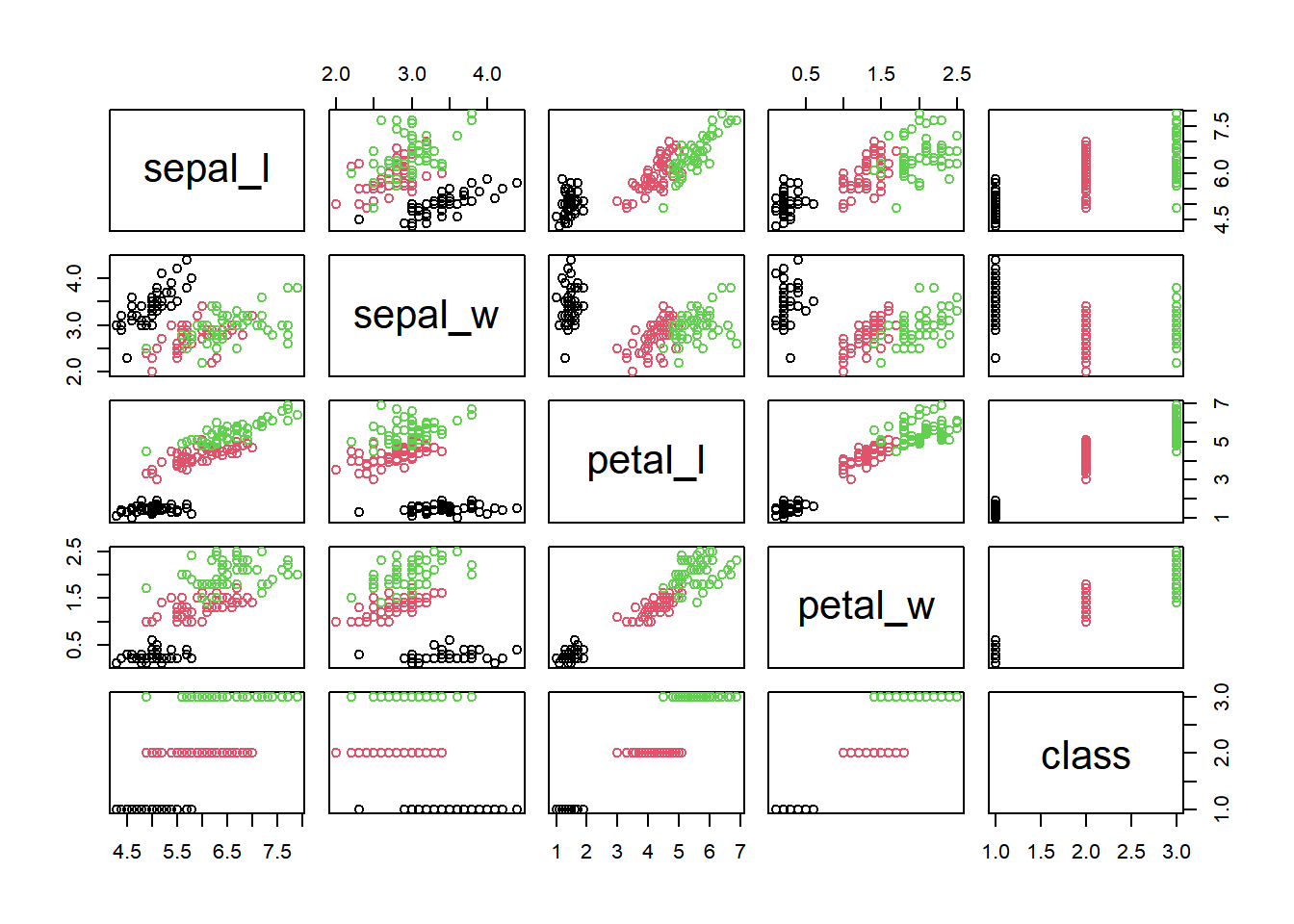
4.1.3 Histogramas
Para fazer um histograma usamos a função hist.
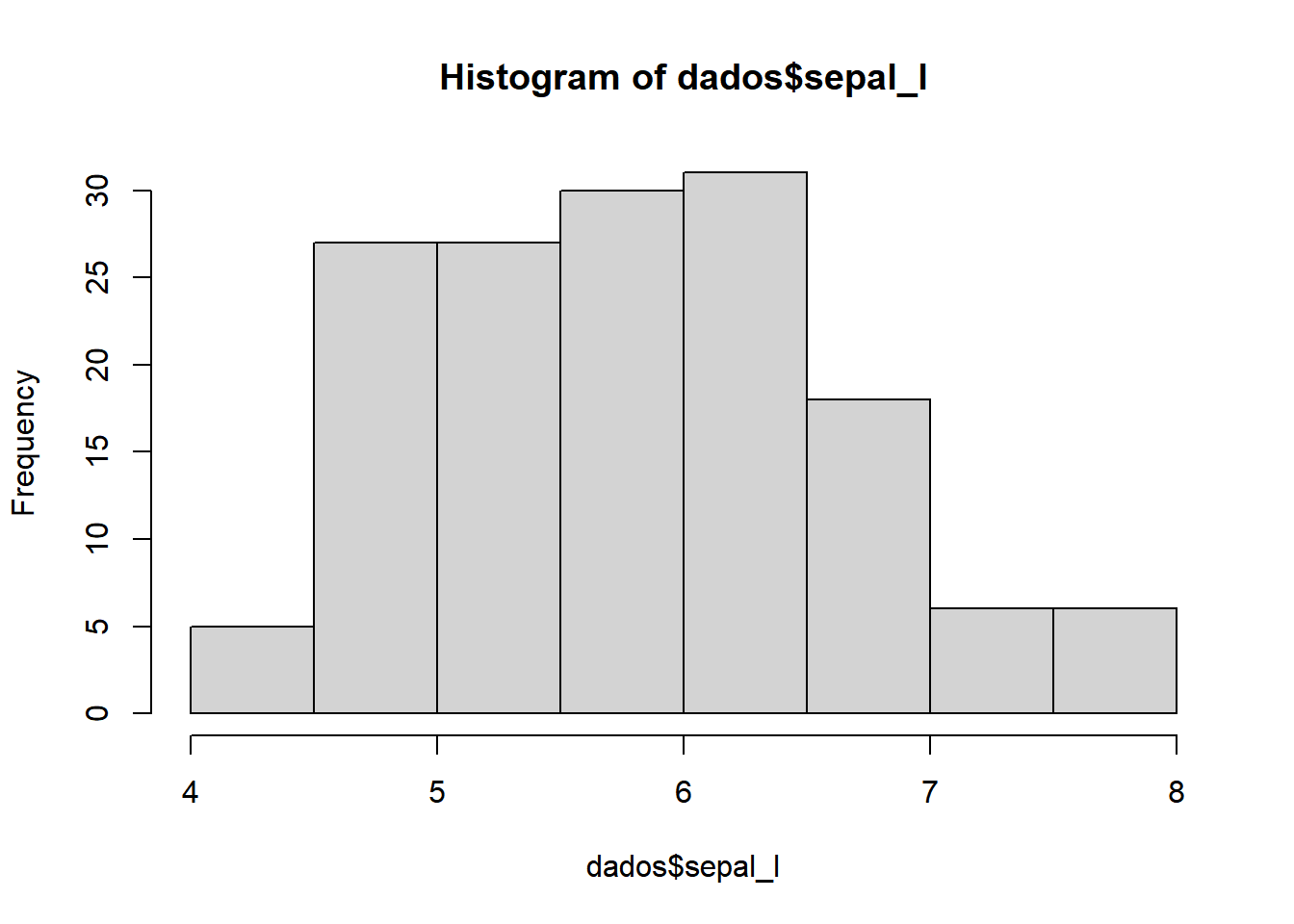
Também podemos personalizar o histograma:
hist(dados$sepal_l,
main="Folha",
xlab="comprimento sépala",
ylab="Frequência",
col=c("gold4","darkseagreen4"),
border="black",
adj=0, #alinhamento do texto
col.axis="forestgreen")#cor do texto nos eixos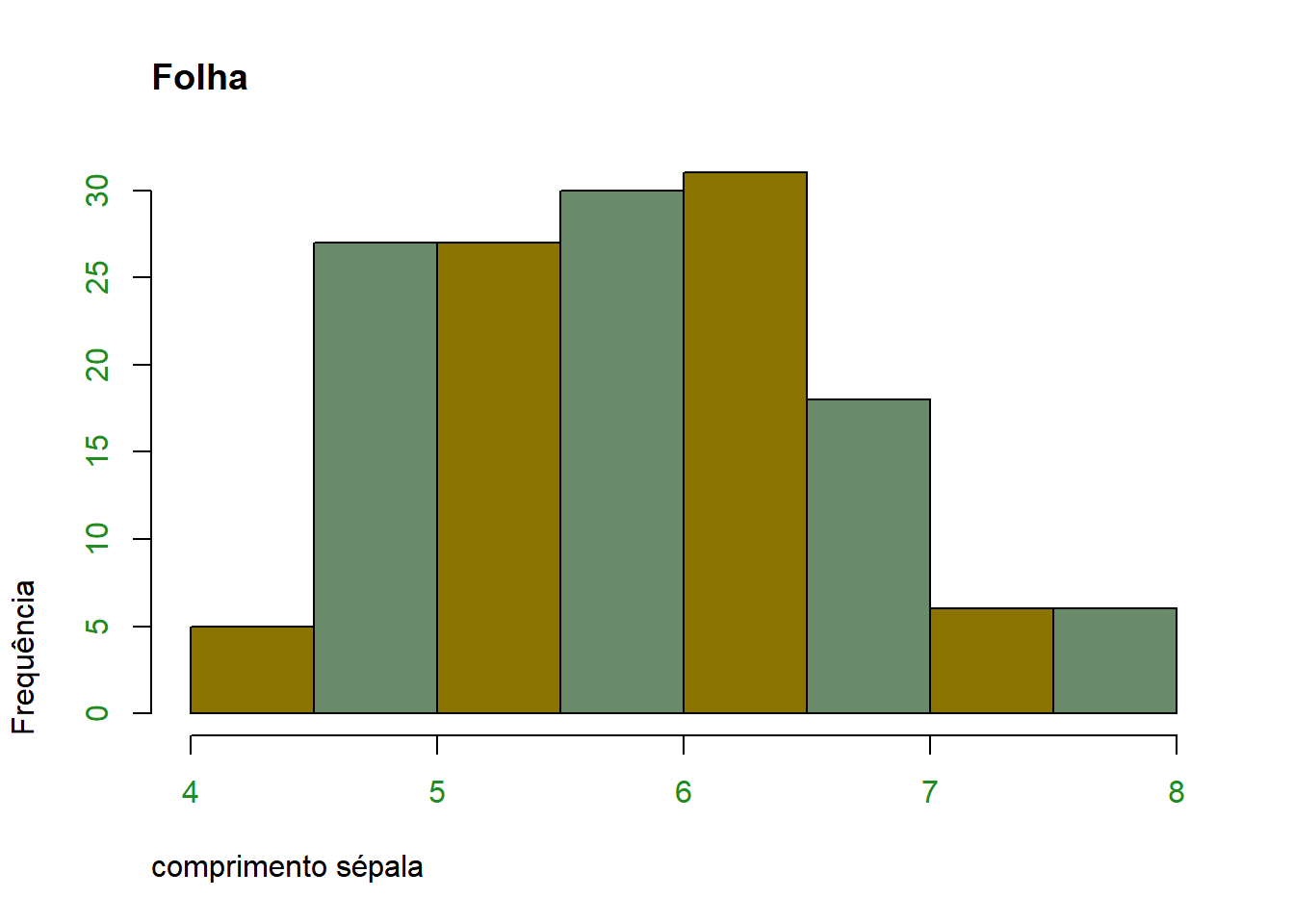
4.1.4 Boxplot
Outro tipo de gráfico bastante usado é o boxplot
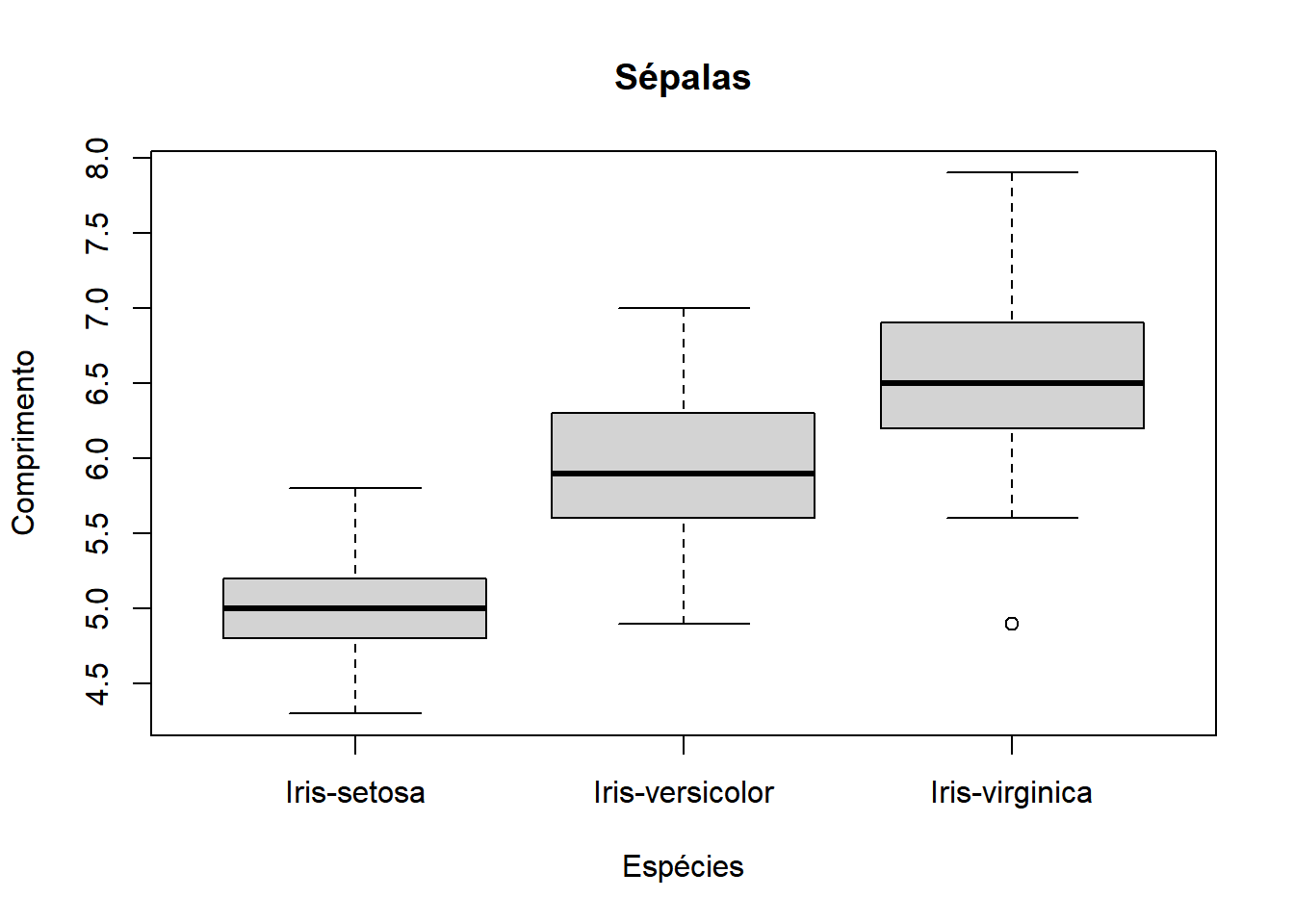
Podemos personalizar o boxplot:
boxplot(dados$sepal_l ~ dados$class,
main="Sépalas", xlab="Espécies",
ylab="Comprimento",
notch=T,
col=c("brown1","khaki1", "blue"))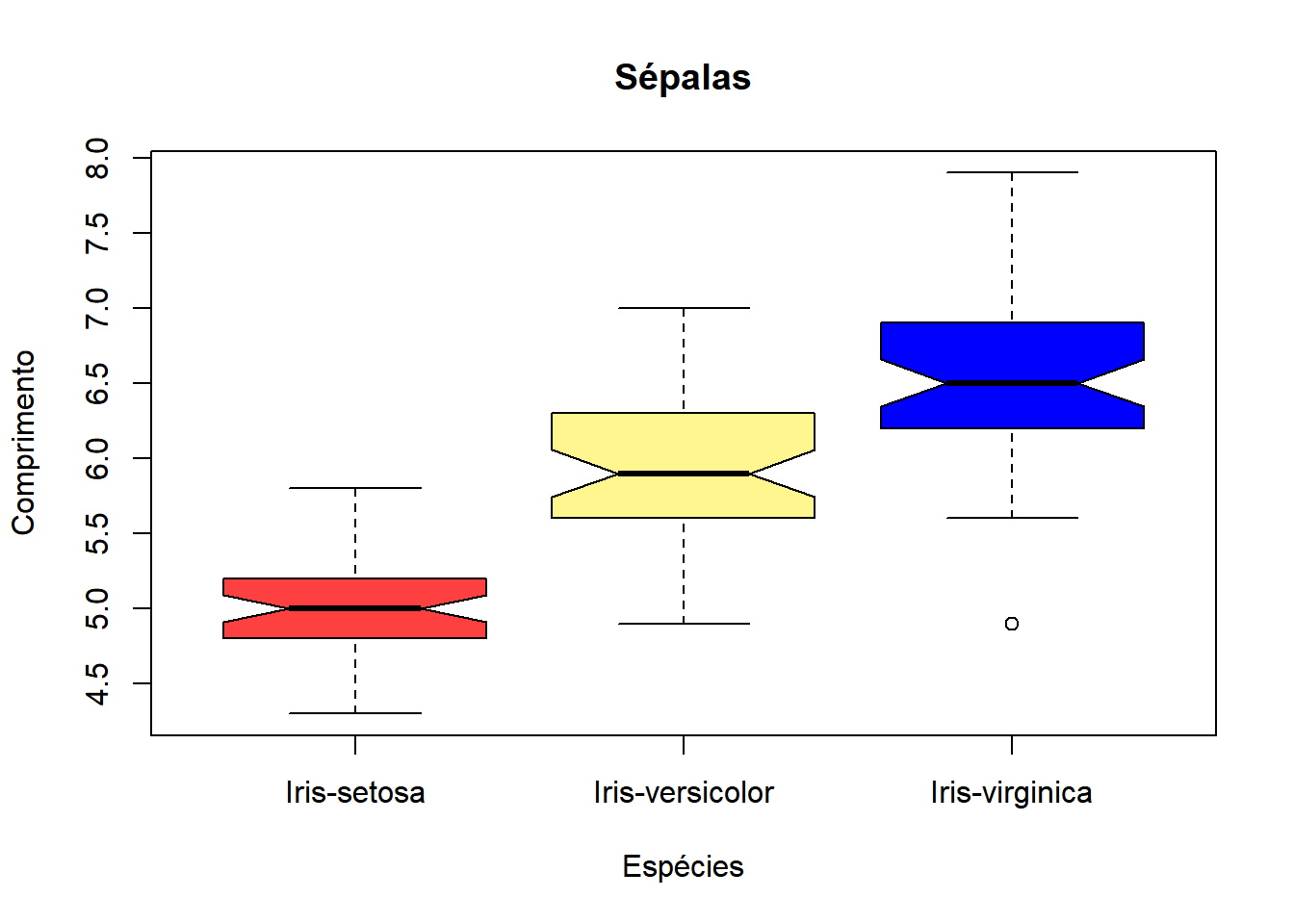
As cores podem ser especificadas pelo nome da cor ou pelo código hex. Os nomes e os códigos podem ser encontrados em vários sites como:
4.1.5 Barplot
Também temos o gráfico de barras. Embora tenha semelhanças com o histograma, os histogramas são barras conectadas umas às outras, visualizando a distribuição de uma variável quantitativa contínua e os gráficos de barras usam retângulos de tamanho proporcional para visualizar algum tipo de dados categóricos.
Podemos criar uma tabela de frequência com a função table e usar o resultado para criar um gráfico de barras:
4.2 O pacote ggplot2
O pacote ggplot2 (Wickham 2016) é um dos principais pacotes gráficos do R.
Os elementos de um gráfico (dados, sistema de coordenadas, rótulos, anotações, entre outros) são as camadas e a construção de um gráfico se dá pela sobreposição dessas camadas.
4.2.1 Gráficos de pontos (dispersão)
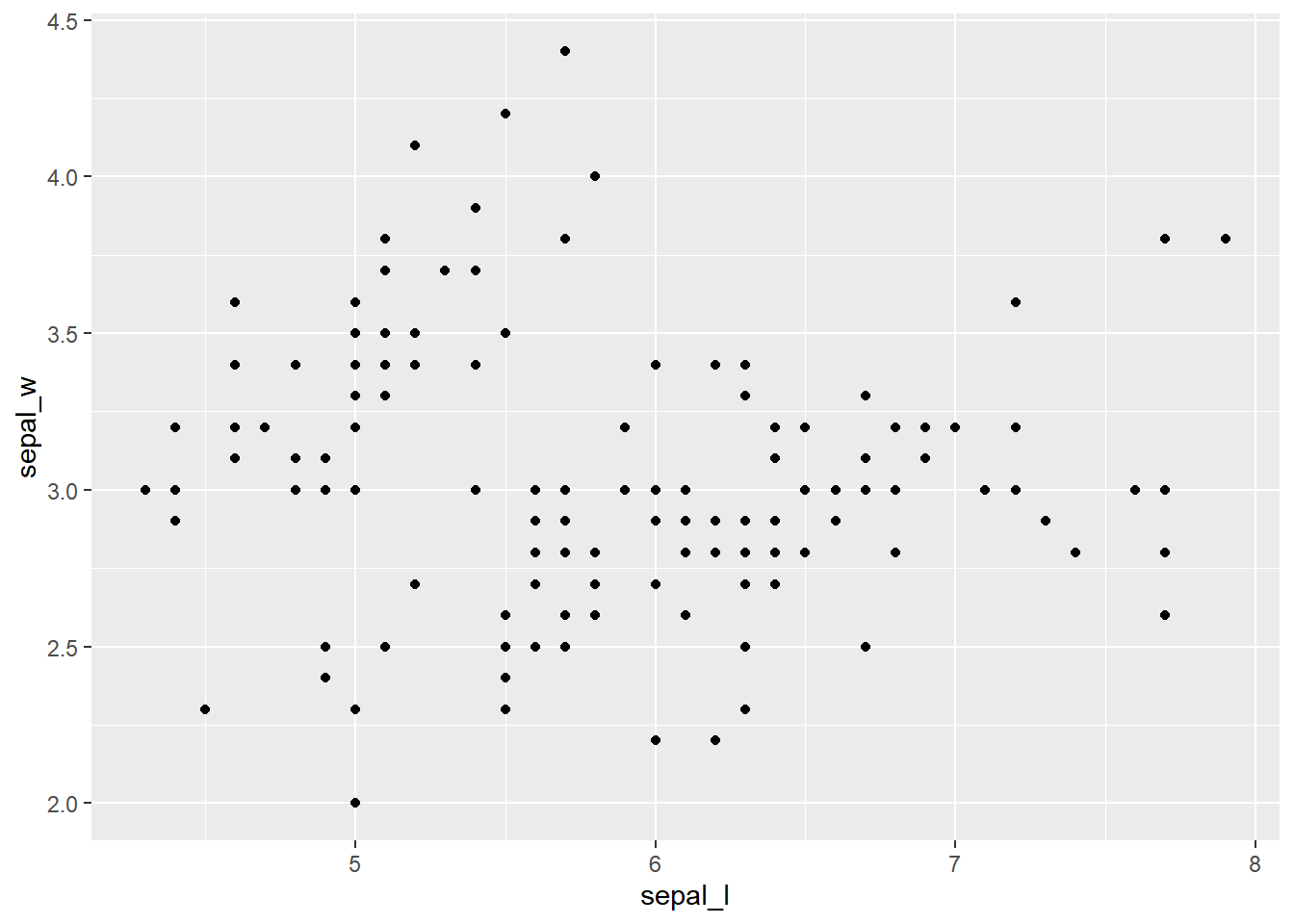
- A primeira camada é dada pela função ggplot() e recebe a nossos dados;
- A segunda camada é dada pela função geom_point(), especificando a forma geométrica utilizada no mapeamento das observações (pontos);
- As camadas são unidas com um +;
- O mapeamento na função geom_point() recebe a função aes(), responsável por descrever como as variáveis serão mapeadas nos aspectos visuais dos pontos (a forma geométrica escolhida);
- Neste caso, os aspectos visuais mapeados são a posição do ponto no eixo x e a posição do ponto no eixo y;
Podemos usar pipes:
Podemos criar um objeto ggplot e depois adicionar camadas. Aqui criamos mais uma camada, labs, com a qual podemos personalisar títulos, legendas, entre outros aspectos:
p <- ggplot(dados)
p + geom_point(aes(sepal_l, sepal_w,color= class))+
labs(title="Iris", x = "Comprimento das sépalas", y = "Largura das sépalas", color = "Espécies")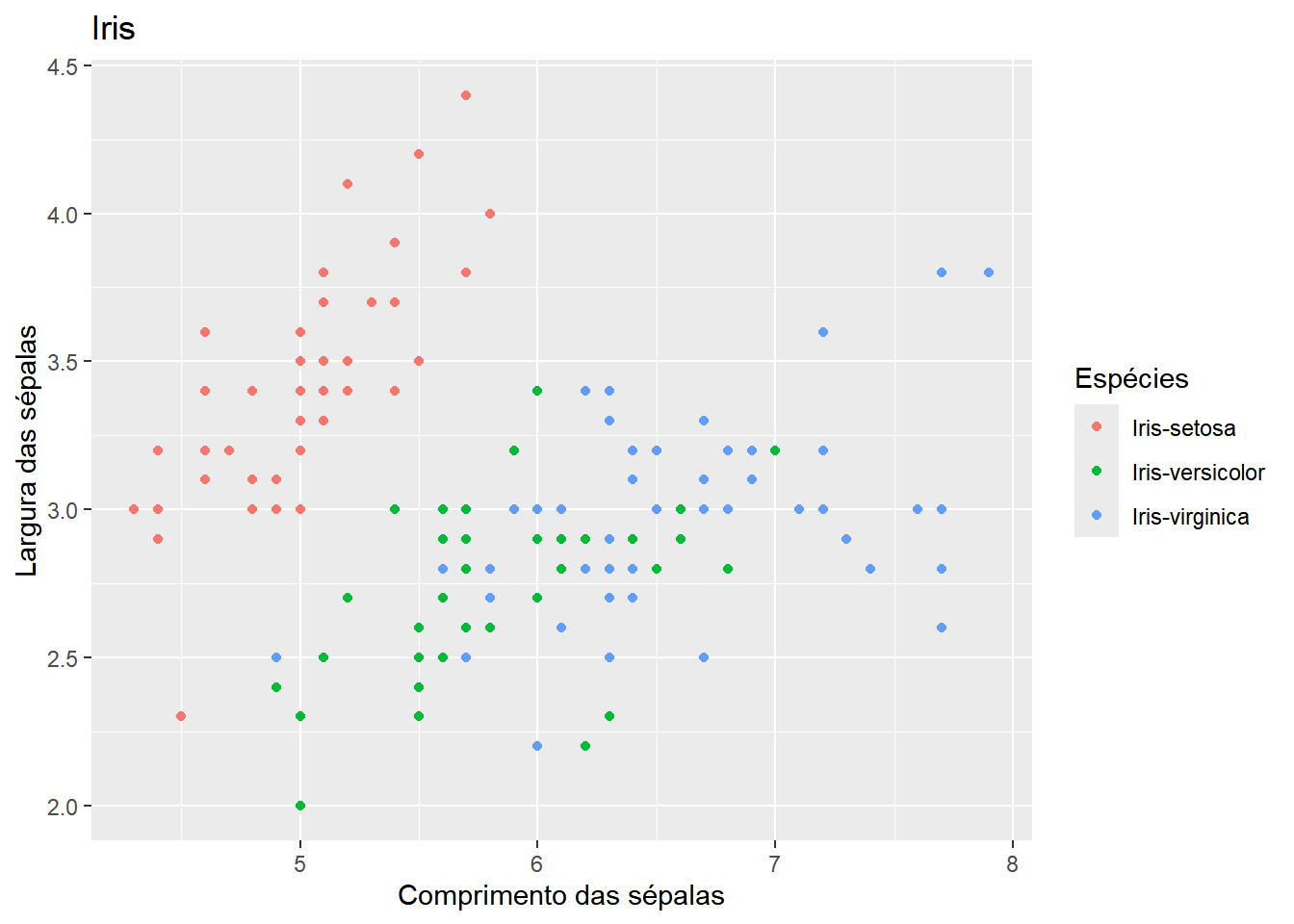
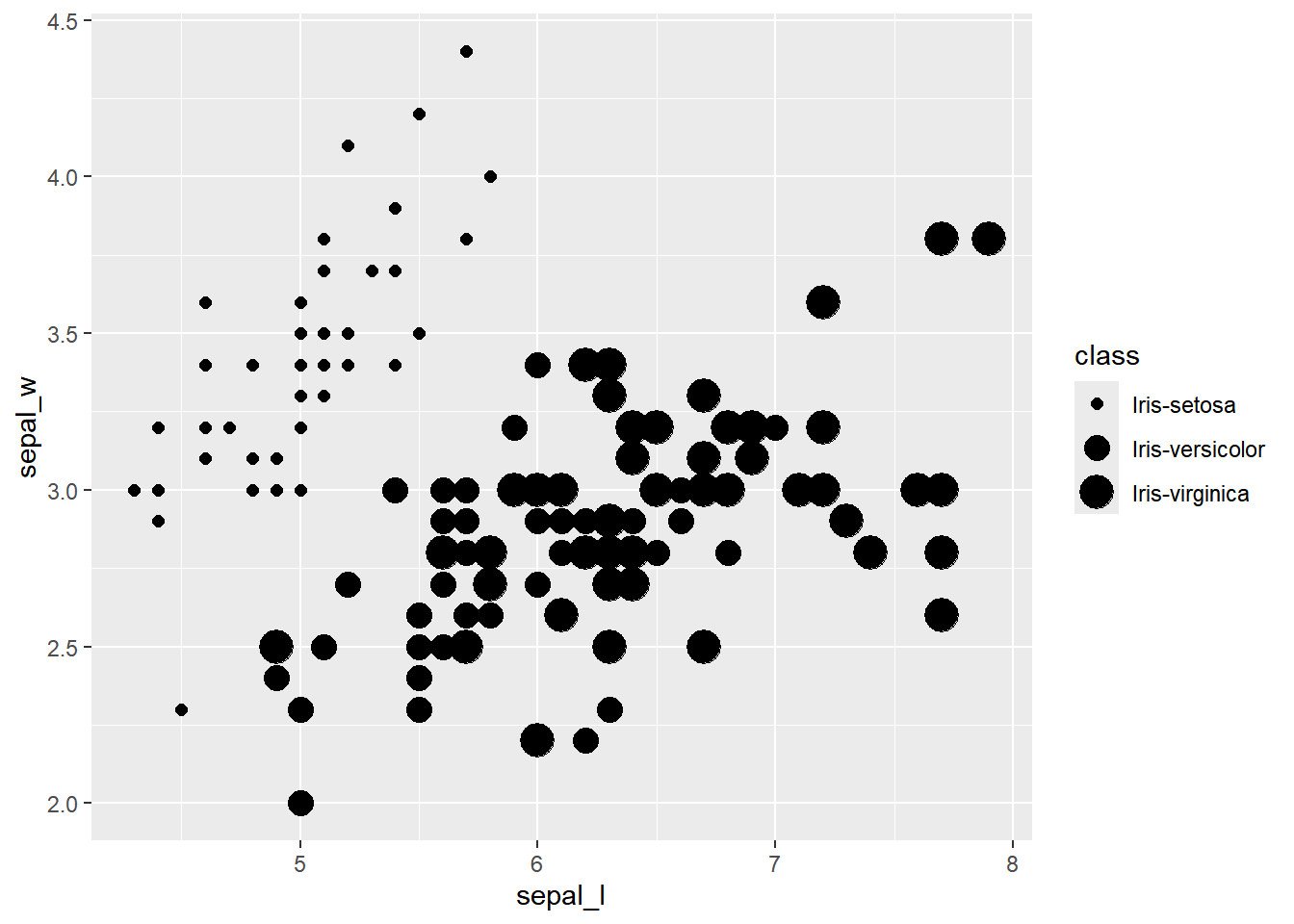
Também podemos alterar o tamanho:
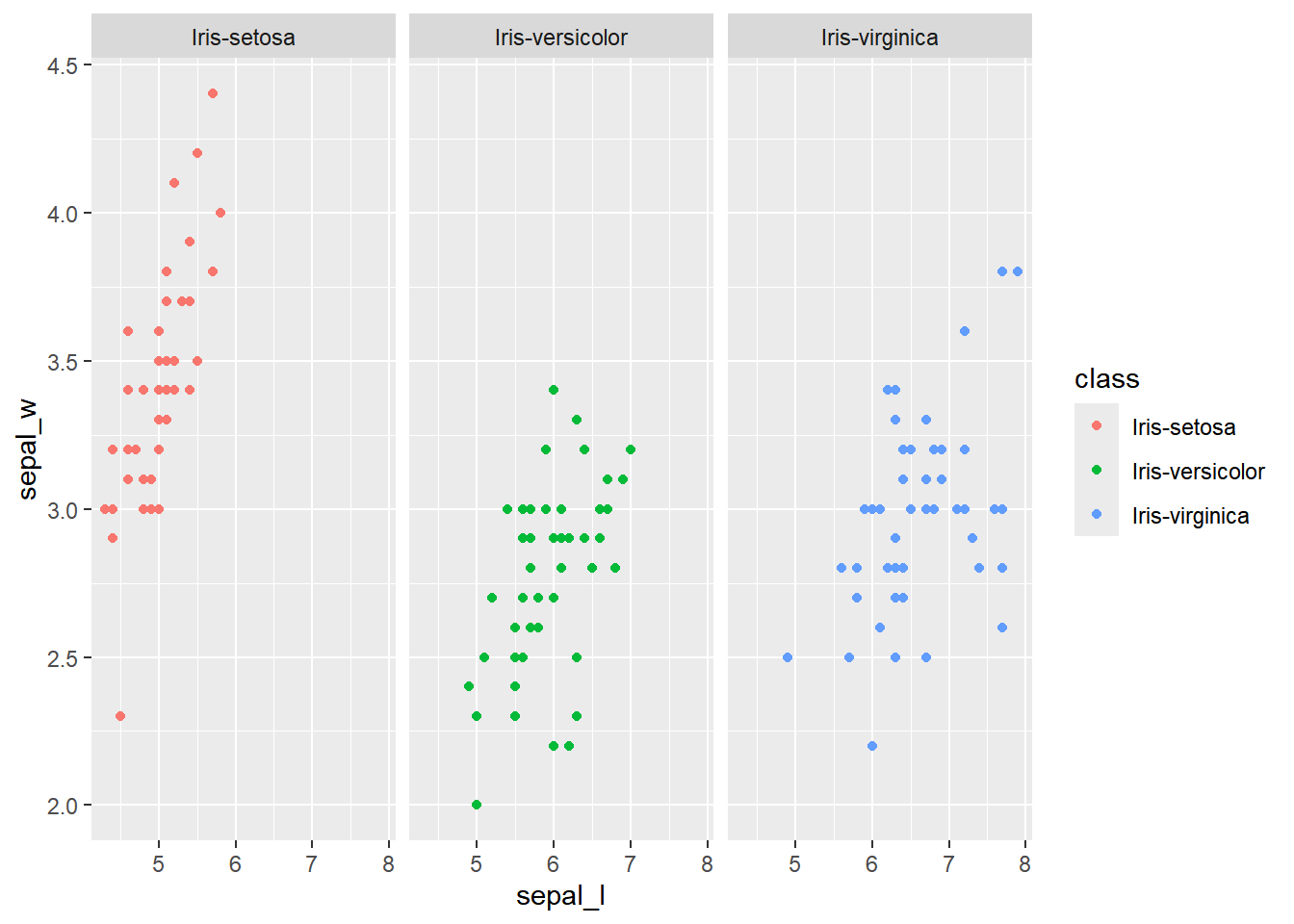
Podemos adicionar mais camadas, como facet_wrap, com a qual criamos mais painéis, com base em uma varável categórica:
4.2.2 Salvar
Para salvar usamos a função ggsave, que vai salvar o último gráfico gerado:
## Saving 7 x 5 in imageVamos carregar outra planilha de dados:
library(openxlsx)
dados2<-read.xlsx("dados/Data_Cortex_Nuclear.xlsx", sheet = 1, colNames = T)
head(dados2)## MouseID DYRK1A_N ITSN1_N BDNF_N NR1_N NR2A_N pAKT_N pBRAF_N
## 1 309_1 0.5036439 0.7471932 0.4301753 2.816329 5.990152 0.2188300 0.1775655
## 2 309_2 0.5146171 0.6890635 0.4117703 2.789514 5.685038 0.2116362 0.1728170
## 3 309_3 0.5091831 0.7302468 0.4183088 2.687201 5.622059 0.2090109 0.1757222
## 4 309_4 0.4421067 0.6170762 0.3586263 2.466947 4.979503 0.2228858 0.1764626
## 5 309_5 0.4349402 0.6174298 0.3588022 2.365785 4.718679 0.2131059 0.1736270
## 6 309_6 0.4475064 0.6281758 0.3673881 2.385939 4.807635 0.2185778 0.1762334
## pCAMKII_N pCREB_N pELK_N pERK_N pJNK_N PKCA_N pMEK_N
## 1 2.373744 0.2322238 1.750936 0.6879062 0.3063817 0.4026984 0.2969273
## 2 2.292150 0.2269721 1.596377 0.6950062 0.2990511 0.3859868 0.2813189
## 3 2.283337 0.2302468 1.561316 0.6773484 0.2912761 0.3810025 0.2817103
## 4 2.152301 0.2070042 1.595086 0.5832768 0.2967287 0.3770870 0.3138320
## 5 2.134014 0.1921579 1.504230 0.5509601 0.2869612 0.3635021 0.2779643
## 6 2.141282 0.1951875 1.442398 0.5663396 0.2898239 0.3638930 0.2668369
## pNR1_N pNR2A_N pNR2B_N pPKCAB_N pRSK_N AKT_N BRAF_N CAMKII_N
## 1 1.0220603 0.6056726 1.877684 2.308745 0.4415994 0.8593658 0.4162891 0.3696080
## 2 0.9566759 0.5875587 1.725774 2.043037 0.4452219 0.8346593 0.4003642 0.3561775
## 3 1.0036350 0.6024488 1.731873 2.017984 0.4676679 0.8143294 0.3998469 0.3680888
## 4 0.8753903 0.5202932 1.566852 2.132754 0.4776707 0.7277046 0.3856387 0.3629700
## 5 0.8649120 0.5079898 1.480059 2.013697 0.4834161 0.6877937 0.3675305 0.3553109
## 6 0.8591209 0.5213066 1.538244 1.968275 0.4959000 0.6724022 0.3694045 0.3571717
## CREB_N ELK_N ERK_N GSK3B_N JNK_N MEK_N TRKA_N RSK_N
## 1 0.1789443 1.866358 3.685247 1.537227 0.2645263 0.3196770 0.8138665 0.1658460
## 2 0.1736797 1.761047 3.485287 1.509249 0.2557270 0.3044187 0.7805042 0.1571935
## 3 0.1739047 1.765544 3.571456 1.501244 0.2596135 0.3117467 0.7851540 0.1608954
## 4 0.1794489 1.286277 2.970137 1.419710 0.2595358 0.2792181 0.7344917 0.1622099
## 5 0.1748355 1.324695 2.896334 1.359876 0.2507050 0.2736672 0.7026991 0.1548274
## 6 0.1797285 1.227450 2.956983 1.447910 0.2508402 0.2840436 0.7043958 0.1568759
## APP_N Bcatenin_N SOD1_N MTOR_N P38_N pMTOR_N DSCR1_N
## 1 0.4539098 3.037621 0.3695096 0.4585385 0.3353358 0.8251920 0.5769155
## 2 0.4309403 2.921882 0.3422793 0.4235599 0.3248347 0.7617176 0.5450973
## 3 0.4231873 2.944136 0.3436962 0.4250048 0.3248517 0.7570308 0.5436197
## 4 0.4106149 2.500204 0.3445093 0.4292113 0.3301208 0.7469798 0.5467626
## 5 0.3985498 2.456560 0.3291258 0.4087552 0.3134148 0.6919565 0.5368605
## 6 0.3910472 2.467133 0.3275978 0.4044899 0.2962764 0.6744186 0.5397231
## AMPKA_N NR2B_N pNUMB_N RAPTOR_N TIAM1_N pP70S6_N NUMB_N
## 1 0.4480993 0.5862714 0.3947213 0.3395706 0.4828639 0.2941698 0.1821505
## 2 0.4208761 0.5450973 0.3682546 0.3219592 0.4545193 0.2764306 0.1820863
## 3 0.4046298 0.5529941 0.3638799 0.3130859 0.4471972 0.2566482 0.1843877
## 4 0.3868603 0.5478485 0.3667707 0.3284919 0.4426497 0.3985340 0.1617677
## 5 0.3608164 0.5128240 0.3515510 0.3122063 0.4190949 0.3934470 0.1602002
## 6 0.3542143 0.5143164 0.3472241 0.3031321 0.4128243 0.3825783 0.1623303
## P70S6_N pGSK3B_N pPKCG_N CDK5_N S6_N ADARB1_N AcetylH3K9_N
## 1 0.8427252 0.1926084 1.443091 0.2947000 0.3546045 1.339070 0.1701188
## 2 0.8476146 0.1948153 1.439460 0.2940598 0.3545483 1.306323 0.1714271
## 3 0.8561658 0.2007373 1.524364 0.3018807 0.3860868 1.279600 0.1854563
## 4 0.7602335 0.1841694 1.612382 0.2963818 0.2906795 1.198765 0.1597991
## 5 0.7681129 0.1857183 1.645807 0.2968294 0.3093450 1.206995 0.1646503
## 6 0.7796946 0.1867930 1.634615 0.2880373 0.3323671 1.123445 0.1756929
## RRP1_N BAX_N ARC_N ERBB4_N nNOS_N Tau_N GFAP_N
## 1 0.1591024 0.1888517 0.1063052 0.1449893 0.1766677 0.1251904 0.1152909
## 2 0.1581289 0.1845700 0.1065922 0.1504709 0.1783090 0.1342751 0.1182345
## 3 0.1486963 0.1905322 0.1083031 0.1453302 0.1762129 0.1325604 0.1177602
## 4 0.1661123 0.1853235 0.1031838 0.1406558 0.1638042 0.1232096 0.1174394
## 5 0.1606870 0.1882214 0.1047838 0.1419830 0.1677096 0.1368377 0.1160478
## 6 0.1505939 0.1838235 0.1064762 0.1395645 0.1748445 0.1305147 0.1152432
## GluR3_N GluR4_N IL1B_N P3525_N pCASP9_N PSD95_N SNCA_N
## 1 0.2280435 0.1427556 0.4309575 0.2475378 1.603310 2.014875 0.1082343
## 2 0.2380731 0.1420366 0.4571562 0.2576322 1.671738 2.004605 0.1097485
## 3 0.2448173 0.1424450 0.5104723 0.2553430 1.663550 2.016831 0.1081962
## 4 0.2349467 0.1450682 0.4309959 0.2511031 1.484624 1.957233 0.1198832
## 5 0.2555277 0.1408705 0.4812265 0.2517730 1.534835 2.009109 0.1195244
## 6 0.2368495 0.1364536 0.4785775 0.2444853 1.507777 2.003535 0.1206872
## Ubiquitin_N pGSK3B_Tyr216_N SHH_N BAD_N BCL2_N pS6_N pCFOS_N
## 1 1.0449792 0.8315565 0.1888517 0.1226520 NA 0.1063052 0.1083359
## 2 1.0098831 0.8492704 0.2004036 0.1166822 NA 0.1065922 0.1043154
## 3 0.9968476 0.8467087 0.1936845 0.1185082 NA 0.1083031 0.1062193
## 4 0.9902247 0.8332768 0.1921119 0.1327812 NA 0.1031838 0.1112620
## 5 0.9977750 0.8786678 0.2056042 0.1299541 NA 0.1047838 0.1106939
## 6 0.9201782 0.8436793 0.1904695 0.1315752 NA 0.1064762 0.1094457
## SYP_N H3AcK18_N EGR1_N H3MeK4_N CaNA_N Genotype Treatment Behavior
## 1 0.4270992 0.1147832 0.1317900 0.1281856 1.675652 Control Memantine C/S
## 2 0.4415813 0.1119735 0.1351030 0.1311187 1.743610 Control Memantine C/S
## 3 0.4357769 0.1118829 0.1333618 0.1274311 1.926427 Control Memantine C/S
## 4 0.3916910 0.1304053 0.1474442 0.1469011 1.700563 Control Memantine C/S
## 5 0.4341538 0.1184814 0.1403143 0.1483799 1.839730 Control Memantine C/S
## 6 0.4398331 0.1166572 0.1407664 0.1421804 1.816389 Control Memantine C/S
## class
## 1 c-CS-m
## 2 c-CS-m
## 3 c-CS-m
## 4 c-CS-m
## 5 c-CS-m
## 6 c-CS-mEsses dados podem ser baixados do seguinte endereço:
https://archive.ics.uci.edu/dataset/342/mice+protein+expression
(Higuera and Cios 2015)
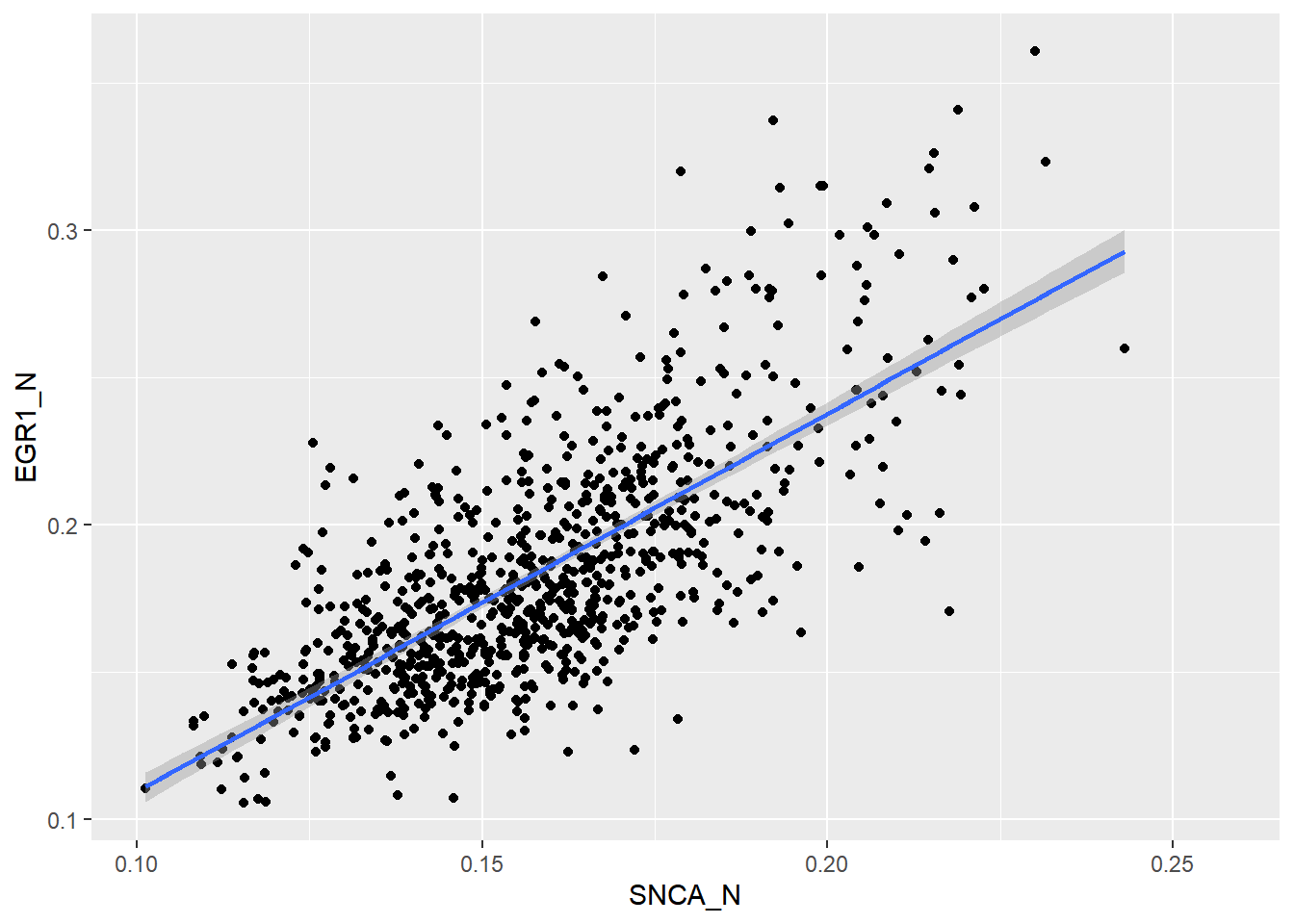
Podemos selecionar algumas colunas e criar um objeto ggplot:
p <- dados2 %>%
select(DYRK1A_N:pAKT_N,Genotype:class)%>%
ggplot(aes(DYRK1A_N, ITSN1_N, color = Genotype))E criar um gráfico de dispersão:
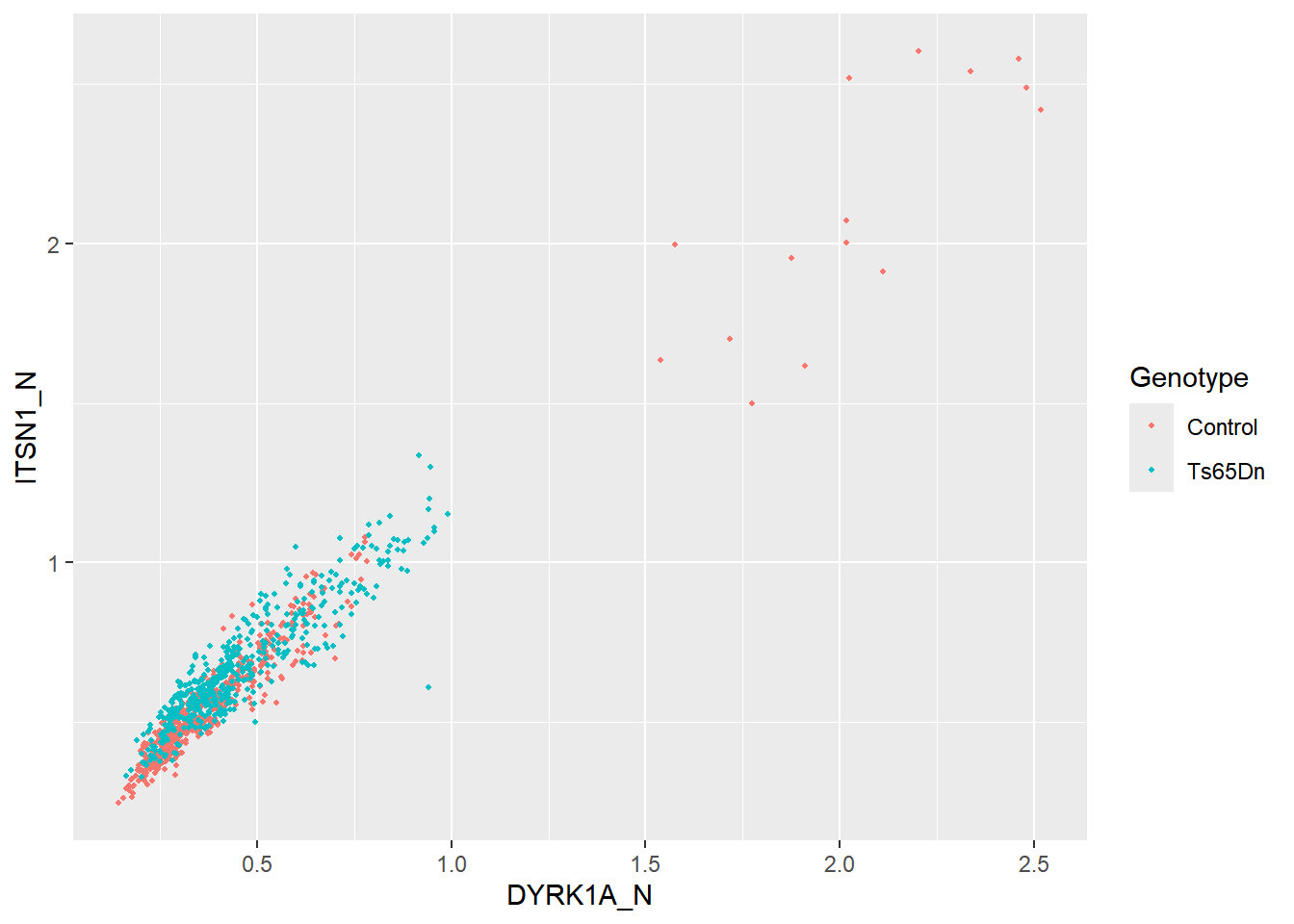
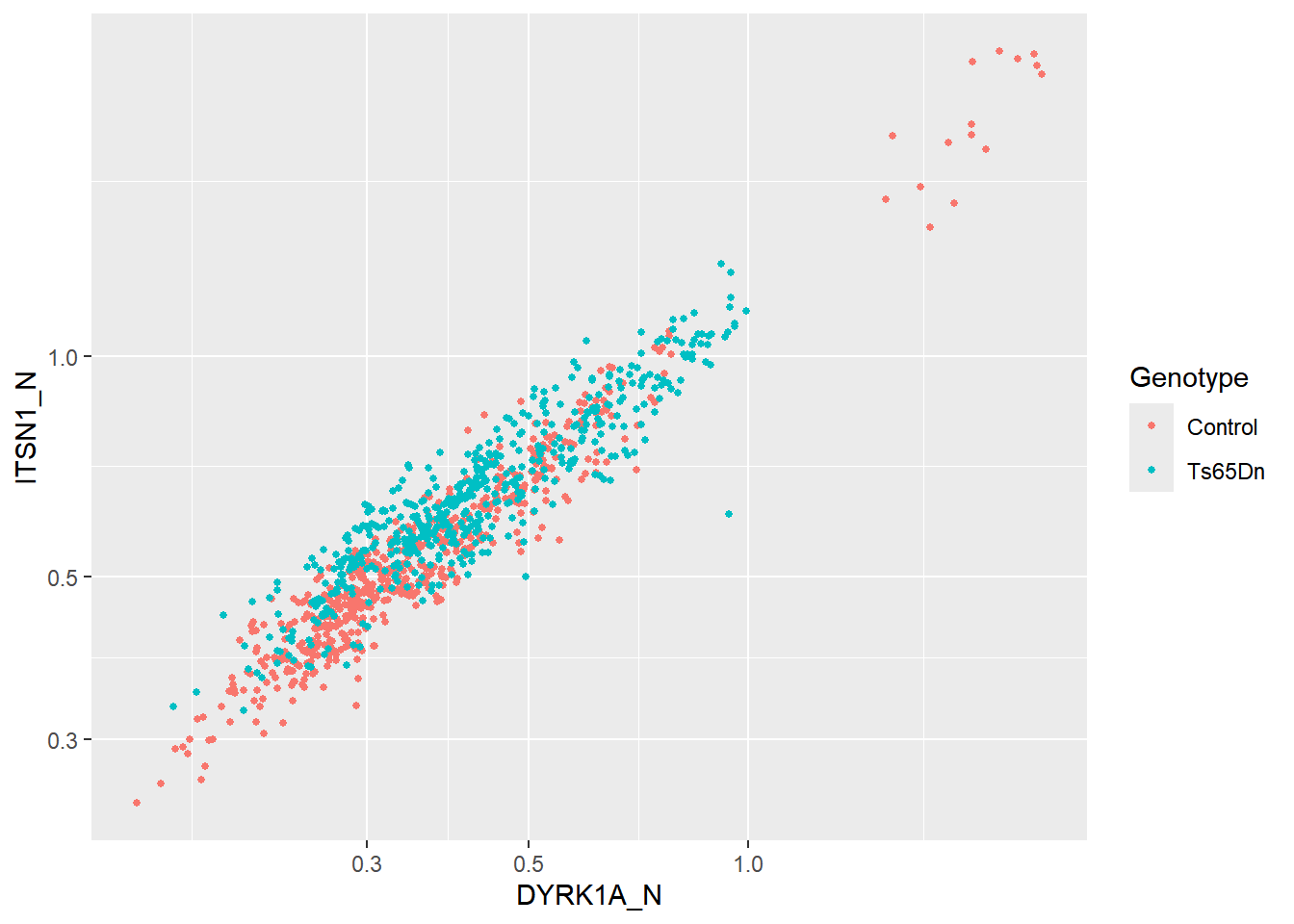
Podemos mudar as escalas dos eixos x e y. Por exemplo, vamos usar uma escala logarítmica (log na base 10):
4.2.3 Histogramas
Criando um objeto ggplot:
Histograma básico:
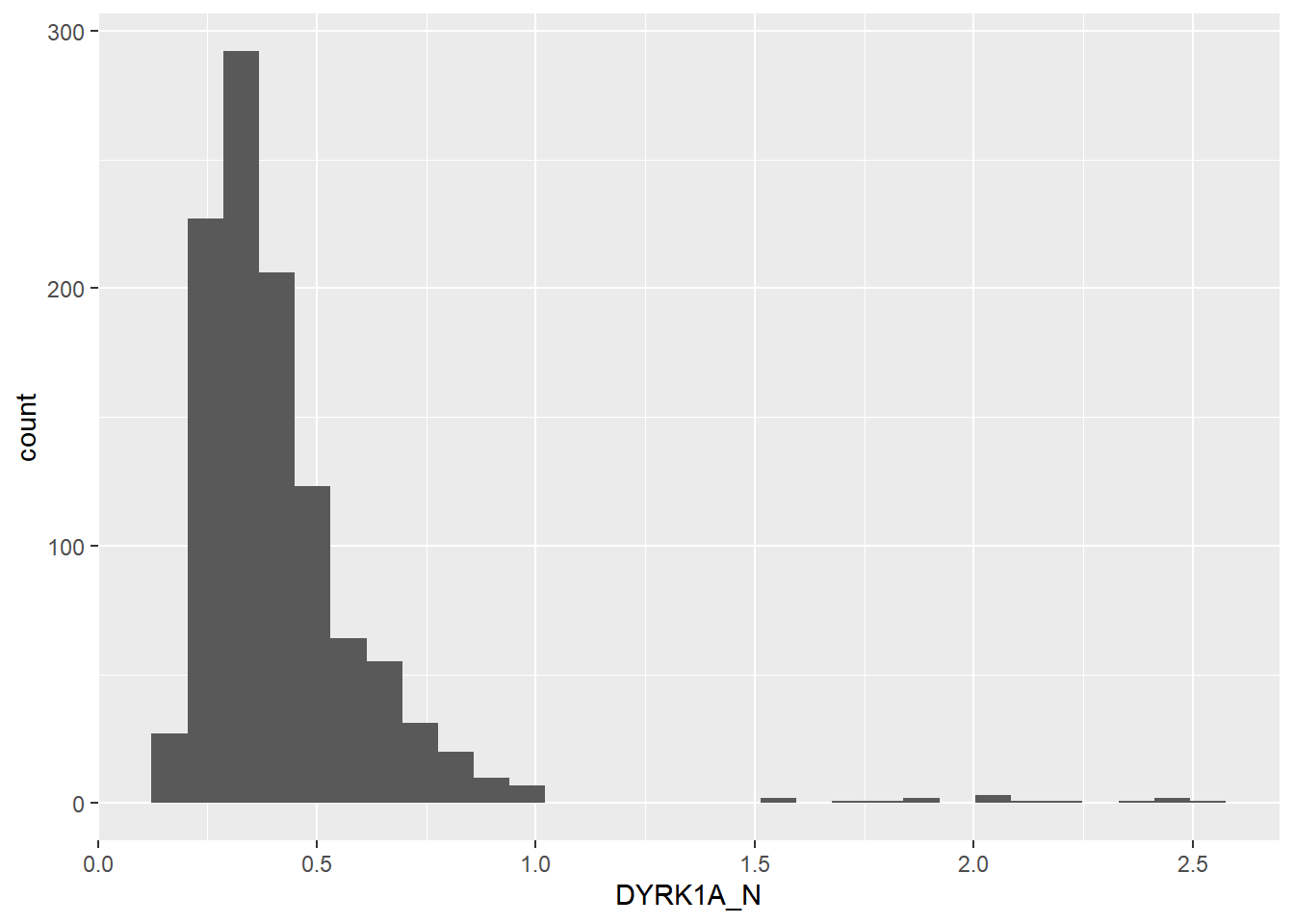
Personalizando a cor de preenchimento, cor da linha e legendas:
p + geom_histogram(binwidth = 0.1, fill = "blue", col = "black") +
xlab("expressão") +
ggtitle("Histograma")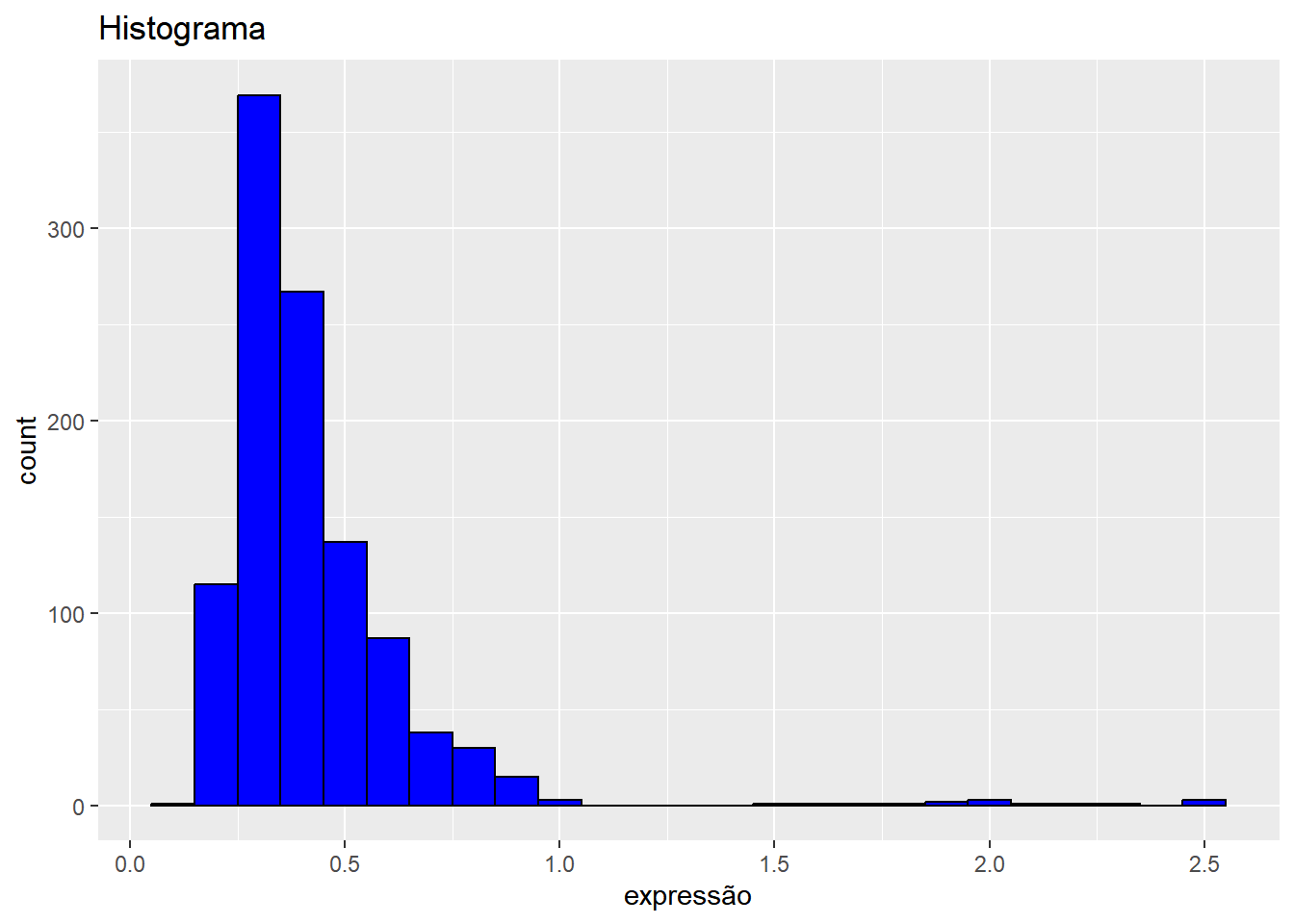
4.2.4 Density plot
Um gráfico de densidade é uma representação da distribuição de uma variável numérica. É uma versão suavizada do histograma e é usado no mesmo conceito.
Podemos usar cores:
Transparência
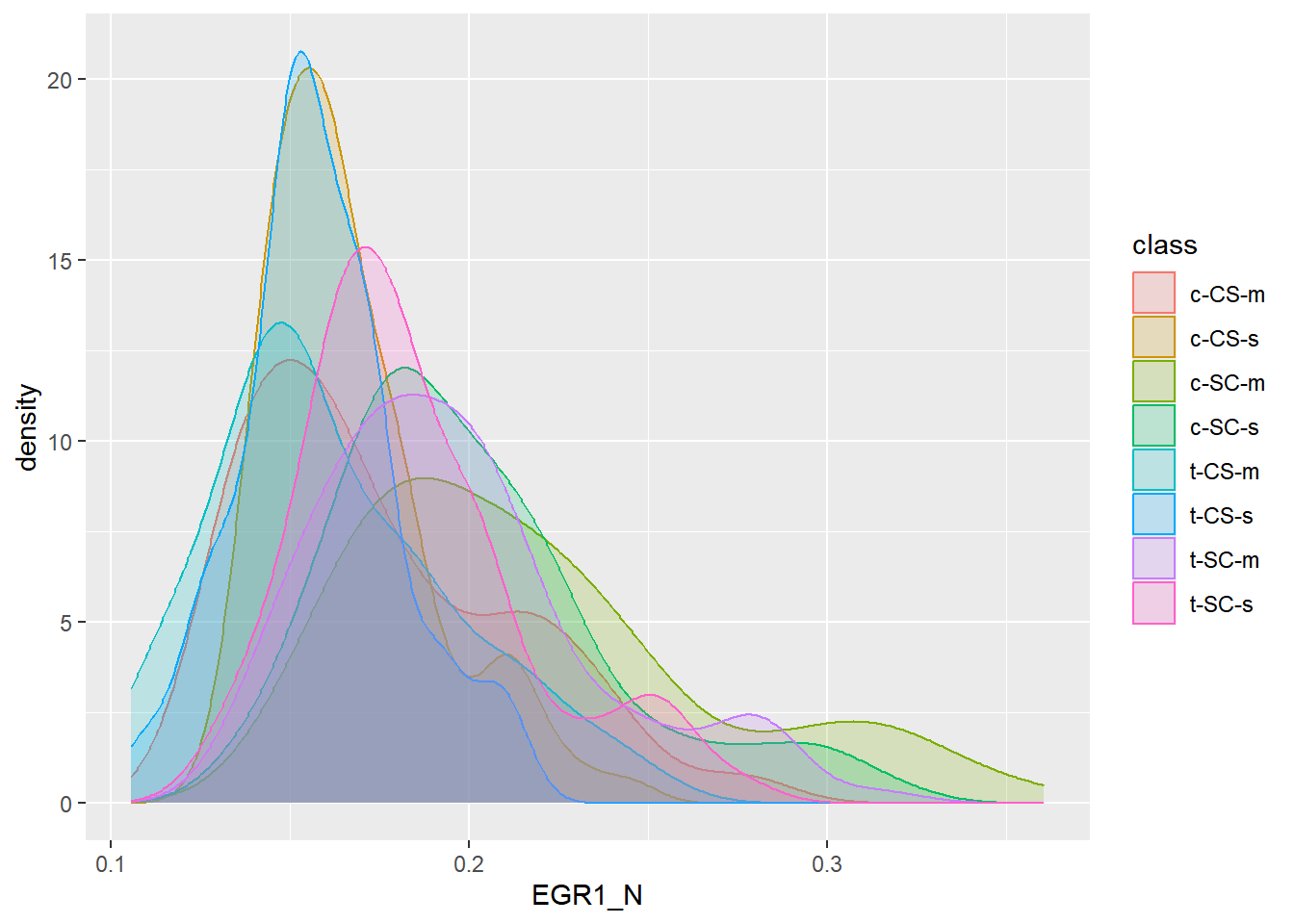
4.2.5 Boxplot
O boxplot é um gráfico onde a parte central contém os valores que estão entre o primeiro quartil e o terceiro quartil. As hastes inferiores e superiores se estendem, respectivamente, do primeiro quartil até o menor valor, limite inferior, e do terceiro quartil até o maior valor.
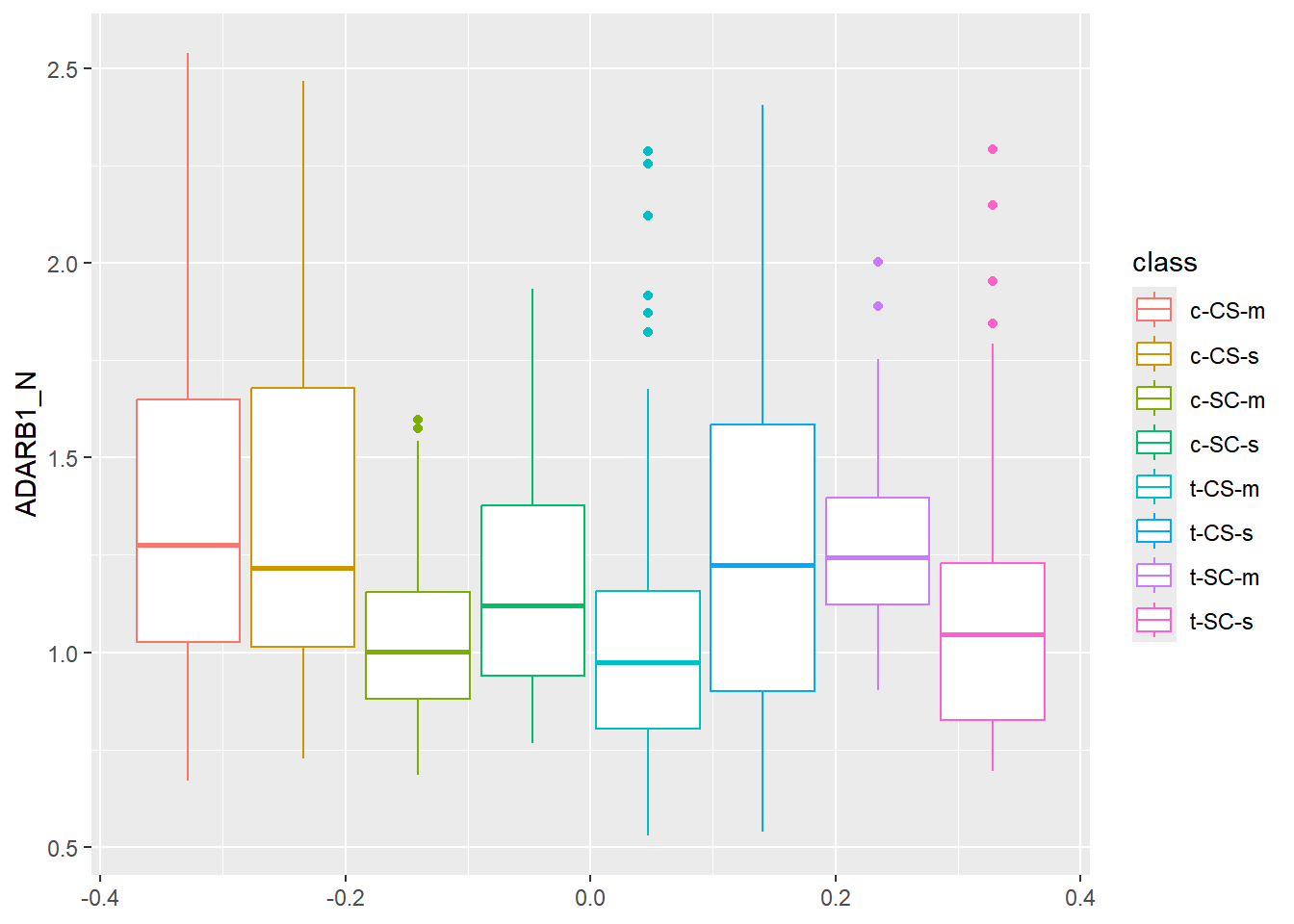
4.2.6 Gráfico Q-Q
O gráfico Q-Q é um gráfico de probabilidades, usado para comparar duas distribuições de probabilidade, traçando seus quantis uns contra os outros.
QQ-plot básico no ggplot2:
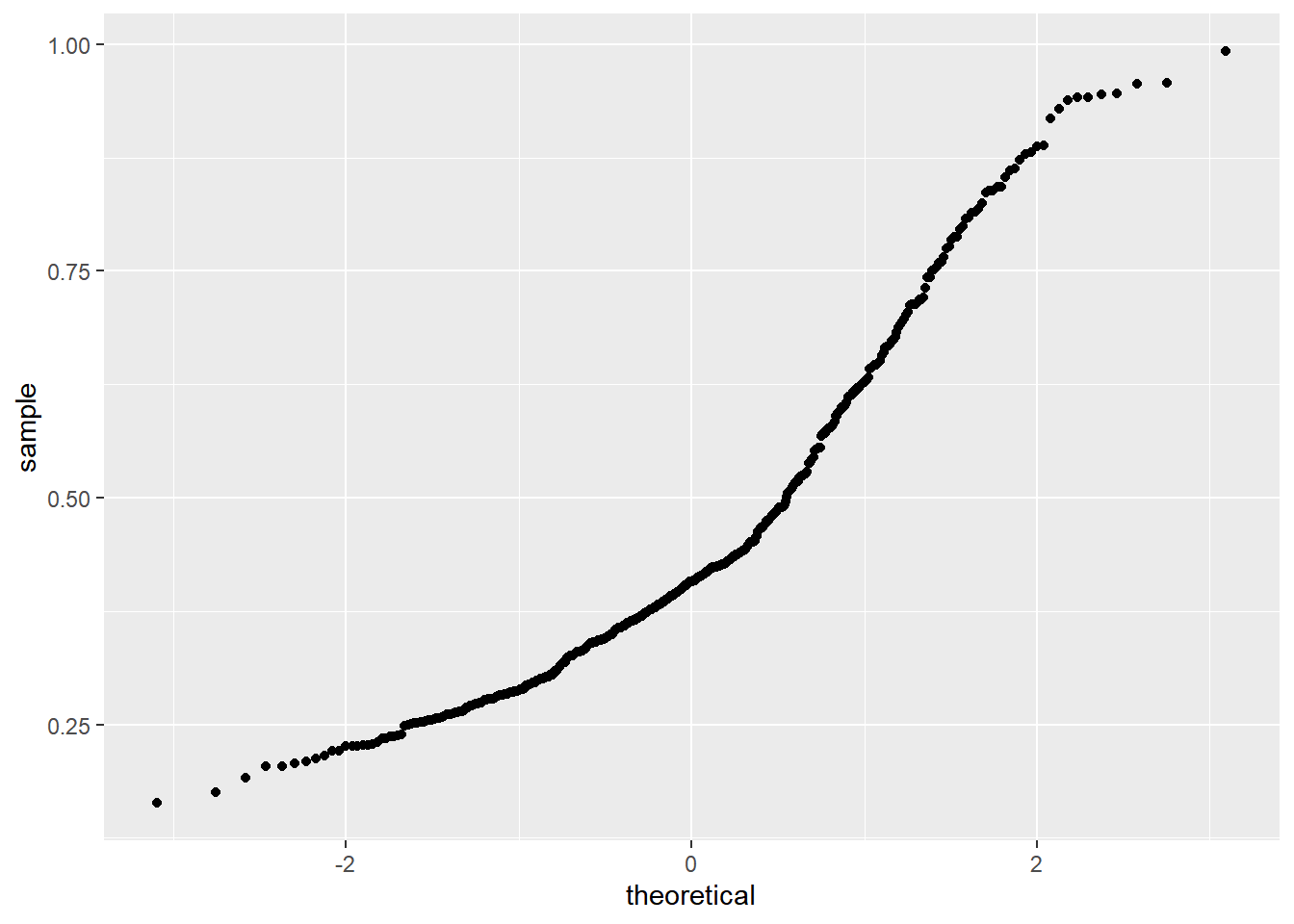
Podemos fazer um QQ-plot contra uma distribuição normal com a mesma média e desvio padrão:
params <- dados2 %>%
filter(Genotype =="Ts65Dn")%>%
summarize(mean = mean(DYRK1A_N, na.rm = T), sd = sd(DYRK1A_N, na.rm=T))
p + geom_qq(dparams = params) +
geom_abline()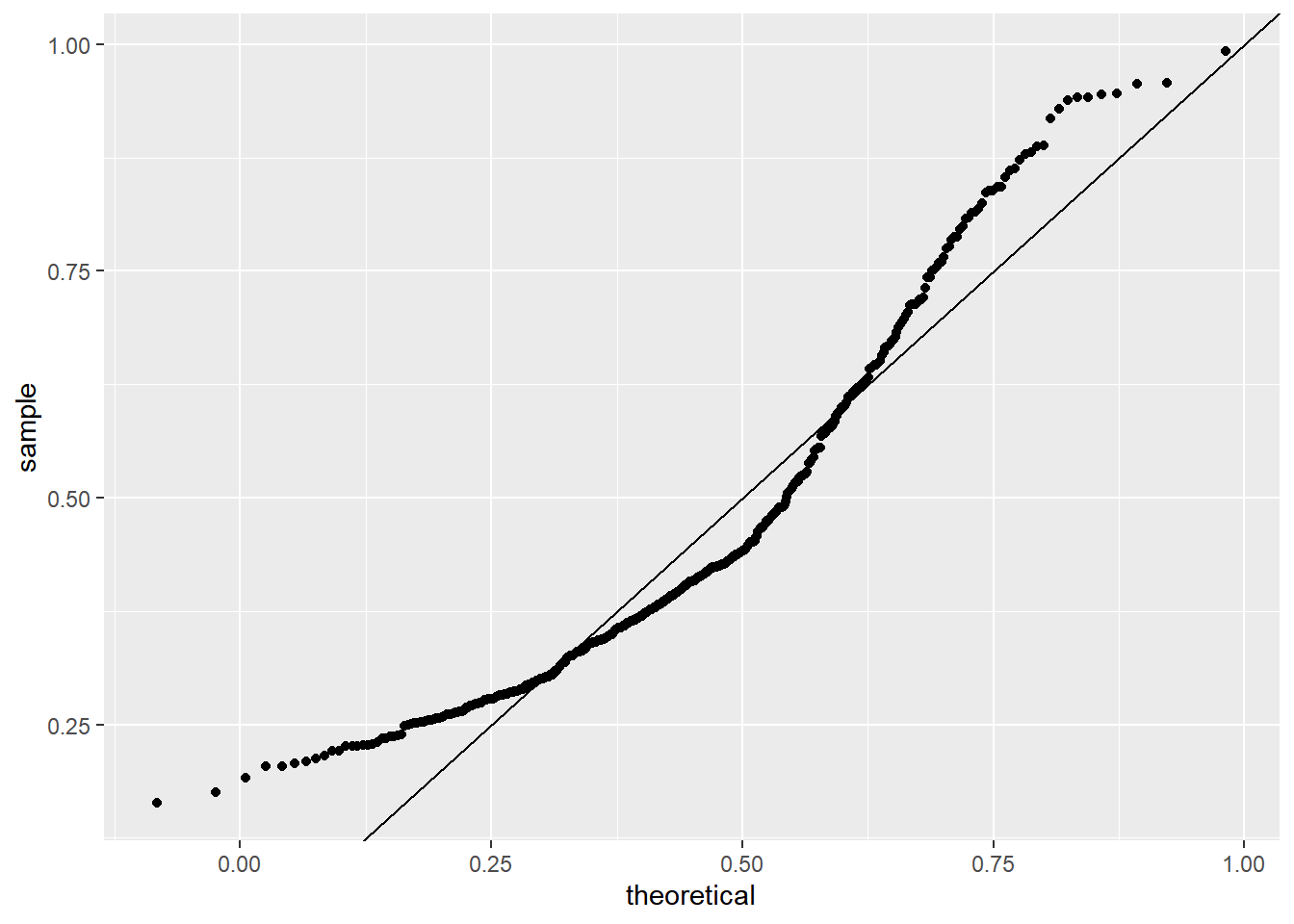
4.2.7 Figura com vários gráficos
Para juntar vários gráficos do ggplot2 em uma imagem usamos o pacote gridExtra
Vamos primeiro criar os gráficos e armazer em p1, p2, p3:
p <- dados2 %>%
ggplot(aes(x = DYRK1A_N))
p1 <- p + geom_histogram(binwidth = 0.1, fill = "blue", col = "black")
p2 <- p + geom_histogram(binwidth = 0.2, fill = "red", col = "black")
p3 <- p + geom_histogram(binwidth = 0.3, fill = "green", col = "black")Então usamos o pacote gridExtra (Auguie 2017) para compor a imagem. Nesse exemplo, vamos dispor as imagens em uma linha e três colunas:
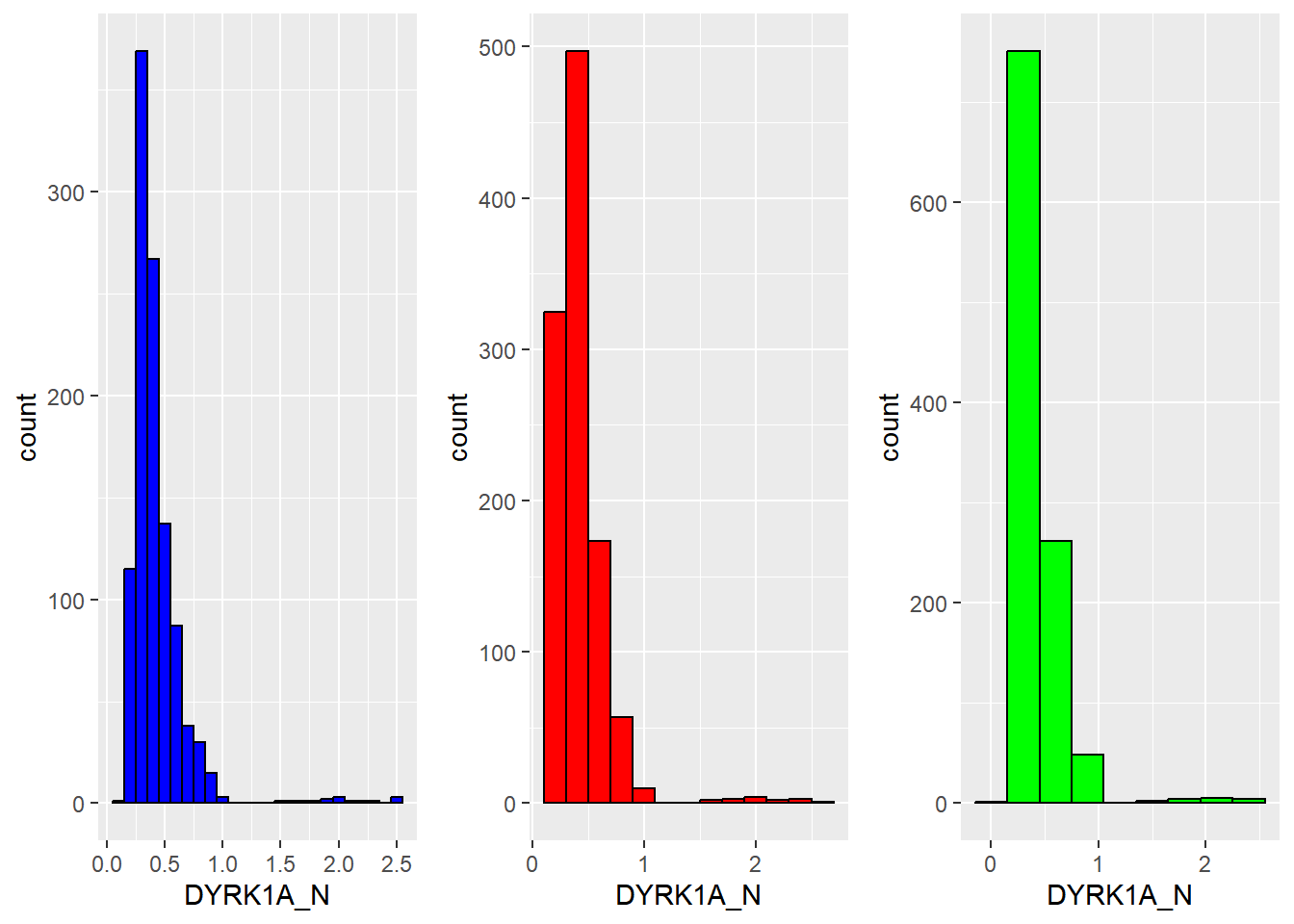
4.2.8 Ajustes de Posição
position = “identity” irá colocar cada objeto na posição exata em que ele cairia no contexto do gráfico
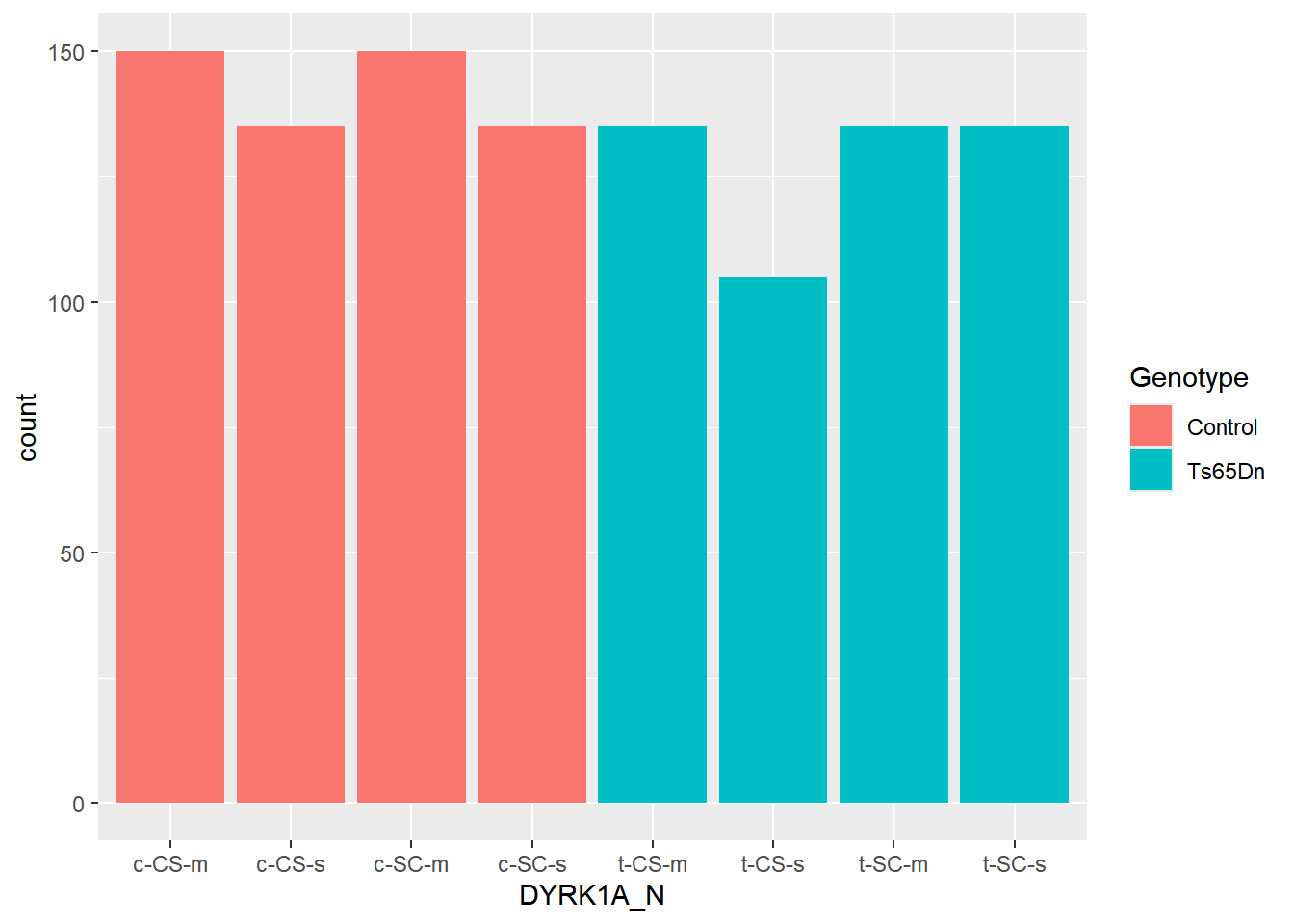
position = “dodge” coloca objetos sobrepostos um ao lado do outro. Isto torna mais fácil a comparação de valores individuais
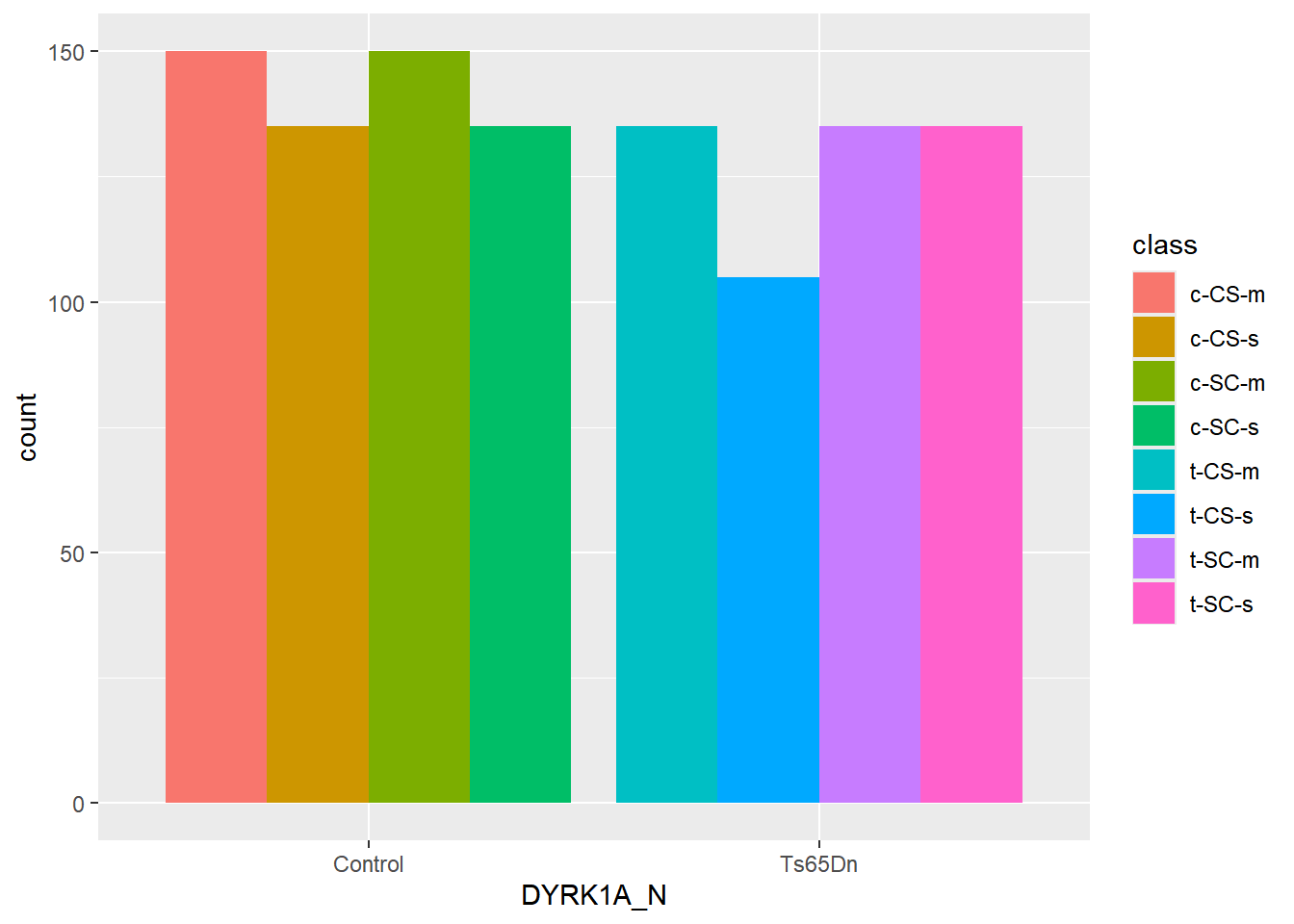
position = “fill” irá empilhar os elementos um sobre o outro, mas normalizando a altura. Isso é muito útil para comparar proporções entre os grupos
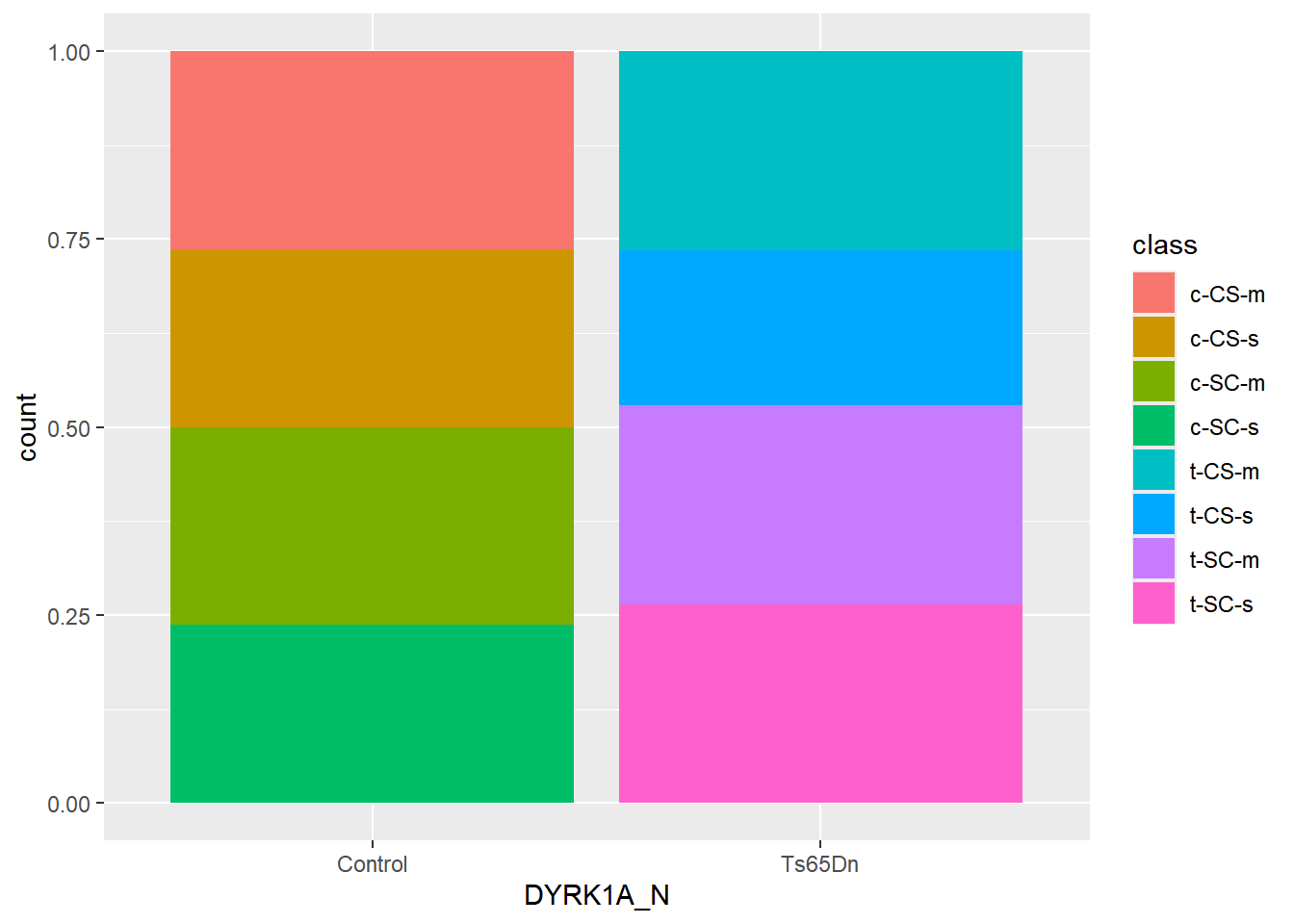
4.3 Outros pacotes gráficos
Vamos carregar os dados e ativar os pacotes:
library(ggplot2)
library(RColorBrewer)
library(openxlsx)
library(dplyr)
dados<-read.xlsx("dados/pinguim.xlsx", sheet = 1, colNames = T)
head(dados)## species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g
## 1 Adelie Torgersen 39.1 18.7 181 3750
## 2 Adelie Torgersen 39.5 17.4 186 3800
## 3 Adelie Torgersen 40.3 18.0 195 3250
## 4 Adelie Torgersen NA NA NA NA
## 5 Adelie Torgersen 36.7 19.3 193 3450
## 6 Adelie Torgersen 39.3 20.6 190 3650
## sex year
## 1 male 2007
## 2 female 2007
## 3 female 2007
## 4 <NA> 2007
## 5 female 2007
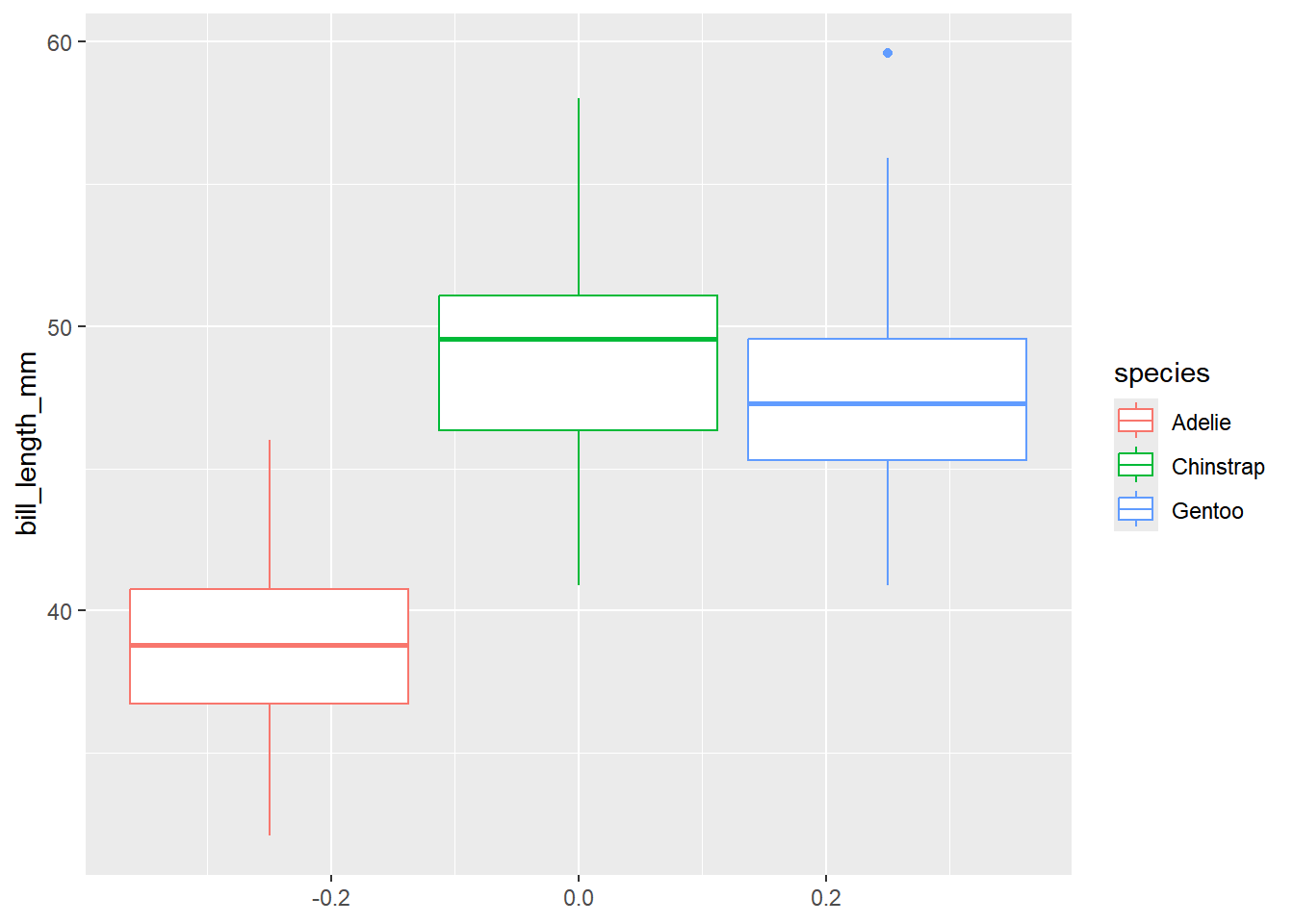
## 6 male 20074.3.1 Usando as cores
Primeiro vamos fazer um gráfico, sem especificar quais cores usar:
No ggplot2, é possível armazenar a paleta de cores em uma variável e usá-la posteriormente.
Por exemplo:
cbPalette <- c("#999999", "#E69F00", "#56B4E9", "#009E73", "#F0E442", "#0072B2", "#D55E00", "#CC79A7")Então podemos usar essas cores no gráfico:
p <- dados %>%
ggplot(aes(y = bill_length_mm, group=species, fill=species))
p + geom_boxplot()+
scale_fill_manual(values=cbPalette)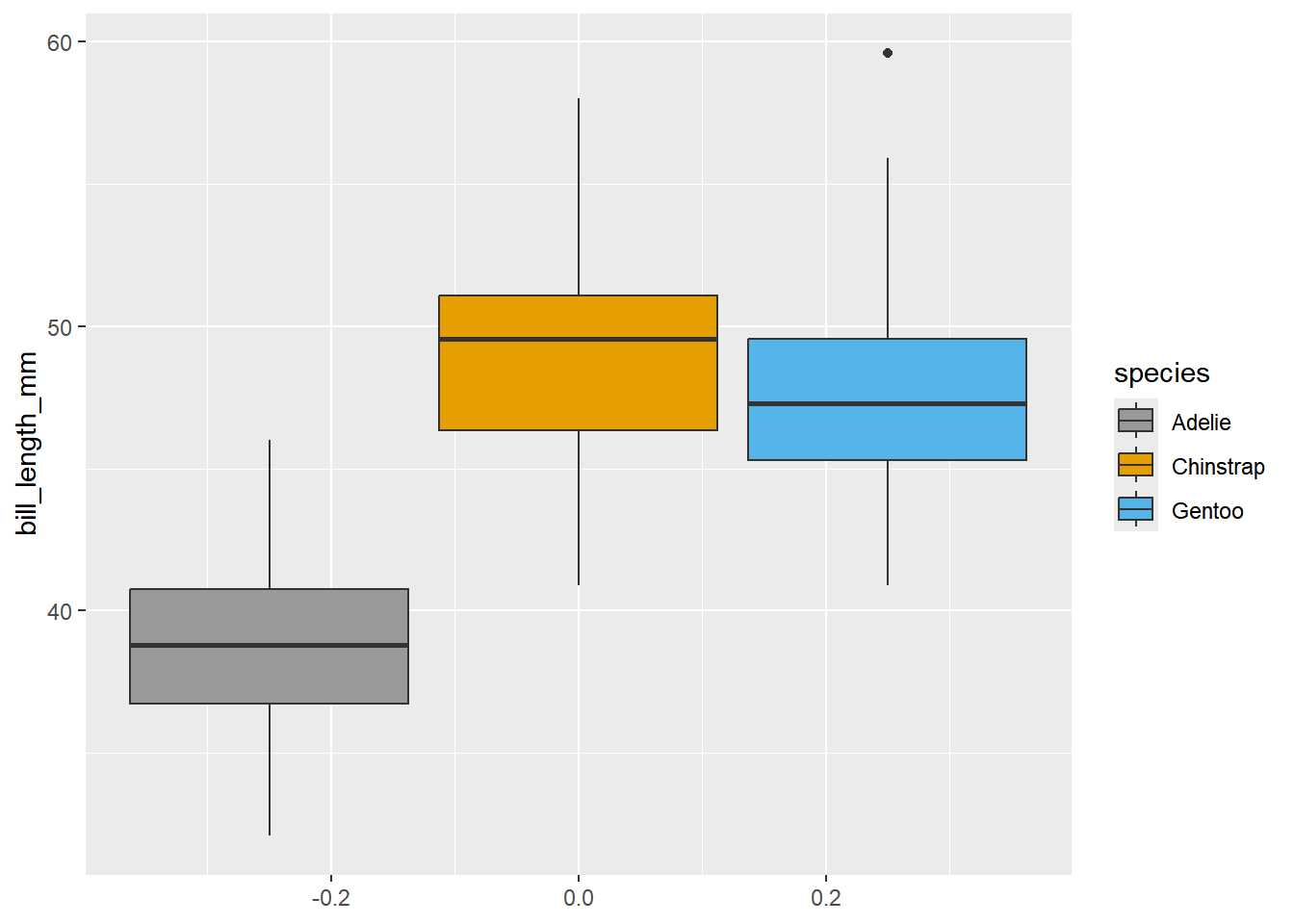
Para o preenchimento adicionamos:
scale_fill_manual(values=cbPalette)
Para cor da linha ou pontos adicionamos:
scale_colour_manual(values=cbPalette)
Também podemos usar outras escalas de cores, como as retiradas do pacote RColorBrewer
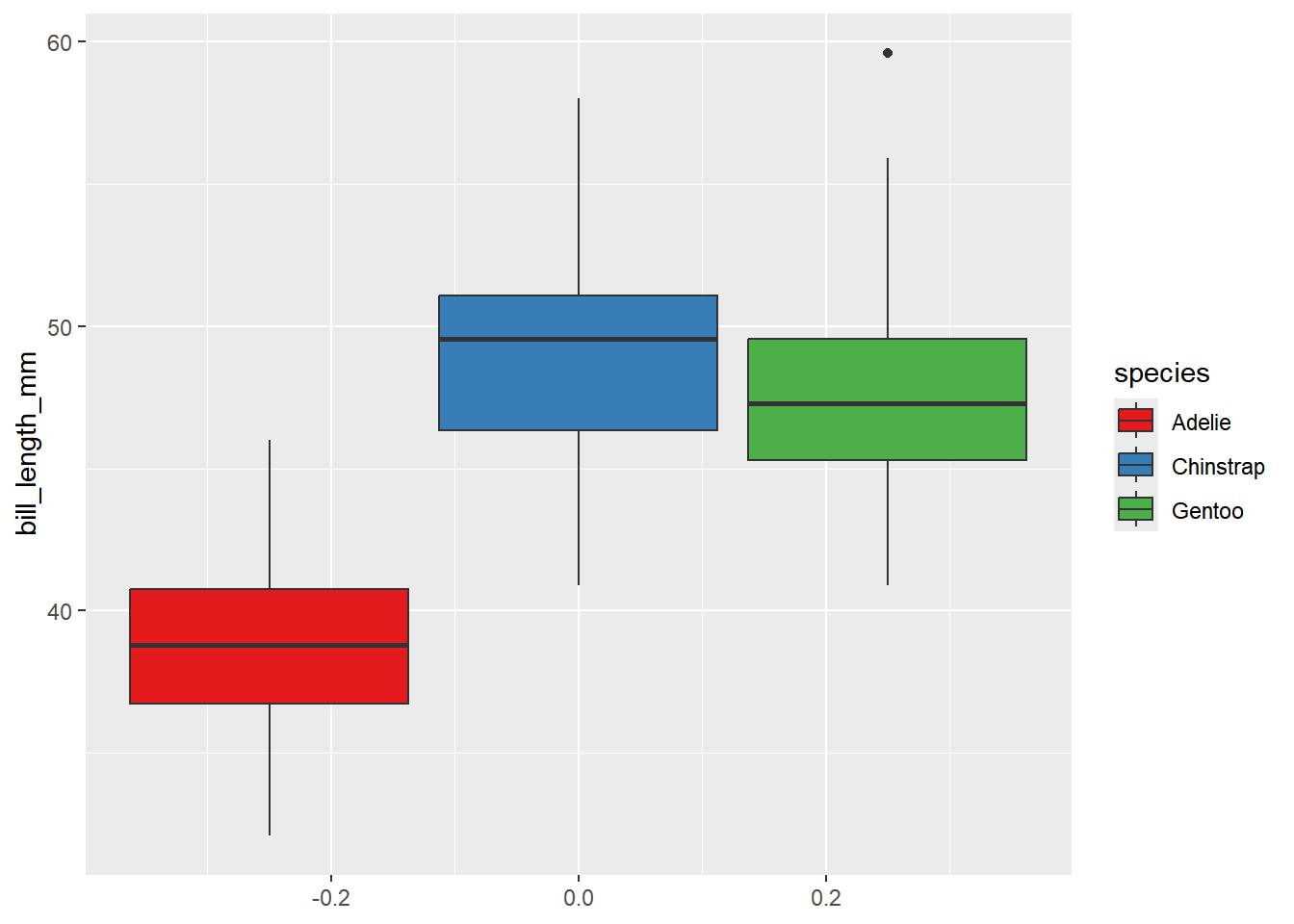
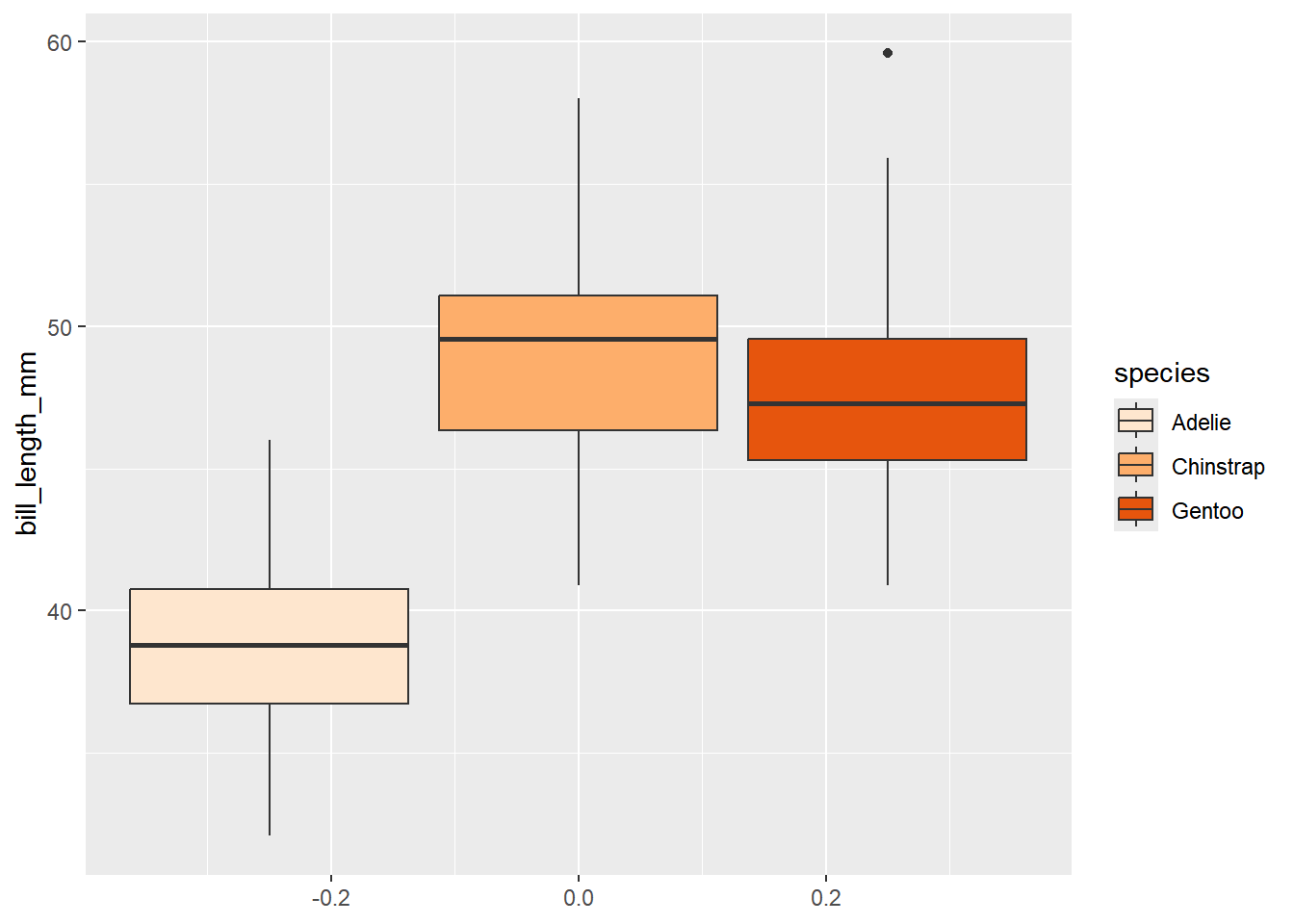
Podem consultar as opções em: https://r-graph-gallery.com/38-rcolorbrewers-palettes.html
4.3.2 Análise gráfica exploratória
Para isso usaremos o pacote GGally (Schloerke et al. 2021), que é uma extensão para o ggplot2.
Vamos usar a função ggpairs e selecionar uma variável categórica para color:
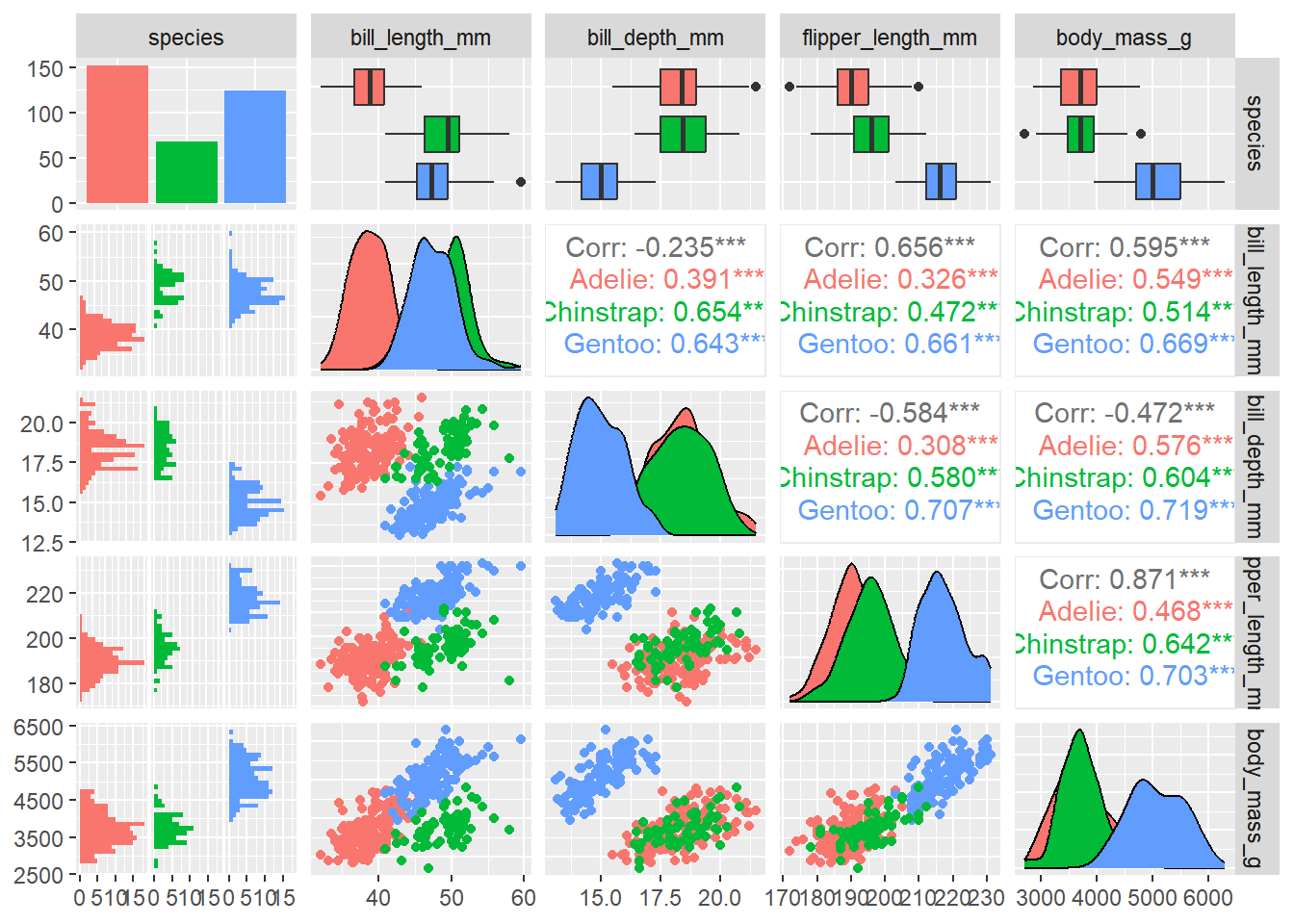
4.3.3 GGPLOT2: mais parâmetros
Vamos usar outro geom, o geom_segment:
ggplot(dados, aes(x=bill_length_mm , y=bill_depth_mm )) +
geom_segment( aes(x=bill_length_mm , xend=bill_length_mm , y=10, yend=bill_depth_mm), color="grey") +
geom_point(color="orange", size=4) +
theme_light() +
theme(
panel.grid.major.x = element_blank(),
panel.border = element_blank(),
axis.ticks.x = element_blank()
) +
xlab("") +
ylab("Value of Y")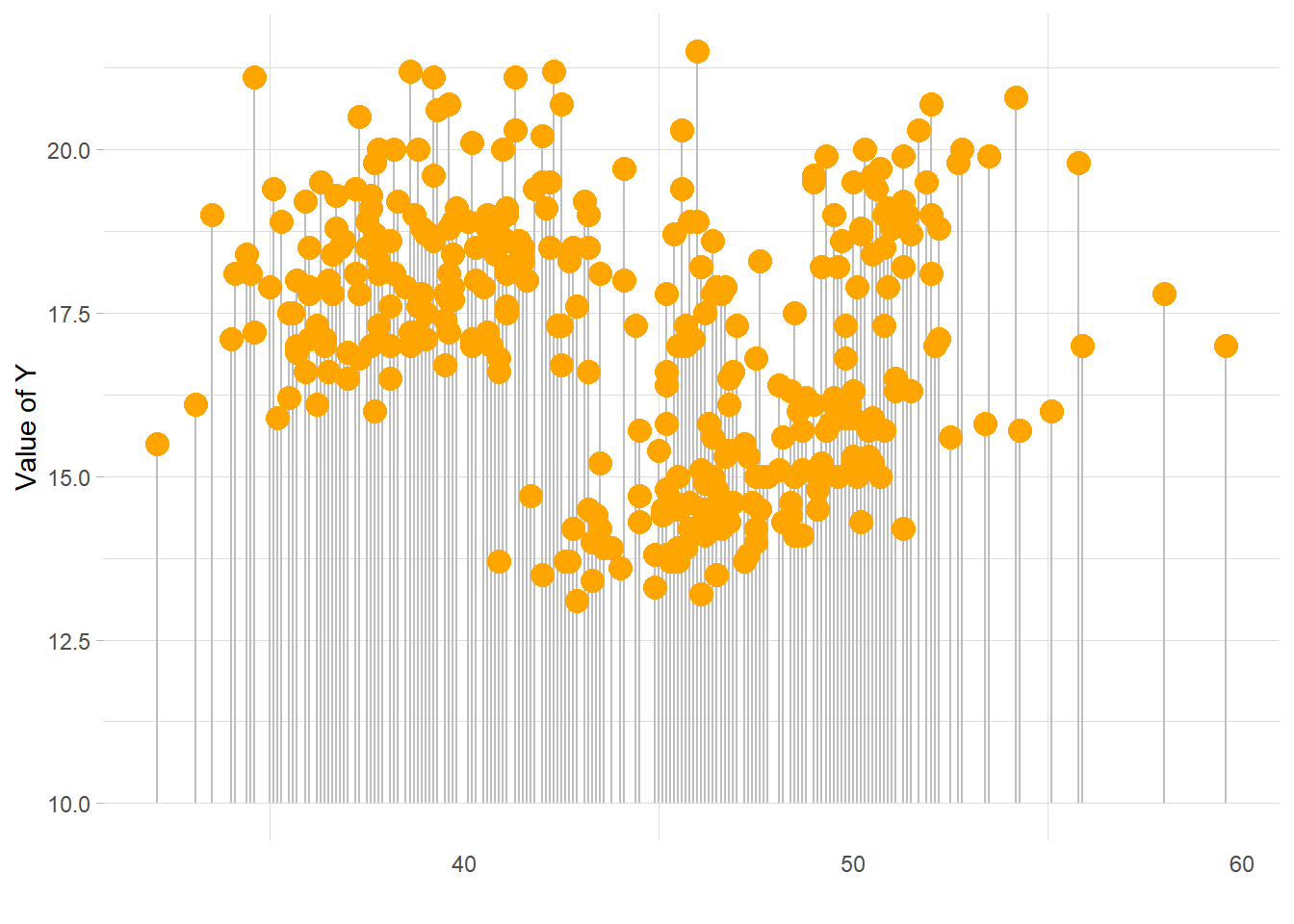
4.3.4 Diagrama de Venn
Vamos criar uma lista com dados aleatórios
set.seed(654925)
list_venn <- list(A = sort(sample(1:100, 20)),
B = sort(sample(1:100, 20)),
C = sort(sample(1:100, 20)),
D = sort(sample(1:100, 20)))
head(list_venn) ## $A
## [1] 1 3 4 11 19 20 22 32 34 36 47 48 58 59 60 64 69 72 97 98
##
## $B
## [1] 4 17 18 23 32 33 34 41 45 52 53 56 58 59 66 67 74 78 91 92
##
## $C
## [1] 3 10 28 31 34 38 46 47 51 57 58 65 67 70 72 74 80 89 94 97
##
## $D
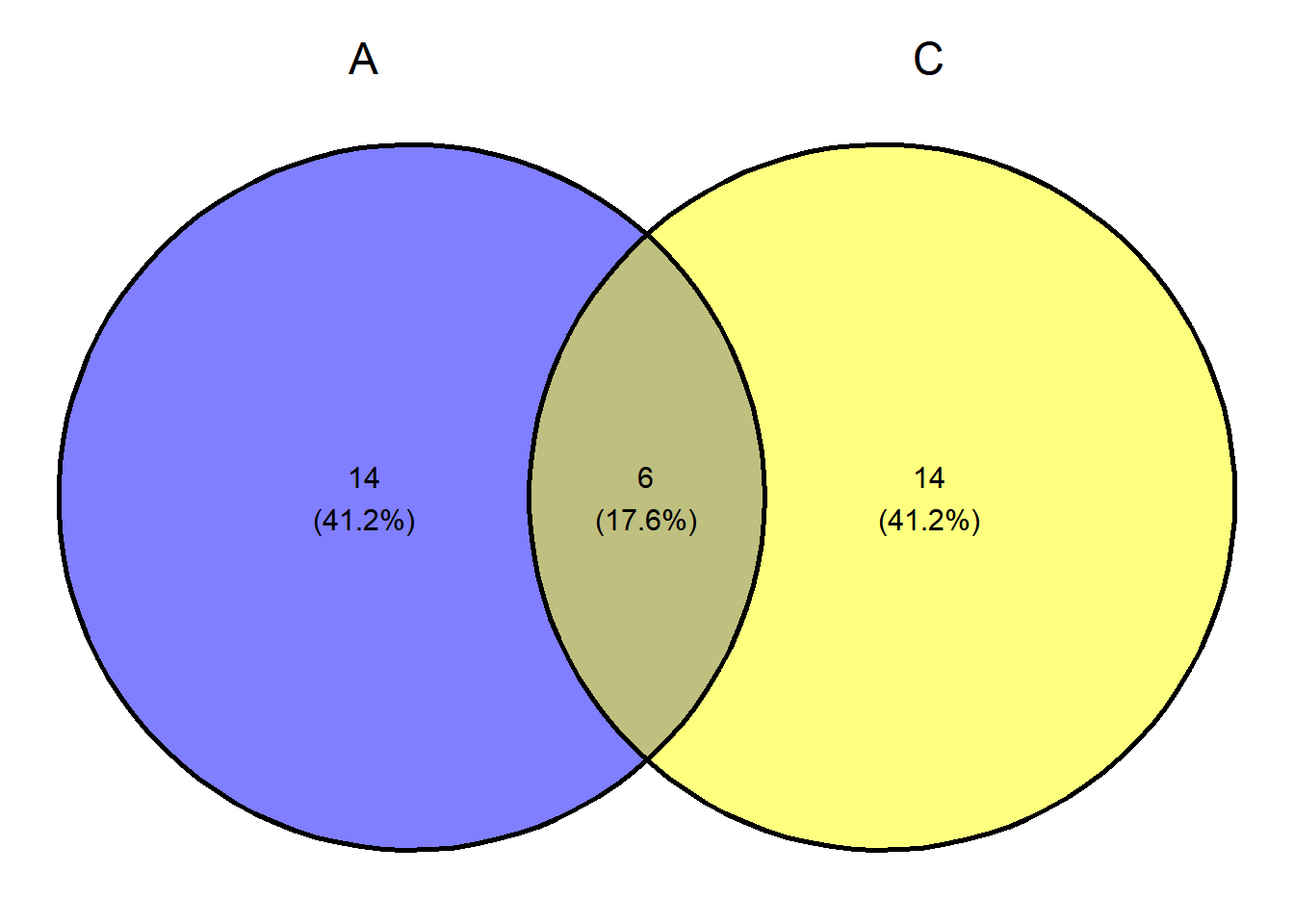
## [1] 8 11 14 15 17 18 19 33 34 47 51 59 66 68 73 77 78 82 86 87Carregar o pacote ggvenn (Yan 2023) e fazer o diagrama com A e C:
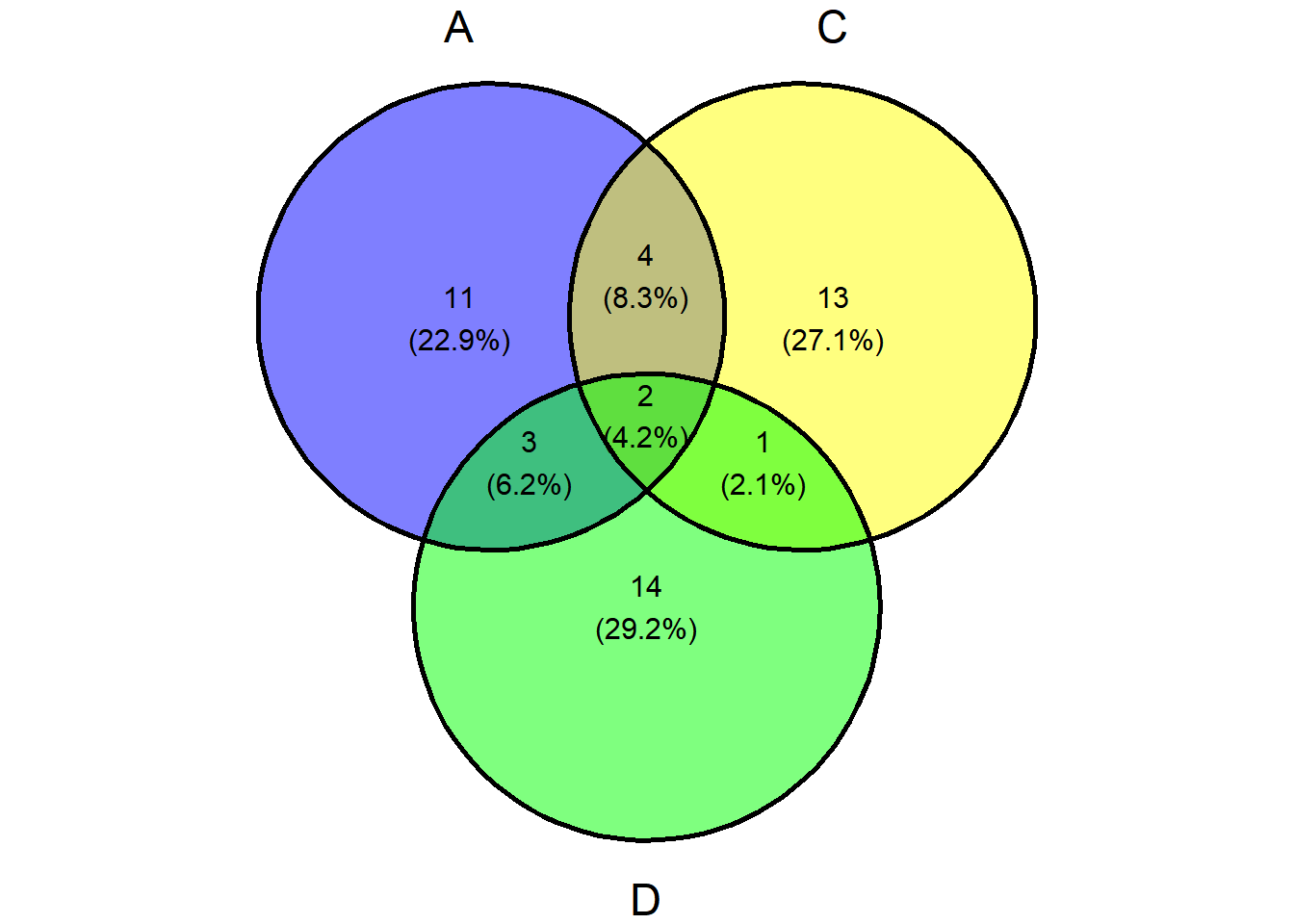
Fazer o diagrama com A, C e D:
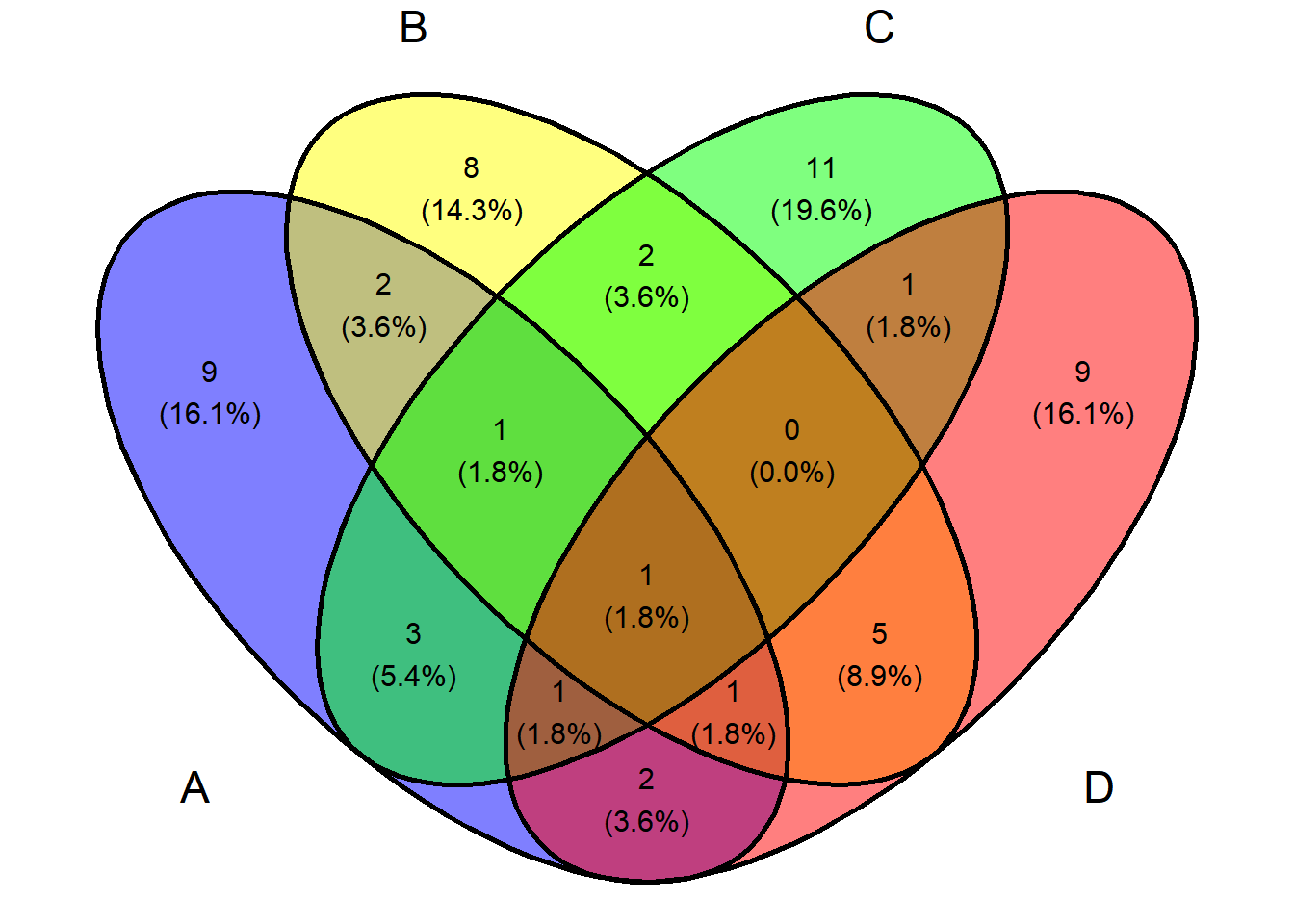
Com todos os dados:
4.3.5 Diagramas
Muitas vezes precisamos fazer um diagrama, ou fluxograma, para mostrar as etapas da pesquisa e podemos fazer isso usando o pacote DiagrammeR (Iannone 2023)
library(DiagrammeR)
figura1<-DiagrammeR::grViz("
digraph a_nice_graph {
node [fontname = Helvetica, color = green]
a [label = 'Ratos', color =blue, fontname=Impact]
b [label = 'caso, n = 20']
c [label = 'controle, n = 20']
d [label = 'medicamento']
e [label = 'salina']
f [label = '30 days']
g [label = 'córtex', color= blue]
h [label = 'hipocampo' , color= blue]
i [label = 'fígado', color =blue]
j [label = 'atividade enzimática', color =red,fontname=Impact]
# edge definitions with the node IDs
a -> {b c}
b -> d
c -> e
{d e} -> f
f -> {g h i}
{g h i} -> j
}
")
figura1Para salvar esse diagrama é preciso carregar mais 2 pacotes: rsvg (Ooms 2022) e DiagrammeRsvg (Iannone 2016)
library(rsvg)
library(DiagrammeRsvg)
figura1 = DiagrammeRsvg::export_svg(figura1)
figura1 = charToRaw(figura1) # flatten
rsvg_png(figura1, file = "figura1b.png", width = 5961, height = 7016)Outra opção é o pacote ggflowchart (Rennie 2023)
Para usar esse pacote tem um tutorial no endereço abaixo:
(https://nrennie.rbind.io/blog/introducing-ggflowchart/)
library(ggflowchart)
data <- tibble::tibble(from = c("A", "A", "A", "B", "C", "F"),
to = c("B", "C", "D", "E", "F", "G"))
ggflowchart(data)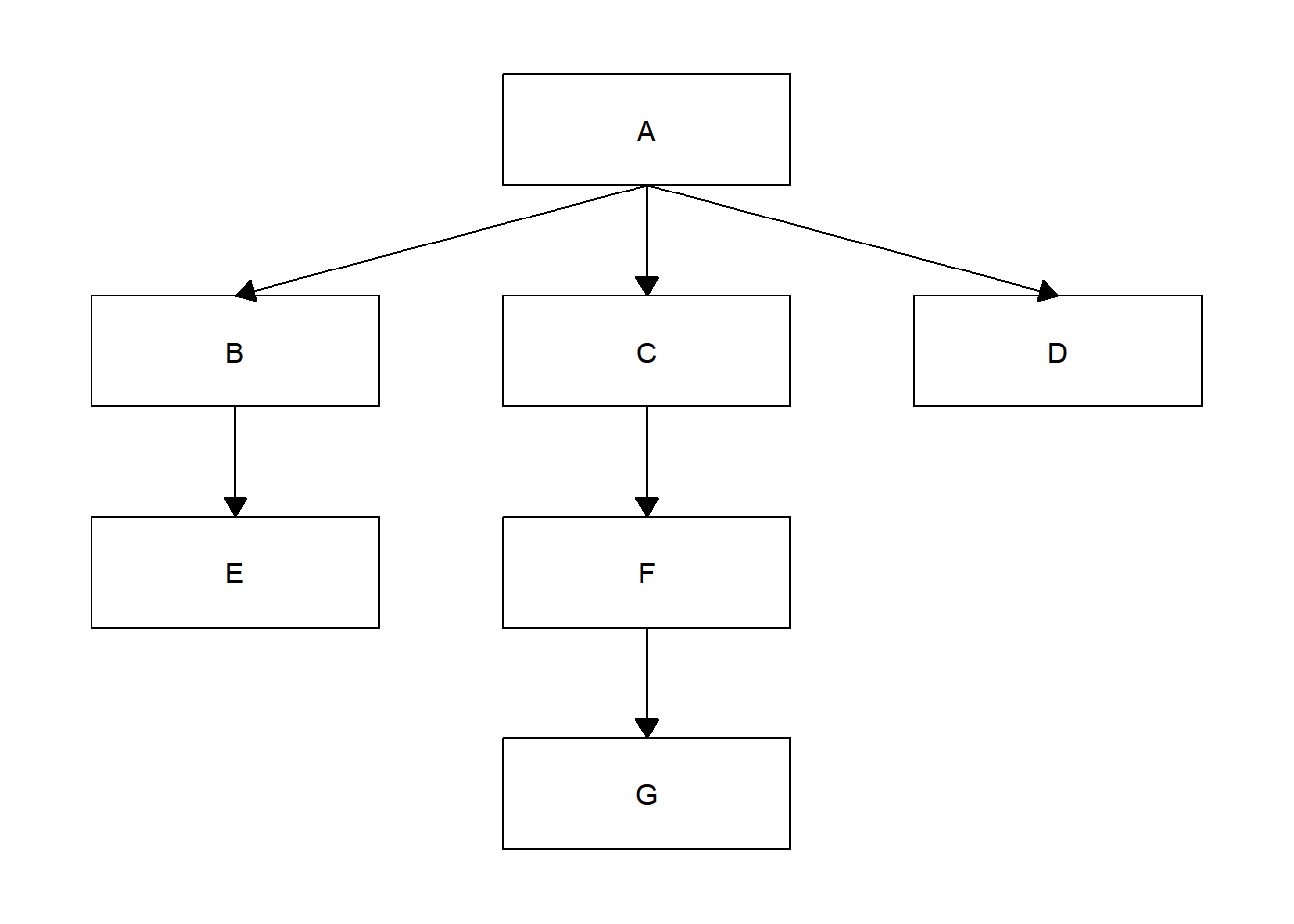
Podemos usar os seguintes parâmetros:
- fill = “white”. A cor de preenchimento.
- colour = “black”. A cor da caixa de texto.
- text_colour = “black”. A cor do texto.
- text_size = 3.88. O tamanho do texto.
- arrow_colour = “black”. A cor das flechas.
- arrow_size = 0.3. O tamanho das flechas.
- family = “sans”. A fonte do texto.
- x_nudge = 0.35. A largura da caixa de texto.
- y_nudge = 0.25. A altura da caica de texto.
- horizontal = FALSE. A direção do fluxograma.
Alguns exemplos:
ggflowchart(data,
colour = "blue",
text_colour = "red",
arrow_colour = "green",
family = "serif",
x_nudge = 0.4,
y_nudge = 0.2,
arrow_size = 0.3)
Com fluxograma na horizontal:
ggflowchart(data,
colour = "blue",
text_colour = "black",
arrow_colour = "orange",
family = "serif",
x_nudge = 0.4,
y_nudge = 0.2,
arrow_size = 0.3,
horizontal = T)
Utilizando tipos diferentes:
node_data <- tibble::tibble(
name = c("A", "B", "C", "D", "E", "F", "G"),
type = c("Type 1", "Type 1", "Type 1", "Type 1",
"Type 2", "Type 2", "Type 2")
)
ggflowchart(data, node_data, fill = type)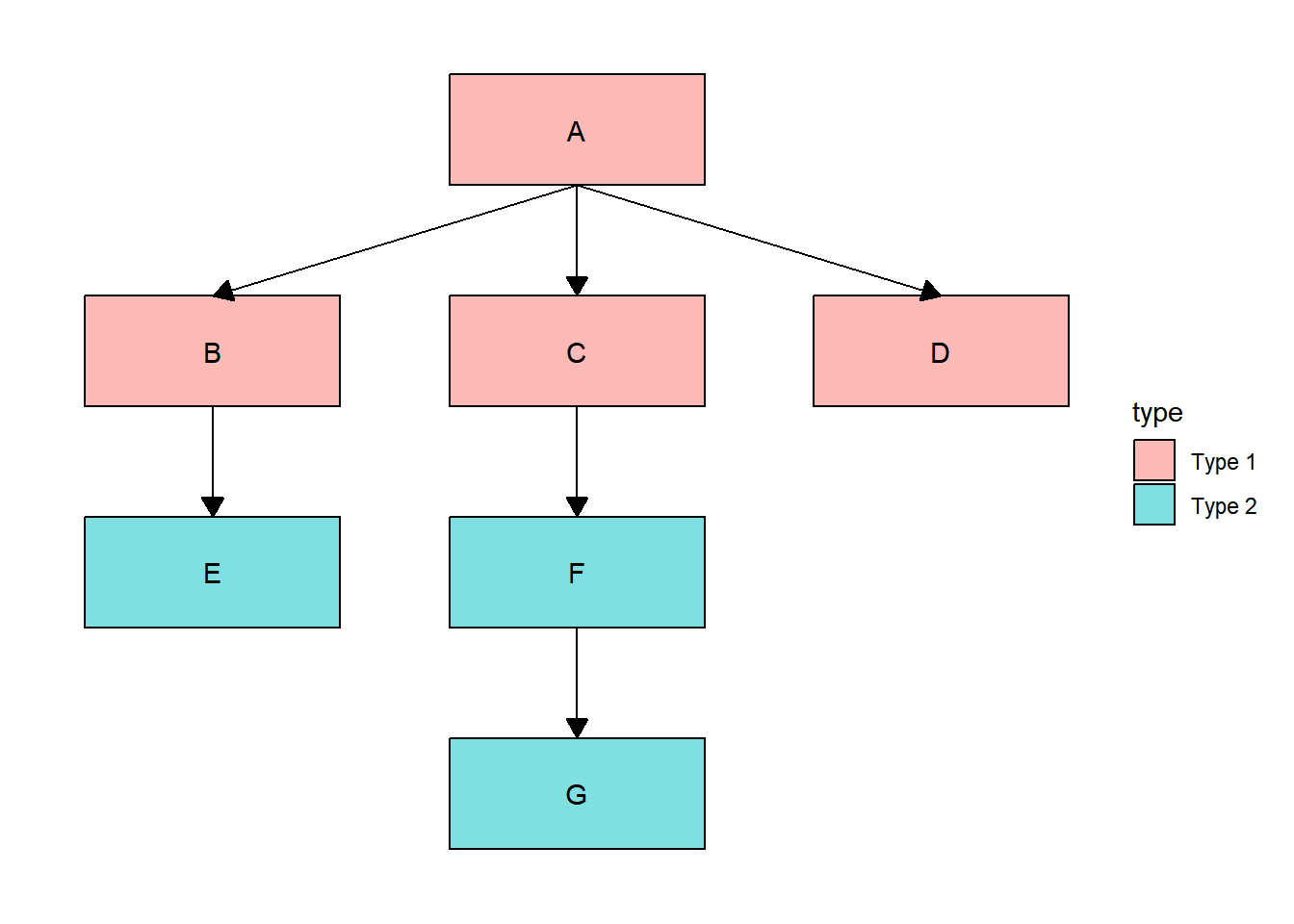
4.3.6 Datas e séries temporais
Vamos plotar uma variável (peso) ao longo do tempo. Primeiro vamos carregar os dados:
## ID grupo tempo peso
## 1 1 controle t1 270
## 2 2 controle t1 287
## 3 3 controle t1 279
## 4 4 controle t1 286
## 5 5 controle t1 275
## 6 6 controle t1 282Nesse arquivo temos a variável grupo (caso e controle), uma variável tempo (t1 a t6) e a variável peso. São 2 grupos de animais, que foram pesados em 6 momentos.
Agora ativaremos uma função (http://www.sthda.com/english/wiki/ggplot2-error-bars-quick-start-guide-r-software-and-data-visualization) para organizar os dados para poder fazer o gráfico:
data_summary <- function(data, varname, groupnames){
require(plyr)
summary_func <- function(x, col){
c(mean = mean(x[[col]], na.rm=TRUE),
sd = sd(x[[col]], na.rm=TRUE))
}
data_sum<-ddply(data, groupnames, .fun=summary_func,
varname)
data_sum <- rename(data_sum, c("mean" = varname))
return(data_sum)
}Agora que a função data_summary está ativa, vamos preparar os dados:
## grupo tempo peso sd
## 1 caso t1 287.1429 32.28207
## 2 caso t2 262.3571 13.78186
## 3 caso t3 265.5000 27.38262
## 4 caso t4 268.5714 26.69115
## 5 caso t5 293.7143 28.22554
## 6 caso t6 327.7857 36.65049
## 7 controle t1 294.0000 32.87011
## 8 controle t2 308.6000 37.88931
## 9 controle t3 330.2000 21.04915
## 10 controle t4 347.1000 20.14641
## 11 controle t5 375.1000 21.74320
## 12 controle t6 408.6000 17.94560E finalmente o gráfico:
p<- ggplot(df, aes(x=tempo, y=peso, group=grupo, color=grupo)) +
geom_line() +
geom_point() +
geom_errorbar(aes(ymin=peso-sd, ymax=peso+sd), width=.2,
position=position_dodge(0.05))
p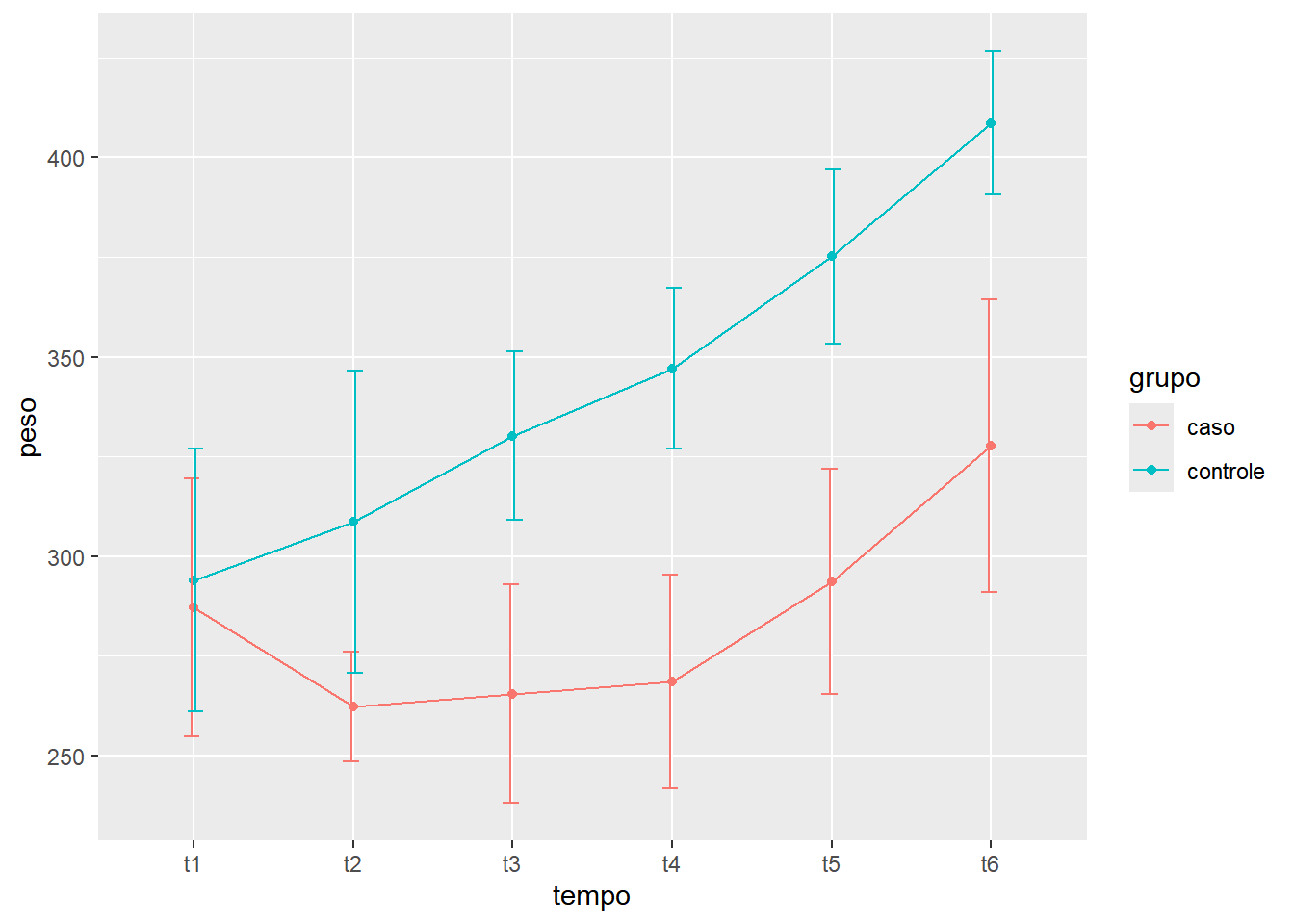
Personalizando o gráfico:
p+labs(title="Weight variation along 30 days", x="Time", y = "Weight")+
theme_classic() +
scale_color_manual(values=c('#999999','#E69F00'))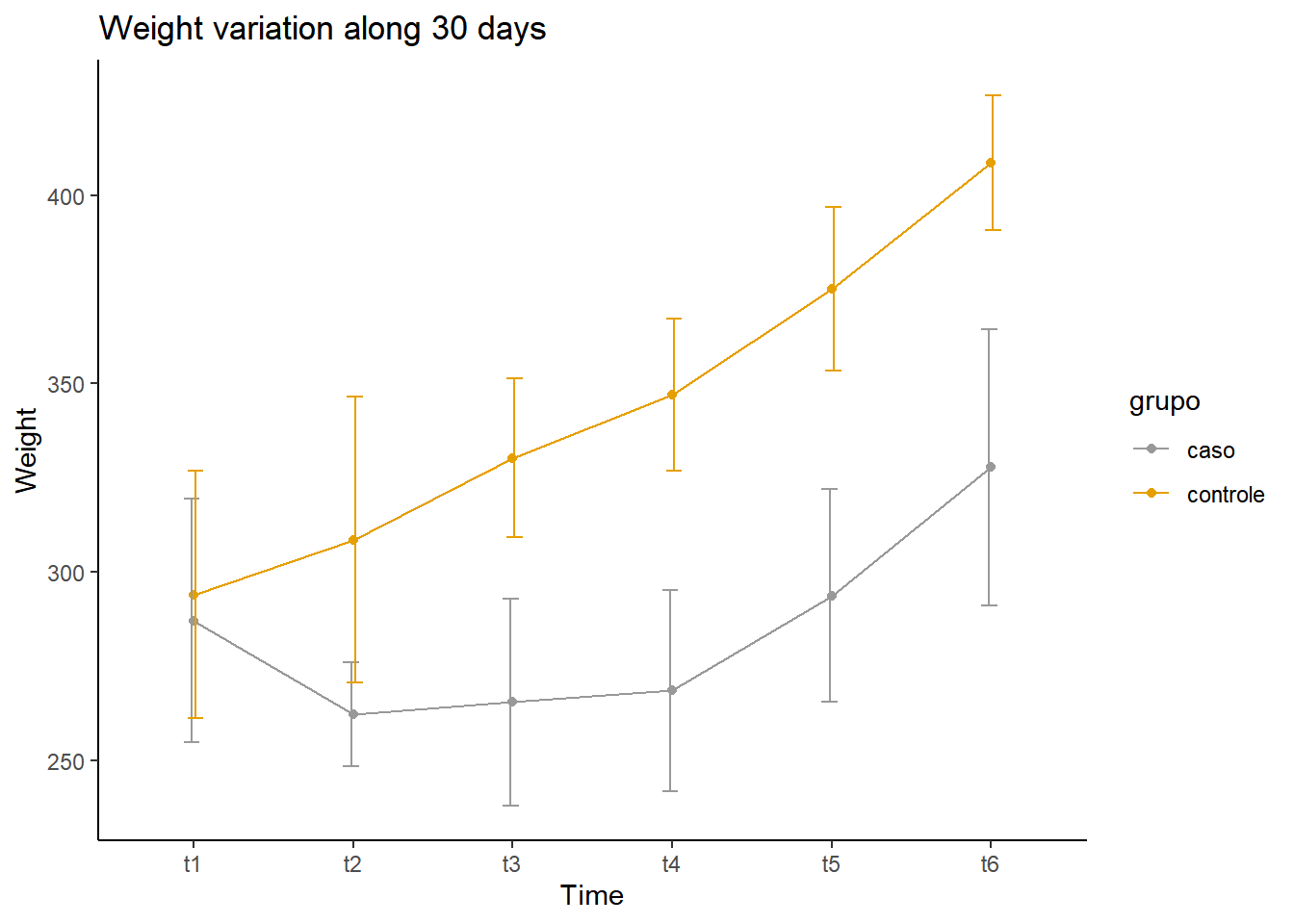
Além da definição das cores, que já vimos, aqui usamos também o theme_classic()
Esses temas controlam outros aspectos visuais do gráfico.
Tem vários temas:
theme_grey()
theme_gray()
theme_bw()
theme_linedraw()
theme_light()
theme_dark()
theme_minimal()
theme_classic()
theme_void()
theme_test()
4.3.7 Time series - Dados de casos de COVID no Paraná
Os dados podem ser baixados de: https://www.saude.pr.gov.br/Pagina/Coronavirus-COVID-19 Como exemplo, vou usar os dados de março de 2022, porque quanto mais recente, maior o arquivo e demora mais para carregar e processar os dados.
Carregar os dados:
dados<-read.csv("dados/informe_epidemiologico_16_03_2022_geral.csv", header = T, sep = ";")
library(dplyr)
head(dados)## IBGE_RES_PR IBGE_ATEND_PR UF_RESIDENCIA SEXO IDADE_ORIGINAL
## 1 4118501 4118501 PR M 96
## 2 4106902 4106902 PR F 77
## 3 4125704 4108304 PR M 73
## 4 4106902 4106902 PR F 95
## 5 4119905 4119905 PR M 81
## 6 4116802 4116802 PR M 56
## MUN_RESIDENCIA MUN_ATENDIMENTO EXAME DATA_DIAGNOSTICO
## 1 PATO BRANCO PATO BRANCO 167 2022-03-02
## 2 CURITIBA CURITIBA 167 2022-02-22
## 3 SAO MIGUEL DO IGUACU FOZ DO IGUACU 27 2022-02-21
## 4 CURITIBA CURITIBA 27 2022-01-20
## 5 PONTA GROSSA PONTA GROSSA 27 2022-01-31
## 6 NOVA CANTU NOVA CANTU 1 2022-03-07
## DATA_CONFIRMACAO_DIVULGACAO DATA_INICIO_SINTOMAS ÓBITO.COVID.19 DATA_OBITO
## 1 2022-03-16 2022-02-24 SIM 2022-02-28
## 2 2022-03-16 SIM 2022-03-14
## 3 2022-03-16 2022-02-21 SIM 2022-03-07
## 4 2022-03-16 2022-01-14 SIM 2022-02-06
## 5 2022-03-16 2022-01-21 SIM 2022-03-04
## 6 2022-03-16 2022-03-01 SIM 2022-03-14
## DATA_OBITO_DIVULGACAO STATUS DATA_RECUPERADO_DIVULGACAO
## 1 2022-03-16 Óbito por COVID-19
## 2 2022-03-16 Óbito por COVID-19
## 3 2022-03-16 Óbito por COVID-19
## 4 2022-03-16 Óbito por COVID-19
## 5 2022-03-16 Óbito por COVID-19
## 6 2022-03-16 Óbito por COVID-19
## ORIGEM_NOTIFICACAO
## 1 Vigilância Epidemiológica de Pato Branco
## 2 e-Saúde
## 3 Notifica COVID-19
## 4 e-Saúde
## 5 Notifica COVID-19
## 6 Notifica COVID-19Os dados tem colunas com data, no entanto, para o R entender que são datas devemos usar o pacote lubridate. Devemos olhar como está o formato da data. Nesse caso estão no formato ano, mês, dia, então usamos função ymd:
library(lubridate)
dados$DATA_INICIO_SINTOMAS <-ymd(dados$DATA_INICIO_SINTOMAS)
dados$DATA_DIAGNOSTICO <-ymd(dados$DATA_DIAGNOSTICO)
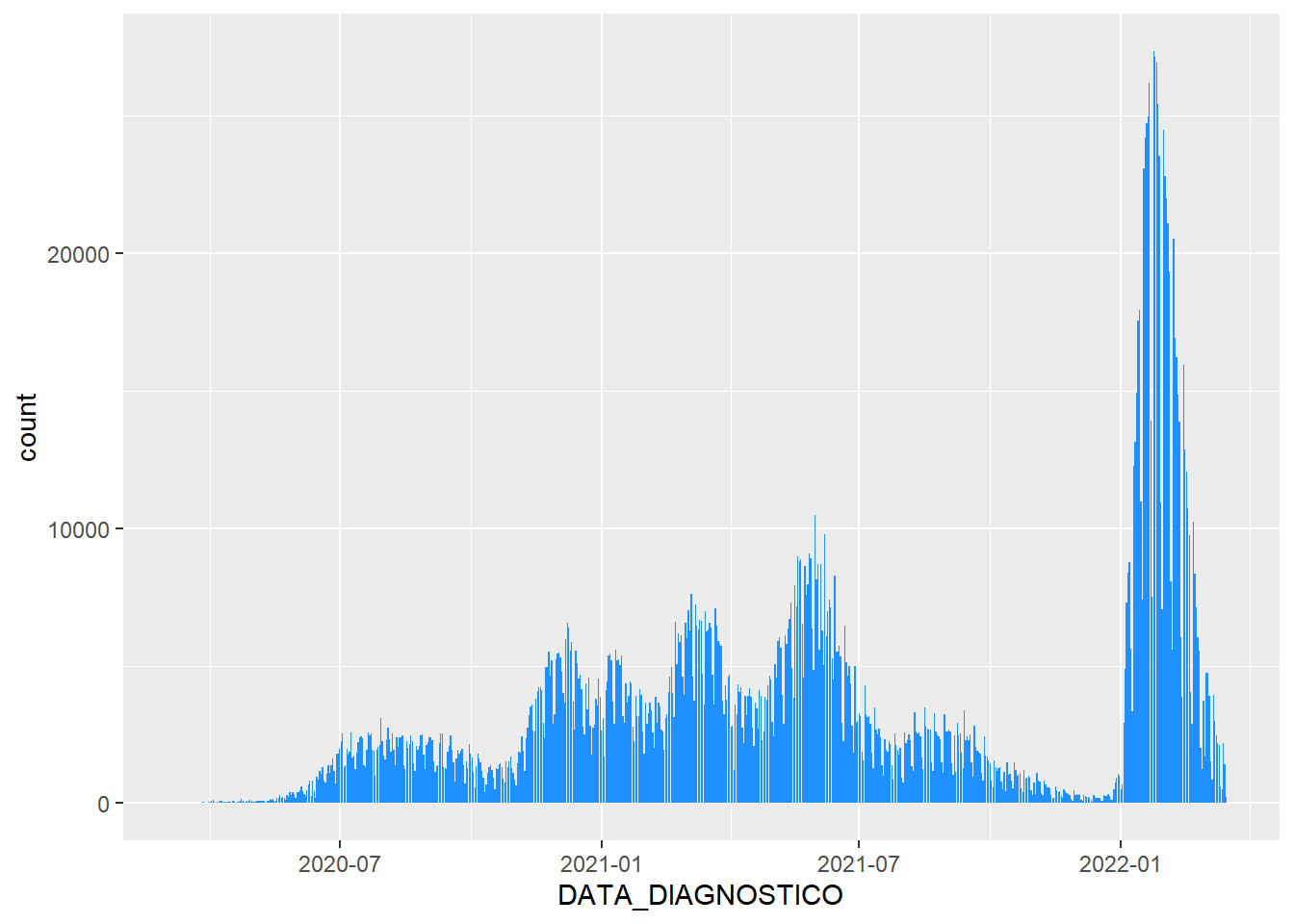
dados$DATA_OBITO <-ymd(dados$DATA_OBITO)Podemos plotar o número de casos por data de diagnóstico:
Podemos ver a idade dos casos positivos:
dados %>%
filter(IDADE_ORIGINAL>=0)%>%
ggplot(mapping = aes(IDADE_ORIGINAL )) +
geom_bar(fill="goldenrod1", col="black")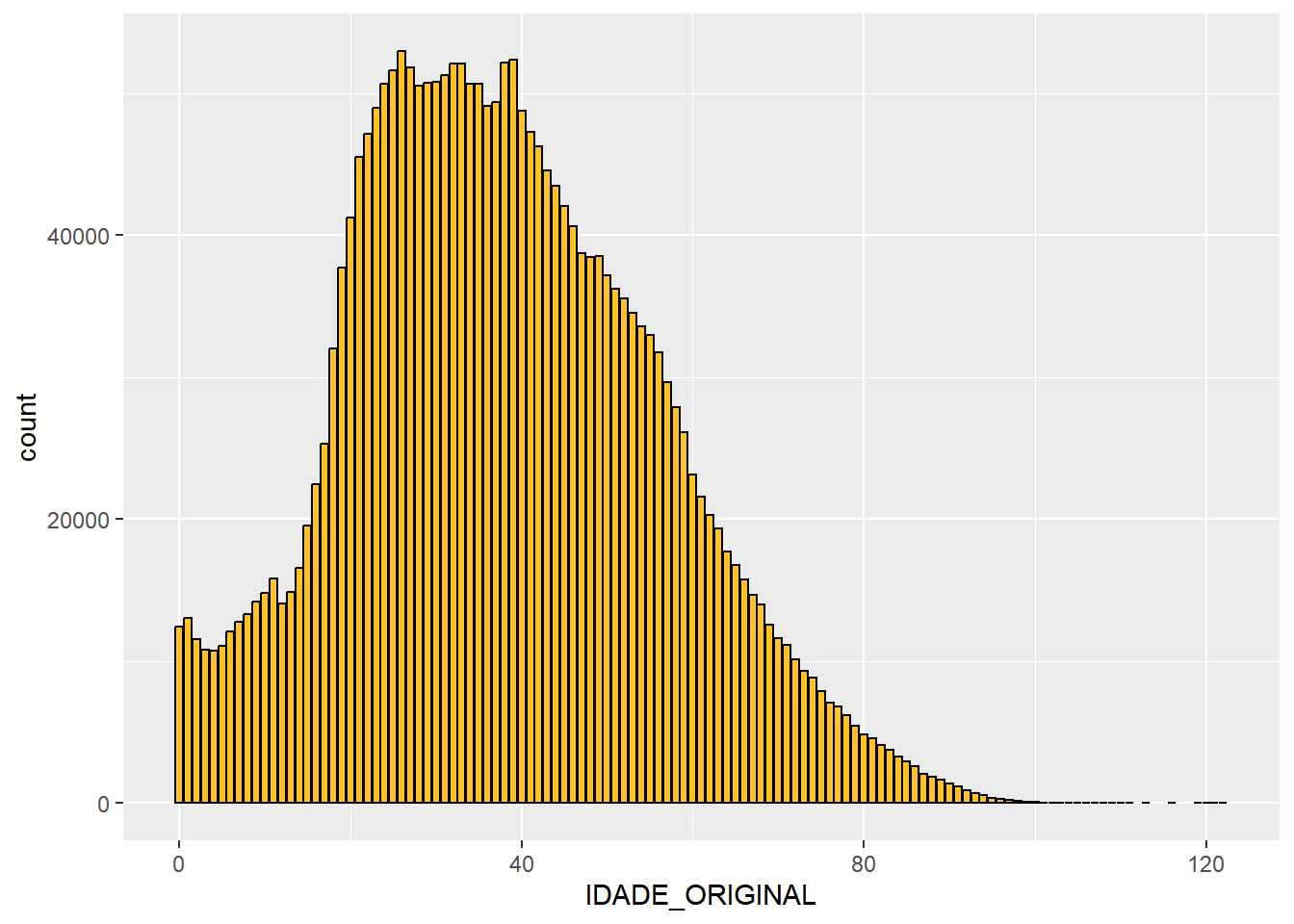
Ou a idade dos casos de óbito:
dados %>%
filter(!is.na(DATA_OBITO))%>%
ggplot(mapping = aes(x =IDADE_ORIGINAL )) +
geom_bar(fill="firebrick1", col="black")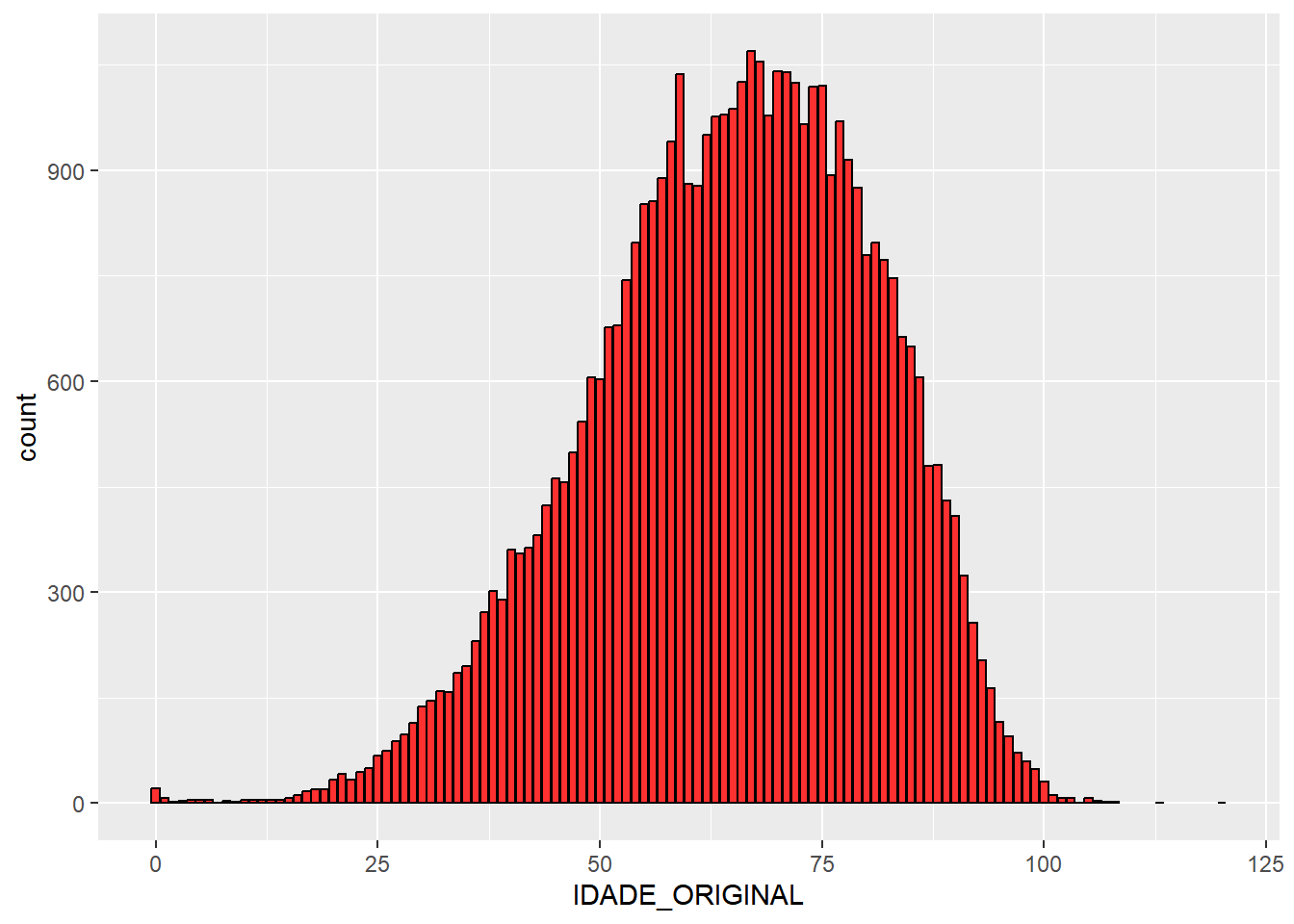
Podemos separa por sexo:
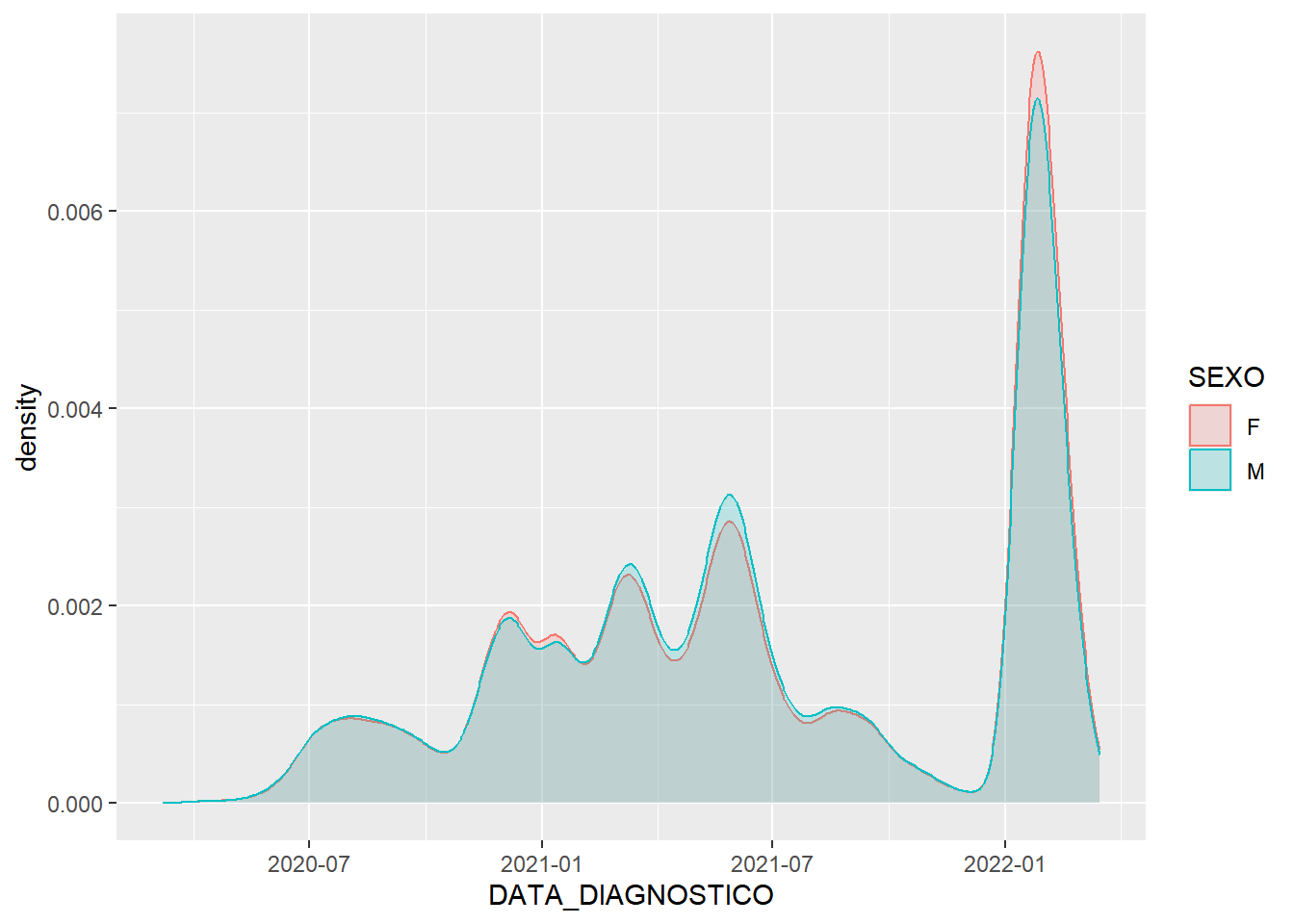
Filtros
Podemos usar filtros, por exemplo, por data:
p <- dados %>%
filter(DATA_DIAGNOSTICO>as.Date("2022-02-15"))%>%
ggplot(aes(DATA_DIAGNOSTICO))
p + geom_bar(fill = "dodgerblue")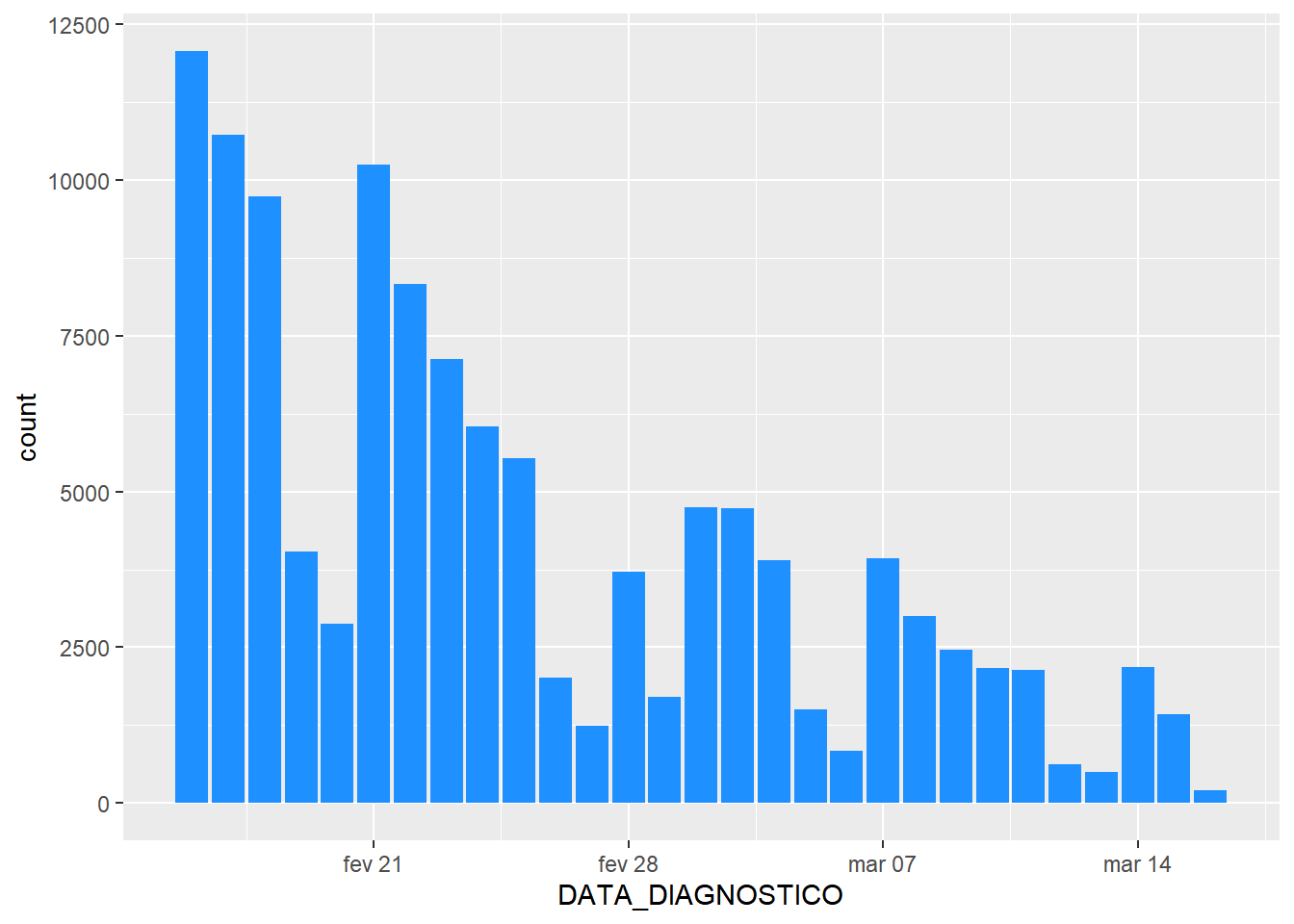
Filtrando por município:
p <- dados %>%
filter(MUN_RESIDENCIA=="PONTA GROSSA")%>%
ggplot(aes(x = DATA_DIAGNOSTICO))
p + geom_bar(fill = "blue")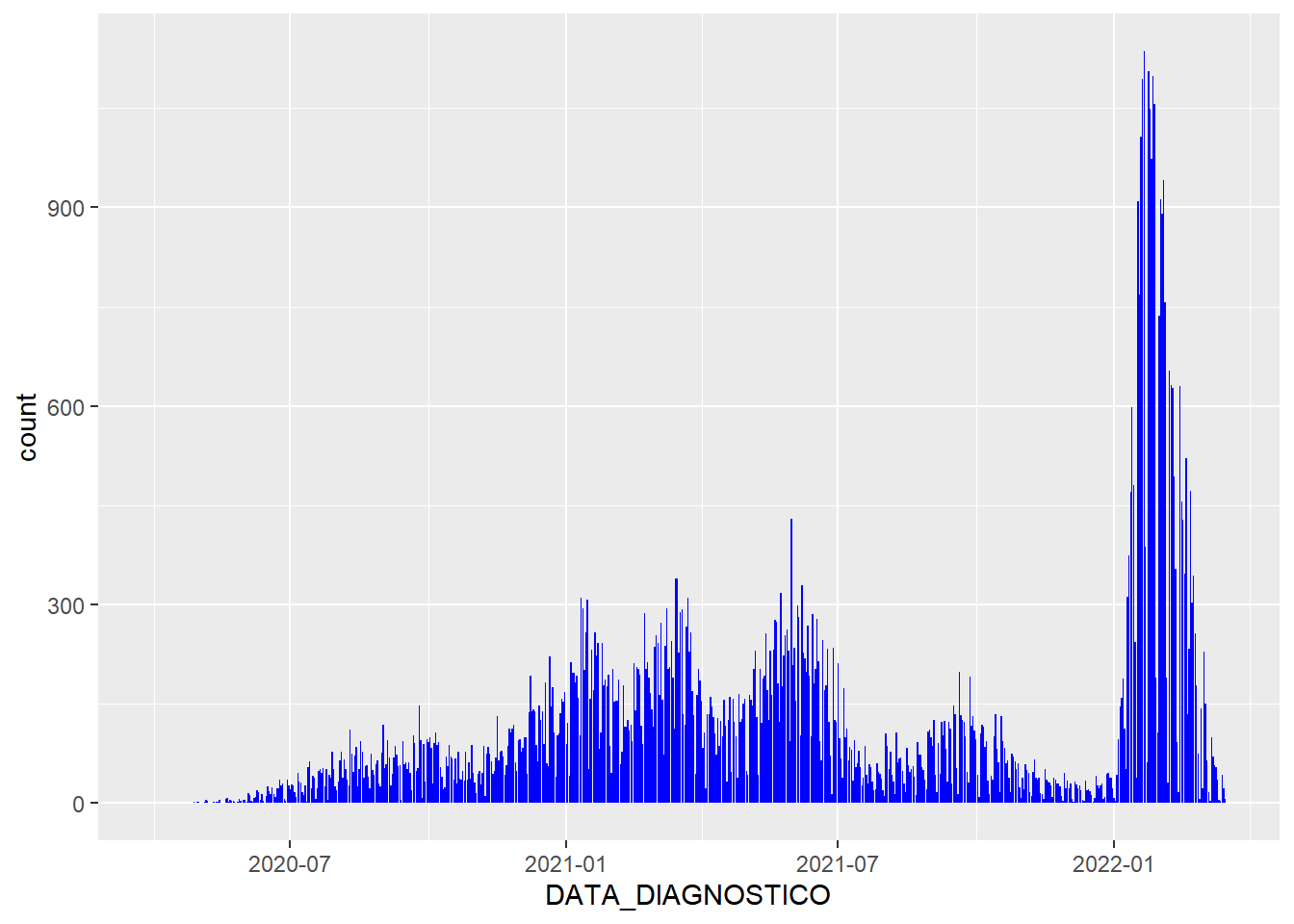
Além desses gráficos todos apresentados aqui, existem muitos outros, os quais vocês podem procurar conforme a necessidade.
Mais detalhes podem ser obtidos em Wickham, Çetinkaya-Rundel, and Grolemund (2023)
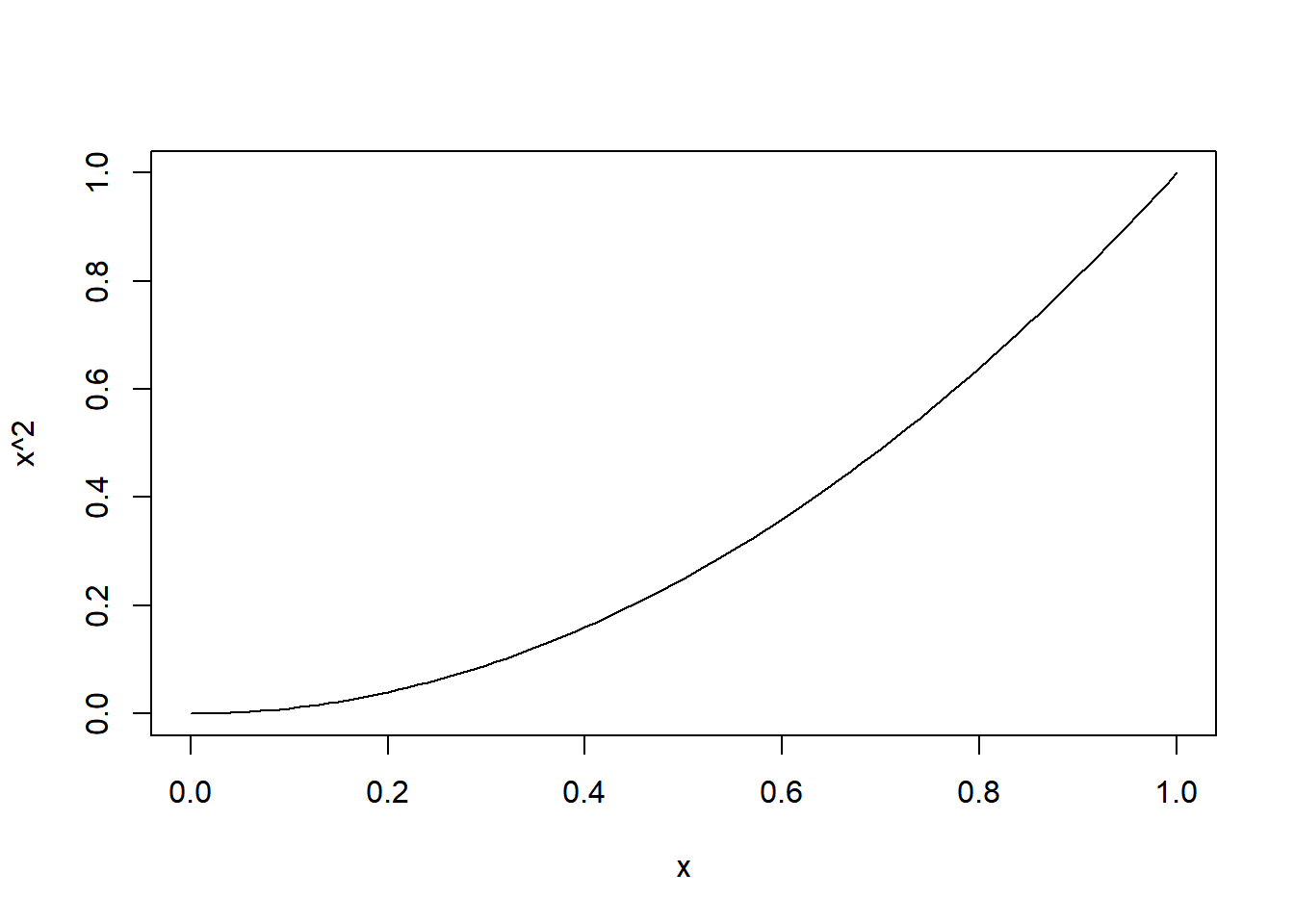 Podemos especificar os valores inicial e final de x, assim como a cor da curva:
Podemos especificar os valores inicial e final de x, assim como a cor da curva: Também podemos sobrepor várias curvas no mesmo gráfico:
Também podemos sobrepor várias curvas no mesmo gráfico: