Capítulo 8 Network
Em várias áreas temos dados complexos de interações entre diferentes grupos de elementos que estão sendo analisados e para esses casos, um gráfico de rede ajuda a visualizar essas interações, que podem ser espécies, genes ou pessoas.
Conceitos básicos: Vértices (Vertices) e arestas (edges)
Vértices: é um ponto em que duas ou mais curvas, retas ou arestas se encontram. Arestas: segmento de reta que liga dois vértices.
Os objetos para fazer uma rede é uma matriz de adjacências, que é uma matriz quadrada, onde os nomes das linha e colunas são os vértices e os valores dentro da matriz indicam uma conexão (1) dos vértices ou não (0). No entanto, podemos criar um objeto para rede a partir de um dataframe, como veremos depois.
8.1 Pacote Igraph
Um dos principais pacotes para criar gráficos de redes é o igraph (Csárdi et al. 2023), cujo manual pode ser consultado em https://igraph.org/r/
Existem muitas funções para criar diferentes estruturas de gráficos no Igraph.
Conceitos básicos: Vértices (Vertices) e arestas (edges)
Vértices: é um ponto em que duas ou mais curvas, retas ou arestas se encontram Arestas: segmento de reta que liga dois vértices
Exemplo:
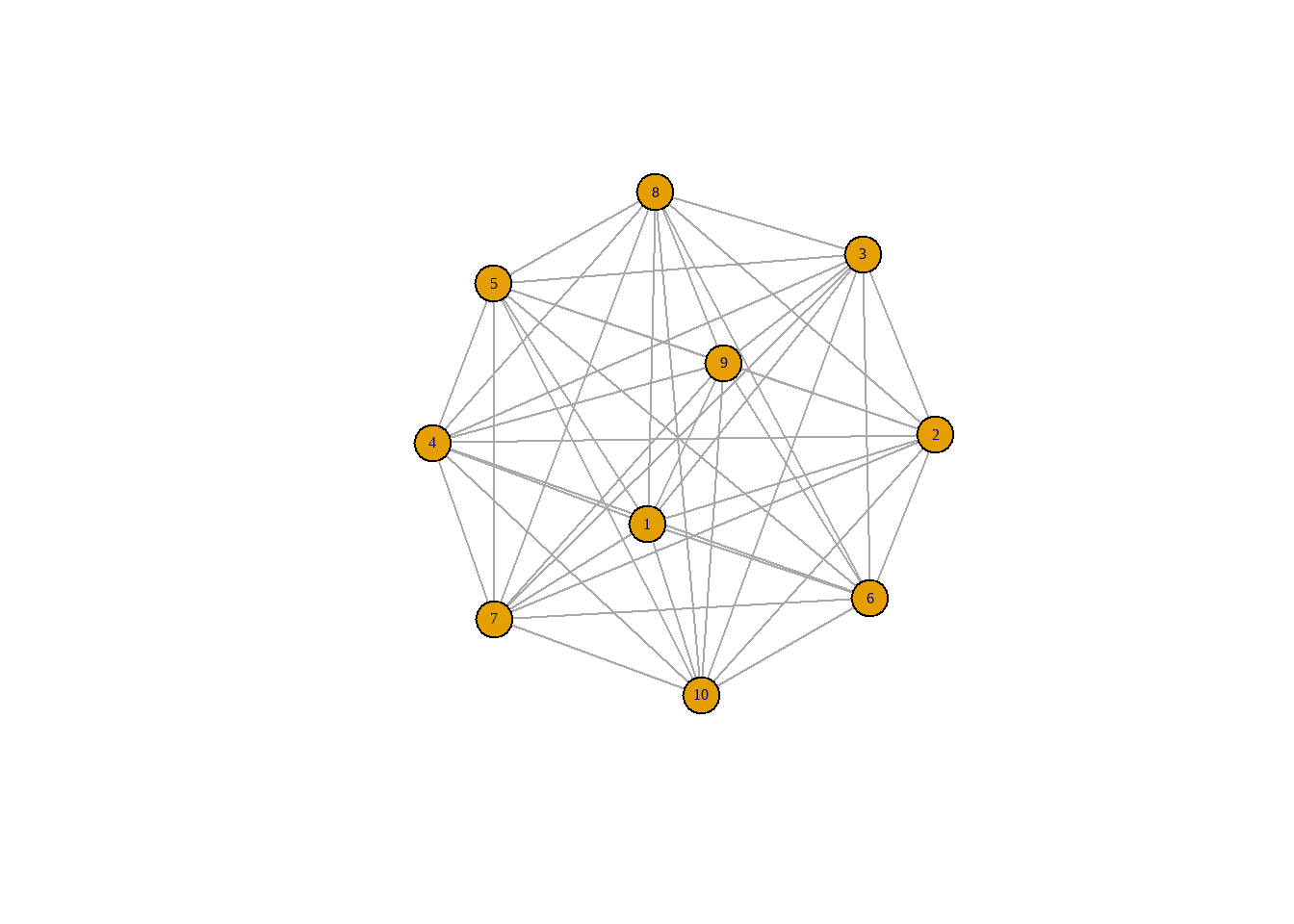
Gráfico completo: cada par de vértices tem uma aresta conectando-os, o argumento LOOPS = FALSE significa que as arestas próprias não são adicionadas.
Gráficos com a lista de arestas (edges) fornecida:
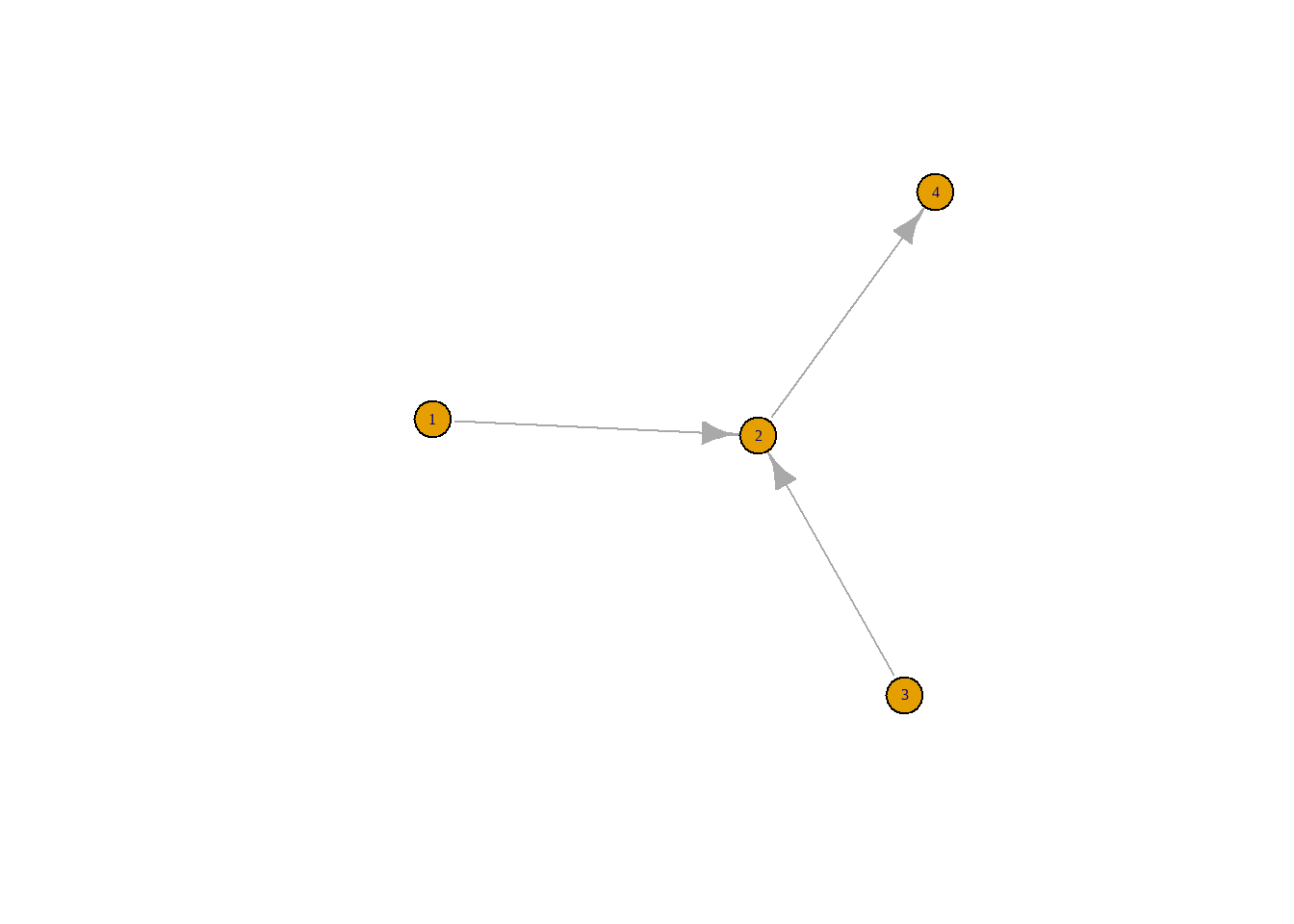
Podemos montar um arquivo com os vértices e arestas
library(openxlsx)
arestas<-read.xlsx("dados/miRNA2.xlsx", sheet=1,colNames = T)
vertices<-read.xlsx("dados/miRNA2.xlsx", sheet=2,colNames = T)
head(arestas)## from to weight type
## 1 1 23 1 3UTR
## 2 1 24 1 3UTR
## 3 1 25 1 3UTR
## 4 1 26 1 3UTR
## 5 1 27 1 3UTR
## 6 2 25 1 3UTR## id nome tipo type.label
## 1 1 miRNA1 1 miRNA
## 2 2 miRNA2 1 miRNA
## 3 3 miRNA3 1 miRNA
## 4 4 miRNA4 1 miRNA
## 5 5 miRNA5 1 miRNA
## 6 6 miRNA6 1 miRNAVamos criar o objet net com a função graph_from_data:
## IGRAPH e5588e5 DNW- 27 51 --
## + attr: name (v/c), nome (v/c), tipo (v/n), type.label (v/c), weight
## | (e/n), type (e/c)
## + edges from e5588e5 (vertex names):
## [1] 1 ->23 1 ->24 1 ->25 1 ->26 1 ->27 2 ->25 2 ->26 3 ->23 3 ->27 4 ->24
## [11] 4 ->25 4 ->26 5 ->26 5 ->27 6 ->24 6 ->26 6 ->27 7 ->24 7 ->27 8 ->24
## [21] 8 ->26 9 ->24 9 ->25 10->23 10->26 10->27 11->24 11->25 12->24 12->26
## [31] 13->24 13->26 13->27 14->25 14->27 15->24 15->27 16->25 16->26 17->25
## [41] 17->26 18->24 18->27 19->25 19->27 20->25 20->27 21->24 21->26 22->24
## [51] 22->26E gerar o gráfico:
plot(net, edge.arrow.size=.2,vertex.label=vertices$nome, vertex.label.color="blue", vertex.label.dist=3, vertex.size=5)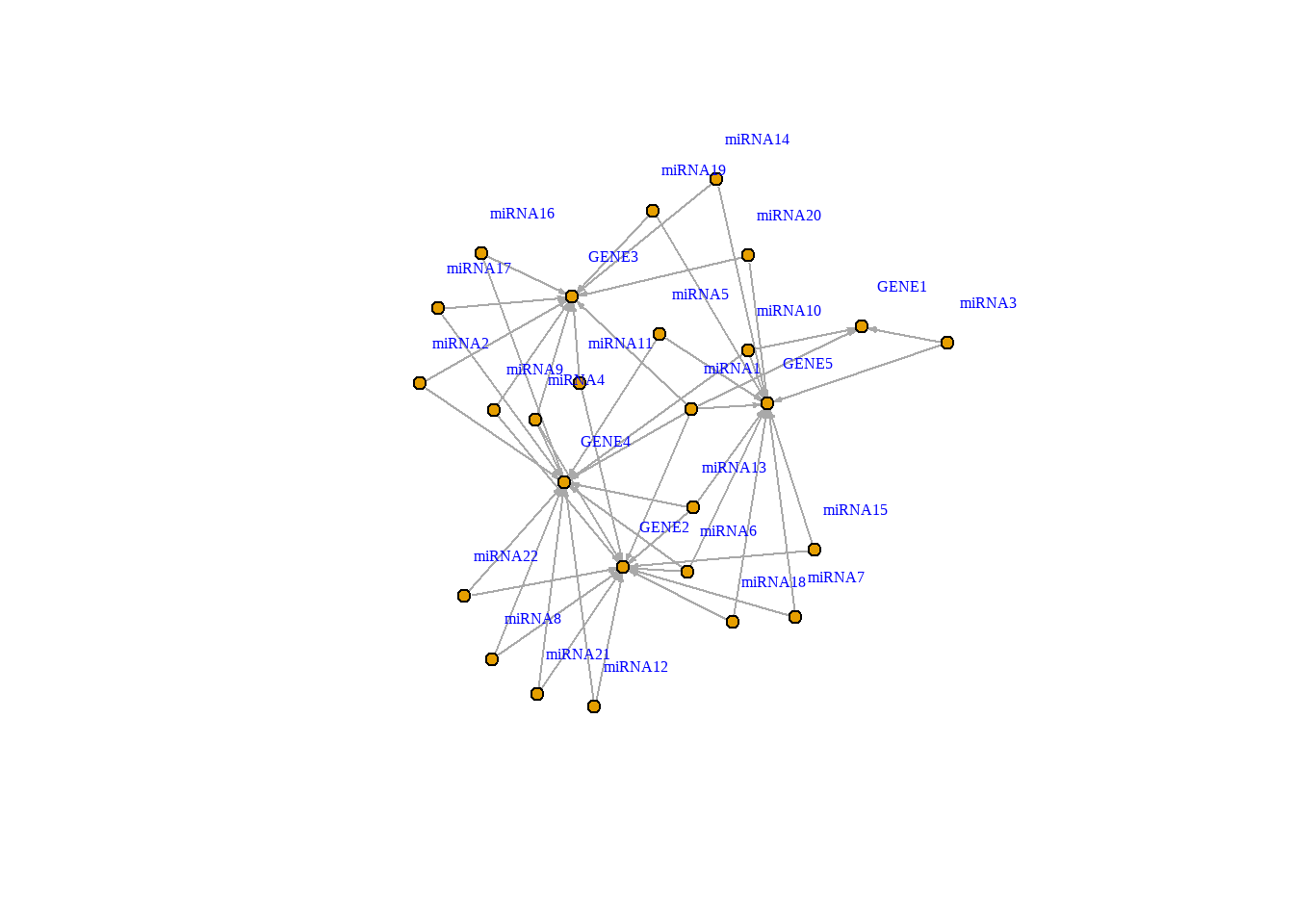
Podemos ainda mudar configurações:
l <- layout_with_kk(net)
l <- norm_coords(l, ymin=-1, ymax=1, xmin=-1, xmax=1)
plot(net, rescale=F, layout=l*1.2, edge.arrow.size=.2,vertex.label=vertices$nome, vertex.label.color=vertices$tipo, vertex.label.dist=3, vertex.size=5, vertex.color=vertices$tipo)