Capítulo 2 Carregar e manipular dados
Dados
Uma etapa muito importante é planejar as planilhas de dados. Se os dados estiverem bem organizados, isso vai poupar muito tempo.
Como os dados devem estar organizados: os casos nas linhas e as variáveis nas colunas.
Os dados podem estar em planilhas do excel ou em arquivos de texto. Vamos ver como carregar esses dados.
2.1 Ler arquivos txt e csv
Para ler um arquivo txt usamos a função read.table. No exemplo abaixo, o arquivo iris.txt está dentro pasta dados:
## sepal_l sepal_w petal_l petal_w class
## 1 5.1 3.5 1.4 0.2 Iris-setosa
## 2 4.9 3.0 1.4 0.2 Iris-setosa
## 3 4.7 3.2 1.3 0.2 Iris-setosa
## 4 4.6 3.1 1.5 0.2 Iris-setosa
## 5 5.0 3.6 1.4 0.2 Iris-setosa
## 6 5.4 3.9 1.7 0.4 Iris-setosa## sepal_l sepal_w petal_l petal_w class
## 145 6.7 3.3 5.7 2.5 Iris-virginica
## 146 6.7 3.0 5.2 2.3 Iris-virginica
## 147 6.3 2.5 5.0 1.9 Iris-virginica
## 148 6.5 3.0 5.2 2.0 Iris-virginica
## 149 6.2 3.4 5.4 2.3 Iris-virginica
## 150 5.9 3.0 5.1 1.8 Iris-virginicaOnde head=T indica que os dados tem uma linha de cabeçalho e sep define o separador das variáveis, nesse caso é a vírgula.
Use help(“read.table”) para ver outros parâmetros.
Podemos ver as primeiras linhas do objeto head e as últimas linhas com tail.
2.2 Ler arquivos xlsx
Instale e ative o pacote openxlsx (Schauberger and Walker 2023). Temos duas maneiras de instalar um pacote. No RStudio, podemos clicar em Install na aba Packages e então digitar o nome do pacote a ser instalado. Ou podemos usar a função install.packages(), especificando o nome do pacote dentro do parêntesis.
Sempre que formos utilizar um pacote, precisamos ativá-lo. Também temos duas maneiras de fazer isso. A primeira, usando o RStudio, na aba Packages procuramos o pacote e clicamos na caixa de seleção. A segunda maneira, a qual recomendo usar, é com a função library(). Dessa forma, a ativação já estará no script e não corremos o risco de esquecer de ativar o pacote.
O pacote openxlsx permite ler e gravar arquivo xlsx, como no exemplo abaixo:
## species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g
## 1 Adelie Torgersen 39.1 18.7 181 3750
## 2 Adelie Torgersen 39.5 17.4 186 3800
## 3 Adelie Torgersen 40.3 18.0 195 3250
## 4 Adelie Torgersen NA NA NA NA
## 5 Adelie Torgersen 36.7 19.3 193 3450
## 6 Adelie Torgersen 39.3 20.6 190 3650
## sex year
## 1 male 2007
## 2 female 2007
## 3 female 2007
## 4 <NA> 2007
## 5 female 2007
## 6 male 2007Onde sheet especifica qual planilha do arquivo xlsx deve ser lida e colNames = T indica que na primeira linha estão os títulos das colunas. Podemos definir outros parâmetros também, como por exemplo, especificar em qual linha começar a ler os dados com startRow =, ou definir quais colunas pegar com cols =.
Também é possível usar a opção do RStudio para importar dados para isso, na aba environment, clique em import Dataset tem opções de importar dados do texto, do excel, do SPSS e outros formatos.
2.4 Manipulação de dados (R base)
Com a função summary podemos ver um resumo sobre uma variável:
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 4.300 5.100 5.800 5.843 6.400 7.900Para especificar a variável, usamos o nome do objeto seguido de $ e o nome da coluna
Podemos ver um resumo de todas as variáveis:
## sepal_l sepal_w petal_l petal_w
## Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
## 1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
## Median :5.800 Median :3.000 Median :4.350 Median :1.300
## Mean :5.843 Mean :3.054 Mean :3.759 Mean :1.199
## 3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
## Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
## class
## Length:150
## Class :character
## Mode :character
##
##
## Podemos ver as características das variáveis usando a função str:
## 'data.frame': 150 obs. of 5 variables:
## $ sepal_l: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## $ sepal_w: num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## $ petal_l: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## $ petal_w: num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## $ class : chr "Iris-setosa" "Iris-setosa" "Iris-setosa" "Iris-setosa" ...Indexação
Podemos usar indexação para acessar dados específicos.
## [1] 1.4## [1] 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 1.5 1.6 1.4 1.1 1.2 1.5 1.3 1.4
## [19] 1.7 1.5 1.7 1.5 1.0 1.7 1.9 1.6 1.6 1.5 1.4 1.6 1.6 1.5 1.5 1.4 1.5 1.2
## [37] 1.3 1.5 1.3 1.5 1.3 1.3 1.3 1.6 1.9 1.4 1.6 1.4 1.5 1.4 4.7 4.5 4.9 4.0
## [55] 4.6 4.5 4.7 3.3 4.6 3.9 3.5 4.2 4.0 4.7 3.6 4.4 4.5 4.1 4.5 3.9 4.8 4.0
## [73] 4.9 4.7 4.3 4.4 4.8 5.0 4.5 3.5 3.8 3.7 3.9 5.1 4.5 4.5 4.7 4.4 4.1 4.0
## [91] 4.4 4.6 4.0 3.3 4.2 4.2 4.2 4.3 3.0 4.1 6.0 5.1 5.9 5.6 5.8 6.6 4.5 6.3
## [109] 5.8 6.1 5.1 5.3 5.5 5.0 5.1 5.3 5.5 6.7 6.9 5.0 5.7 4.9 6.7 4.9 5.7 6.0
## [127] 4.8 4.9 5.6 5.8 6.1 6.4 5.6 5.1 5.6 6.1 5.6 5.5 4.8 5.4 5.6 5.1 5.1 5.9
## [145] 5.7 5.2 5.0 5.2 5.4 5.1## sepal_l sepal_w petal_l petal_w class
## 2 4.9 3 1.4 0.2 Iris-setosa## [1] 3.5 3.0 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 3.7 3.4 3.0 3.0 4.0 4.4 3.9 3.5
## [19] 3.8 3.8 3.4 3.7 3.6 3.3 3.4 3.0 3.4 3.5 3.4 3.2 3.1 3.4 4.1 4.2 3.1 3.2
## [37] 3.5 3.1 3.0 3.4 3.5 2.3 3.2 3.5 3.8 3.0 3.8 3.2 3.7 3.3 3.2 3.2 3.1 2.3
## [55] 2.8 2.8 3.3 2.4 2.9 2.7 2.0 3.0 2.2 2.9 2.9 3.1 3.0 2.7 2.2 2.5 3.2 2.8
## [73] 2.5 2.8 2.9 3.0 2.8 3.0 2.9 2.6 2.4 2.4 2.7 2.7 3.0 3.4 3.1 2.3 3.0 2.5
## [91] 2.6 3.0 2.6 2.3 2.7 3.0 2.9 2.9 2.5 2.8 3.3 2.7 3.0 2.9 3.0 3.0 2.5 2.9
## [109] 2.5 3.6 3.2 2.7 3.0 2.5 2.8 3.2 3.0 3.8 2.6 2.2 3.2 2.8 2.8 2.7 3.3 3.2
## [127] 2.8 3.0 2.8 3.0 2.8 3.8 2.8 2.8 2.6 3.0 3.4 3.1 3.0 3.1 3.1 3.1 2.7 3.2
## [145] 3.3 3.0 2.5 3.0 3.4 3.0## [1] "sepal_l" "sepal_w" "petal_l" "petal_w" "class"Selecionando dados.
Como exemplo, vamos selecionar apenas as linhas que
correspondem à espécie Iris virginica. Os dados referentes à espécie estão na variável class.
A indexação é linha, coluna. A vírgula no final sem nada depois significa que é para pegar todos as colunas.
Se quisermos pegar só a coluna 3 usamos assim:
Podemos selecionar algumas colunas. No exemplo abaixo selecionamos as colunas 3 a 5:
Como a indexação é linha, coluna, dentro dos [] tem uma vírgula e nada antes da vírgula, isso significa pegar todas as linhas, depois da vírgula está especificado para pegar as colunas de 3 a 5.
Criando uma coluna
Criando uma coluna chamada índice com NA:
## [1] NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
## [26] NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
## [51] NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
## [76] NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
## [101] NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA
## [126] NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NA NAAdicionando dados:
## sepal_l sepal_w petal_l petal_w class indice
## 1 5.1 3.5 1.4 0.2 Iris-setosa 17.85
## 2 4.9 3.0 1.4 0.2 Iris-setosa 14.70
## 3 4.7 3.2 1.3 0.2 Iris-setosa 15.04
## 4 4.6 3.1 1.5 0.2 Iris-setosa 14.26
## 5 5.0 3.6 1.4 0.2 Iris-setosa 18.00
## 6 5.4 3.9 1.7 0.4 Iris-setosa 21.06Operadores
Podemos usar operadores:
igual ==
diferente !=
x ou y => x | y
x e y => x & y
Então, além de selecionar um parâmetro da variável class, aqui também selecionamos por outro parâmetro da variável sepal_l, usando o operador &.
## [1] 7.1 7.6 7.3 6.7 7.2 6.8 7.7 7.7 6.9 7.7 6.7 7.2 7.2 7.4 7.9 7.7 6.9 6.7 6.9
## [20] 6.8 6.7 6.72.5 Transformação de dados
Muitos valores das características tem escalas muito diferentes. Isso pode gerar dificuldades para algoritmos que usam todos esses valores para compor um valor único de comparação. Para resolver isso pode ser utilizada a normalização ou conversão de dados.
2.6 Técnicas de normalização
Min-max: altera valores extremos e organiza os internos dentro de um novo intervalo.
v’=((v-min(v))/(max(v) - min(v)))*(maxnovo - minnovo) + minnovo
Para exemplificar, vamos transformar a variável body_mass_g em uma escala de 0 a 1 e armazenar o resultado em uma nova variável chamada body_norm:
maxnovo<-1
minnovo<-0
dados2$body_norm<-NA
dados2$body_norm<-((dados2$body_mass_g-(min(dados2$body_mass_g, na.rm = T)))/(max(dados2$body_mass_g,na.rm = T) - (min(dados2$body_mass_g,na.rm = T)))*(maxnovo - minnovo) + minnovo)
head(dados2$body_norm)## [1] 0.2916667 0.3055556 0.1527778 NA 0.2083333 0.2638889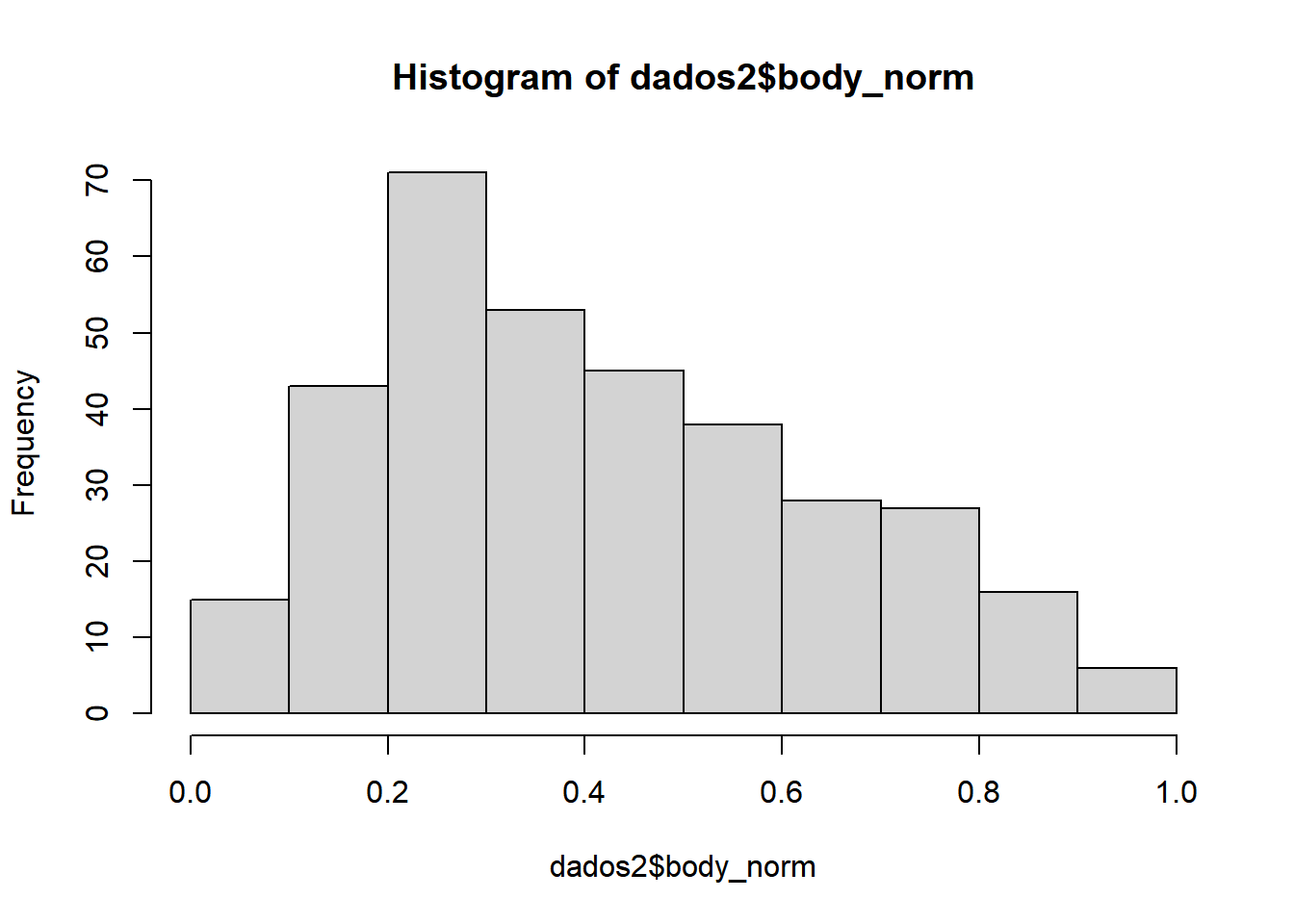
Outra opção é o Z-score, no qual os valores são reorganizados pela média e desvio-padrão do conjunto original:
v1=(v - média(v))/desvio padrão (v)
Vamos usar a mesma variável e transformar para Z-score:
dados2$body_norm2<-NA
dados2$body_norm2<-(dados2$body_mass_g - mean(dados2$body_mass_g, na.rm = T))/sd(dados2$body_mass_g, na.rm=T)
head(dados2$body_norm2)## [1] -0.5633167 -0.5009690 -1.1867934 NA -0.9374027 -0.6880121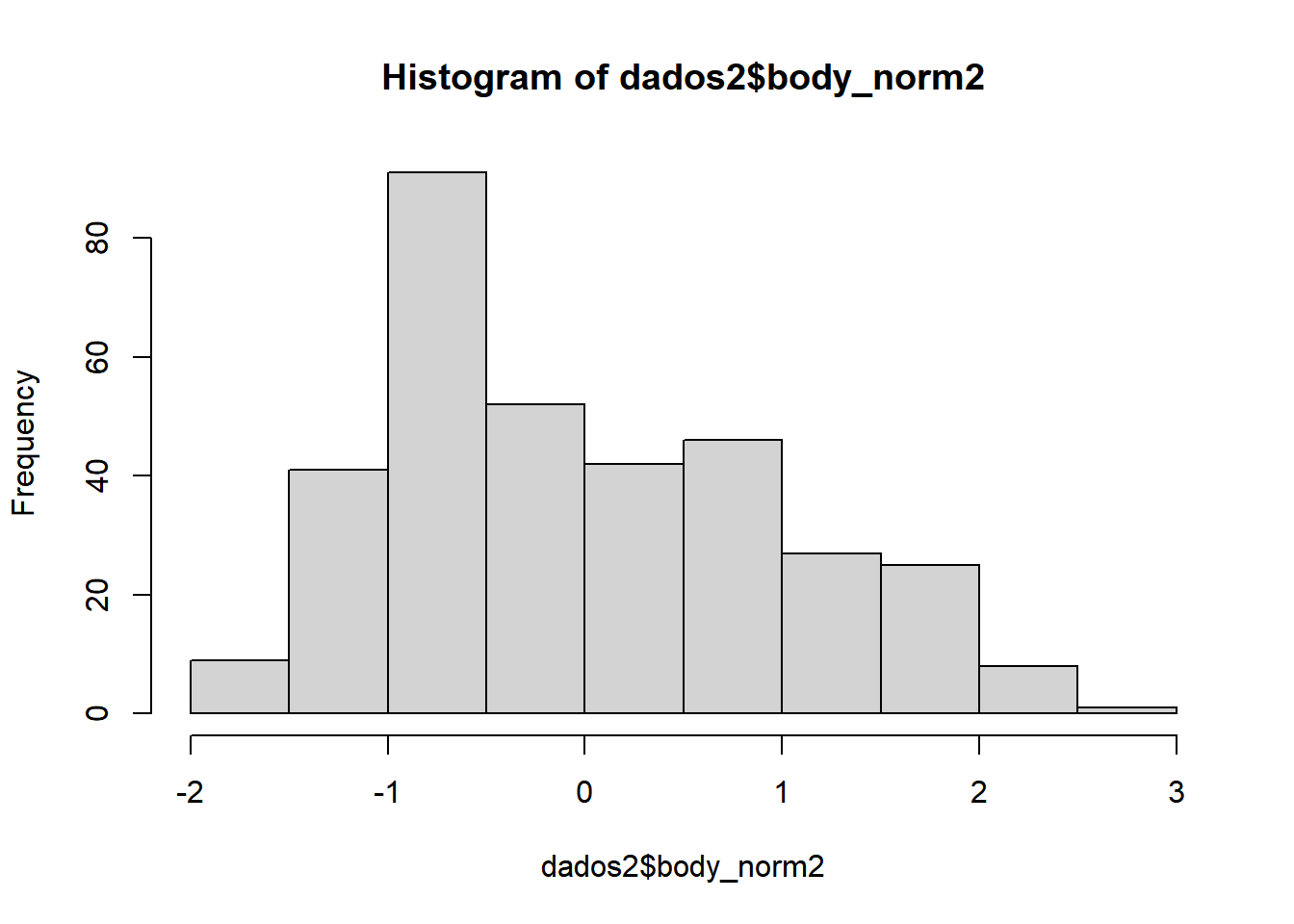
Para identificando o tipo de variável, podemos usar algumas funções:
## [1] TRUE## [1] FALSE## [1] FALSE## [1] TRUETransformando tipos de variáveis
Tem outras opções como as.numeric() e as.matrix()
## bill_length_mm bill_depth_mm flipper_length_mm
## 1 39.1 18.7 181
## 2 39.5 17.4 186
## 3 40.3 18.0 195
## 4 NA NA NA
## 5 36.7 19.3 193
## 6 39.3 20.6 190## [1] TRUE2.7 Juntar dados (R base)
Podemos usar o R para juntar dados de diferentes arquivos.
Primeiro vamos carregar alguns dados:
dados3<-read.xlsx("dados/dados_bioq.xlsx", sheet=1, colNames=T)
dados4<-read.xlsx("dados/dados_bioq.xlsx", sheet=2, colNames=T)
dados5<-read.xlsx("dados/dados_bioq.xlsx", sheet=3, colNames=T)Esses dados tem diferentes informações da mesma amostra:
## ID PN RON
## 1 1 71.14 NA
## 2 2 71.54 NA
## 3 3 68.63 86.55
## 4 4 68.79 86.52
## 5 5 68.83 86.61
## 6 6 70.17 NA## ID atividade
## 1 1 2.47
## 2 4 3.35
## 3 5 3.99
## 4 6 3.24
## 5 7 3.75
## 6 9 3.46## ID K
## 1 1 GA
## 2 2 <NA>
## 3 3 <NA>
## 4 4 GG
## 5 5 GA
## 6 6 AAJuntando os dados3 e dados4:
## ID PN RON atividade
## 1 1 71.14 NA 2.47
## 2 4 68.79 86.52 3.35
## 3 5 68.83 86.61 3.99
## 4 6 70.17 NA 3.24
## 5 7 74.75 NA 3.75
## 6 9 71.07 88.07 3.46Em dados6 tem as amostras que estão presentes em dados3 e também em dados4.
Juntando o objeto resultante com dados5:
## ID PN RON atividade K
## 1 1 71.14 NA 2.47 GA
## 2 4 68.79 86.52 3.35 GG
## 3 5 68.83 86.61 3.99 GA
## 4 6 70.17 NA 3.24 AA
## 5 7 74.75 NA 3.75 <NA>
## 6 9 71.07 88.07 3.46 GAMas também podemos manter apenas os dados de um ou outro objeto:
## ID PN RON atividade K
## 1 1 71.14 NA 2.47 GA
## 2 4 68.79 86.52 3.35 GG
## 3 5 68.83 86.61 3.99 GA
## 4 6 70.17 NA 3.24 AA
## 5 7 74.75 NA 3.75 <NA>
## 6 9 71.07 88.07 3.46 GA## ID PN RON atividade K
## 1 1 71.14 NA 2.47 GA
## 2 2 NA NA NA <NA>
## 3 3 NA NA NA <NA>
## 4 4 68.79 86.52 3.35 GG
## 5 5 68.83 86.61 3.99 GA
## 6 6 70.17 NA 3.24 AAOu ainda manter todas as amostras dos 2 objetos
dados10<-merge.data.frame(dados6, dados5, by.x = "ID", by.y = "ID", all.x = T, all.y = T)
head(dados10)## ID PN RON atividade K
## 1 1 71.14 NA 2.47 GA
## 2 2 NA NA NA <NA>
## 3 3 NA NA NA <NA>
## 4 4 68.79 86.52 3.35 GG
## 5 5 68.83 86.61 3.99 GA
## 6 6 70.17 NA 3.24 AA