16 報告と再現性

こ れまでの章では、 R でメタ分析を行うために使用できる様々なテクニック、アプローチ、戦略について説明してきた。しかし、統計解析の実行は、実際にはメタ分析「プロセス」全体のごく一部の割合を占めるに過ぎない。「現場」では、以下のような「事件」が発生する。
R のコードにエラーが見つかったため、解析の一部を変更してやり直さなければならない。
共同研究者や査読者は、別のアプローチやモデルの使用、あるいは追加の感度分析を行うことを提案している。
分析作業の一部を共同研究者の一人に委任する必要があり、現在の作業状況を送らなければならない。
プロジェクトをしばらく中断していたため、再開するころにはいろいろなことを忘れてしまっている。
解析結果をプロジェクトの共同研究者と共有したいが、共同研究者は R を知らず、R Studio はインストールすらされていない。
これらはほんの一部のシナリオであるが、 R でメタ分析を行う際の再現可能なワークフローが、あなたや一緒に働く人々にとって有益であることを説明している。また、再現性を目指すことは、オープンサイエンスの実践の基礎でもある。完全に再現可能なメタ分析は、私たちがどのように結果に至ったかを他の人に可能な限り明らかにするものである。
R Studio は、再現性のあるワークフローを作成し、協力を得るために最適なツールである。本章では、分析の再現、報告、普及のための3つのツールを紹介する R Projects、R Markdown および Open Science Framework である。
16.1 R Project の利用
解析を始めるには、まずR Studioで R Project を立ち上げるのがよいだろう R Projectは、コンピュータ上のフォルダーに新しい環境を作成する。このフォルダには、分析に必要なすべてのデータと R コードが保存される R Project で分析を行うということは、作成したすべてのオブジェクトが Project 環境に一時的に保存され、次に開いたときにアクセスできるようになることを意味する。新しい R Project を作成するには、R Studio ウィンドウの右上にある R Project フィールドをクリックし、ドロップダウンメニューから New Project… を選択する。
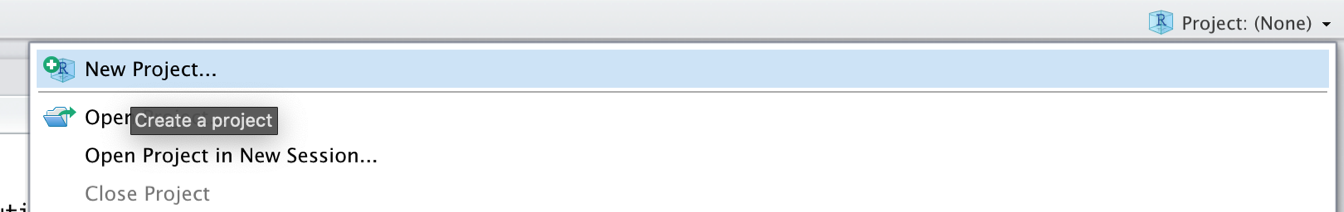
次に、コンピュータ上に新しいフォルダである New Directory を作成する。これがプロジェクトの作業ディレクトリとなる。
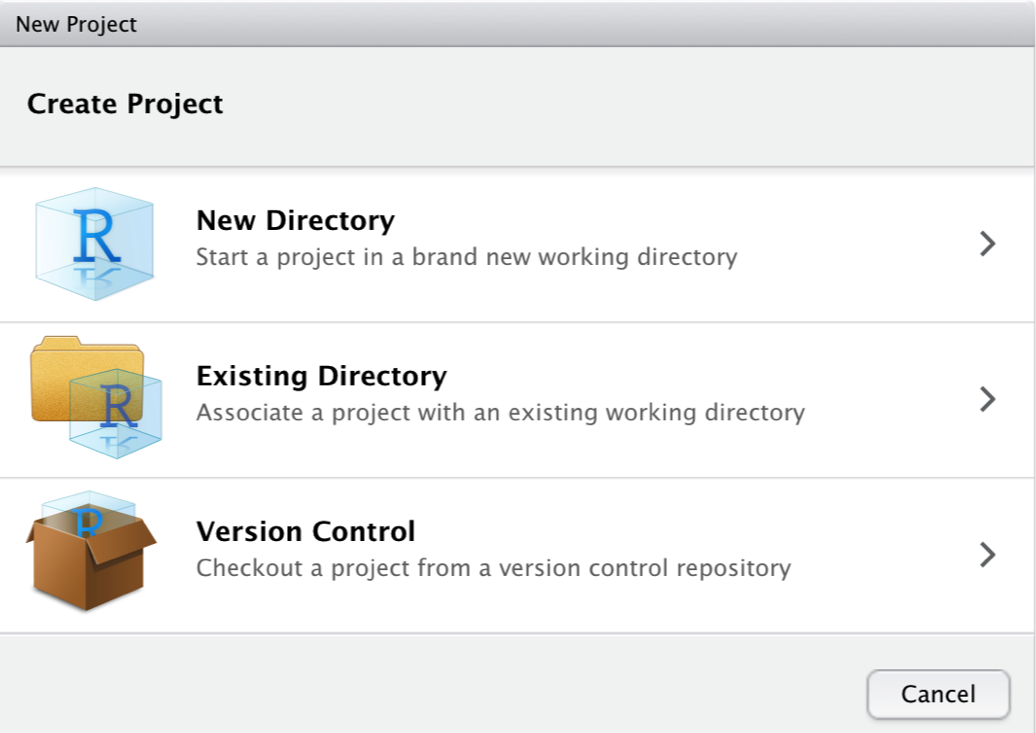
そして、New Project をクリックする。
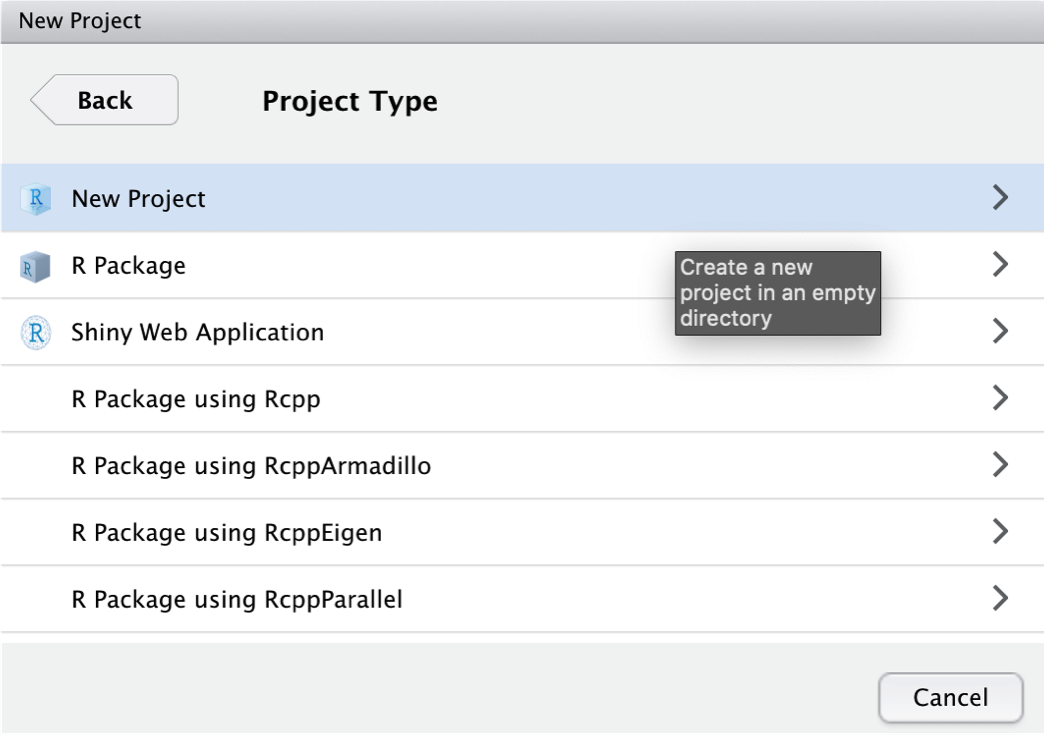
新しいプロジェクトに「Meta-Anallysis Project」という名前をつける。プロジェクトフォルダは、~Documents/R に格納される。
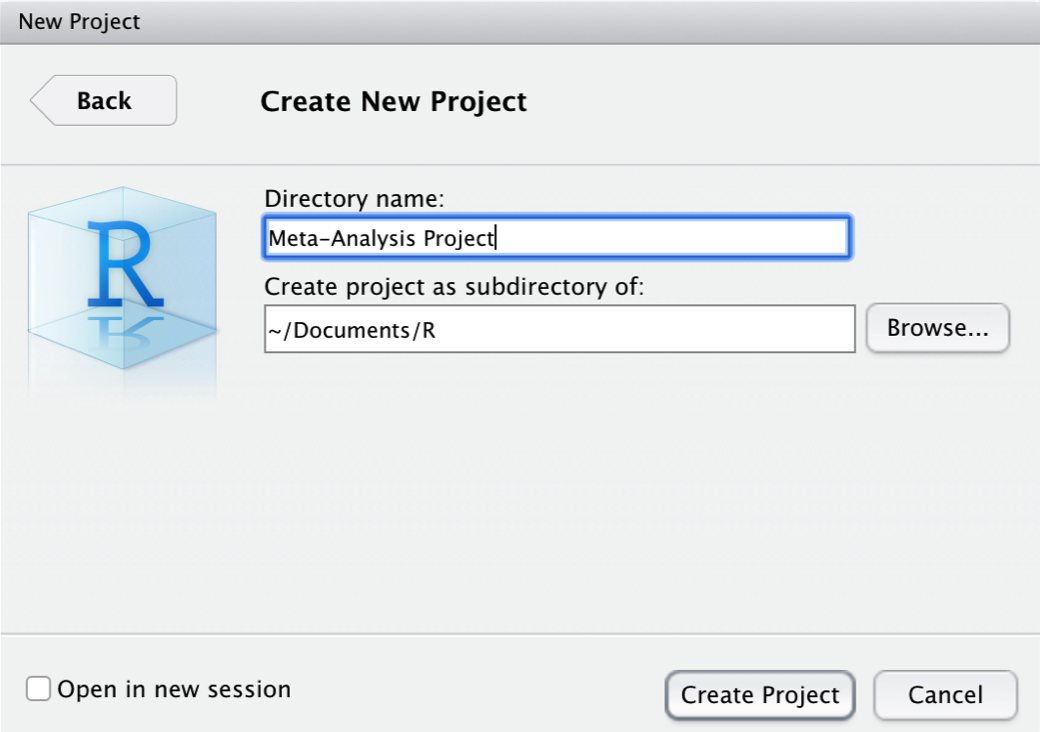
プロジェクトの作成をクリックすると、 R Project が設定される。 R Project の大きな特徴は、参照したいファイルへの絶対パスを使用する必要がないことである。ファイル名、またはファイルが(サブ)フォルダーにある場合は、フォルダー名とファイル名だけを使用する。例えば、データセット data.xlsx をサブフォルダー “data” に格納する。{openxlsx} パッケージ(Chapter 2.4)を使用すると、相対パスでデータセットをインポートすることができる。
read_excel("data/data.xlsx")16.2 R Markdown で再現性のあるレポートを作成
Markdown はテキストフォーマットのためのシンプルなマークアップ言語である。R Markdown (Xie, Allaire, and Grolemund 2018)は Markdown の拡張機能で、プレーンテキスト、 R コード、 R 出力を1つのドキュメントに簡単にまとめることができるようになっている。このため、R Markdown は非常に便利なレポート作成ツールとなっている。R Markdown を使用すると、分析で使用したすべてのコード、コードによって生成された出力を含む HTML または PDF ファイルを作成でき、各分析ステップで行ったことに関する詳細な情報を追加することが可能にある。
R Markdown ファイルを R Studio で構築するのはとても簡単である。R Studio ウィンドウの左上隅にある、緑色の「プラス」記号のついた白いシンボルをクリックすればよいのである。そして、ドロップダウンメニューから、R Markdown… をクリックする。
新しい R Markdown ドキュメントの名前を定義すると、R Studio ウィンドウの左上隅にポップアップ表示されるはずである。
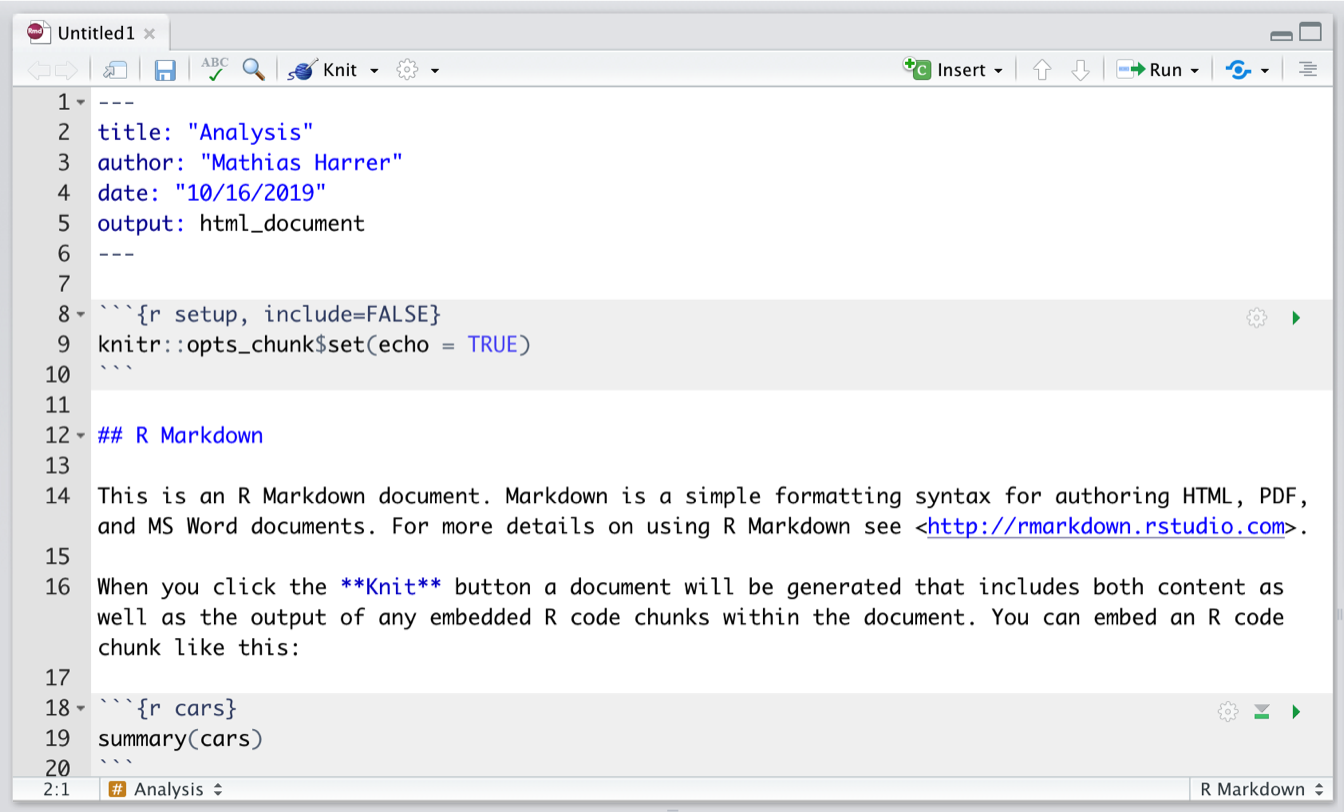
このファイルには、すでにいくつかの例示的な内容が含まれているが、最初の6行を除いて削除することが可能である。
---
title: "Analysis"
author: "Author Name"
date: "10/16/2020"
output: html_document
---この部分はいわゆる YAML ヘッダーである。これは、ドキュメントのタイトル、著者、日付、エクスポート形式を記述する。出力形式は html_document を選択した。これはドキュメントがレンダリングされると HTML ページとしてエクスポートされることを意味している。
R Markdown ドキュメント はすべて、2つの部分から構成されている。 普通の Markdown テキスト、そして、グレーで示されたいわゆる R チャンクである。R Markdown ドキュメントのテキスト部分がどのようにフォーマットされるかについては詳しく説明しない。オンラインの cheat sheet は、Markdown 構文を学び始める素晴らしいリソースである(20分程度で読むことができる)。一方、 R コードのチャンクは、通常コンソールに入力するコードを含んでいるだけである。ドキュメントの右上にある Insert フィールドをクリックすることで、新しいコードチャンクを追加することができる。各チャンクの上にある小さな緑の三角形をクリックし、コードを実行することができる。
文書を書き終えたら、左上の編み目のマークをクリックして、HTML、PDF、MS Word 文書として書き出すことができる。これにより、すべてのテキスト、コード、出力を含む文書がレンダリングされ、定義されたフォーマットでエクスポートされる。最終的な文書は、自動的にプロジェクトフォルダに保存される。

16.3 OSF レポジトリ
オープンサイエンス・フレームワーク(Open Science Framework, OSF)は、研究におけるコラボレーションと再現性を促進するためのオープンソースのオンラインプラットフォームである。OSF にはオンラインリポジトリがあり、研究者が研究資料を預けて共同研究を行い、研究プロセスのすべてのステップを(より)透明化することが可能である。OSF は、過去10年間に大きな勢いを得たオープンサイエンス運動の急先鋒である。
すべてのメタ分析者は、収集したデータと分析に使用した R コードにオープンアクセスすることで、研究と分析プロセスを一般に公開することが推奨される。OSFはこれを行うための素晴らしいツールである。自分で作成したすべてのリポジトリは、デフォルトで非公開になっており、いつ、何を公開するかは、あなた次第なのである。以下では、 R で OSF リポジトリを設定する方法、ファイルのアップロードとダウンロード、共同研究者を追加する方法を紹介する。
16.3.1 アクセス・トークン
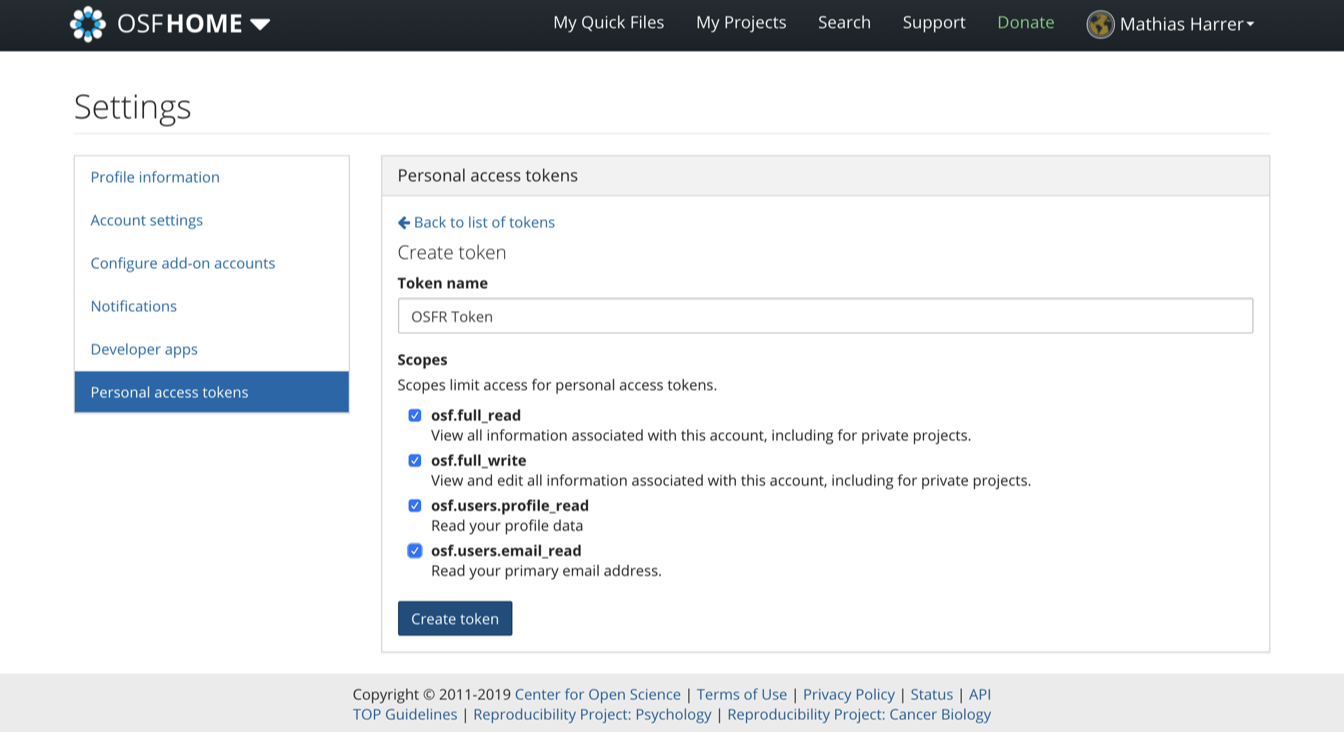
OSF を使い始めるには、まずウェブサイトで個人アカウントを作成する必要がある。アカウントが作成されたら、 R を使って直接リポジトリを操作できるように、アクセストークンを生成する必要がある。アクセストークンを取得するには、Profile > Settings > Personal access tokens に移動する必要がある。そこで、Create token をクリックする。
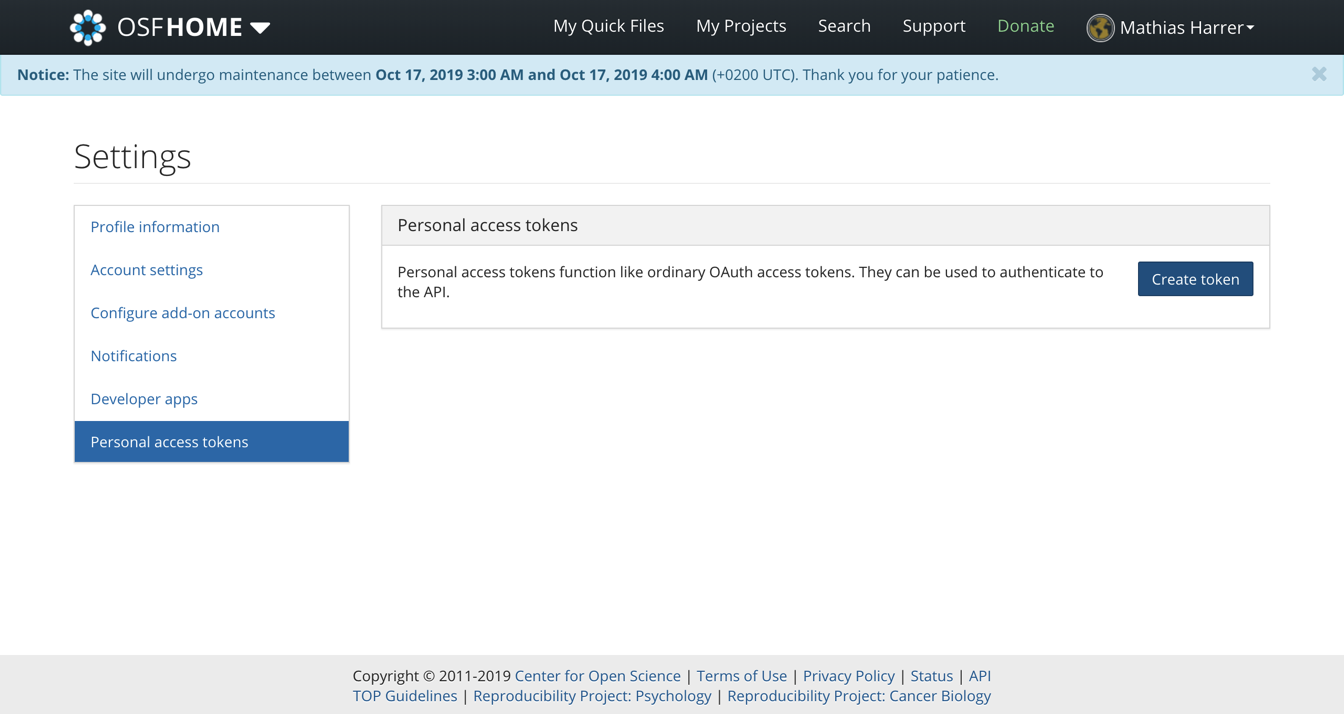
次に、Scopes の下にあるすべてのボックスにチェックを入れ、Create token を再度クリックする。すると、個人用のアクセストークンが表示されるはずである。このトークンをコピーして保存しておく。
16.3.2 パッケージと認証について
{OSF} リポジトリに R 経由で直接アクセスするには、{osfr} パッケージ (Wolen et al. 2020) を使用する。このパッケージの機能を使う前に、まずアクセストークンを使って認証する必要がある。これを行うには、osf_auth 関数を使用して、受け取ったばかりのアクセストークンを渡す (以下に表示されるトークンはデタラメ)。
16.3.3 レポジトリの設定
{osfr} を使うと、 R を使った OSF リポジトリを初期化することが可能である。新しいメタ分析プロジェクトに取り組んでいて、データとR Markdown スクリプトを OSF リポジトリにアップロードしたいと想像してみよう。リポジトリの名前は “Meta-Analysis Project” とする。
新しいリポジトリを作成するには、osf_create_project 関数を使用する。新しい OSF リポジトリを R に meta_analysis_project という名前で保存する。
meta_analysis_project <- osf_create_project("Meta-Analysis Project")osf_open 関数を使用すると、新しく作成したリポジトリにオンラインでアクセスできるようになる。
osf_open(meta_analysis_project)
リポジトリが作成されたので、次に コンポーネントを追加していく。OSF では、コンポーネントはコンピュータのフォルダのように動作した。例えば、データセット用のコンポーネントと R Markdown スクリプト用のコンポーネントの2つを作成したいとする。これを行うには、 osf_create_component 関数を使用することが可能である。この関数に R のリポジトリオブジェクト( meta_analysis_project )を渡し、新しいコンポーネントのタイトルを設定しなければならない。
scripts <- osf_create_component(meta_analysis_project,
title = "Analysis Scripts")
datasets <- osf_create_component(meta_analysis_project,
title = "Datasets")リポジトリのオンラインページに行くと、2つのコンポーネントが追加されていることがわかる。
16.3.4 アップロードとダウンロード
OSF リポジトリにデータをアップロードするには、 osf_upload 関数を使用する。この関数では、ファイルを追加するコンポーネントと、アップロードするファイルのパスを指定する必要がある。例えば、“Analysis.rmd” というR Markdownスクリプトをアップロードしたい場合、現在 R プロジェクトのサブフォルダ “scripts” に保存されているものとする。アップロードするには、次のコードを使用する。
osf_upload(scripts, "scripts/Analysis.rmd")ファイルが正常にアップロードされたかどうかを確認するには、 osf_ls_files 関数を使用してコンポーネントのコンテンツにアクセスする。
osf_ls_files(scripts)## # A tibble: 2 x 3
## name id meta
## <chr> <chr> <list>
## 1 Analysis.rmd 1db74s7bfcf91f0012567572l <named list [3]>アップロードが成功したことが出力で確認可能である。ファイルをダウンロードするには、 osf_ls_files 関数の出力から行を選択し、osf_download 関数でそれを使用して、ファイルをコンピュータのプロジェクトフォルダにダウンロードしなおせばよい。
osf_download(osf_ls_files(scripts)[1,])16.3.5 コラボレーション、オープンアクセス、事前登録
OSF のリポジトリサイトでは、Contributorsという項目で共同研究者を追加することも可能である。
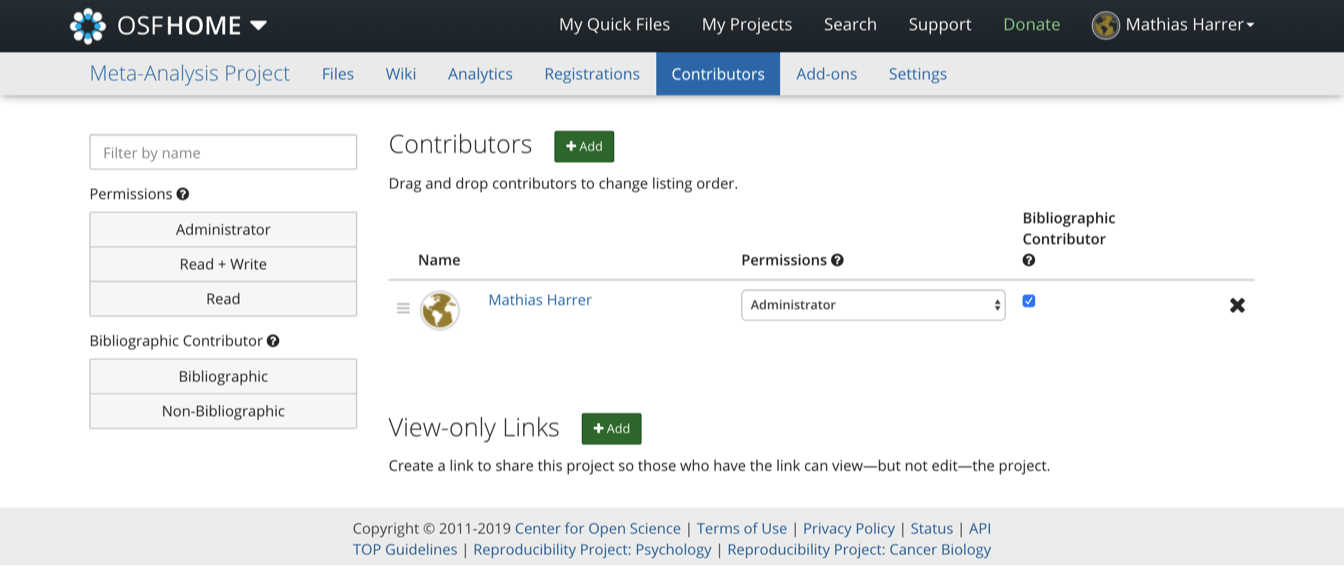
ウェブサイト右上の Make Public ボタンをクリックすることで、いつでもリポジトリを public にすることが可能である。

Chapter 1.4.2 で、解析計画と事前登録が高品質なメタ分析に不可欠な部分であることを説明してきた。OSFでは、私たちのプロジェクトのために、オープンにアクセスできる事前登録を作成することができ、とても便利である。上部にある登録ボタンをクリックし、新規登録を作成するだけでよいのである。これにより、OSF Registries のウェブサイトが表示され、分析計画など、計画中の研究についての詳細情報を提供することが可能である。
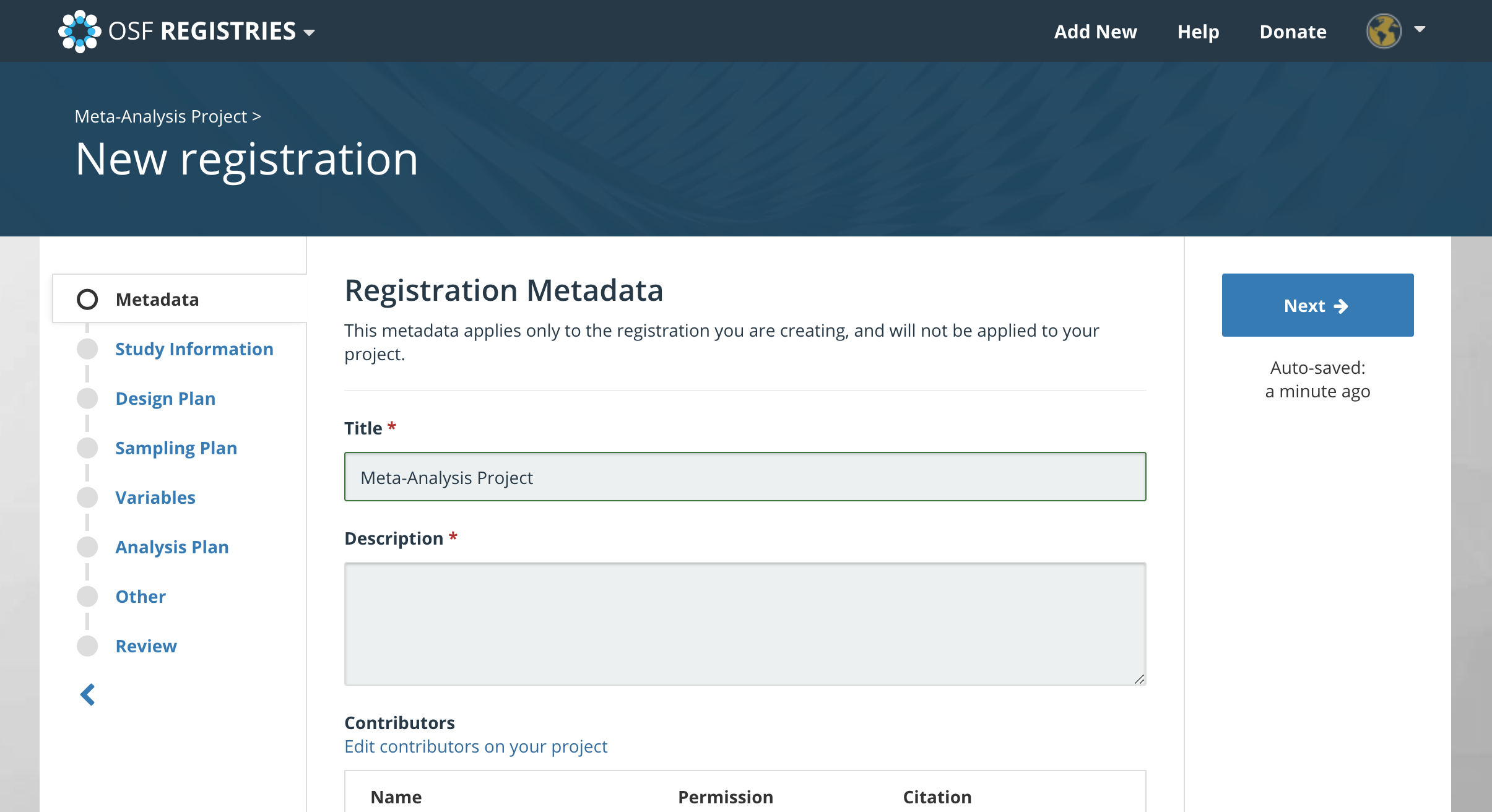
必要な情報をすべて指定した後、試験を登録することが可能である。これにより、一意のID(例:osf.io/q2jp7)でアクセス可能な登録項目が作成される。登録が完了した後は、検索計画、仮説、分析戦略を変更することはできない。
\[\tag*{$\blacksquare$}\]