Capítulo 2 Técnicas de Manipulación de Datos
2.1 Ordenación y estructuración de los datos (Data Tidying)
Como en toda área profesional, existen algunos principios básicos que deben respetar quienes trabajan en ciencia de datos. A esto se le denomina Data Tidying. Es el primer paso del flujo de trabajo e involucra la limpieza y organización de los datos con una estructura estándar, lo cual facilita su posterior manipulación, modelado y visualización.
Principios generales
- Cada variable está en una columna.
- Cada observación diferente de esa variable está en una fila.
- Debe haber una tabla distinta para cada “clase” de variable, p.e. secuencias, datos ambientales, etc
- Si tienes múltiples tablas, debe existir una columna en cada tabla que permita enlazarlas (identificador).
- Se debe generar archivo texto complementario con descripción de la fuente de la base de datos, diseño experimental, variables, unidades, etc.

Imagen tomada de https://r4ds.hadley.nz/data-tidy.html
2.2 Manipulación de datos con data.table
El programa data.table es un paquete R que proporciona una versión mejorada para el manejo de data.frames. Los data.frames son una estructura de datos estándar de manejo de datos en R. Este programa simplifica la programación y reduce tremendamente el tiempo de cálculo. Mantiene juntas operaciones de manipulación de datos, como subconjuntos, grupos, actualizaciones, uniones, etc. Las principales características incluyen:
- Una sintaxis concisa y consistente, independientemente del conjunto de operaciones que le gustaría realizar para lograr su objetivo final.
- La realización de los análisis de forma fluida.
- La optimización automática de las operaciones internamente y de manera muy eficaz, al conocer con precisión los datos necesarios para cada operación, lo que lleva a un código muy rápido y eficiente en memoria.
A continuación se presenta un esquema de la estructura básica de operación de data.table:
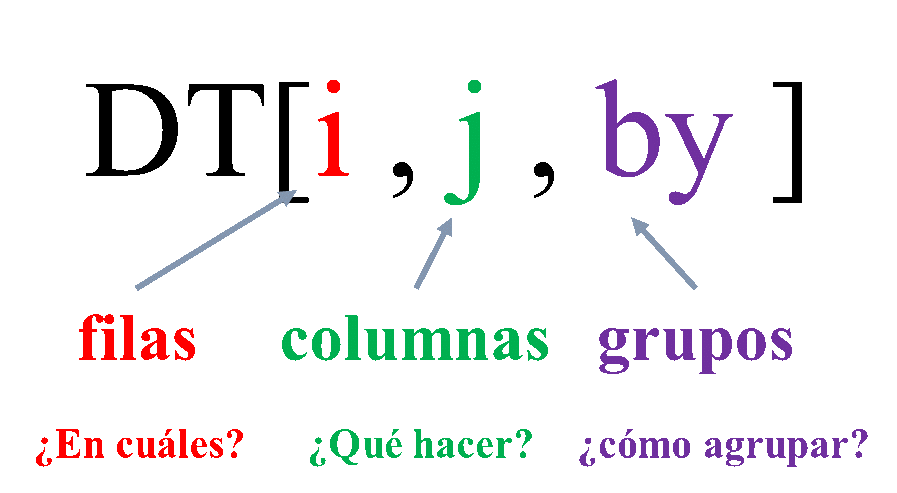
2.2.1 Creación de un conjunto de datos con la función data.table
library(data.table)
datatable <- data.table(x =c("a", "b", "c", "d", "e"),
y = c(1, 2, 3, 4, 5))
str(datatable)
#> Classes 'data.table' and 'data.frame': 5 obs. of 2 variables:
#> $ x: chr "a" "b" "c" "d" ...
#> $ y: num 1 2 3 4 5
#> - attr(*, ".internal.selfref")=<externalptr>Se pueden autocompletar datos
DT = data.table(x=rep(c("b","a","c"),each=3), y=c(1,3,6), v=1:9) #repite 3 veces cada valor en x
DT
#> x y v
#> 1: b 1 1
#> 2: b 3 2
#> 3: b 6 3
#> 4: a 1 4
#> 5: a 3 5
#> 6: a 6 6
#> 7: c 1 7
#> 8: c 3 8
#> 9: c 6 92.2.2 Operaciones básicas para manejo de filas
DT[2] # visualiza la segunda fila
#> x y v
#> 1: b 3 2
DT[3:2] # visualiza de la tercera a la segunda fila
#> x y v
#> 1: b 6 3
#> 2: b 3 2
DT[c(2, 3)] # visualiza la fila2 y la fila3
#> x y v
#> 1: b 3 2
#> 2: b 6 3
DT[order(x)] # ordena de acuerdo a la columna x
#> x y v
#> 1: a 1 4
#> 2: a 3 5
#> 3: a 6 6
#> 4: b 1 1
#> 5: b 3 2
#> 6: b 6 3
#> 7: c 1 7
#> 8: c 3 8
#> 9: c 6 9
DT[y>2] # llama todas las filas que tengan valores de y > 2
#> x y v
#> 1: b 3 2
#> 2: b 6 3
#> 3: a 3 5
#> 4: a 6 6
#> 5: c 3 8
#> 6: c 6 9
DT[y>2 & v>5] # llama filas de y > 2 y(&) valores de v > 5
#> x y v
#> 1: a 6 6
#> 2: c 3 8
#> 3: c 6 9
DT[!2:4] # llama todas las filas excepto de la 2 a la 4
#> x y v
#> 1: b 1 1
#> 2: a 3 5
#> 3: a 6 6
#> 4: c 1 7
#> 5: c 3 8
#> 6: c 6 9
DT[-(2:4)] # llama todas las filas excepto de la 2 a la 4
#> x y v
#> 1: b 1 1
#> 2: a 3 5
#> 3: a 6 6
#> 4: c 1 7
#> 5: c 3 8
#> 6: c 6 92.2.3 Selección de subconjuntos de filas
DT["a", on="x"] # busca las filas con “a” dentro de columna x
#> x y v
#> 1: a 1 4
#> 2: a 3 5
#> 3: a 6 6
DT["a", on=.(x)] # igual pero sin paréntesis en columnas
#> x y v
#> 1: a 1 4
#> 2: a 3 5
#> 3: a 6 6
DT[2:3, sum(v)] # calcula suma de valores en v, filas 2 y 3
#> [1] 5
DT[2:3, .(sum(v))] # igual pero nombrar columna como V1
#> V1
#> 1: 5
DT[2:3, .(sv=sum(v))] # igual pero nombra columna como “sv”
#> sv
#> 1: 5
colnames(DT) # visualiza los nombres de las columnas
#> [1] "x" "y" "v"
dim(DT) # visualiza las dimensiones de DT
#> [1] 9 32.2.4 Creación de nuevas variables.
Se pueden crear nuevas bases de datos con nuevas variables a partir de la data.table existente. Para la creación de nuevas variables se pueden realizar por ejemplo operaciones aritméticas entre variables existentes.
nueva_DT <- DT[, .(y, z=y*v)]
nueva_DT
#> y z
#> 1: 1 1
#> 2: 3 6
#> 3: 6 18
#> 4: 1 4
#> 5: 3 15
#> 6: 6 36
#> 7: 1 7
#> 8: 3 24
#> 9: 6 542.2.5 Operaciones básicas para el manejo de columnas
DT[, v] # visualiza los elementos de la columna v como vector
#> [1] 1 2 3 4 5 6 7 8 9
DT[["v"]] # visualiza los elementos de la columna v como vector
#> [1] 1 2 3 4 5 6 7 8 9
DT[, list(v)] # visualiza los elementos de la columna v en formato data.table
#> v
#> 1: 1
#> 2: 2
#> 3: 3
#> 4: 4
#> 5: 5
#> 6: 6
#> 7: 7
#> 8: 8
#> 9: 9
DT[, .(v)] # visualiza columna v. El parámetro .() es un alias para list()
#> v
#> 1: 1
#> 2: 2
#> 3: 3
#> 4: 4
#> 5: 5
#> 6: 6
#> 7: 7
#> 8: 8
#> 9: 9
DT[, 2, with=FALSE] # visualiza segunda columna
#> y
#> 1: 1
#> 2: 3
#> 3: 6
#> 4: 1
#> 5: 3
#> 6: 6
#> 7: 1
#> 8: 3
#> 9: 6
DT[, sum(v)] # calcula la suma de la columna v, resultado como vector
#> [1] 45
DT[, .(sum(v))] # calcula sumatoria, crea un data.table con V1 como nueva columna
#> V1
#> 1: 45
DT[, .(v, v*2)] # visualiza una tabla con la columna v y otra con los valores de v*2
#> v V2
#> 1: 1 2
#> 2: 2 4
#> 3: 3 6
#> 4: 4 8
#> 5: 5 10
#> 6: 6 12
#> 7: 7 14
#> 8: 8 16
#> 9: 9 182.2.6 Selección de subconjuntos de columnas
DT[c(1,3), y:v] # visualiza datos de filas 1 y 3 de las columnas de y a v.
#> y v
#> 1: 1 1
#> 2: 6 3
DT[, sum(v), by=x] # suma valores de v agrupados por x, preserva orden original
#> x V1
#> 1: b 6
#> 2: a 15
#> 3: c 24
DT[, sum(v), keyby=x] # suma valores de v agrupados por x, ordena ascendente
#> x V1
#> 1: a 15
#> 2: b 6
#> 3: c 24
DT["b", sum(v*y), on="x"] # para valores de "b" en x calcula la suma de v*y
#> [1] 25
DT[x!="a", sum(v), by=x] # para cada subgrupo dentro de “x” excepto “a” calcula suma de v
#> x V1
#> 1: b 6
#> 2: c 242.2.7 Conversión a formato data.table
Para el siguiente ejemplo se puede convertir la popular base de datos sobre la variación morfológica de la flor Iris, pasando de un data.frame a un data.table con la función data.table().
data(iris)
class(iris)
#> [1] "data.frame"
DT <- as.data.table(iris)
class(DT)
#> [1] "data.table" "data.frame"2.2.8 Realización de cálculos en columnas y agrupación
data(iris)
str(iris)
#> 'data.frame': 150 obs. of 5 variables:
#> $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
#> $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
#> $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
#> $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
#> $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
DT[, mean(Sepal.Length), by=Species] # calcula la media de variable Sepal.Length agrupada por especie
#> Species V1
#> 1: setosa 5.006
#> 2: versicolor 5.936
#> 3: virginica 6.588
DT[, list(.N, mean(Sepal.Length), sd(Sepal.Length)), by=Species] # se pueden listar varias métricas a calcular manteniendo agrupación
#> Species N V2 V3
#> 1: setosa 50 5.006 0.3524897
#> 2: versicolor 50 5.936 0.5161711
#> 3: virginica 50 6.588 0.6358796
DT[, .(mean=(mean(Sepal.Length)), sd=(sd(Sepal.Length))), by=Species] # se puede asignar encabezado para variable calculada
#> Species mean sd
#> 1: setosa 5.006 0.3524897
#> 2: versicolor 5.936 0.5161711
#> 3: virginica 6.588 0.6358796Con estes programa se pueden realizar cálculos más complejos sobre las variables en la base de datos. Se puede, por ejemplo, crear una nueva variable (Sepal.Area)producto de multiplicar Sepal.Length por Sepal.Width. El valor obtenido se puede redondear al valor más cercano a los 10 cm2. Finalmente se puede realizar un conteo con el parámetro .N de la cantidad de individuos en cada rango de area.
DT[, .N, by = 10 * round(Sepal.Length*Sepal.Width / 10)]
#> round N
#> 1: 20 117
#> 2: 10 29
#> 3: 30 42.2.9 Fusión de tablas
Una de las funciones más importantes en el manejo de bases de datos se relaciona con la capacidad de fusionar diferentes bases de datos a partir de identificadores o elementos comunes en ellas
DT = data.table(x=rep(c("b","a","c"),each=3), y=c(1,3,6), v=1:9)
NT = data.table(x=c("c","b"), v=8:7, k=c(4,2))
NT
#> x v k
#> 1: c 8 4
#> 2: b 7 2
DT[NT, on="x"] # elementos de x en NT presentes en DT, autocompleta
#> x y v i.v k
#> 1: c 1 7 8 4
#> 2: c 3 8 8 4
#> 3: c 6 9 8 4
#> 4: b 1 1 7 2
#> 5: b 3 2 7 2
#> 6: b 6 3 7 2
NT[DT, on="x"] # elementos de x en DT presentes en NT, autocompleta incluye NA
#> x v k y i.v
#> 1: b 7 2 1 1
#> 2: b 7 2 3 2
#> 3: b 7 2 6 3
#> 4: a NA NA 1 4
#> 5: a NA NA 3 5
#> 6: a NA NA 6 6
#> 7: c 8 4 1 7
#> 8: c 8 4 3 8
#> 9: c 8 4 6 9
DT[!NT, on="x"] # elementos ausentes en NT que están en DT
#> x y v
#> 1: a 1 4
#> 2: a 3 5
#> 3: a 6 6
DT[NT, on="x", mult="first"] # primera fila de cada grupo luego de fusión
#> x y v i.v k
#> 1: c 1 7 8 4
#> 2: b 1 1 7 2
DT[NT, sum(v), by=.EACHI, on="x"] # fusiona y calcula suma de v para cada grupo de x
#> x V1
#> 1: c 24
#> 2: b 62.2.10 Algunos atributos especiales de data.table
DT <- data.table( a = c(1L, 2L), b = LETTERS[1:10], c = c(1:10))
DT
#> a b c
#> 1: 1 A 1
#> 2: 2 B 2
#> 3: 1 C 3
#> 4: 2 D 4
#> 5: 1 E 5
#> 6: 2 F 6
#> 7: 1 G 7
#> 8: 2 H 8
#> 9: 1 I 9
#> 10: 2 J 10
DT[.N ] # visualiza última fila
#> a b c
#> 1: 2 J 10
DT[.N - 1] # visualiza penultima fila
#> a b c
#> 1: 1 I 9
DT[, .N] # realiza conteo del número filas en DT
#> [1] 10
DT[, .N, by=a] # cuenta los elementos en cada grupo de "a"
#> a N
#> 1: 1 5
#> 2: 2 5
DT[, .SD[1], by=a] # visualiza primera fila para cada grupo en “a”
#> a b c
#> 1: 1 A 1
#> 2: 2 B 2
DT[, .I[1], by=a] # visualiza número de fila donde empieza cada grupo de “a”
#> a V1
#> 1: 1 1
#> 2: 2 2También se pueden generar nuevas columnas con el parámetro grp numerando por ejemplo los grupos de “a” de 1…n
DT[, grp := .GRP, by=a]
DT
#> a b c grp
#> 1: 1 A 1 1
#> 2: 2 B 2 2
#> 3: 1 C 3 1
#> 4: 2 D 4 2
#> 5: 1 E 5 1
#> 6: 2 F 6 2
#> 7: 1 G 7 1
#> 8: 2 H 8 2
#> 9: 1 I 9 1
#> 10: 2 J 10 2También es posible remover la nueva columna creada
DT[, z:=NULL] # remueve columna creada
#> Warning in `[.data.table`(DT, , `:=`(z, NULL)): Column 'z'
#> does not exist to remove
DT
#> a b c grp
#> 1: 1 A 1 1
#> 2: 2 B 2 2
#> 3: 1 C 3 1
#> 4: 2 D 4 2
#> 5: 1 E 5 1
#> 6: 2 F 6 2
#> 7: 1 G 7 1
#> 8: 2 H 8 2
#> 9: 1 I 9 1
#> 10: 2 J 10 22.3 Manipulación de datos con dplyr
El paquete dplyr es quizás una de las herramientas más útiles en la actualidad para manipular, limpiar y resumir datos. Hace que la exploración y la manipulación de datos sean fáciles y rápidas. El paquete dplyr es uno de los paquetes más poderosos y populares de R. Fue escrito por Hadley Wickham, quien ha escrito otros paquetes del Tidyverse como ggplot2 y tidyr.Proporciona un gramática consistente, basada en verbos, ayudan a resolver los desafíos de manipulación de datos más comunes. A continuación, algunos ejemplos:
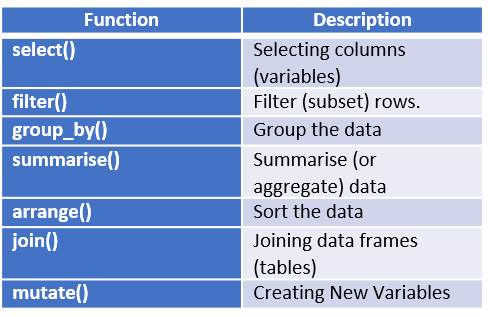
2.3.1 Algunas funciones básicas
Para la práctica con dplyr se utilizará un conjunto de datos del paquete palmerpenguins sobre mediciones morfológicas en tres especies de pinguinos, tomadas en la Estación Palmer, Antarctica.

Imagen creada por1 (https://allisonhorst.github.io/palmerpenguins/)
library(dplyr)
#>
#> Attaching package: 'dplyr'
#> The following objects are masked from 'package:data.table':
#>
#> between, first, last
#> The following objects are masked from 'package:stats':
#>
#> filter, lag
#> The following objects are masked from 'package:base':
#>
#> intersect, setdiff, setequal, union
library(palmerpenguins)
data(penguins)
str(penguins)
#> tibble [344 x 8] (S3: tbl_df/tbl/data.frame)
#> $ species : Factor w/ 3 levels "Adelie","Chinstrap",..: 1 1 1 1 1 1 1 1 1 1 ...
#> $ island : Factor w/ 3 levels "Biscoe","Dream",..: 3 3 3 3 3 3 3 3 3 3 ...
#> $ bill_length_mm : num [1:344] 39.1 39.5 40.3 NA 36.7 39.3 38.9 39.2 34.1 42 ...
#> $ bill_depth_mm : num [1:344] 18.7 17.4 18 NA 19.3 20.6 17.8 19.6 18.1 20.2 ...
#> $ flipper_length_mm: int [1:344] 181 186 195 NA 193 190 181 195 193 190 ...
#> $ body_mass_g : int [1:344] 3750 3800 3250 NA 3450 3650 3625 4675 3475 4250 ...
#> $ sex : Factor w/ 2 levels "female","male": 2 1 1 NA 1 2 1 2 NA NA ...
#> $ year : int [1:344] 2007 2007 2007 2007 2007 2007 2007 2007 2007 2007 ...
glimpse(penguins)
#> Rows: 344
#> Columns: 8
#> $ species <fct> Adelie, Adelie, Adelie, Adelie, ~
#> $ island <fct> Torgersen, Torgersen, Torgersen,~
#> $ bill_length_mm <dbl> 39.1, 39.5, 40.3, NA, 36.7, 39.3~
#> $ bill_depth_mm <dbl> 18.7, 17.4, 18.0, NA, 19.3, 20.6~
#> $ flipper_length_mm <int> 181, 186, 195, NA, 193, 190, 181~
#> $ body_mass_g <int> 3750, 3800, 3250, NA, 3450, 3650~
#> $ sex <fct> male, female, female, NA, female~
#> $ year <int> 2007, 2007, 2007, 2007, 2007, 20~
sample_n(penguins,3) # selección aleatoria de un número de datos
#> # A tibble: 3 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Dream 32.1 15.5
#> 2 Gentoo Biscoe 51.1 16.3
#> 3 Gentoo Biscoe 50.5 15.9
#> # ... with 4 more variables: flipper_length_mm <int>,
#> # body_mass_g <int>, sex <fct>, year <int>
sample_frac(penguins,0.01) # selección aleatoria de un porcentaje de datos
#> # A tibble: 3 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Gentoo Biscoe 50 15.9
#> 2 Gentoo Biscoe 42.6 13.7
#> 3 Adelie Dream 37.2 18.1
#> # ... with 4 more variables: flipper_length_mm <int>,
#> # body_mass_g <int>, sex <fct>, year <int>
2.3.2 Uso del operador binario %>% del paquete magrittr
Las capacidades de dplyr son potenciadas por algunas librerías como magrittr, que introduce al ambiente de R el operador pipe o también llamado operador de tuberías %>% (ctrl+shift+m), el cual una se leería en voz alta como un “y luego” que permite enlazar las diferentes acciones a realizar. Permite realizar operaciones complejas en bases de datos siguiendo una lógica simple y fluida. A continuación una comparación:
La manera tradicional en R sería así:
summary(subset(penguins,bill_length_mm >=40))
#> species island bill_length_mm
#> Adelie : 51 Biscoe :139 Min. :40.10
#> Chinstrap: 68 Dream : 85 1st Qu.:43.33
#> Gentoo :123 Torgersen: 18 Median :46.50
#> Mean :46.66
#> 3rd Qu.:49.77
#> Max. :59.60
#> bill_depth_mm flipper_length_mm body_mass_g
#> Min. :13.10 Min. :176.0 Min. :2700
#> 1st Qu.:15.00 1st Qu.:195.0 1st Qu.:3812
#> Median :16.60 Median :208.0 Median :4400
#> Mean :16.75 Mean :206.1 Mean :4468
#> 3rd Qu.:18.57 3rd Qu.:216.0 3rd Qu.:5038
#> Max. :21.50 Max. :231.0 Max. :6300
#> sex year
#> female: 99 Min. :2007
#> male :138 1st Qu.:2007
#> NA's : 5 Median :2008
#> Mean :2008
#> 3rd Qu.:2009
#> Max. :2009En dplyr con los %>% sería de esta manera:
penguins %>%
subset(bill_length_mm >=40) %>%
summary()
#> species island bill_length_mm
#> Adelie : 51 Biscoe :139 Min. :40.10
#> Chinstrap: 68 Dream : 85 1st Qu.:43.33
#> Gentoo :123 Torgersen: 18 Median :46.50
#> Mean :46.66
#> 3rd Qu.:49.77
#> Max. :59.60
#> bill_depth_mm flipper_length_mm body_mass_g
#> Min. :13.10 Min. :176.0 Min. :2700
#> 1st Qu.:15.00 1st Qu.:195.0 1st Qu.:3812
#> Median :16.60 Median :208.0 Median :4400
#> Mean :16.75 Mean :206.1 Mean :4468
#> 3rd Qu.:18.57 3rd Qu.:216.0 3rd Qu.:5038
#> Max. :21.50 Max. :231.0 Max. :6300
#> sex year
#> female: 99 Min. :2007
#> male :138 1st Qu.:2007
#> NA's : 5 Median :2008
#> Mean :2008
#> 3rd Qu.:2009
#> Max. :2009
2.3.3 Uso de la función select()
Se utiliza la función select() para seleccionar columnas. La información está bien ordenada las columnas son equivalentes de las variables.
penguins %>% select(species,bill_length_mm,body_mass_g) #selecciona por nombre de columna
#> # A tibble: 344 x 3
#> species bill_length_mm body_mass_g
#> <fct> <dbl> <int>
#> 1 Adelie 39.1 3750
#> 2 Adelie 39.5 3800
#> 3 Adelie 40.3 3250
#> 4 Adelie NA NA
#> 5 Adelie 36.7 3450
#> 6 Adelie 39.3 3650
#> 7 Adelie 38.9 3625
#> 8 Adelie 39.2 4675
#> 9 Adelie 34.1 3475
#> 10 Adelie 42 4250
#> # ... with 334 more rows
penguins %>% select(c(1,3,6)) # selecciona por número de columna
#> # A tibble: 344 x 3
#> species bill_length_mm body_mass_g
#> <fct> <dbl> <int>
#> 1 Adelie 39.1 3750
#> 2 Adelie 39.5 3800
#> 3 Adelie 40.3 3250
#> 4 Adelie NA NA
#> 5 Adelie 36.7 3450
#> 6 Adelie 39.3 3650
#> 7 Adelie 38.9 3625
#> 8 Adelie 39.2 4675
#> 9 Adelie 34.1 3475
#> 10 Adelie 42 4250
#> # ... with 334 more rows
penguins %>% select(-c(island,sex,year)) # remueve por nombre de columna
#> # A tibble: 344 x 5
#> species bill_length_mm bill_depth_mm flipper_length_mm
#> <fct> <dbl> <dbl> <int>
#> 1 Adelie 39.1 18.7 181
#> 2 Adelie 39.5 17.4 186
#> 3 Adelie 40.3 18 195
#> 4 Adelie NA NA NA
#> 5 Adelie 36.7 19.3 193
#> 6 Adelie 39.3 20.6 190
#> 7 Adelie 38.9 17.8 181
#> 8 Adelie 39.2 19.6 195
#> 9 Adelie 34.1 18.1 193
#> 10 Adelie 42 20.2 190
#> # ... with 334 more rows, and 1 more variable:
#> # body_mass_g <int>
penguins %>% select(-c(2,7:8)) # remueve por número de columna
#> # A tibble: 344 x 5
#> species bill_length_mm bill_depth_mm flipper_length_mm
#> <fct> <dbl> <dbl> <int>
#> 1 Adelie 39.1 18.7 181
#> 2 Adelie 39.5 17.4 186
#> 3 Adelie 40.3 18 195
#> 4 Adelie NA NA NA
#> 5 Adelie 36.7 19.3 193
#> 6 Adelie 39.3 20.6 190
#> 7 Adelie 38.9 17.8 181
#> 8 Adelie 39.2 19.6 195
#> 9 Adelie 34.1 18.1 193
#> 10 Adelie 42 20.2 190
#> # ... with 334 more rows, and 1 more variable:
#> # body_mass_g <int>Otras funciones adicionales para seleccionar columnas según características de los nombres de las variables
penguins %>% select(contains("bill")) # selecciona columnas que contienen "bill"
#> # A tibble: 344 x 2
#> bill_length_mm bill_depth_mm
#> <dbl> <dbl>
#> 1 39.1 18.7
#> 2 39.5 17.4
#> 3 40.3 18
#> 4 NA NA
#> 5 36.7 19.3
#> 6 39.3 20.6
#> 7 38.9 17.8
#> 8 39.2 19.6
#> 9 34.1 18.1
#> 10 42 20.2
#> # ... with 334 more rows
penguins %>% select(ends_with("mm")) # selecciona columnas que terminan con "bill"
#> # A tibble: 344 x 3
#> bill_length_mm bill_depth_mm flipper_length_mm
#> <dbl> <dbl> <int>
#> 1 39.1 18.7 181
#> 2 39.5 17.4 186
#> 3 40.3 18 195
#> 4 NA NA NA
#> 5 36.7 19.3 193
#> 6 39.3 20.6 190
#> 7 38.9 17.8 181
#> 8 39.2 19.6 195
#> 9 34.1 18.1 193
#> 10 42 20.2 190
#> # ... with 334 more rows
penguins %>% select(species, starts_with("flipper")) # se puede seleccionar dentro de alguna variable
#> # A tibble: 344 x 2
#> species flipper_length_mm
#> <fct> <int>
#> 1 Adelie 181
#> 2 Adelie 186
#> 3 Adelie 195
#> 4 Adelie NA
#> 5 Adelie 193
#> 6 Adelie 190
#> 7 Adelie 181
#> 8 Adelie 195
#> 9 Adelie 193
#> 10 Adelie 190
#> # ... with 334 more rowsSe pueden seleccionar y a la vez asignar un nuevo orden a las columnas
penguins %>% select(year,species,body_mass_g,species)
#> # A tibble: 344 x 3
#> year species body_mass_g
#> <int> <fct> <int>
#> 1 2007 Adelie 3750
#> 2 2007 Adelie 3800
#> 3 2007 Adelie 3250
#> 4 2007 Adelie NA
#> 5 2007 Adelie 3450
#> 6 2007 Adelie 3650
#> 7 2007 Adelie 3625
#> 8 2007 Adelie 4675
#> 9 2007 Adelie 3475
#> 10 2007 Adelie 4250
#> # ... with 334 more rowsSe puede usar la función wherepara distinguir por atributos de las columnas
penguins %>% select(where(is.factor), body_mass_g)
#> # A tibble: 344 x 4
#> species island sex body_mass_g
#> <fct> <fct> <fct> <int>
#> 1 Adelie Torgersen male 3750
#> 2 Adelie Torgersen female 3800
#> 3 Adelie Torgersen female 3250
#> 4 Adelie Torgersen <NA> NA
#> 5 Adelie Torgersen female 3450
#> 6 Adelie Torgersen male 3650
#> 7 Adelie Torgersen female 3625
#> 8 Adelie Torgersen male 4675
#> 9 Adelie Torgersen <NA> 3475
#> 10 Adelie Torgersen <NA> 4250
#> # ... with 334 more rows2.3.4 Otras funciones para el manejo de columnas
La función rename() se utiliza para cambiar de nombre a las columnas
penguins %>% rename(isla = island)
#> # A tibble: 344 x 8
#> species isla bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen NA NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Se puede cambiar la localización de las columnas con la función relocate() y los parámetros .before y .after
penguins %>% relocate(year, .after = species)
#> # A tibble: 344 x 8
#> species year island bill_length_mm bill_depth_mm
#> <fct> <int> <fct> <dbl> <dbl>
#> 1 Adelie 2007 Torgersen 39.1 18.7
#> 2 Adelie 2007 Torgersen 39.5 17.4
#> 3 Adelie 2007 Torgersen 40.3 18
#> 4 Adelie 2007 Torgersen NA NA
#> 5 Adelie 2007 Torgersen 36.7 19.3
#> 6 Adelie 2007 Torgersen 39.3 20.6
#> 7 Adelie 2007 Torgersen 38.9 17.8
#> 8 Adelie 2007 Torgersen 39.2 19.6
#> 9 Adelie 2007 Torgersen 34.1 18.1
#> 10 Adelie 2007 Torgersen 42 20.2
#> # ... with 334 more rows, and 3 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>La función distinct()se utiliza para ver los factores dentro de una columna
Mientras que la función n_distinct()se utiliza para contar el número de factores diferentes dentro de una columna. Favor notar que en el siguiente ejemplo se utiliza la función pull()para escoger la columna a analizar.
penguins %>% pull(year) %>% n_distinct()
#> [1] 3
2.3.5 Uso de la función filter()
La función filter()se utiliza para seleccionar filas que cumplen con una determinada condición o criterio lógico. Notar que esta función permite el uso de dos tipos de sintaxis, con y sin el operador binario %>%.
library(palmerpenguins)
filter(penguins,species== "Adelie")
#> # A tibble: 152 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen NA NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> # ... with 142 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>
penguins %>% filter(bill_length_mm >= 50)
#> # A tibble: 57 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Gentoo Biscoe 50 16.3
#> 2 Gentoo Biscoe 50 15.2
#> 3 Gentoo Biscoe 50.2 14.3
#> 4 Gentoo Biscoe 50 15.3
#> 5 Gentoo Biscoe 59.6 17
#> 6 Gentoo Biscoe 50.5 15.9
#> 7 Gentoo Biscoe 50.5 15.9
#> 8 Gentoo Biscoe 50.1 15
#> 9 Gentoo Biscoe 50.4 15.3
#> 10 Gentoo Biscoe 54.3 15.7
#> # ... with 47 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Un aspecto importante de la función filter() es que permite el uso de declaraciones lógicas y booleanas. Por ejemplo, se puede usar el operador & (del inglés AND) y el operador | (del inglés OR).
penguins %>% filter(bill_length_mm >= 50 & island == "Dream")
#> # A tibble: 31 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Chinstrap Dream 50 19.5
#> 2 Chinstrap Dream 51.3 19.2
#> 3 Chinstrap Dream 52.7 19.8
#> 4 Chinstrap Dream 51.3 18.2
#> 5 Chinstrap Dream 51.3 19.9
#> 6 Chinstrap Dream 51.7 20.3
#> 7 Chinstrap Dream 52 18.1
#> 8 Chinstrap Dream 50.5 19.6
#> 9 Chinstrap Dream 50.3 20
#> 10 Chinstrap Dream 58 17.8
#> # ... with 21 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>El operador binario %in% permite el filtrado con múltiples criterios, es decir, se pueden seleccionar diferentes items dentro de una columna o variable.
penguins %>% filter(island %in% c("Torgersen","Dream"))
#> # A tibble: 176 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen NA NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> # ... with 166 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Notar que se escribe el nombre de la columna a analizar, el operador y luego el conjunto de items seleccionados. También se puede incluir otros operadores lógicos.
penguins %>% filter(island %in% c("Torgensen","Dream") & body_mass_g >= 3900)
#> # A tibble: 43 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Dream 37.2 18.1
#> 2 Adelie Dream 40.9 18.9
#> 3 Adelie Dream 39.2 21.1
#> 4 Adelie Dream 38.8 20
#> 5 Adelie Dream 39.8 19.1
#> 6 Adelie Dream 40.8 18.4
#> 7 Adelie Dream 44.1 19.7
#> 8 Adelie Dream 39.6 18.8
#> 9 Adelie Dream 42.3 21.2
#> 10 Adelie Dream 38.3 19.2
#> # ... with 33 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>El parámetro near() permite escoger dentro de un rango de valores numéricos cercanos, estableciendo un valor de tolerancia.
penguins %>% filter(near(x = body_mass_g, y = 3500, tol = 500))
#> # A tibble: 154 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen 36.7 19.3
#> 5 Adelie Torgersen 39.3 20.6
#> 6 Adelie Torgersen 38.9 17.8
#> 7 Adelie Torgersen 34.1 18.1
#> 8 Adelie Torgersen 37.8 17.1
#> 9 Adelie Torgersen 37.8 17.3
#> 10 Adelie Torgersen 41.1 17.6
#> # ... with 144 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>El parámetro between() permite escoger un rango de valores numéricos dentro de un límite específico.
penguins %>% filter(between(bill_length_mm, left = 43, right = 46))
#> # A tibble: 52 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 46 21.5
#> 2 Adelie Dream 44.1 19.7
#> 3 Adelie Torgersen 45.8 18.9
#> 4 Adelie Dream 43.2 18.5
#> 5 Adelie Biscoe 43.2 19
#> 6 Adelie Biscoe 45.6 20.3
#> 7 Adelie Torgersen 44.1 18
#> 8 Adelie Torgersen 43.1 19.2
#> 9 Gentoo Biscoe 45.4 14.6
#> 10 Gentoo Biscoe 43.3 13.4
#> # ... with 42 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Ejercicio 1. Dentro de la base de datos penguins, filtrar las de la especie “Chinstrap”, que tengan una masa corporal menor a 3100 g, y un longitud de pico mayor o igual a 40. Además, seleccionar las variables que contengan “mm” y las que sean factores. ¿De qué isla provienen y qué sexo tienen?
2.3.6 Manejo de NAs
Los NAs son datos faltantes en celdas específicas. Se utiliza ese acrónimo para denominar datos Not Available en inglés. Hay distintas maneras de cómo eliminarlos o incluso reemplazarlos. La manera más simple, pero más grocera, es usar la función drop_na()que remueve toda la fila donde se encuentre al menos un NA.
library(tidyr)
penguins %>% drop_na()
#> # A tibble: 333 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen 36.7 19.3
#> 5 Adelie Torgersen 39.3 20.6
#> 6 Adelie Torgersen 38.9 17.8
#> 7 Adelie Torgersen 39.2 19.6
#> 8 Adelie Torgersen 41.1 17.6
#> 9 Adelie Torgersen 38.6 21.2
#> 10 Adelie Torgersen 34.6 21.1
#> # ... with 323 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Otra manera es remover los valores faltantes dentro de una columna
penguins %>% drop_na(bill_depth_mm)
#> # A tibble: 342 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen 36.7 19.3
#> 5 Adelie Torgersen 39.3 20.6
#> 6 Adelie Torgersen 38.9 17.8
#> 7 Adelie Torgersen 39.2 19.6
#> 8 Adelie Torgersen 34.1 18.1
#> 9 Adelie Torgersen 42 20.2
#> 10 Adelie Torgersen 37.8 17.1
#> # ... with 332 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Se pueden remplazar los NAs con la media de los valores de la columna
penguins %>%
replace_na(
list(bill_length_mm=mean(penguins$bill_length_mm,na.rm=TRUE)))
#> # A tibble: 344 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen 43.9 NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>
2.3.7 Uso de la función arrange()
Esta función permite ordenar de manera ascendente o descendente los elementos de una columna, sea alfabéticamente o numéricamente.
penguins %>% arrange(body_mass_g)
#> # A tibble: 344 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Chinstrap Dream 46.9 16.6
#> 2 Adelie Biscoe 36.5 16.6
#> 3 Adelie Biscoe 36.4 17.1
#> 4 Adelie Biscoe 34.5 18.1
#> 5 Adelie Dream 33.1 16.1
#> 6 Adelie Torgersen 38.6 17
#> 7 Chinstrap Dream 43.2 16.6
#> 8 Adelie Biscoe 37.9 18.6
#> 9 Adelie Dream 37.5 18.9
#> 10 Adelie Dream 37 16.9
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>
penguins %>% arrange(desc(body_mass_g))
#> # A tibble: 344 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Gentoo Biscoe 49.2 15.2
#> 2 Gentoo Biscoe 59.6 17
#> 3 Gentoo Biscoe 51.1 16.3
#> 4 Gentoo Biscoe 48.8 16.2
#> 5 Gentoo Biscoe 45.2 16.4
#> 6 Gentoo Biscoe 49.8 15.9
#> 7 Gentoo Biscoe 48.4 14.6
#> 8 Gentoo Biscoe 49.3 15.7
#> 9 Gentoo Biscoe 55.1 16
#> 10 Gentoo Biscoe 49.5 16.2
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Para escoger los individuos que presentaron los cinco valores más altos de una variables se podría hacer los siguiente. El parámetro wt se utiliza para definir la columna.
penguins %>%
arrange(desc(bill_length_mm)) %>%
top_n(n=5, wt=bill_length_mm)
#> # A tibble: 5 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Gentoo Biscoe 59.6 17
#> 2 Chinstrap Dream 58 17.8
#> 3 Gentoo Biscoe 55.9 17
#> 4 Chinstrap Dream 55.8 19.8
#> 5 Gentoo Biscoe 55.1 16
#> # ... with 4 more variables: flipper_length_mm <int>,
#> # body_mass_g <int>, sex <fct>, year <int>También se pueden realizar operaciones más complejas que involucran el uso de varios verbos de dplyr encadenados por el operador pipe %>%. Se puede, por ejemplo, seleccionar las columnas que contenga el texto “mm”, que sean numéricas y que tenga una media menor a 50 (removiendo los NAs), para luego ordenarlos de manera descendente según los valores de la variable bill_length_mm
penguins %>%
select(contains("mm")) %>%
select(where(is.numeric))%>%
select(where(~mean(., na.rm=TRUE) < 50))%>%
arrange(desc(bill_depth_mm))
#> # A tibble: 344 x 2
#> bill_length_mm bill_depth_mm
#> <dbl> <dbl>
#> 1 46 21.5
#> 2 38.6 21.2
#> 3 42.3 21.2
#> 4 34.6 21.1
#> 5 39.2 21.1
#> 6 41.3 21.1
#> 7 54.2 20.8
#> 8 42.5 20.7
#> 9 39.6 20.7
#> 10 52 20.7
#> # ... with 334 more rows
2.3.8 Uso de la función summarise()
Se utiliza esta función para resumir los datos mediante operaciones de cálculo básicas
penguins %>%
summarise(
avg_bill_length = mean(bill_length_mm, na.rm=TRUE),
avg_body_mass = mean(body_mass_g,na.rm=TRUE))
#> # A tibble: 1 x 2
#> avg_bill_length avg_body_mass
#> <dbl> <dbl>
#> 1 43.9 4202.Se puede utilizar la función across() dentro de summarise()para aplicar funciones a múltiples columnas. Notar que se usa el parámetro .cols para seleccionar columnas y el parámetro .fns para asignar la función a realizar.
penguins %>%
summarise(
across(.cols = bill_length_mm:body_mass_g,
.fns = ~mean(., na.rm = TRUE)))
#> # A tibble: 1 x 4
#> bill_length_mm bill_depth_mm flipper_length_mm body_mass_g
#> <dbl> <dbl> <dbl> <dbl>
#> 1 43.9 17.2 201. 4202.En la gramática de dplyr el uso del punto antes del parámetro permite diferenciarlo respecto a lo que podría ser una variable con el mismo nombre. Además, es importante señalar que la sintaxis “~mean(.)” se utiliza para especificar el cálculo de la media es para cada una de las variables seleccionadas. Por supuesto, también se pueden incluir diferentes funciones a la vez.
penguins %>%
drop_na() %>%
summarise(
across(.cols = bill_length_mm:body_mass_g,
.fns = c(mean,sum)))
#> # A tibble: 1 x 8
#> bill_length_mm_1 bill_length_mm_2 bill_depth_mm_1
#> <dbl> <dbl> <dbl>
#> 1 44.0 14650. 17.2
#> # ... with 5 more variables: bill_depth_mm_2 <dbl>,
#> # flipper_length_mm_1 <dbl>, flipper_length_mm_2 <int>,
#> # body_mass_g_1 <dbl>, body_mass_g_2 <int>Incluso, se le puede asignar un nuevo nombre a las columnas generadas
penguins %>%
drop_na() %>%
summarise(
across(.cols = c(where(is.numeric),-year),
.fns = list(avg=mean,sd=sd),
.names = "{.col}.{.fn}"))
#> # A tibble: 1 x 8
#> bill_length_mm.avg bill_length_mm.sd bill_depth_mm.avg
#> <dbl> <dbl> <dbl>
#> 1 44.0 5.47 17.2
#> # ... with 5 more variables: bill_depth_mm.sd <dbl>,
#> # flipper_length_mm.avg <dbl>,
#> # flipper_length_mm.sd <dbl>, body_mass_g.avg <dbl>,
#> # body_mass_g.sd <dbl>También se pueden realizar operaciones más complejas con funciones personalizadas
2.3.9 Uso de la función group_by()
Una de las funciones más útilizadas en dplyr. Permite agrupar diferentes variables categóricas, a partir de las cuales se pueden realizar diferentes operaciones de cálculo
penguins %>%
group_by(species) %>%
summarise(avg_mass = mean(body_mass_g, na.rm=TRUE)) %>%
arrange(desc(avg_mass))
#> # A tibble: 3 x 2
#> species avg_mass
#> <fct> <dbl>
#> 1 Gentoo 5076.
#> 2 Chinstrap 3733.
#> 3 Adelie 3701.Se pueden realizar agrupaciones y cálculos para diferentes variables
penguins %>%
group_by(species) %>%
summarise(
across(
.cols = c(where(is.numeric),-year),
.fns = ~mean(., na.rm = TRUE)))
#> # A tibble: 3 x 5
#> species bill_length_mm bill_depth_mm flipper_length_mm
#> <fct> <dbl> <dbl> <dbl>
#> 1 Adelie 38.8 18.3 190.
#> 2 Chinstrap 48.8 18.4 196.
#> 3 Gentoo 47.5 15.0 217.
#> # ... with 1 more variable: body_mass_g <dbl>Se pueden realizar agrupaciones con dos o más variables categóricas
penguins %>%
group_by(species,island) %>%
summarise(avg_flipper = mean(flipper_length_mm,na.rm=TRUE)) %>%
arrange(desc(avg_flipper))
#> # A tibble: 5 x 3
#> # Groups: species [3]
#> species island avg_flipper
#> <fct> <fct> <dbl>
#> 1 Gentoo Biscoe 217.
#> 2 Chinstrap Dream 196.
#> 3 Adelie Torgersen 191.
#> 4 Adelie Dream 190.
#> 5 Adelie Biscoe 189.Con el parámetro n() se pueden contar los elementos dentro de las variables agrupadas
penguins %>%
group_by(species,island) %>%
summarise(n())
#> # A tibble: 5 x 3
#> # Groups: species [3]
#> species island `n()`
#> <fct> <fct> <int>
#> 1 Adelie Biscoe 44
#> 2 Adelie Dream 56
#> 3 Adelie Torgersen 52
#> 4 Chinstrap Dream 68
#> 5 Gentoo Biscoe 124Otra forma rápida de realizar conteos es con la función count() donde se suprime el uso las funciones group_by()y summarise(). La desventaja es que solamente aplica para una sola variable.
penguins %>% count(species, sort = TRUE)
#> # A tibble: 3 x 2
#> species n
#> <fct> <int>
#> 1 Adelie 152
#> 2 Gentoo 124
#> 3 Chinstrap 68También se puede hacer construcción de modelos lineales automatizada. En la lógica gramatical de Tidyverse se utilizan verbos para ejecutar las diferentes acciones. Esto incluye las funciones auxiliares del propio dplyr, de R-base o de otros paquetes. Incluso, si no existe una función apropiada, se puede utilizar la función do().
penguins %>%
group_by(species) %>%
do(mod = lm(body_mass_g ~ bill_length_mm, data = .)) %>%
summarise(rsq = summary(mod)$r.squared)
#> # A tibble: 3 x 1
#> rsq
#> <dbl>
#> 1 0.301
#> 2 0.264
#> 3 0.448Ejercicio 2. De la base de datos iris, calcule la media y la desviación estándar por especie, pero solamente de las variables que contengan la palabra “Length”. Asegúrese que los nombres de las columnas especifiquen el tipo de valor calculado.
2.3.10 Uso de la función mutate()
Permite crear nuevas variables y realizar operaciones matemáticas entre ellas.
penguins %>% mutate(mass_kg = body_mass_g / 1000, .before=bill_length_mm)
#> # A tibble: 344 x 9
#> species island mass_kg bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl> <dbl>
#> 1 Adelie Torgersen 3.75 39.1 18.7
#> 2 Adelie Torgersen 3.8 39.5 17.4
#> 3 Adelie Torgersen 3.25 40.3 18
#> 4 Adelie Torgersen NA NA NA
#> 5 Adelie Torgersen 3.45 36.7 19.3
#> 6 Adelie Torgersen 3.65 39.3 20.6
#> 7 Adelie Torgersen 3.62 38.9 17.8
#> 8 Adelie Torgersen 4.68 39.2 19.6
#> 9 Adelie Torgersen 3.48 34.1 18.1
#> 10 Adelie Torgersen 4.25 42 20.2
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Se utiliza la función transmute()cuando se quiere generar y mostrar solamente la columna resultante de una operación matemática entre variables
penguins %>%
transmute(bill_ratio = bill_length_mm/bill_depth_mm)
#> # A tibble: 344 x 1
#> bill_ratio
#> <dbl>
#> 1 2.09
#> 2 2.27
#> 3 2.24
#> 4 NA
#> 5 1.90
#> 6 1.91
#> 7 2.19
#> 8 2
#> 9 1.88
#> 10 2.08
#> # ... with 334 more rowsCon el parámetro if_else() se pueden definir categorías arbitrarias de clasificación dentro de variables numéricas. Esta función literalmente se leería como “si cumple con la siguiente característica lo clasifica como tal cosa, sino, como tal otra cosa”.
library(tidyr)
penguins %>%
drop_na() %>%
mutate(size = if_else(body_mass_g>4000,"BIG","SMALL")) %>%
count(size)
#> # A tibble: 2 x 2
#> size n
#> <chr> <int>
#> 1 BIG 167
#> 2 SMALL 166Para concatenar dos variables, asignarles nuevo nombre y posición, se puede hacer los siguiente
sp_si =
penguins %>%
mutate(
sp_sitio=paste(
species,
island,
sep="_"),
.before =bill_length_mm)
sp_si
#> # A tibble: 344 x 9
#> species island sp_sitio bill_length_mm bill_depth_mm
#> <fct> <fct> <chr> <dbl> <dbl>
#> 1 Adelie Torgersen Adelie_To~ 39.1 18.7
#> 2 Adelie Torgersen Adelie_To~ 39.5 17.4
#> 3 Adelie Torgersen Adelie_To~ 40.3 18
#> 4 Adelie Torgersen Adelie_To~ NA NA
#> 5 Adelie Torgersen Adelie_To~ 36.7 19.3
#> 6 Adelie Torgersen Adelie_To~ 39.3 20.6
#> 7 Adelie Torgersen Adelie_To~ 38.9 17.8
#> 8 Adelie Torgersen Adelie_To~ 39.2 19.6
#> 9 Adelie Torgersen Adelie_To~ 34.1 18.1
#> 10 Adelie Torgersen Adelie_To~ 42 20.2
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>La función separate()se puede utilizar para separar elementos dentro de una variable categórica
sp_si %>%
separate(col = sp_sitio,
into= c("especie", "isla"),
sep="_")
#> # A tibble: 344 x 10
#> species island especie isla bill_length_mm bill_depth_mm
#> <fct> <fct> <chr> <chr> <dbl> <dbl>
#> 1 Adelie Torge~ Adelie Torg~ 39.1 18.7
#> 2 Adelie Torge~ Adelie Torg~ 39.5 17.4
#> 3 Adelie Torge~ Adelie Torg~ 40.3 18
#> 4 Adelie Torge~ Adelie Torg~ NA NA
#> 5 Adelie Torge~ Adelie Torg~ 36.7 19.3
#> 6 Adelie Torge~ Adelie Torg~ 39.3 20.6
#> 7 Adelie Torge~ Adelie Torg~ 38.9 17.8
#> 8 Adelie Torge~ Adelie Torg~ 39.2 19.6
#> 9 Adelie Torge~ Adelie Torg~ 34.1 18.1
#> 10 Adelie Torge~ Adelie Torg~ 42 20.2
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>2.3.11 Fusión de bases de datos
Dentro del paquete dplyr existen numerosas funciones para unir bases de datos. Una de las funciones utilizadas para unir multiples bases de datos por filas para obtener una sola ordenándolas es con bind_rows(). Para el ejemplo dividiremos la base de datos penguins en dos imaginando que serían dos bases de datos distintas.
mitad1 <- penguins[1:172, ]
mitad2 <- penguins[173:344, ]
bind_rows(mitad1, mitad2)
#> # A tibble: 344 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen NA NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>También se puede usar una lista enumerando las bases de datos a unir. De hecho, ese sería el proceso más recomendado para unir más de los bases de datos.
bind_rows(list(mitad1, mitad2))
#> # A tibble: 344 x 8
#> species island bill_length_mm bill_depth_mm
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen NA NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> # ... with 334 more rows, and 4 more variables:
#> # flipper_length_mm <int>, body_mass_g <int>, sex <fct>,
#> # year <int>Para unir columnas se utiliza la función bind_cols(). Una salvedad importante es las bases de datos tienen que tener el mismo número de filas para ser fusionadas.
bind_cols(list(mitad1, mitad2))
#> # A tibble: 172 x 16
#> species...1 island...2 bill_length_mm..~ bill_depth_mm..~
#> <fct> <fct> <dbl> <dbl>
#> 1 Adelie Torgersen 39.1 18.7
#> 2 Adelie Torgersen 39.5 17.4
#> 3 Adelie Torgersen 40.3 18
#> 4 Adelie Torgersen NA NA
#> 5 Adelie Torgersen 36.7 19.3
#> 6 Adelie Torgersen 39.3 20.6
#> 7 Adelie Torgersen 38.9 17.8
#> 8 Adelie Torgersen 39.2 19.6
#> 9 Adelie Torgersen 34.1 18.1
#> 10 Adelie Torgersen 42 20.2
#> # ... with 162 more rows, and 12 more variables:
#> # flipper_length_mm...5 <int>, body_mass_g...6 <int>,
#> # sex...7 <fct>, year...8 <int>, species...9 <fct>,
#> # island...10 <fct>, bill_length_mm...11 <dbl>,
#> # bill_depth_mm...12 <dbl>, flipper_length_mm...13 <int>,
#> # body_mass_g...14 <int>, sex...15 <fct>, year...16 <int>Dentro de dplyr hay varias funciones asociadas al verbo join.
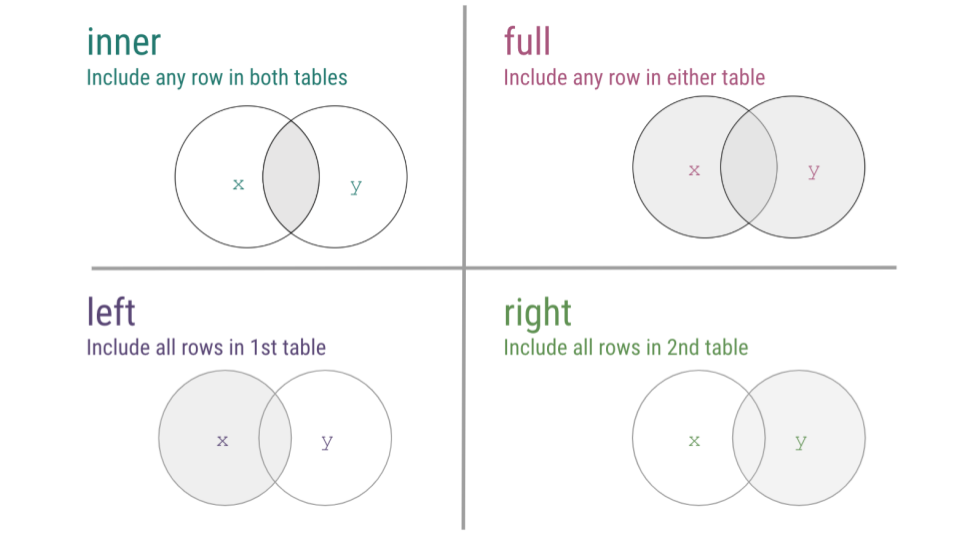
La función inner_join() crea una nueva tabla a partir de coincidencias.
df1 = data.frame(ID = c(1, 2, 3, 4, 5),
w = c('a', 'b', 'c', 'd', 'e'),
z=letters[1:5])
df2 = data.frame(ID = c(1, 7, 3, 6, 8),
a = c('z', 'b', 'k', 'd', 'l'),
d =letters[2:6])
inner_join(df1, df2, by = "ID")
#> ID w z a d
#> 1 1 a a z b
#> 2 3 c c k dLa función left_join() devuelve todas las filas de la tabla izquierda, incluso si no hay coincidencias. Imputa NA cuando hay información faltante
left_join(df1, df2, by = "ID")
#> ID w z a d
#> 1 1 a a z b
#> 2 2 b b <NA> <NA>
#> 3 3 c c k d
#> 4 4 d d <NA> <NA>
#> 5 5 e e <NA> <NA>Se puede combinar verticalmente usando intersect()donde se selecciona filas únicas que son comunes a ambas tablas
mtcars$model <- rownames(mtcars)
first <- mtcars[1:20, ]
second <- mtcars[10:32, ]
intersect(first, second)
#> mpg cyl disp hp drat wt qsec vs
#> Merc 280 19.2 6 167.6 123 3.92 3.440 18.30 1
#> Merc 280C 17.8 6 167.6 123 3.92 3.440 18.90 1
#> Merc 450SE 16.4 8 275.8 180 3.07 4.070 17.40 0
#> Merc 450SL 17.3 8 275.8 180 3.07 3.730 17.60 0
#> Merc 450SLC 15.2 8 275.8 180 3.07 3.780 18.00 0
#> Cadillac Fleetwood 10.4 8 472.0 205 2.93 5.250 17.98 0
#> Lincoln Continental 10.4 8 460.0 215 3.00 5.424 17.82 0
#> Chrysler Imperial 14.7 8 440.0 230 3.23 5.345 17.42 0
#> Fiat 128 32.4 4 78.7 66 4.08 2.200 19.47 1
#> Honda Civic 30.4 4 75.7 52 4.93 1.615 18.52 1
#> Toyota Corolla 33.9 4 71.1 65 4.22 1.835 19.90 1
#> am gear carb model
#> Merc 280 0 4 4 Merc 280
#> Merc 280C 0 4 4 Merc 280C
#> Merc 450SE 0 3 3 Merc 450SE
#> Merc 450SL 0 3 3 Merc 450SL
#> Merc 450SLC 0 3 3 Merc 450SLC
#> Cadillac Fleetwood 0 3 4 Cadillac Fleetwood
#> Lincoln Continental 0 3 4 Lincoln Continental
#> Chrysler Imperial 0 3 4 Chrysler Imperial
#> Fiat 128 1 4 1 Fiat 128
#> Honda Civic 1 4 2 Honda Civic
#> Toyota Corolla 1 4 1 Toyota Corolla2.4 Reordenamiento de datos con Tidyr
Los datos tidy generalmente existen en dos formas: 1)datos a lo ancho y 2)datos a lo largo. Ambos tipos de datos se usan y se necesitan en el análisis de datos y, afortunadamente, existen herramientas que pueden llevar de un formato a otro. Obtener los datos en el formato correcto será crucial más adelante al resumir los datos y visualizarlos.
Los datos a lo ancho tienen una columna para cada variable y una fila para cada observación. Los datos a menudo se ingresan y almacenan de esta manera. Los datos ordenados a lo largo, por otro lado, tienen una columna que indica el tipo de variable contenida en esa fila y luego, en una columna separada, el valor de esa variable. Cada fila contiene una sola observación para una sola variable. Sigue siendo un conjunto de datos ordenado, pero la información se almacena en un formato largo.
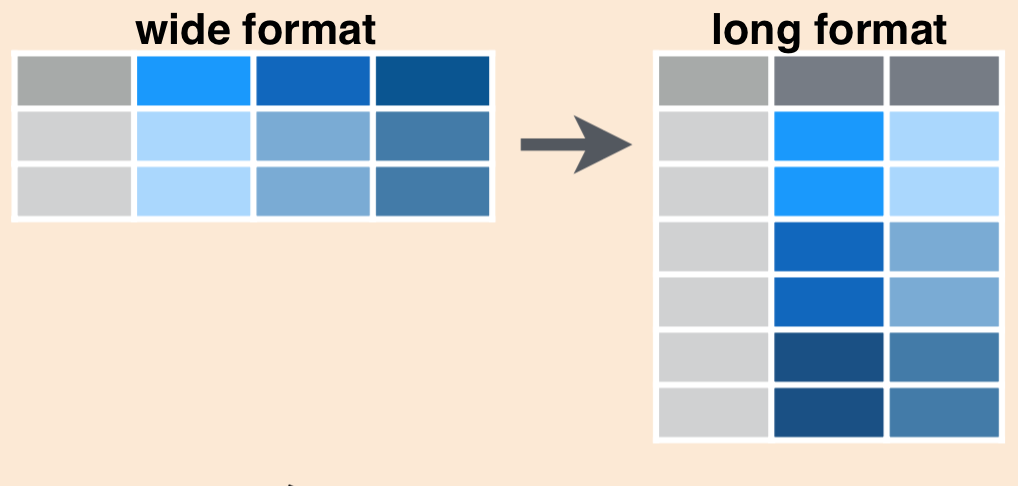
Imagen tomada de https://jules32.github.io/r-for-excel-users/tidying.html
Si bien los formatos de datos largos son menos legibles que los datos a lo ancho, a menudo es mucho más fácil trabajar con estos últimos durante el análisis. Por lo tanto, para pasar de cómo se almacenan los datos a menudo (a lo ancho) a trabajar con los datos durante el análisis (a lo largo), podemos utilizar algunas funciones del paquete tidyr
2.4.1 Uso de la función pivot_longer()
Como los datos a menudo se almacenan en formatos a lo ancho, es probable que la función pivot_longer() sea muy utilizada. Esto le permitirá obtener los datos en un formato a lo largo que será fácil de usar para análisis posteriores. De ejemplo usaremos la base de datos airquality sobre mediciones en la calidad del aire en Nueva York en 1973. Notar que se puede usar el parámetro names_to para asignar un nombre a la nueva columna generada y el parámetro values_to para la columna con los valores.
data("airquality")
head(airquality)
#> Ozone Solar.R Wind Temp Month Day
#> 1 41 190 7.4 67 5 1
#> 2 36 118 8.0 72 5 2
#> 3 12 149 12.6 74 5 3
#> 4 18 313 11.5 62 5 4
#> 5 NA NA 14.3 56 5 5
#> 6 28 NA 14.9 66 5 6
largo <- airquality %>%
pivot_longer(c(Ozone, Solar.R, Wind, Temp),
names_to = "variable", values_to = "value")
largo
#> # A tibble: 612 x 4
#> Month Day variable value
#> <int> <int> <chr> <dbl>
#> 1 5 1 Ozone 41
#> 2 5 1 Solar.R 190
#> 3 5 1 Wind 7.4
#> 4 5 1 Temp 67
#> 5 5 2 Ozone 36
#> 6 5 2 Solar.R 118
#> 7 5 2 Wind 8
#> 8 5 2 Temp 72
#> 9 5 3 Ozone 12
#> 10 5 3 Solar.R 149
#> # ... with 602 more rows
2.4.2 Uso de la función pivot_wider()
Para volver del formato largo a su forma original puede usar pivot_wider(). Se deben especificar dos columnas: la columna que contiene los nombres de lo que deberían ser sus columnas en formato ancho, usando el parámetro names_from y la columna que contiene los valores values_from.
ancho <- largo %>%
pivot_wider(names_from = "variable",
values_from = "value")
ancho
#> # A tibble: 153 x 6
#> Month Day Ozone Solar.R Wind Temp
#> <int> <int> <dbl> <dbl> <dbl> <dbl>
#> 1 5 1 41 190 7.4 67
#> 2 5 2 36 118 8 72
#> 3 5 3 12 149 12.6 74
#> 4 5 4 18 313 11.5 62
#> 5 5 5 NA NA 14.3 56
#> 6 5 6 28 NA 14.9 66
#> 7 5 7 23 299 8.6 65
#> 8 5 8 19 99 13.8 59
#> 9 5 9 8 19 20.1 61
#> 10 5 10 NA 194 8.6 69
#> # ... with 143 more rows2.4.3 Aplicaciones más elaboradas de las tablas dinámicas
En el siguiente ejemplo usaremos pivot_longer()para resumir y visualiar datos de vegetación en los prados de dunas en Holanda contenida en el paquete para manejo estadistico de datos ecológicos vegan. Este set de datos se compone de dos tablas, la primera contiene la abundancia de 30 especies en 20 sitios y la segunda contiene la información ambiental correspondiente a cada sitio (Jongman et al. 1987).
library(vegan)
library(tidyr)
data("dune","dune.env")
dune2= bind_cols(list(dune, dune.env))
tab.din <- dune2 %>%
pivot_longer(Achimill:Callcusp,
names_to = "especie", values_to = "abundancia")
tab.din
#> # A tibble: 600 x 7
#> A1 Moisture Management Use Manure especie abundancia
#> <dbl> <ord> <fct> <ord> <ord> <chr> <dbl>
#> 1 2.8 1 SF Hayp~ 4 Achimi~ 1
#> 2 2.8 1 SF Hayp~ 4 Agrost~ 0
#> 3 2.8 1 SF Hayp~ 4 Airapr~ 0
#> 4 2.8 1 SF Hayp~ 4 Alopge~ 0
#> 5 2.8 1 SF Hayp~ 4 Anthod~ 0
#> 6 2.8 1 SF Hayp~ 4 Bellpe~ 0
#> 7 2.8 1 SF Hayp~ 4 Bromho~ 0
#> 8 2.8 1 SF Hayp~ 4 Chenal~ 0
#> 9 2.8 1 SF Hayp~ 4 Cirsar~ 0
#> 10 2.8 1 SF Hayp~ 4 Comapa~ 0
#> # ... with 590 more rowsLuego se puede visualizar de la siguiente manera
library(ggplot2)
ggplot(data = tab.din,aes(x = especie,y = abundancia)) +
geom_point(size=0.5) + xlab(label = "especie") +
facet_grid(Management~ ., scales = "free_x", space = "free_x")+
theme_bw() +
theme(axis.text.x = element_text(size=7,angle = 90,vjust = 0.5, hjust=1),
axis.text.y = element_text(size = 6),
axis.title.y = element_text(size = 8),
axis.title.x = element_blank())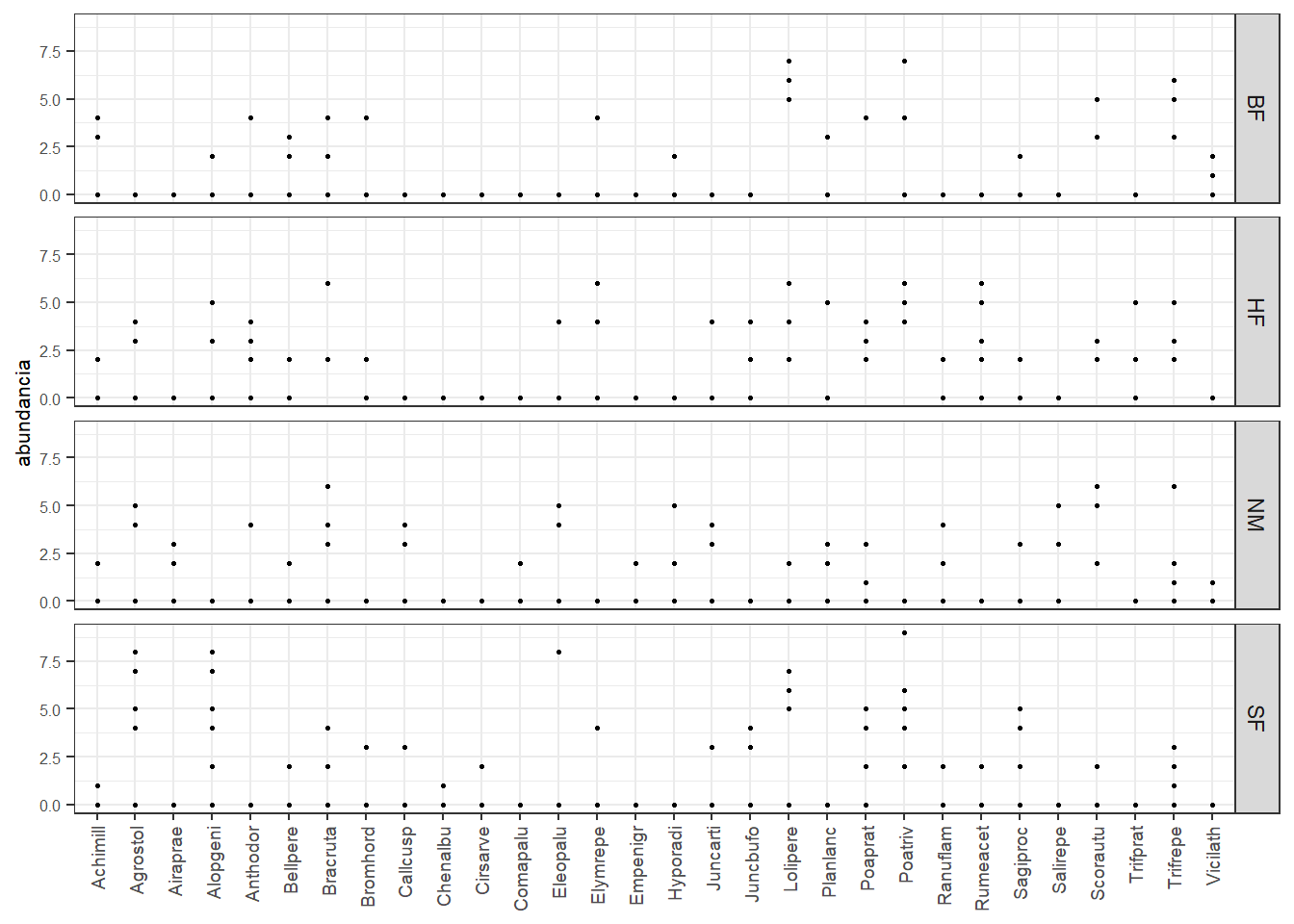
Figure 2.1: 2
2.5 Programación funcional con el paquete purr
La programación funcional es un enfoque de programación donde el código evaluado se trata como una función matemática. A menudo se promociona y utiliza debido al hecho de que se puede escribir un código más limpio, más corto y con menos redundancias. En última instancia, el objetivo es tener un código más simple que minimice el tiempo necesario para la depuración, prueba y mantenimiento. En el enfoque básico de R, se utilizaban loops (bucles) para realizar iteraciones, por ejemplo, de una operación en cada columna de un data.frame. Este enfoque ha sido criticado por ser lento e innecesario.
2.5.1 Enfoque clásico de loops
En el siguiente ejemplo se puede ver la utilización de loops para calcular la mediana en la base de datos iris.
library(dplyr)
data(iris)
iris2= select(iris,1:4)
output <- vector("double", ncol(iris2))
for (i in seq_along(iris2)) {
output[[i]] <- median(iris2[[i]])
}
output
#> [1] 5.80 3.00 4.35 1.30Otra alternativa es crear una función. La ventaja de crear funciones es que esta se puede utilizar para cualquier otro data.frame, es decir, permite generalizar el código.
2.5.2 El enfoque de purrr
purrr mejora el conjunto de herramientas de programación funcional de R al proporcionar un conjunto completo y consistente de herramientas para trabajar con funciones y vectores. Por ejemplo, la familia de funciones map() permiten reemplazar muchos “for loops” con código que es más breve y más fácil de leer.
En general, la función map()permite llevar a cabo la operación de interés para una sola ocurrencia (p.e. calcular la mediana para una sola columna). Luego, realiza esa operación en todo el data.frame, por lo que a esta función también se le denomina como un “iterador”.
Otras funciones más específicas de la familia map() son las siguientes:
-
map()- genera una lista -
map_lgl()- genera vector lógico -
map_int()- genera vector de enteros -
map_dbl()- genera vector de doubles -
map_chr()- genera vector de caracteres -
map_dfr()- genera un data frame -
map2()- iteración sobre dos conjuntos de datos -
pmap()- itera sobre una lista de argumentos
El uso genérico de la familia de funciones map()es el siguiente:
map(.x, .f, ...)
map(INPUT, FUNCTION_TO_APPLY, OPTIONAL_OTHER_STUFF)Para el caso en particular que estabamos analizando, usamos la función map_dbl() para iterar sobre las columnas y calcular la mediana. Notar que incluso se muestra el nombre de la variable en la salida.
library(purrr)
map_dbl(iris2, median)
#> Sepal.Length Sepal.Width Petal.Length Petal.Width
#> 5.80 3.00 4.35 1.30Además de la función principal, se pueden pasar argumentos adicionales. Estos van después de la función principal especificada. Por ejemplo, a continuación, especificamos que nos gustaría eliminar los NA, especificando un argumento que se pasará como parte de la función mean()
2.5.3 Manejo de múltiples archivos
Hasta el momento hemos trabajado generalmente con datos en un solo archivo. Sin embargo, muchas veces hay que trabajar con archivos que están en múltiples carpetas.
En el siguiente ejemplo veremos cómo se puede separar un data.frame en múltiples archivos que se pueden guardar en un nuevo directorio. Luego volveremos a combinar estos múltiples archivos en un solo data.frame. Para esto estaremos utilizando los paquetes fs, purrr y readr y la base de datos iris.
library(dplyr)
library(readr)
library(fs)
library(purrr)
carpeta= dir_create("iris_sep/")
iris %>%
group_by(Species) %>%
group_walk(~ write.csv(.x, file = file.path(carpeta, paste0(.y$Species,".csv"))))
csv <- dir_ls(path = carpeta, regexp = "\\.csv$")
csv
#> iris_sep/setosa.csv iris_sep/versicolor.csv
#> iris_sep/virginica.csvCon la función dir_create() del paquete fs se crea un nuevo subdirectorio en la carpeta de trabajo. Con la función group_by() se divide iris en tres data.frames según la especie. Con la función group_walk() generamos una función que permite escribir cada data.frame como un nuevo archivo csv, al cual se le asigna el nombre de la especie. La función dir_ls() permite ver la lista de archivos en la subcarpeta que tengan extensión csv.
Ahora, volveremos a leer y combinar los tres archivos .csv con las funciones map_dfr() y read_csv(). Además se utiliza el argumento .id para rastrear la fuente de cada fila del data.frame generado.
csv %>%
map_dfr(read_csv, .id= "source")
#> New names:
#> Rows: 50 Columns: 5
#> -- Column specification
#> ------------------------------------ Delimiter: "," dbl
#> (5): ...1, Sepal.Length, Sepal.Width, Petal.Length, ...
#> i Use `spec()` to retrieve the full column specification
#> for this data. i Specify the column types or set
#> `show_col_types = FALSE` to quiet this message.
#> New names:
#> Rows: 50 Columns: 5
#> -- Column specification
#> ------------------------------------ Delimiter: "," dbl
#> (5): ...1, Sepal.Length, Sepal.Width, Petal.Length, ...
#> i Use `spec()` to retrieve the full column specification
#> for this data. i Specify the column types or set
#> `show_col_types = FALSE` to quiet this message.
#> New names:
#> Rows: 50 Columns: 5
#> -- Column specification
#> ------------------------------------ Delimiter: "," dbl
#> (5): ...1, Sepal.Length, Sepal.Width, Petal.Length, ...
#> i Use `spec()` to retrieve the full column specification
#> for this data. i Specify the column types or set
#> `show_col_types = FALSE` to quiet this message.
#> * `` -> `...1`
#> # A tibble: 150 x 6
#> source ...1 Sepal.Length Sepal.Width Petal.Length
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 iris_sep/set~ 1 5.1 3.5 1.4
#> 2 iris_sep/set~ 2 4.9 3 1.4
#> 3 iris_sep/set~ 3 4.7 3.2 1.3
#> 4 iris_sep/set~ 4 4.6 3.1 1.5
#> 5 iris_sep/set~ 5 5 3.6 1.4
#> 6 iris_sep/set~ 6 5.4 3.9 1.7
#> 7 iris_sep/set~ 7 4.6 3.4 1.4
#> 8 iris_sep/set~ 8 5 3.4 1.5
#> 9 iris_sep/set~ 9 4.4 2.9 1.4
#> 10 iris_sep/set~ 10 4.9 3.1 1.5
#> # ... with 140 more rows, and 1 more variable:
#> # Petal.Width <dbl>2.6 Recursos complementarios
- https://jhudatascience.org/tidyversecourse/get-data.html
- https://benwhalley.github.io/just-enough-r/index.html
- https://static-bcrf.biochem.wisc.edu/courses/Tabular-data-analysis-with-R-and-Tidyverse/book/
- https://r4ds.had.co.nz/index.html
- https://mdsr-book.github.io/mdsr2e/
- https://www.institutomora.edu.mx/testU/SitePages/martinpaladino/manipulacion_de_datos_con_r_dplyr_y_tidyr.html
- https://www.uv.es/pjperez/curso_R/index.html
- https://www.rstudio.com/resources/cheatsheets/