3.2 Importing data in readr
3.2.1 Introduction
readr 是 tidyverse 的核心 R 包之一,作用是将平面数据(flat files)快速读取至 R 中,并转换为易用的 tibble 格式。相对于 base R 中有的数据读取函数,它有以下的优点:
- 一般来说,它们比基础模块中的函数速度更快(约快 10 倍)。第 ?? 章中的
fread()和第 10 章中的vroom()提供了更快速的数据读取
- 它们可以生成
tibble,而且不会将字符串向量转换为因子,不使用行名称,也不会随意改动列名称,这些都是 Base R 读取的痛点 - 它们更容易重复使用。base R 中的函数会继承操作系统的功能,并依赖环境变量,因此,可以在你的计算机上正常运行的代码未必适用于他人的计算机
主要函数有:
read_csv()读取逗号分割文件、read_csv2()读取分号分隔文件(这在用逗号表示小数点的国家非常普遍),read_tsv()读取制表符分隔文件,read_delim()函数可以读取使用任意分隔符的文件(通过指定delim参数)
read_fwf()读取固定宽度的文件(fixed width file)。既可以使用fwf_width()函按照宽度来设定域,也可以使用fwf_positions()函数按照位置来设定域。read_table()读取固定宽度文件的一种常用变体,其中使用空白字符来分隔各列
重要参数:
delim指定分隔符
col_names = T:以上的函数默认将第一行用作列名,如果设定col_names = F则列名为X1、X2、···。也可以指定一个字符向量。
col_types用一个字符串指定各列的数据类型
| 字符 | 含义 |
|---|---|
| “i” | integer |
| “d” | double |
| “c” | character |
| “l” | logical |
| "_" | 舍弃该列 |
n_max最大读取行数
na表明文件中缺失值的表示方法
下面用一些例子演示read_delim()函数的用法,因为它是最一般的形式,一旦掌握它,我们就可将从中学到的经验轻松应用于readr的其他函数。
## column names
properties <- c("area", "temp", "size", "storage", "method",
"texture", "flavor", "moistness")
## 文件中用制表符为分隔符
potatoes <- read_delim("data\\potatoes.txt",delim = "\t",
col_names = properties)
potatoes
#> # A tibble: 160 x 8
#> area temp size storage method texture flavor moistness
#> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 1 1 1 1 1 2.9 3.2 3
#> 2 1 1 1 1 2 2.3 2.5 2.6
#> 3 1 1 1 1 3 2.5 2.8 2.8
#> 4 1 1 1 1 4 2.1 2.9 2.4
#> 5 1 1 1 1 5 1.9 2.8 2.2
#> 6 1 1 1 2 1 1.8 3 1.7
#> # ... with 154 more rows当运行readr中的数据导入函数时,会打印出一份数据了说明,给出每个列的名称和类型。后面我们学习解析时,还会继续讨论这项功能。
通过col_types指定前五列为integer类型:
properties <- c("area", "temp", "size", "storage", "method",
"texture", "flavor", "moistness")
potatoes <- read_delim("data\\potatoes.txt",delim = "\t",
col_names = properties,
col_types = "iiiiiddd")
potatoes
#> # A tibble: 160 x 8
#> area temp size storage method texture flavor moistness
#> <int> <int> <int> <int> <int> <dbl> <dbl> <dbl>
#> 1 1 1 1 1 1 2.9 3.2 3
#> 2 1 1 1 1 2 2.3 2.5 2.6
#> 3 1 1 1 1 3 2.5 2.8 2.8
#> 4 1 1 1 1 4 2.1 2.9 2.4
#> 5 1 1 1 1 5 1.9 2.8 2.2
#> 6 1 1 1 2 1 1.8 3 1.7
#> # ... with 154 more rows我们还可以创建一个行内 csv 文件。这种文件非常适合用readr进行实验,以及与他人分享可重现的例子:
read_delim("a, b, c
1, 2 , 3
4, 5, 6", delim = ",")
#> # A tibble: 2 x 3
#> a ` b` ` c`
#> <chr> <chr> <chr>
#> 1 " 1" " 2 " " 3"
#> 2 " 4" " 5" " 6"有时文件开头会有几行元数据。可以使用 skip = n 来跳过前 n 行;或者使用 comment = "#" 丢弃所有以 # 开头的行:
read_delim("The first line metadata
The second line of metadata
x,y,z
1,2,3",
delim = ",", skip = 2)
#> # A tibble: 1 x 3
#> ` x` y z
#> <chr> <dbl> <dbl>
#> 1 " 1" 2 3
read_delim("# A comment I want to skip
x,y,z
1,2,3",
delim = ",", comment = "#")
#> # A tibble: 1 x 3
#> ` x` y z
#> <chr> <dbl> <dbl>
#> 1 " 1" 2 3设置na:
3.2.2 Writing data
readr还提供了两个非常有用的函数,用于将数据写回到磁盘:write_csv() 和 write_tsv(),这两个函数输出的文件能够顺利读取的概率更高,因为:
- 它们总使用 UTF-8 对字符串进行编码
- 它们都是用 ISO 8601 日期格式来保存日期和日期时间数据
如果想要将 CSV 文件导为 Excel 文件,可以使用 write_excel_csv() 函数,该函数会在文件开头写入一个特殊字符(字节顺序标记),告诉 Excel 这个文件采用的是 UTF-8 编码。
这几个函数中最重要的参数是 x (要保存的数据框)和path(保存文件的位置)。还可以使用 na 参数设定如何写入缺失值。默认情况下,写入函数会创建一个新文件或清空原有的文件再导入数据(Python中open()函数的"mode = w"模式),如果想要追加到现有的文件,可以设置 append = T (Python中的mode = "a"模式):
打开对应的文件:
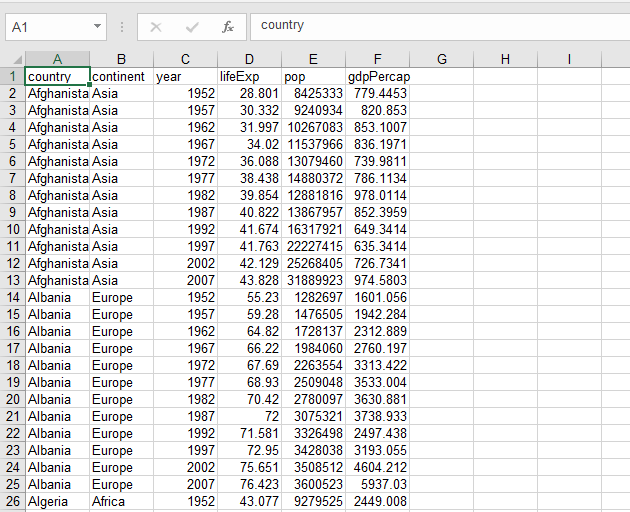
3.2.3 Exercises
应该使用read_delim(path, delim = "|")
read_fwf() 中最重要的参数是什么?
read_fwf()用于固定宽度文件(fixed width files)。在固定宽度文件中,每一列的的宽度是固定的(不足的用某种填充符号填充),如第一列总是10个字符长度,第二列 5 个字符长度,第三列8个字符长度,每列内采取统一的对齐方式。readr安装时附带了一个固定宽度文件的示例,我们用一个变量存储它的路径:
fwf_sample <- readr_example("fwf-sample.txt")
fwf_sample
#> [1] "C:/Users/Lenovo/Documents/R/win-library/3.6/readr/extdata/fwf-sample.txt"txt 文件内的内容:

读取固定宽度文件时,最重要的是告诉 R 每列的位置,参数 col_positions 用于这项工作,有几种不同的表示方式:
# You can specify column positions in several ways:
# 1. Guess based on position of empty columns
read_fwf(fwf_sample,
col_positions = fwf_empty(fwf_sample, col_names = c("first", "last", "state", "ssn")))
#> # A tibble: 3 x 4
#> first last state ssn
#> <chr> <chr> <chr> <chr>
#> 1 John Smith WA 418-Y11-4111
#> 2 Mary Hartford CA 319-Z19-4341
#> 3 Evan Nolan IL 219-532-c301
# 2. A vector of field widths
read_fwf(fwf_sample,
col_positions = fwf_widths(c(20, 10, 12), col_names = c("name", "state", "ssn")))
#> # A tibble: 3 x 3
#> name state ssn
#> <chr> <chr> <chr>
#> 1 John Smith WA 418-Y11-4111
#> 2 Mary Hartford CA 319-Z19-4341
#> 3 Evan Nolan IL 219-532-c301
# 3. Paired vectors of start and end positions
read_fwf(fwf_sample,
col_positions = fwf_positions(c(1, 30), c(20, 42), col_names = c("name", "ssn")))
#> # A tibble: 3 x 2
#> name ssn
#> <chr> <chr>
#> 1 John Smith 418-Y11-4111
#> 2 Mary Hartford 319-Z19-4341
#> 3 Evan Nolan 219-532-c301
# 4. Named arguments with start and end positions
read_fwf(fwf_sample,
col_positions = fwf_cols(name = c(1, 20), ssn = c(30, 42)))
#> # A tibble: 3 x 2
#> name ssn
#> <chr> <chr>
#> 1 John Smith 418-Y11-4111
#> 2 Mary Hartford 319-Z19-4341
#> 3 Evan Nolan 219-532-c301
# 5. Named arguments with column widths
read_fwf(fwf_sample,
col_positions = fwf_cols(name = 20, state = 10, ssn = 12))
#> # A tibble: 3 x 3
#> name state ssn
#> <chr> <chr> <chr>
#> 1 John Smith WA 418-Y11-4111
#> 2 Mary Hartford CA 319-Z19-4341
#> 3 Evan Nolan IL 219-532-c301