6 R Modulo 11
6.1 Organização básica
dev.off() #apaga os graficos, se houver algum
rm(list=ls(all=TRUE)) #limpa a memória
cat("\014") #limpa o consoleInstalando os pacotes necessários para esse módulo
install.packages("vegan3d")
install.packages("geometry")
install.packages("magick")Agora vamos definir o diretório de trabalho. Esse código é usado para obter e definir o diretório de trabalho atual no R. O comando getwd() retorna o caminho do diretório onde o R está lendo e salvando arquivos. O comando setwd() muda esse diretório de trabalho para o caminho especificado entre aspas. No seu caso, você deve ajustar o caminho para o seu próprio diretório de trabalho. Lembre de usar a barra “/” entre os diretórios. E não a contra-barra “\”.
Definindo o diretório de trabalho e installando os pacotes necessários:
getwd()
setwd("C:/Seu/Diretório/De/Trabalho")6.1.1 Sobre os dados do PPBio
A planilha ppbio contém os dados de abundância de espécies em diferentes unidades amostrais (UA’s). A base teórica dos dados do PPBio para o presente estudo pode ser vista em Base Teórica. Leia antes de prosseguir.
6.1.2 A planilha PPBio Grupos
Para esse módulo também usaremos a planilha ppbio06-grupos. Esta é uma tabela de agrupamentos, guardados no arquivo ppbio06-grupos.xlsx, que traz os agrupamentos das UA’s definidos a priori no delineamento amostral do PPBio. Essa tabela contem as ~26 localidades (UAs) em períodos diferentes (linhas) x ~5 tipos de grupos aos quais cada UA foi atribuida (colunas) (dados publicados por Medeiros, Silva, and Ramos (2008)). As bases teóricas dos dados do PPBio para o presente estudo pode ser vista em Base Teórica. Leia antes de prosseguir.
6.1.3 Importando as planilhas
Note que o símbolo # em programação R significa que o texto que vem depois dele é um comentário e não será executado pelo programa. Isso é útil para explicar o código ou deixar anotações.
- Ajuste a primeira linha do código abaixo para refletir “C:/Seu/Diretório/De/Trabalho/Planilha.xlsx”.
- Ajuste o parâmetro sheet = "" para refletir a aba correta do arquivo .xlsx a ser importado.
Alternativamente você pode ir na barra de tarefas e escolhes as opções:
SESSION -> SET WORKING DIRECTORY -> CHOOSE DIRECTORY
library(openxlsx)
ppbio <- read.xlsx("D:/Elvio/OneDrive/Disciplinas/_EcoNumerica/5.Matrizes/ppbio06p-peixes.xlsx",
rowNames = T,
colNames = T,
sheet = "Sheet1")
t_grps <- read.xlsx("D:/Elvio/OneDrive/Disciplinas/_EcoNumerica/5.Matrizes/ppbio06-grupos.xlsx",
rowNames = T,
colNames = T,
sheet = "peixesp")
m_hab <- read.xlsx("D:/Elvio/OneDrive/Disciplinas/_EcoNumerica/5.Matrizes/ppbio06p-habitat.xlsx",
rowNames = T,
colNames = T,
sheet = "ano1")
ppbio[1:5,1:5] #[1:5,1:5] mostra apenas as linhas e colunas de 1 a 5.
t_grps## ap-davis as-bimac as-fasci ch-bimac ci-ocela
## S-R-CT1 0 194 55 0 0
## S-R-CP1 0 19 0 0 0
## S-A-TA1 0 23 1 13 0
## S-R-CT2 0 142 3 3 0
## S-R-CP2 0 5 1 0 40
## area ambiente UA coleta
## S-R-CT1 Serido rio CT 1
## S-R-CP1 Serido rio CP 1
## S-A-TA1 Serido acude TA 1
## S-R-CT2 Serido rio CT 2
## S-R-CP2 Serido rio CP 2
## S-A-TA2 Serido acude TA 2
## S-R-CT3 Serido rio CT 3
## S-R-CP3 Serido rio CP 3
## S-A-TA3 Serido acude TA 3
## S-R-CT4 Serido rio CT 4
## S-R-CP4 Serido rio CP 4
## S-A-TA4 Serido acude TA 4
## B-A-MU1 Buique acude MU 1
## B-A-GU1 Buique acude GU 1
## B-R-PC2 Buique rio PC 2
## B-A-MU2 Buique acude MU 2
## B-A-GU2 Buique acude GU 2
## B-R-PC3 Buique rio PC 3
## B-A-MU3 Buique acude MU 3
## B-A-GU3 Buique acude GU 3
## B-R-PC4 Buique rio PC 4
## B-A-MU4 Buique acude MU 4
## B-A-GU4 Buique acude GU 46.2 REINÍCIO 1
m_bruta <- (ppbio) # <1>- Aqui usaremos as matrizes relativizadas/transformadas/particionadas, etc
Podemos exibir a planilha depois de ter sido importada para o ambiente R/RStudio usando as funções View(), print() ou head(). Note que essas funções são case-sensitive. A função ignore.case() é uma função do pacote stringr que modifica um padrão para que ele não considere o caso das letras nas correspondências. Por exemplo, se você quiser encontrar todas as ocorrências da letra “a” em um vetor de caracteres, independente de ser “A” ou “a”, você pode usar essa função.
6.3 REINÍCIO 2
m_trab <- (m_bruta) # <1>- Aqui usaremos as matrizes relativizadas/transformadas/particionadas, etc
6.4 DCA
https://www.davidzeleny.net/anadat-r/doku.php/en:ordiagrams_examples
library (vegan)
#veg.data <- read.delim ('https://raw.githubusercontent.com/zdealveindy/anadat-r/master/data/vltava-spe.txt', row.names = 1)
#env.data <- read.delim ('https://raw.githubusercontent.com/zdealveindy/anadat-r/master/data/vltava-env.txt')
com.data <- m_trab
env.data <- t_grps
env.data
#env.data <- within(env.data, ambiente <- factor(ambiente, labels = c(1,2)))
#lvs <- factor(env.data$area, labels = c(2,1))
grp <- env.data$area
env.data$lvs <- factor(env.data$area, labels = c(2,1)) #adiciona uma coluna chamada levels
env.data$lvs <- as.numeric(as.character(env.data$lvs)) #lvs como numeros
lvs <- env.data$lvs
DCA <- decorana(com.data)
DCA
summary(DCA)
plot(DCA, choices = c(1,2), display = "sites", type = "text")
points(DCA)
par(mfrow=c(2,2))
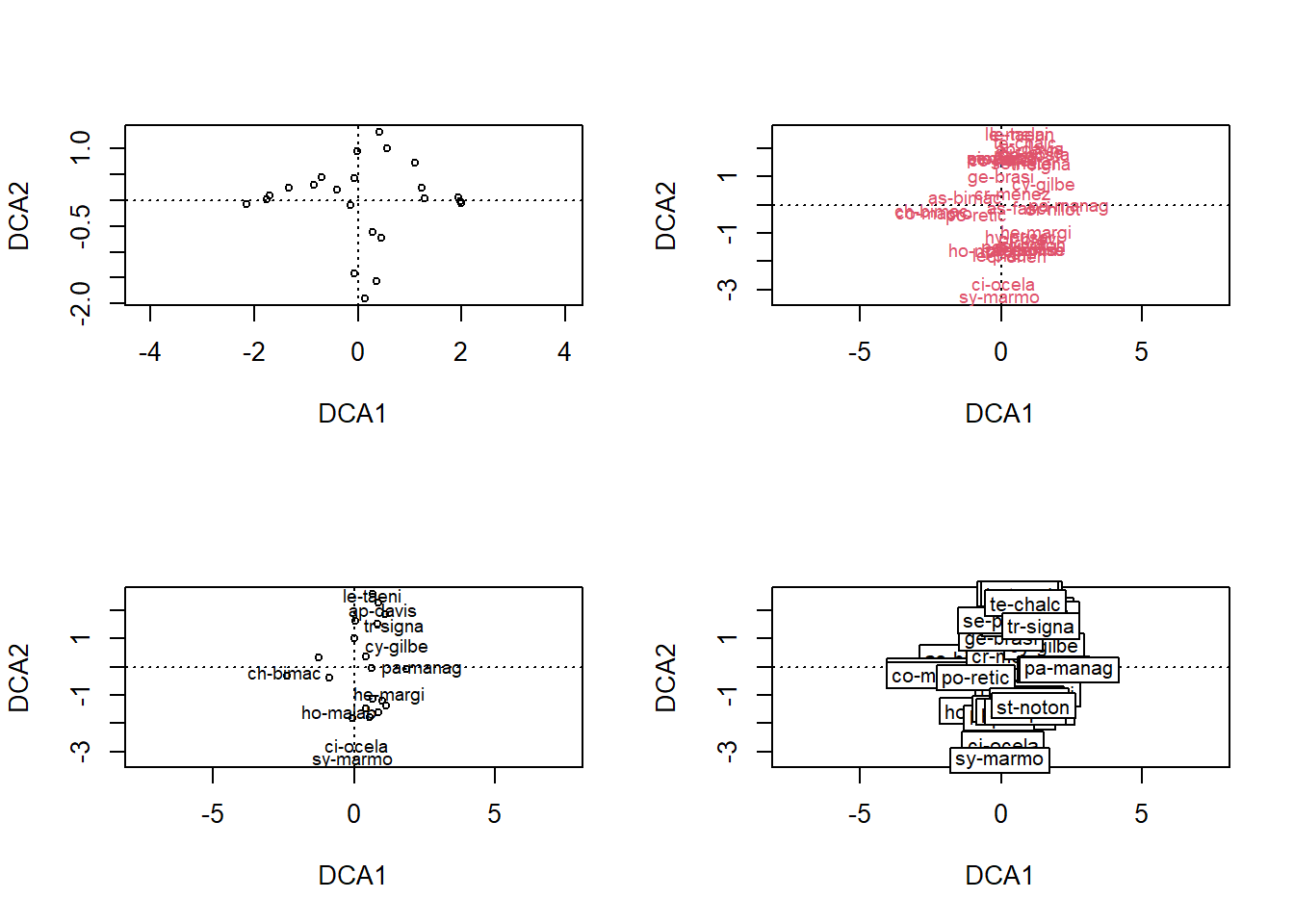
ordiplot(DCA, display = 'sites', type = 'p')
ordiplot(DCA, display = 'species', type = 't')
ordiplot(DCA, display = 'sp', type = 'n')
orditorp(DCA, display = 'sp')
ordiplot(DCA, display = 'sp', type = 'n')
ordilabel(DCA, display = 'sp')
par(mfrow=c(1,1))
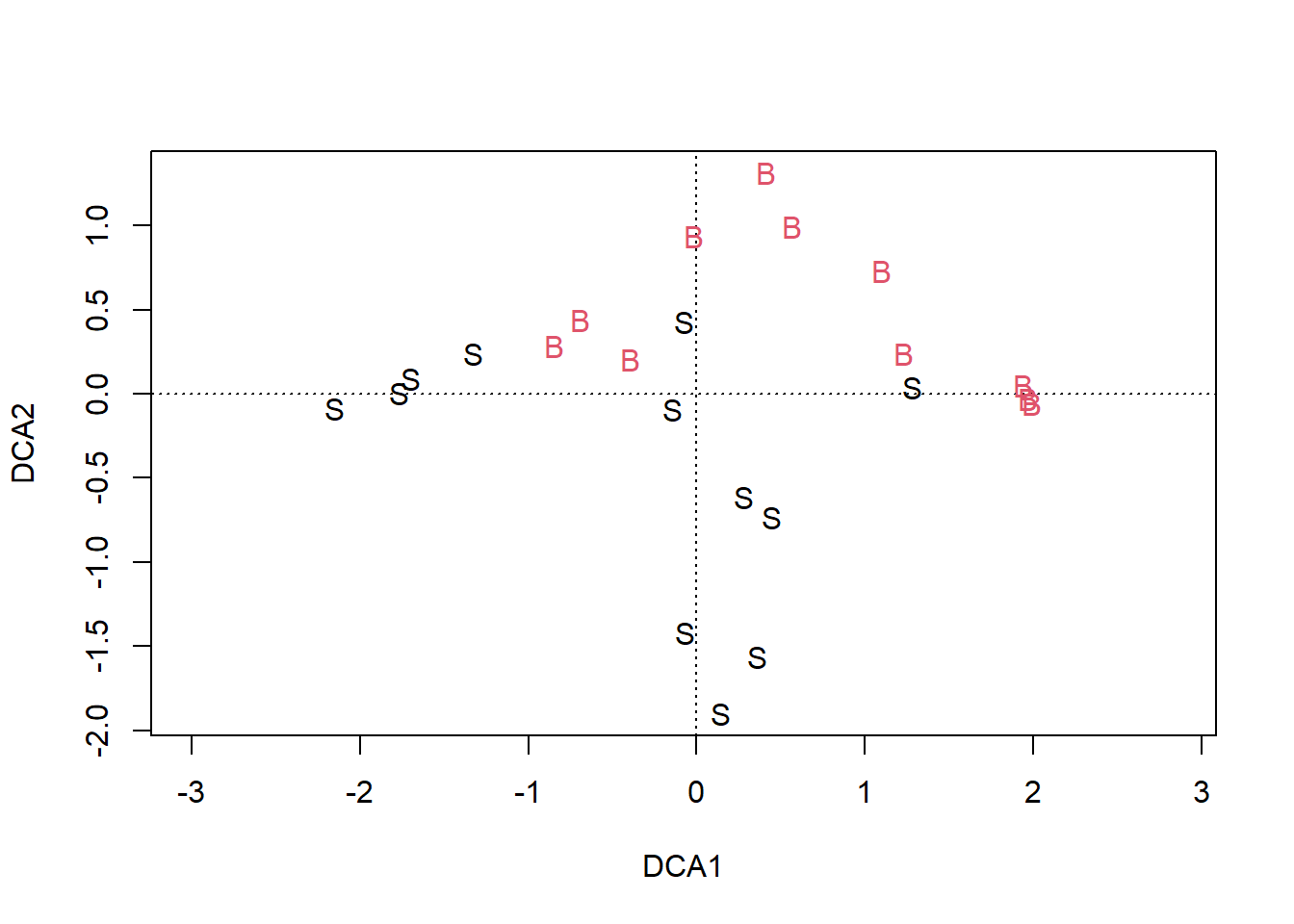
ordiplot(DCA, display = 'si', type = 'n')
points(DCA, col = lvs, pch = grp)
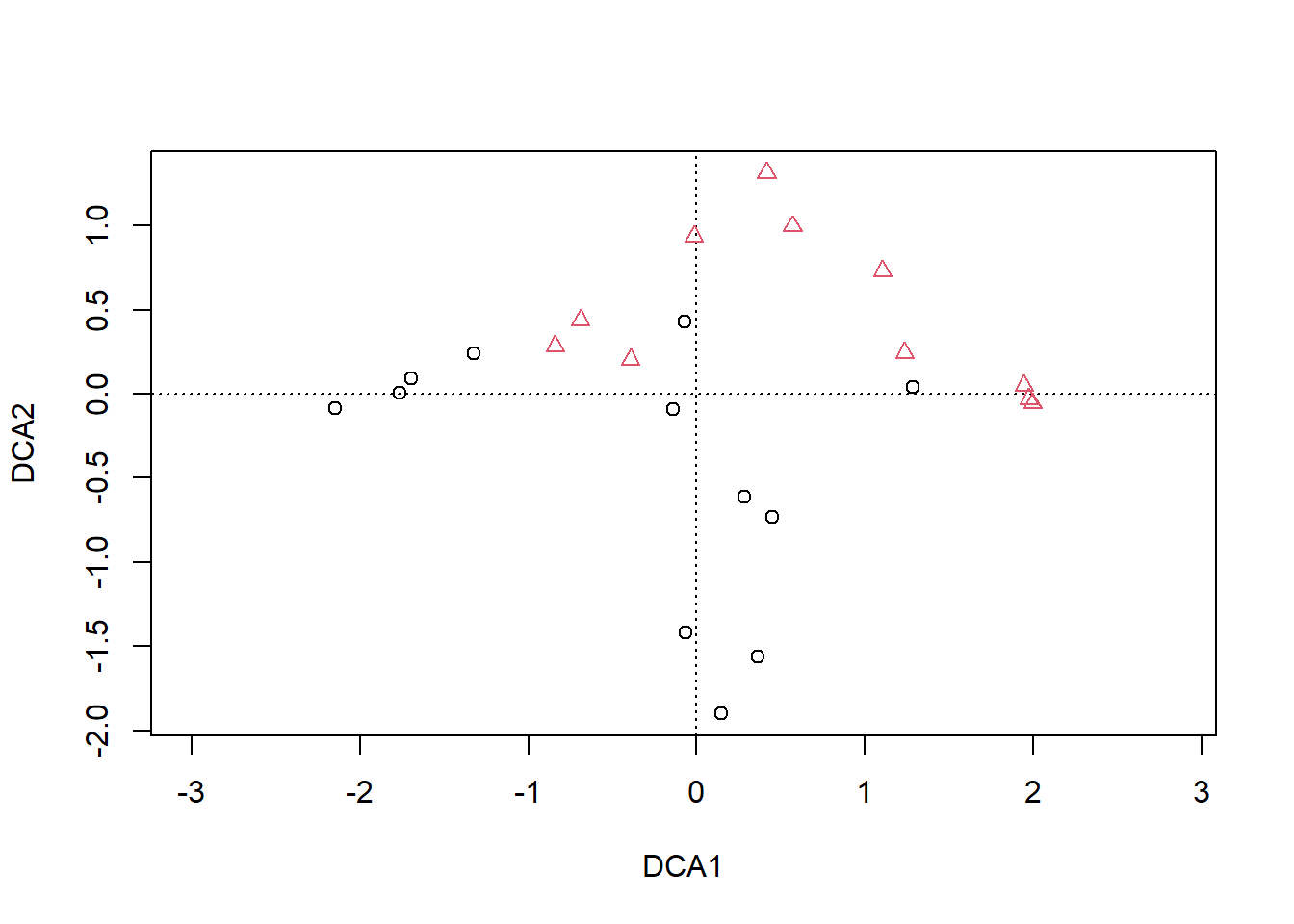
ordiplot(DCA, display = 'si', type = 'n')
points(DCA, col = lvs, pch = lvs)
ordiplot(DCA, display = 'si', type = 'n')
for (i in seq (1, 2)) ordispider (DCA, groups = lvs, show.groups = i, col = i, label = T)
for (i in seq (1, 2)) ordihull (DCA, groups = lvs, show.groups = i, col = i, lty = 'dotted')
ordiplot(DCA, display = 'si', type = 'n')
points(DCA, col = lvs, pch = lvs)
for (i in unique (lvs)) ordihull (DCA, groups = lvs, show.group = i, col = i, draw = 'polygon', label = T)
source('http://www.davidzeleny.net/anadat-r/doku.php/en:customized_functions:ordicenter?do=export_code&codeblock=0')
ordiplot(DCA, display = 'si', type = 'n')
ordicenter(DCA, groups = grp, col = 'red', cex = 2)
ordiplot(DCA, display = 'si', type = 'n')
scaling.parameter <- as.vector(table(lvs))/max(as.vector(table(lvs)))
for (i in 1:length (unique (lvs)))
ordicenter (DCA, groups = lvs, show.groups = i, col = i, cex = 4*scaling.parameter[i])
colnames(m_hab)
ordiplot(DCA, display = 'si', type = 'n')
ordiarrows(DCA, groups = env.data$area, order.by = grp, startmark = 1, label = TRUE, length = .1) #integers## area ambiente UA coleta
## S-R-CT1 Serido rio CT 1
## S-R-CP1 Serido rio CP 1
## S-A-TA1 Serido acude TA 1
## S-R-CT2 Serido rio CT 2
## S-R-CP2 Serido rio CP 2
## S-A-TA2 Serido acude TA 2
## S-R-CT3 Serido rio CT 3
## S-R-CP3 Serido rio CP 3
## S-A-TA3 Serido acude TA 3
## S-R-CT4 Serido rio CT 4
## S-R-CP4 Serido rio CP 4
## S-A-TA4 Serido acude TA 4
## B-A-MU1 Buique acude MU 1
## B-A-GU1 Buique acude GU 1
## B-R-PC2 Buique rio PC 2
## B-A-MU2 Buique acude MU 2
## B-A-GU2 Buique acude GU 2
## B-R-PC3 Buique rio PC 3
## B-A-MU3 Buique acude MU 3
## B-A-GU3 Buique acude GU 3
## B-R-PC4 Buique rio PC 4
## B-A-MU4 Buique acude MU 4
## B-A-GU4 Buique acude GU 4
##
## Call:
## decorana(veg = com.data)
##
## Detrended correspondence analysis with 26 segments.
## Rescaling of axes with 4 iterations.
## Total inertia (scaled Chi-square): 3.8345
##
## DCA1 DCA2 DCA3 DCA4
## Eigenvalues 0.6772 0.3542 0.3524 0.22035
## Additive Eigenvalues 0.6772 0.3520 0.3686 0.21920
## Decorana values 0.7171 0.3430 0.1673 0.03794
## Axis lengths 4.1449 3.2131 2.7721 1.76694
##
##
## Call:
## decorana(veg = com.data)
##
## Detrended correspondence analysis with 26 segments.
## Rescaling of axes with 4 iterations.
## Total inertia (scaled Chi-square): 3.8345
##
## DCA1 DCA2 DCA3 DCA4
## Eigenvalues 0.6772 0.3542 0.3524 0.22035
## Additive Eigenvalues 0.6772 0.3520 0.3686 0.21920
## Decorana values 0.7171 0.3430 0.1673 0.03794
## Axis lengths 4.1449 3.2131 2.7721 1.76694
##
## Species scores:
##
## DCA1 DCA2 DCA3 DCA4 Totals
## ap-davis 1.02221 1.98581 1.54783 0.97680 27
## as-bimac -1.25430 0.31830 0.70008 0.36371 2283
## as-fasci 0.62664 -0.05284 1.80207 -0.92737 158
## ch-bimac -2.43457 -0.16336 -0.24672 0.11676 705
## ci-ocela 0.06836 -2.75464 -1.07216 1.84959 70
## ci-orien 0.54231 -1.78864 -0.64528 2.09638 143
## co-macro -2.36880 -0.31585 0.16658 0.81494 2
## co-heter 0.04013 1.63747 2.92555 -2.51734 1
## cr-menez 0.40767 0.36859 1.94856 -1.37148 28
## cu-lepid 1.09467 1.85208 1.71132 1.00871 21
## cy-gilbe 1.51716 0.72004 1.66175 1.77251 131
## ge-brasi 0.00303 1.00732 -1.57442 -0.53060 1020
## he-margi 1.25370 -0.97209 -1.01562 1.26984 2
## ho-malab -0.53496 -1.55983 -0.17666 1.45752 109
## hy-pusar 0.64816 -1.14840 1.01223 1.68117 71
## le-melan 0.66235 2.57426 -1.13478 0.80265 2
## le-piau -0.06989 -1.82065 1.37399 -0.41094 15
## le-taeni 0.66235 2.57426 -1.13478 0.80265 1
## mo-costa 1.09467 1.85208 1.71132 1.00871 1
## mo-lepid 0.05841 1.62472 2.87740 -2.39035 40
## or-nilot 1.86365 -0.09015 0.21326 -0.17812 838
## pa-manag 2.41173 -0.11494 -0.49638 0.43929 553
## pimel-sp 0.04013 1.63747 2.92555 -2.51734 6
## po-retic -0.89266 -0.38665 -0.92709 -0.09085 347
## po-vivip 0.40644 -1.49198 -0.32826 -0.66872 978
## pr-brevi 0.99059 -1.20902 -0.79831 -1.16156 270
## ps-rhomb 0.85392 -1.60142 -0.84458 -1.35054 1
## ps-genise 0.85392 -1.60142 -0.84458 -1.35054 1
## se-heter 0.82778 1.53488 1.68472 1.44012 296
## se-piaba 0.04012 1.63747 2.92555 -2.51735 68
## se-spilo 0.85392 -1.60142 -0.84458 -1.35054 1
## st-noton 1.14624 -1.37592 -0.95243 -1.11405 205
## sy-marmo -0.04511 -3.32486 -1.28164 2.07808 1
## te-chalc 0.86594 2.26168 0.60829 0.89962 134
## tr-signa 1.40721 1.42576 -0.56360 -1.45080 208
##
## Site scores:
##
## DCA1 DCA2 DCA3 DCA4 Totals
## S-R-CT1 -0.072166 0.427833 1.180734 -0.496695 545
## S-R-CP1 -0.136811 -0.089812 0.601908 0.503481 55
## S-A-TA1 -1.323091 0.239878 0.491361 0.322733 42
## S-R-CT2 0.286326 -0.610759 0.130103 0.195213 717
## S-R-CP2 0.144642 -1.900141 -0.507305 0.789985 108
## S-A-TA2 -2.149733 -0.087604 -0.059923 0.167128 228
## S-R-CT3 0.447823 -0.731616 0.000618 -0.351221 1144
## S-R-CP3 -0.063382 -1.420951 -0.306451 1.160429 68
## S-A-TA3 -1.764436 0.006795 0.228571 0.273823 501
## S-R-CT4 1.286243 0.040435 -0.219933 -0.606515 436
## S-R-CP4 0.363410 -1.562958 -0.331514 -0.136156 104
## S-A-TA4 -1.693769 0.088877 0.303842 0.265948 684
## B-A-MU1 -0.015834 0.935908 -1.391628 -0.468836 208
## B-A-GU1 1.235436 0.241895 -0.433478 -0.021747 25
## B-R-PC2 0.414912 1.312989 0.348081 0.328698 185
## B-A-MU2 -0.689885 0.438438 -0.258269 -0.024308 186
## B-A-GU2 1.945079 0.050928 -0.491704 0.162680 161
## B-R-PC3 0.568935 0.996155 0.832976 0.634867 371
## B-A-MU3 -0.840642 0.284921 0.095272 0.091237 762
## B-A-GU3 1.973505 -0.031507 -0.219811 0.095384 535
## B-R-PC4 1.104079 0.729371 1.380474 1.064008 156
## B-A-MU4 -0.393996 0.205616 -0.797919 -0.270823 1174
## B-A-GU4 1.995217 -0.056304 -0.214747 0.175175 342
##
## [1] "h.macroph" "h.grass" "h.subveg" "h.overhveg" "h.litter"
## [6] "h.filalgae" "h.attalgae" "h.roots" "h.lrgdeb" "h.smldeb"
## [11] "s.mud" "s.sand" "s.smlgrav" "s.lrggrav" "s.cobbles"
## [16] "s.rocks" "s.bedrock" "m.elev" "m.river" "m.stream"
## [21] "m.distsource" "m.distmouth" "m.maxslope" "m.maxdepth" "m.habdepth"
## [26] "m.width" "a.veloc" "a.temp" "a.do" "a.transp"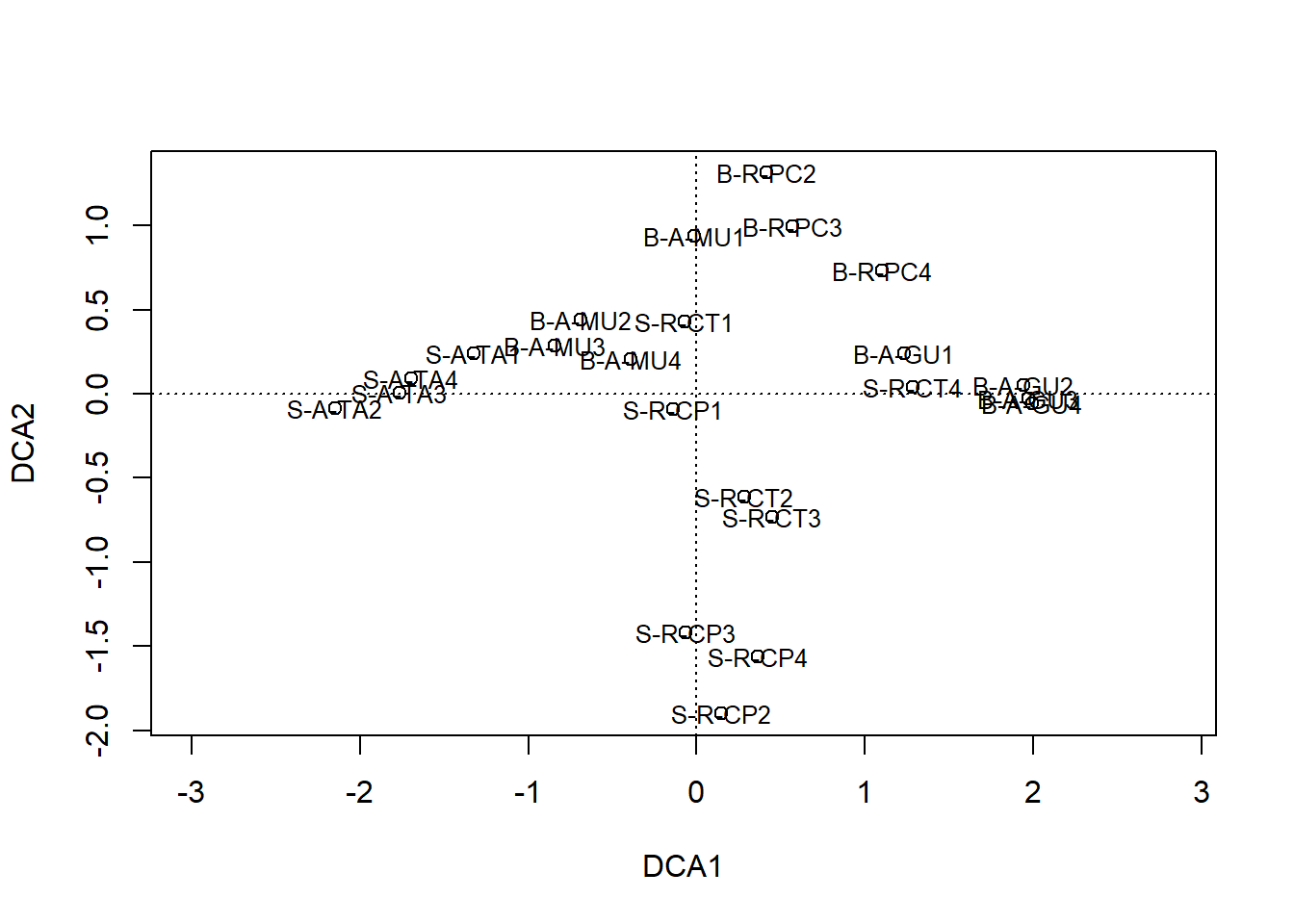
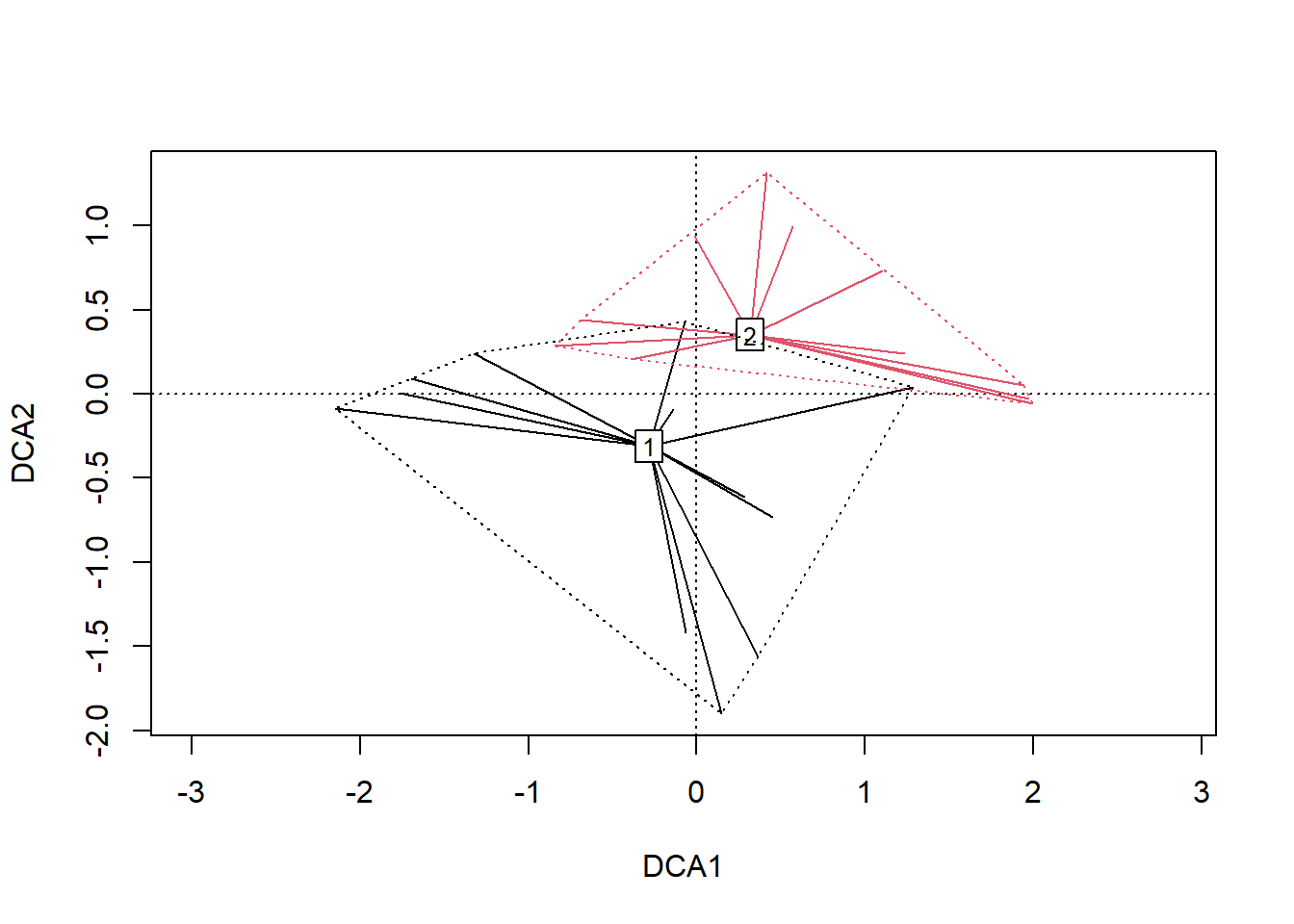
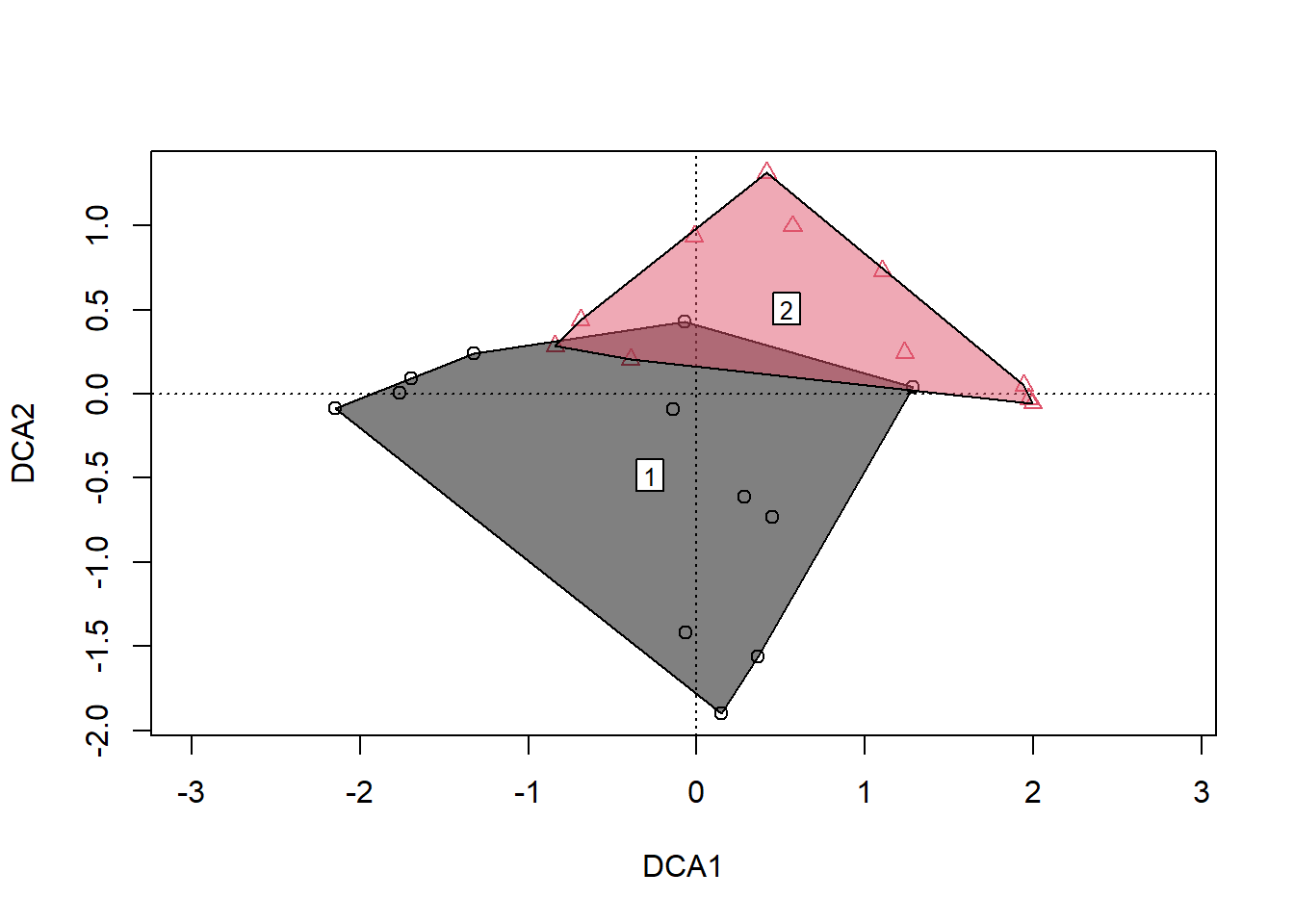

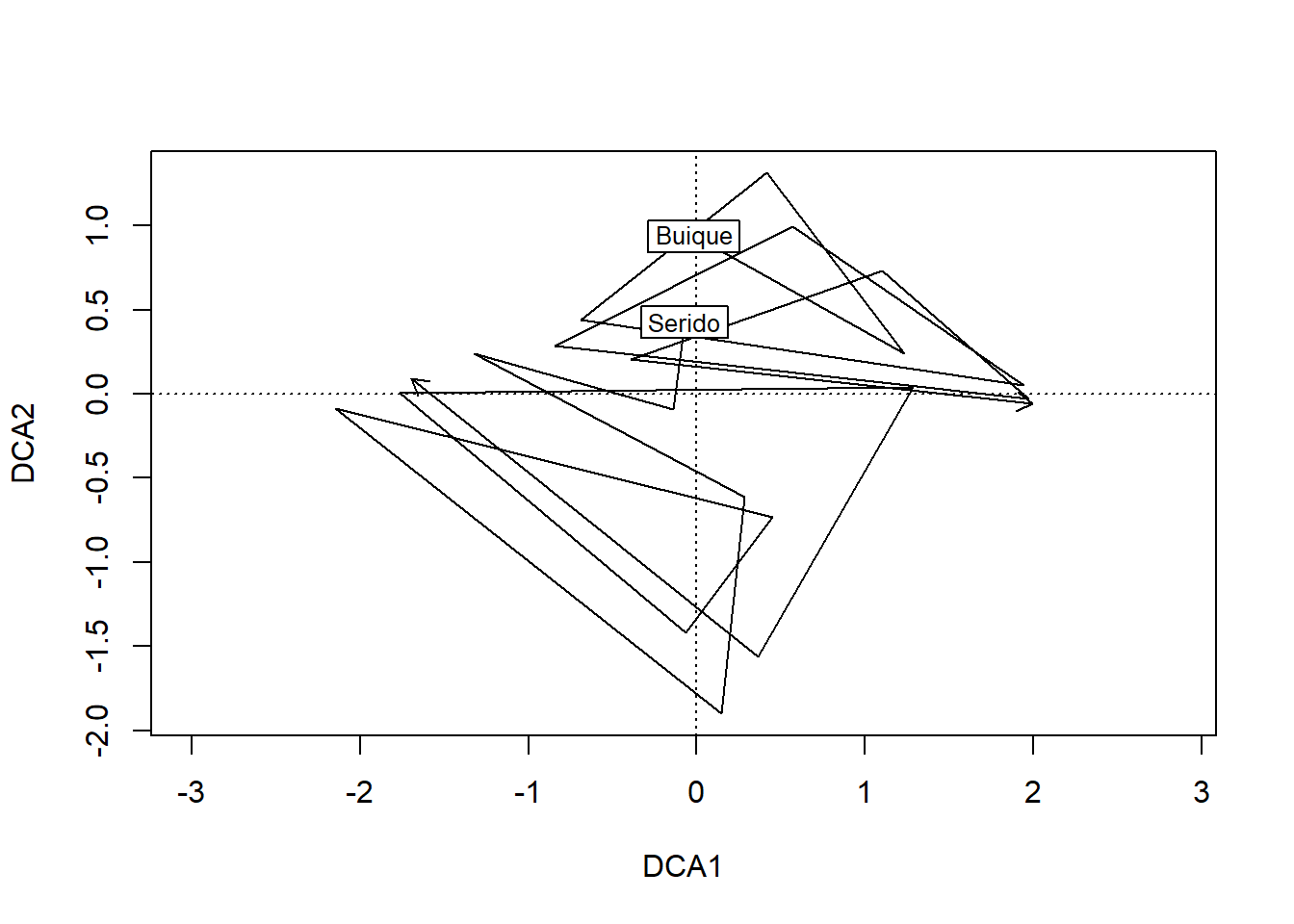
6.4.1 Fazendo gráficos em 3D
Ao executar a função ´ordirgl()´ procure pelo widget criado usando o pacote rgl e abra-o em uma segunda tela.
library (vegan3d)## Registered S3 methods overwritten by 'vegan3d':
## method from
## plot.orditkplot vegan
## points.orditkplot vegan
## scores.orditkplot vegan
## text.orditkplot vegan##
## Attaching package: 'vegan3d'## The following object is masked from 'package:vegan':
##
## orditkplotlibrary(rgl)
ordirgl(DCA)
orglspider(DCA, groups = grp)
source('http://www.davidzeleny.net/anadat-r/doku.php/en:customized_functions:orglhull?do=export_code&codeblock=1')
orglhull(DCA, groups = grp, col = 'tomato', alpha = 0.5)## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11] [,12] [,13] [,14]
## [1,] 9 10 6 6 6 4 4 4 4 5 5 5 5 5
## [2,] 11 11 3 9 9 10 10 3 1 10 10 9 1 9
## [3,] 7 7 7 7 3 7 1 7 3 11 1 11 3 3
## [,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10] [,11] [,12]
## [1,] 5 3 3 9 9 11 11 11 12 12 12 12
## [2,] 10 10 1 5 1 5 1 1 3 9 3 9
## [3,] 6 6 10 6 5 10 10 5 1 1 6 66.4.2 Fazendo a animação do gráfico
#veg.data <- read.delim ('https://raw.githubusercontent.com/zdealveindy/anadat-r/master/data/vltava-spe.txt', row.names = 1)
#env.data <- read.delim ('https://raw.githubusercontent.com/zdealveindy/anadat-r/master/data/vltava-env.txt')
library(magick)
temp.dir <- tempdir ()
DCA <- decorana(veg = com.data)
rgl.bg(color = 'white') # makes the background white
ordirgl(DCA)
movie3d(spin3d(axis = c(0,1,0)), duration=60/5, movie = "ordirgl", fps = 20, dir = temp.dir)
orglspider(DCA, groups = grp)
movie3d(spin3d(axis = c(0,1,0)), duration=60/5, movie = "orglspider", fps = 20, dir = temp.dir)
library(geometry)
source('http://www.davidzeleny.net/anadat-r/doku.php/en:customized_functions:orglhull?do=export_code&codeblock=1')
orglhull(DCA, groups = grp, col = 'tomato', alpha = 0.5)
movie3d(spin3d(axis = c(0,1,0)), duration=60/5, movie = "orglhull", fps = 20, dir = temp.dir)
# Gif files are stored in tempdir:
temp.dir