7 R Modulo 1
RESUMO
A importação de planilhas do Excel para o ambiente de programação R é uma tarefa fundamental para análise de dados e estatísticas. Através da importação de planilhas do Excel, é possível transformar dados armazenados em formatos familiares em estruturas que podem ser manipuladas e exploradas de maneira eficaz no R. Isso permite a aplicação de diversas técnicas estatísticas e criação de visualizações informativas, contribuindo para a tomada de decisões embasadas em dados. Neste contexto, entender como importar dados do Excel para o R é um passo crucial para realizar análises de alta qualidade e obter insights significativos a partir dos conjuntos de dados disponíveis.
Apresentação
A importação de planilhas do Excel para o ambiente de programação R é uma tarefa fundamental para análise de dados e estatísticas. O R é uma linguagem de programação amplamente utilizada por cientistas de dados, pesquisadores e analistas para manipular, visualizar e modelar informações. Através da importação de planilhas do Excel, é possível transformar dados armazenados em formatos familiares em estruturas que podem ser manipuladas e exploradas de maneira eficaz no R. Isso permite a aplicação de diversas técnicas estatísticas e criação de visualizações informativas, contribuindo para a tomada de decisões embasadas em dados. Neste contexto, entender como importar dados do Excel para o R é um passo crucial para realizar análises de alta qualidade e obter insights significativos a partir dos conjuntos de dados disponíveis.
7.2 R base
- O primeiro passo é entrar na página do projeto CRAN (Comprehensive R Archive Network) (https://www.r-project.org/).
- Do lado esquerdo da página clique sobre o link CRAN abaixo de Download. 3. Uma nova página com uma série de links irá se abrir. Esses links são chamados de “espelhos” e servem para que você possa escolher o local mais próximo de onde você está para fazer o download do programa. Escolha um espelho no Brasil.
- Na seção Download and Install R, clique sobre o link
Download R for Windowspara baixar a versão para esse sistema… (MacOS??… o que é isso?)
- Clique sobre o link
base. - Clique sobre o link
Download R 4.x.x for Windowspara fazer o download do arquivo R.exe. - A instalação segue o formato padrão de instalação de programas no Windows, e portanto não são necessários maiores detalhes.
7.3 RStudio
- Para baixar o RStudio entre no endereço (https://posit.co/download/rstudio-desktop/)
- Clique no link
Products > RStudio
- Selecione a versão Desktop.
- Clique em
DOWNLOAD RSTUDIO DESKTOP
- Será exibida uma página com a recomendação para você baixar o RStudio FREE versão mais recente - Windows
- Clicando nesse link, você irá baixar o arquivo RStudio atual (.exe)
- Depois é só clicar e instalar da forma convencional do Windows.
Após a instalação, você pode abrir o RStudio pelo seu respectivo ícone, e o RStudio estará pronto para ser utilizado. O “R base” continuará instalado mas será acessado pelo RStudio. Não o desinstale.
7.3.1 RStudio na nuvem
Usar o RStudio Cloud (https://login.rstudio.cloud/) é uma opção para quem não quer instalar a versão para PC. O RStudio Cloud é uma plataforma online que fornece um ambiente de desenvolvimento integrado para o R, permitindo que os usuários executem análises, desenvolvam código e colaborem com outras pessoas, sem a necessidade de instalar o R e o RStudio em seus próprios computadores. É uma solução conveniente e acessível, especialmente para iniciantes ou usuários que desejam compartilhar projetos e colaborar de forma eficiente.
7.4 Sobre os dados do PPBio
Usaremos para esse tutorial dados coletados no Programa de Pesquisa em Biodiversidade - PPBio (Veja Programa de Pesquisa em Biodiversidade – PPBio). Nesta base de dados estão armazenadas informações sobre diversos grupos taxonômicos dstribuidos em diversas unidades amostrais (UA’s ou sítios), como peixes, macroinverbrebrados bentônicos, quironomídeos e zooplâncotn, além de dados do habitat, como variáveis físicas e químicas, morfologia do habitat, composição do substrato, estrutura de habitat marginal, entre outros (Figura 7.1). Essa é a matriz bruta de dados, porque os valores ainda não foram ajustados para os valores de Captura Por Unidade de Esforço (CPUE), nem foram relativizados ou transformados (Tabela 7.1).
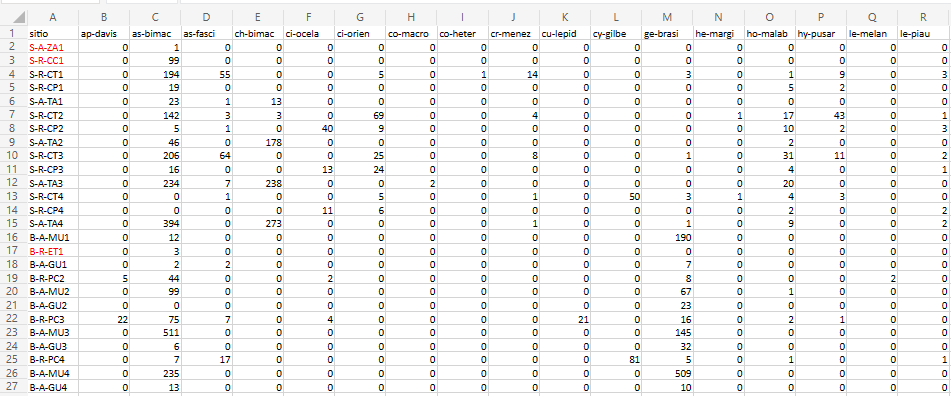
Figura 7.1: Parte da planilha de dados brutos do PPBio.
| Arquivo (.xlsx) | Tipo de matriz | Descrição | Tipo de dados |
|---|---|---|---|
| ppbio06c | Matriz comunitária | O arquivo ppbio06 traz os dados brutos que serão usados nas análises. A matriz de dados brutos contendo 26 localidades em estações do ano diferentes (objetos) x 35 espécies (atributos), antes de qualquer modificação. | Contagens de indivíduos com alta amplitude de variação, sugerido uso de matriz relativizada. |
| ppbio06h | Matriz ambiental | O arquivo ppbio06h traz os dados brutos que serão usados nas análises. A matriz de dados brutos contendo 26 localidades em estações diferentes (objetos) x 35 variáveis ambienteis (atributos) medidas em diferentes escalas espaciais, antes de qualquer modificação. | Unidades de medição diferentes (cm, m, °C, mg/L, etc.), com uma alta amplitude de variação, sugerido uso de matriz transformada e/ou reescalada. |
| ppbio06 | Matriz comunitária | O arquivo ppbio06 traz os dados brutos que serão usados nessa análise. A matriz de dados brutos contendo 26 locais/ocasiões (objetos) x 35 espécies (atributos), antes de qualquer modificação. | Contagens de indivíduos com alta amplitude de variação, sugerido uso de matriz relativizada. |
| ppbio06cpue | Matriz comunitária | O arquivo ppbio06cpue traz os valores depois de terem sidos ajustados pela Captura Por Unidade de Esforço (CPUE), onde o número de indivíduos de cada espécie em uma determinada UA é dividido pelo esforço de captura daquela UA. Isso significa que os dados foram relativizados pela CPUE. A matriz de dados brutos contendo 26 localidades em estações do ano diferentes (objetos) x 35 espécies (atributos), antes de qualquer modificação. | Densidades de indivíduos (no. de indivíduos por Unidade de Esforço de Captura) com alta amplitude de variação, sugerido uso de matriz relativizada. |
A planilha ppbio contém o delineamento amostral de um dos estudos do Projeto PPBio (Figura 7.2). Nas linhas são apresentadas as abreviações dos nomes das unidades amostrais (UA’s) e nas colunas são apresentados os nomes abreviados das espécies - temos portando uma matriz comunitária (Tabela 7.1). No corpo da planilha temos os valores para o tipo de dados amostrado. Quantitativo, semi-quatitativo ou qualitativo.
Qual desses tipos de dados você acha que é apresentado na planilha?
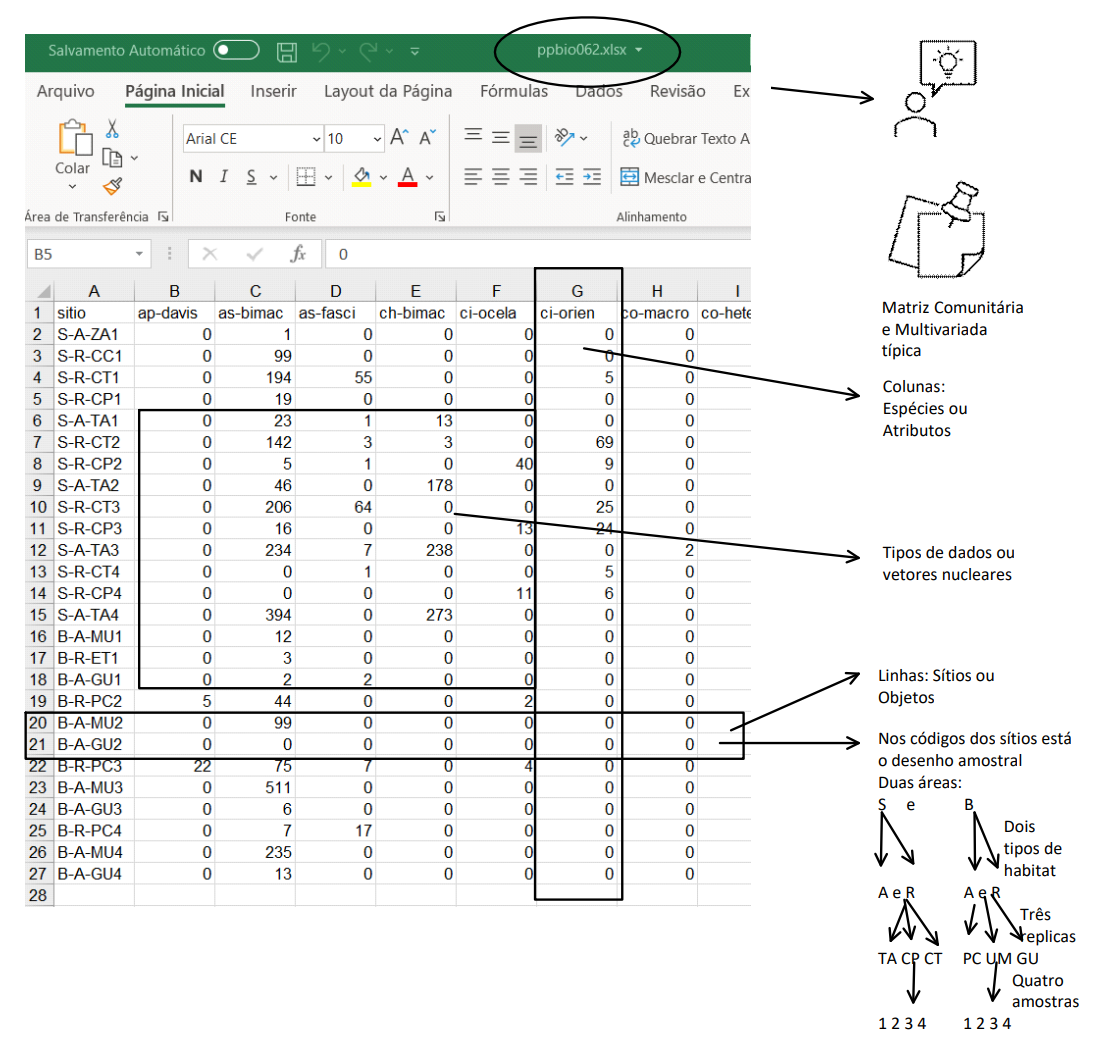
Figura 7.2: Associação entre a planilha de dados brutos do PPBio e o delineamento amostral do estudo.
Várias das espécies nessa matriz tem grande importância ecológica, como é o caso de Astyanax bimaculatus 11 (Figura 7.3), que é muito comum em rios intermitentes e serve de alimento para predadores maiores como a espécie Hoplias malabaricus 12 (Figura 7.4).

Figura 7.3: Astyanax bimaculatus, a espécie mais comum da matriz de dados ppbio. Peru, by Eakins, R. Fonte: https://www.fishbase.se/summary/Astianax-bimaculatus.html

Figura 7.4: Hoplias malabaricus, espécie que cresce para se tornar um importante predador. Brazil, by Roselet, F.F.G. Fonte: https://www.fishbase.se/summary/Hoplias-malabaricus.html
7.5 Importando a planilha de trabalho
Para começar a usar o R e analisar os dados do Projeto PPBio, abra o RStudio, verifique sua interface (Figura 7.5) e siga as instruções a seguir.
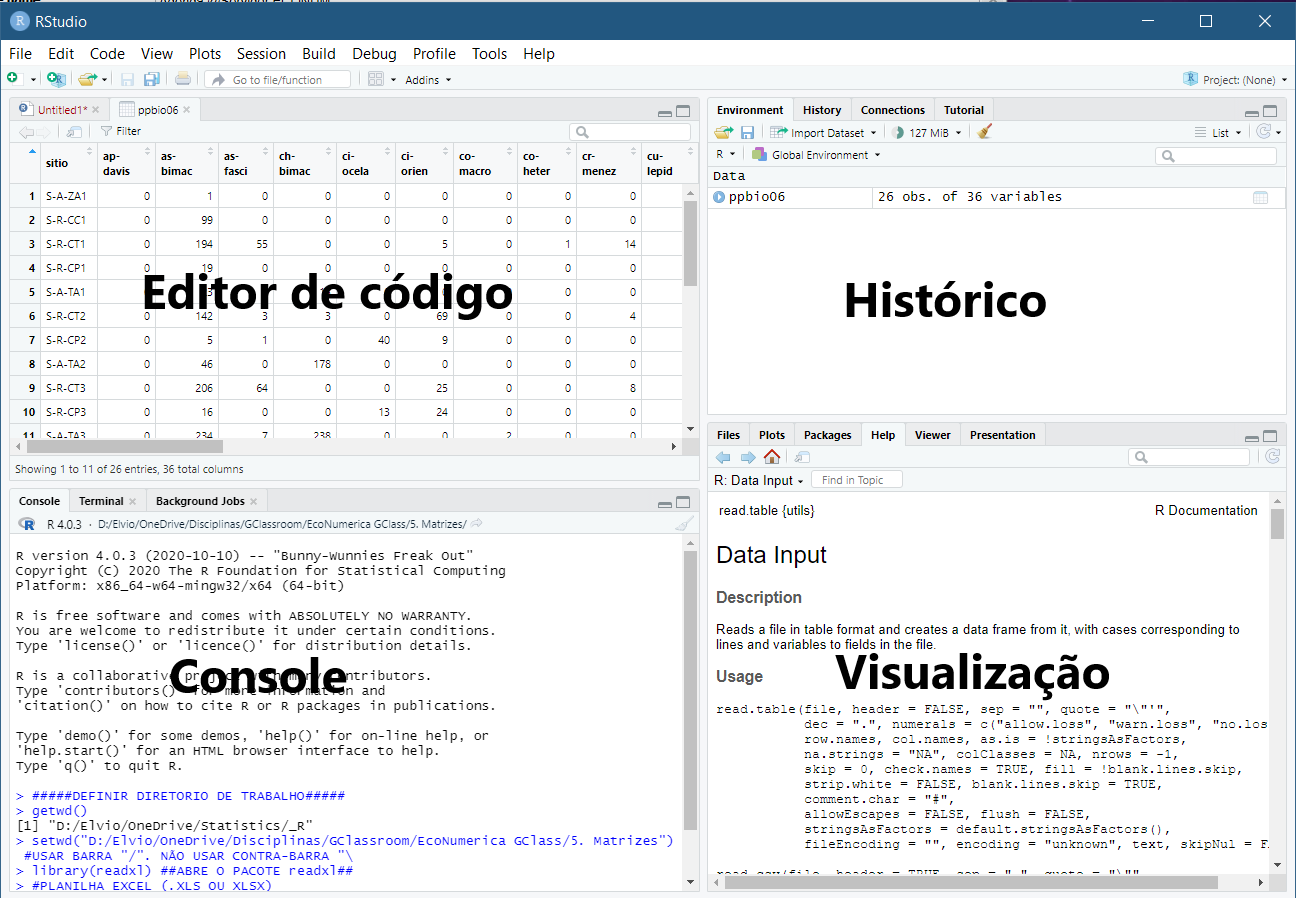
Figura 7.5: Interface típica do RStudio e nome dos paineis ou janelas.
7.6 Mensagens de erro e avisos no R
No contexto da linguagem de programação R, mensagens de erro (errors) e mensagens de aviso (warnings) que aparecem em vermelho no painel de console. Elas são formas de feedback do sistema que indicam problemas ou situações potencialmente problemáticas durante a execução do código. Aqui está uma breve explicação de cada um:
- Erro (Error):
- Um erro ocorre quando algo no código não está correto ou não pode ser executado como esperado.
- Isso pode ser causado por sintaxe incorreta, uso incorreto de funções, operações inválidas, referências a objetos que não existem, entre outros problemas.
- Quando ocorre um erro, a execução do código é interrompida e uma mensagem de erro é exibida no console em vermelho, indicando o tipo de erro e, muitas vezes, a linha onde ocorreu.
- Aviso (Warning):
- Não indica erro. Um aviso é emitido quando algo no código pode resultar em um comportamento indesejado ou em resultados inesperados, mas não interrompe necessariamente a execução do código.
- Os avisos geralmente indicam situações que merecem atenção, como conversões de tipos de dados que podem perder informações ou funções que estão sendo usadas de maneira que pode levar a resultados questionáveis.
- Os avisos são exibidos em vermelho no console e fornecem informações sobre a natureza do aviso e, possivelmente, como abordá-lo.
É importante prestar atenção a mensagens de erro e avisos, pois eles fornecem insights sobre problemas em seu código ou potenciais fontes de comportamento inesperado. Resolver erros é fundamental para que o código funcione conforme o esperado. Embora os avisos não interrompam a execução, investigá-los pode ajudar a evitar problemas futuros ou melhorar a qualidade do código.
7.7 Organização básica
No ambiente do RStudio no painel de edição de código execute (Ctrl+Enter com o teclado ou Run no editor de código) os comandos a seguir, para instalar os pacotes necessários para este módulo.
E em seguida,
Os códigos acima, são usados para instalar e carregar os pacotes necessários para este módulo. Esses códigos são comandos para instalar pacotes no R. Um pacote é uma coleção de funções, dados e documentação que ampliam as capacidades do R (R CRAN) (R. C. Team 2017), e RStudio (R. S. Team 2022). No exemplo acima, o pacote readxl permite ler e escrever arquivos Excel no R.
Para instalar um pacote no R, você precisa usar a função install.packages(). Depois de instalar um pacote, você precisa carregá-lo na sua sessão R com a função library().
Por exemplo, para carregar o pacote readxl, você precisa executar a função library(readxl). Isso irá permitir que você use as funções do pacote na sua sessão R. Você precisa carregar um pacote toda vez que iniciar uma nova sessão R e quiser usar um pacote instalado.
Agora vamos definir o diretório de trabalho. Esse código é usado para obter e definir o diretório de trabalho atual no R. O comando getwd() retorna o caminho do diretório onde o R está lendo e salvando arquivos. O comando setwd() muda esse diretório de trabalho para o caminho especificado entre aspas. No seu caso, você deve ajustar o caminho para o seu próprio diretório de trabalho. Lembre de usar a barra “/” entre os diretórios. E não a contra-barra “\”.
Usaremos uma matriz multivariada (sítios x espécies, matriz comunitária) do Projeto PPBio chamada ppbio**.xlsx que está no diretório “C:/Meu/Diretório/De/Trabalho/Planilha.xlsx”
Note que o símbolo # em programação R significa que o texto que vem depois dele é um comentário e não será executado pelo programa. Isso é útil para explicar o código ou deixar anotações.
Ajuste a segunda linha do código abaixo para refletir “C:/Seu/Diretório/De/Trabalho/Planilha.xlsx”.
Definindo o diretório de trabalho e installando os pacotes necessários:
Alternativamente você pode ir na barra de tarefas e escolhes as opções:\SESSION -> SET WORKING DIRECTORY -> CHOOSE DIRECTORY
7.8 Importando a planilha
library(readxl)
ppbio06 <- read_excel("D:/Elvio/OneDrive/Disciplinas/_EcoNumerica/5.Matrizes/ppbio06-peixes.xlsx",
sheet = "Sheet1",
na = "NA")
str(ppbio06)
class(ppbio06)Com essas linhas de código a primeira coluna da matriz importada apresenta texto. Não queremos assim porque vamos fazer cálculos matemáticos na matriz.
Resolvemos o problema com mais algumas linhas de código.
ppbio06 <- as.data.frame(ppbio06)
class(ppbio06)
rownames(ppbio06) <- ppbio06[,1] #tem que ser um df
ppbio06[,1] <- NULLOu podemos instalar esse pacote de importação de arquivos .xlsx para o R.
library(openxlsx)
ppbio <- read.xlsx("D:/Elvio/OneDrive/Disciplinas/_EcoNumerica/5.Matrizes/ppbio06-peixes.xlsx",
rowNames = T,
colNames = T,
sheet = "Sheet1")
str(ppbio)
class(ppbio)
ppbio_ma <- as.matrix(ppbio) #lê ppbio como uma matriz
str(ppbio_ma)
class(ppbio_ma)
#ppbio
#ppbio_maCompare as diferenças. Agora podemos exportar os dados como uma matriz de dados em formato de valores separados por vírgula (.csv).
write.table(ppbio, "ppbiocsv.txt", append = F, quote = T, ";", row.names = T)
dir <- getwd()
shell.exec(dir) #abre o diretorio de trabalho no Windows ExplorerPodemos abrir o arquivo .csv criado ppbiocsv.txt usando os códigos abaixo.
ppbiocsv <- read.csv("ppbiocsv.txt",
sep = ";", dec = ",", #definimos o dígito separador
header = T,
row.names = 1,
na.strings = NA)
str(ppbiocsv)
ppbiocsvLembre de prestar atenção no dígito separador de decimais ” , ” ou ” . ” . Além disso, só estaamos usando ppbio**.*** porque o diretório de trabalho ja fo definido no início. Se não deveríamos estar usando C:/Seu/Diretório/De/Trabalho/ppbio**.***
Alguns comandos para exibir a planilha são “case-sensitive” (ignore.case(object))
7.8.1 Outra forma de achar e importar uma planilha
Essa forma é desaconselhavel porque é demorada e sujeita a erros. Além de precisar ser refeita sempre que se quiser abrir uma nova planilha ou reabrir a última planilha importada. Veja o tópico Prefira sempre códigos e scripts do que mouse e menus de janelas no R
7.9 Importando .ods do LibreOffice Calc
A planilha a seguir pode ser encontrada AQUI
library(readODS)
rebio23.ods <- read_ods("D:/Elvio/OneDrive/Disciplinas/_EcoNumerica/5.Matrizes/rebio23-zoopl_5-20.ods",
row_names = TRUE,
col_names = TRUE,
sheet = "densidade_hab_indL",
as_tibble = FALSE,
na = "n/a") # porque existem celulas vazias (n/a)
rebio23.ods <- na.omit(rebio23.ods)
str(rebio23.ods)
class(rebio23.ods)
rebio23.ods_ma <- as.matrix(rebio23.ods) #lê como uma matriz
str(rebio23.ods_ma)
class(rebio23.ods_ma)
#rebio23.ods
#rebio23.ods_ma7.9.1 Colunas com o mesmo nome
Em algumas situações é necessário encontrar colunas com o mesmo nome em um conjunto de dados e soma-las ou fazer sua média. Isso pode ocorrer quando se está trabalhando com dados de várias fontes e é necessário combinar esses diferentes conjuntos de dados.
Considere por exemplo um cenário onde se está trabalhando em um projeto de análise de dados ecológicos e você recebeu conjuntos de dados de diferentes locais de amostragens enviados por diferentes pesquisadores, e cada pesquiador enviou seu conjunto de dados que contém informações sobre as mesmas espécies (ou variáveis ambientais). Devido a diferentes sistemas de registro ou a falta de comunicação entre os pesquisadores, pode haver repetição nos nomes das colunas.
#Dados de mamíferos roedores
df <- data.frame(
Rato = c(1, 2, 3),
Musaranho = c(4, 5, 6),
Rato = c(7, 8, 9), #nome duplicado
Esquilo = c(0, 0, 1),
Ratão = c(1, 1, 0),
Castor = c(1, 0, 0),
Tâmia = c(11, 12, 13),
Marmota = c(1, 2, 0),
Castor = c(2, 1, 1), #nome duplicado
check.names = FALSE
)
df## Rato Musaranho Rato Esquilo Ratão Castor Tâmia Marmota Castor
## 1 1 4 7 0 1 1 11 1 2
## 2 2 5 8 0 1 0 12 2 1
## 3 3 6 9 1 0 0 13 0 1Por exemplo, no estudo sobre mamíferos roedores acima, quando se combina o conjunto geral de dados para realizar uma análise abrangente, depara-se com colunas duplicadas, onde a matriz com o conjunto total de dados contém espécies repetidas.
Nesse caso, encontrar e resolver colunas com o mesmo nome é crucial para garantir a integridade dos dados e realizar uma análise precisa. Você deve consolidar essas colunas duplicadas, somando-as ou fazendo sua média.
# Achando colunas com nomes duplicados
dup_cols <- names(df)[duplicated(names(df))]
# Somando colunas com o mesmo nome
for (col_name in unique(dup_cols)) {
# Get indices of columns with the same name
col_indices <- which(names(df) == col_name)
# Sum columns with the same name
df[[col_name]] <- rowSums(df[, col_indices, drop = FALSE])
}
# Remove as colunas duplicadas originais e mantem as novas colunas que são a soma ("except for the first occurrence")
df <- df[, !duplicated(names(df))]
# Mostra a nova tabela com colunas repetidas somadas
print(df)## Rato Musaranho Esquilo Ratão Castor Tâmia Marmota
## 1 8 4 0 1 3 11 1
## 2 10 5 0 1 1 12 2
## 3 12 6 1 0 1 13 07.9.3 Criando uma matriz de médias
Por razões diferentes o precedimento anterior pode vir a ser necessário de ser aplicado às linhas. Por exemplo, quando se quer somar ou fazer a média de amostras diferentes do mesmo ambiente de coleta. Veja a matriz abaixo.
data <- read.table(text = "
Sp1 Sp2 Sp3 Sp4 Sp5 Sp6 Sp7 Sp8
A1 0 0 0 0 0 0 6 1
A2 0 0 0 2 0 0 10 2
B1 93 2 0 177 0 260 2 5
B2 0 4 0 8 0 0 83 7
C1 0 0 0 0 1 0 0 1
C2 0 0 1 0 0 1 0 1
C3 0 2 0 2 0 0 0 1
", header = TRUE, row.names = 1)
data## Sp1 Sp2 Sp3 Sp4 Sp5 Sp6 Sp7 Sp8
## A1 0 0 0 0 0 0 6 1
## A2 0 0 0 2 0 0 10 2
## B1 93 2 0 177 0 260 2 5
## B2 0 4 0 8 0 0 83 7
## C1 0 0 0 0 1 0 0 1
## C2 0 0 1 0 0 1 0 1
## C3 0 2 0 2 0 0 0 1library("tidyverse")
#Inserindo coluna para agrupamentos
nrow(data); ncol(data) #no. de N colunas x M linhas
data_g <- cbind(Grupos = rownames(data), data)
data_g
grps <- substr(data_g[, 1], 1,3)
grps
data_g <- data_g %>% mutate(Grupos=c(grps))
#data_avg <- aggregate(data_g[, 9:9], list(data_g$Grupos), mean)
#data_avg
data_avg <- data_g %>%
group_by(Grupos) %>%
summarise(across(.cols = everything(), ~ mean(.x, na.rm = TRUE)))
data_avg
data_dp <- data_g %>%
group_by(Grupos) %>%
summarise(across(.cols = everything(), list(mean = mean, sd = sd)))
#?across
data_dp
#Primeira coluna para nomes das linhas
data_dp <- as.data.frame(data_dp)
class(data_dp)
rownames(data_dp) <- data_dp[,1]
data_dp[,1] <- NULL
data_dp <- round(data_dp, 1)
data_dp
#Salvando a matriz
write.table(data_dp,
"data_dp.csv",
append = F,
quote = TRUE,
sep = ";", dec = ",",
row.names = T)
data_dp_csv <- read.csv("data_dp.csv",
sep = ";", dec = ",",
header = T,
row.names = 1,
na.strings = NA)Apêndices
Sites consultados
[https://youtu.be/U6ksXvvY6Q0]
[https://youtu.be/a7EJE_2mtGk]
Script limpo
Aqui apresento o scrip na íntegra sem os textos ou outros comentários. Você pode copiar e colar no R para executa-lo. Lembre de remover os # ou ## caso necessite executar essas linhas.
## install.packages("readxl") #importa arquivos do excel
## library(readxl)
## getwd()
## setwd("C:/Seu/Diretório/De/Trabalho")
library(readxl)
ppbio06 <- read_excel("D:/Elvio/OneDrive/Disciplinas/_EcoNumerica/5.Matrizes/ppbio06.xlsx",
sheet = "Sheet1",
na = "NA")
str(ppbio06)
class(ppbio06)
ppbio06 <- as.data.frame(ppbio06)
class(ppbio06)
rownames(ppbio06) <- ppbio06[,1] #tem que ser um df
ppbio06[,1] <- NULL
#install.packages("openxlsx")
library(openxlsx)
ppbio <- read.xlsx("D:/Elvio/OneDrive/Disciplinas/_EcoNumerica/5.Matrizes/bentos06.xlsx",
rowNames = T,
colNames = T,
sheet = "contagem")
str(ppbio)
class(ppbio)
ppbio_ma <- as.matrix(ppbio) #lê ppbio como uma matriz
str(ppbio_ma)
class(ppbio_ma)
#ppbio
#ppbio_ma
## write.table(ppbio, "ppbiocsv.txt", append = F, quote = T, ";", row.names = T)
## dir <- getwd()
## shell.exec(dir) #abre o diretorio de trabalho no Windows Explorer
## ppbiocsv <- read.csv("ppbiocsv.txt",
## sep = ";", dec = ",", #definimos o dígito separador
## header = T,
## row.names = 1,
## na.strings = NA)
## str(ppbiocsv)
## ppbiocsv
## #View(ppbio)
## print(ppbio)
## ppbio
## str(ppbio)
## #?View
## #?view
## #?remove
## getwd()
## ppbio <- read.xlsx(file.choose(), #abre o windows explorer
## rowNames = T, colNames = T,
## sheet = "Sheet1")