12 Pruebas de hipótesis para variables cuantitativas
- Autor: Edsaúl Emilio Pérez Guerrero
- Afiliación: Universidad de Guadalajara
- mail: edsaul.perezg@academicos.udg.mx
- Última actualización: 2024-09-11
En este capítulo se revisarán las pruebas estadísticas básicas para la comparación de variables de tipo cuantitivo entre dos o más grupos, a saber:
- Prueba t para variables independientes
- Prueba t para variables dependientes
- ANOVA de una vía
- Prueba U-Mann-Whitney
- Prueba de los rangos de Wilcoxon
- Prueba de Kruskall-Wallis
12.1 Prueba t y distritución t
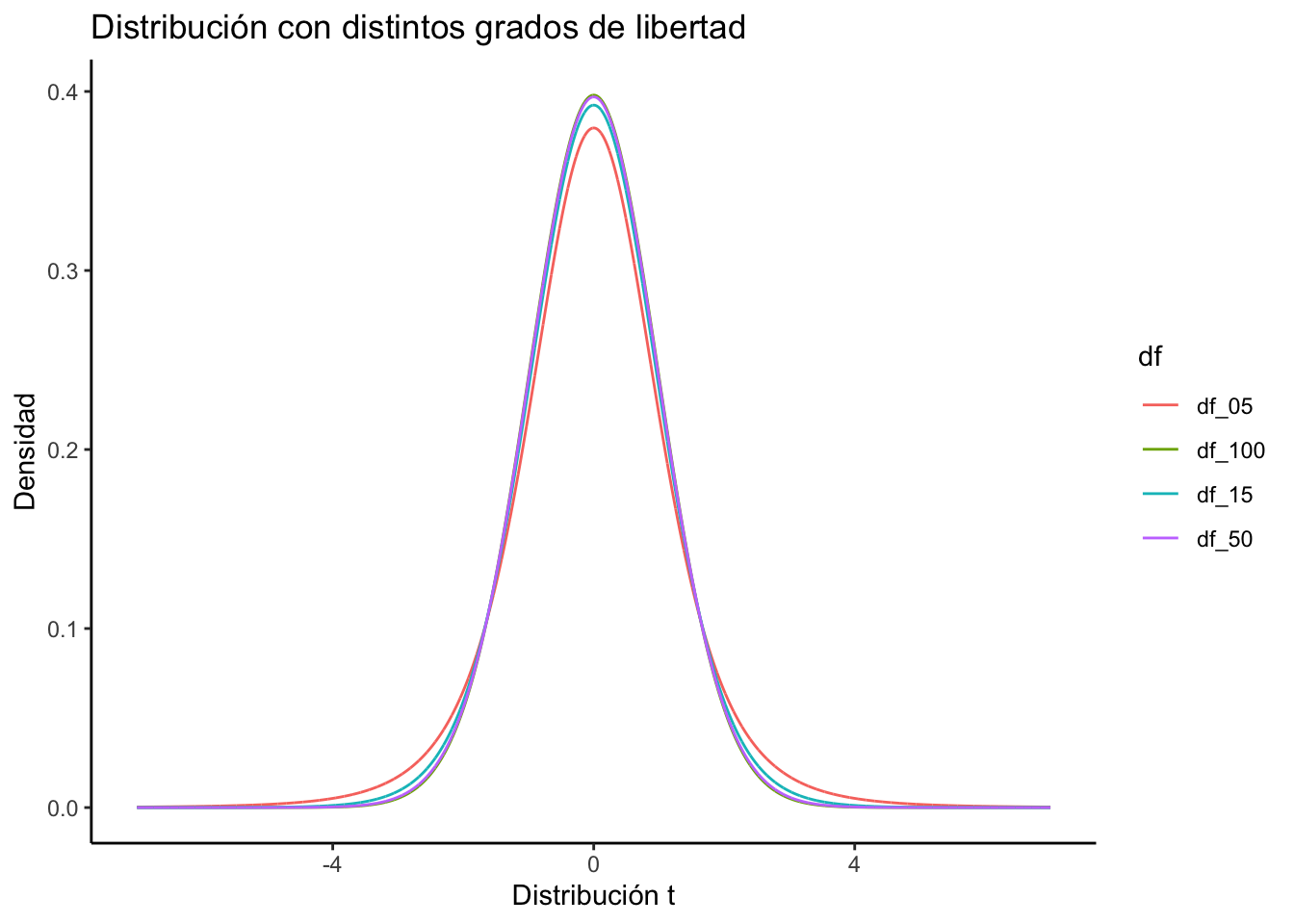
La prueba t de student se basa en la distribución t, que presenta las siguientes características:
- Tiene media de 0.
- Es simétrica con respecto a la media.
- Varianza mayor que 1. Aunque tiene a 1 cuando la \(n\) aumenta.
- La variable \(t\) va de.
- Es una familia de distribuciones. Hay una distribución para cada valor de \(n\).
- Es menos espigada y mas alargada que la distribución normal.
- Se aproxima a la distribución normal a medida que \(n-1\) se aproxima al infinito.
En la Figura 12.1 se muestran distintas distribuciones t de acuerdo con sus grados de libertad.

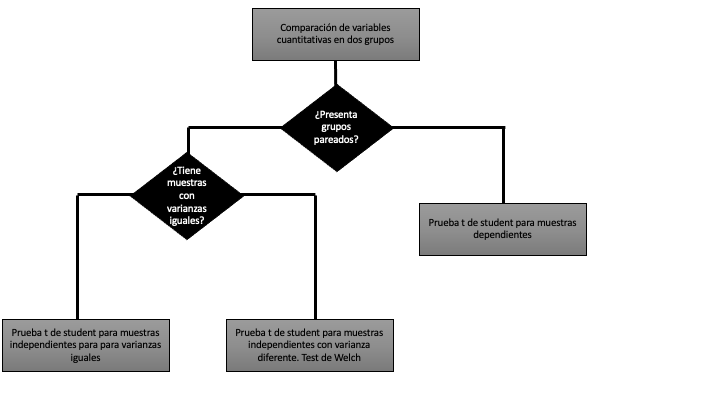
Dentro de las pruebas t, existen diferente variantes, cada una de ella para situaciones en particular. Por ejemplo, existe una prueba t para grupos pareados, otra para grupos independientes etc. La Figura 12.2 muestra un resumen de las diferentes pruebas t.
12.2 Prueba t student para muestras independientes
La prueba t de student para muestras independientes se utiliza para la comparación de medias de grupos independientes. En general las hipótesis que se pueden plantear son:
| \(H_O\) | \(H_A\) | Tipo de hipótesis | |
|---|---|---|---|
| 1 | \(\mu_1- \mu_2 =0\) | \(\mu_1- \mu_2 \neq0\) | Bilateral |
| 2 | \(\mu_1- \mu_2 >=0\) | \(\mu_1- \mu_2 \ <0\) | Unilateral |
| 3 | \(\mu_1- \mu_2 <=0\) | \(\mu_1- \mu_2 \ >0\) | Unilateral |
En R podemos utilizar la función t.test() que posee los siguientes argumentos:
X: un vector numérico (no vacío) de valores de datos.
y: un vector numérico opcional (no vacío) de valores de datos.
alternative: una cadena de caracteres que especifica el tipo de hipótesis. Las opciones a saber son: “two.sided” (default) para una hipótesis bilateral, “greater” cuando se quiere probar si la media 1 es mayor que la media 2 o “less” (lo contrario a greater). Puede especificar solo la letra inicial.
paired: una indicación lógica si desea una prueba t pareada.
var.equal: una variable lógica que indica si se deben tratar las dos varianzas como si fueran iguales. Si es VERDADERO, la varianza agrupada se usa para estimar la varianza; de lo contrario, se usa la aproximación de Welch (o Satterthwaite) a los grados de libertad.
conf.level: nivel de confianza del intervalo.
formula: se utiliza para introducir los datos a manera de formula.
data: Argumento que se utilza para identificar el data frame del que provienen los datos. Si se usó la función
attach()o el simbolo de$no es necesario utilizar este argumento.
En R la función t.test se puede utilizar de dos formas:
- Utilizando “x” y “y”: t.test(x=objeto1, y=objeto2)
- Utilizando la formula: t.test(Variable de prueba~Variable agrupación)
La primera opción se utiliza cuando las variables a comparar se encuentran en columnas separadas, mientras que la segunda, se utiliza cuando mi variable cuantitativa se encuentra en un columna para ambos grupos, y mi variable de agrupación (factor) en otra columna.
Vamos a resolver un ejempl.
Ejemplo 12.1 Un estudio de los investigadores Eidelman et al.tiene como objetivo examinar las características de destrucción pulmonar en personas que fuman cigarros antes de desarrollar un marcado enfisema pulmonar. Se practicaron mediciones de tres indices de destrucción pulmonar en los pulmones de personas longevas que no fumaban y en personas con tabaquismo que murieron repentinamente fuera del hospital por causas no respiratorias. Una calificación alta indica un mayor daño pulmonar. Los datos fueron guardados en dos objetos para uno de los índices de destrucción pulmonar de una muestra de 9 personas que no fuman y 16 fumadores. Se pretende saber si es posible concluir, con base en los datos, que las personas que sí fuman, en general, tienen los pulmones mas dañados que las personas no fumadoras, como lo indican las mediciones. No se conocen las variancias poblacionales, pero se supone que son iguales. Utilice un nivel de confianza del 98%. (ejercio obtenido del libro de Bioestadística de Daniels)
Para la resolución de este ejercicio vamos a crear dos objetos, uno para la calificación de los fumadores y otro para la calificación de los no fumadores:
Calif_No_Fumadores<-c(18.1,6.0,10.8,11.0,7.7,17.9,8.5,13.0,18.9)
Calif_Fumadores<- c(16.6,13.9,11.3,26.5, 17.4, 15.3, 15.8, 12.3,
18.6, 12.0, 24.1, 16.5, 21.8, 16.3, 23.4,
18.8)Lo primero es graficar, podemos hacerlo mediante un boxplot. Más adelante utilizaremos la función plotmeans()
boxplot(Calif_Fumadores, Calif_No_Fumadores, ylab="Calificaciones",
main="Calificación de daño pulmonar", col=paleta,
names = c("Fumadores", "No fumadores"), frame = FALSE) 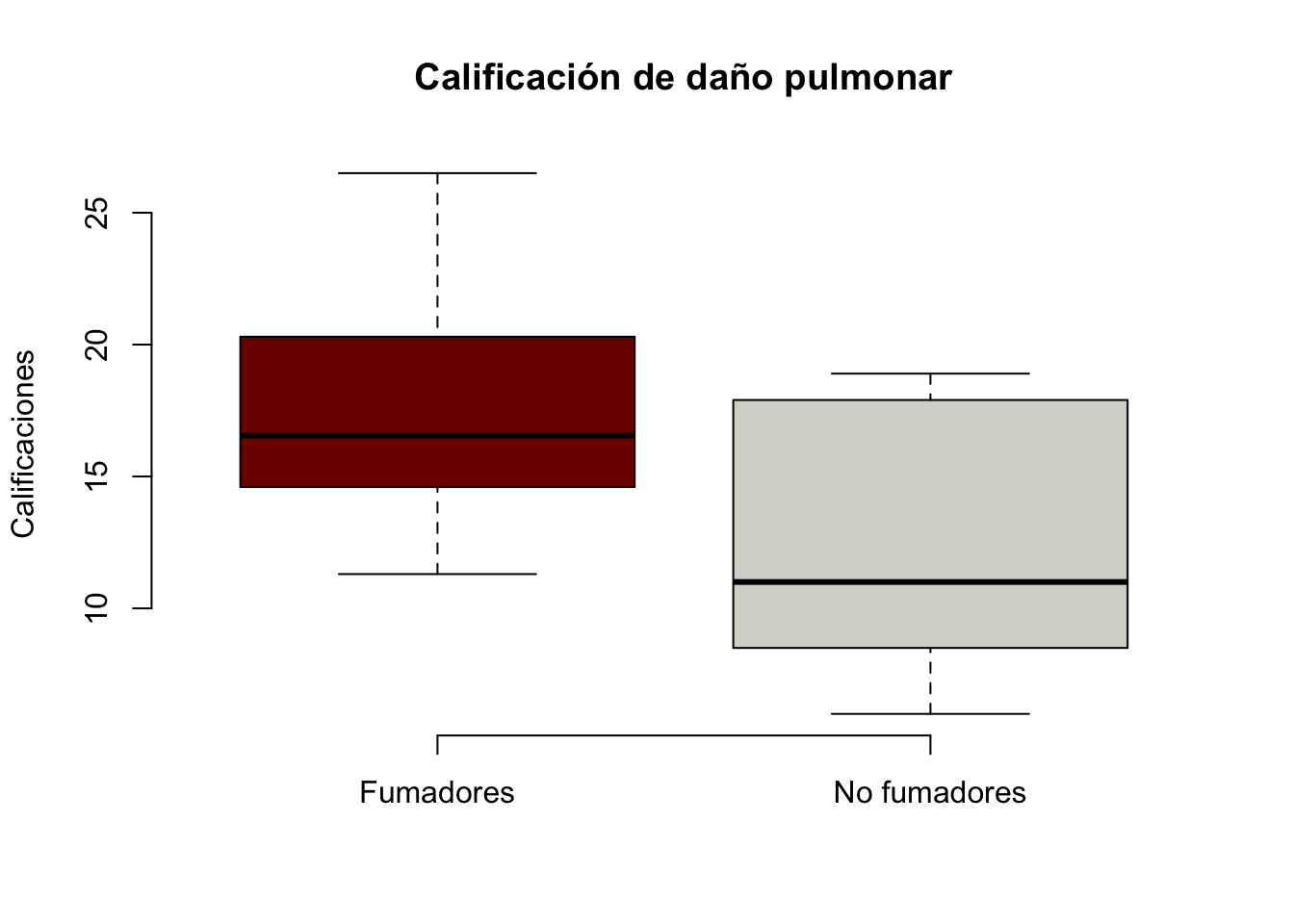
También podemos hacer un gráfico de violín:
vioplot::vioplot(Calif_Fumadores, Calif_No_Fumadores, ylab="Calificaciones",
main="Calificación de daño pulmonar", col=paleta,
names = c("Fumadores", "No fumadores")) 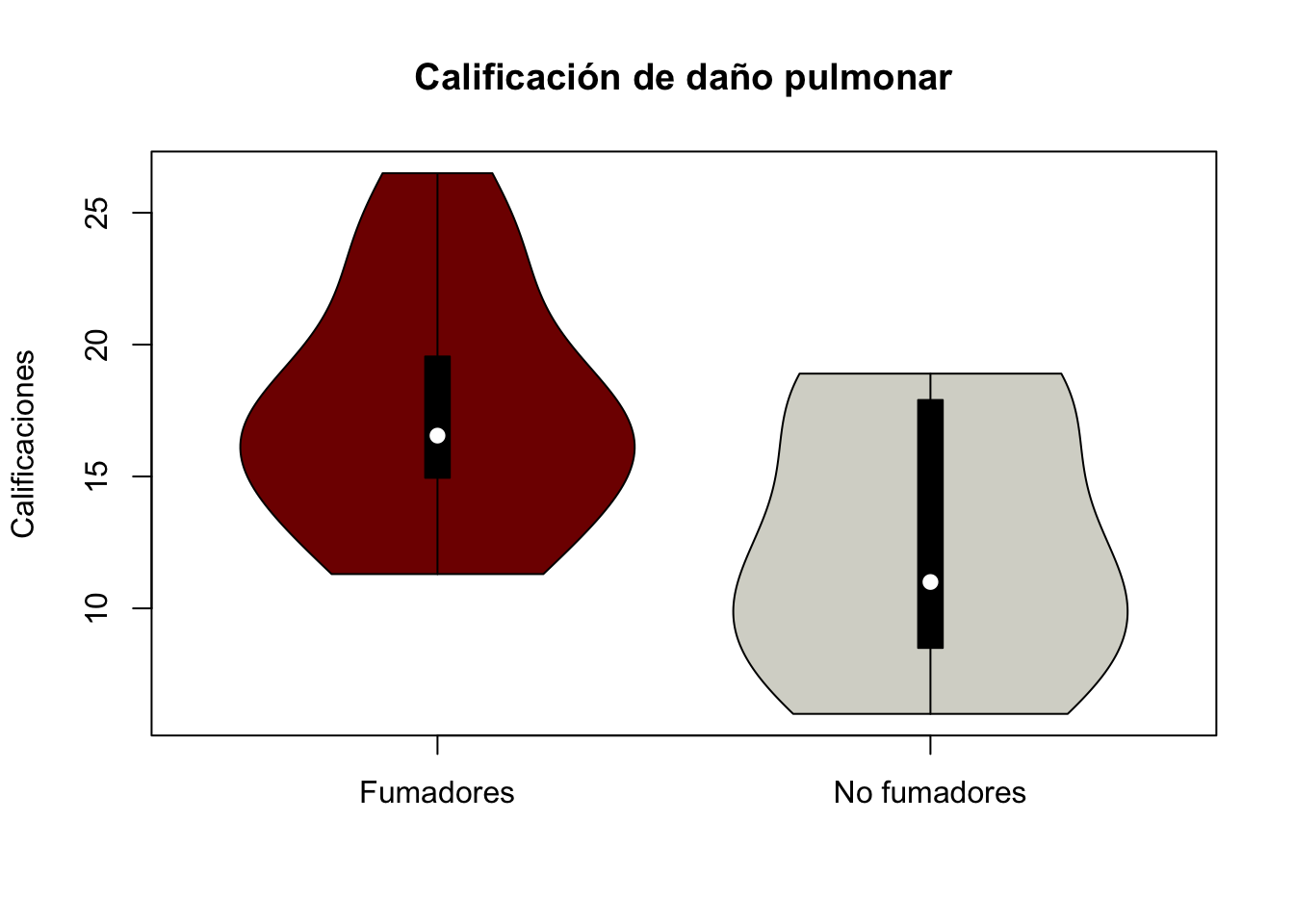
La gráfica anterior parece indicar que la calificación en el grupo de fumadores es mayor que en el grupo de los no fumadores. Vamos a comprobarlo mediante la prueba t de student.
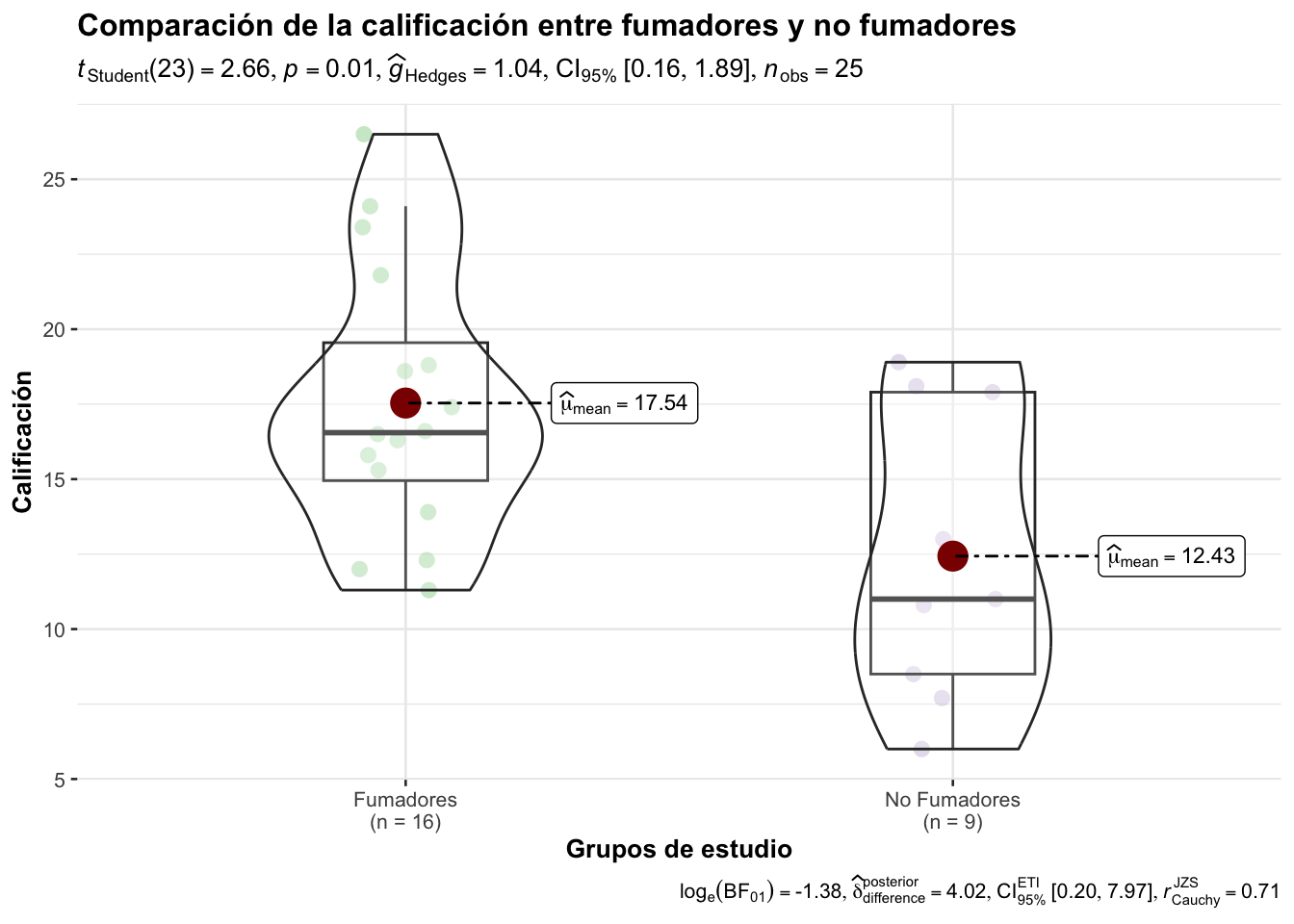
t.test(Calif_Fumadores, Calif_No_Fumadores, alternative = "greater",
conf.level = 0.98, var.equal = T)
Two Sample t-test
data: Calif_Fumadores and Calif_No_Fumadores
t = 2.658, df = 23, p-value = 0.007027
alternative hypothesis: true difference in means is greater than 0
98 percent confidence interval:
0.9237087 Inf
sample estimates:
mean of x mean of y
17.53750 12.43333 Utilizamos el argumento alternative="greater" ya que nos interesa probar que la calificación de los fumadores es mayor que los no fumadores (la media del grupo es mayor que la media del grupo 2). Además, utilizamos un nivel de confianza del 98%. De momento asumimos que ambos grupos tienen varianzas iguales.
Supongamos ahora que es nuestro interés conocer que simplemente si las medias son diferentes y cambiamos el argumento alternative="two.sided".
t.test(Calif_Fumadores, Calif_No_Fumadores, alternative = "two.sided",
conf.level = 0.98, var.equal = T)
Two Sample t-test
data: Calif_Fumadores and Calif_No_Fumadores
t = 2.658, df = 23, p-value = 0.01405
alternative hypothesis: true difference in means is not equal to 0
98 percent confidence interval:
0.3036205 9.9047128
sample estimates:
mean of x mean of y
17.53750 12.43333 Aunque podría omitir el argumento alternative="two.sided" ya que por se encuentra predefinido. El siguiente código dará el mismo resultado:
t.test(Calif_Fumadores, Calif_No_Fumadores,
conf.level = 0.98, var.equal = T)
Two Sample t-test
data: Calif_Fumadores and Calif_No_Fumadores
t = 2.658, df = 23, p-value = 0.01405
alternative hypothesis: true difference in means is not equal to 0
98 percent confidence interval:
0.3036205 9.9047128
sample estimates:
mean of x mean of y
17.53750 12.43333 Otra opción para utilizar la función t.test() es mediante el uso de formula. Para ello, vamos a importar la base de datos Calif_Fum utilizando el menú de RStudio. Asegúrese de que su objeto fue nombrado como Calif_Fum.
Ahora ya podemos utilizar la función t.test() como formula:
t.test(Calif_Fum$Calificacion~Calif_Fum$Grupo, conf.level = 0.98, var.equal = T)
Two Sample t-test
data: Calif_Fum$Calificacion by Calif_Fum$Grupo
t = 2.658, df = 23, p-value = 0.01405
alternative hypothesis: true difference in means between group Fumadores and group No Fumadores is not equal to 0
98 percent confidence interval:
0.3036205 9.9047128
sample estimates:
mean in group Fumadores mean in group No Fumadores
17.53750 12.43333 También ahora ya podemos hacer un gráficos de medias, si es necesario instale la librería gplots
library(gplots)
Attaching package: 'gplots'The following object is masked from 'package:stats':
lowessplotmeans(Calif_Fum$Calificacion~Calif_Fum$Grupo, xlab="Grupos de estudio", ylab="Calificación")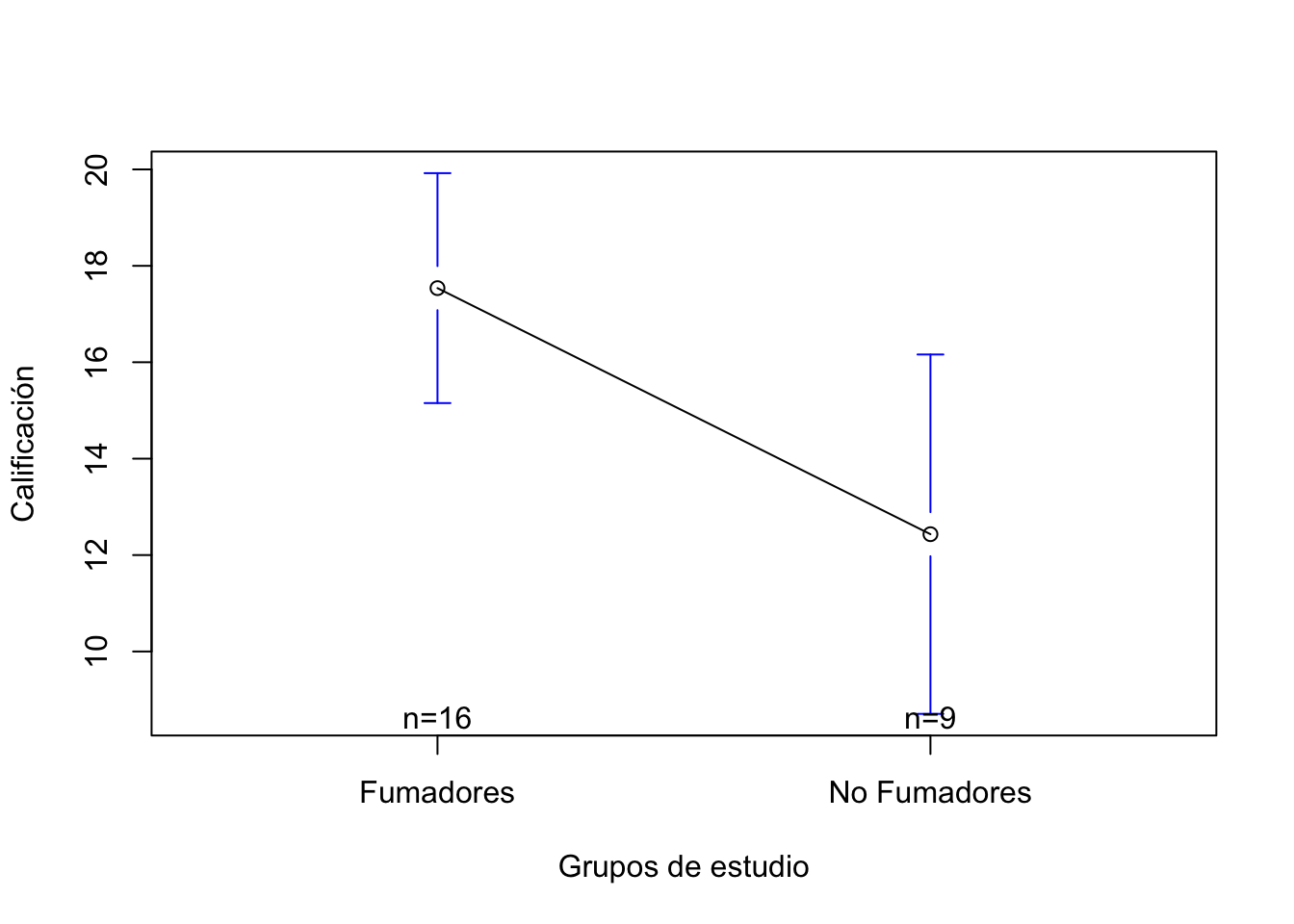
Al final del capitulo hay una explicación más profunda de cómo utilizar la librería ggstastplot para resolver estos ejercicios. En este apartado solo se muestra el código.
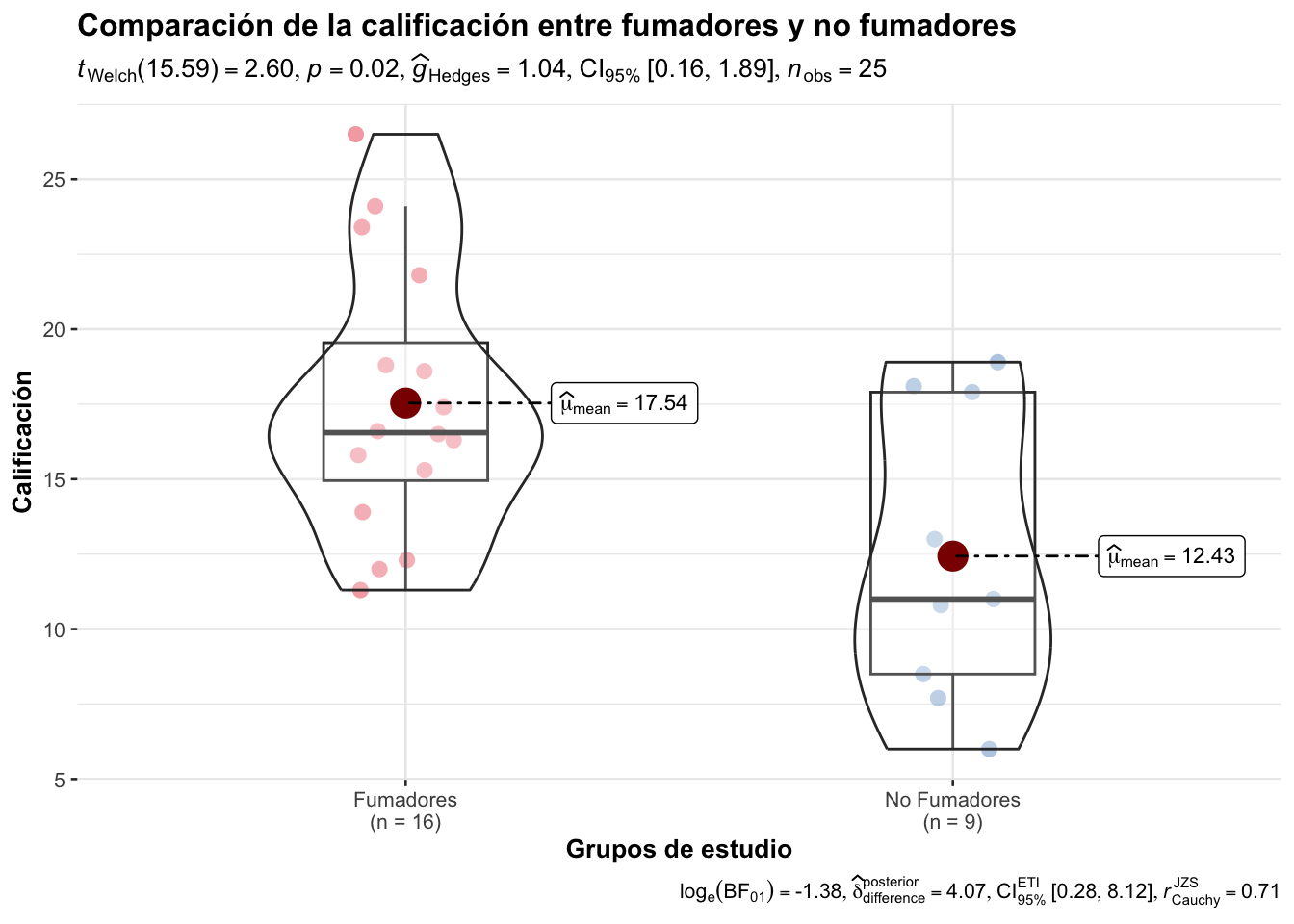
ggstatsplot::ggbetweenstats(
data=Calif_Fum, #Origen de los datos
x=Grupo, # Variuables de agrupación
y=Calificacion, # Variable de respuesta, númericas
plot.type = "box", # Tipo de gráfico, otras opciones son: "violin", "boxviolin"
type= "parametric", # Paramétrica para t student
xlab="Grupos de estudio", # Titulo de las x
ylab = "Calificación", # Título de la Y
title = "Comparación de la calificación entre fumadores y no fumadores",
subtitle = "Ejemplo utilizando librería ggstatsplot",
var.equal = F, # Argumento lógico para indicar varianzas iguales
palette = "Set1" # Paleta de la librería RColorBrewer
)
12.2.1 Test de welch
En estadística, la prueba t de Welch, o prueba t de varianzas desiguales, es una prueba de ubicación de dos muestras que se utiliza para probar la hipótesis de que dos poblaciones tienen medias iguales. Lleva el nombre de su creador, Bernard Lewis Welch, y es una adaptación de la prueba t de Student, y es más confiable cuando las dos muestras tienen varianzas desiguales y/o tamaños de muestra desiguales.
Ejemplo 12.2 Para evaluar la igualdad de varianzas existen varias opciones, sin embargo, en este curso utilizaremos el test de barttlet mediante la función bartlett.test() también se puede utilizar la función car::leveneTest().
Utilizamos la base Calif_Fum.
bartlett.test(Calif_Fum$Calificacion~Calif_Fum$Grupo)
Bartlett test of homogeneity of variances
data: Calif_Fum$Calificacion by Calif_Fum$Grupo
Bartlett's K-squared = 0.065041, df = 1, p-value = 0.7987Note como es necesario introducir los datos como formula. La prueba nos dice que las varianzas son iguales entre los grupos.
A pesar del resultado, asumamos que las varianzas no son iguales para ejemplificar como realizar el test de welch cambiando el argumento var.equal = F
t.test(Calif_Fum$Calificacion~Calif_Fum$Grupo, conf.level = 0.98, var.equal = F)
Welch Two Sample t-test
data: Calif_Fum$Calificacion by Calif_Fum$Grupo
t = 2.5964, df = 15.593, p-value = 0.01978
alternative hypothesis: true difference in means between group Fumadores and group No Fumadores is not equal to 0
98 percent confidence interval:
0.01088222 10.19745111
sample estimates:
mean in group Fumadores mean in group No Fumadores
17.53750 12.43333 Otra manera de evaluar la homogenidad de varianzas es utilizar la prueba de levene con la función leveneTest de la librería car
car::leveneTest(Calif_Fum$Calificacion~Calif_Fum$Grupo)Warning in leveneTest.default(y = y, group = group, ...): group coerced to
factor.Levene's Test for Homogeneity of Variance (center = median)
Df F value Pr(>F)
group 1 0.1704 0.6836
23 Utilizando ggstastplot el código sería:
ggstatsplot::ggbetweenstats(
data=Calif_Fum, #Origen de los datos
x=Grupo, # Variuables de agrupación
y=Calificacion, # Variable de respuesta, númericas
plot.type = "box", # Tipo de gráfico, otras opciones son: "violin", "boxviolin"
type= "parametric", # Paramétrica para t student
xlab="Grupos de estudio", # Titulo de las x
ylab = "Calificación", # Título de la Y
title = "Comparación de la calificación entre fumadores y no fumadores",
subtitle = "Ejemplo utilizando librería ggstatsplot",
var.equal = T, # Argumento lógico para indicar varianzas iguales
palette = "Accent" # Paleta de la librería RColorBrewer
)
12.2.2 Ejercicios para la prueba t de student para muestras independientes:
Ejercicio 12.1 Utilizando la base de datos “Pima.tr” describa si hay diferencias en los niveles de glucosa, la presión arterial, en sikin, índice de masa corporal, en ped y edad entre las mujeres con diabetes y las mujeres sin diabetes. Para este problema utilice únicamente la función t.test de R no realice la prueba de hipótesis por pasos. Para cada una de las variables entregue los siguiente:
- Realice un boxplot para cada variable agrupadando entre las mujeres con diabetes y sin diabetes
- Realice un gráfico de violín para cada variable agrupando entre mujeres con diabetes y sin diabetes
- Realice un gráfico de
plotmeanspara cada variable agrupando entre mujeres con diabetes y sin diabetes - Resultado de la prueba \(t-student\)
- Conclusión
Ejercicio 12.2 Utilizando la base de datos “Pima.tr” describa si hay diferencias en los niveles de glucosa, la presión arterial entre las mujeres con más de 40 años y las que mujeres con 40 años o menos.
Ejercicio 12.3 Compruebe las hipótesis que puedan ser comprobadas mediante una prueba t generadas en el EDA de Melanoma. Asuma normalidad de los datos.
Ejercicio 12.4 Compruebe las hipótesis que puedan ser comprobadas mediante una prueba t generadas en el EDA de birthwt. Asuma normalidad de los datos.
Ejercicio 12.5 Compruebe las hipótesis que puedan ser comprobadas mediante una prueba t generadas en el EDA de biopsy Asuma normalidad de los datos.
12.3 Prueba t para variables dependientes
Un método que se utiliza con frecuencia para averiguar la eficacia de un tratamiento o procedimiento experimental es aquel que hace uso de observaciones relacionadas que resultan de muestras no independientes.
- Mediciones a través del tiempo
- Dos mediciones en el mismo sujeto
- Diseño cruzados
En estos casos la prueba de hipótesis más adecuada es la prueba t de student para muestras pareadas. Dado que se trabaja con la diferencia de las medias, no es necesario realizar prueba de homogeneidad de varainzas. !Es la misma muestra!
Vamos a resolver un ejemplo
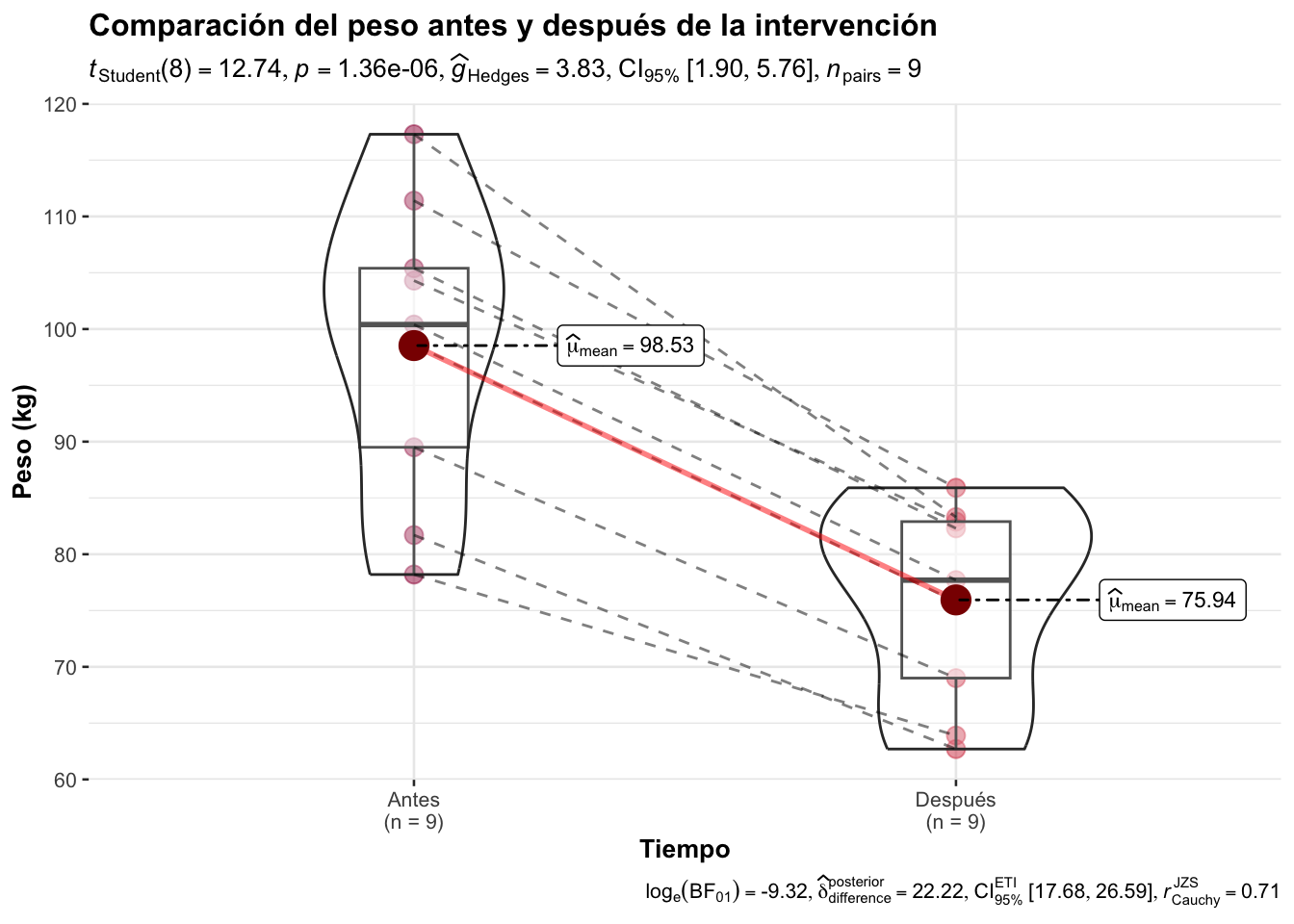
Ejemplo 12.3 Nancy Stearns Burgess condujo un estudio para determinar la perdida de peso, la composición corporal, la distribución de grasa corporal y la tasa metabólica en reposo en individuos obesos antes y después de 12 semanas de tratamiento con dieta muy baja en calorías (DMBC), y comparar la hidrodensitometría con el análisis de impedancia bioeléctrica. Los 17 individuos (nueve mujeres y ocho hombres) que participaron en el estudio eran pacientes externos de un programa de tratamiento con base hospitalaria para la obesidad. Los pesos de las mujeres antes y después del tratamiento de 12 semanas de DMBC se muestran en dos objetos. Se pretende saber si estos datos ofrecen suficiente evidencia que permita concluir que el tratamiento es eficaz para reducir el peso en mujeres obesas.
Se crean dos objetos para ello:
antes<-c(117.3, 111.4,98.6,104.3,105.4,100.4, 81.7,89.5,78.2)
despues<-c(83.3,85.9,75.8,82.9,82.3,77.7,62.7,69.0,63.9)Tratamos de probar si:
- Lo que se busca es saber si existe la suficiente evidencia para concluir que el programa de dietas es eficaz.
- Si es posible rechazar la hipótesis nula que indica que el cambio en la media de la población \(\mu_d\) es cero o positivo.
Podemos graficar, el siguiente código tiene objetivo mostrar todos los argumentos de la función boxplot, usted puede copiar y pegar o hacer un gráfico más sencillo:
boxplot(antes, despues, # Datos
horizontal = FALSE, # Horizontal or vertical plot
lwd = 2, # Lines width
col = c("#76EEC6", "#FFE4C4"), # Color
xlab = "Grupos", # X-axis label
ylab = "Peso", # Y-axis label
main = "Comparación del peso antes y después de la intervención", # Title
border = "black", # Boxplot border color
outpch = 25, # Outliers symbol
outbg = "green", # Outliers color
whiskcol = "blue", # Whisker color
whisklty = 2, # Whisker line type
names=c("Peso antes", "Peso despues"), #Nombres de los grupos
lty = 1) # Line type (box and median)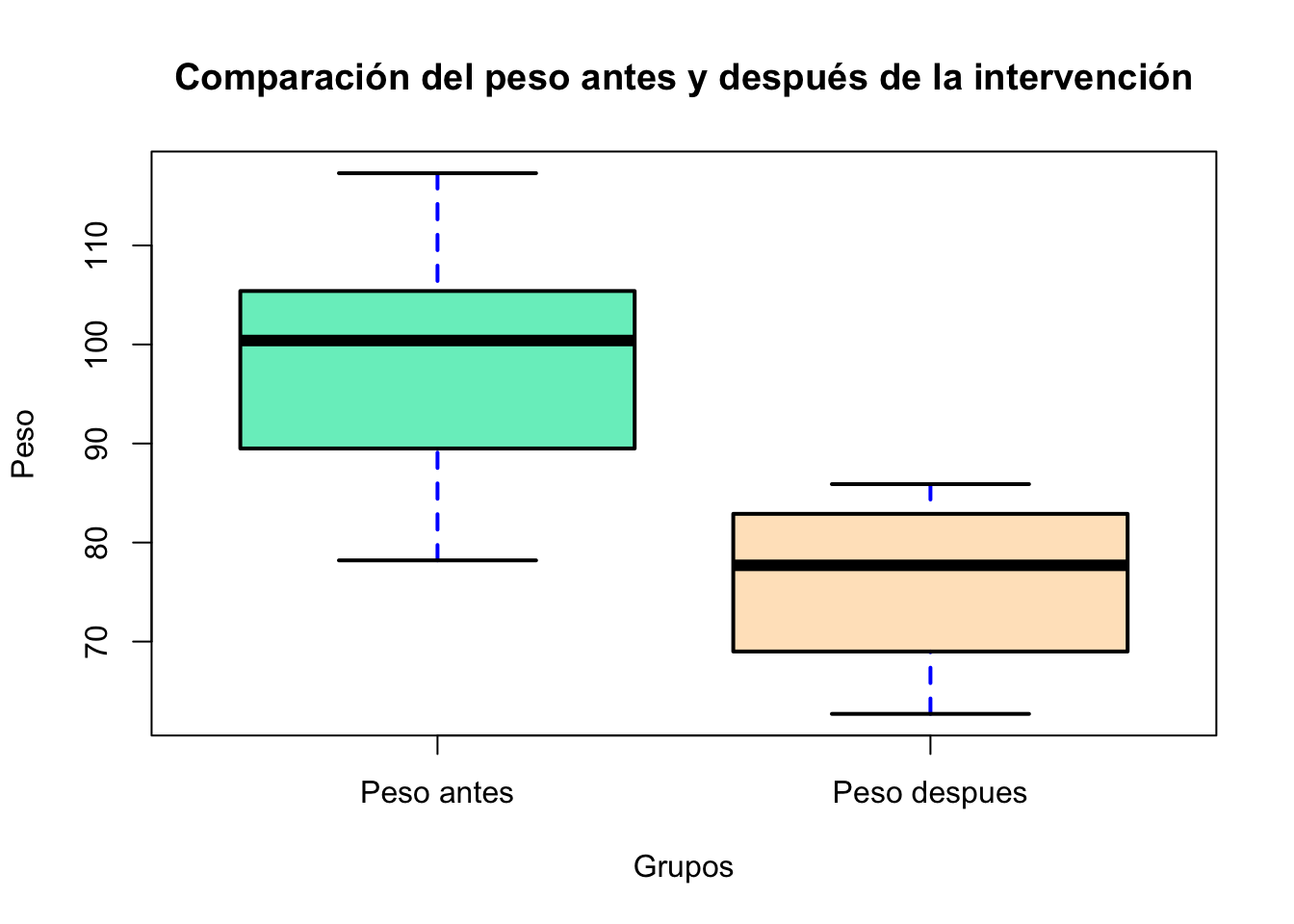
Para realizar la prueba t, puede emplear el siguiente código:
t.test(x=antes, y=despues, alternative = "greater",
paired = T, var.equal = T) # Se tienen que considerar varianzas iguales
Paired t-test
data: antes and despues
t = 12.74, df = 8, p-value = 6.787e-07
alternative hypothesis: true mean difference is greater than 0
95 percent confidence interval:
19.29166 Inf
sample estimates:
mean difference
22.58889 En el caso que nuestra hipótesis sea a dos colas:
t.test(x=antes, y=despues, alternative = "two.sided",
paired=T, var.equal = T)
Paired t-test
data: antes and despues
t = 12.74, df = 8, p-value = 1.357e-06
alternative hypothesis: true mean difference is not equal to 0
95 percent confidence interval:
18.50003 26.67775
sample estimates:
mean difference
22.58889 Note que el único argumento que se debe de cambiar para indicarle a R que la muestra es pareada es: paired=T. Es la misma varianza.
Ahora vamos a crear un data frame con los datos anteriores para utilizar la librería ggstastplot. Mi recomendación es que copie y pegue el código.
# Creación del df
Pesos<-c(antes, despues)
Tiempo <- rep(x=c("Antes", "Después"), times=1, each=9 )
df <- data.frame(Tiempo, Pesos)ggstatsplot::ggwithinstats(
data=df, # El dataframe que contiene los datos a graficar
x=Tiempo, # Nombre de la variable para el eje X
y=Pesos, # Nombre de la variable para el eje Y
type = "parametric", # Tipo de prueba estadística a realizar (paramétrica en este caso)
xlab = "Tiempo", # Etiqueta para el eje X
ylab = "Peso (kg)", # Etiqueta para el eje Y
title = "Comparación del peso antes y después de la intervención", # Título del gráfico
subtitle = "Ejemplo de como utilizar ggstastsplot", # Subtítulo del gráfico
palette = "Spectral" # Paleta de colores
)
12.3.1 Ejercicios prueba t pareada
Utilizando las base de datos “Base_Prueba_t_pareada” que se encuentra adjunta a esta a tarea o que puede descargar de la carpeta de bases de datos, resuelva los ejercicios utilizando la función t.test. Además, realice gráficos de caja y bigotes y concluya en cada uno de los ejercicios.
La “Base_Prueba_t_pareada” es una base de datos que contiene la mediciones de adipocinas en pacientes con insuficiencia hepática. Contiene mediciones de estas adipocinas séricas a distintos tiempos: Basales, 3 meses, 6 y 12 meses. Puede descargar la base de datos aquí
Ejercicio 12.6 ¿Existe evidencia para demostrar que los pacientes con insuficiencia hepática presentaron algún cambio entre las mediciones basales de leptina (Leptin) y las mediciones de leptina a los 12 meses (Leptin12M)?
- Realice los gráficos adecuados
- Realice la pruea de hipótesis pertinente para responder la pregunta
- Responda este ejercicio utilizando la librería `ggstastsplot``
Ejercicio 12.7 Los investigadores asignaron una intervención para reducir las concentraciones de adiponectina en los pacientes con insuficiencia hepática, para ello realizaron mediciones basales de esta adipocina (Adiponectin) y mediciones a los 6 meses (Adiponectin6M) y a los 12 meses (Adiponectin12M). ¿Pueden concluir los investigadores que se presentó una disminución de las concentraciones séricas de adiponectina?
- Realice los gráficos adecuados
- Realice la prueba de hipótesis pertinente para responder la pregunta
- Responda este ejercicio utilizando la librería `ggstastsplot``
Ejercicio 12.8 Los investigadores a cargo del estudio realizaron la medición de un biomarcador que se asocia a una enfermedad más grave en los pacientes con insuficiencia hepática, se ha validado que a mayores concentraciones de este biomarcador se presenta una enfermedad más grave. ¿Existe evidencia para demostrar que los pacientes empeoraron a los 6 meses y a los 12 meses con respecto a la medición basal?. La variable “Biomarcador” tiene las mediciones basales del biomarcador de interés. Mientras que variables “Biomarcador6M” y “Biomarcador12M” las mediciones a los 6 y 12 meses
- Realice los gráficos adecuados
- Realice la pruea de hipótesis pertinente para responder la pregunta
- Responda este ejercicio utilizando la librería `ggstastsplot``
12.4 ANOVA de una vía
La prueba de ANOVA permite la comparación de variables cuantitativas entre más de dos grupos. Se basa en la comparación de las varianzas entre los grupos y dentro de los grupos. La hipótesis nula es que las medias de los grupos son iguales.
Ejemplo 12.4 En R podemos emplear la función aov para realizar pruebas de ANOVA. Vamos a resolver un ejemplo utilizando la base de datos cushings de la librería MASS
El conjunto de datos de Cushings [2], que está disponible en el paquete MASS. El síndrome de Cushing es un trastorno hormonal asociado con un alto nivel de cortisol secretado por la glándula suprarrenal. El conjunto de datos de Cushings incluye 27 observaciones (n = 27). Para cada individuo de la muestra, se registran las tasas de excreción urinaria de dos metabolitos de esteroides. Estos son la tasa de excreción urinaria (mg/24 h) de tetrahidrocortisona y la tasa de excreción urinaria (mg/24 h) de pregnanetriol. La variable Tipo en el conjunto de datos muestra el tipo de síndrome subyacente, que puede ser una de cuatro categorías: adenoma (a), hiperplasia bilateral (b), carcinoma (c) y desconocido (u)
Adjuntar las base de datos y observar los primeros 6 datos
library(MASS)
Attaching package: 'MASS'The following object is masked from 'package:dplyr':
selectdata(Cushings)
attach(Cushings) # Evita el uso de $
head(Cushings) # Primeros 6 datos Tetrahydrocortisone Pregnanetriol Type
a1 3.1 11.70 a
a2 3.0 1.30 a
a3 1.9 0.10 a
a4 3.8 0.04 a
a5 4.1 1.10 a
a6 1.9 0.40 aLos nombres de esta base, son un tanto complicados. Podemos obtener los nombres de un objeto con la función names
names(Cushings)[1] "Tetrahydrocortisone" "Pregnanetriol" "Type" ¿Existe diferencia entre los niveles de tetrahidrocortisona entre los tipos de Cushings?
Vamos a graficar
plotmeans(Tetrahydrocortisone~Type)Warning in arrows(x, li, x, pmax(y - gap, li), col = barcol, lwd = lwd, :
zero-length arrow is of indeterminate angle and so skippedWarning in arrows(x, ui, x, pmin(y + gap, ui), col = barcol, lwd = lwd, :
zero-length arrow is of indeterminate angle and so skipped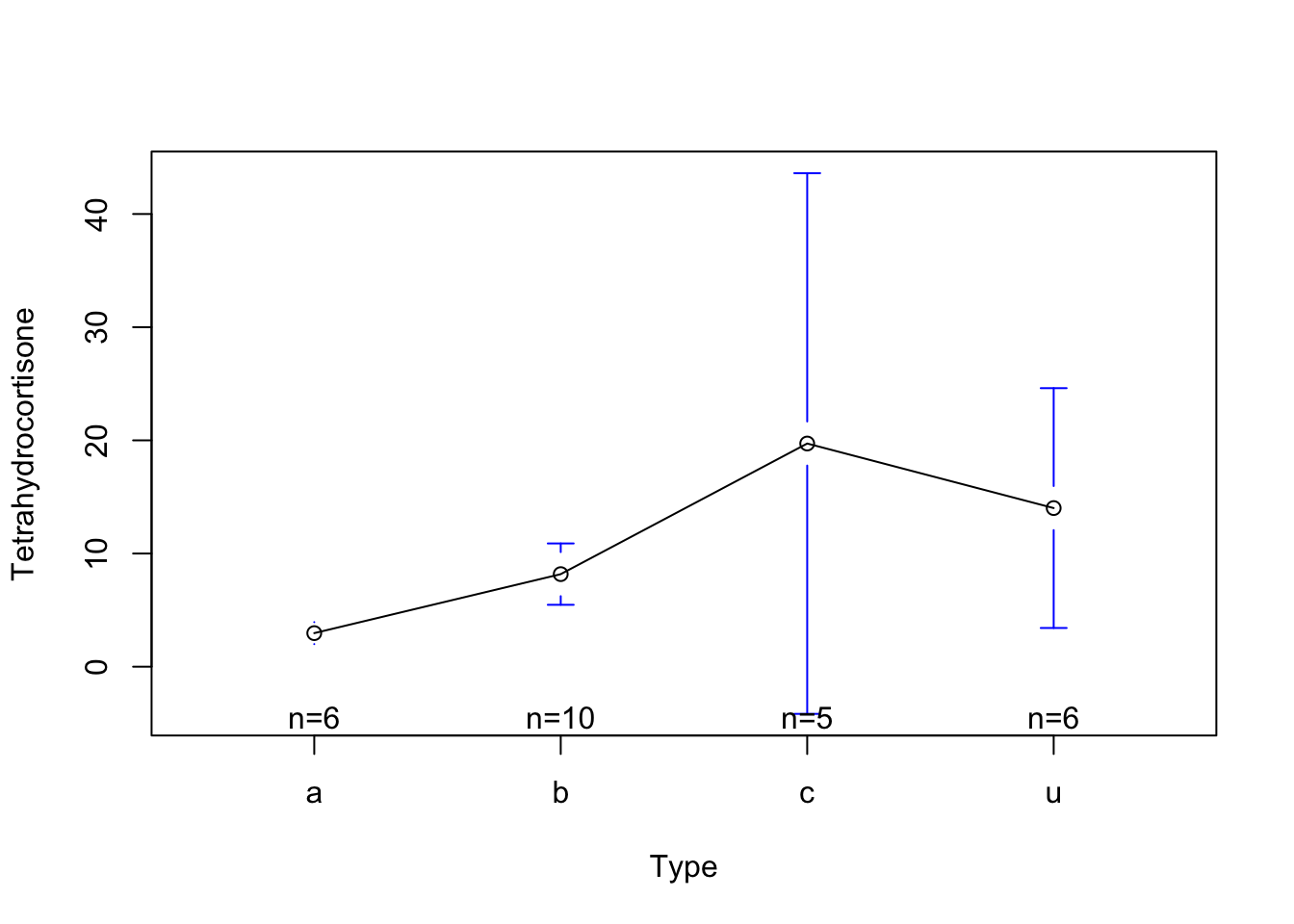
Para realizar el ANOVA se utiliza la función aov.
aov(Tetrahydrocortisone~Type)|>
summary()## Con esto vemos el resultado del ANOVA Df Sum Sq Mean Sq F value Pr(>F)
Type 3 893.5 297.84 3.226 0.0412 *
Residuals 23 2123.6 92.33
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Los resultados anteriores nos indican que si hay diferencia entre los niveles de tetrahidrocortisona entre los tipos de Cushings.
Podemos una prueba posthoc utilizando la función TukeyHSD alimentada por el objeto de nuestro ANOVA.
aov(Tetrahydrocortisone~Type)|>
TukeyHSD()# Para hacer la prueba de tukey, se alimenta con el objeto del ANOVA Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = Tetrahydrocortisone ~ Type)
$Type
diff lwr upr p adj
b-a 5.213333 -8.5181810 18.94485 0.7219961
c-a 16.753333 0.6517055 32.85496 0.0393613
u-a 11.050000 -4.3022998 26.40230 0.2200574
c-b 11.540000 -3.0244704 26.10447 0.1551716
u-b 5.836667 -7.8948477 19.56818 0.6473881
u-c -5.703333 -21.8049612 10.39829 0.7619359Podemos realizar una gráfica de nuestra prueba posthoc con el siguiente código:
aov(Tetrahydrocortisone~Type)|>
TukeyHSD()|>
plot()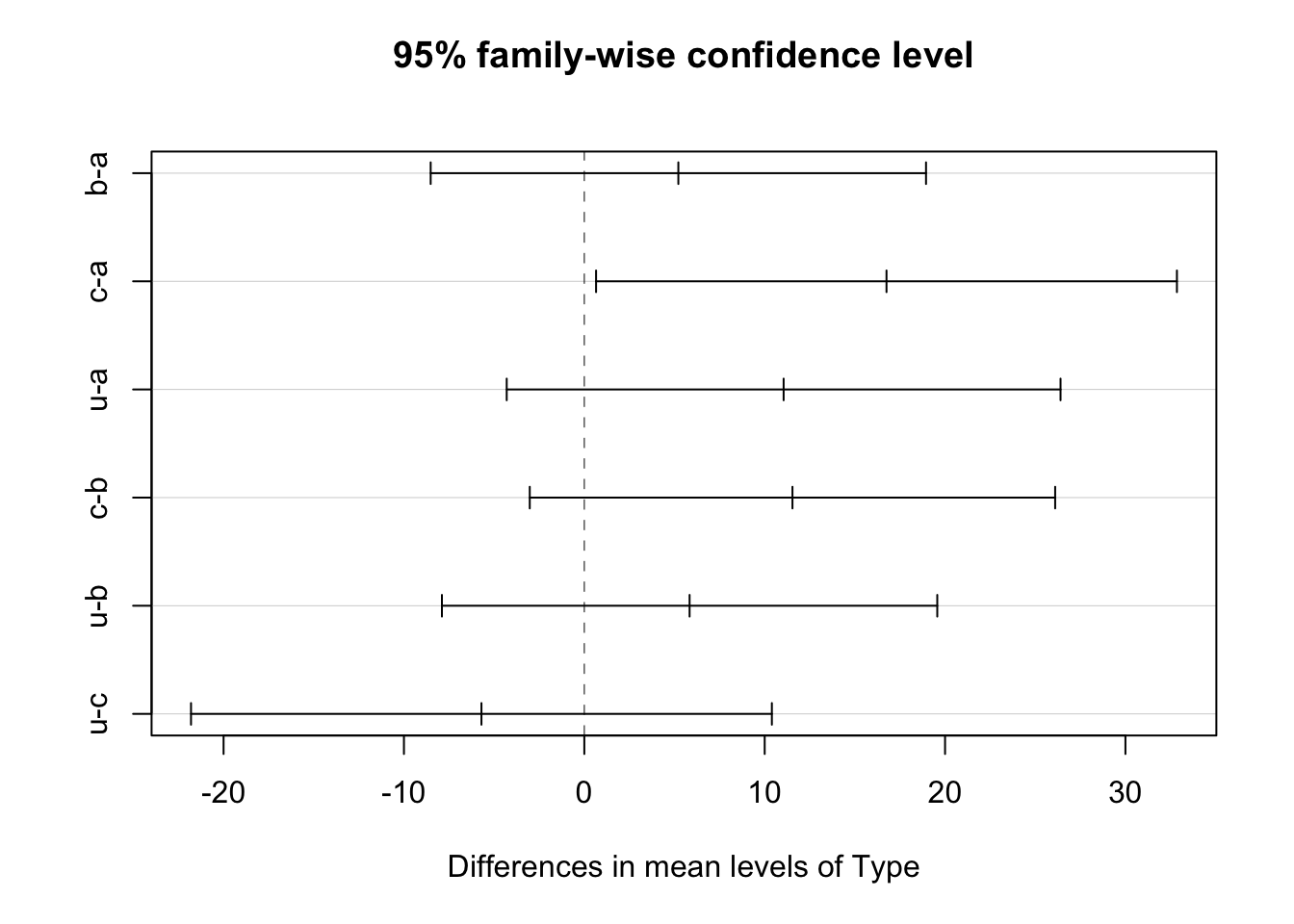
En el caso de que no se cumpla el supuesto de homogeneidad de varianzas, se puede utilizar la función oneway.test() del paquete base de R.
12.4.1 Ejercicios para ANOVA
Ejercicio 12.9 Utilizando la base de datos Cushing responda:
- ¿la variable “Pregnanetriol” es diferente entre los grupos?.
- Realice un boxplot
- Realice un gráfico de medias
- ¿Cual de los grupos es diferente?
- Cree una gráfica para la prueba posthoc
- Pruebe si tienen variancias iguales o no
Ejercicio 12.10 La base de datos original de articulo: Serum levels of adiponectin and leptin as biomarkers of proteinuria in lupus nephritis, se encuentra publicada como material adicional en (https://ndownloader.figstatic.com/files/9307021). Con esta base, utilizando la variable Groups_NLSLEvsSLE y utilizando ANOVA de una vía compare:
- Age
- Leptin
- Leptin_BMI
- Adiponectin
- Adiponectin_BMI
- Realice gráficos, estadística descriptiva y la prueba de hipótesis utilizando ANOVA de una vía
- Responda este ejercicio utilizando la librería
ggstastsplot - Responda este ejercicio utilizando JASP
Ejercicio 12.11 Compare las variables cuantitativas de su base de datos Melanoma utilizando ANOVA de una vía. Realice gráficos, estadística descriptiva y la prueba de hipótesis utilizando ANOVA de una vía. Utilice la variable Status (original) como variable de agrupación.
12.5 Pruebas no paramétricas
Cuando se quiere comparar una variables de tipo cuantitativo u ordinal entre dos o entre más de dos grupos pero esta no presenta una distribución normal no se puede emplear la prueba t-student o las prueba de ANOVA de una vía. Ya estas pruebas no son libres de parámetros y requieren que se cumplan algunos supuestos. Por lo tanto debe de recurrirse a ciertas alternativas de pruebas no-paramétricas (libres de parámetros) para poder realizar esta comparación. Las pruebas a saber son:
- Prueba U-Mann-Whitney. Prueba que compara la suma de rangos de variables en dos grupos independientes.
- Prueba Prueba de los rangos de wilcoxon. Prueba que compara la suma de rangos de dos variables medidas en dos grupos dependientes.
- Prueba de Kruskkal-Wallis. Prueba que compara la suma de rangos en más de dos grupos independientes.
Se describen a continuación como realizar cada una de estás pruebas.
12.5.1 Prueba U-Mann-Whitney
La prueba U de Mann-Whitney es una prueba estadística que se utiliza para determinar si 2 grupos son significativamente diferentes entre sí en la variable de interés (cuantitiva). La variable a comparar debe ser por lo menos de tipo ordinal. Además los grupos deberán ser independientes (no relacionados entre sí) y se deben de tener una muestra suficiente (más de 5 valores en cada grupo, aunque también depende de qué tan grande sea la diferencia entre los grupos).
La prueba U de Mann-Whitney es el equivalente a la prueba t-student para muestras independientes. Necesita el cumplimiento de los siguientes supuestos: - 1) las muestras son aleatorias e independientes y - 2) la escala de medición es ordinal o cuantitativa - 3) la forma de los datos debe ser similar entre los grupos - 4) se deben de tener datos suficientes
Un investigador desea conocer si las horas de sueño son distintas en dos grupos en grupo de pacientes tratados con benzodicepinas. El grupo 1 corresponde a pacientes medicados con clonazepam y el grupo 2 a pacientes con lorazepam. La tabla 1 muestra las horas de sueño de estos pacientes.
| clonzepam | lorazepam |
|---|---|
| 10 | 13 |
| 6 | 17 |
| 8 | 14 |
| 10 | 12 |
| 12 | 10 |
| 13 | 9 |
| 11 | 15 |
| 9 | 16 |
| 5 | 11 |
| 11 | 8 |
| 9 | 7 |
Vamos a crear dos objetos con estos datos
clonazepam <- c(10,6,8,10,12,13,11,9,5,11,9)
lorazepam <- c(13, 17,14,12,10,9,15,16,11,8,7)Se describe a continuación como realizar esta prueba en R
Las prueba de U-Mann-Whitney fue desarrollada en dos versiones independientes que conducen a la misma conclusión: la de Mann y Whitney, y la Wilcoxon. Por ello se conoce como prueba de Mann-Whitney-Wilcoxon. Este antecedente es la razón por la que en R se utiliza la función wilcox.test.
Utilizando los objetos creados anteriormente el código quedaría:
wilcox.test(clonazepam, lorazepam, paired = F)# Es necesario especificar que los datos no son pareados Warning in wilcox.test.default(clonazepam, lorazepam, paired = F): cannot
compute exact p-value with ties
Wilcoxon rank sum test with continuity correction
data: clonazepam and lorazepam
W = 34.5, p-value = 0.09265
alternative hypothesis: true location shift is not equal to 0En el ejemplo anterior se utilizó la coma para indicar las comparaciones, dado que estamos trabajando con dos objetos diferentes.
Finalmente podremos comprobar las diferencias visualmente realizando un boxplot.
boxplot(lorazepam, clonazepam,
main="Comparación de las horas de sueño entre dos benzodiacepinas",
col = c("#76EEC6", "#FFE4C4"),
xlab = "Grupos",
ylab = "Horas de sueño")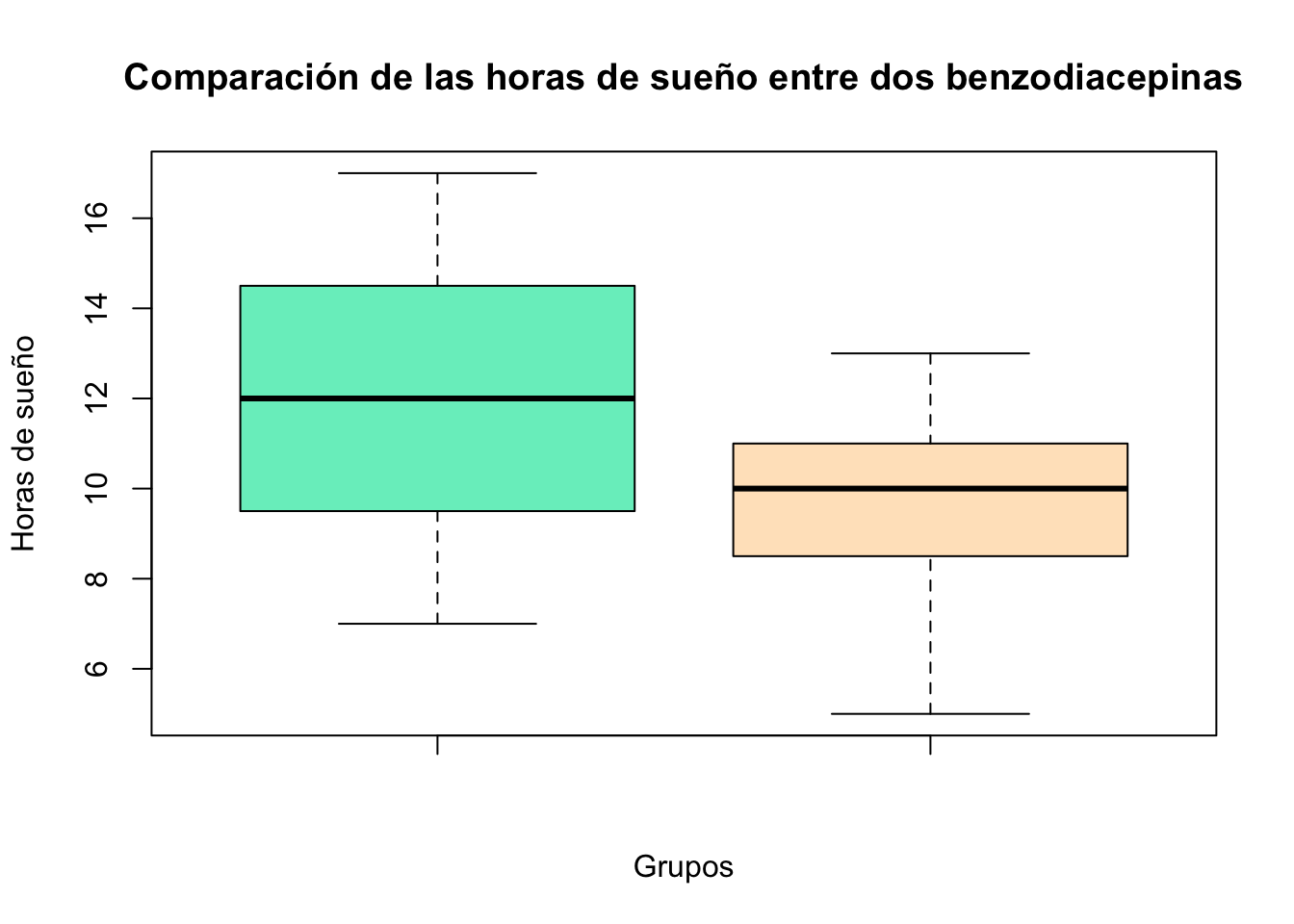
Ejemplo 12.5 Importe la base Non-Parametric utilizando el menú de Rstudio. Esta base es un archivo .xlxs que contiene datos de biomarcadores entre un grupo de casos y controles y entre un grupo 1 y un grupo 2.
Utilizando la viraibles Patients que indica si se trata de un caso o un control compare la edad (Age) utilizando al prueba de U-Mann-Whitney los siguiente:
Después de importar la base de datos, podemos crear una gráfica y luego realizar nuestra prueba de hipótesis:
boxplot(Non_Parametric$Biomarcador1~Non_Parametric$Patients,
main="Comparación del Biomarcador 1 entre casos y controles",
col = c("#76EEC6", "#FFE4C4"),
xlab = "Grupos",
ylab = "Concentracioens de biomarcadore",
frame=F)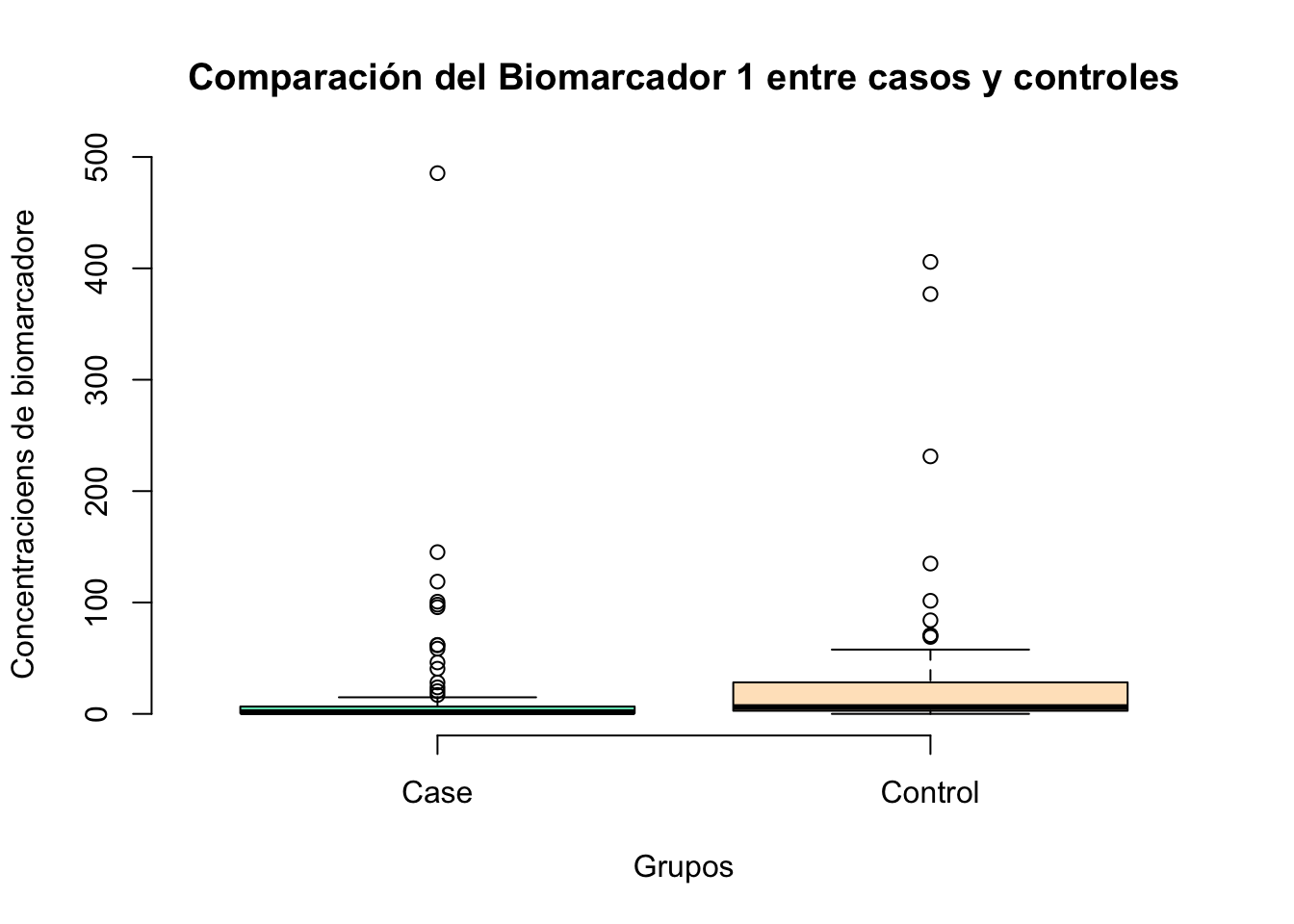
vioplot::vioplot(Non_Parametric$Biomarcador1~Non_Parametric$Patients,
main="Comparación del Biomarcador 1 entre casos y controles",
col = c("#76EEC6", "#FFE4C4"),
xlab = "Grupos",
ylab = "Concentracioens de biomarcadore",
frame=F)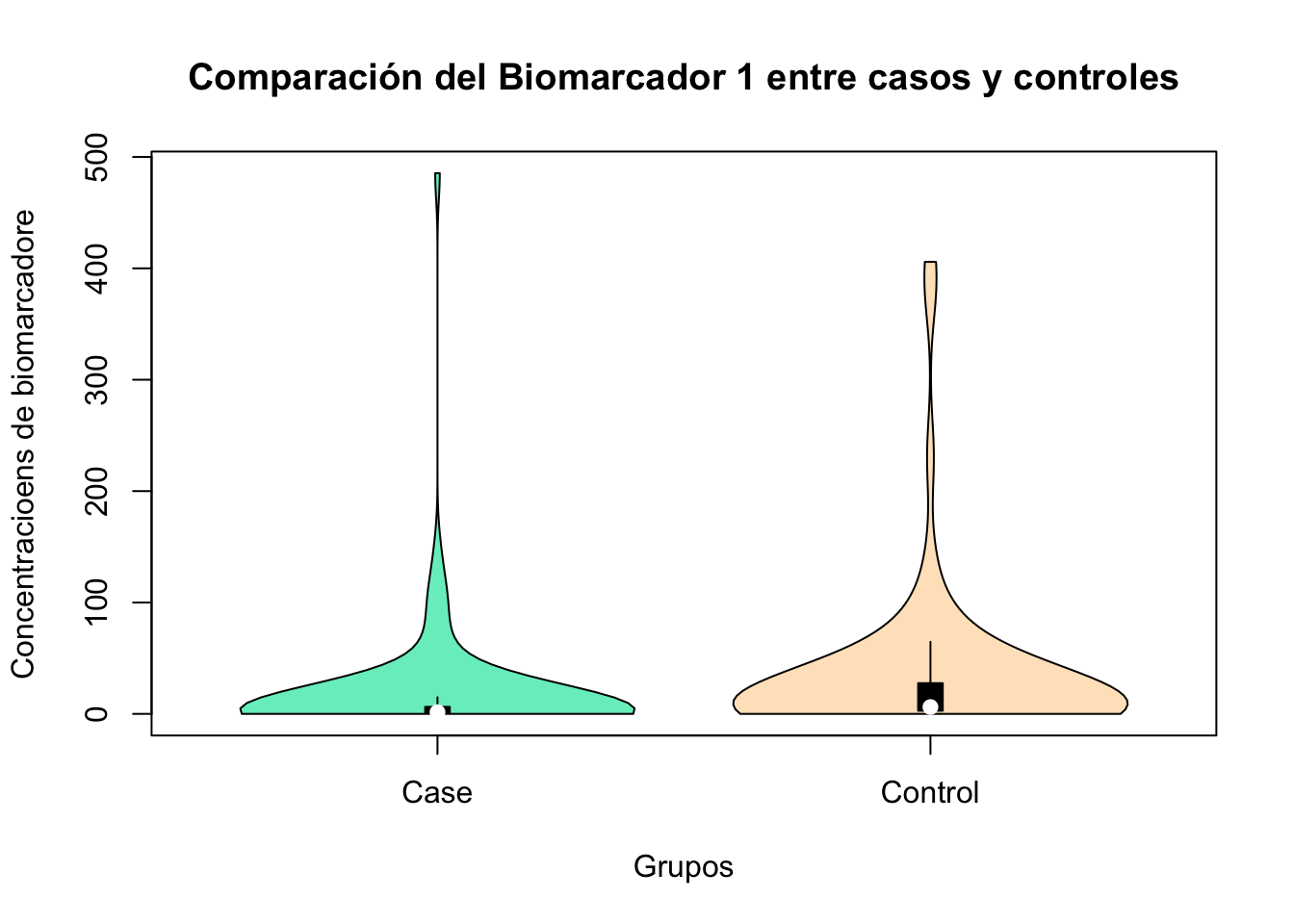
wilcox.test(Non_Parametric$Biomarcador1~Non_Parametric$Patients)
Wilcoxon rank sum test with continuity correction
data: Non_Parametric$Biomarcador1 by Non_Parametric$Patients
W = 2117, p-value = 8.644e-06
alternative hypothesis: true location shift is not equal to 012.5.2 Ejercicios para la prueba U-Mann-Whitney
Ejercicio 12.12 Con la bae de datos Non-Parametric y Utilizando la viraibles Patients que indica si se trata de un caso o un control compare utilizando al prueba de U-Mann-Whitney los siguiente:
- BiomarkerA
- BiomarkerB
- Biomarcador1
- Biomarcador2
- Biomarcador3
Ejercicio 12.13 Repita la resolución del Ejercicio 12.3 pero esta vez, utilice estadística no paramétrica
12.5.3 Prueba de los rangos de Wilcoxon
La prueba de Wilcoxon para rangos con signo de pares comparados es alternativa a la prueba de los signos, con la ventaja de que reconoce la magnitud de las diferencias observadas en cada par muestreado. Es el equivalente no paramétrico de la prueba t-student para muestras pareadas.
Para realizar esta prueba se deben de cumplir con los siguientes supuestos:
Cada par de sujetos muestreados es aleatorio.
Las observaciones proceden de una población con distribución simétrica.
La variable de interés es cuantitativa u ordinal
Unos investigadores desean conocer si el tratamiento experimental influye en el peso de dos grupos de ratones. La siguiente tabla muestra los pesos de los ratones antes y después del tratamiento experimental
| peso antes | peso después |
|---|---|
| 90 | 80 |
| 95 | 89 |
| 80 | 72 |
| 79 | 84 |
| 89 | 94 |
| 95 | 80 |
| 99 | 89 |
| 84 | 72 |
| 78 | 84 |
| 69 | 64 |
| 74 | 80 |
| 95 | 89 |
| 99 | 72 |
| 84 | 84 |
| 78 | 94 |
| 69 | 84 |
| 74 | 94 |
Se crean dos objetos, uno para peso antes y otro para peso después
Peso_Antes <- c(74, 78, 69, 78, 74, 79, 89, 84, 69, 95, 95, 80, 90, 99,84,95, 99)
Peso_Despues <- c(94, 94, 84, 84, 80, 84, 94, 84, 64, 89, 89, 72, 80, 89, 72, 80, 72)Se utiliza la función wilcox.test con el argumento paired=T
wilcox.test(Peso_Antes, Peso_Despues, paired = T, conf.int = T)Warning in wilcox.test.default(Peso_Antes, Peso_Despues, paired = T, conf.int =
T): cannot compute exact p-value with tiesWarning in wilcox.test.default(Peso_Antes, Peso_Despues, paired = T, conf.int =
T): cannot compute exact confidence interval with tiesWarning in wilcox.test.default(Peso_Antes, Peso_Despues, paired = T, conf.int =
T): cannot compute exact p-value with zeroesWarning in wilcox.test.default(Peso_Antes, Peso_Despues, paired = T, conf.int =
T): cannot compute exact confidence interval with zeroes
Wilcoxon signed rank test with continuity correction
data: Peso_Antes and Peso_Despues
V = 79.5, p-value = 0.5685
alternative hypothesis: true location shift is not equal to 0
95 percent confidence interval:
-5.499992 9.000023
sample estimates:
(pseudo)median
1.849605 - Hacer un boxplot para visualizar sus resultados
boxplot(Peso_Despues, Peso_Antes, main="Comparación de los peso antes y después del tratamiento")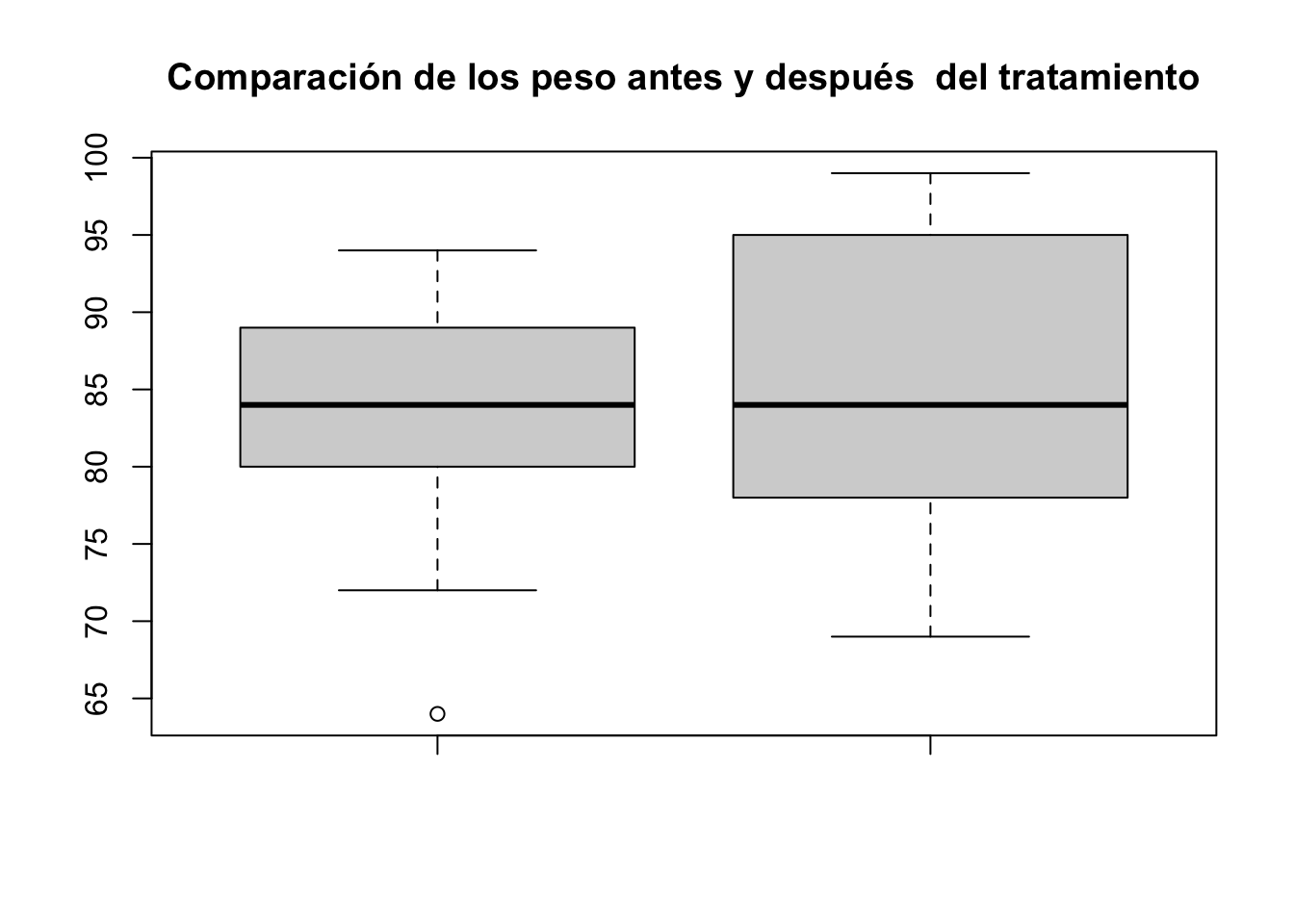
Ejemplo 12.6 Importe la base Non-Parametric utilizando el menú de Rstudio. Esta base es un archivo .xlxs que contiene datos de biomarcadores medidos en diferente tiempos.
Responda lo siguiente:
- ¿Los niveles de BiomarkerA son iguales a los 6 y 12 meses?. Cree además una gráfica de boxplot. Utilice las variables BiomarkerA_6M y BiomarkerA_12M para responder esta pregunta
boxplot(Non_Parametric$BiomarkerB_6M, Non_Parametric$BiomarkerB_12M,
main="Comparación del Biomarcador B a los 6 y 12 meses",
col = c("#76EEC6", "#FFE4C4"),
xlab = "Grupos",
ylab = "Concentracioens de biomarcador B",
frame=F,
names = c("Biomarcador B 6 meses", "Biomarcador B 12 meses"))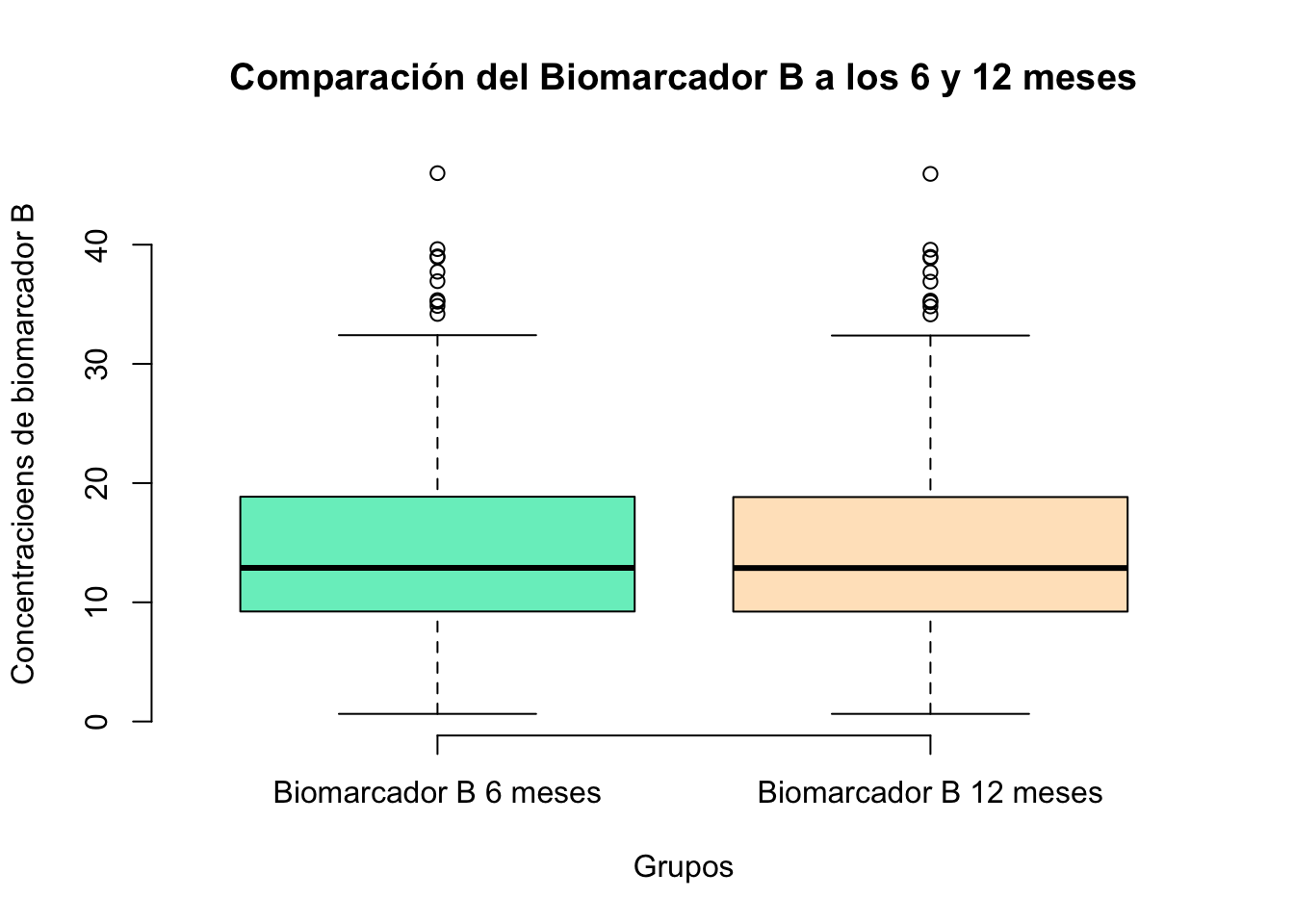
wilcox.test(Non_Parametric$BiomarkerB_6M, Non_Parametric$BiomarkerB_12M, paired = T)
Wilcoxon signed rank test with continuity correction
data: Non_Parametric$BiomarkerB_6M and Non_Parametric$BiomarkerB_12M
V = 15225, p-value < 2.2e-16
alternative hypothesis: true location shift is not equal to 012.5.4 Ejercicios para la prueba de los rangos de Wilcoxon
Ejercicio 12.14 ¿Los niveles de BiomarkerB son iguales a los 3 y 6 meses?. Utilice las variables BiomarkerB_3M y BiomarkerB_6M?
Ejercicio 12.15 ¿Los niveles de BiomarkerB son iguales a los 6 y 12 meses?. Utilice las variables BiomarkerB_6M y BiomarkerB_12M.?
12.6 Prueba de Kruskall-Wallis
Cuando se desea comparar una variable cuantitativa u ordinal en tres o más grupos y esta variable no se comporta de forma normal o la muestra es muy pequeño se utiliza la prueba de Kruskal-Wallis. Esta prueba es el equivalente no paramétrico del AONVA de una vía.
Los supuestos de esta prueba son:
Cada una de las muestras ha sido tomada al azar de su población.
Hay independencia entre las observaciones dentro de cada muestra, así como entre las muestras.
Los datos representan medidas por lo menos en una escala ordinal.
En Rse utiliza la función kruskal.test. La función tiene los siguientes argumentos:
x: Es un un vector numérico.
g: Es un vector numérico o un factor.
- a vector or factor object giving the group for the corresponding elements of x. Ignored with a warning if x is a list.
formula: Esta función se puede utilizar a manera de formula (response ~ group)
Ejemplo 12.7 Utilizando la base de datos Non-Parametric, responda la siguientes preguntas:
- Utilizando la variable
groupsindique si se presentan diferencias en:
- BiomarkerA
Realice un boxplot y agregue el valore de para cada una de las comparaciones anteriores.
Comparación de biomarkerA
boxplot(Non_Parametric$BiomarkerA~Non_Parametric$Groups,
main="Comparación del Biomarcador entre
grupo 1, grupo 2 y Controles",
col = 2:5, #Colores del 2 a 5
xlab = "Grupos",
ylab = "Concentracioens de biomarcador A",
frame=T,
names = c("Control", "Grupo 1", "Gropo 2"))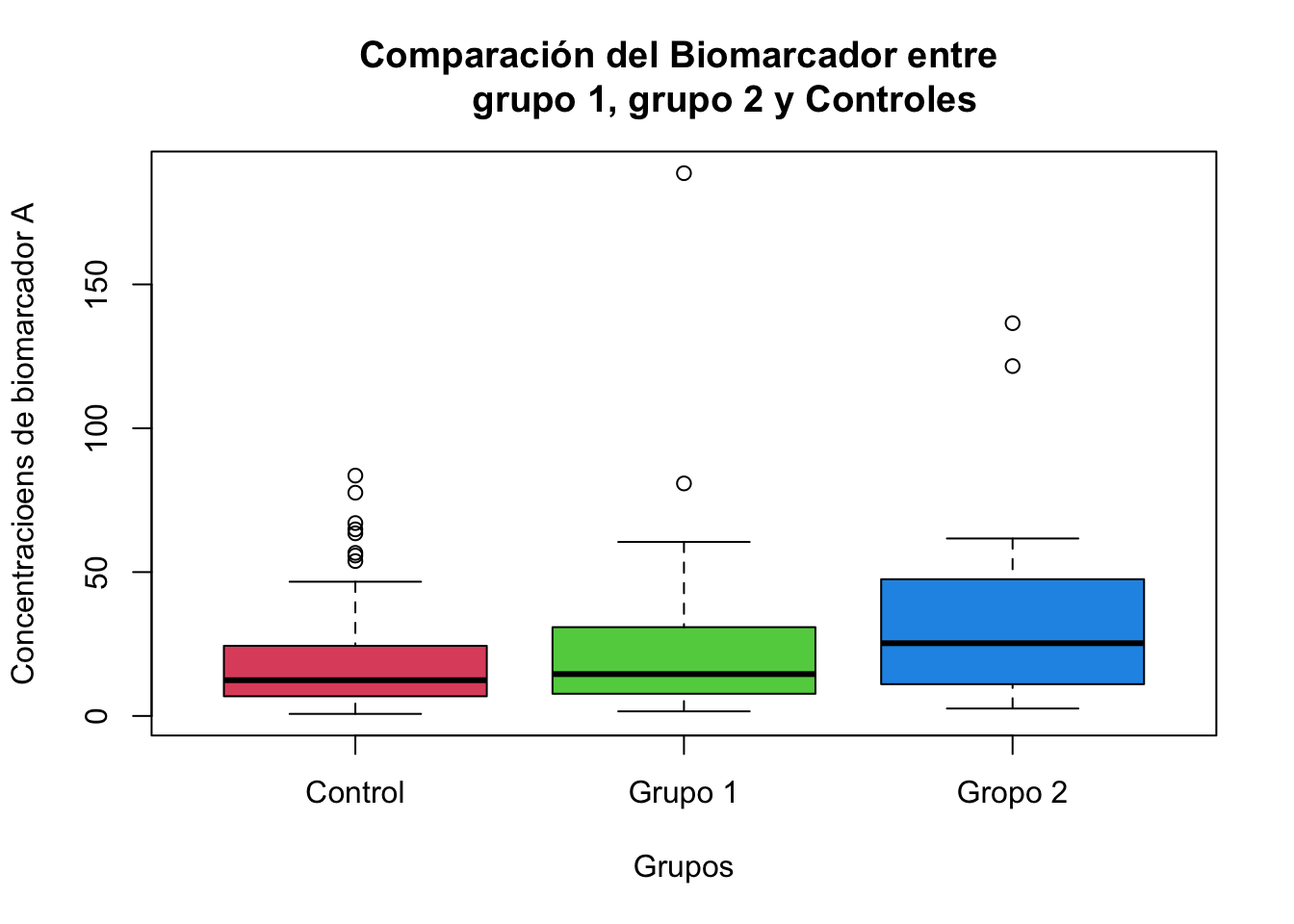
# Puede utilizar la función table o names para conocer los nombres
# table (Non_Parametric$Groups)
# names (Non_Parametric$Groups)kruskal.test(Non_Parametric$BiomarkerA~Non_Parametric$Groups)
Kruskal-Wallis rank sum test
data: Non_Parametric$BiomarkerA by Non_Parametric$Groups
Kruskal-Wallis chi-squared = 5.5597, df = 2, p-value = 0.06205# Este código llega al mismo resultado
# kruskal.test(BiomarkerA~Groups, data=Non_Parametric)Para agregar el valor p utilice la función text
boxplot(Non_Parametric$BiomarkerA~Non_Parametric$Groups,
main="Comparación del Biomarcador entre
grupo 1, grupo 2 y Controles",
col = 2:5, #Colores del 2 a 5
xlab = "Grupos",
ylab = "Concentracioens de biomarcador A",
frame=T,
names = c("Control", "Grupo 1", "Gropo 2"))
text(x=2, y=150, label = "p-value = 0.06")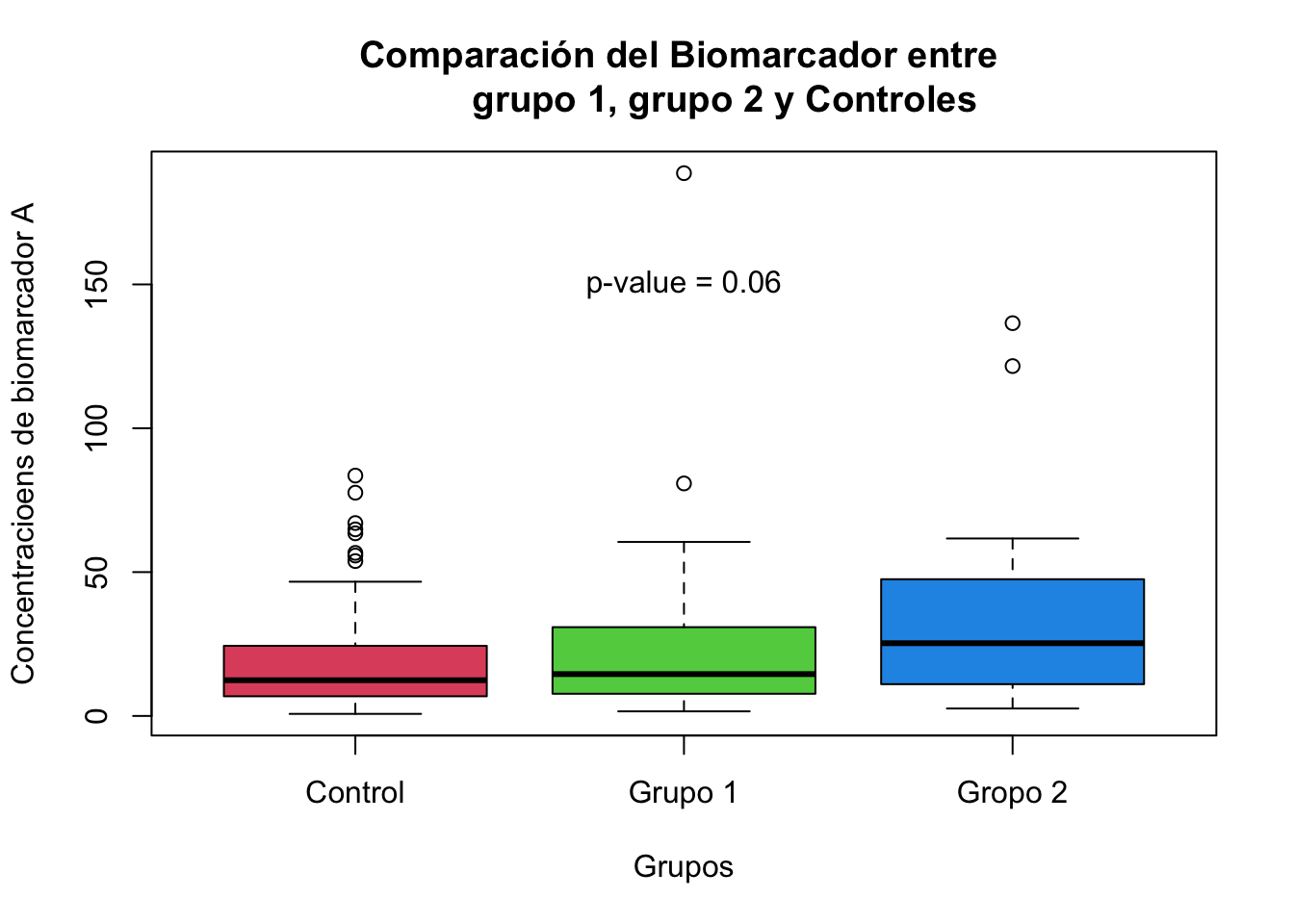
Ejercicio 12.16 Utilizando la base de datos Non-Parametric, responda la siguientes preguntas:
- Utilizando la variable
groupsindique si se presentan diferencias en:
- BiomarkerA
- BiomarkerB
- BMI
- Age
- Realice un boxplot y agregue el valore de para cada una de las comparaciones anteriores.
- Utilice la librería ggstatsplot para realizar la comparación de las variables anteriores
Ejercicio 12.17 Utilizando la base de datos Melanoma y asuimiendo estadística no paramétrica responda la siguientes preguntas: a) ¿El tamaño del tumor se asocia con el estatus del paciente (variable original)? b) ¿La edad se asocia con el estatus del paciente (variable original)?