library(MASS)
data("Melanoma")10 Análisis exploratorio de datos
- Autor: Edsaúl Emilio Pérez Guerrero
- Afiliación: Universidad de Guadalajara
- mail: edsaul.perezg@academicos.udg.mx
- Última actualización: 2024-09-11
Este laboratorio de R esta pensado en la exploración de datos. El paso previo a la estadística inferencial.
10.1 Introducción
De forma general se puede decir que los pasos para el análisis estadístico son los siguientes:
Preparación de la base de datos
Elección del software estadístico
Búsqueda de errores y depuración de la base de datos
Exploración de la base de datos (estadística descriptiva)
Formular hipótesis, responder preguntas iniciales
Análisis estadístico para cumplir con los objetivos del proyecto de investigación
Conclusiones.
En este compendio nos enfocaremos en los puntos del 3 al 5. Estas etapas tendrán como objetivo:
Resumir y presentar datos.
Tener una compresión profunda de las variables y datos contenidos en el data frame
Identificar valores atípicos y errores en la captura de los datos.
Identificar la distribución o las distribuciones de los datos
Eliminar variables que no necesitamos (con precaución)
Extraer respuestas y formular hipótesis. Incluso se pueden responder algunas preguntas iniciales.
En conjunto estos pasos pertenecen a la exploración de datos.
10.2 Exploración de datos
La exploración de datos se podría decir que es el proceso de analizar conjuntos de datos para encontrar patrones y relaciones. En ocasiones se denomina más formalmente análisis exploratorio de datos (EDA). El EDA puede ayudarlo a desarrollar hipótesis sobre cómo se relacionan las diferentes variables. Además, puede ayudarnos a identificar qué variables son las más importantes para predecir un resultado en particular. Explorar los datos puede ayudarlo a comprender mejor los datos y desarrollar la intuición sobre cómo se comportan los datos. Además, la exploración de datos en muy útil para la identificación de errores en nuestra base de datos.
Es recomendable familiarizarse y entender los datos antes de trabajar con ellos. El EDA tiene como objetivo entender y descifrar la información contenida en los datos antes de realizar intervenciones adicionales. Por ejemplo, imagine que se le encomienda realizar una excavación en un playa. Puede intuir que es lo que espera encontrar, sin embargo, en el camino puede encontrar diferentes objetos que le permitan tomar decisiones sobre a donde concentrar su búsqueda. Algo similar sucede con la EDA.
La exploración de datos incluye:
- Gráficas
- Tablas
- Resúmenes de datos
- Otros
10.2.1 Pasos para realizar un EDA
En este compendio para realizar un EDA seguiremos los siguientes pasos.
- Recodificación de variables
- Identificación de valores de perdidos y errores en la base de datos
- Evaluar la normalidad de los datos
- Realizar una exploración gráfica
- Para variables cualitativas
- gráficos de barras, sectores, etc.
- Para variables cuantitativas
- Boxplot, densidad, histogramas etc.
- Estadística descriptiva según el tipo de variable
- Identificación de relaciones y formulación de hipótesis
Estos pasos no son únicos y puede agregar los considere necesarios, además no siempre se debe de seguir el mismo orden que el planteado arriba. Sin embargo, los ejercicios de este compendio serán realizados utilizando este orden.
10.3 EDA del DataFrame Melanoma
El data frame Melanoma contiene datos de 205 pacientes en Dinamarca con melanoma maligno.
Este data frame contiene las siguientes columnas:
time: Tiempo de supervivencia en días, posiblemente censurado.status: 1 = fallecido por melanoma, 2 = vivo, 3 = fallecido por otras causas.sex: 1 = masculino, 0 = femenino.age: Edad en años.year: Año de la operación.thickness: Espesor del tumor en mm.ulcer: 1 = presencia, 0 = ausencia.
Vamos a proceder a realizar la explorarción de la base de datos
Ejemplo 10.1 Realice la exploración de la base de datos Melanoma de la libreía MASS. Puede tomar como base la siguient pregunta: ¿El tamaño del tumor influirá sobre el desenlace de los pacientes? Utilice los pasos para EDA descritos en la sección anterior
Para importar la base de datos utilice el siguiente código:
Paso 1: Recodificación de variables
Puede visualizar la ayuda para identificar las variables que no están bien codificadas
help(Melanoma)Si hacemos una visualización de la base con la función str podemos notar que: las variables status, sex, y ulcer requieren codificación, R la está leyendo como variables numéricas cuando deberías ser factores.
También puede utilizar la función vis_dat de la librería visdat. Esta función muestra gráficamente la estructura de nuestro data frame.
# install.packages("visdat") # Si lo requiere
visdat::vis_dat(Melanoma)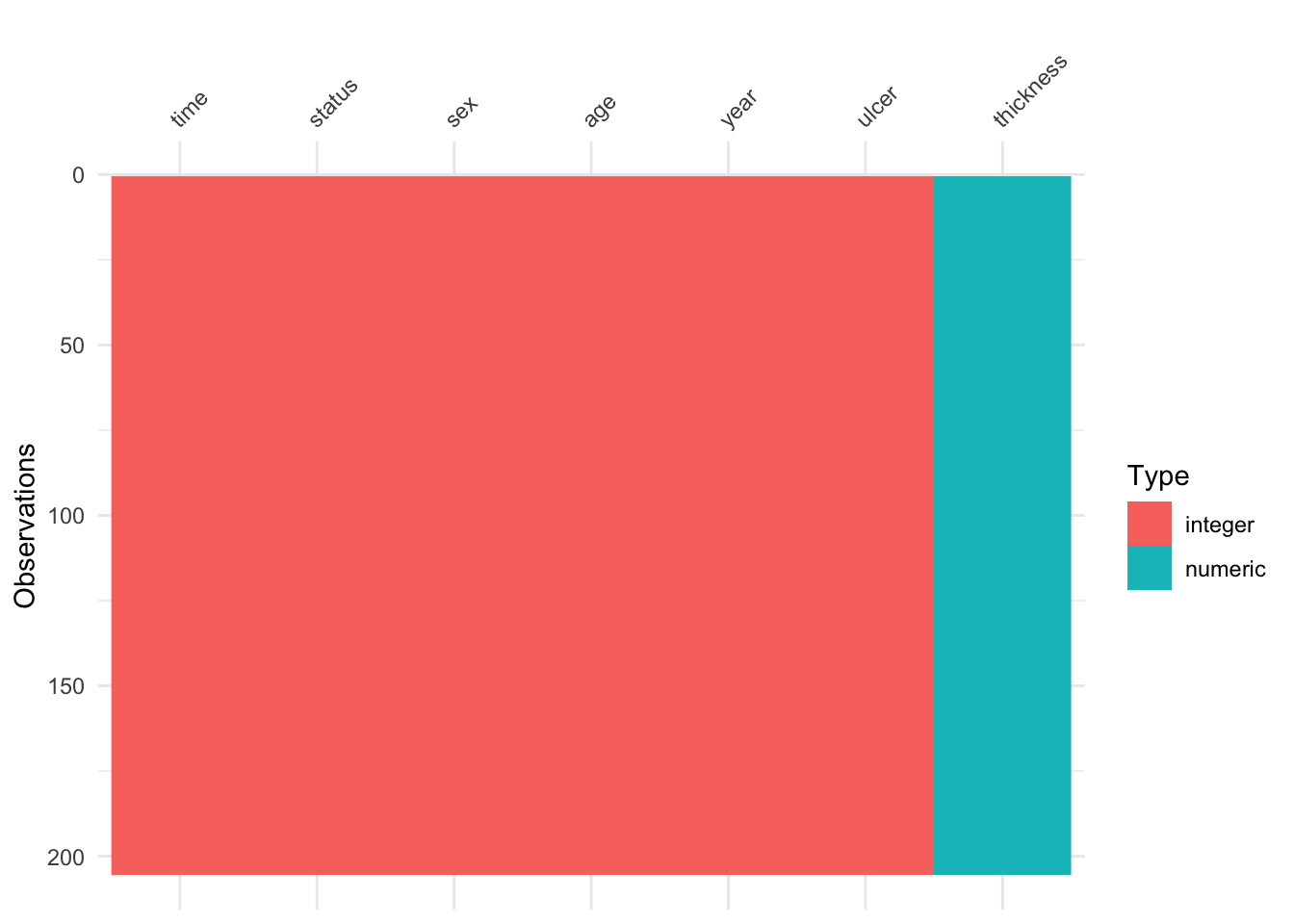
Para recodificar las variables, puede utilizar la forma clásica para la re codificación como lo visto en Capítulo 4
Melanoma$status <- as.factor(Melanoma$status)Sin embargo una mejor opción es cambiar los valores de los factores por etiquetas. Puede emplear el siguiente código:
Melanoma$status2 <- factor(Melanoma$status,
levels=c(1,2,3), # Si los niveles están ordenado se puede omitir
labels = c("died from melanoma", "alive", "dead from other causes"))
Melanoma$ulcer <- factor(Melanoma$ulcer,levels = c(1,0),
labels = c("presence", "absence"))
Melanoma$sex<-factor(Melanoma$sex,
levels=c(1,0),
labels=c("male","famale"))La función factor permite cambiar una variable a factor y establecer etiquetas para los valores numéricos. Note como cambiamos directamente los objetos en la base de datos, sin embargo, creamos una variable status2 para conservar la original. Posteriormente explicaremos el porqué de esta acción.
Una vez realizado el cambio de la codificación de los factores podemos comprobar la estructura de la base utilizando la función str
str(Melanoma)'data.frame': 205 obs. of 8 variables:
$ time : int 10 30 35 99 185 204 210 232 232 279 ...
$ status : int 3 3 2 3 1 1 1 3 1 1 ...
$ sex : Factor w/ 2 levels "male","famale": 1 1 1 2 1 1 1 2 1 2 ...
$ age : int 76 56 41 71 52 28 77 60 49 68 ...
$ year : int 1972 1968 1977 1968 1965 1971 1972 1974 1968 1971 ...
$ thickness: num 6.76 0.65 1.34 2.9 12.08 ...
$ ulcer : Factor w/ 2 levels "presence","absence": 1 2 2 2 1 1 1 1 1 1 ...
$ status2 : Factor w/ 3 levels "died from melanoma",..: 3 3 2 3 1 1 1 3 1 1 ...o con el siguiente código:
# install.packages("visdat") # Si lo requiere
visdat::vis_dat(Melanoma)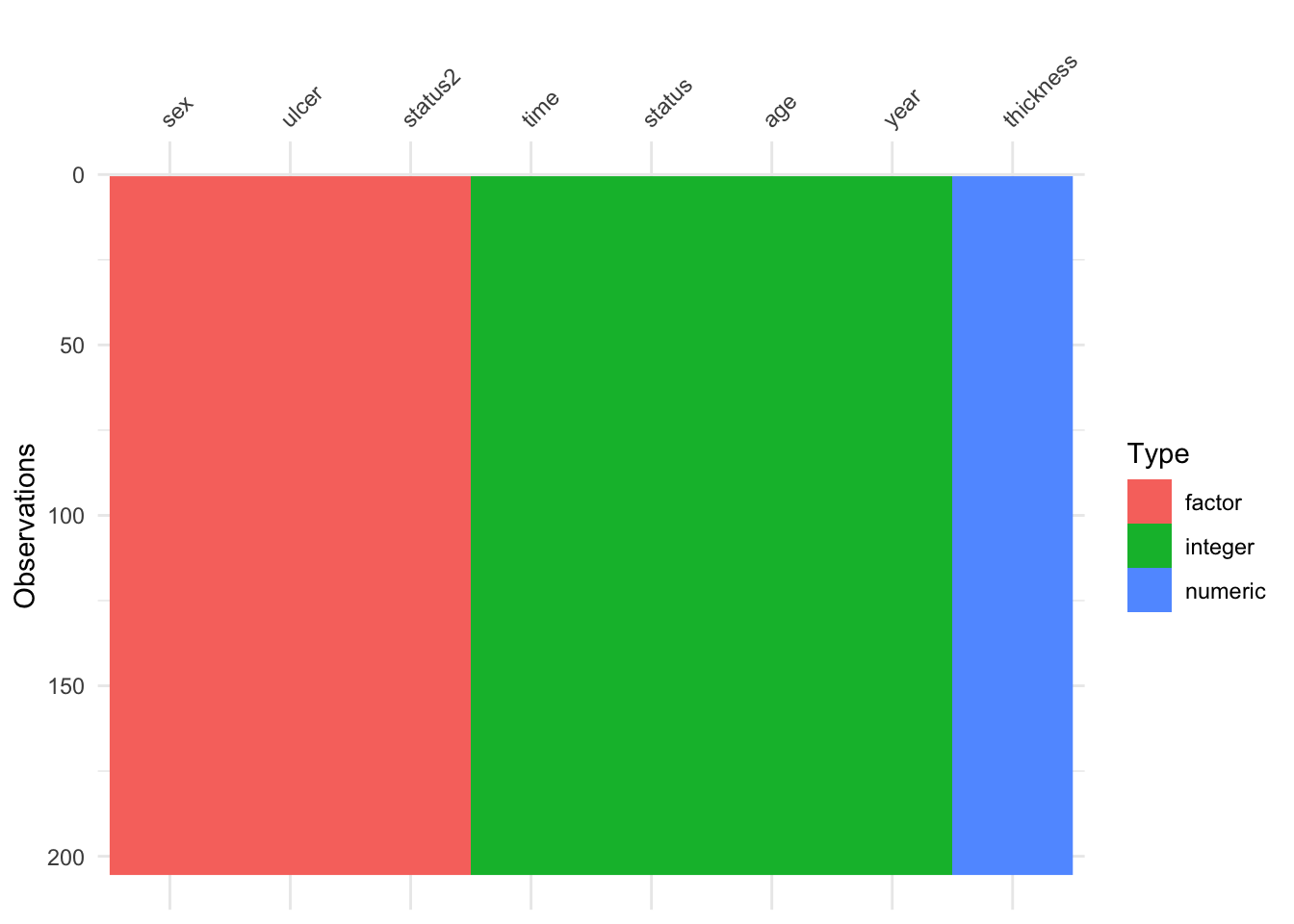
Paso 2: Indentificación de errores y valores perdidos
Para la identificación de errores y valores perdidos tenemos varias opciones: utilizar la función summary, hacer gráficos en las variables cuantitativas y preguntar a R directamente si existen valores perdidos.
summary(Melanoma) time status sex age year
Min. : 10 Min. :1.00 male : 79 Min. : 4.00 Min. :1962
1st Qu.:1525 1st Qu.:1.00 famale:126 1st Qu.:42.00 1st Qu.:1968
Median :2005 Median :2.00 Median :54.00 Median :1970
Mean :2153 Mean :1.79 Mean :52.46 Mean :1970
3rd Qu.:3042 3rd Qu.:2.00 3rd Qu.:65.00 3rd Qu.:1972
Max. :5565 Max. :3.00 Max. :95.00 Max. :1977
thickness ulcer status2
Min. : 0.10 presence: 90 died from melanoma : 57
1st Qu.: 0.97 absence :115 alive :134
Median : 1.94 dead from other causes: 14
Mean : 2.92
3rd Qu.: 3.56
Max. :17.42 lapply(Melanoma, is.na)$time
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[37] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[97] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[109] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[121] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[133] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[145] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[157] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[193] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[205] FALSE
$status
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[37] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[97] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[109] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[121] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[133] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[145] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[157] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[193] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[205] FALSE
$sex
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[37] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[97] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[109] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[121] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[133] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[145] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[157] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[193] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[205] FALSE
$age
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[37] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[97] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[109] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[121] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[133] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[145] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[157] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[193] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[205] FALSE
$year
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[37] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[97] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[109] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[121] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[133] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[145] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[157] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[193] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[205] FALSE
$thickness
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[37] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[97] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[109] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[121] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[133] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[145] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[157] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[193] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[205] FALSE
$ulcer
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[37] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[97] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[109] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[121] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[133] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[145] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[157] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[193] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[205] FALSE
$status2
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[13] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[25] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[37] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[49] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[61] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[73] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[85] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[97] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[109] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[121] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[133] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[145] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[157] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[169] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[181] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[193] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
[205] FALSEAunque un código más estilizado es:
lapply(Melanoma, is.na)|>
table(), , sex = FALSE, age = FALSE, year = FALSE, thickness = FALSE, ulcer = FALSE, status2 = FALSE
status
time FALSE
FALSE 205También con la función visdat::vis_dat(Melanoma) pude visualizar los NA.
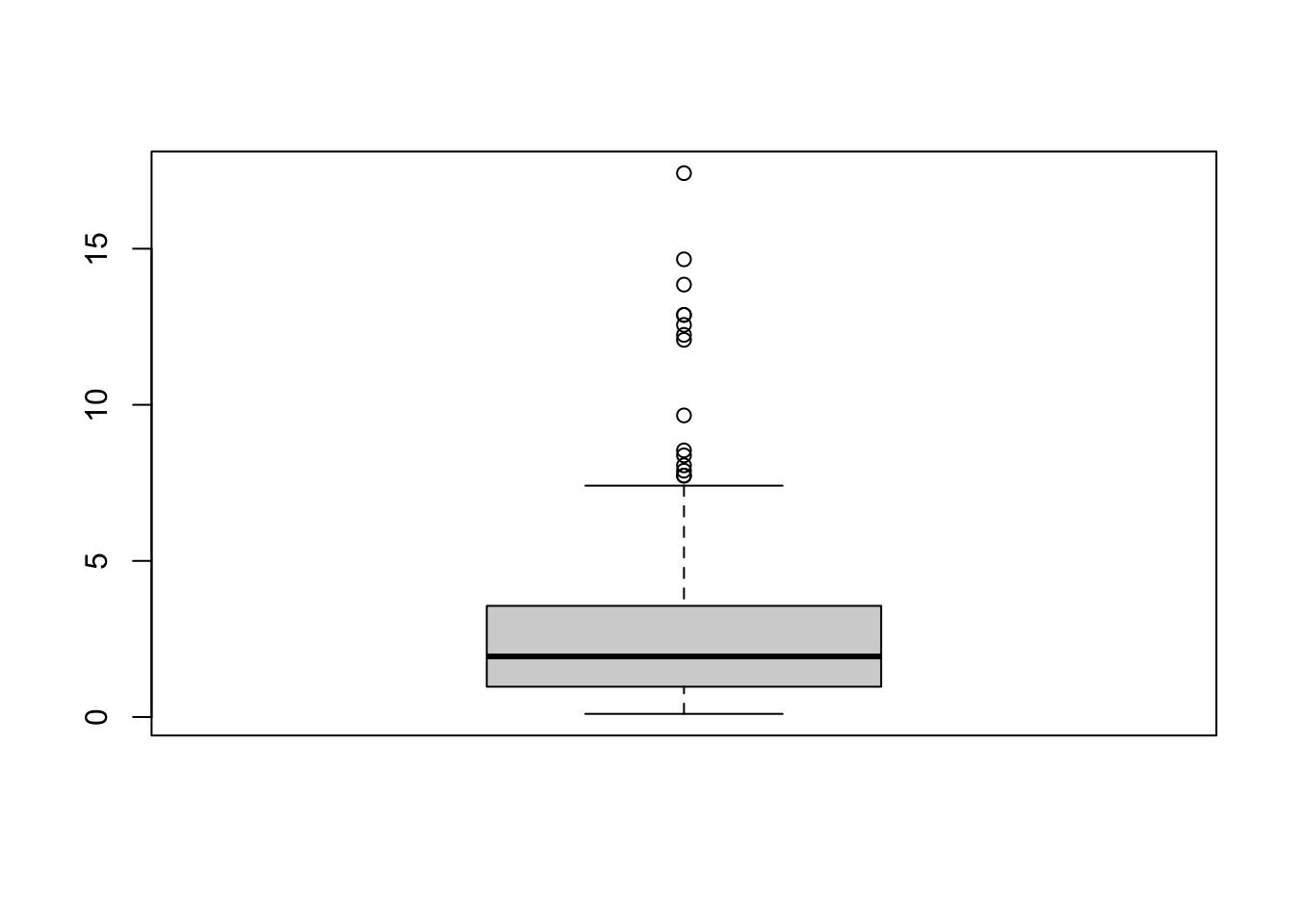
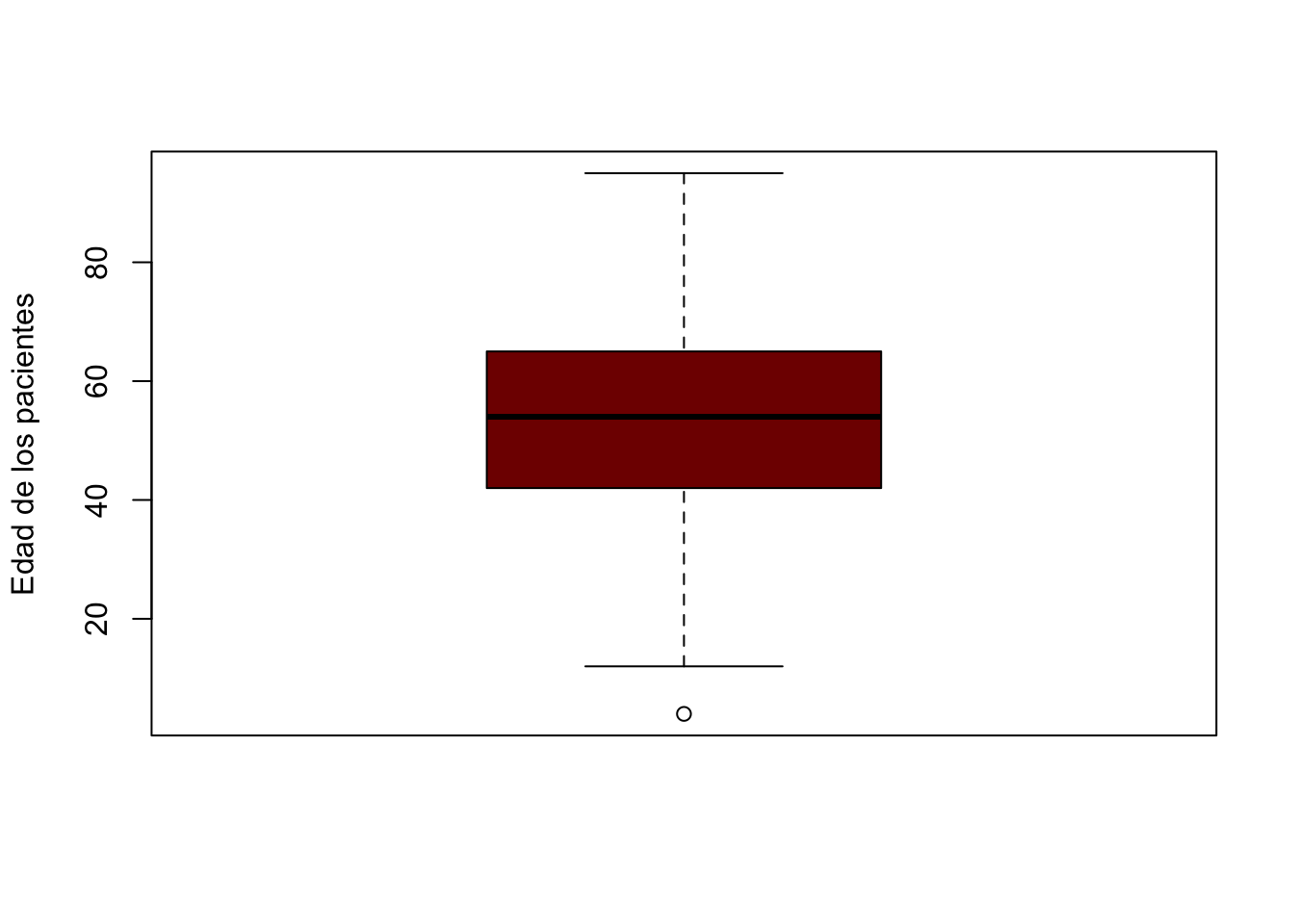
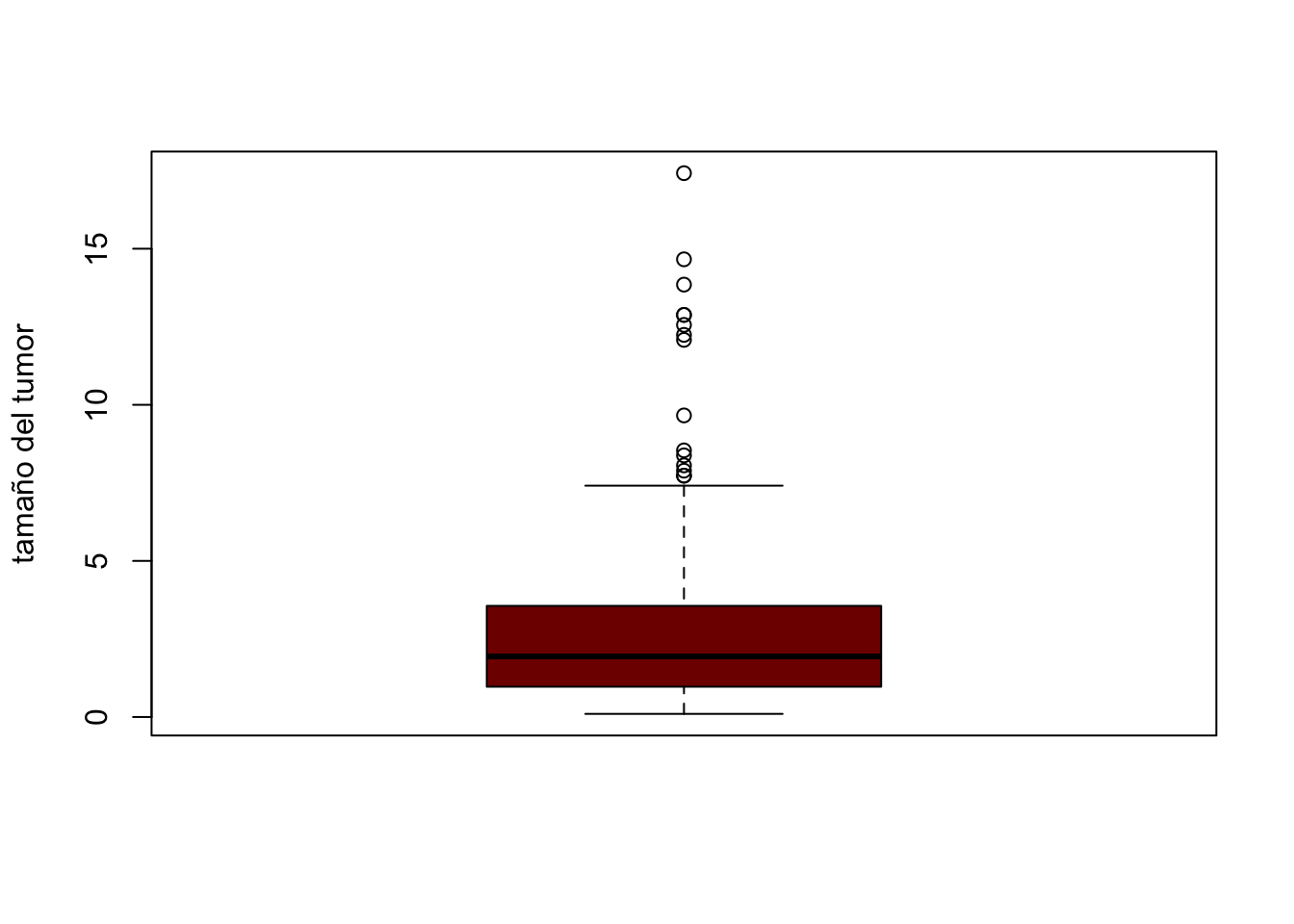
Para identificar errores podemos emplear gráficos de boxplot, si hay algún valor fuera de lugar será visible en el boxplot. Esto, solo aplica para variables cuantitativas.
apply(Melanoma[, c(4,6)], 2, boxplot)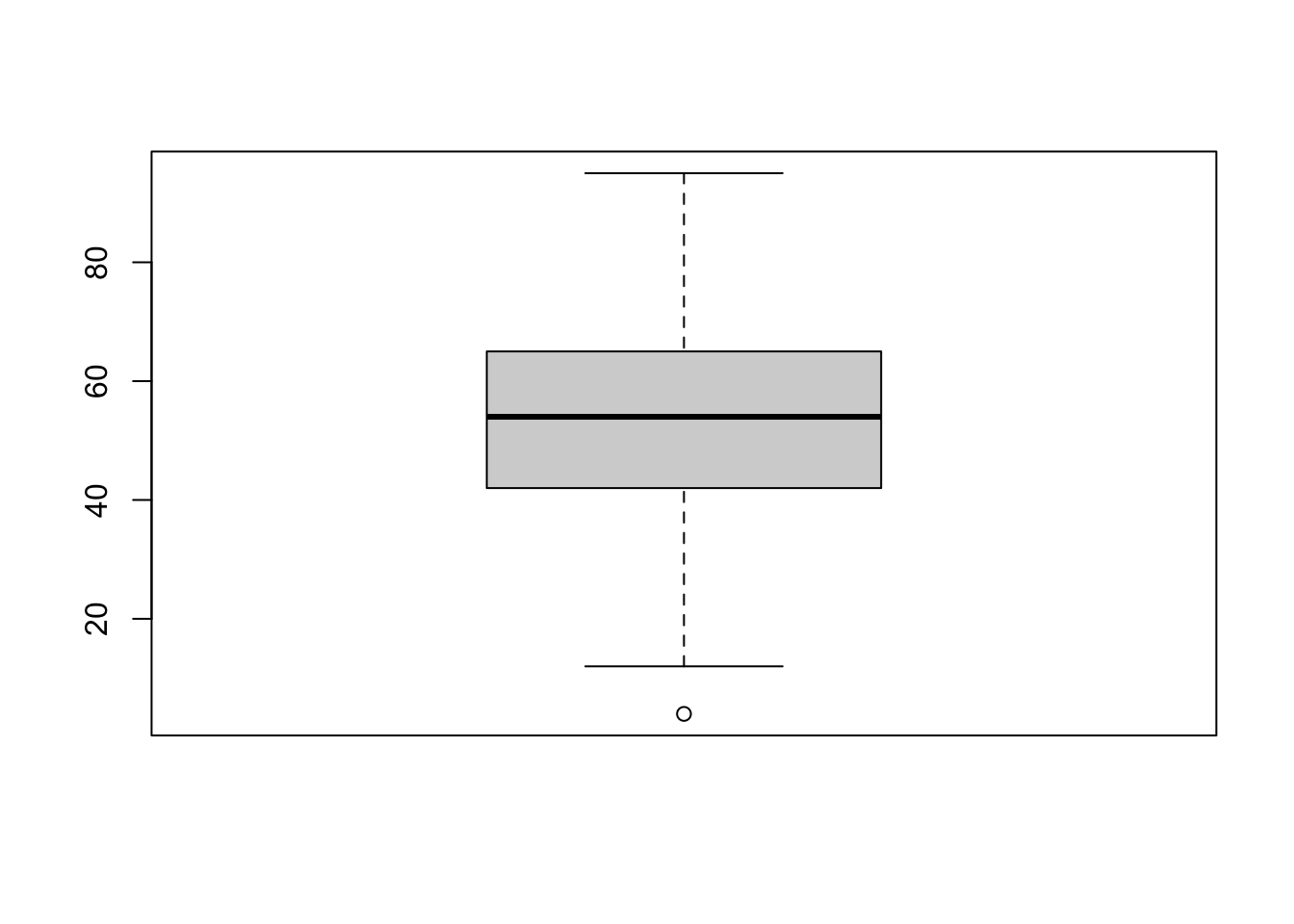
$age
$age$stats
[,1]
[1,] 12
[2,] 42
[3,] 54
[4,] 65
[5,] 95
$age$n
[1] 205
$age$conf
[,1]
[1,] 51.4619
[2,] 56.5381
$age$out
174
4
$age$group
[1] 1
$age$names
[1] "1"
$thickness
$thickness$stats
[,1]
[1,] 0.10
[2,] 0.97
[3,] 1.94
[4,] 3.56
[5,] 7.41
$thickness$n
[1] 205
$thickness$conf
[,1]
[1,] 1.654188
[2,] 2.225812
$thickness$out
5 9 16 20 21 27 35 43 48 98 100 123 141
12.08 12.88 12.56 7.73 13.85 8.54 14.66 17.42 8.38 7.73 12.88 9.66 7.89
190 191
12.24 8.06
$thickness$group
[1] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
$thickness$names
[1] "1"Paso 3. Normalidad de los datos
Evaluar la normalidad me puede ayudar, entre otras cosas, para indicarme cual es la manera correcta de presentar y analizar mis resultados. Para evaluar la normalidad podemos utilizar gráficos y pruebas de normalidad.
Para evaluar la normalidad gráficamente puede utilizar la función qqPlot de la librería car
car::qqPlot(Melanoma$age, main="Normalidad para la variable edad")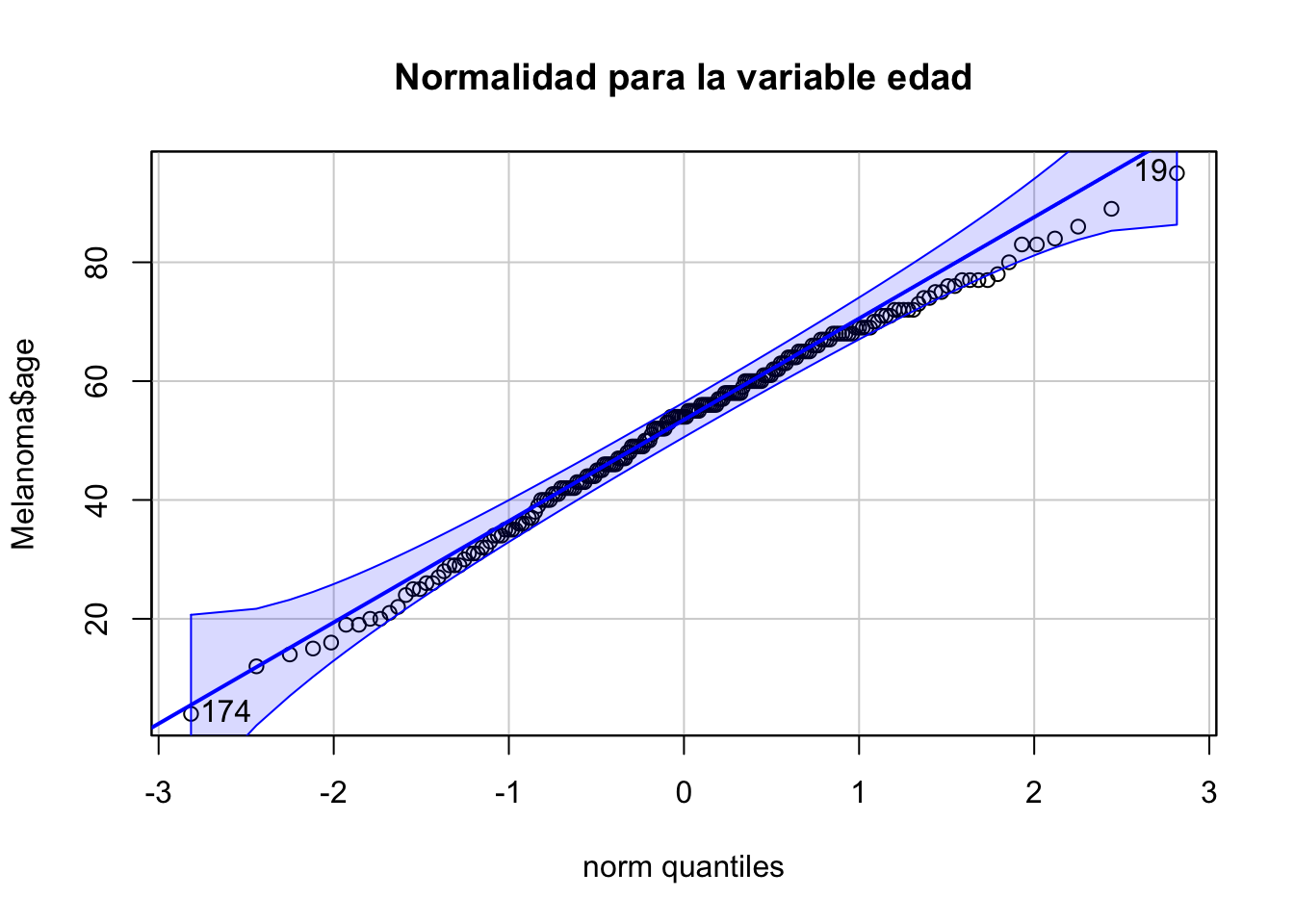
[1] 174 19Melanoma$thickness|>
car::qqPlot(main="Normalidad para el tamaño del tumor")
[1] 43 35Una maner más efectiva es utilizar rainclouds. Para ello, utilice el siguient código
install.packages("ggrain") # Si es necesariolibrary(ggrain)
library(ggplot2)
Melanoma|>
ggplot(mapping = aes(x = 1, y = thickness))+
geom_rain(fill = "#725663FF", alpha=0.5)+
theme_linedraw()
Este análisis lo podemos complementar con una prueba de hipótesis.
shapiro.test(Melanoma$thickness)
Shapiro-Wilk normality test
data: Melanoma$thickness
W = 0.76818, p-value < 2.2e-16Si requiere utilizar la pureba de Lilliefors utilice:
# install.packages("nortes")
nortest::lillie.test(Melanoma$thickness)
Lilliefors (Kolmogorov-Smirnov) normality test
data: Melanoma$thickness
D = 0.17498, p-value < 2.2e-16Paso 4 Exploración gráfica
Puede recurrir a los gráficos explorados en Capítulo 8
Los gráficos a saber son:
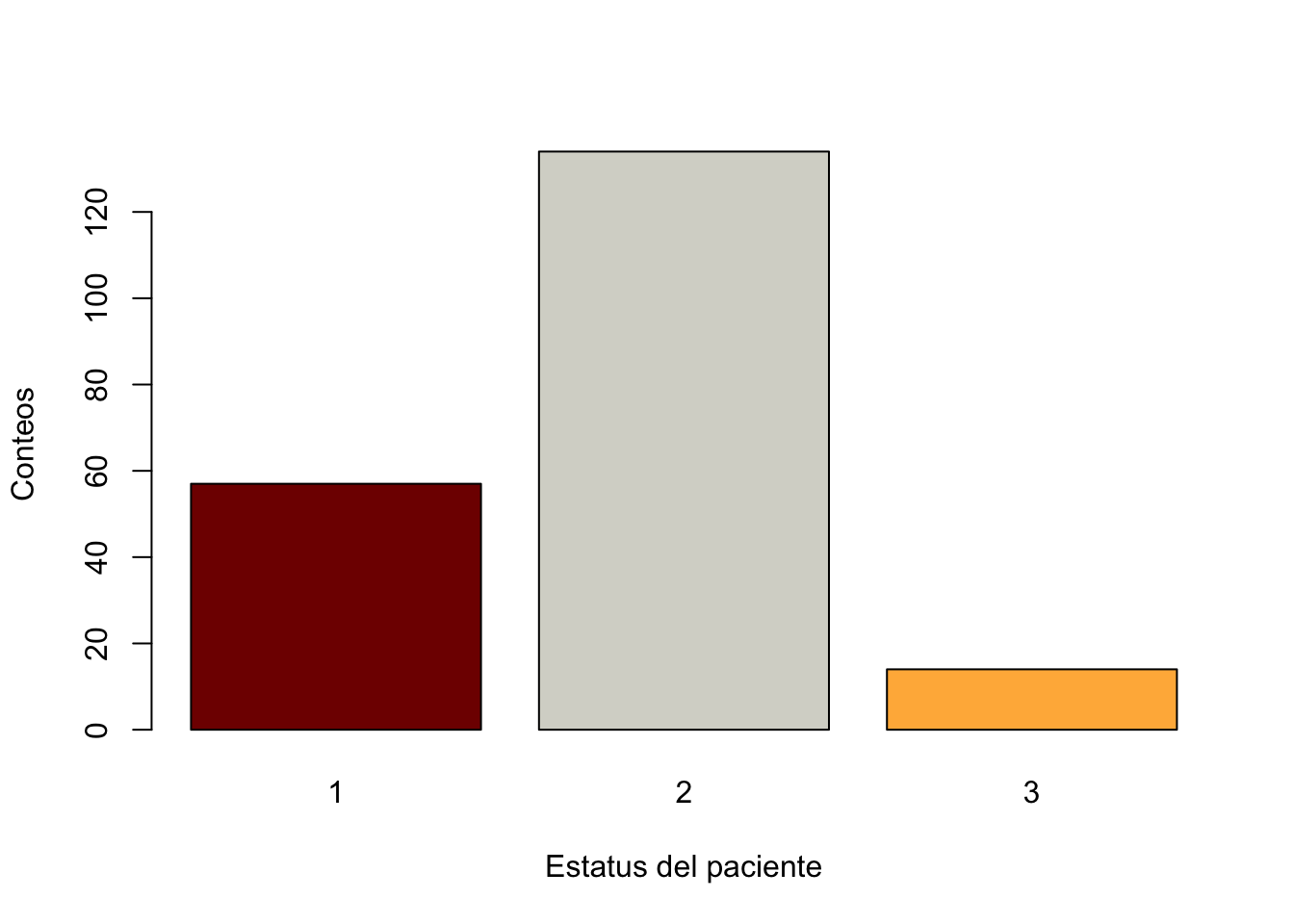


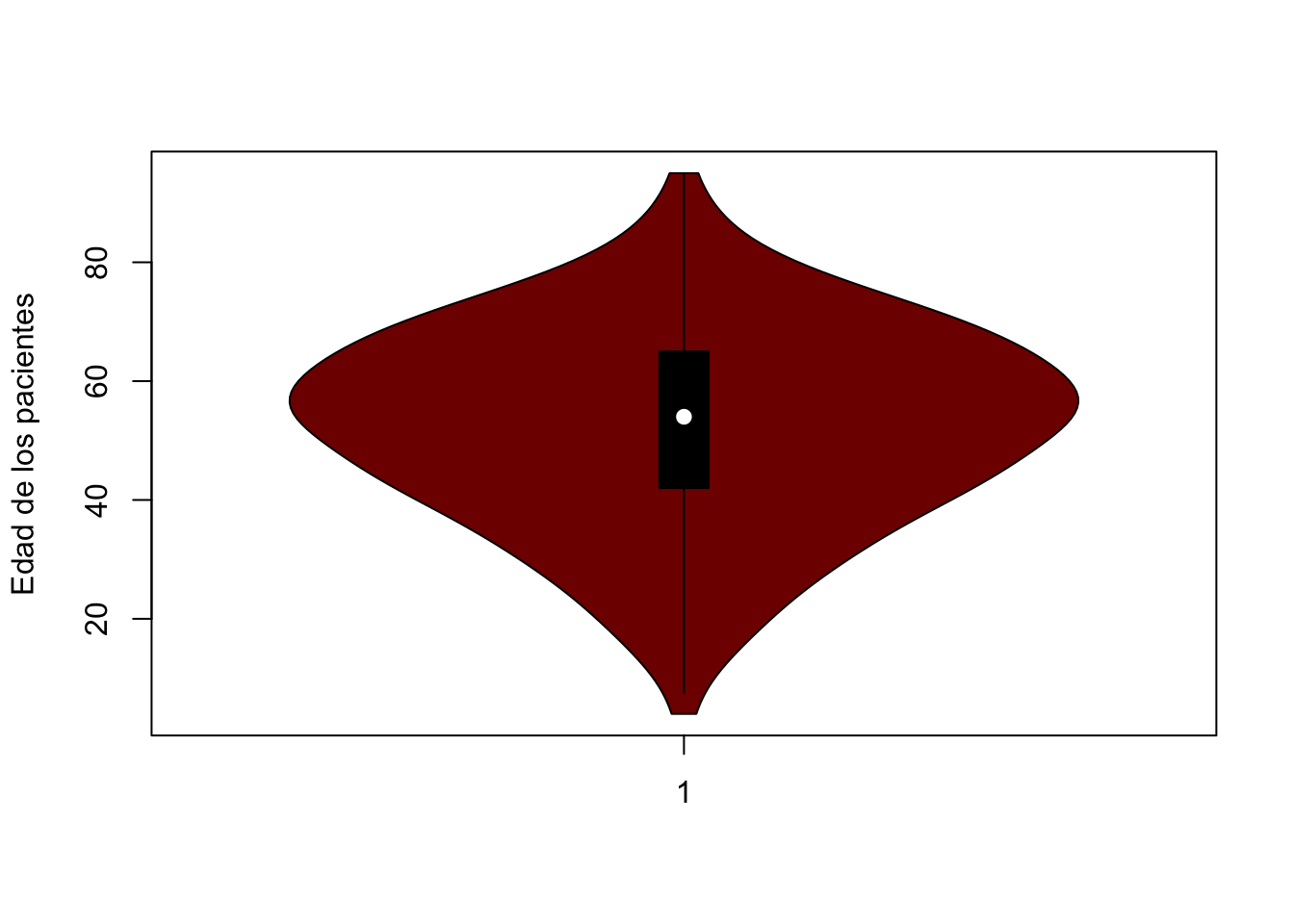
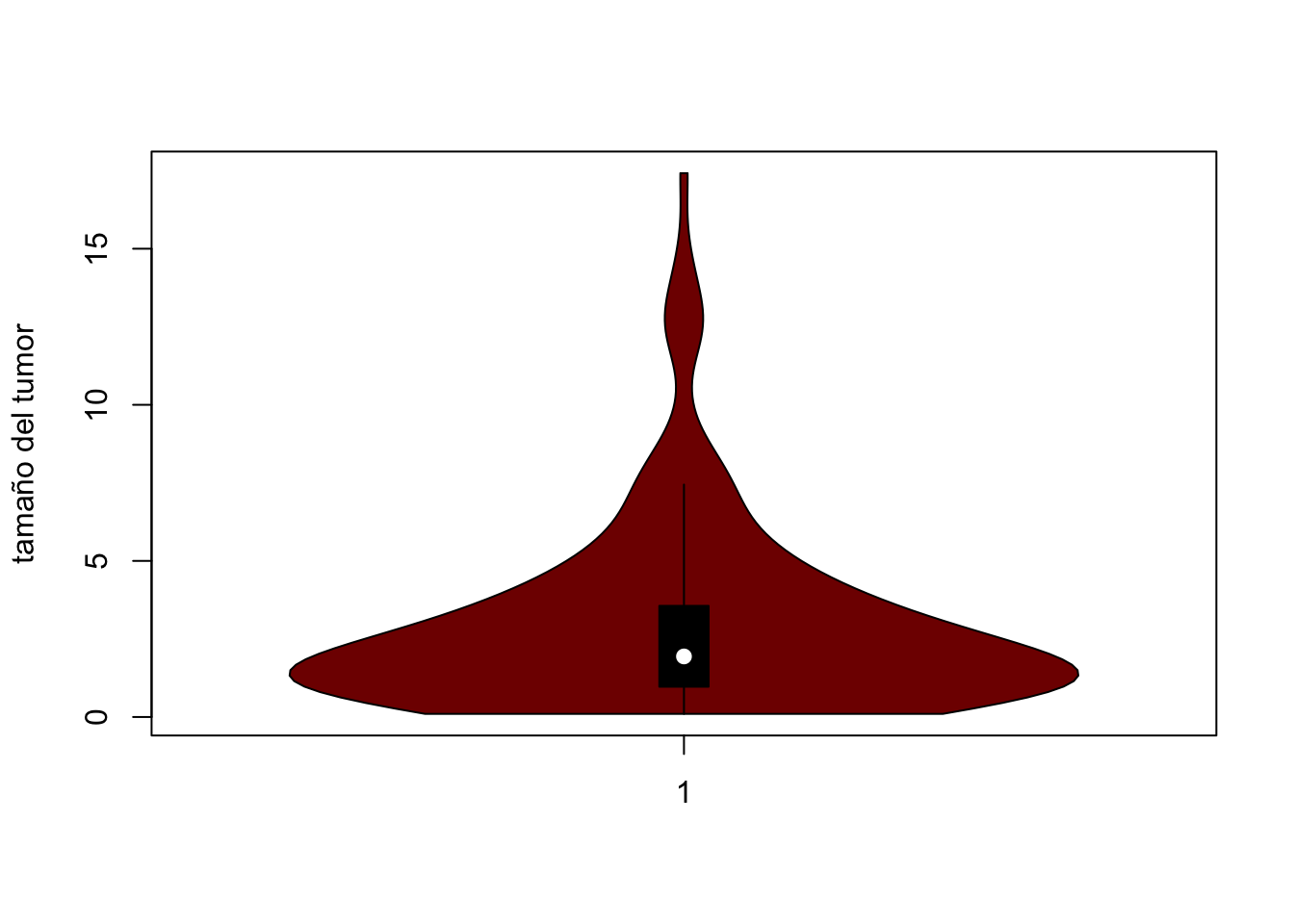
Lo ideal sería utilizar ggplot2 para ello, los gráficos se pueden construir con los siguientes códigos:
ggplot(Melanoma, aes(x = status2, fill = status2)) +
geom_bar() +
labs(title = "Distribución de Status en Melanoma",
x = "Status",
y = "Frecuencia") +
theme_minimal()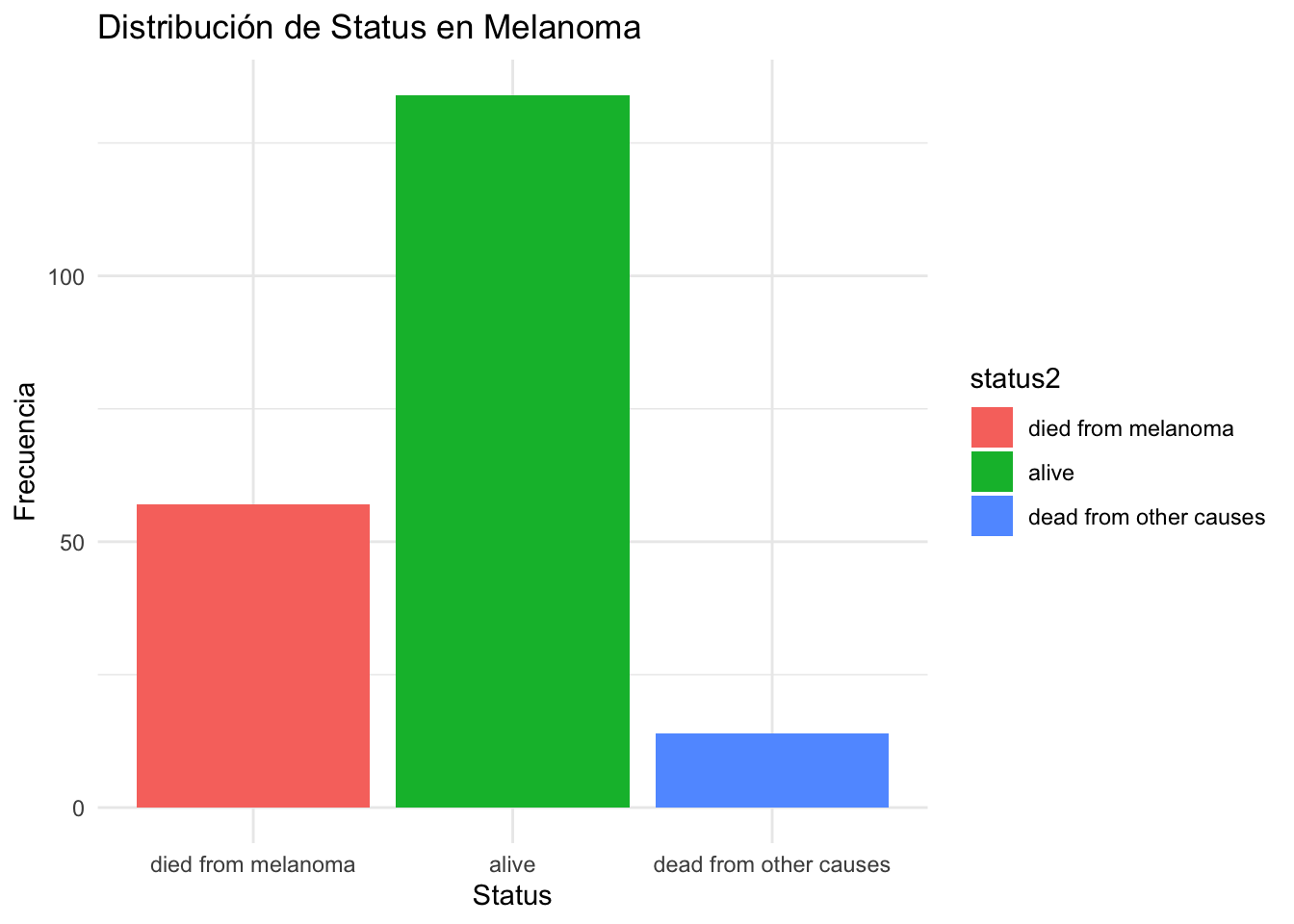
Si quiero utilizar otros colores:
Paleta <- c("#800000FF", "#D6D6CEFF", "#FFB547FF", "#ADB17DFF", "#5B8FA8FF",
"#B1746FFF", "#8A8B79FF", "#725663FF")Melanoma|>
ggplot(mapping = aes(x=status2, fill = status2))+
geom_bar()+
scale_fill_manual(values = Paleta) +
labs(title = "Distribución de Status en Melanoma",
x = "Status",
y = "Frecuencia") +
theme_minimal()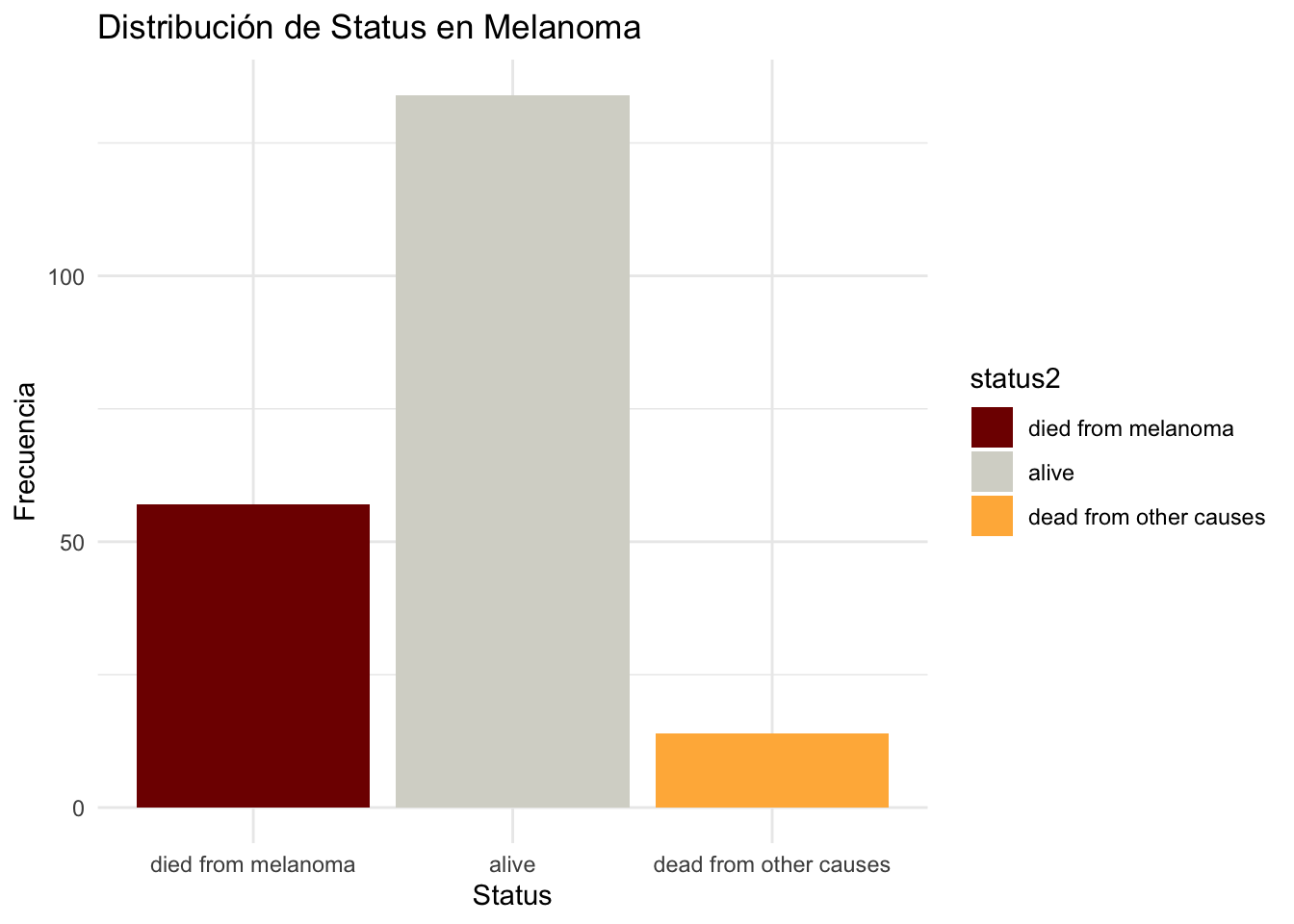
Ejercicio 10.1 Complete el resto de las gráfica utilizando ggplot2
Paso 5. Estadística descriptiva
Ejercicio 10.2 Realice estadística descriptiva de cada una de las variables
Paso 6. Formulación de hipótesis
El conocer si hay relación o no entre determinadas variables depende de los conocimientos que se tengan de las variables y del entendimiento medico-biológico de las mismas. Sin embargo, la EDA en ocasiones permite conocer si estas relaciones están presentes o no. Solo debe asegurarse de que si encuentra una posible relación esta tenga sentido biológico.
Para responder la pregunta planteada en el ejemplo (¿El tamaño del tumor influirá sobre el desenlace de los pacientes?) puede utilizar gráficas
vioplot::vioplot(Melanoma$thickness~Melanoma$status,
ylab="Tamano del tumor", xlab="Estatus del paciente",
col=paleta)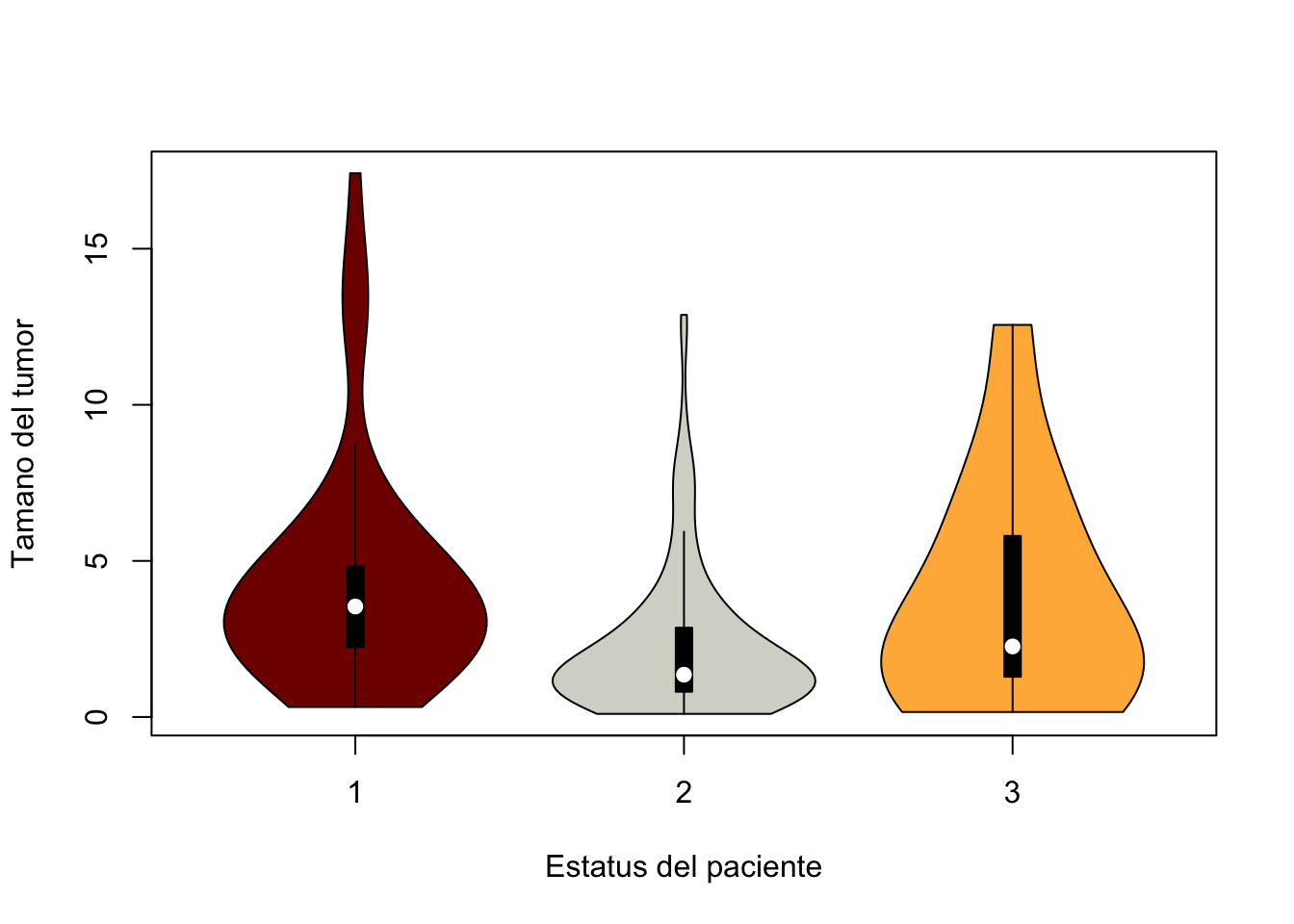
boxplot(Melanoma$thickness~Melanoma$status,
ylab="Tamano del tumor", xlab="Estatus del paciente",
col=paleta)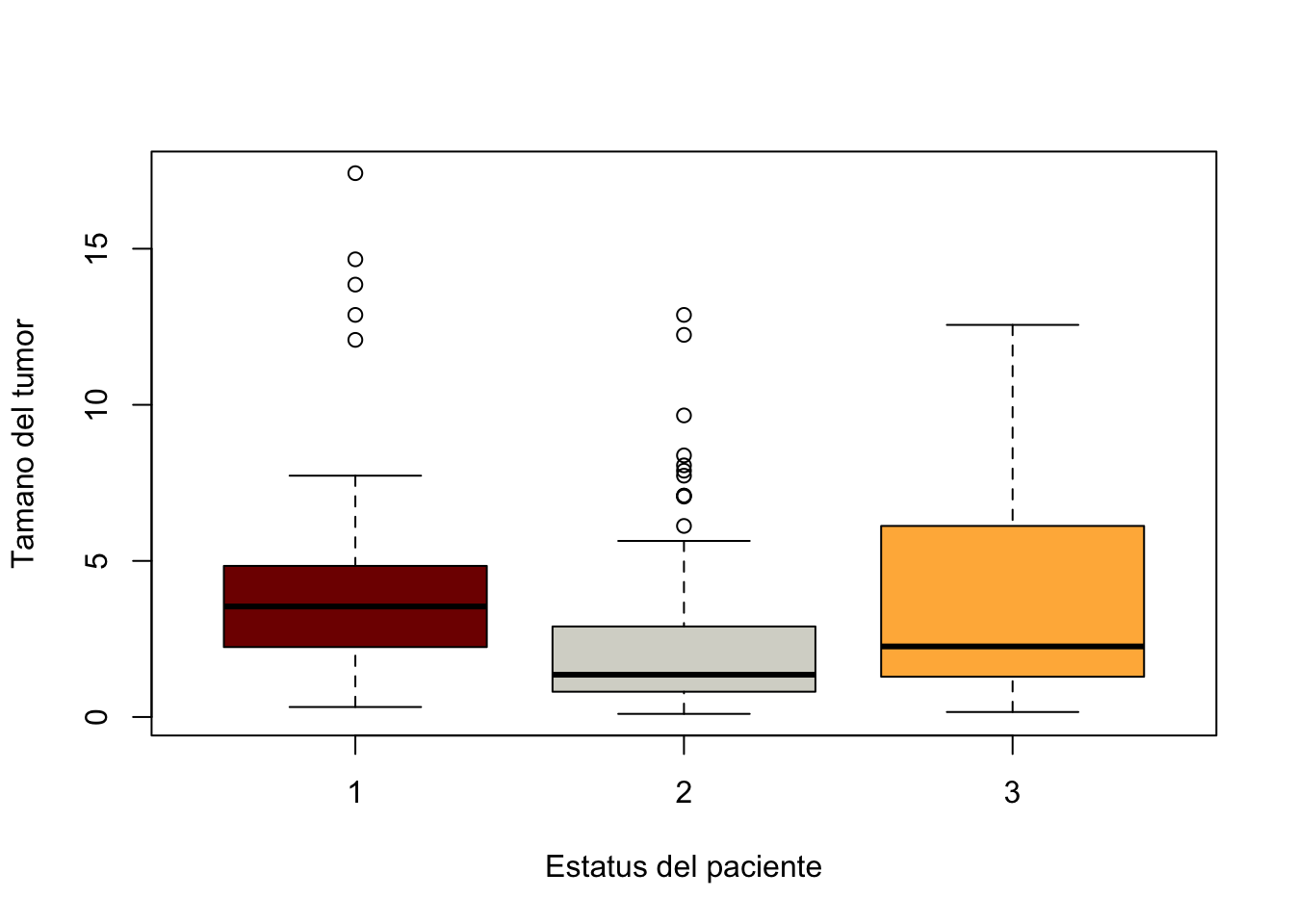
Este paso puede repetirse cuantas veces sea necesario para indentificar todas la posibles hipótesis.
10.4 EDA con otras librerías
En R hay librerías que facilitan la exploración de datos una de ellas es: DataExplorer. Es sencillo crear un reporte del Data Frame:
install.packages("DataExplorer") ## En caso de ser necesario
DataExplorer::create_report(Melanoma)10.5 Ejercicios del ?sec-EDA
Ejercicio 10.3 Utilizando la base de datos Melanoma identifique si ¿Hay relación con el tamaño del tuor la edad?.
Ejercicio 10.4 Utilizando la base de datos Melanoma identifique si ¿Hay relación con el tamaño del tumor y el sexo de los pacientes?.
Ejercicio 10.5 Utilizando la base de datos Melanoma identifique si ¿Hay relación con el tamaño del tumor y la presencia de ulceras?.
Ejercicio 10.6 Utilizando la base de datos Melanoma identifique si ¿Identifica otra relación?
Ejercicio 10.7 Realice EDA para la base datos birthwt de la librería MASS
Ejercicio 10.8 Realice EDA para la base datos biopsy de la librería MASS.
La base deo biopsy es una base de datos de cáncer de mama se obtuvo de los Hospitales de la Universidad de Wisconsin, Madison del Dr. William H. Wolberg. Evaluó biopsias de tumores de mama de 699 pacientes hasta el 15 de julio de 1992; cada uno de los nueve atributos se ha puntuado en una escala del 1 al 10, y también se conoce el resultado. Hay 699 filas y 11 columnas. Puede encontrar mayor información sobre el significado de las varibles en ayuda o en el siguiente link: https://rdrr.io/cran/MASS/man/biopsy.html
Ejercicio 10.9 Realice un EDA de la base de datos Breast Cancer Survival. Esta base de datos contiene los datos clínicos de 400 pacientes con cáncer de mama que se sometieron a cirugía para el tratamiento del cáncer. A continuación, se describe la información de todas las columnas en el conjunto de datos:
- Patient_ID: ID del paciente
- Age: Edad del paciente
- Gender: Género del paciente
- Protein1, Protein2, Protein3, Protein4: Niveles de expresión
- Tumor_Stage: Etapa del cáncer de mama del paciente
- Histology: Carcinoma ductal infiltrante, Carcinoma lobulillar infiltrante, Carcinoma mucinoso
- ER status: Positivo/Negativo
- PR status: Positivo/Negativo
- HER2 status: Positivo/Negativo
- Surgery_type: Lumpectomía, Mastectomía simple, Mastectomía radical modificada, Otro
- DateofSurgery: Fecha de la cirugía
- DateofLast_Visit: Fecha de la última visita del paciente
- Patient_Status: Vivo/Muerto
Este conjunto de datos se construyó con el objetivo de identificar variables asociadas a la sobervida de las pacientes.
La base se enceuntra disponible en:https://raw.githubusercontent.com/amankharwal/Website-data/master/BRCA.csv
Para importarla a su ambiente de trabajo solo necesita leer el archivo .csv, por ejemplo:
df <- read.csv(file = "https://raw.githubusercontent.com/amankharwal/Website-data/master/BRCA.csv")