5 Regression logistique
Objectif
Expliquer une variable réponse binaire en fonction de variables explicatives quantitatives et / ou qualitatives + effets d’ordres plus élevés (interactions…).
La fonction de modélisation principale utilisée ici est la fonction glm() qui permet d’estimer des modèles linéaires généralisés très généraux. Elle s’utilise de façon très semblable à la fonction lm() et , comme pour lm(), de multiples fonctions générique s’appliquent aux résulats de glm().
5.1 Modélisation GLM
Régression logistique simple X variable explicative qualitative ou quantitative
# si les réponses sous la forme d'un tableau nombre succès / échec / total
mod <- glm( cbind(succes,echec) ~ X, data = .., family = binomial)
# si les réponses sont sous la forme d'un vecteur de 1 et 0 : 1 ligne = 1 individu
mod <- glm( Y ~ X, data = .., family = binomial) Régression logistique multiple
Quand il y a plusieurs variables explicatives avec ou sans effets d’ordres plus élevés (interactions…), l’équation du modèle s’écrit comme dans le cas des modèles linéaires lm().
5.2 Visualiser les données avant modélisation GLM
Si X quantitative
- Plot p ~ X où p est la proportion de Y = 1 (=succès).
- Table de fréquence.
Si X qualitative
- Barplot Y ~ X
- Table de fréquence.0
5.3 Fonctions utiles sur un objet de type mod <- glm()
La plupart des fonction disponible sur les modèles de type lm() s’appliquent aussi aux modèles glm().
Pour rappel :
| Résumé du modèle | summary(mod) |
| Mise à jour du modèle | drop1(mod, ..) + update(mod, ..) |
| Coefficients + IC |
coef(mod) + confint(mod) ou car::Confint(mod) |
| Résidus / Valeurs ajustées | residuals(mod, ..) / fitted(mod) |
| Matrice Var-Cov des paramètres | vcov(mod) |
| Matrice X du modèle | mod.matrix(mod) |
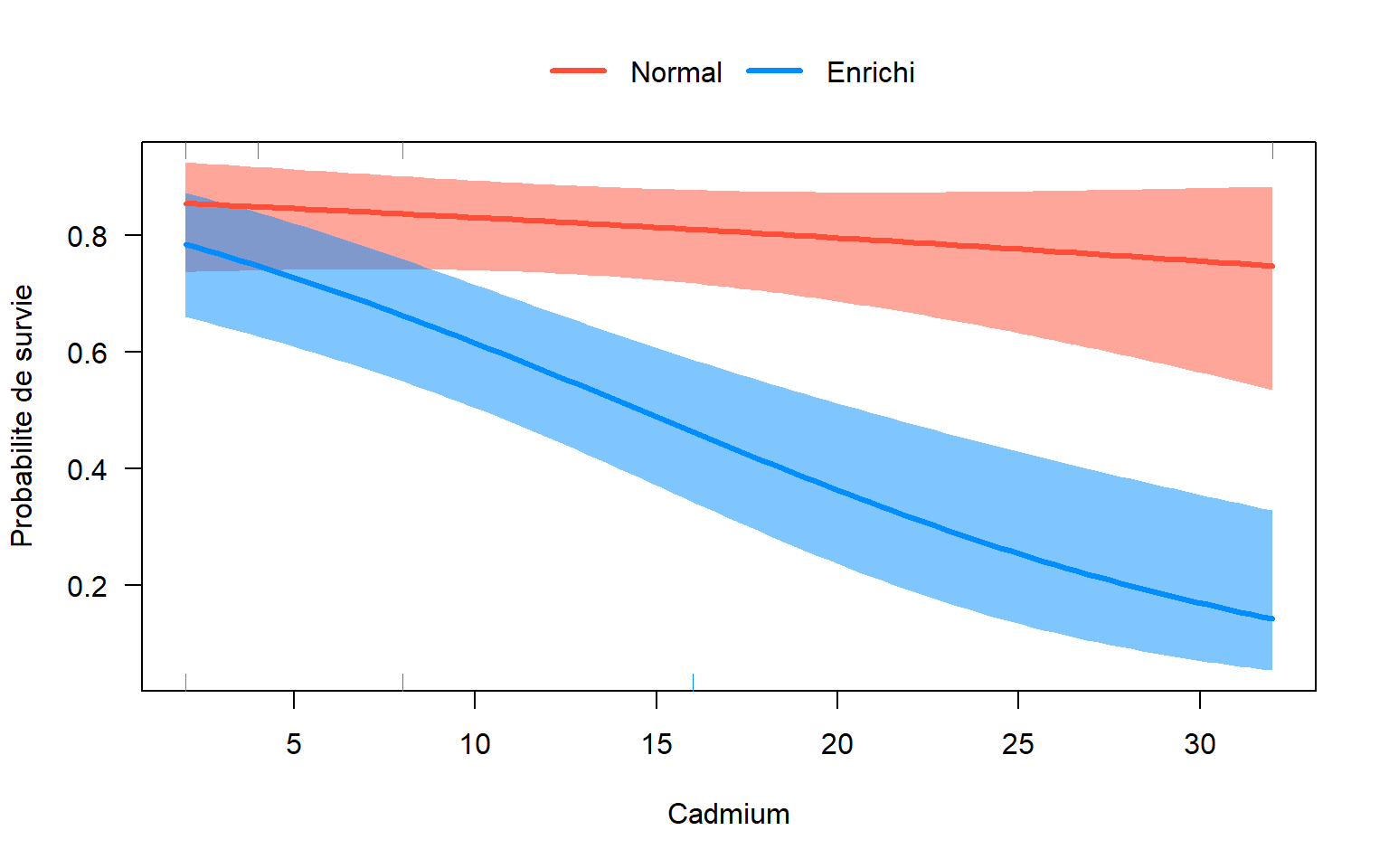
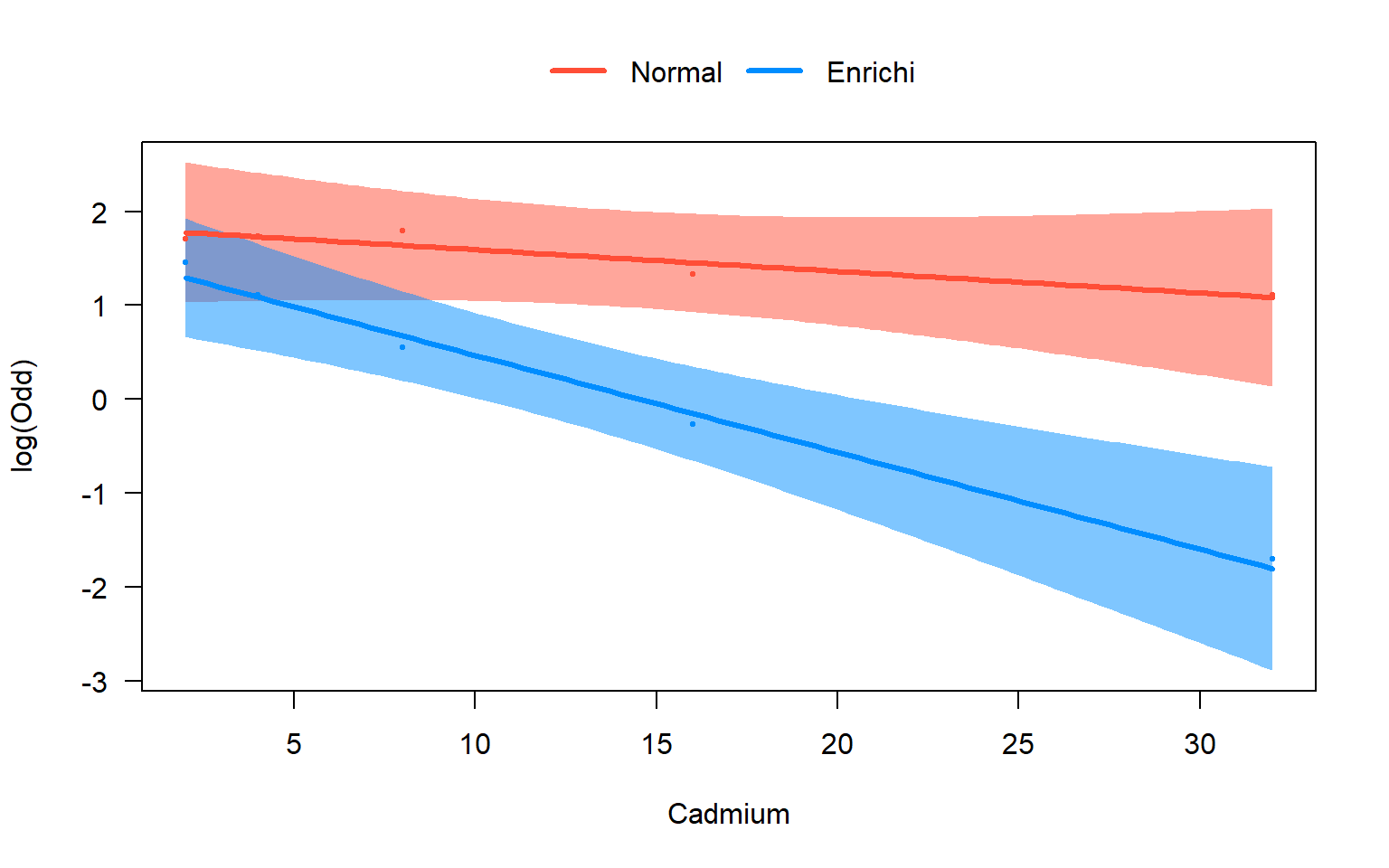
| Visualiser la modélisation | visreg::visreg(mod, ..) |
| Prédictions | predict(mod, ..) |
5.4 Données du chapitre GLM
omega3Cadmium.txt |
Etude de l’interaction entre nutriments bénéfiques pour la santé Omega3 (1 var. quali. à 2 niv.) et contaminants a priori nocifs Cadmium (1 var. quanti.) sur la survie de cellules. |
Ces données ne sont pas des données individuelles mais sont agrégées par groupe.
# Représentation graphique des données
PS=100*data[,"Survie_Oui"]/(data[,"Survie_Oui"]+data[,"Survie_Non"])
OM=as.numeric(data[,"Omega3"])
plot(data[,"Cadmium"],PS,col=OM,pch=OM,xlab="X = Cadmium",ylab="% succès",main="Données pour régression logistique")
legend(1,40,legend=levels(data[,"Omega3"]),col=1:2,pch=1:2)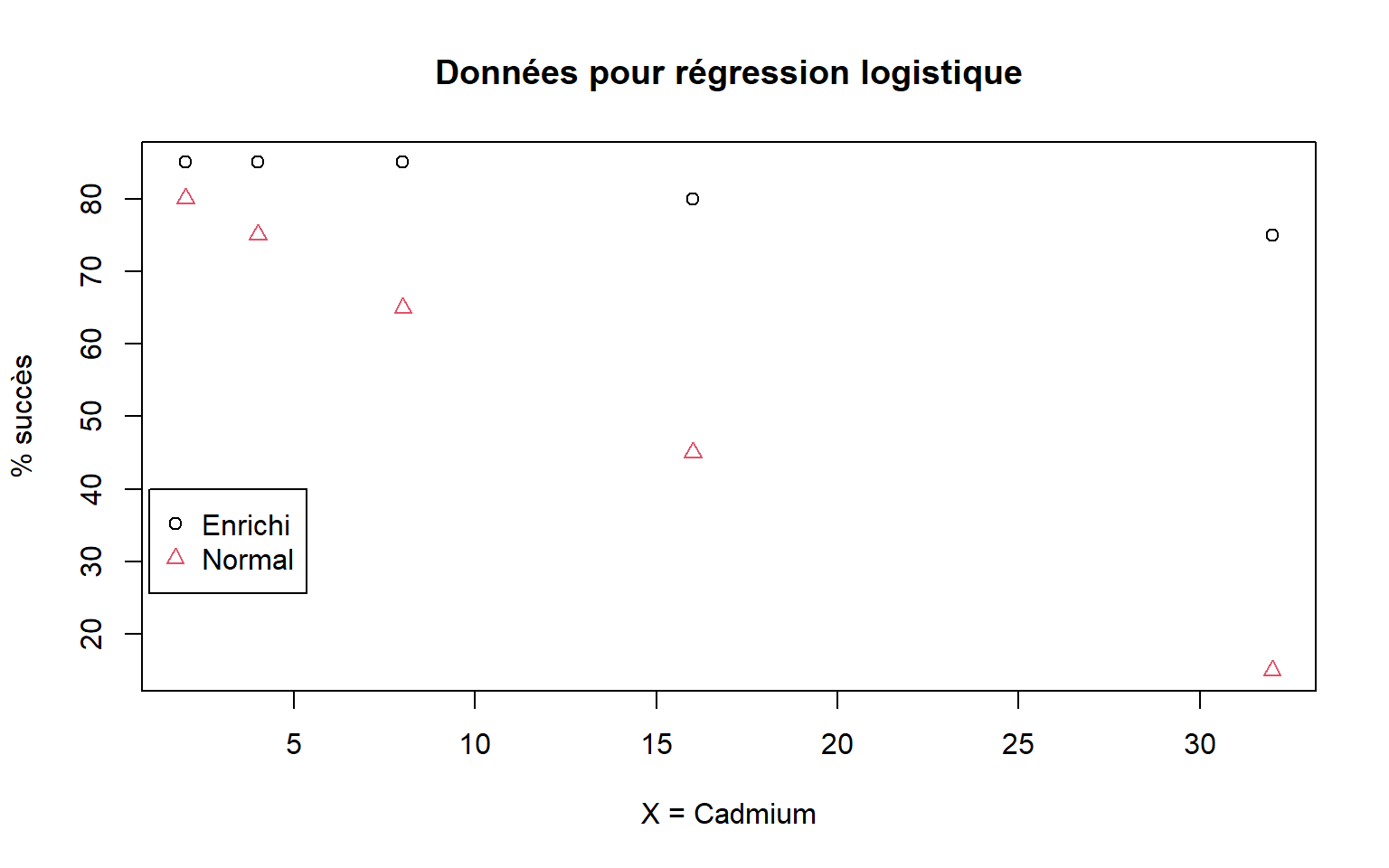
5.5 Utiliser summary() sur glm
Call:
glm(formula = cbind(Survie_Oui, Survie_Non) ~ Cadmium * Omega3,
family = binomial, data = data)
Deviance Residuals:
Min 1Q Median 3Q Max
-0.12008 -0.10088 0.01398 0.08504 0.16546
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 1.82160 0.41425 4.397 1.1e-05 ***
Cadmium -0.02298 0.02267 -1.014 0.3107
Omega3Normal -0.32059 0.54736 -0.586 0.5581
Cadmium:Omega3Normal -0.08021 0.03323 -2.413 0.0158 *
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 41.56439 on 9 degrees of freedom
Residual deviance: 0.10979 on 6 degrees of freedom
AIC: 38.758
Number of Fisher Scoring iterations: 45.6 Utiliser drop1()/update() sur glm
Utilisation similaire aux modèles linéaires lm(). La seule différence est dans les tests disponibles:
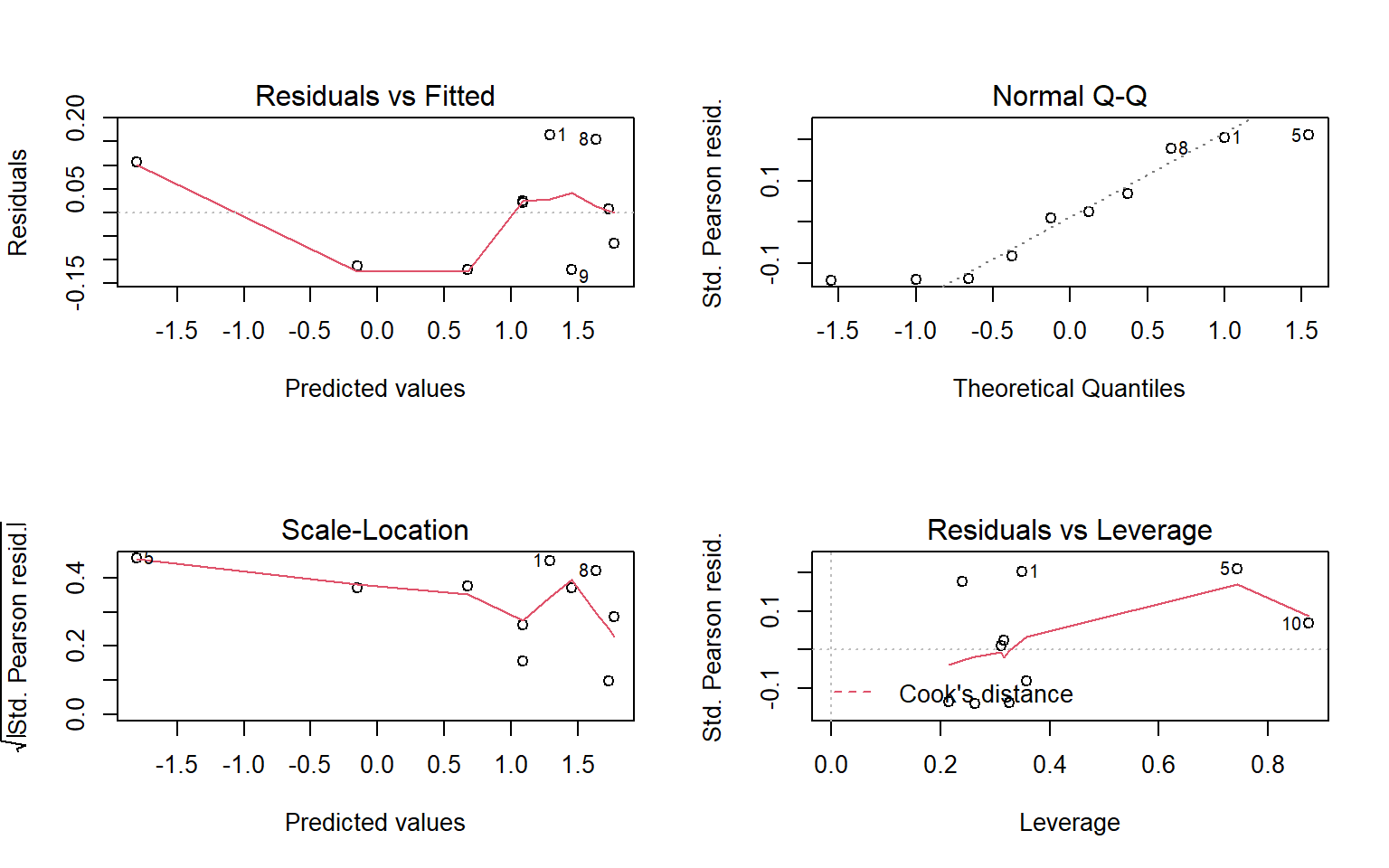
5.7 Utiliser residuals() sur glm
Pour obtenir les valeurs numériques des résidus
residuals(mod, type = "deviance") # Résidus de déviance
residuals(mod, type = "pearson") # Résidus de PearsonGraphe des résidus de pearson
5.8 Utiliser visreg() sur glm
5.9 Utiliser predict() sur glm
Obtenir les prédictions pour (Omega3, Cadmium) = (“Normal”, 12) , (“Enrichi”,12)
xnew <- data.frame("Omega3" = c("Normal", "Enrichi"), "Cadmium" = c(12, 12))
pred <- predict(mod, newdata = xnew, type = "response", se.fit = TRUE)
res <- data.frame(xnew,
"Fit" = pred$fit,
"Lwr" = pred$fit - 1.96 * pred$se.fit,
"Upr" = pred$fit + 1.96 * pred$se.fit)
pander(res)| Omega3 | Cadmium | Fit | Lwr | Upr |
|---|---|---|---|---|
| Normal | 12 | 0.5653 | 0.4546 | 0.676 |
| Enrichi | 12 | 0.8243 | 0.7487 | 0.8999 |