5 Image Preprocessing of fMRI
5.1 先備知識
5.1.1 相關軟體
RadiAnt DICOM Viewer:用以瀏覽 DICOM 的軟體
MRIcro:亦為用以瀏覽 DICOM 的軟體
Statistical Parametric Mapping (SPM 12):一個用以資料前處理、分析的 MATLAB toolbox
5.1.2 常見的 fMRI protocol
同節 2.2.3,常見的 fMRI protocol 如下:
Single‐Shot 2D EPI (GRE‐EPI), T2* weighting
Repetition Time \(= \rm 2000 ms\)
Echo Time \(=\rm 20 ms\)
Flip Angle \(\rm = 70\)–\(90^\circ\)
NEX \(= 1\)
Slice thickness \(\rm = 3.4 mm\)
Field of View \(\rm = 220 \times 220 mm^2\)
Matrix size \(= 64 \times 64\)
Slice number \(= 40\)
Volume number \(= 240\)–\(360\)(取決於實驗設計)
5.1.3 DICOM (1993) 與 DICOM format
DICOM 全名為 Digital Imaging and COmmunication in Medicine,是醫學設備的共通格式,在 1993 年由 ACR 與 NEMA 提出。
DICOM 格式由 header 與 image data 組成,前者即包含了各式的參數,後者則是影像部分。 一開始我們有 DICOM format (*.dcm, *.ima),但為了分析資料,要將其轉成 NIfTI (.nii) 或 Analyze75 (*.hdr, *.img)。
5.2 資料前處理
5.2.1 前處理程序
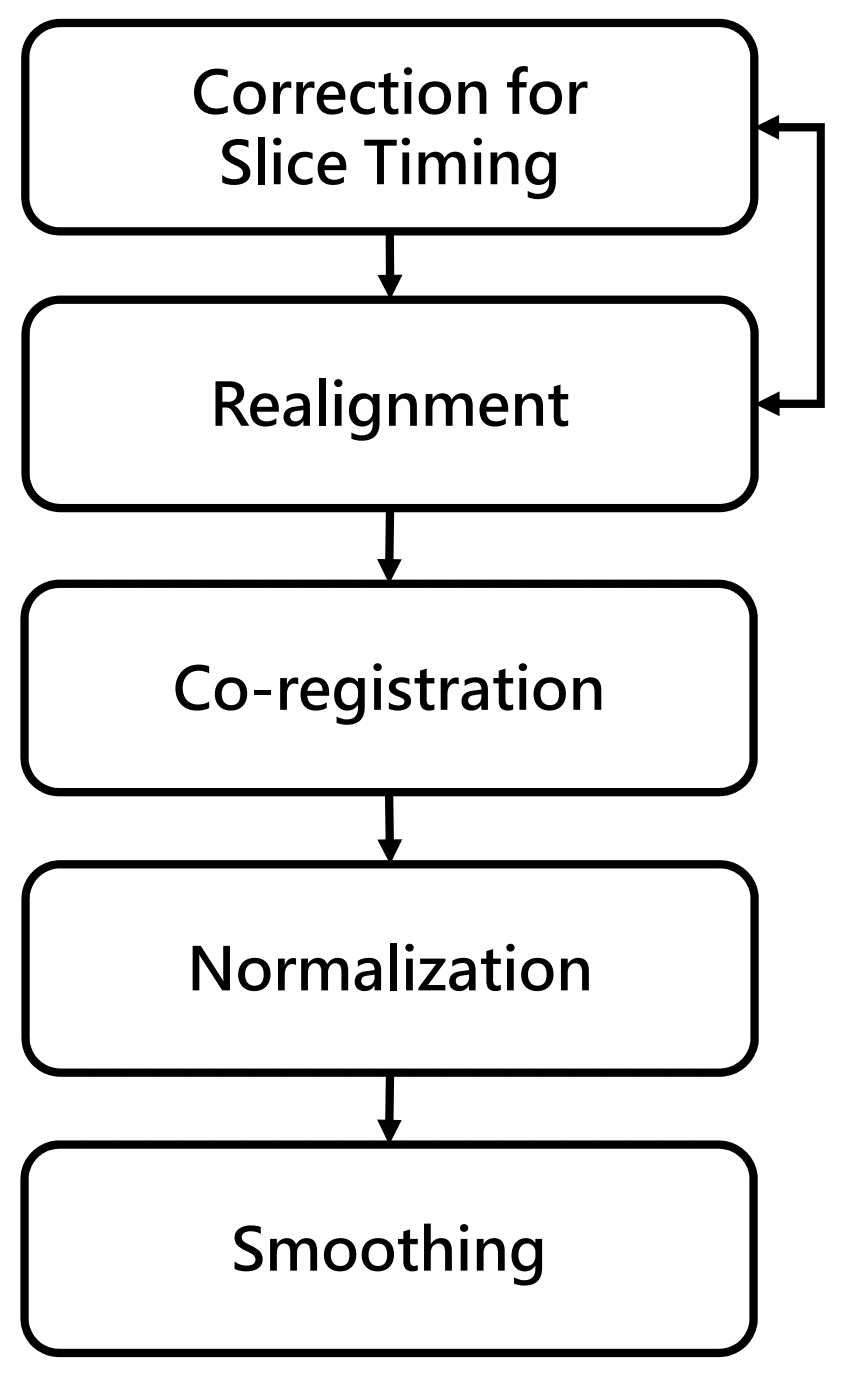
Figure 5.1: 資料前處理的程序。
Slice timing:校正時間的差異。會在檔名前面加上 “a”,即
a*.nii。Realignment:校正腦的位置。會在檔名前面加上 “r”,即
ra*.nii,還會產生一個rp_*.txt檔。Co‐registration (with anatomical images):fMRI 犧牲掉的空間解析度,在此步驟透過解剖影像可以部分挽回。檔名不變。
Normalization:把不同大小、形狀的頭轉換到統一的座標上。會在檔名前面加上 “w”,即
wra*.nii。Smoothing:會降低空間解析度,但可以降低 noise(增進 single noise ratio)。會在檔名前面加上 “s”,即
swra*.nii。Segment (tissue classification; optional):分類或識別各個不同的組織或腦區。會產生
c1\*.nii、c2\*.nii、c3\*.nii、mws\*.nii、BiasField_\*.nii。
5.2.2 匯入 DICOM
安裝完 SPM 12 以後,在 MATLAB 的 command line 輸入 spm fmri,以開啟 SPM 12。此外,先在 MATLAB 的路徑列輸入資料所在的資料夾,方便等會使用 SPM 時選取資料。
Figure 5.2: SPM 12’s Menu.
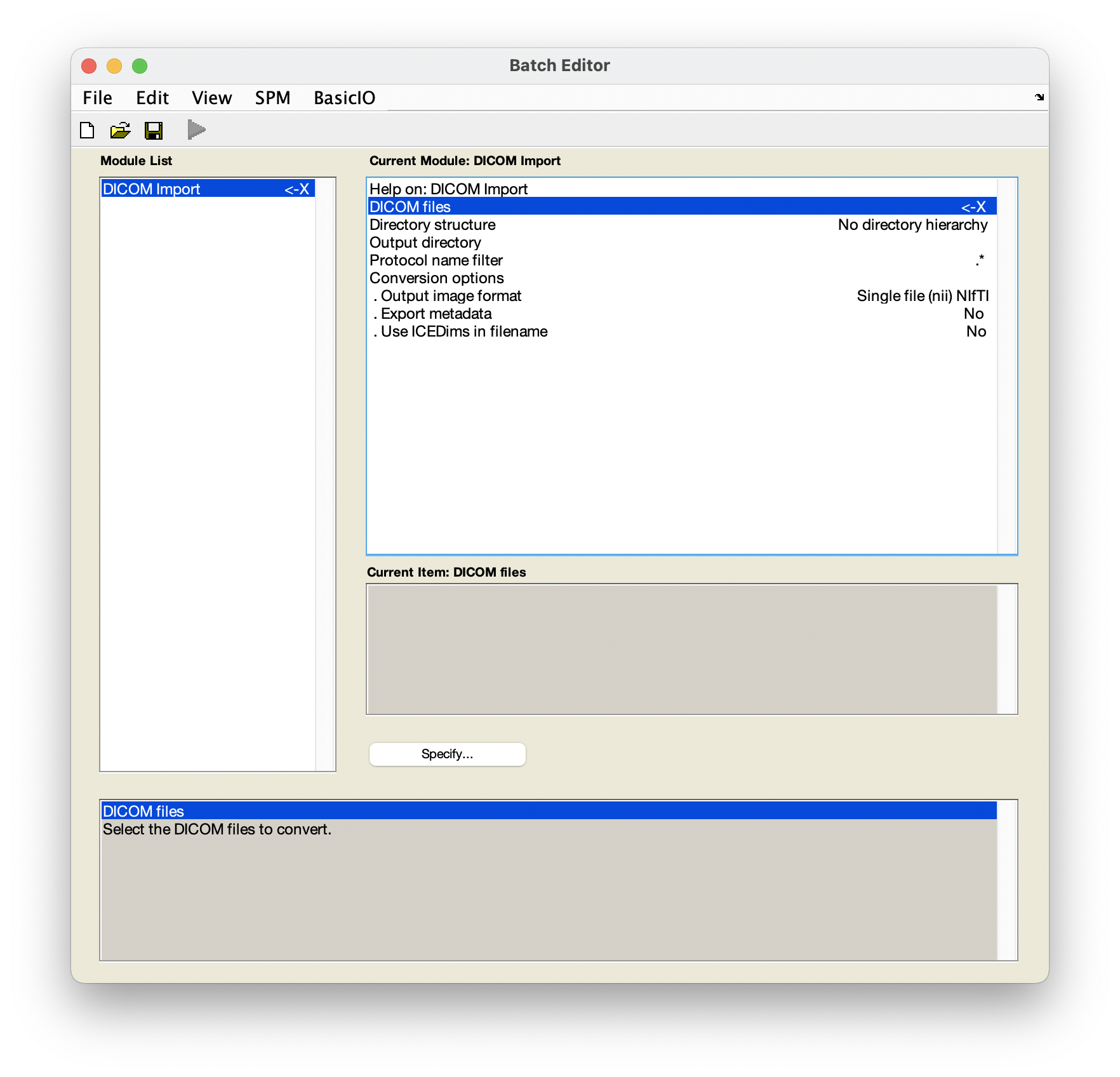
Figure 5.3: SPM 12’s Batch Editor.
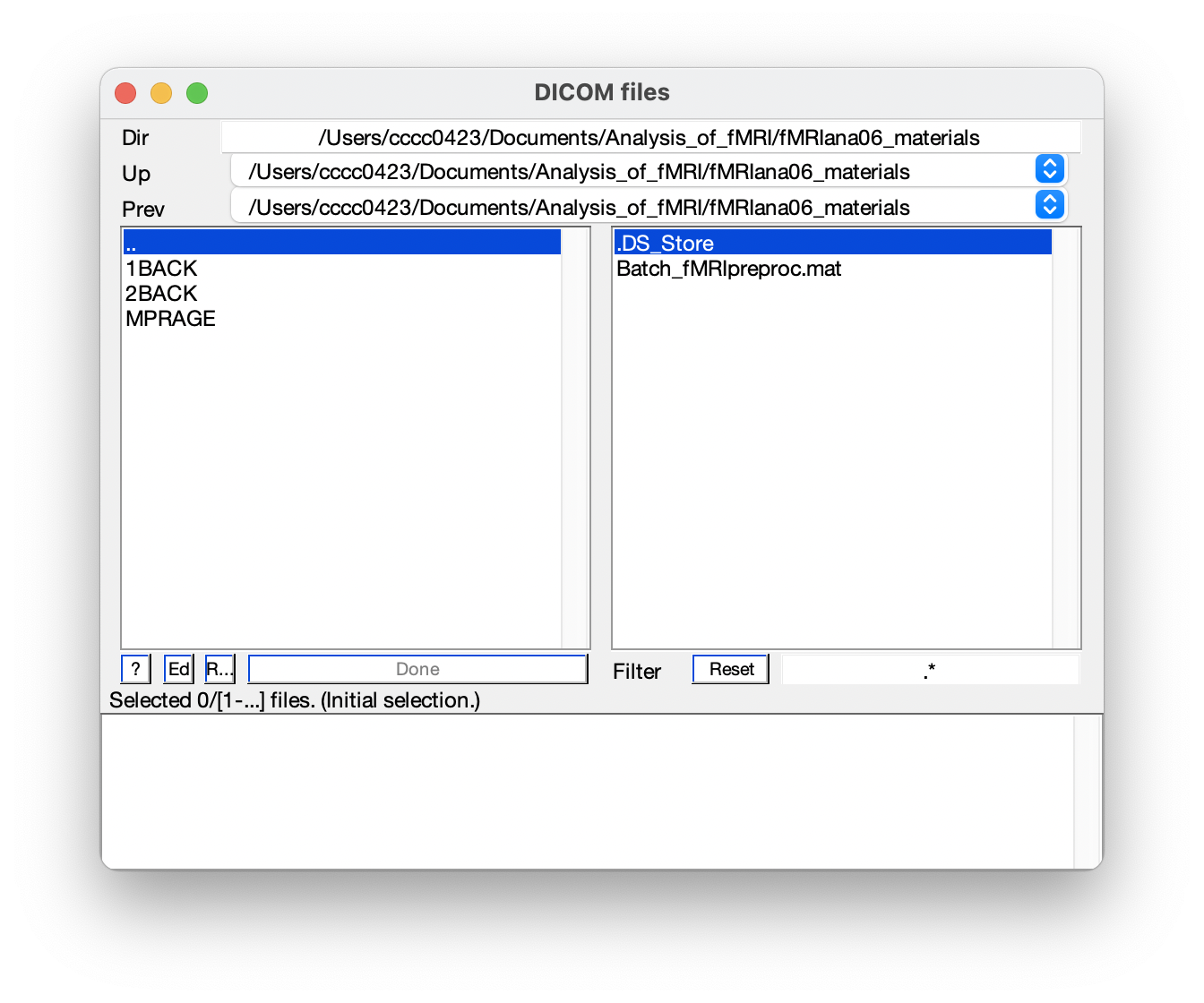
Figure 5.4: SPM 12’s DICOM files.
要注意的是,在 DICOM files 視窗中,要選取檔案只需要單擊(而不是雙擊)。
..意味著上層資料夾;.則是現在的資料夾。如果要選取整個資料夾的檔案,只需要在右欄點擊右鍵,再按下Select All即可。然後按下Done可以回到原先的 Batch Editor 視窗。在 Batch Editor 視窗中,如圖 5.3,有 output directory 選項。我們可以在原路徑下新增一個資料夾,作為輸出的路徑,並在其中新增幾個子資料夾來分類資料,如圖 5.5。然後選取剛剛新增的資料夾作為 output directory。
Figure 5.5: 新增用以輸出檔案的資料夾。
- 接著,我們還要匯入另一種 DICOM 檔,即結構影像(structure image)。要加入另一個 DICOM 檔,得從 Batch Editor 的工具列依序選取
SPM、Util、Import與DICOM Import,如圖 5.6。從此我們可以得到另一個可供我們輸入 DICOM 檔的函數。此函數用以輸入 MPRAGE 檔。仿效前述步驟,在 DICOM files 選取 MPRAGE 檔,並把 output directory 改成剛剛新增的資料夾。
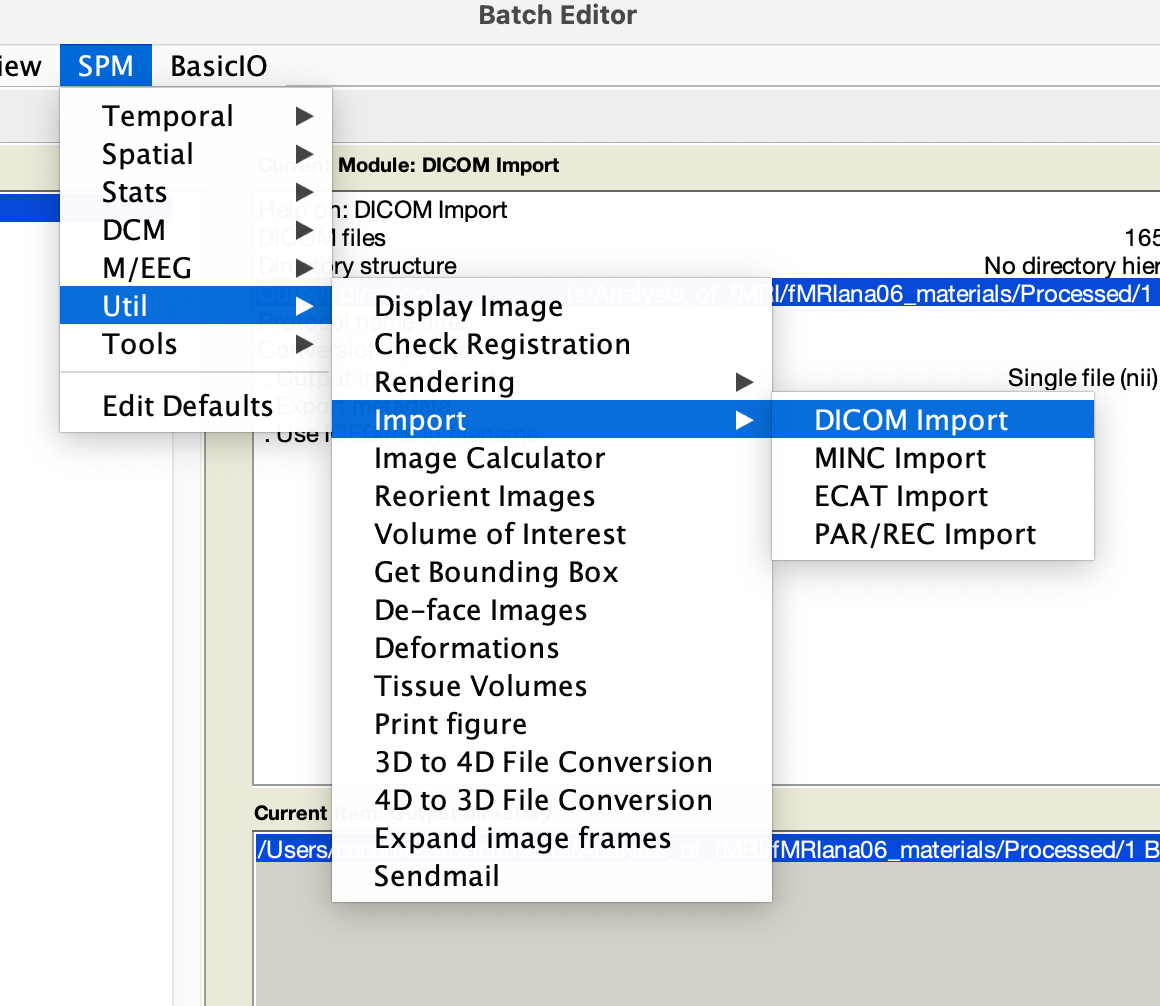
Figure 5.6: 新增另一個輸入 DICOM 的函數。
Figure 5.7: 轉檔中。
- 除了可以用 Menu(圖 5.2)中左下角的
Display查看輸出的檔案,我們也可以使用 MRIcro,其功能更多。
5.2.3 Batch of SPM fMRI preprocessing
- 點擊 Menu(圖 5.2)中右下角的
Batch,開啟 Batch Editor。我們可以在左欄載入我們需要的 modules。在此,我們在左上角的Load Batch中載入現成的 batch process 檔Batch_fMRIpreproc.mat,得到如圖 5.8 的畫面。我們同時可以發現,只有三個 modules 為<-X,即需要輸入檔案;其他都是DEP,即 “dependency”,不需輸入檔案。
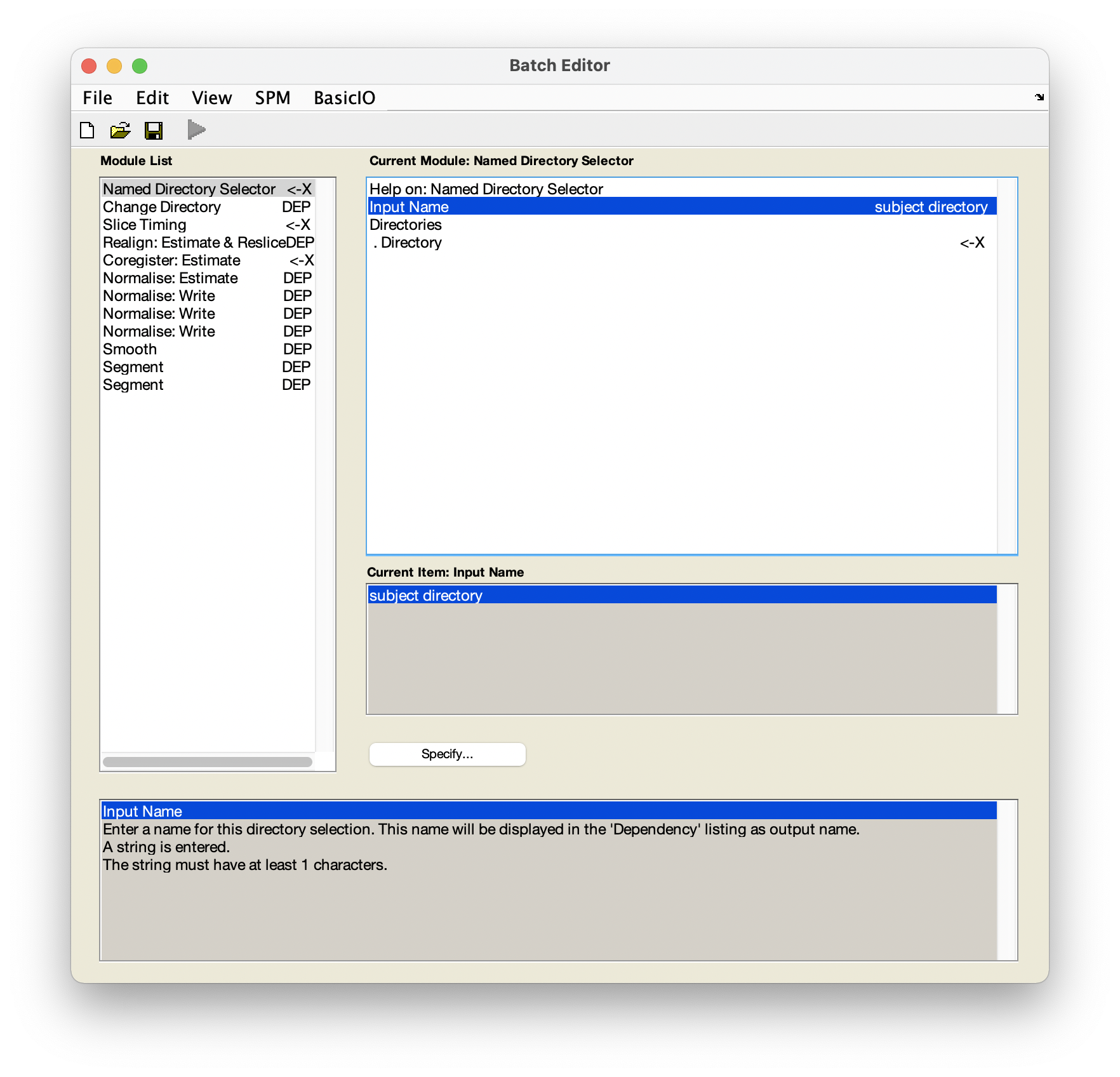
Figure 5.8: 載入現成的 batch process 檔。
點選 Batch Editor(圖 5.8)中左欄的
Normalise: Estimate,並其中的Tissue probability map的路徑修改為 SPM 12 的資料夾下的子資料夾tpm中的TPM.nii。仿效以上步驟,點選 Batch Editor(圖 5.8)中左欄的兩個
Segment,並修改各個Tissue probability map的路徑。要注意的是,Segment中的每個TPM.nii必須是依序的不同的 6 個 maps。我們可以選取 Tissue probability map 視窗中右下角的空格輸入1:6,按下鍵盤Enter後會變成[1 2 3 4 5 6],而右上欄會出現TPM.nii,1至TPM.nii,6的六個檔案,如圖 5.9。然後依序修改六個Tissue probability map的路徑到該六個檔案。
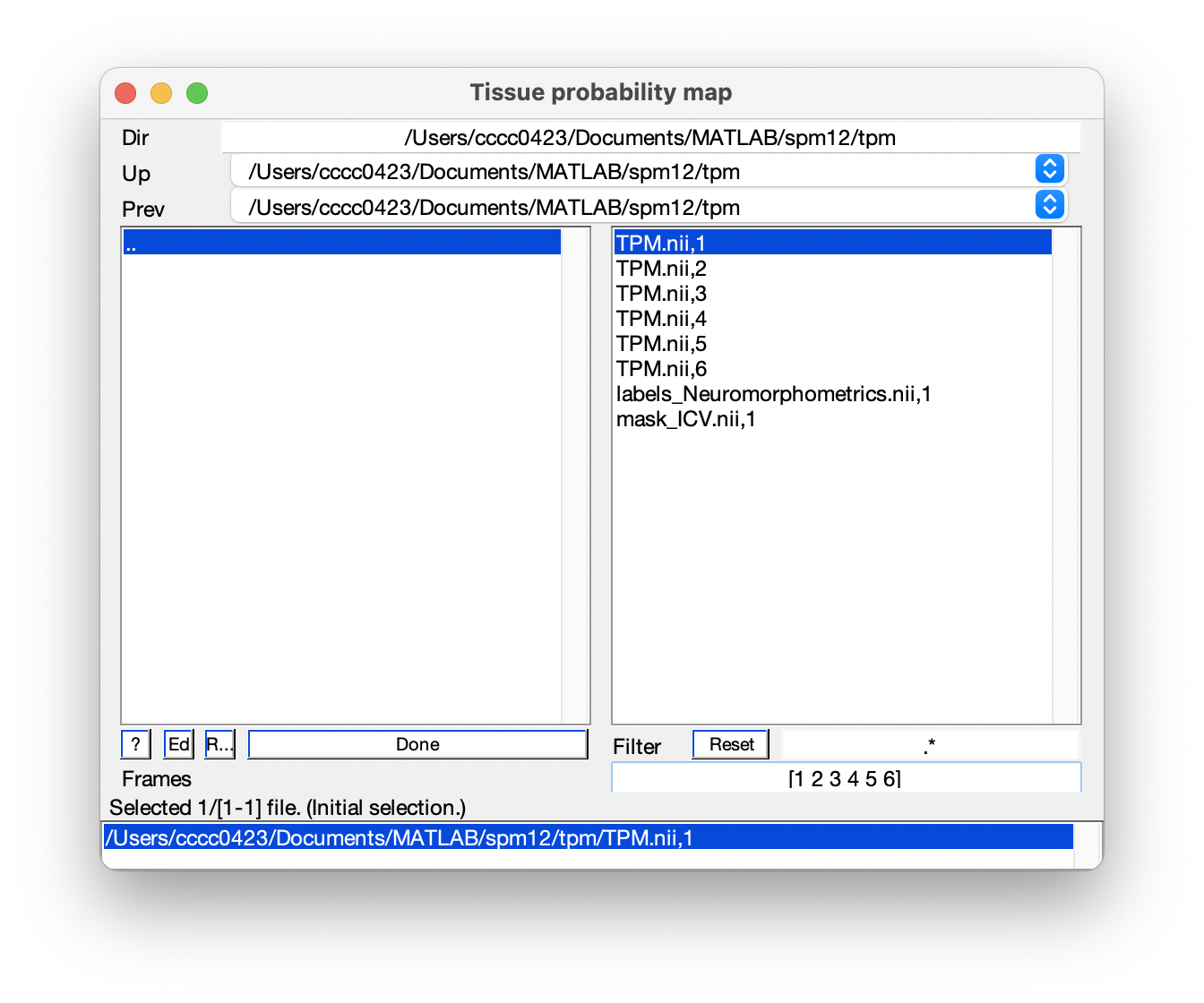
Figure 5.9: Segment 中的每個 TPM.nii 必須是依序的不同的六個 maps。
完成之後,點選 Batch Editor(圖 5.8)的工具列下方的
Save Batch,儲存這個 Batch 檔,避免每次都要重新修改路徑。此時有左欄還有三個 modules 有
<-X,表示待輸入檔案。選擇Named Directory Selector。把Named Directory Selector中的Directory所輸入的路徑改成剛剛轉檔後輸出的資料夾的路徑,如圖 5.10。
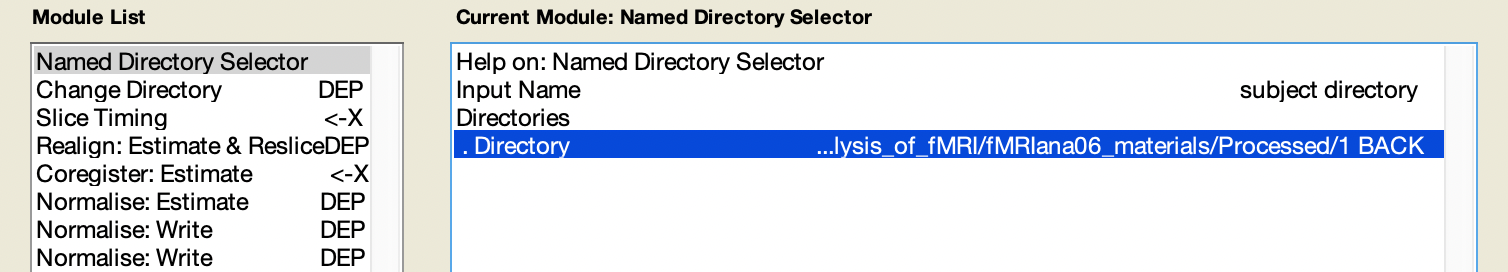
Figure 5.10: Named Directory Selector 中的 Directory。
- 在
Slice Timing的Session輸入剛剛轉檔後的那 165 個.nii,如圖 5.11。
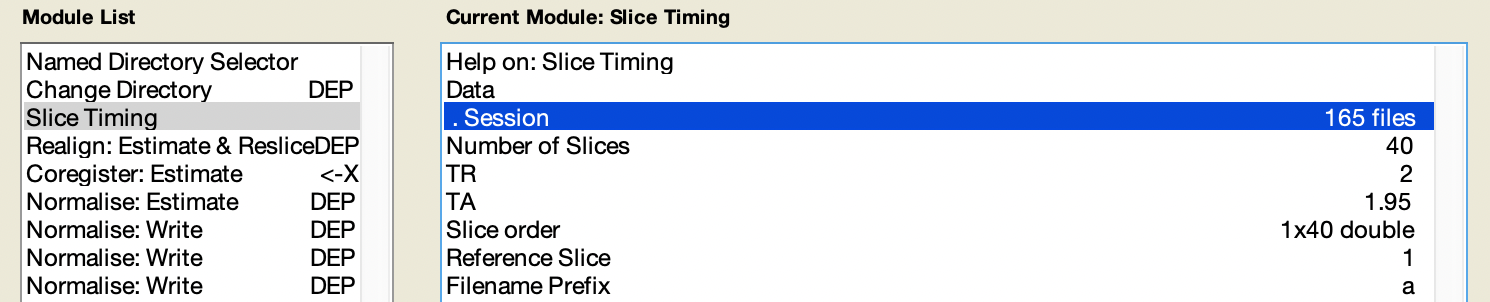
Figure 5.11: Slice Timing 中的 Session。
- 接著處理
Coregister: Estimate的部分。此步驟要輸入的是結構影像。所以我們先複製一個剛剛處理過的MPRAGE資料夾,然後丟到1 BACK資料夾中,避免等等各個不同 Sessions 的檔案混在一團。然後我們把Coregister: Estimate中的Source Image的路徑改成剛剛複製到1 BACK的MPRAGE中的結構影像檔,如圖 5.12。
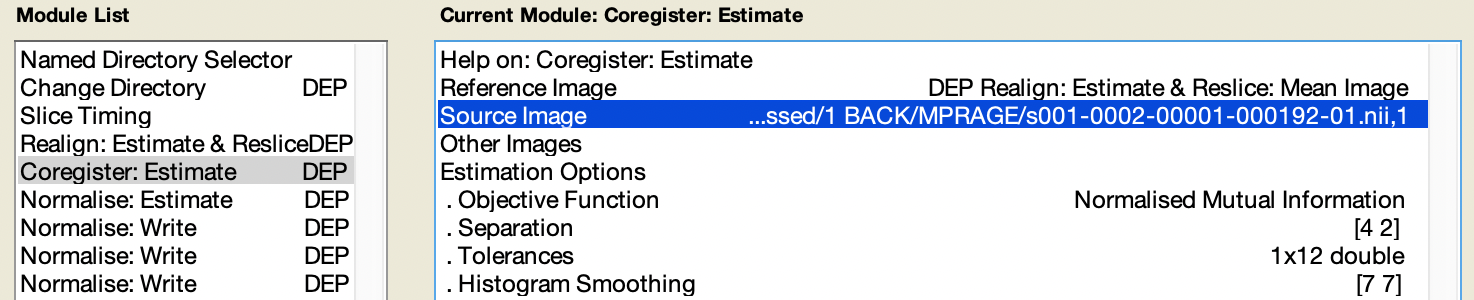
Figure 5.12: Coregister: Estimate 中的 Source Image。
- 按下 Batch Editor 上方的綠色播放鍵圖示
Run Batch。我們可以在 MATLAB 的 Command Window 查看處理進度。
這些步驟背後的意義何在?
5.2.4 Correction for slice timing
即便我們聲稱我們有時間解析度 2 秒,意思是我們每 2 秒可以獲取一張全腦的影像。但即使在同一個時點,每個 slice 所獲取到的時間還是有差別,例如也許每張 slice 之間都都差個 5 毫秒左右。
進行 slice timing 的校正就如圖 5.13。縱軸是不同的 slice,經過轉換會稍微移動其對應到的橫軸座標。
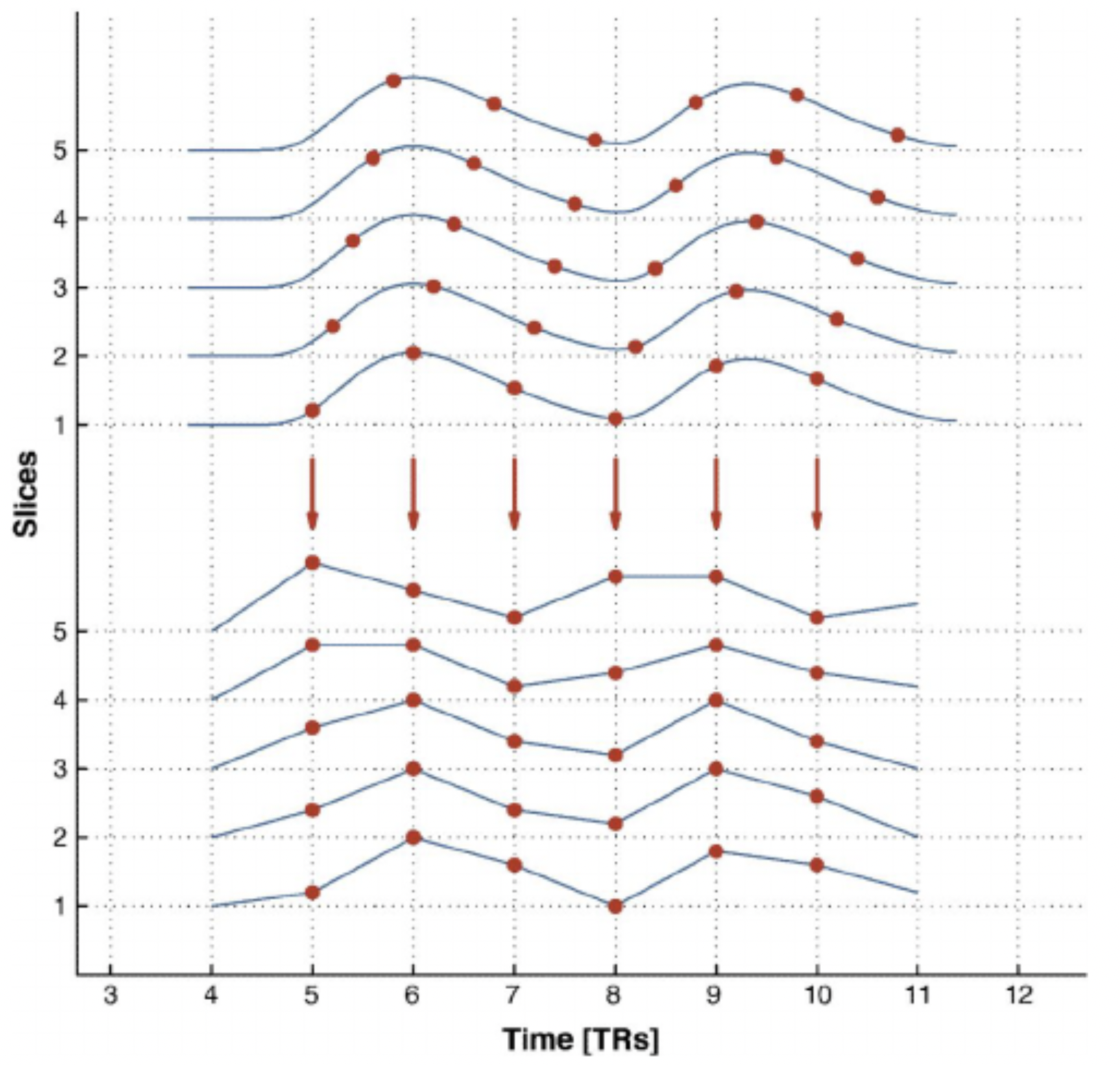
Figure 5.13: Correction for slice timing. 圖源:Sladky et al (2011).
5.2.5 Realignment of head motion
因為受試者的頭可能會動,所以我們需要重新將其重新對齊。
我們可以將頭移動的程度量化成 translation 與 rotation,如圖 5.14(縱軸是長度與角度,橫軸是不同的時點),而前者即 head movement,分為 \(x\)、\(y\)、\(z\) 三種方向;後者即頭旋轉的方向,分成 pitch、roll、yaw 三種方向。三種 translations 與三種 rotations 如圖 5.15。
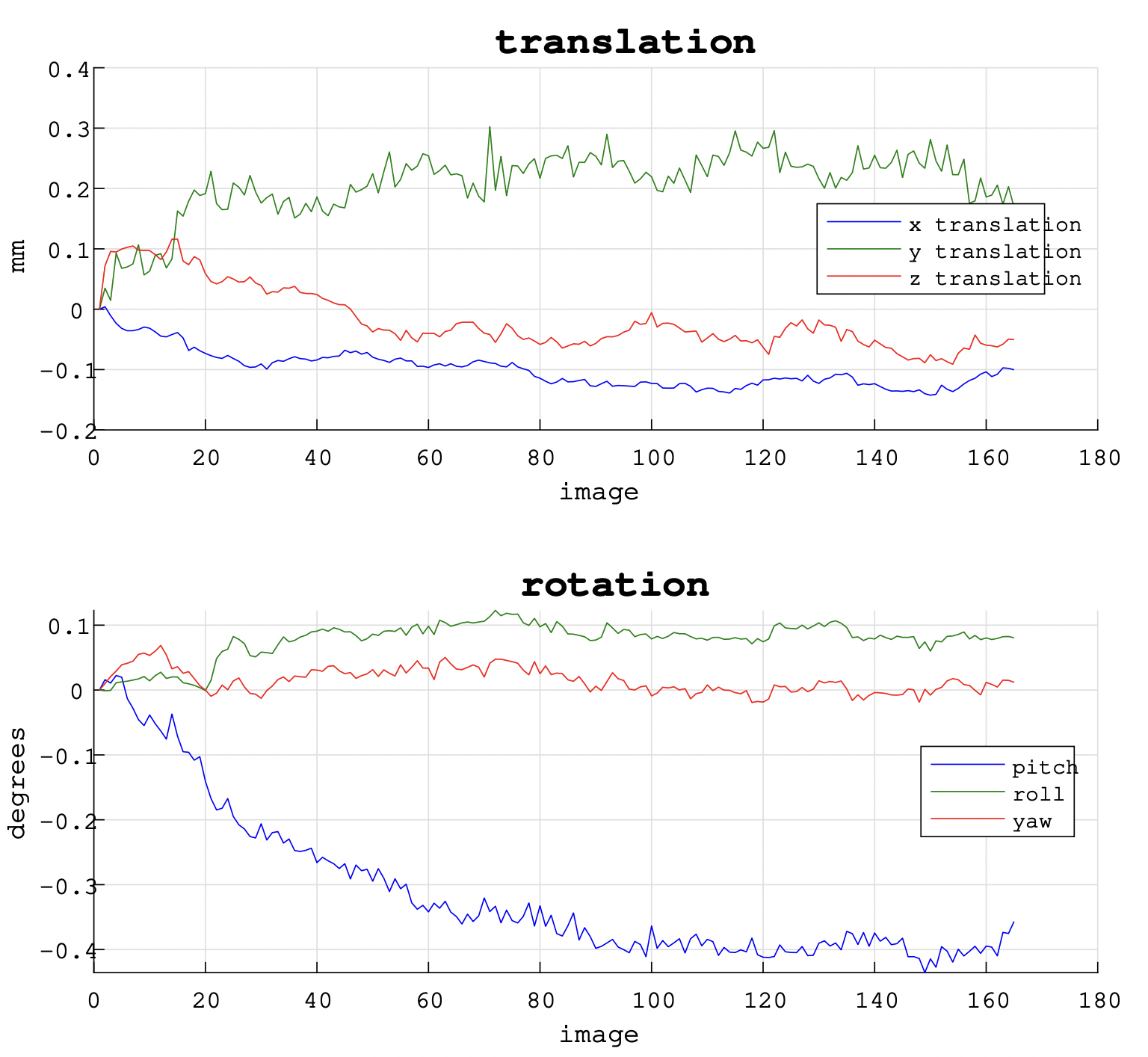
Figure 5.14: Translation and rotation.
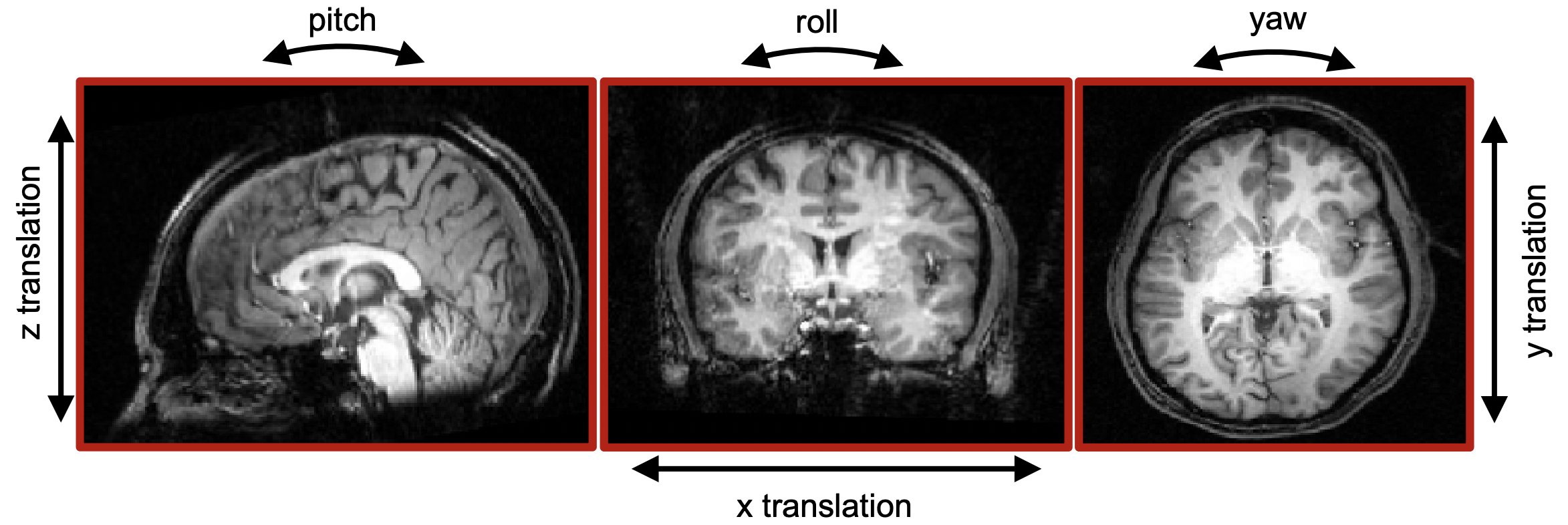
Figure 5.15: 三種 translations 與三種 rotations 的方向。
如果受試者的 translation 大於 \(\rm 3mm\),或者 rotation 超過 \(\rm 3^\circ\),那我們就得排除掉此資料。因為太過嚴重的 head motion 會大過 hemodynamic response,造成分析上的困難。
5.2.6 Co‐registration
把結構影像(T1W)與 fMRI 影像(EPI)合併。結構影像的空間解析度高,而 fMRI 的影像的空間解析度低。在此步驟即將這兩種資料合併到同一個座標上,如圖 5.16。而 SPM 使用 rigid body transformation,rigid body 意即不會真的去改變或調整內在的結構,只會改變 translation 或 rotation。因為兩種資料是來自同一個受試者,所以這種轉換並不會改變形狀。SPM 也能讓使用者手動調整,但一般而言使用自動的轉換就夠了。
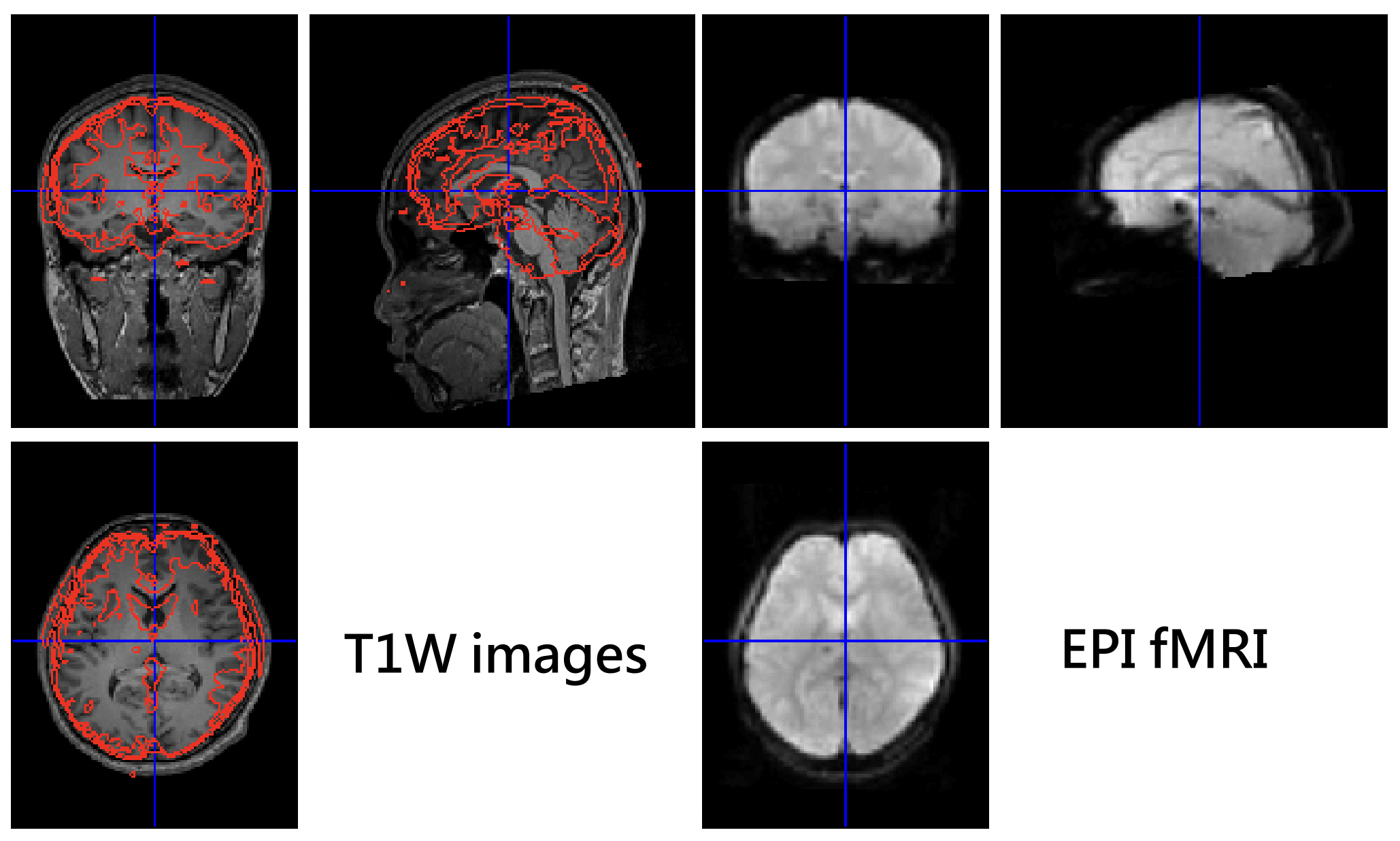
Figure 5.16: 合併 T1W 與 EPI 到同一個座標上。
5.2.7 Spatial normalization
之所以要進行 spatial normalization,是因為如果要在受試者之間比較的話,每個受試者的腦的座標都有點差異,我們必須進行標準化。我們可以用 T1 或 EPI 資料來進行 spatial normalization。雖然 SPM 也能使用後者來進行標準化,但前者會得到更好的結果,因為其空間解析度更高。而圖 5.17 中的 T1 template 之所以那麼糊,是因為其 “over smoothing” 了,以 152 個樣本的腦代表人腦平均的樣貌。此步驟與 “co‐registration” 中的轉換不同,“co‐registration” 中只有線性的轉換,但此步驟中為了統一空間的座標,所以將進行非線性的轉換。
我們在進行 T1 的轉換以後,會有一個 transformation matrix,記錄了我們如何把一個特定的座標轉換成標準化的座標,就能將其應用在 EPI 的轉換上。
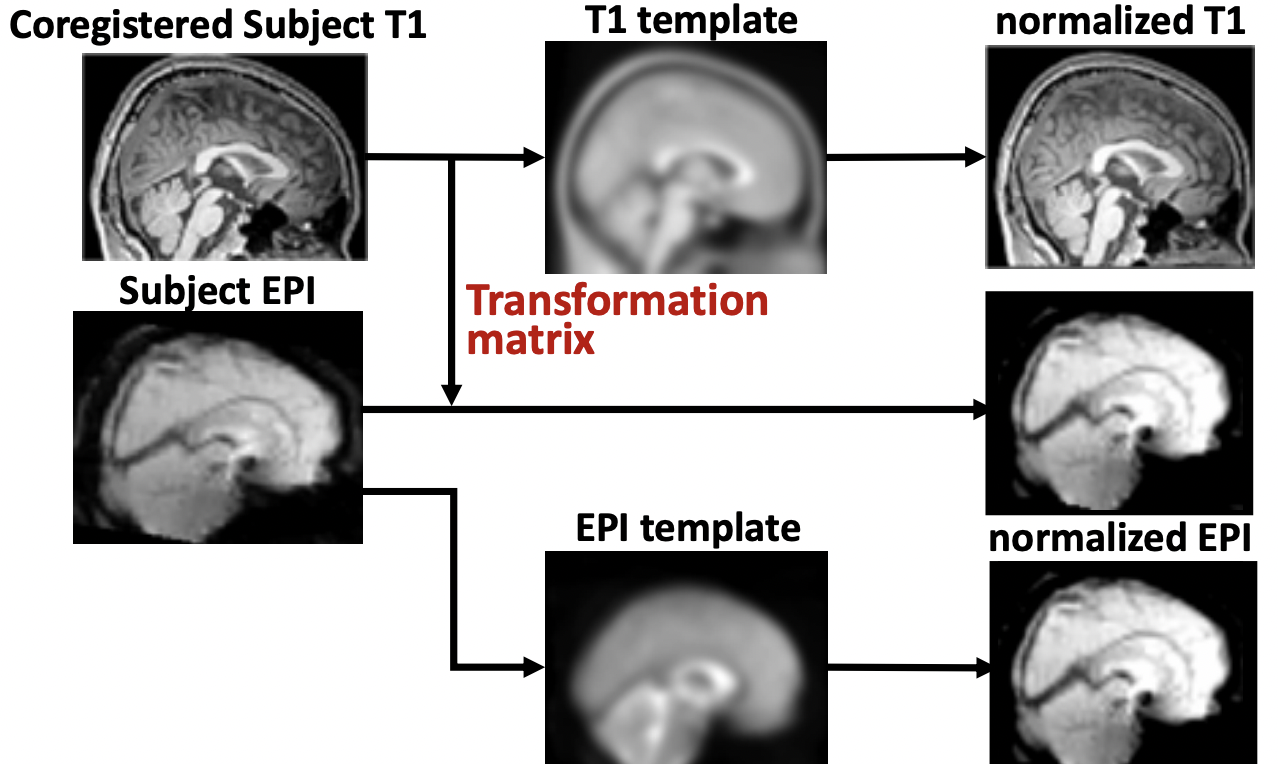
Figure 5.17: Spatial normalization 的途徑。
5.2.8 Gaussian spatial smoothing
標準化以後,我們要進行 spatial smoothing,如圖 5.18 右,結果將會變得比原本模糊。此步驟即應用 3D Gaussian kernel,進行 convolution。在此步驟以後,影像會失去一些空間解析度,不過因為我們還有結構影像,所以成本不算太大,卻有降低 noise 的效益。
在 smoothing 中,每個 voxel 都會變成其周圍 voxels 的加權平均,這會讓鄰近的 voxels 有相近的值,所以相比起未 smoothing 的資料才會比較模糊。此步驟可以增加 signal-to-noise 比,故能增加 statistical power,更容易進行統計推論。
不過,smoothing 並非在所有 fMRI 分析中都是必備的步驟,例如在 MVPA 中就不能從事 smoothing。
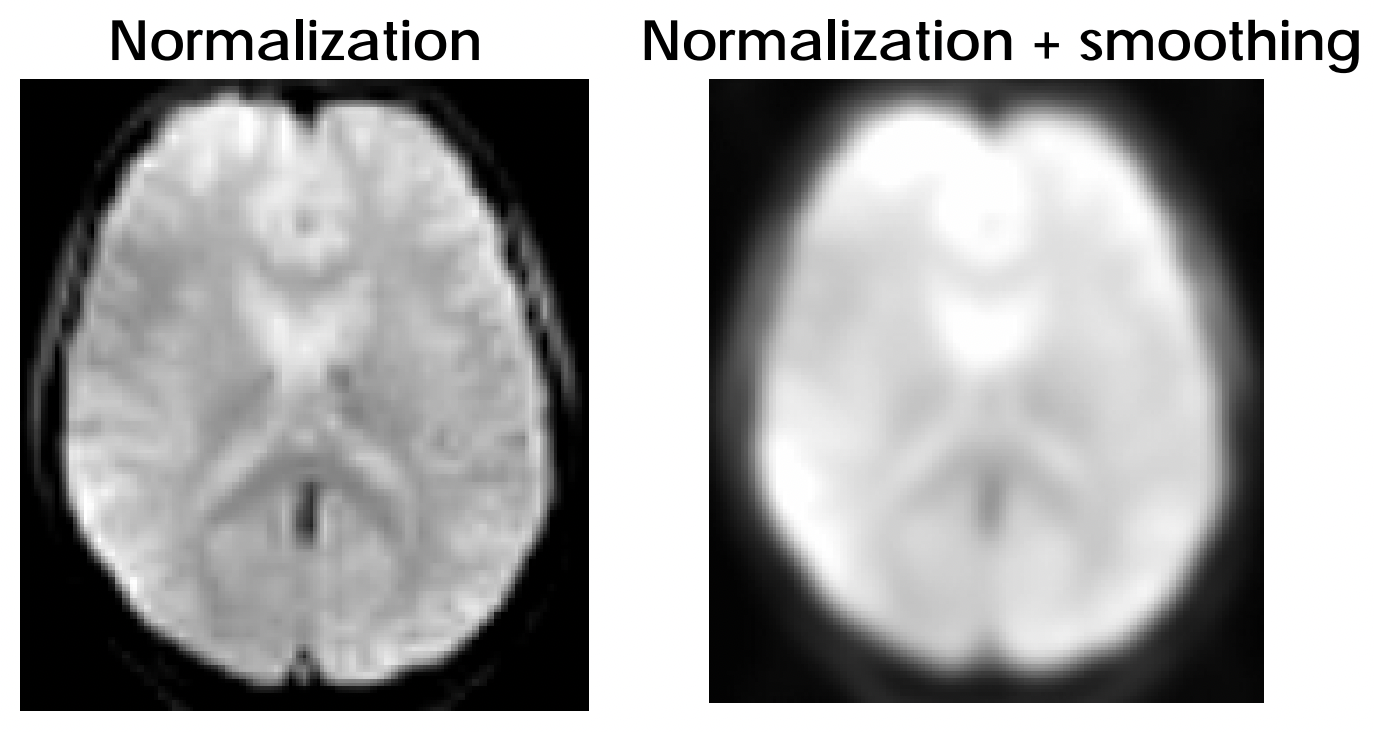
Figure 5.18: Gaussian spatial smoothing.
5.2.9 Tissue segmentation
在此步驟,我們可以識別哪些區域是灰質、白質或 CSF,如圖 5.19。要注意的是,此步驟所產生的檔案會存在剛剛的 1 BACK/MPRAGE 資料夾裡。
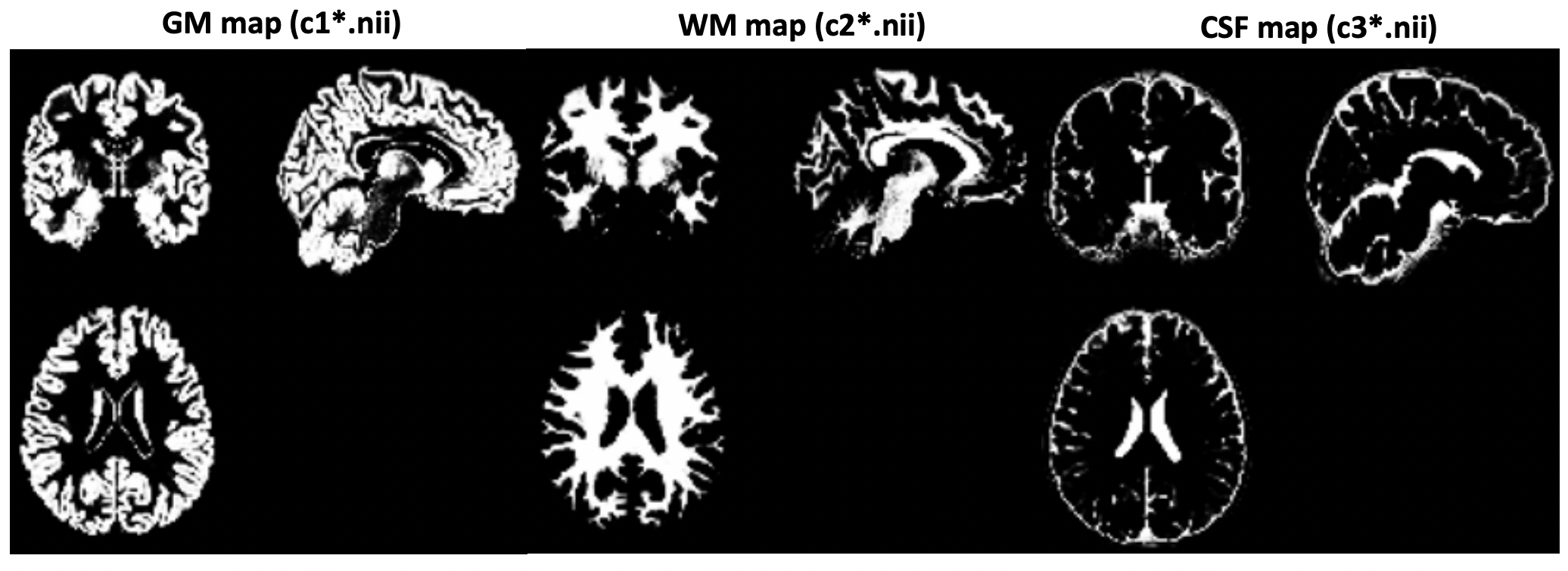
Figure 5.19: Tissue probability map.
5.3 Parameter Setup of SPM Batch
在此,要接續節 5.2 沒有討論到的 Batch Editors 中的參數到底怎麼設定。
- 按下 SPM Menu 右下方的
Batch,以開啟 Batch Editor 視窗。
5.3.1 Correction for Slice Timing
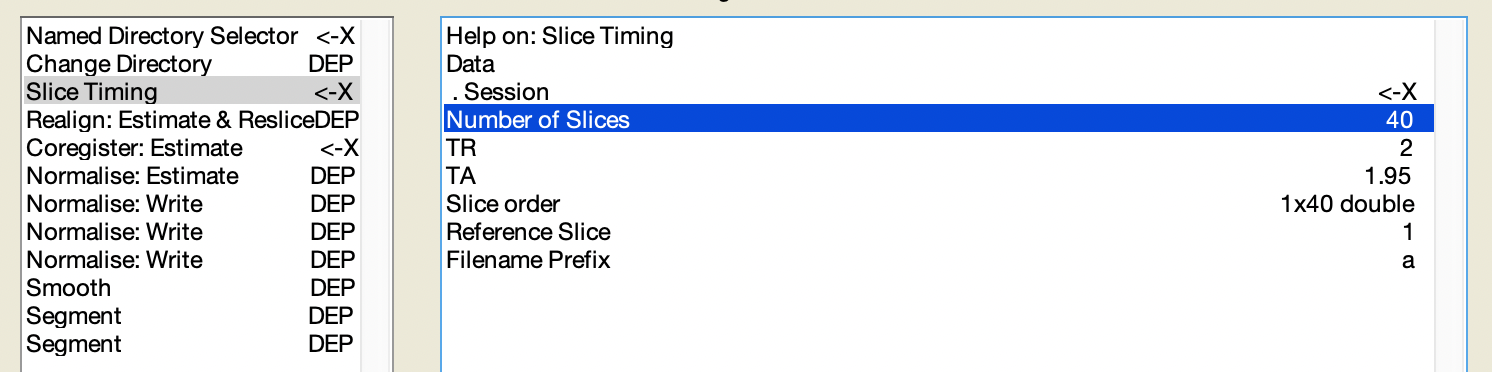
Figure 5.20: Batch Editor 中的 Slice Timing.
- 如圖 5.20,選取
Module List中的Slice Timing。 - 其中的
Number of Slices要設成 40。 TR意即 “Repetition Time”,要注意的是此處以秒為單位,而 2000 ms = 2 s,故此處應該設為 2。TA的計算方式是 \(\rm TA = TR-\frac{TR}{nslices}=2-\frac{2}{40}=1.95\)。可見 Batch Editor 視窗下方的資訊欄。Slice order在西門子機器的真實的設定方式2 4 6 8 10 12 14 16 18 20 22 24 26 28 30 32 34 36 38 40 1 3 5 7 9 11 13 15 17 19 21 23 25 27 29 31 33 35 37 39。Reference Slice設置成中間的 slice,即 40 或 1,此處設為 1。
5.3.2 Realignment of head motion
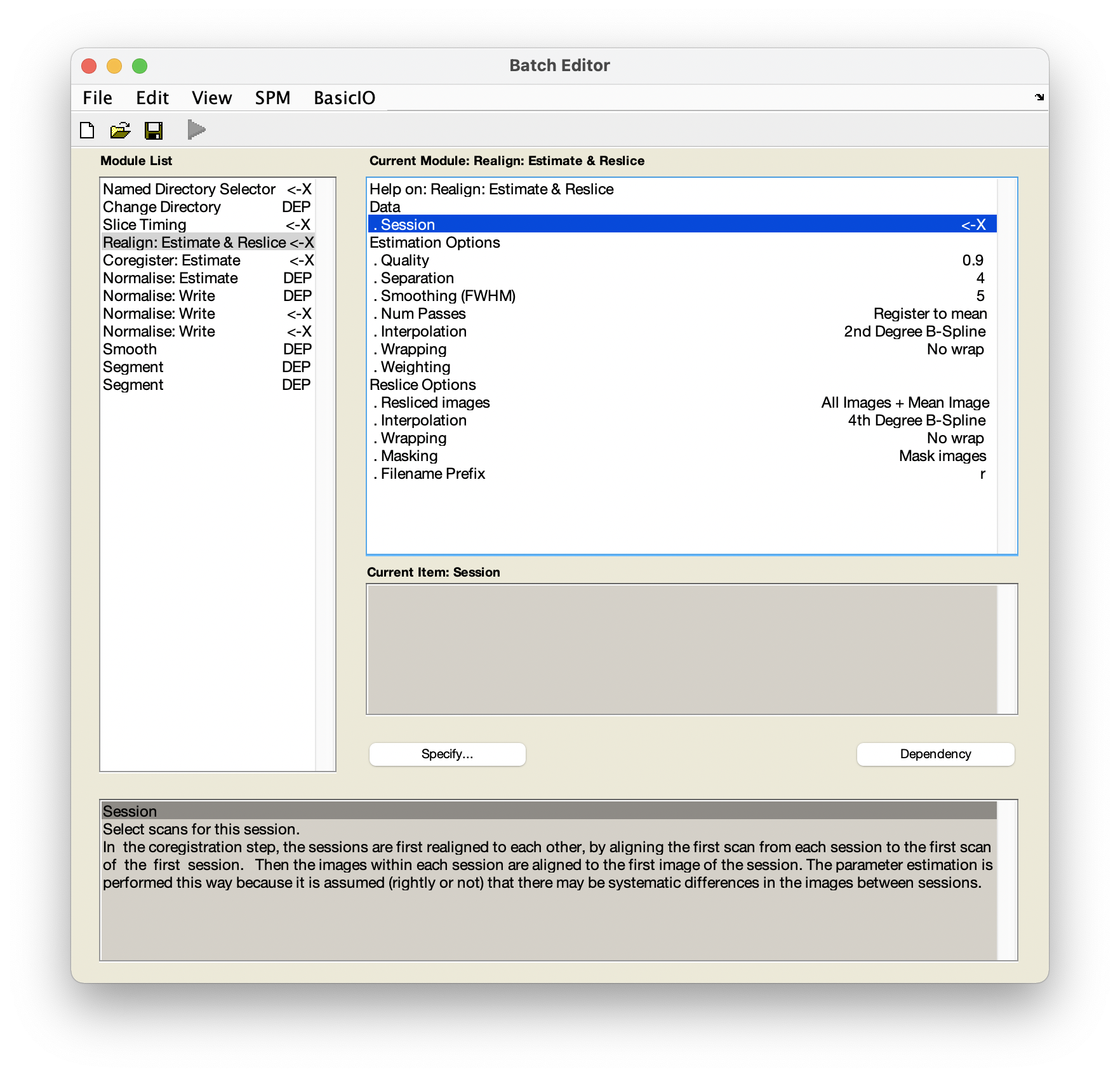
Figure 5.21: Realign: Estimate & Reslice 中的 Session。
此步驟我們都使用預設的參數就好。值得一提的是,當點選 Realign: Estimate & Reslice 中的 Session 時,欄位右下方會有兩個按鈕,分別是 Specify 與 Dependency,如圖 5.21。前者正如選取一個檔案丟入 Session,點選後者則會跑出如圖 5.22 的視窗。我們可以使用此函數,來使用前一個步驟 Slice Timing 過後的檔案。
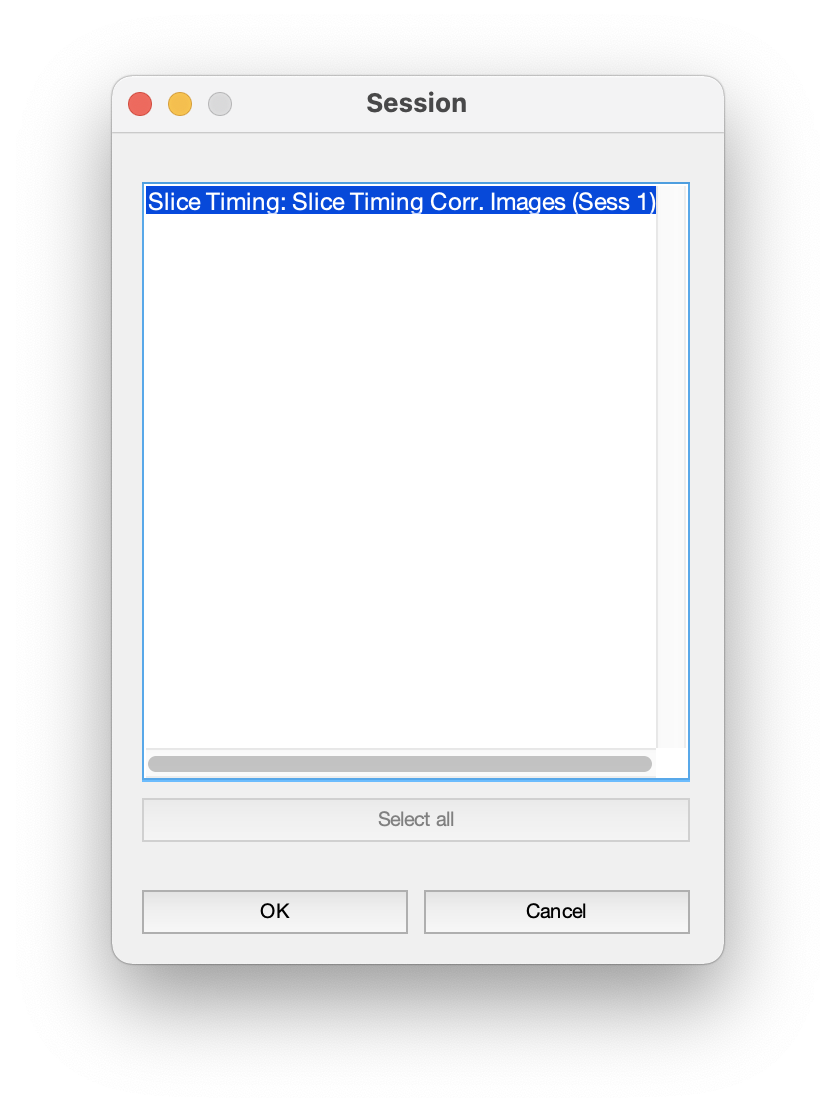
Figure 5.22: 依序點選 Session 與欄位右下方的 Dependency,將會出現此視窗。
5.3.3 Co‐registration
此步驟要對齊結構影像(T1W)與 fMRI 資料(EPI)。點選 Module List 中的 Coregister: Estimate,裡頭的 Reference Image 與 Source Image 呈現 <-X,表示待輸入檔案。Reference Image 要輸入的是 fMRI 檔,而 Source Image 要輸入的是結構影像檔。
此時,Reference Image 可以仿照上述步驟,點選下方的 Dependency,並選取其中的 Realign: Estimate & Reslice: Mean Image,如圖 5.23。而 Source Image 就要乖乖 Specify 一個 T1 檔了。
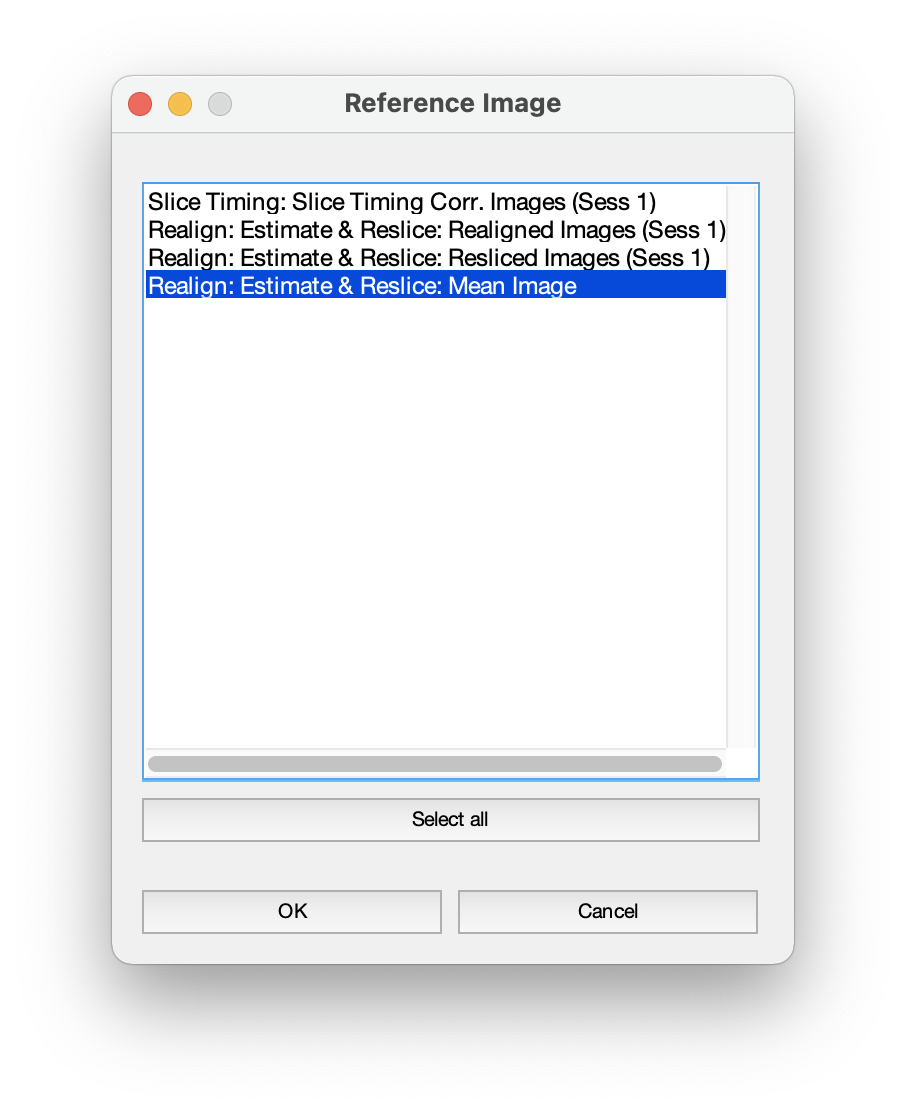
Figure 5.23: 依序點選 Reference Image 與欄位右下方的 Dependency,將會出現此視窗。選取其中的 Realign: Estimate & Reslice: Mean Image。
此外,在此步驟中 SPM 不會產生新檔案,但是會估計一個用以 image co-registration 的最適的 transformation matrix。
5.3.4 Spatial normalization
在 Normalise: Estimate 中,Image to Align 一樣要 specify 前個步驟的 coregistered images,亦可以使用前述的 Dependency。之所以要輸入一個檔案,是為了要估計用以 spatial normalization 的 transformation matrix。而在 Tissue probability map 中,記得要放進 SPM 12 的子資料夾下的 TPM.nii。Affine Regularisation 則因為在臺灣收取資料,故要設置成 “East Asian brains”。

Figure 5.24: 注意 Normalise: Estimate 中的 Image to Align、Tissue probability map 與 Affine Regularisation。

Figure 5.25: 注意 Normalise: Estimate 中的 Image to Align、Tissue probability map 與 Affine Regularisation。
此外,還有三個 Normalise: Write,如圖 5.25。第一個 Normalise: Write 的 Deformation Field 即是需要輸入一個 transformation matrix,即要設置 Dependency 為前個步驟所製造出來的 transformation matrix,在選單中即 Normalise: Estimate: Deformation,如圖 5.26;在 Images to Write 則要設置成 coregister 出來的結構影像,即設置 Dependency 為 Coregister: Estimate: Coregisterd Images。
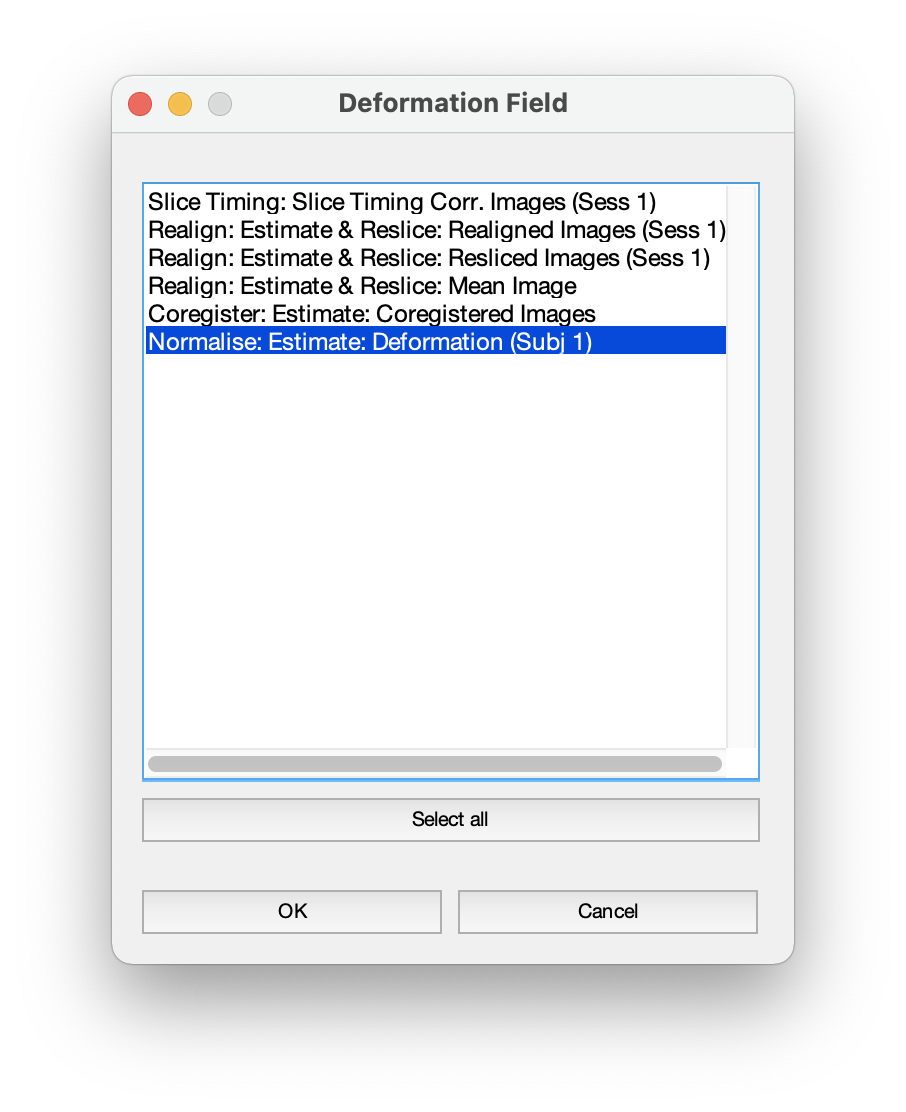
Figure 5.26: 三個 Normalise: Write。
第二個 Normalise: Write 的 Deformation Field 亦同,但 Images to Write 的 Dependency 則要設置成 realign 出來的 fMRI 資料,即 Realign: Estimate & Reslice: Resliced Images (Sess 1);最後一個 Normalise: Write 的 Deformation Field 亦同,但 Images to Write 的 Dependency 則要設置成另一個 realign 出來的 fMRI 資料,即 Realign: Estimate & Reslice: Mean Image。此 “mean” 是平均各個時點的影像而來。
另外,注意圖 5.25,在 Writing Options 下有兩個函數 Bounding box 與 Voxel sizes。後者即輸入一個向量,例如 [2 2 2],單位是毫米(本來我們的每個 voxel 並非這個大小,在標準化後才成為 \(2\times 2 \times2\))。
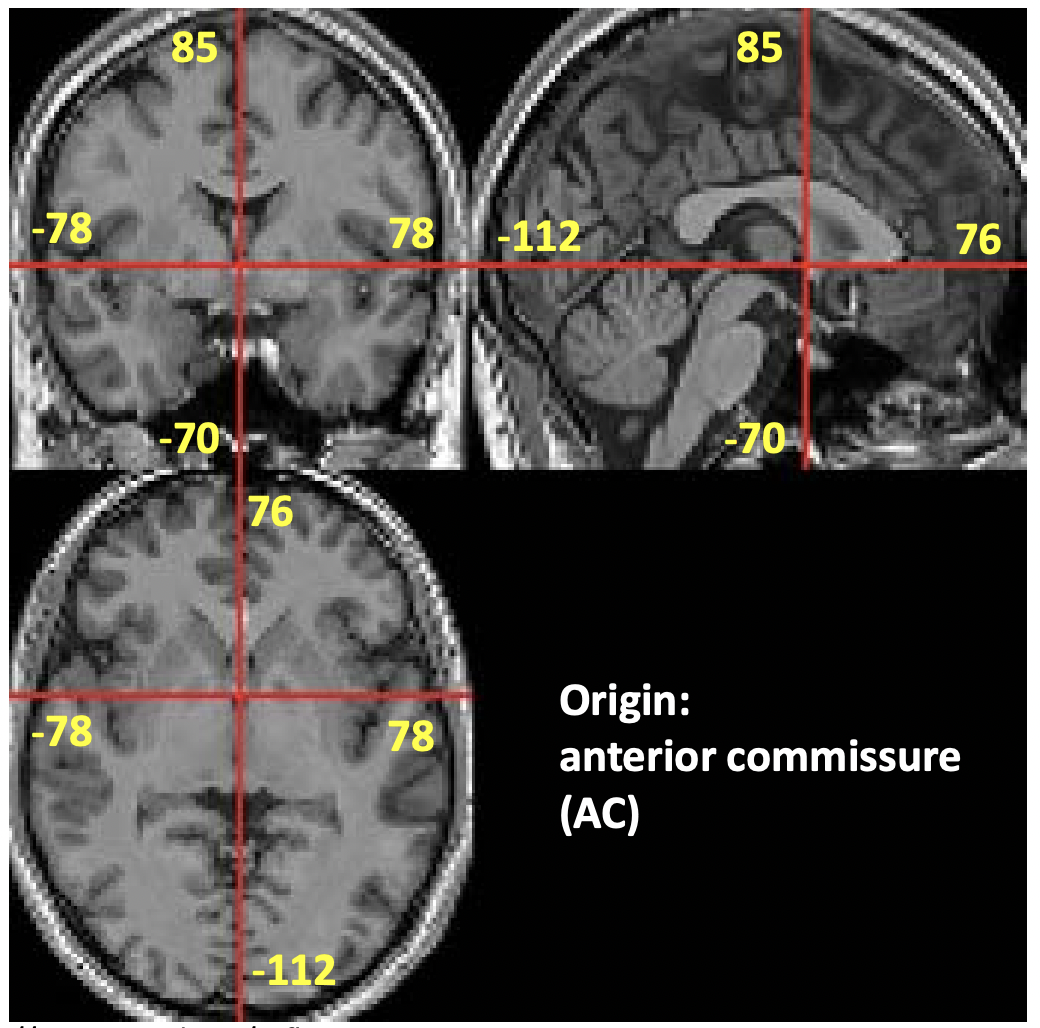
Figure 5.27: Bounding box.
至於 Bounding box,如圖 5.27,在空間標準化以後,我們就已經設定了一個原點,位於 anterior commissure (AC)。而 Bounding box 事實上是一個 \(2\times 3\) 的矩陣。第一個 column 即 \(x\) 的範圍,以此類推:第二個 column 即 \(y\) 的範圍,第三個 column 即 \(z\) 的範圍。以圖 5.27 而言,此矩陣為
\[
\begin{pmatrix}
-78 & -112 & -70\\
78 & 76 & 85
\end{pmatrix}
\]
同樣以毫米為單位。可以想像,如果範圍設得太小,則看到的影像就不會是全貌。當使用其他種類的腦,如小孩時,就要調整 bounding box 的大小。
5.3.5 Gaussian spatial smoothing
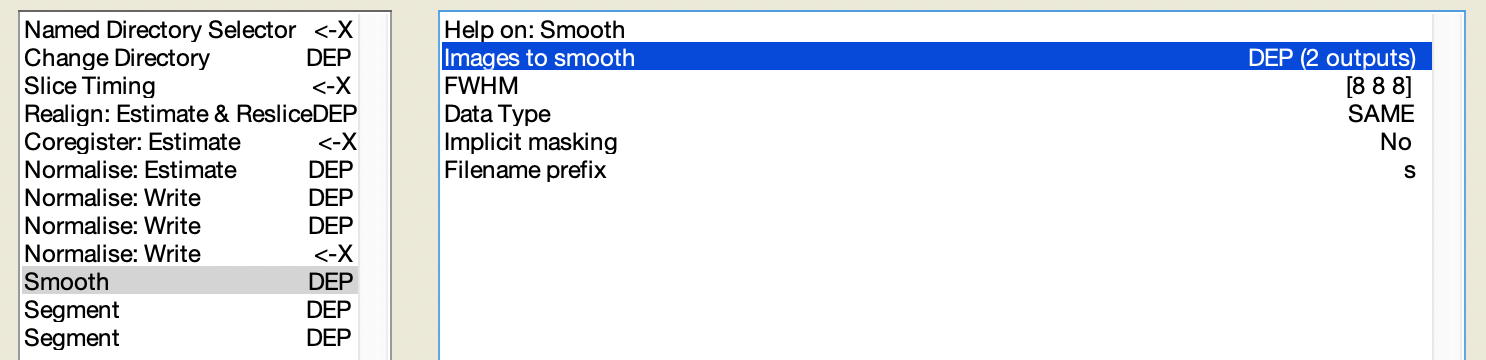
Figure 5.28: Spatial smoothing.
如圖 5.28 中的 Smooth 中的 Images to smooth 中輸入兩個檔案,如圖 5.29,分別是前兩個步驟處理完的 fMRI 資料,前者要放的是 resliced 版,後者要放的是 mean 版。此外,從圖 5.28 中可以看到還有一個需要設定的選項是 FWHM,其全名是 “Full width at half maximum (of the
Gaussian smoothing kernel in mm)”。這裡的值設定得越大,則 smoothing 效果會越強,但值一般都設在 4–8 之間,而三個向度的值要相同。
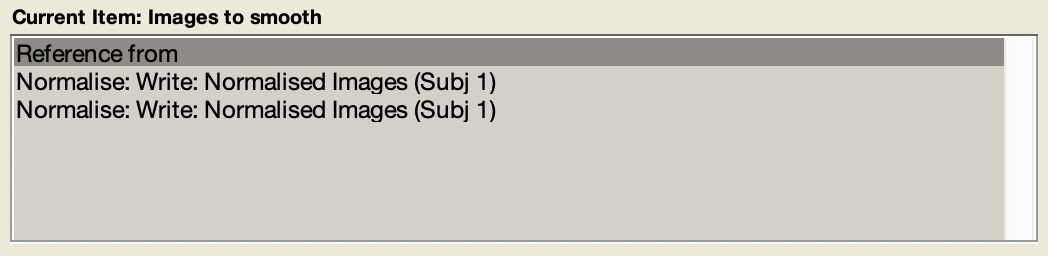
Figure 5.29: Images to smooth 中要輸入兩個檔案。
5.3.6 Tissue segmentation
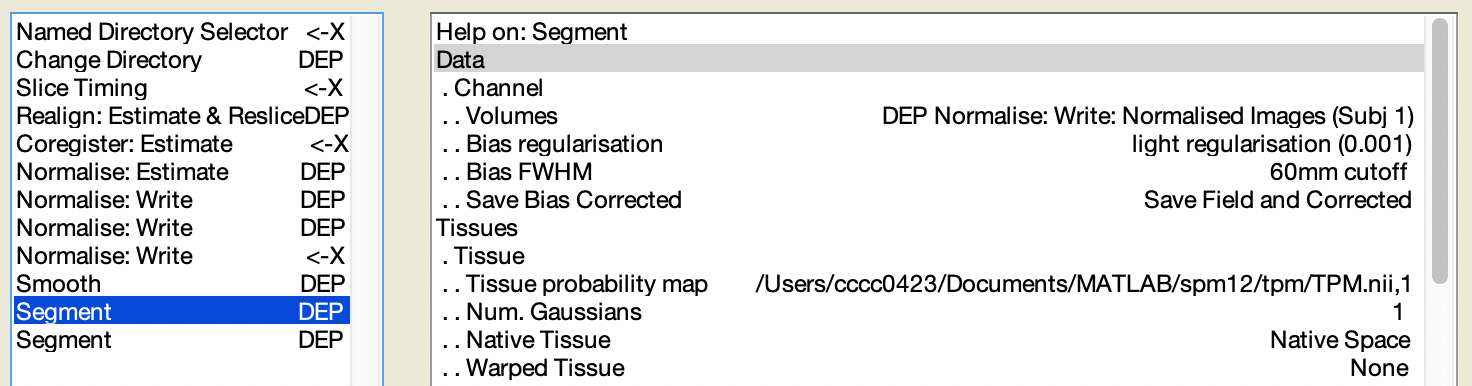
Figure 5.30: Segment 中的 Volumes 與 Save Bias Corrected。
如圖 5.30。在此步驟中,第一個 Segment 中的 Volumes 要輸入的是第一個 Normalise: Write 的空間標準化後的結構影像,切記不要選到後兩個空間標準化後的 EPI 資料。除外,Save Bias Corrected 的選項要設為 Save Field and Corrected。但在第二個 Segment 中的 Volumes 要輸入的是第一個 Normalise: Write 的空間標準化後的 mean EPI。
要注意到在此 SPM 並未做 bias field correction。在課程材料 fMRIana07_materials 中有一檔案 ApplyBiasfield_CFLu.m 為 MATLAB script。我們要在 MATLAB 中開啟此檔案,然後按下 MATLAB 視窗右上角的綠色播放按鈕 Run,將會跳出一視窗,選取 Change Folder 即可。執行此段指令的作用在自動在前處理過的資料進行 biad field correction。
執行以後,會跳出一個選取檔案的視窗,此時所要選取的的是前處理過後的資料夾。例如我們選取前處理過的 1BACK 資料夾,將會跳出如圖 5.31 的視窗。此時有兩個選項,此處我們選擇第一個。接著,當執行完成,將會調出如圖 5.32 的視窗,表示 bias field correction 已完成。
Figure 5.31: 執行進行 bias field correction 的指令並選取前處理後的資料夾後所跳出的視窗。
Figure 5.32: Bias field correction 已完成。
在完成 bias correction 以後,原本那個資料夾還會出現 “m” 開頭的 .nii,如圖 5.33。
Figure 5.33: 出現 “m” 開頭的檔案。
從此才算是完成了所有前處理程序!
功能影像之所以會分成那麼多檔案是因為有許多不同時點,然而結構影像只有一個時點。↩︎