6 Brain Activation: General Linear Model
資料前處理以後,現在要做的是分析 brain activation,即以 general linear model (GLM) 找出活躍的腦區。
6.1 先備知識
6.1.1 相關軟體
MRIcro
Statistical Parametric Mapping (SPM 12)
xjView (A viewing program for SPM)
6.2 General Linear Model
6.2.1 N‐Back fMRI Data Acquisition
In short, in the N-Back task, participants are presented a sequence of stimuli one-by-one. For each stimulus, they need to decide if the current stimulus is the same as the one presented N trials ago. The N can be 1 trials, 2 trials, 3 trials, etc. The higher the number, the more difficult the task. The factors that seem to influence the performance are not only the N, but also the speed of presentation and the size of the set of stimuli.
— https://www.psytoolkit.org/experiment-library/touch_nback2.html
6.2.2 GLM 模型
首先,我們會測量各個 voxel 的時間序列的 BOLD signals,正如圖 6.1 的最左。而在圖 6.1 中,實驗設計為有 4 個 tasking blocks 的 block design。而 \(x_1\) 與 \(x_2\) 可能分別是在一個 fMRI session 中左手跟右手的抓握。此外,我們還會有一個 \(x_3\),為未知的變數。然後,GLM 的目的就在估計最能適配觀察到的資料的係數。
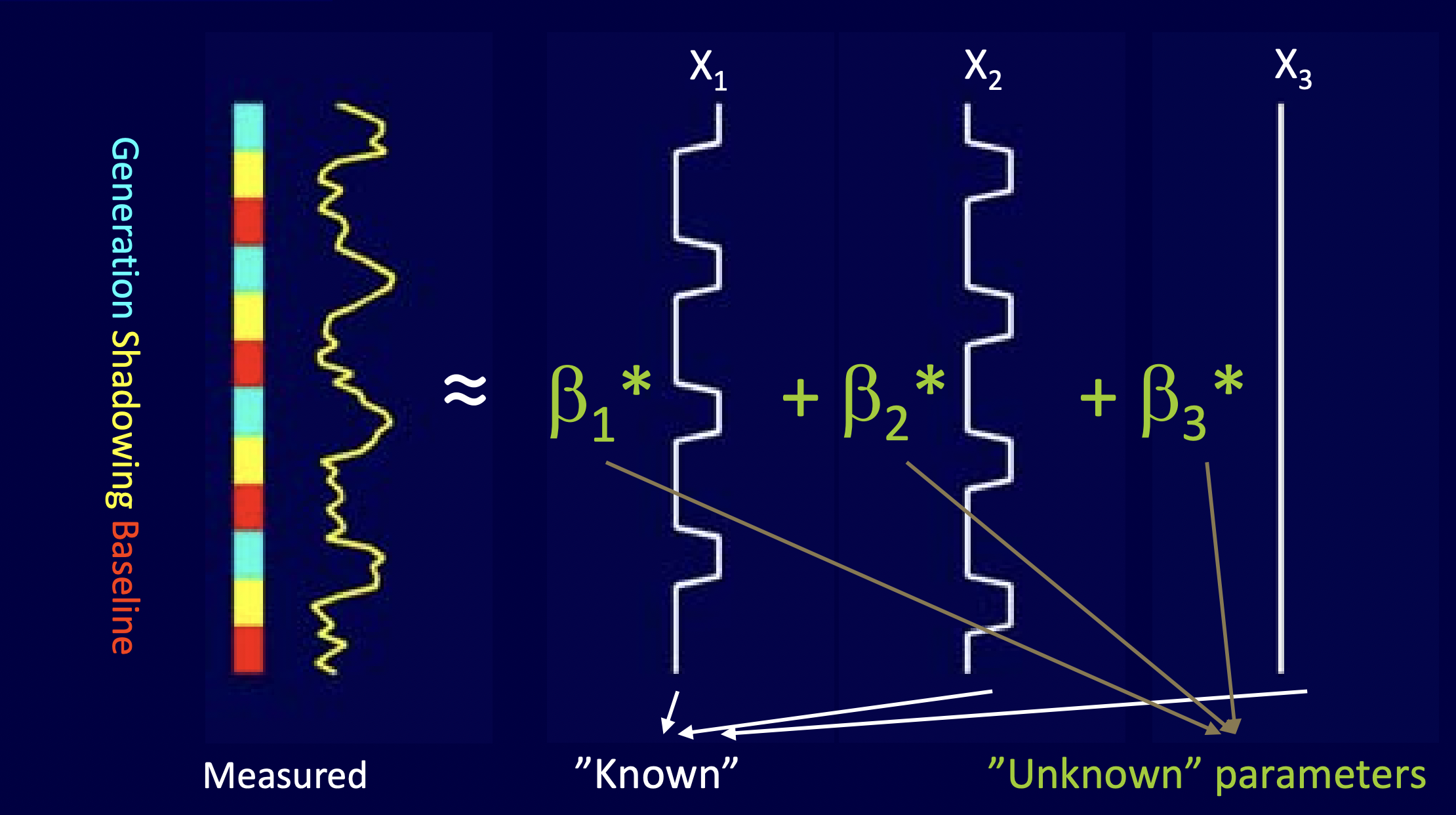
Figure 6.1: GLM 模型。
6.2.3 參數估計
估計出的係數可能如圖 6.2。
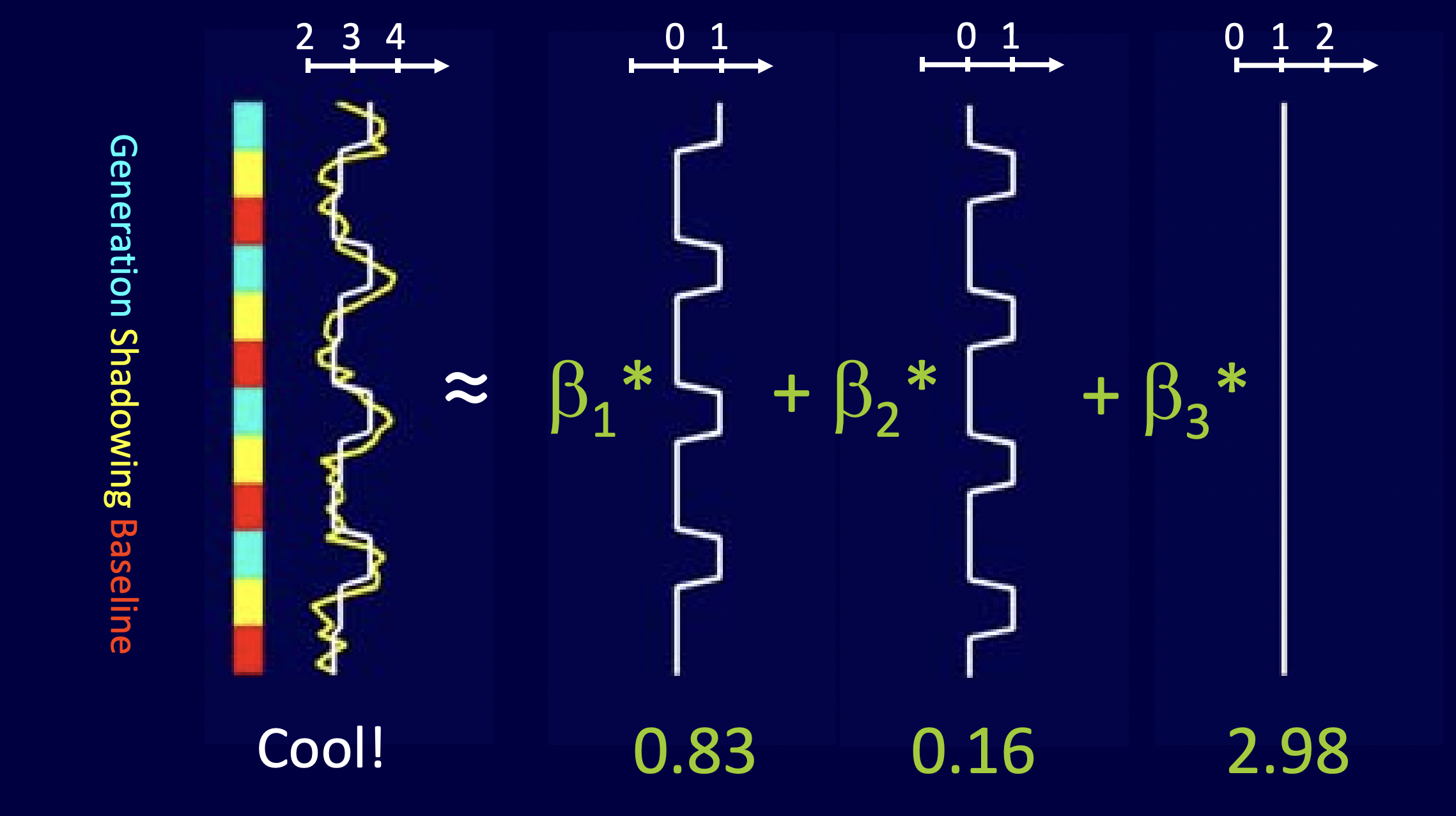
Figure 6.2: 參數估計。
在我們估計出各個 voxel 的係數後,如圖 6.3,我們會得到很多個 \(\vec{\beta}\),其中有許多 \(\beta_1\)、\(\beta_2\) 與 \(\beta_3\)。我們要把這些 \(\beta_1\)、\(\beta_2\) 與 \(\beta_3\) 分別結合起來,成為不同的 task conditions 的輿圖(map)。
接著,我們就可以進行統計檢定,看看在各個 task condition 下,究竟哪些腦區特別活躍了。
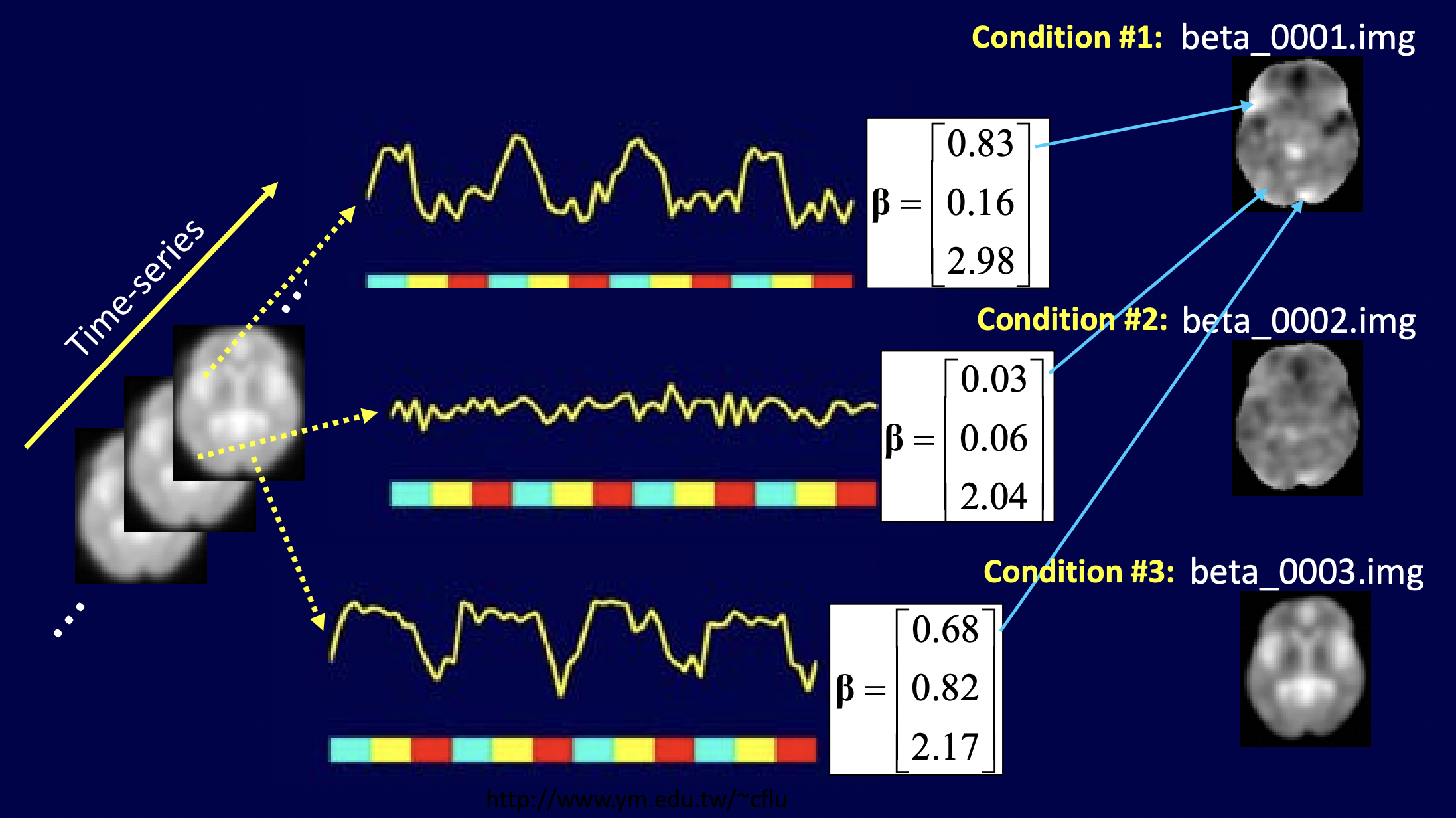
Figure 6.3: 結合各 voxel 的係數為一大腦輿圖。
6.2.4 Specify 1st‐level Model
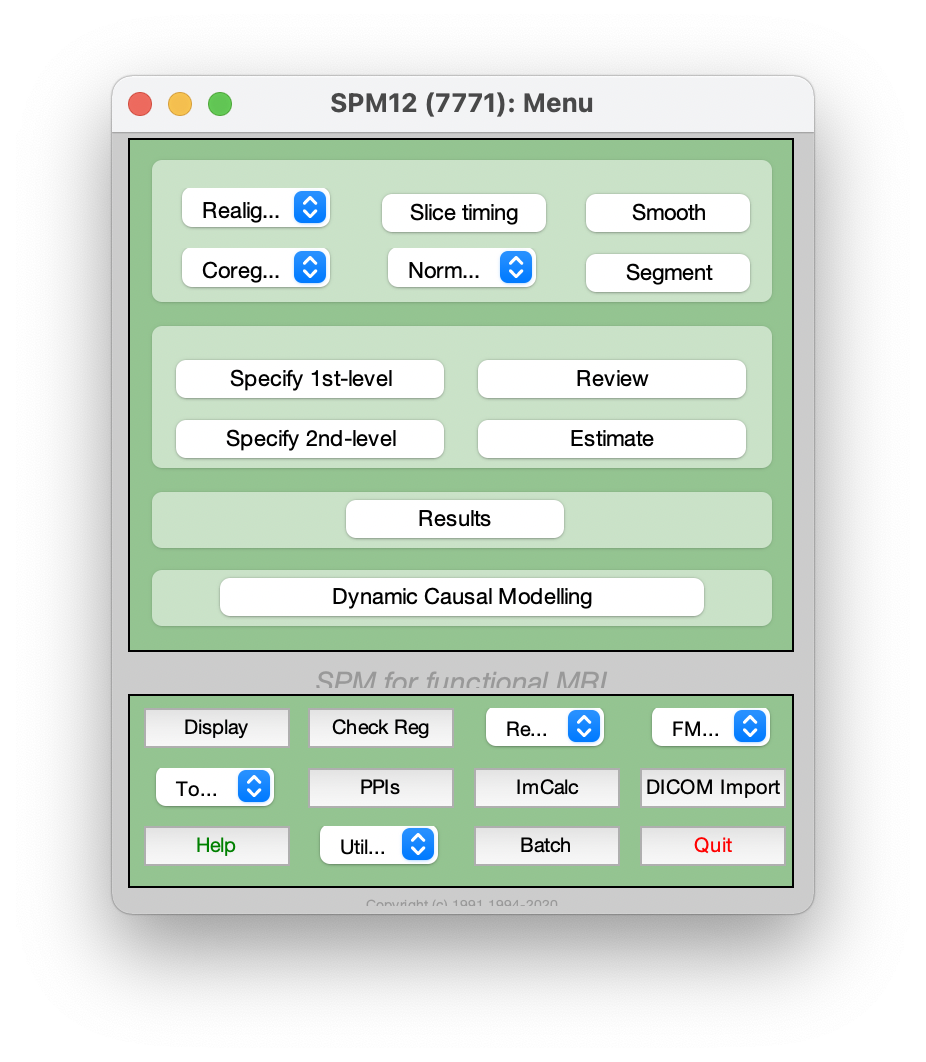
Figure 6.4: SPM 12: Menu.
按下圖 6.4 中的 “Specify 1st‐level” 鈕後,將會開啟 Batch Editor。在 Directory 中,我們可以選擇待會 SPM.mat 將要寫入/輸出的資料夾。我們在此選擇 fMRIana06_materials 中已經前處理後的 2BACK 資料夾。
此外,還有幾個需要指定的參數:
在
Units for design,選擇 scans 會更容易操作。Interscan interval此例應該設置為 2。Microtime resolution此例應該設置為 40 (Slice number)Microtime onset此例應該設置為 1 (Reference Slice)
接著,雙擊 Data & Design 會一個新的 Subject/Session,而:
在
Scans中,要使用經過 bias field correction、smooth、normalize 等步驟的影像,因此正則表達式要輸入^ms.*,然後全選這些影像。在
Conditions中,因為本影像資料只有一個 experimental condition,所以只要雙擊兩次Conditions來產生一個Condition區塊;而如圖 6.1 的實驗則有兩個 experimental conditions(可能是左右手),所以我們要雙擊四次Conditions或點擊下方的New: Condition兩次,來產生兩個Condition區塊。- 其中,
Name可以設為 2_BACK。 Onsets取決於我們的 block design,在此例應該設置為 [16 46 76 106 136]。受試者在第 16 個 scan 才第一次看到刺激(6 個數字),以此類推,如圖 6.5。Duration應該設為 15,因為事件的時長都是 15 個 scans,如圖 6.5。- 因為 head motion 可能會比 hemodynamic change 來得更大,為了消除這種潛在的隱憂,要選取 6 個 column parameters 作為 multiple regressors。我們要進到前處理完畢的
2BACK資料夾,會發現有個 rp 開頭的.txt檔。選取它作為 mulitiple regreesors,其記錄了 6 個 column parameters。
- 其中,
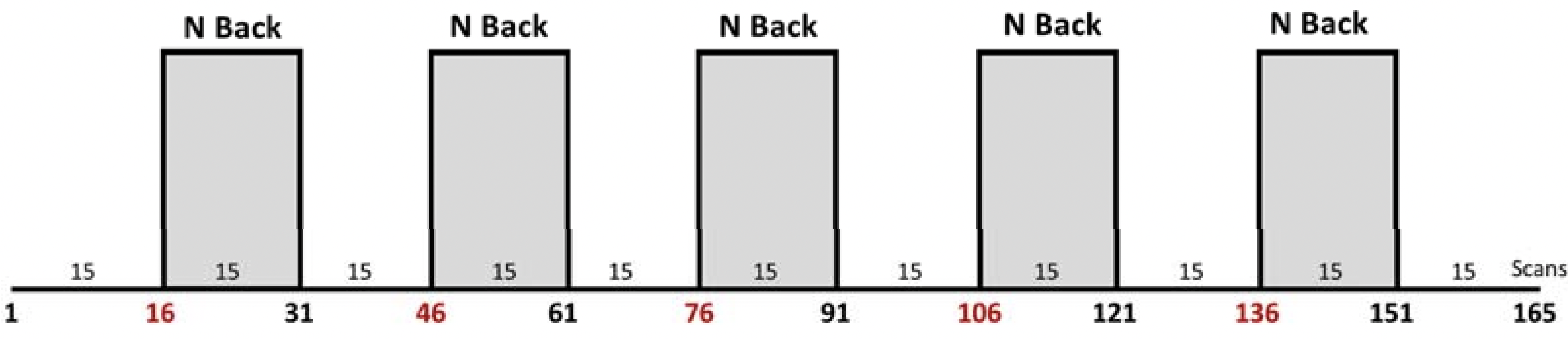
Figure 6.5: 此受試者影像資料所用的實驗設計。
然後我們就可以按下 Run Batch。
6.2.5 Statistical Analysis: Design
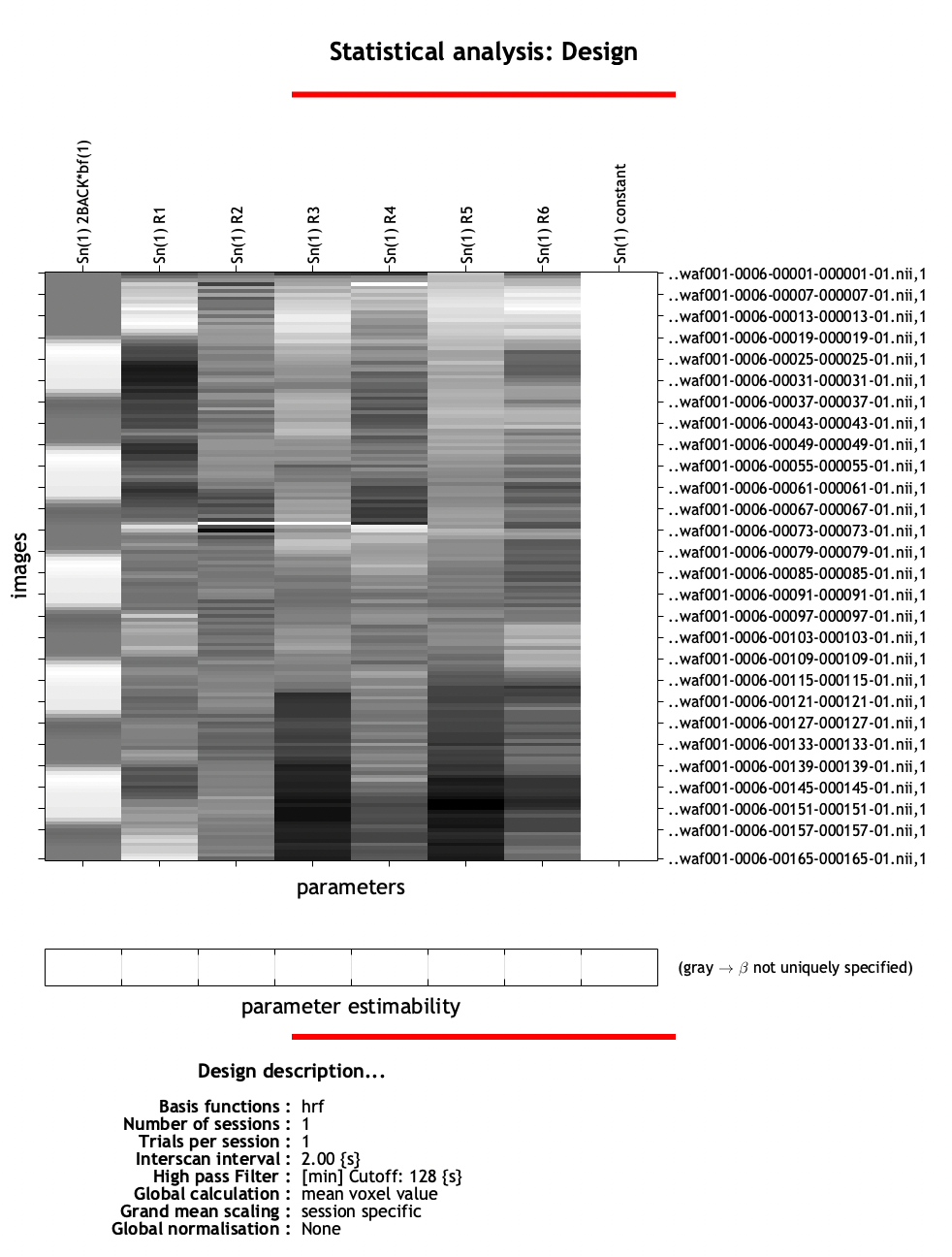
Figure 6.6: SPM: Graphics.
在按下 Run Batch 之後,我們將會得到如圖 6.6 的畫面。最左側的 column 是剛剛指定的 block design;而其右邊 6 個 columns 中,左 2 至左 4 是 translation parameters;右 2 至右 4 則是 rotation parameters;最右側的則是 SPM 自動添加的常數項。
在 6.6 中,我們可以看到底下的參數估計的欄位全部都是空白的。這是一件好事,若否,則表示各個 regressors 之間高度相關,此時就不適合使用 GLM 了,因此要進行其他調整,此處從略。
6.2.6 Estimate Model
下一步是按下 SPM Menu 中的 Estimate。在 Select SPM.mat 中,我們只要回到剛剛使用的資料的那個資料夾,它已經建立了預備估計的模型的檔案了,找到名為 SPM.mat 的檔案並選取之即可,然後也 Run Batch。
要如何查看結果呢?回到 SPM Menu,按下 Results,接著到同樣的路徑,選取 SPM.mat。此時,會開啟一個新視窗,如圖 6.7
Figure 6.7: 在 SPM contrast manager 中查看結果。
我們可以在此查看我們感興趣的 condition。之所以稱為 “contrast”,即我們可以選取一 condition 與另外一 condition 比較。
剛開啟 SPM contrast manager 時會發現,其顯示 no contrasts defined。因此,我們需要定義 contrast,即按下藍色的 Define new contrast 鈕即可。這時候畫面會變成如圖 6.8。
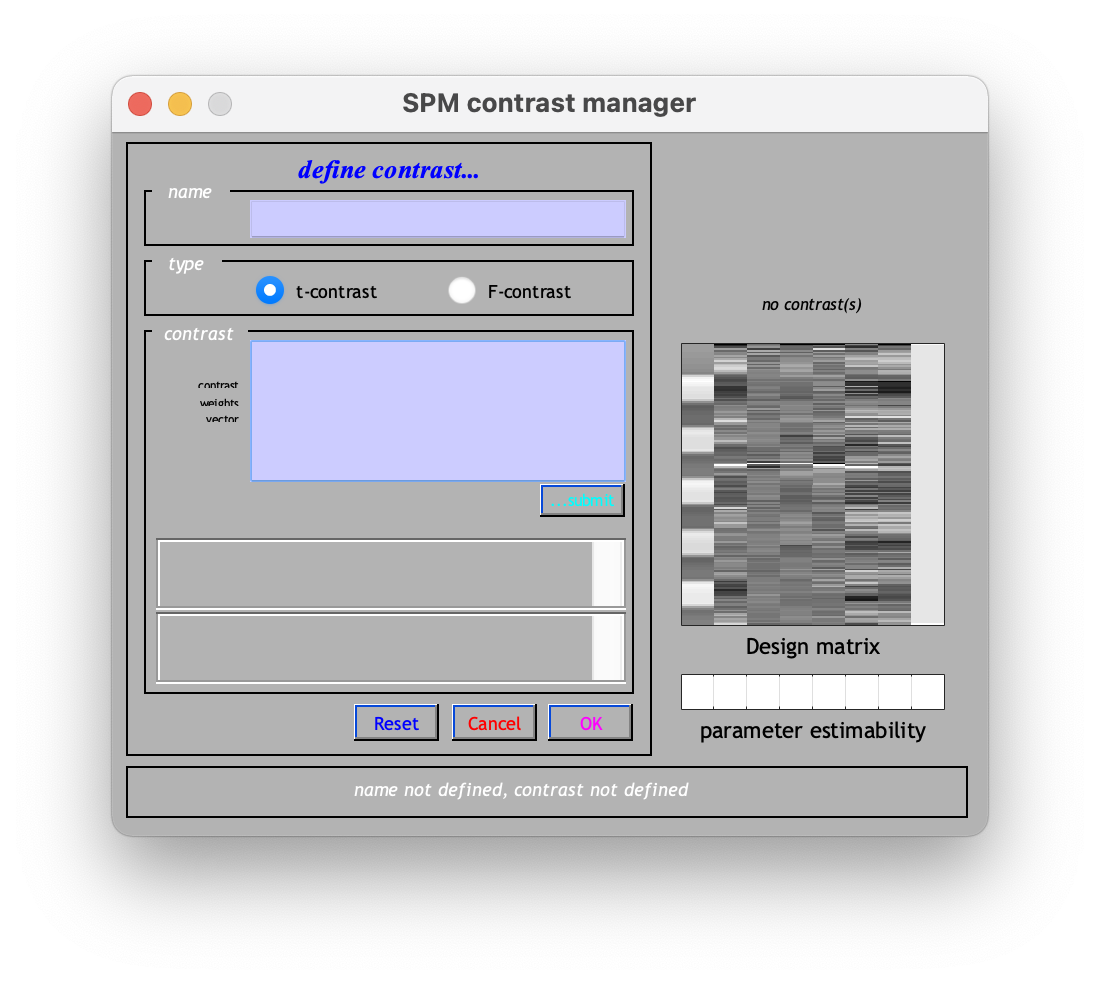
Figure 6.8: Define new contrast.
我們可以為這個 contrast 取個名字。而我們要在下方,把感興趣的 conditions 設為 1。在此例中,因為我們僅對第一個 block design 感興趣,而後面 6 個為不感興趣的動作參數,最後一個常數也是不敢興趣的,所以我們將數值設為 1 0 0 0 0 0 0 0。之後,按下鍵盤上的 Enter,將會如圖 6.9,右上角的白色欄位出現一個小方塊。
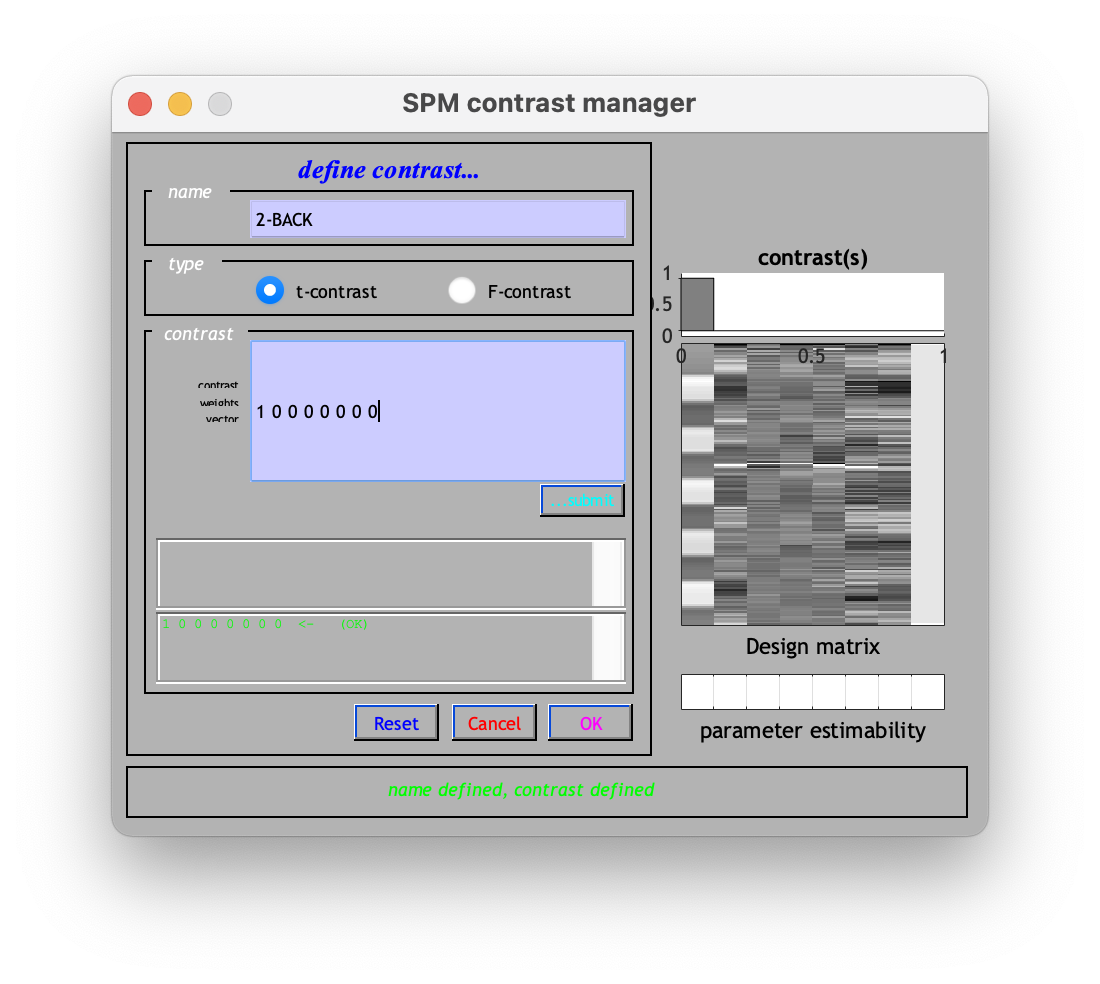
Figure 6.9: 按下 Enter 之後。
然後我們就可以按下 OK,就會回到 SPM contrast manager 的主頁面,剛剛所定義的 contrast 就會成為一個 contrast 選項,下次要查看時就不用重新再定義。
我們現在可以選取剛剛定義的 contrast,然後按下底下的 Done,會跳出如圖 6.10 的視窗。

Figure 6.10: 按下 Enter 之後。
接著,是一連串的問題:
如果我們不是只想看某幾個的話,就沒必要 apply masking,因此這裡選擇
none。如何調整 \(p\) 值?因為我們要進行 multiple comparison,所以不能只是使用 0.05,否則將會出現 family-wise error;我們必須選擇
FWE,即進行 family-wise error correction。選擇了FWE後,在p value (FWE)使用預設的 0.05 即可,而在extent threshold中,我們必須指定 “cluster size”,即在附近的 voxels 都顯著,才能說這是活躍的腦區。我們通常設置為 30。Modality 要設置為 fMRI。之後 SPM 將會開始運作,而得到如圖 6.11 的結果。
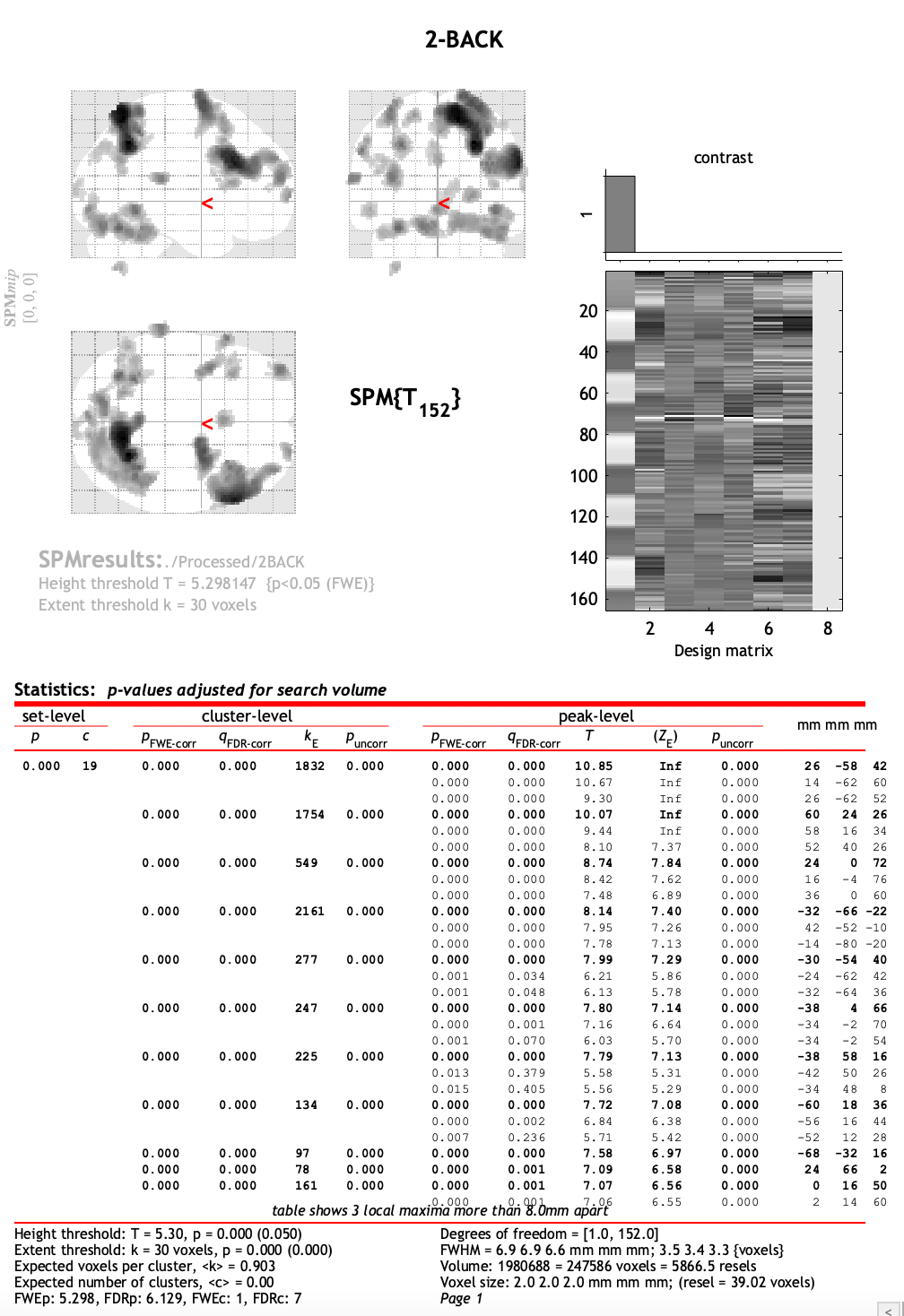
Figure 6.11: 運作結果。
圖 6.11,上方腦的部分,顏色越深代表活動越強烈。而我們如果點擊底下的統計表格的 mm 那欄,上方的腦的區塊的指示箭頭也會隨之改變到剛剛點擊的數字所對應到的腦位置。
不過,這樣的圖還太簡單了!我們需要 xjView!
6.2.7 安裝與啟動 xjView
如同安裝 SPM 一樣,從 MATLAB 主視窗的 Environment 中按下 Set Path 按鈕,如圖 6.12,開啟新視窗如圖 6.13。透過左側的 Add with Subfolders... 按鈕可以將解壓縮後的 xjView 資料夾加入路徑。
Figure 6.12: Environment > Set Path
Figure 6.13: Set Path.
然後只要輸入指令 xjView,就能開啟 xjView,如圖 6.14。
Figure 6.14: xjView.
6.2.8 使用 xjView
我們可以在左上角的 File 中,選擇 Open Images,來開啟影像檔。到剛剛的 2BACK 資料夾中,搜尋 spm,可以發現有一個名稱由 spmT 開頭的檔案(“T” 意味著 t contrast,“0001” 則是編號),選擇該檔案。然後我們會得到如圖 6.15 的畫面。
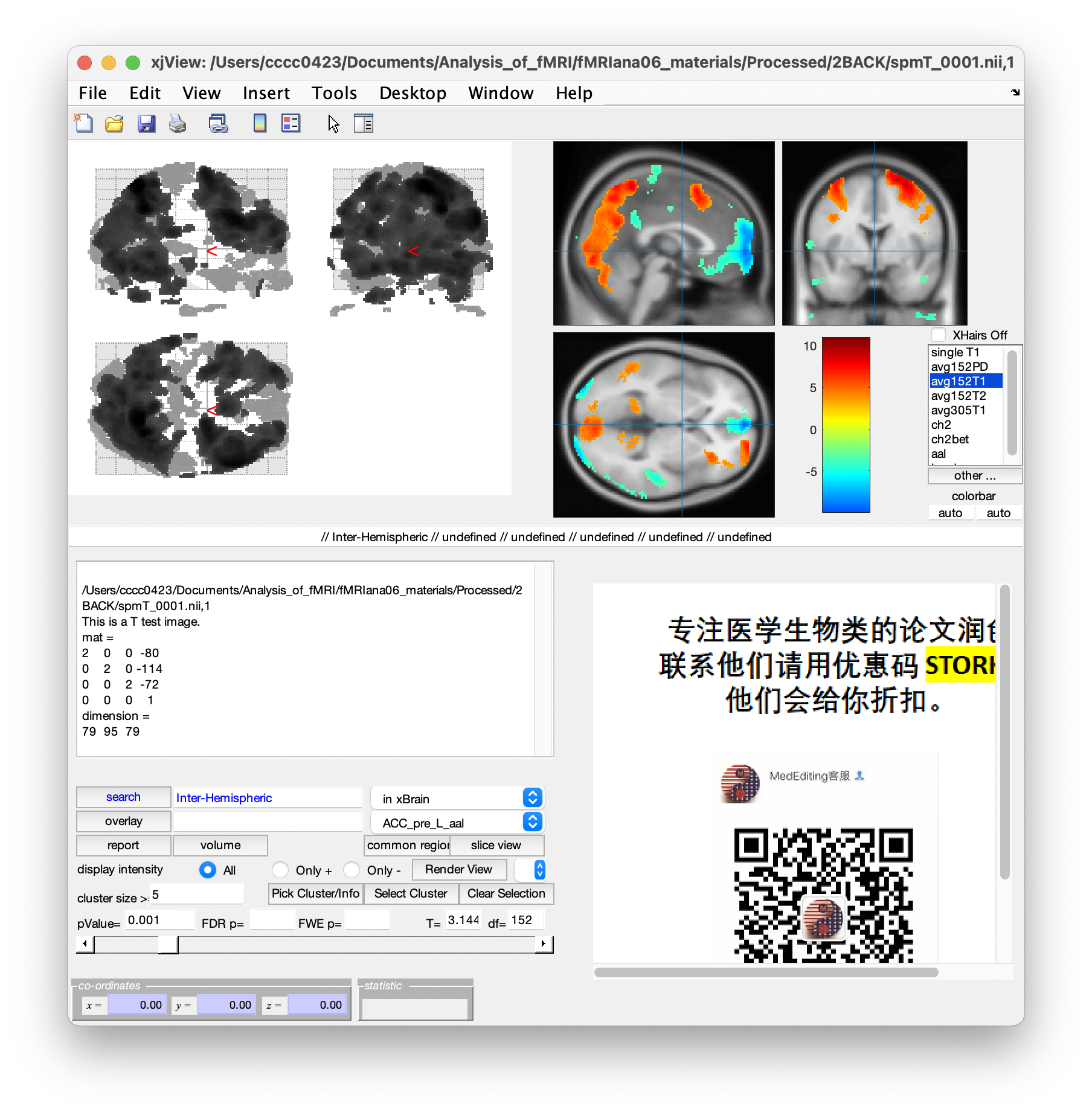
Figure 6.15: 以 xjView 開啟影像檔。
在 display intensity 處,選擇 All 即同時顯示 \(t\) 值為正且為負的區域,另外兩者則分別只顯示正負區域。一般而言,我們只關切正的部分,因此選擇 Only +。我們也可以在下方調整 \(p\) 值,或者 FWE 的 \(p\) 值。此外,FDR 也是一種進行 multiple comparison correction 的方法,如果使用 FWE 發現都沒有腦區是活躍的,或許可以考慮改用 FDR,能夠放寬一點嚴格的程度。
而我們也可以用手去拉右上的腦圖的座標線, xjView 會提示現在是位於哪個腦區。另外,我們也能透過右側的選單,選擇不同的影像檔,例如 Single T1 即結構影像檔。
xjView 還有一功能為 “slice view”,按下左下選單的 slice view,可以開啟如圖 6.16 的畫面。我們也可以透過左下角的選單調整顯示的方式。
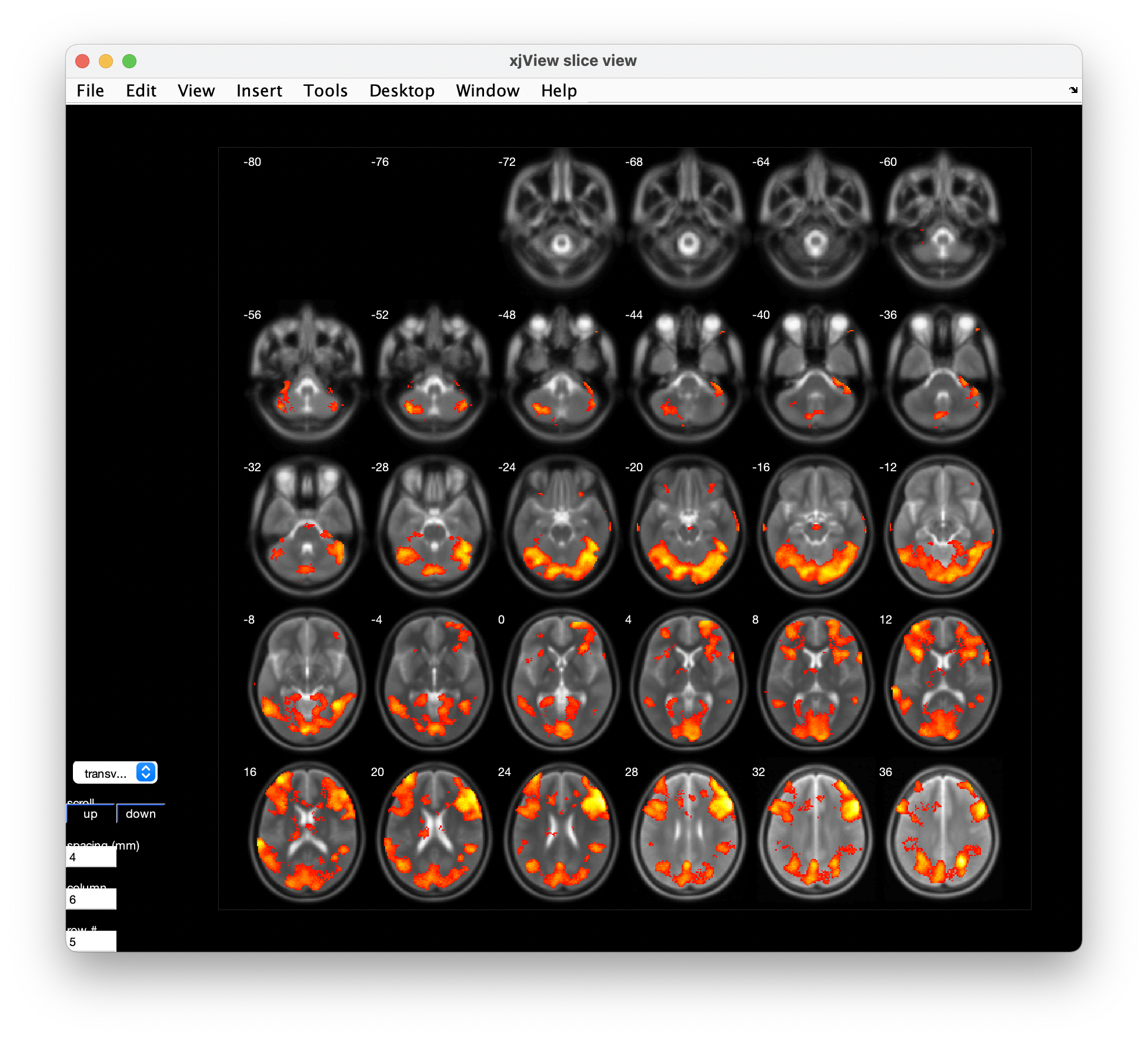
Figure 6.16: Slice view.
另外,也可以在主選單按下 Render view,可以開啟如圖 6.17 的畫面。如果對哪個小圖特別有興趣,也可以點擊放大。
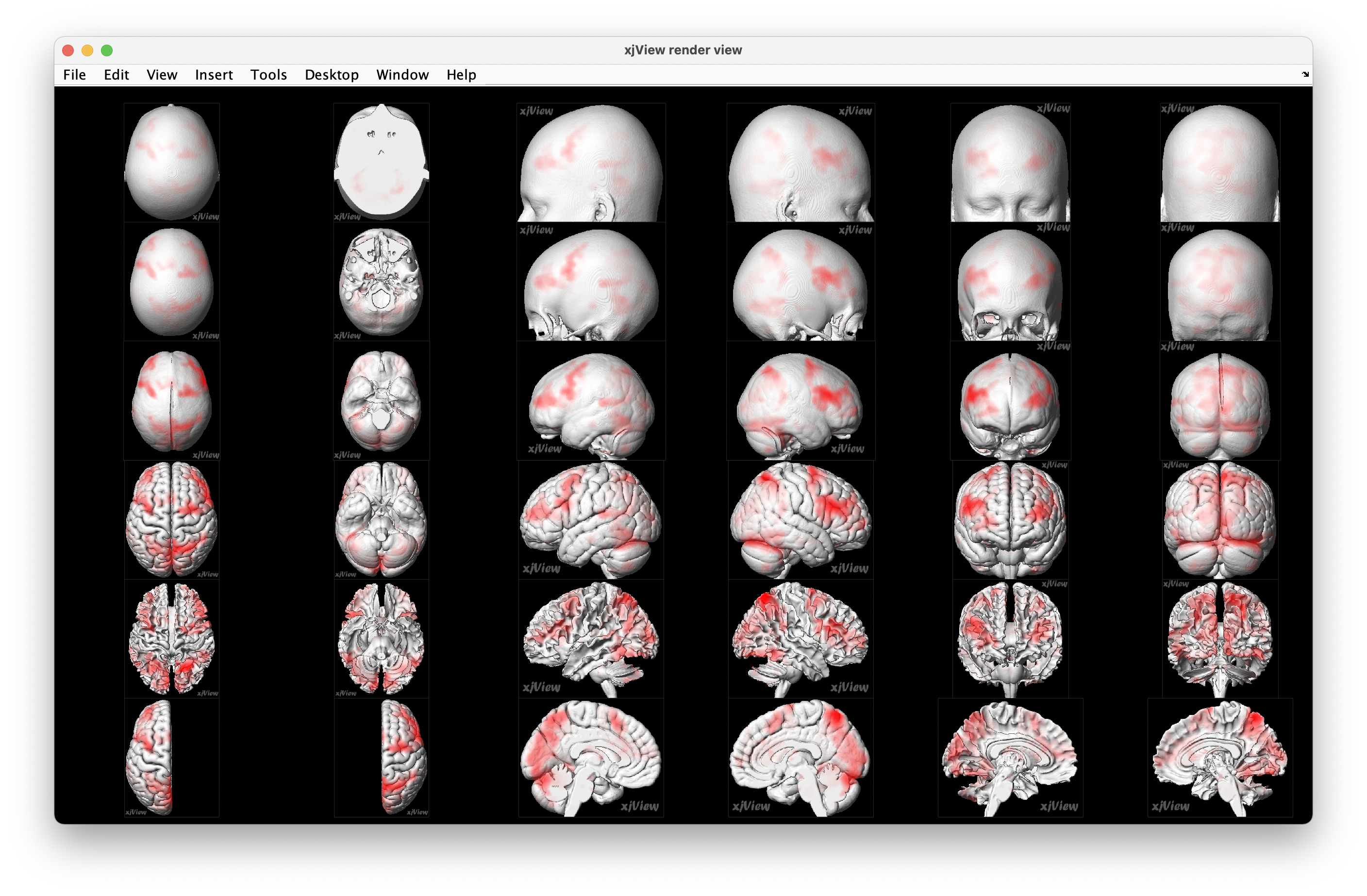
Figure 6.17: Render view.