第 7 章 光谱数据
什么是光谱,简单的说光谱是指光学频谱,简称光谱,是复色光通过色散系统(如光栅、棱镜)进行分光后,依照光的波长(或频率)的大小顺次排列形成的图案。光谱中的一部分可见光谱是电磁波谱中人眼可见的唯一部分,在这个波长范围内的电磁辐射被称作可见光。其他部分光谱根据波段不同可以分为紫外光、红外,近红外,远红外等,如下图7.1:
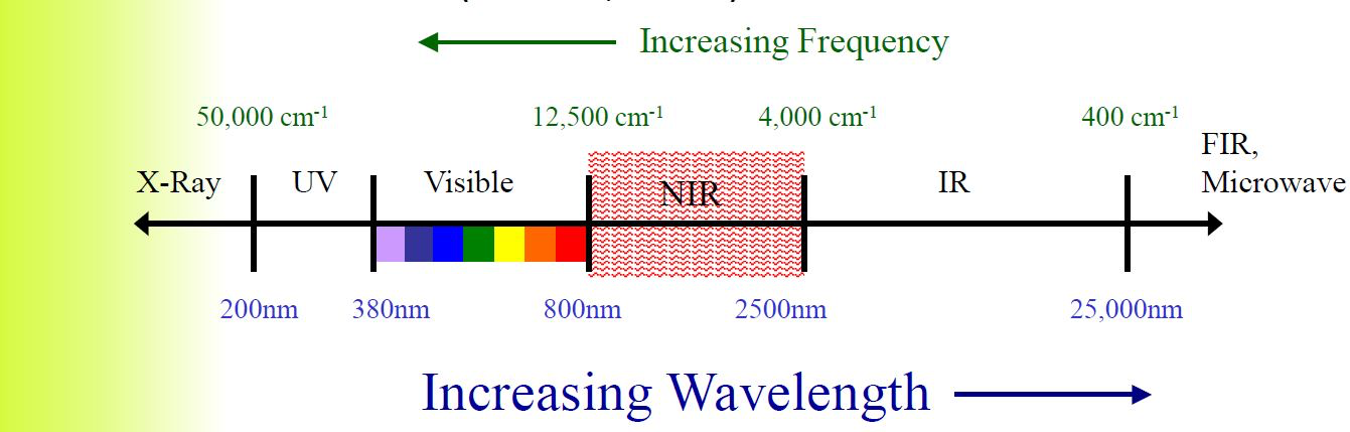
图7.1: 光谱范围
我们通常感兴趣的波段为红外和近红外波段,当光波到达某种物质上时,由于为物质的原子内部电子的运动情况不同,物质的的化学基团和化学键将会与光波的电子产生振动,化学键的振动是量子化的。分子会吸收特定频率的红外线,使化学键由振动基态跃迁至激发态(通常是第一激发态)。在通常状态下,分子的所有共价键几乎全部处于振动的基态。化学键的振动可用简谐振子近似,所以欲使化学键振动能级发生改变其对光谱不同波段将产生不同的反射、折射以及吸收信息将会呈现不同程度的影响,从而得到的反射或吸收光谱将会间接的携带出这个物质的光谱信息,研究不同物质的吸收和反射光情况,具有重要的理论和实际意义,进而衍生出一门专门的学科-光谱学,具体可以参考 维基百科光谱介绍,不同的分子键,其振动方式也更为复杂。例如亚甲基中的碳氢键,就可以以 “对称伸缩”、“非对称伸缩”、“剪刀式摆动”、“左右摇摆”、“前后摇摆”和“扭摆”六种方式振动。前面我们说了理论,接下来让我们看看实例,以我比较感兴趣的近红外波段为例,来看看光谱是什么样子的。这里以4个种源的竹子光谱为例,我们收集了747个来自四个种源地的竹笋样品光谱,想通过近红外光谱对这四个种源进行分类鉴别,首先我们来看一下这批数据。这批数据是近红外数据,其波段为1000-2500 nm.
spec <- readRDS('spec')
#查看数据结构,
dim(spec)
## [1] 747 258
summary(spec$typee)
## BJ G P S
## 270 115 242 120
#有747行,258列,就是说有742个样品,每个样品除去前两列的factor命名列,有256个光谱点,数据量较大
#由于数据较宽,这里只展示前6行前10列
head(spec)[1:10]
## name typee X976.3 X981.7 X987.2 X992.7 X998.3 X1004 X1009.7 X1015.5
## 1 BJ1 BJ 0.37264 0.36946 0.37258 0.37424 0.37214 0.37313 0.37338 0.37324
## 2 BJ1 BJ 0.31845 0.31465 0.31609 0.31694 0.31639 0.31730 0.31853 0.31756
## 3 BJ1 BJ 0.23722 0.23451 0.23447 0.23592 0.23572 0.23577 0.23689 0.23629
## 4 BJ10 BJ 0.37305 0.36960 0.37275 0.37397 0.37356 0.37425 0.37312 0.37329
## 5 BJ10 BJ 0.33899 0.33575 0.33645 0.33723 0.33742 0.33777 0.33672 0.33603
## 6 BJ10 BJ 0.24967 0.24702 0.24831 0.24932 0.24946 0.25022 0.25053 0.25035
#去除列名字里面的X字母,方便后续作图
sub <- sub('^X','',colnames(spec)[-c(1:2)])
colnames(spec)[-c(1:2)] <- sub
head(spec)[1:10]
## name typee 976.3 981.7 987.2 992.7 998.3 1004 1009.7 1015.5
## 1 BJ1 BJ 0.37264 0.36946 0.37258 0.37424 0.37214 0.37313 0.37338 0.37324
## 2 BJ1 BJ 0.31845 0.31465 0.31609 0.31694 0.31639 0.31730 0.31853 0.31756
## 3 BJ1 BJ 0.23722 0.23451 0.23447 0.23592 0.23572 0.23577 0.23689 0.23629
## 4 BJ10 BJ 0.37305 0.36960 0.37275 0.37397 0.37356 0.37425 0.37312 0.37329
## 5 BJ10 BJ 0.33899 0.33575 0.33645 0.33723 0.33742 0.33777 0.33672 0.33603
## 6 BJ10 BJ 0.24967 0.24702 0.24831 0.24932 0.24946 0.25022 0.25053 0.25035下面我们将原始光谱数据进行绘图
matplot(colnames(spec)[-c(1:2)],t(spec[,-c(1:2)]),type = 'l')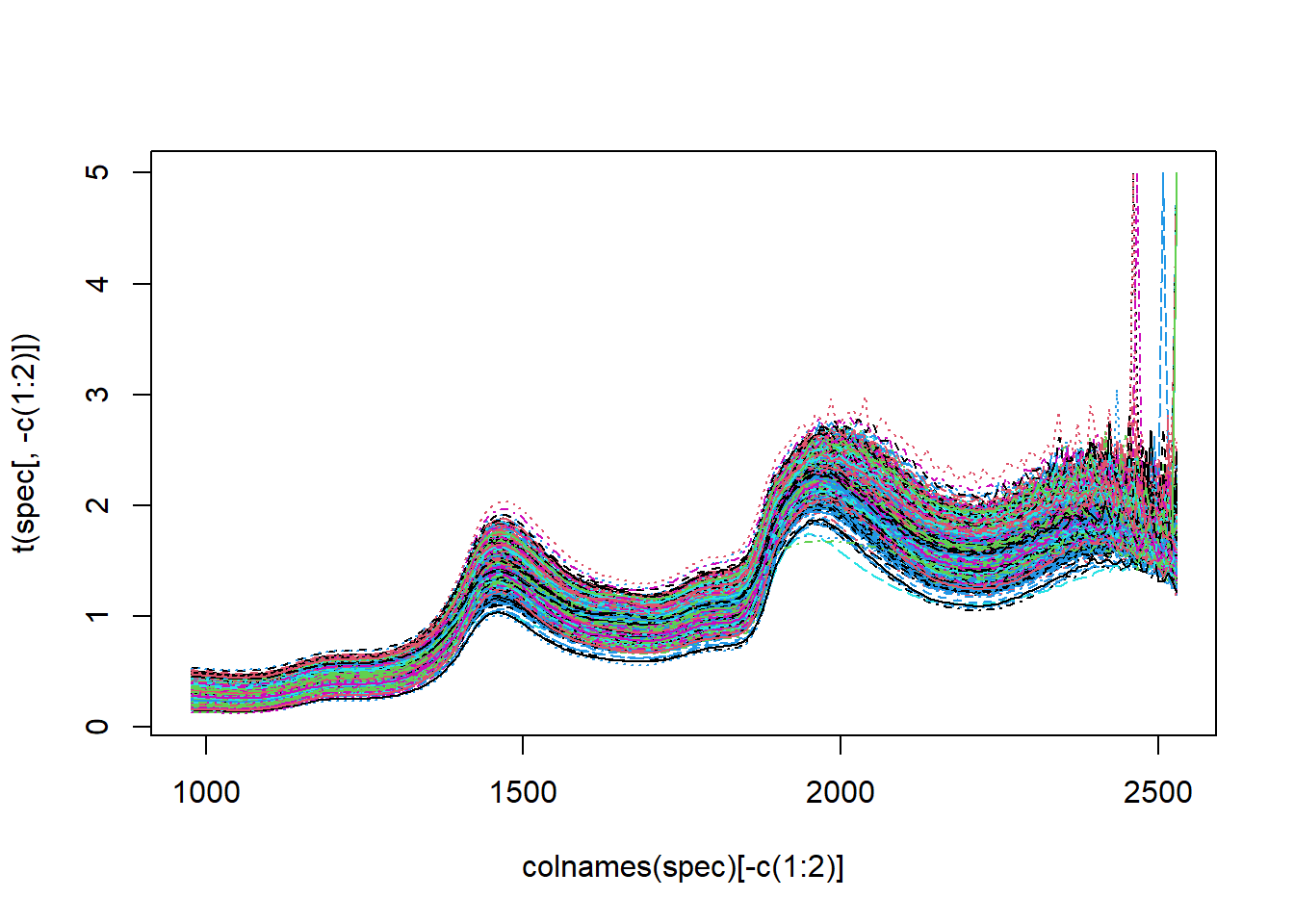
图7.2: 原始光谱
7.1 光谱预处理
可以看出,原始光谱在2500nm左右出现异常值,由于采集光谱的环境,例如温度、湿度、以及采样时的采样角度等影响,会导致光谱出现噪音和散射,尤其是在2500nm附近,因此需要对光谱数据进行有效选择。同时,从图中可以看出,光谱的数据的走势都大致相同,单凭肉眼很难区分各个种源光谱的不同,同时由于光谱的散射与噪音会影响模型预测精度,因此在构建预测模型之前需要对原始光谱进行光谱预处理,
library(e1071)
library(prospectr)## prospectr version 0.2.1 -- 'seville'## check the github repository at: http://github.com/l-ramirez-lopez/prospectrnir <- as.matrix(spec[,-c(1:2,238:258)])
spec_name <- colnames(spec)[-c(1:2,238:258)]
matplot(spec_name,t(nir),type = 'l')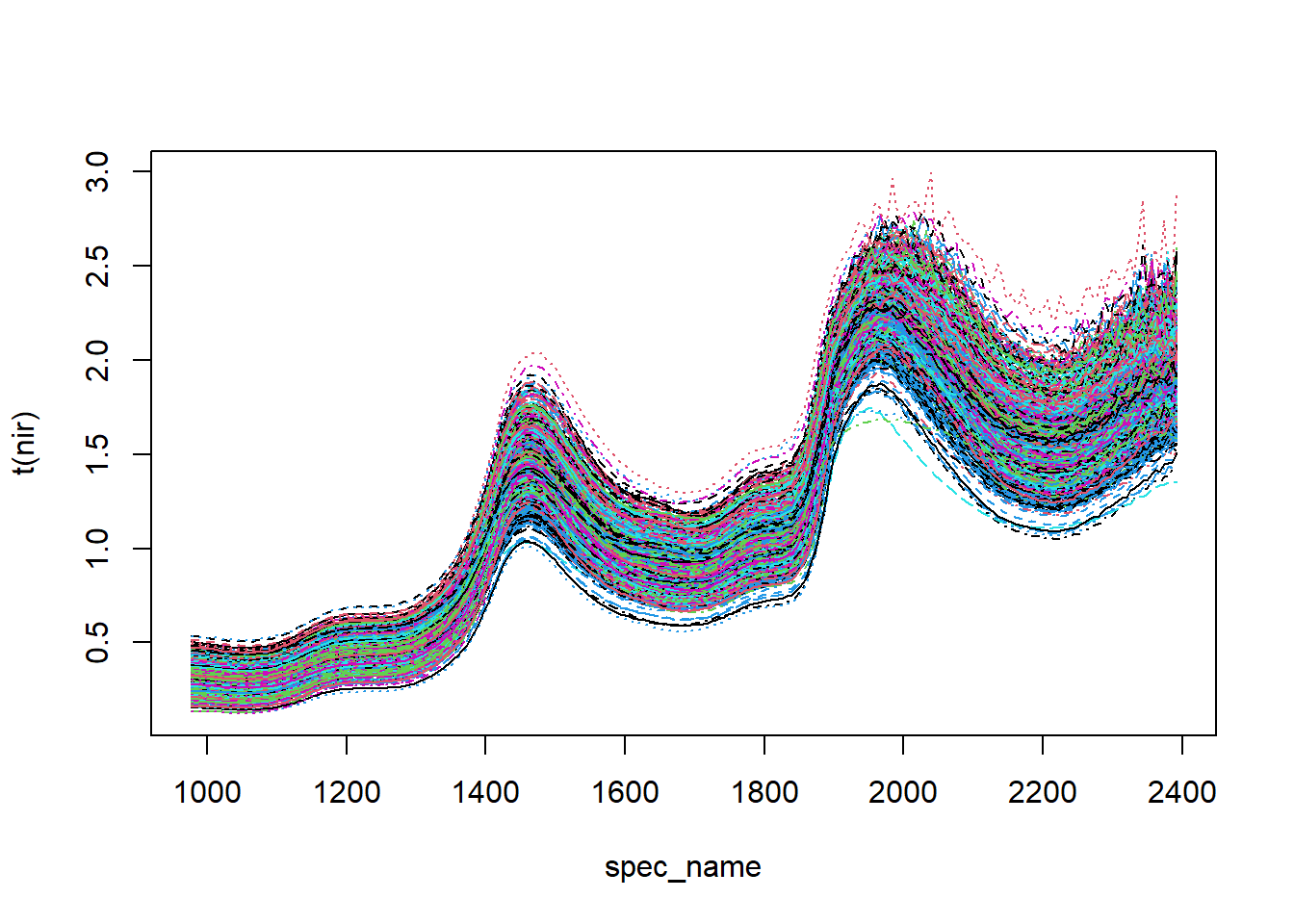
### snv
snv_spec <- standardNormalVariate(nir)
matplot(spec_name,t(snv_spec),type = 'l')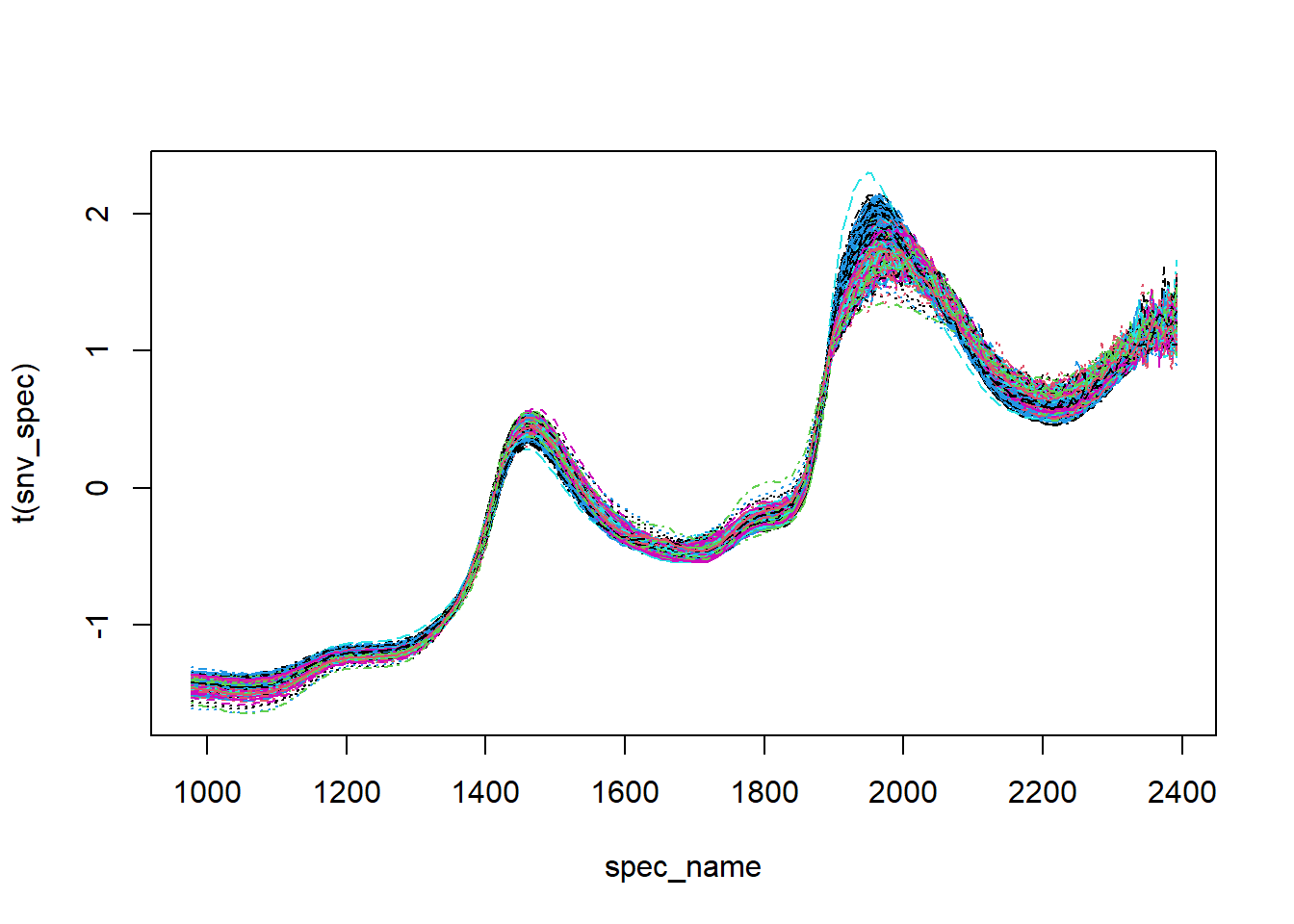
## 1st derivertive
fst_der <- savitzkyGolay(nir,1,2,15)
dim(fst_der)## [1] 747 221matplot(colnames(fst_der),t(fst_der),type = 'l')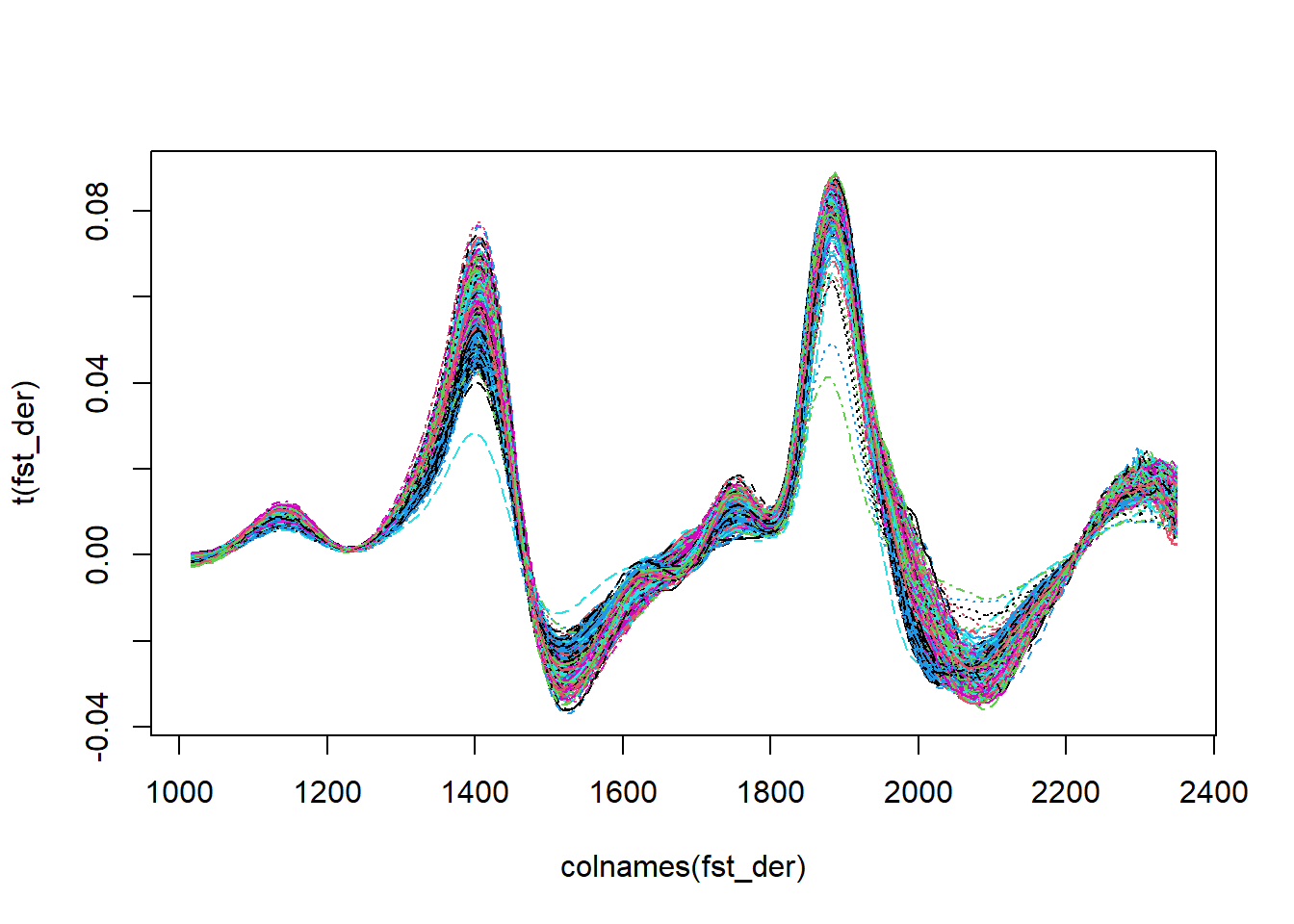
## 2nd derivertive
snd_der <- savitzkyGolay(nir,2,2,15)
matplot(colnames(snd_der),t(snd_der),type = 'l')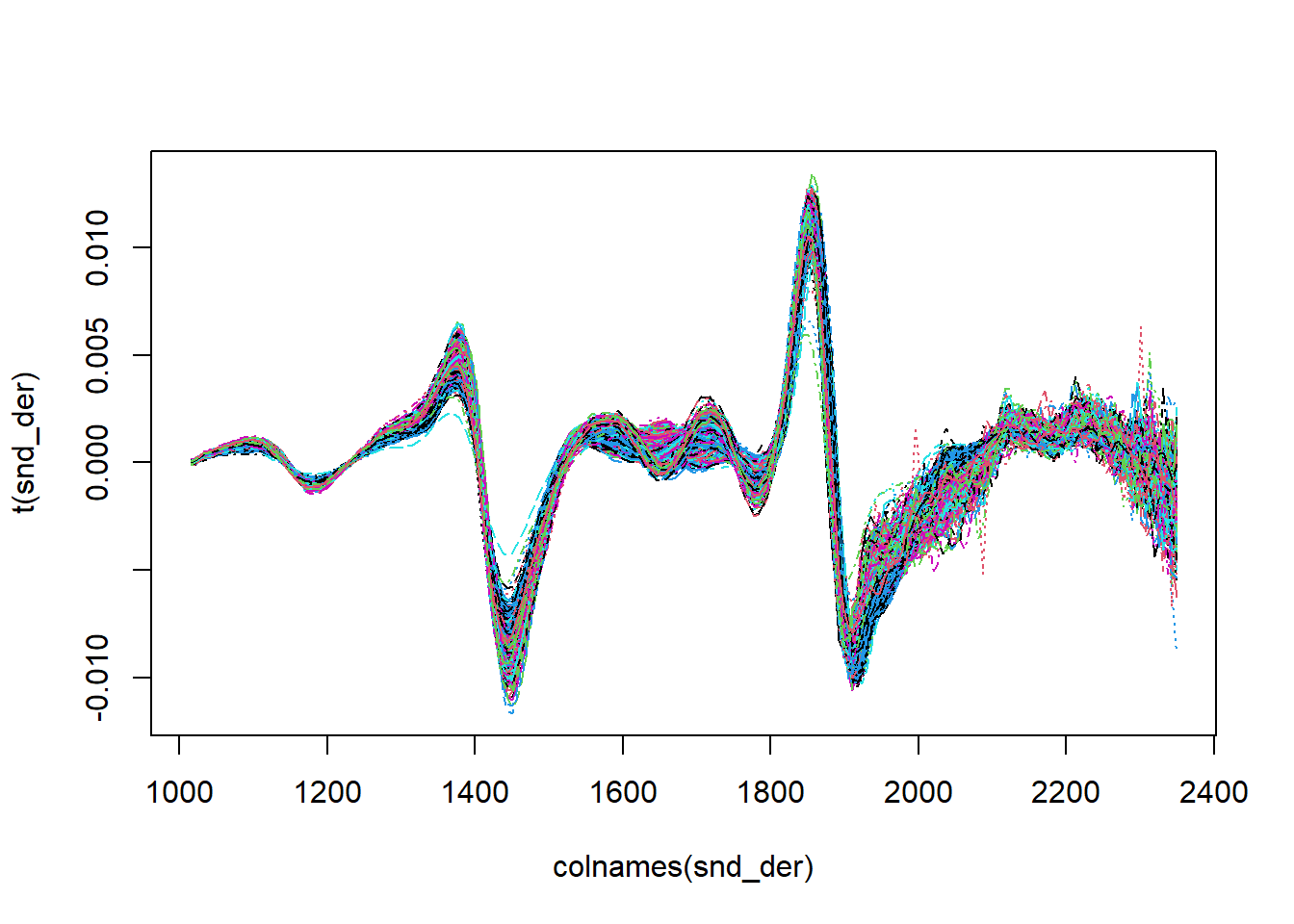
##moving average
snd_der <- savitzkyGolay(nir,0,2,15)
matplot(colnames(snd_der),t(snd_der),type = 'l')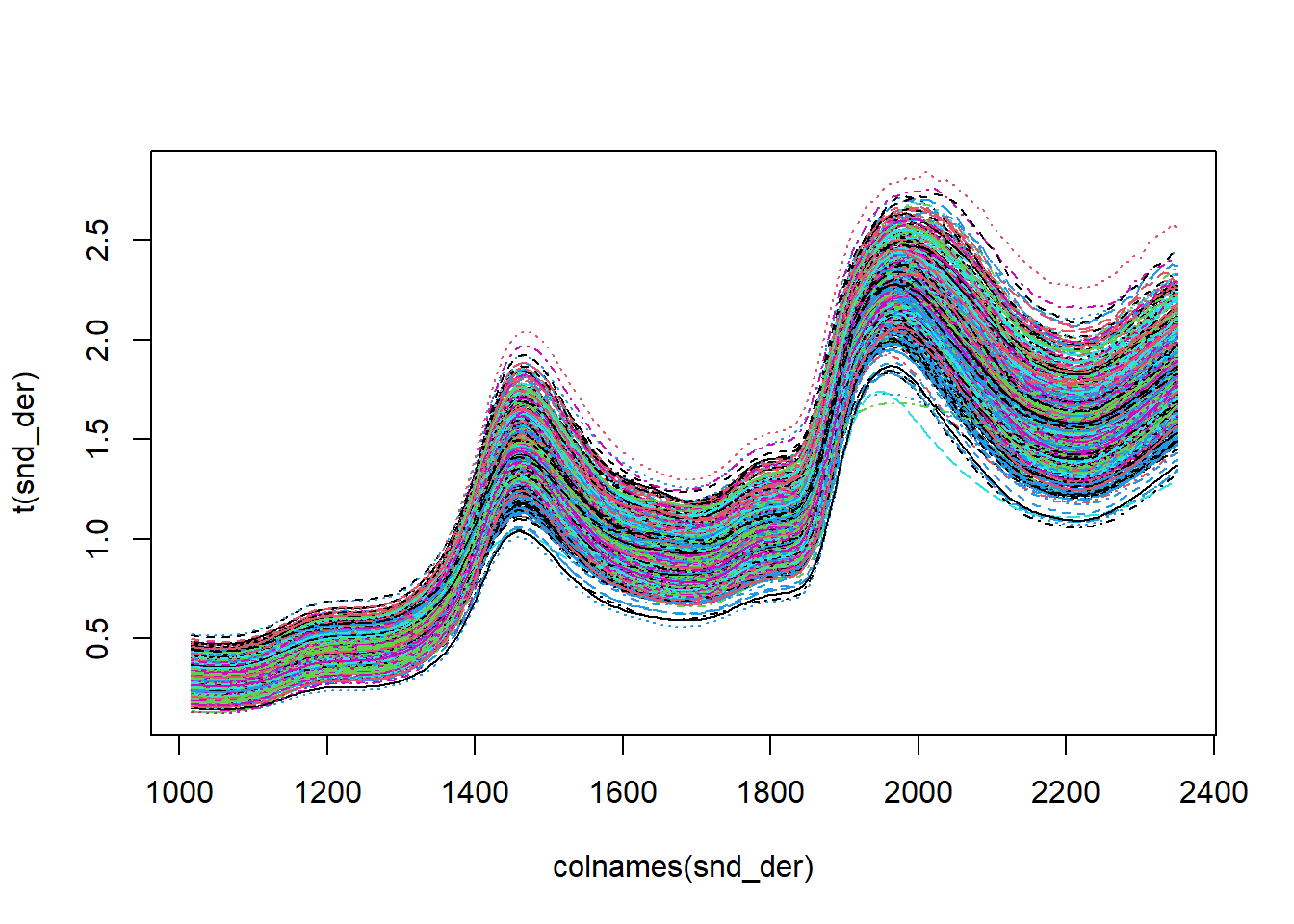
#con rem
cr_spe <- continuumRemoval(nir, type = "A")
matplot(colnames(cr_spe),t(cr_spe),type = 'l')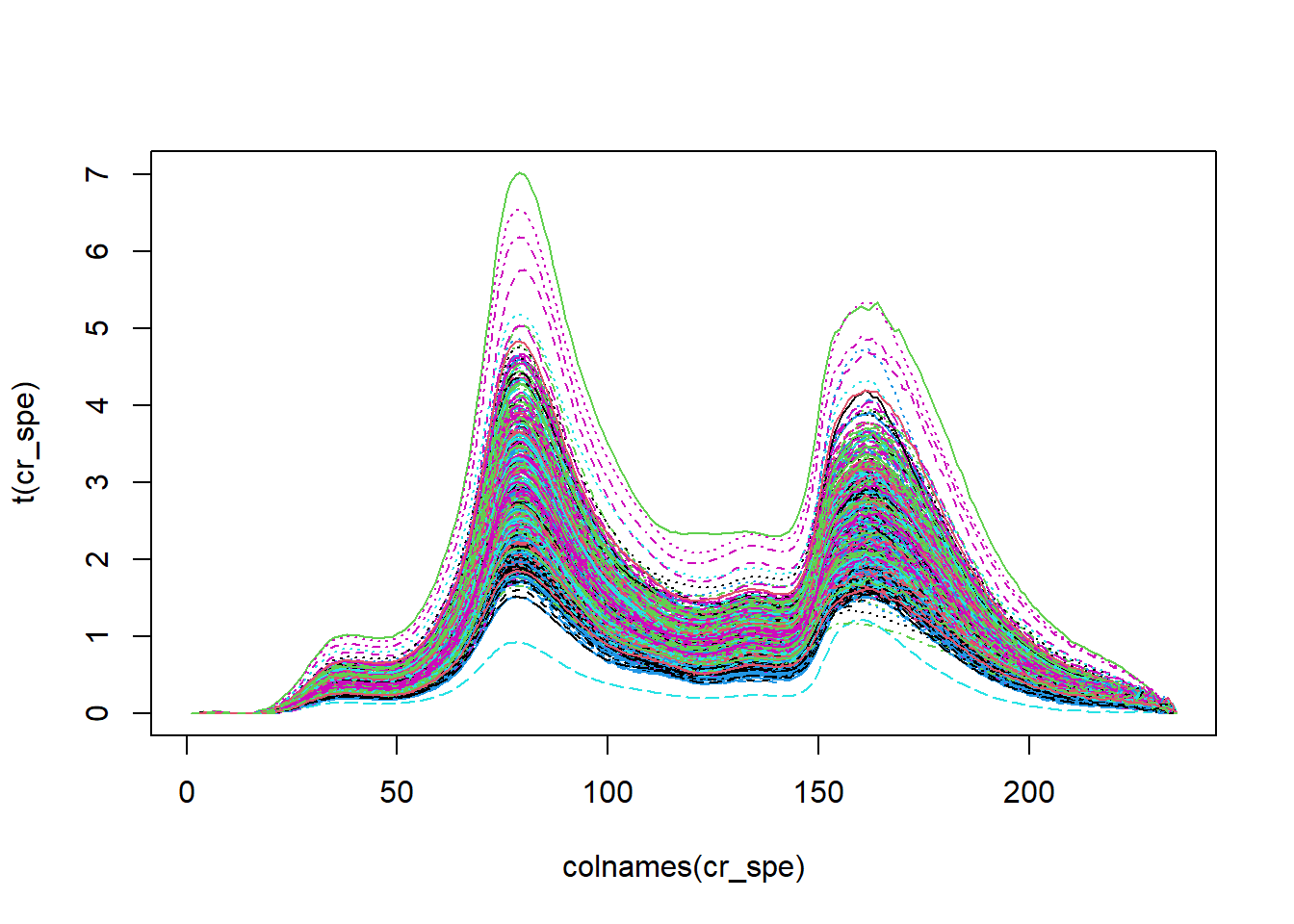
经过光谱预处理有优化后的光谱数据就可以进行模型构建了,然而,不同的预处理光谱所能 达到的模型精度可能会有所不同,我们需要进行一一建模来进行最优模型筛选。