library(cmdstanr)
# 编译模型
mod_state_x77 <- cmdstan_model(
stan_file = "code/state_x77.stan",
compile = TRUE,
cpp_options = list(stan_threads = TRUE)
)
# 准备数据
state_x77 <- data.frame(
x = state.center$x,
y = state.center$y,
state_name = state.name,
state_abb = state.abb,
state_region = state.region,
state_division = state.division,
state.x77, check.names = FALSE
)
state_x77_d <- list(
N = nrow(state_x77), # 观测记录的条数
K = 2, # 协变量个数
x = state_x77[, c("Income", "Murder")], # N x 2 矩阵
y = state_x77[, "Life Exp"] # N 向量
)
nchains <- 4 # 4 条迭代链
# 给每条链设置不同的参数初始值
inits_data <- lapply(1:nchains, function(i) {
list(
alpha = runif(1, 0, 1),
beta = runif(2, 1, 10),
sigma = runif(1, 1, 10)
)
})
# 采样拟合模型
fit_state_x77 <- mod_state_x77$sample(
data = state_x77_d, # 观测数据
init = inits_data, # 迭代初值
iter_warmup = 1000, # 每条链预处理迭代次数
iter_sampling = 2000, # 每条链总迭代次数
chains = nchains, # 马尔科夫链的数目
parallel_chains = 1, # 指定 CPU 核心数,可以给每条链分配一个
threads_per_chain = 1, # 每条链设置一个线程
show_messages = FALSE, # 不显示迭代的中间过程
refresh = 0, # 不显示采样的进度
seed = 20190425 # 设置随机数种子,不要使用 set.seed() 函数
)5 简单线性模型
以一个简单的贝叶斯线性模型为例,介绍贝叶斯统计中的后验分布。继续以 state_x77 数据为例,以贝叶斯线性模型拟合数据,获得参数的后验分布,Stan 语言编码的贝叶斯线性模型如下:
data {
int<lower=0> N; // number of data items
int<lower=0> K; // number of predictors
matrix[N, K] x; // predictor matrix
vector[N] y; // outcome vector
}
parameters {
real alpha; // intercept
vector[K] beta; // coefficients for predictors
real<lower=0> sigma; // error scale
}
model {
// alpha ~ normal(0, 1); // prior
// beta ~ normal(0, 1); // prior
y ~ normal(x * beta + alpha, sigma); // likelihood
}这里采用汉密尔顿蒙特卡洛算法(HMC)做全贝叶斯推断,下面依次编译模型、准备数据、参数初值和迭代设置。Stan 的 R 语言接口 cmdstanr 包可以让这一切在 R 语言环境里做起来比较顺畅。
模型参数估计结果如下:
| variable | mean | median | sd | mad | q5 | q95 | rhat | ess_bulk | ess_tail |
|---|---|---|---|---|---|---|---|---|---|
| alpha | 71.229 | 71.214 | 0.979 | 0.957 | 69.666 | 72.862 | 1.001 | 3029.027 | 3072.625 |
| beta[1] | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | 0.001 | 1.000 | 3506.239 | 3598.459 |
| beta[2] | -0.270 | -0.270 | 0.035 | 0.034 | -0.327 | -0.214 | 1.001 | 2912.894 | 3115.030 |
| sigma | 0.851 | 0.843 | 0.093 | 0.089 | 0.719 | 1.013 | 1.000 | 2948.774 | 3236.519 |
| lp__ | -16.206 | -15.862 | 1.527 | 1.264 | -19.139 | -14.494 | 1.000 | 2436.626 | 2891.515 |
参数的 \(\alpha,\beta_1,\beta_2\) 后验均值估计与普通线性模型的拟合结果非常一致。采样结果可以直接传递给 bayesplot 包(Gabry 等 2019),绘制参数迭代的轨迹图和后验分布图。
library(ggplot2)
library(bayesplot)
library(patchwork)
# 参数的后验分布
p1 <- mcmc_hist(fit_state_x77$draws(c("alpha", "beta[1]", "beta[2]")),
facet_args = list(
labeller = ggplot2::label_parsed,
strip.position = "top",
ncol = 1
)
) + theme_classic()
# 参数的迭代轨迹
p2 <- mcmc_trace(fit_state_x77$draws(c("alpha", "beta[1]", "beta[2]")),
facet_args = list(
labeller = ggplot2::label_parsed,
strip.position = "top",
ncol = 1
)
) + theme_classic() + theme(legend.title = element_blank())
# 绘图
p1 | p2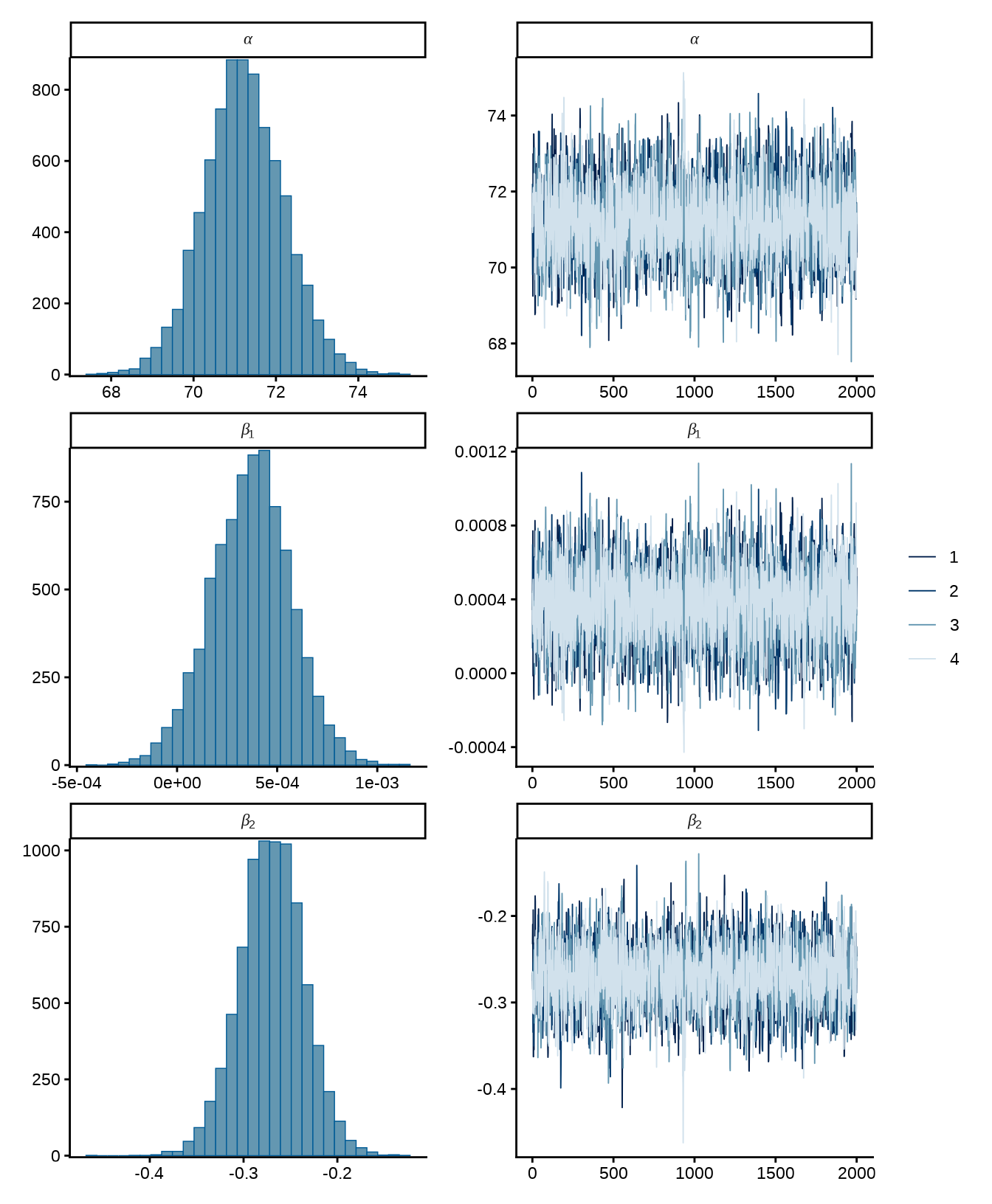
从参数的迭代轨迹可以看出四条马尔可夫链混合得很好,后验分布图主要用来描述参数的迭代结果,后验分布图可以是直方图或密度图的形式。