Chapter 3 Preparation des donnees H2
### Charger les bibliothèques ###
library(ade4)
library(amt)
library(ape)
library(car)
library(data.table)
library(dplyr)
library(gclus)
library(ggplot2)
library(ggpmisc)
library(ggpubr)
library(gridExtra)
library(janitor)
library(lubridate)
library(mapview)
library(pals)
library(performance)
library(readxl)
library(SciViews)
library(sf)
library(tidyverse)
library(vegan)
options(ggrepel.max.overlaps = Inf)
options(scipen=999)
setwd("C:/Users/Sarah/Desktop/Maitrise/Données/")3.1 Aires d’études par région
Le but de cette section est de créer une polygone de chaque aire d’étude en réalisant un MCP 100% sur tous les points d’un secteur/année.
######## 1. Aires d'études par région ########
# Charger les données
locs8 = readRDS("C:/Users/Sarah/Desktop/Maitrise/Données/Data/locs8.rds")
data_fem <- readRDS(file = "C:/Users/Sarah/Desktop/Maitrise/Données/Data/data_hyp1_final.rds")
# Vérifier le CRS des points
st_crs(locs8) # 32198## Coordinate Reference System:
## User input: EPSG:32198
## wkt:
## PROJCRS["NAD83 / Quebec Lambert",
## BASEGEOGCRS["NAD83",
## DATUM["North American Datum 1983",
## ELLIPSOID["GRS 1980",6378137,298.257222101,
## LENGTHUNIT["metre",1]]],
## PRIMEM["Greenwich",0,
## ANGLEUNIT["degree",0.0174532925199433]],
## ID["EPSG",4269]],
## CONVERSION["Quebec Lambert Projection",
## METHOD["Lambert Conic Conformal (2SP)",
## ID["EPSG",9802]],
## PARAMETER["Latitude of false origin",44,
## ANGLEUNIT["degree",0.0174532925199433],
## ID["EPSG",8821]],
## PARAMETER["Longitude of false origin",-68.5,
## ANGLEUNIT["degree",0.0174532925199433],
## ID["EPSG",8822]],
## PARAMETER["Latitude of 1st standard parallel",60,
## ANGLEUNIT["degree",0.0174532925199433],
## ID["EPSG",8823]],
## PARAMETER["Latitude of 2nd standard parallel",46,
## ANGLEUNIT["degree",0.0174532925199433],
## ID["EPSG",8824]],
## PARAMETER["Easting at false origin",0,
## LENGTHUNIT["metre",1],
## ID["EPSG",8826]],
## PARAMETER["Northing at false origin",0,
## LENGTHUNIT["metre",1],
## ID["EPSG",8827]]],
## CS[Cartesian,2],
## AXIS["easting (X)",east,
## ORDER[1],
## LENGTHUNIT["metre",1]],
## AXIS["northing (Y)",north,
## ORDER[2],
## LENGTHUNIT["metre",1]],
## USAGE[
## SCOPE["Topographic mapping (medium and small scale)."],
## AREA["Canada - Quebec."],
## BBOX[44.99,-79.85,62.62,-57.1]],
## ID["EPSG",32198]]## Simple feature collection with 6 features and 2 fields
## Geometry type: POINT
## Dimension: XY
## Bounding box: xmin: -379255 ymin: 394616.8 xmax: -377263.9 ymax: 395095
## Projected CRS: NAD83 / Quebec Lambert
## long_proj lat_proj geometry
## 1...1 -378650.8 394721.7 POINT (-378650.8 394721.7)
## 2...2 -378699.9 394773.4 POINT (-378699.9 394773.4)
## 3...3 -379255.0 394765.7 POINT (-379255 394765.7)
## 4...4 -377263.9 395095.0 POINT (-377263.9 395095)
## 5...5 -379192.0 394733.8 POINT (-379192 394733.8)
## 6...6 -379172.1 394616.8 POINT (-379172.1 394616.8)## [1] 258# Faire les tracks (un par site)
tracks_dv <- make_track(locs8, long_proj, lat_proj, Date_Heure_QC, Site, crs = 32198)
# Faire le MCP 100% par site
mcp_dv <- tracks_dv %>%
nest(data = -Site) %>%
mutate(mcp = map(data, ~ hr_mcp(., levels = 1)), n = map_int(data, nrow)) %>%
hr_to_sf(mcp, Site, n)
st_crs(mcp_dv) # Ok, encore en 32198## Coordinate Reference System:
## User input: EPSG:32198
## wkt:
## PROJCRS["NAD83 / Quebec Lambert",
## BASEGEOGCRS["NAD83",
## DATUM["North American Datum 1983",
## ELLIPSOID["GRS 1980",6378137,298.257222101,
## LENGTHUNIT["metre",1]]],
## PRIMEM["Greenwich",0,
## ANGLEUNIT["degree",0.0174532925199433]],
## ID["EPSG",4269]],
## CONVERSION["Quebec Lambert Projection",
## METHOD["Lambert Conic Conformal (2SP)",
## ID["EPSG",9802]],
## PARAMETER["Latitude of false origin",44,
## ANGLEUNIT["degree",0.0174532925199433],
## ID["EPSG",8821]],
## PARAMETER["Longitude of false origin",-68.5,
## ANGLEUNIT["degree",0.0174532925199433],
## ID["EPSG",8822]],
## PARAMETER["Latitude of 1st standard parallel",60,
## ANGLEUNIT["degree",0.0174532925199433],
## ID["EPSG",8823]],
## PARAMETER["Latitude of 2nd standard parallel",46,
## ANGLEUNIT["degree",0.0174532925199433],
## ID["EPSG",8824]],
## PARAMETER["Easting at false origin",0,
## LENGTHUNIT["metre",1],
## ID["EPSG",8826]],
## PARAMETER["Northing at false origin",0,
## LENGTHUNIT["metre",1],
## ID["EPSG",8827]]],
## CS[Cartesian,2],
## AXIS["easting (X)",east,
## ORDER[1],
## LENGTHUNIT["metre",1]],
## AXIS["northing (Y)",north,
## ORDER[2],
## LENGTHUNIT["metre",1]],
## USAGE[
## SCOPE["Topographic mapping (medium and small scale)."],
## AREA["Canada - Quebec."],
## BBOX[44.99,-79.85,62.62,-57.1]],
## ID["EPSG",32198]]##
## Attachement du package : 'rnaturalearthdata'## L'objet suivant est masqué depuis 'package:rnaturalearth':
##
## countries110canada <- ne_countries(country = "canada")
# Subset des points
pts_verif = locs8 %>%
sample_n(nrow(locs8)/10) # 10%
ggplot(data = canada) +
geom_sf() +
geom_sf(data = mcp_dv, fill = "lightblue") +
geom_sf(data = pts_verif, size = 2, shape = 21, fill = "darkred") +
coord_sf(xlim = c(-78, -64), ylim = c(51, 45), expand = FALSE)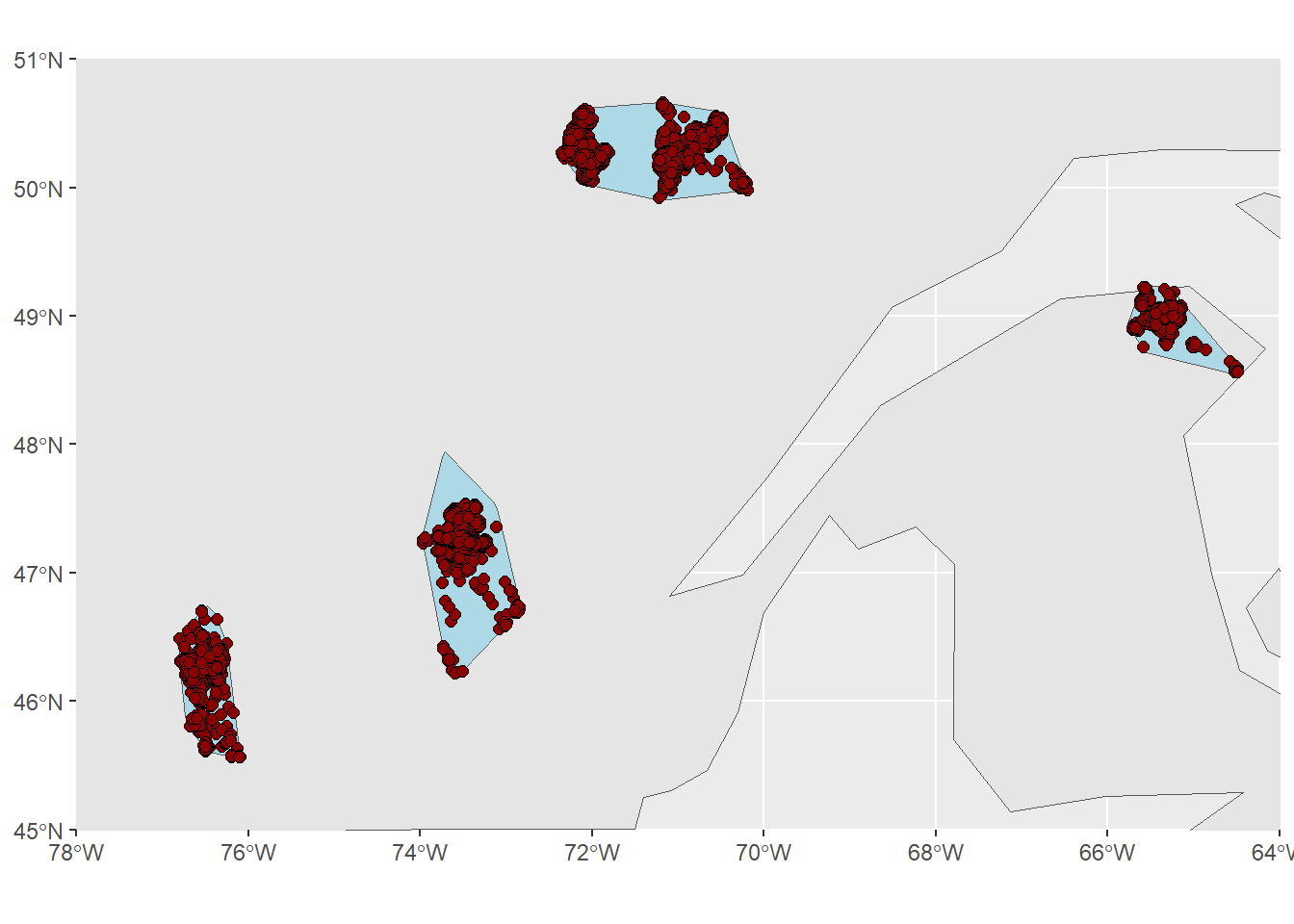
# Ajouter un buffer au tour
# 95% : 0.575km de distance par heure, donc *3 arrondi on va mettre 2km pour être sûrs.
mcp_dv # Units = mètres## Simple feature collection with 4 features and 5 fields
## Geometry type: POLYGON
## Dimension: XY
## Bounding box: xmin: -637960.1 ymin: 205917.9 xmax: 294746.7 ymax: 742987.8
## Projected CRS: NAD83 / Quebec Lambert
## level what area Site n
## 1 1 estimate 10137703532 [m^2] Mauricie 115429
## 2 1 estimate 10015382589 [m^2] SLSJ 108551
## 3 1 estimate 4897277832 [m^2] Outaouais 74605
## 4 1 estimate 3685249817 [m^2] Gaspesie 11869
## geometry
## 1 POLYGON ((-330473.9 314992....
## 2 POLYGON ((-121304.3 671323....
## 3 POLYGON ((-592742.1 206783,...
## 4 POLYGON ((292080 521330.8, ...3.2 Intersection avec les cartes
Ensuite, je fais l’intersection entre l’aire d’étude calculée à la section précédente et la carte écoforestière correspondante pour chaque secteur/année. Puisque c’est le même principe qui se répète, j’ai mis seulement Mauricie 2015 en example.
# Charger les données
scores_hab <- readRDS("C:/Users/Sarah/Desktop/Maitrise/Données/Data/scores_nourriture_final.rds")
mcp_dv2 <- readRDS("C:/Users/Sarah/Desktop/Maitrise/Données/Data/mcp_dv2.rds")
locs8 <- readRDS("C:/Users/Sarah/Desktop/Maitrise/Données/Data/locs8.rds")
mcp_Mauricie = mcp_dv2[1,]
mcp_SLSJ = mcp_dv2[2,]
mcp_Outaouais = mcp_dv2[3,]
mcp_Gaspesie = mcp_dv2[4,]
# Créer les catégories Eau et Agricole dans HABOURS
coter_eau <- c("EAU", "ILE", "INO")
coter_agri <- c("A", "AF")
coter_anthro <- c("ANT", "NF", "GR", "RO", "CFO","AEP", "DEP")
#### 2015: Locs Mauricie ####
locs8 %>% as.data.frame %>%
filter(Annee == "2015") %>%
group_by(Site) %>%
summarise(n = n()) # Mauricie## # A tibble: 1 × 2
## Site n
## <fct> <int>
## 1 Mauricie 13046 # Charger la carte
carte_2015 <- sf::st_read("C:/Users/Sarah/Desktop/Maitrise/Données/Data/Ours_noir_mai_2022/Peup_Ecofor_Sites_Etude_Ours_2016_2020_new.gdb",
layer = "Peupl_Eco_mauricie_2016")## Reading layer `Peupl_Eco_mauricie_2016' from data source
## `C:\Users\sarah\Desktop\Maitrise\Données\Data\Ours_noir_mai_2022\Peup_Ecofor_Sites_Etude_Ours_2016_2020_new.gdb'
## using driver `OpenFileGDB'## Warning in CPL_read_ogr(dsn, layer, query, as.character(options), quiet, : GDAL
## Message 1: organizePolygons() received a polygon with more than 100 parts. The
## processing may be really slow. You can skip the processing by setting
## METHOD=SKIP, or only make it analyze counter-clock wise parts by setting
## METHOD=ONLY_CCW if you can assume that the outline of holes is counter-clock
## wise defined## Simple feature collection with 471679 features and 97 fields
## Geometry type: MULTIPOLYGON
## Dimension: XY
## Bounding box: xmin: -458007.4 ymin: 250841.2 xmax: -320488.3 ymax: 489989.8
## Projected CRS: NAD83 / Quebec Lambert # Corriger le nom des habitats
carte_2015$HABOURS <- recode_factor(carte_2015$HABOURS,
"Coupe_05" = "C_0-5", #1
"Coupe_610" = "C_6-10", #2
"Brulis_020" = "BR_0-20", #3
"Coupe_1120" = "C_11-20", #4
"M_50+" = "M_50", #5
"M_30" = "M_30", #6
"F_30" = "F_30", #7
"F_50+" = "F_50", #8
"PES_30" = "PES_30", #9
"DS_DH" = "DS-DH", #10
"PES_50+" = "PES_50", #11
"SAP_30" = "SAP_30", #12
"SAP_50+" = "SAP_50",
"EAU" = "Eau") %>% #13
droplevels()
# Créer l'intersection entre les MCP et la carte
inter_2015 <- st_intersection(mcp_Mauricie, carte_2015)## Warning: attribute variables are assumed to be spatially constant throughout
## all geometries