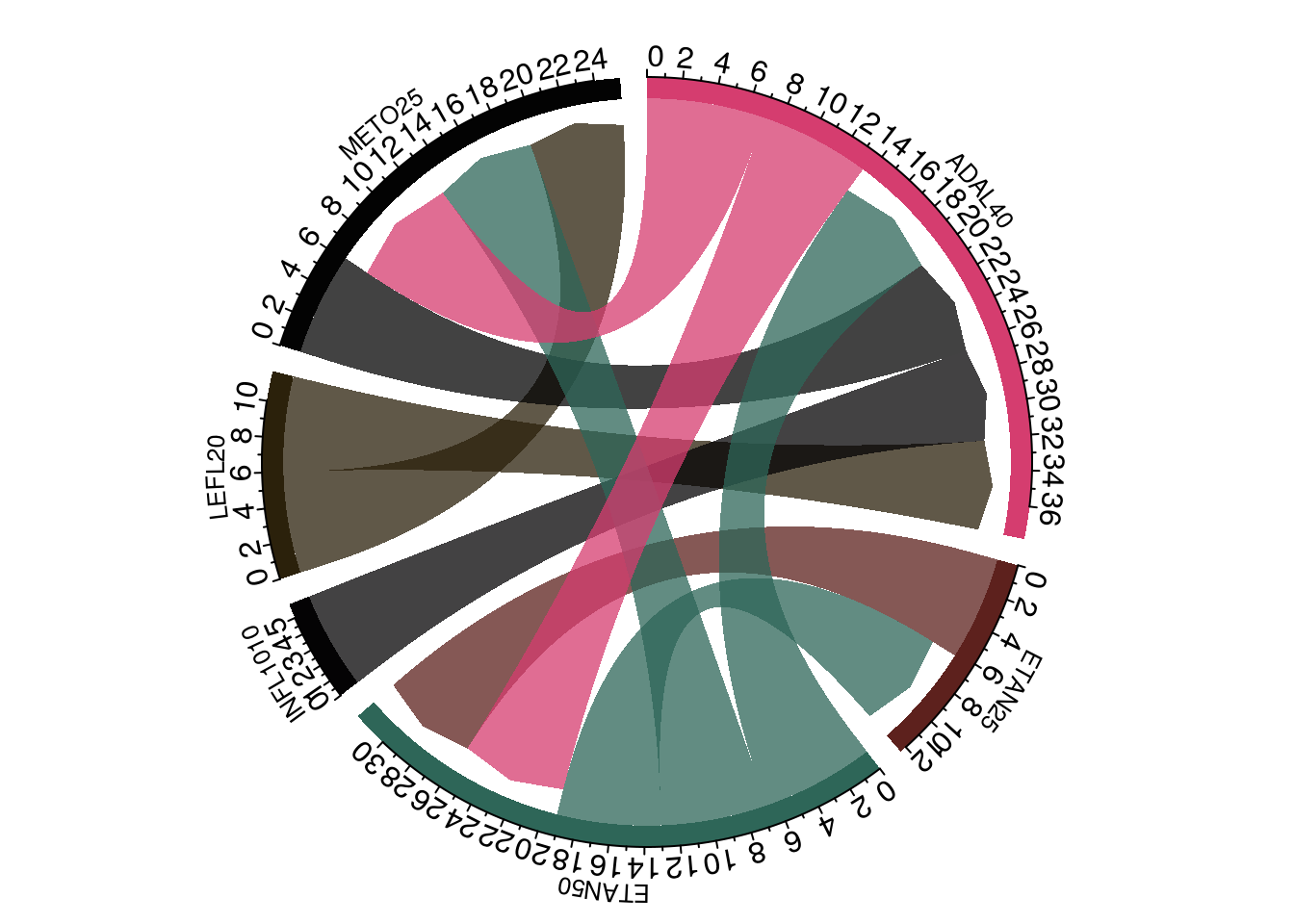
3.9 Explore - O que - Troca de medicamentos - D
3.9.1 Conceituação
Troca de medicamentos relativo ao Protocolo Clínico e Diretrizes Terapêuticas (PCDT).
3.9.2 Interpretação
- As setas indicam a direção da troca.
- Os números indicam o logarítimo do número de usuários.
- As siglas indicam os medicamentos.
3.9.3 Usos
- Identificar o padrão de troca de medicamentos relativo ao número de usuários.
3.9.4 Limitações
- São identificadas trocas de medicamentos, não se foi a terceira ou quarta opção terapêutica, por exemplo.
3.9.5 Fonte
- DATASUS/MS - Ministério da Saúde, Departamento de Informática do SUS [ftp://ftp.datasus.gov.br/dissemin/publicos/SIASUS/200801_/Dados/].
- IBGE - Instituto Brasileiro de Geografia e Estatística [https://educa.ibge.gov.br/jovens/conheca-o-brasil/populacao/18318-piramide-etaria.html]
3.9.6 Métodos de Cálculo
Não há cálculo, apenas a aplicação de filtros.
3.9.7 Categorias Sugeridas para Análise
- Unidade geográfica: região ampliada de saúde e região de saúde.
- Classificação Anatômico-químico-terapêutica (ATC)
- Forma de Organização da SIGTAP - Sistema de Gerenciamento da Tabela de Procedimentos, Medicamentos e OPM do SUS.
- Avaliar o perfil da geral do medicamento em outros PCDT.
3.9.8 Dados Estatísticos e Comentários
Visualizar no painel SABEIS > Explore.
3.9.9 Notas
Descrito conforme metodologia RIPSA. Vide a seção fonte.
3.9.10 Visualização
Fluxo.
3.9.11 Conjunto de dados e Código-fonte
Pré-requisitos
Baixe o arquivo db_sabeis.tf_troca.csv na pasta dataset.
Certifique-se de que os pacotes R abaixo estejam instalados.
library(tidyverse)
library(viridis)
library(patchwork)
library(circlize)R script
tf_troca = read.csv(file = "db_sabeis.tf_troca.csv")
data_long = subset(tf_troca, co_pcdt == 8)
data_long$co_pcdt = NULL
data_long$sg_procedimentob2 = NULL
data_long$qt_cns_paciente = log(data_long$qt_cns_paciente)
circos.clear()
circos.par(
start.degree = 90,
gap.degree = 4,
track.margin = c(-0.1, 0.1),
points.overflow.warning = FALSE
)
par(mar = rep(0, 4))
# color palette
mycolor <- viridis(
10,
alpha = 1,
begin = 0,
end = 1,
option = "D"
)
mycolor <- mycolor[sample(1:10)]
# Base plot
chordDiagram(
x = data_long,
# grid.col = mycolor,
transparency = 0.25,
directional = 1,
direction.type = c("arrows", "diffHeight"),
diffHeight = -0.04,
annotationTrack = "grid",
annotationTrackHeight = c(0.05, 0.1),
link.arr.type = "big.arrow",
link.sort = TRUE,
link.largest.ontop = TRUE
)
# Add text and axis
circos.trackPlotRegion(
track.index = 1,
bg.border = NA,
panel.fun = function(x, y) {
xlim = get.cell.meta.data("xlim")
sector.index = get.cell.meta.data("sector.index")
# Add names to the sector.
circos.text(
x = mean(xlim),
y = 3.2,
labels = sector.index,
facing = "bending",
cex = 0.8
)
# Add graduation on axis
circos.axis(
h = "top",
major.at = seq(
from = 0,
to = xlim[2],
by = ifelse(
test = xlim[2] > 10,
yes = 2,
no = 1
)
),
minor.ticks = 1,
major.tick.percentage = 0.57,
labels.niceFacing = FALSE
)
}
)