BLOQUE 2: Técnicas de modelos supervisados en datos de Sección Cruzada
Clase 1. La validación de modelos
Un paso importante en el proceso de análisis de datos es buscar alguna manera imparcial de analizar el desempeño de un modelo. En general, no es sensato utilizar magnitudes que analizan el grado de ajuste de un modelo (\(R^2\), ECM, etc…) ya que- en general- cuanto mayor sea el conjunto de variables utilizado en un modelo, mayor será el grado de ajuste (y menor su ECM). Sin embargo, ello no quiere decir que el modelo sea más útil para realizar una predicción.
Técnica 1: Entrenamiento+Test
Una técnica sencilla para analizar cómo se desenvuelve un modelo es la partición entre entrenamiento y test. Generalmente, se elige una proporción alta para el entrenamiento (en torno al 70%) y el resto se dedica a test:
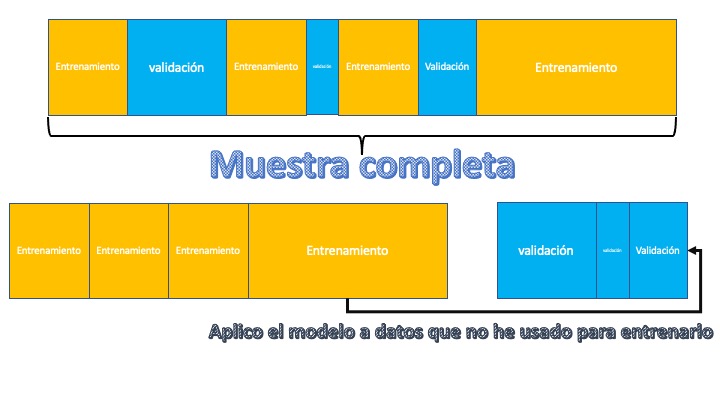
FIG 1: División entre entrenamiento y test.
Esta elección se realiza de manera aleatoria de tal forma que la partición realizada nos permita analizar, de manera adecuada, la población de estudio tanto en el entrenamiento de modelos como en su testado. El modo de trabajo es sencillo:
- Elige la muestra de entrenamiento y validación, así como la variable que quiere predecir (\(y_T,y_V\)) y los predictores (los almacenamos en una matriz: \(\bf{X_T,X_v}\))
- Utiliza la muestra de entrenamiento para estimar los modelos que considere (haciendo, por ejemplo, un análisis descriptivo previo sólo a ese conjunto de datos), de tal forma que tiene, por ejemplo, \(N\) modelos: \(y_T=f_1(\bf{X_T})\),\(y_T=f_2(\bf{X_T})\),…,\(y_T=f_N(\bf{X_T})\), donde \(f_1,f_2,...,f_N\) son los modelos.
- con el modelo que ha entrenado, selecciona la muestra de test y se lo aplica, de tal forma que tendrá, por ejemplo con el modelo 1, \(\hat{y}=f_1(\bf{X_V})\) las predicciones que realiza dicho modelo con los datos de validación de \(\bf{X}\). De esta forma, podrá comparar la predicción del modelo (\(\hat{y}\)) con las observaciones que tiene almacenadas del conjunto de validación (\(y_V\)).
- Puede calcular, entonces, alguna medida de error promedio que le interese.
- Esta técnica podría utilizarla como un complemento a la inferencia estadística tradicional. Si considera que una variable es significativa (o relevante), entonces mediante este análisis, debería proporcionarle un error predictivo menor, por ejemplo, que otra variable menos relevante.
- Esta técnica le puede ayudar a elegir modelos (si tiene varios modelos candidatos y quiere quedarse con uno de ellos, puede elegir el que menor error le proporcione en la validación)
- Sin embargo, esta técnica está muy condicionada por la elección del entrenamiento y la validación.
Técnica 2: Validación Cruzada aleatoria
Una manera de mejorar el último aspecto de la técnica anterior, consistirá en tener más información sobre el error de predicción. Para ello, se puede hacer un muestreo tanto de los datos que se destinarán a entrenamiento como aquellos que se utilizarán para validación. La idea de la validación cruzada es hacer “un montón de veces” la partición entre entrenamiento y validación utilizando para ello extracciones aleatorias (sin reemplazamiento) de las muestras. Visualmente, consiste en el esquema de la FIG2:
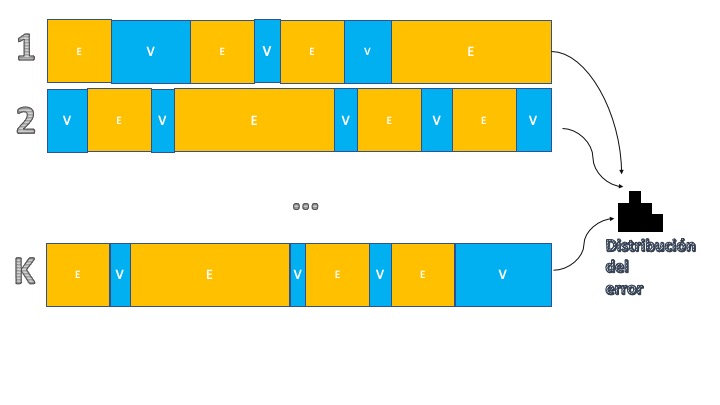
FIG 2: Idea de la validación cruzada aleatoria.
Como se puede ver, consiste en replicar la estructura anterior pero ahora se hará \(K\) veces particionando la muestra entre entrenamiento y validación. Ello nos permitirá tener \(K\) observaciones del error y, por tanto, tener una distribución del error de predicción. Esto es ventajoso, pues nos permite comparar entre modelos de una manera más amplia. Para entender el algoritmo de validación cruzada, tiene aquí un ejemplo en el que se quiere discutir qué modelo es mejor:
\[ Rating=\beta_0+\beta_1Marge+u \] \[ Rating=\beta_0+\beta_1Marge+\beta_2Marge^2+u \] \[ Rating=\beta_0+\beta_1Marge+\beta_2Marge^2+\beta_3Marge^3+u \]
con el objeto de entender cuál es, de esos tres modelos, el que mejor predice el rating:
SIMPSONS<- read_table2("SIMPSONS_FINAL.csv", locale = locale(decimal_mark = ","))
N<-100 #Decido el número de simulaciones que quiero hacer
error1f<-rep(0,N) #Creo un vector para almacenar errores para el modelo 1
error2f<-rep(0,N) #Creo un vector para almacenar errores para el modelo 2
error3f<-rep(0,N) #Creo un vector para almacenar errores para el modelo 3
for(i in 1:N){ #Inicio el bucle
sample <- sample.int(n = nrow(SIMPSONS),size = floor(.7*nrow(SIMPSONS)), replace = F) #me da el sorteo de filas aleatorias
train <- SIMPSONS[sample, ] #Obtengo la muestra de entrenamiento
test <- SIMPSONS[-sample, ] #y la muestra de validación
Mtrain1<-lm(rating_imdb~Marge, data=train)# Entreno el modelo 1 con la muestra de train
Mtrain2<-lm(rating_imdb~Marge+I(Marge^2) data=train) # Entreno el modelo 2 con la muestra de train
Mtrain3<-lm(rating_imdb~Marge+I(Marge^2)+I(Marge^3), data=train) # Entreno el modelo 3 con la muestra de train
prediction_1<-predict(Mtrain1, test) #predigo con el modelo 1 y la muestra de validación
prediction_2<-predict(Mtrain2, test) #predigo con el modelo 2 y la muestra de validación
prediction_3<-predict(Mtrain3, test) #predigo con el modelo 3 y la muestra de validación
error1<-test$rating_imdb-prediction_1 #obtenemos el error de predicción 1 para cada elemento de validación
error2<-test$rating_imdb-prediction_2 #obtenemos el error de predicción 2 para cada elemento de validación
error3<-test$rating_imdb-prediction_3 #obtenemos el error de predicción 3 para cada elemento de validación
error1f[i]<-mean(error1)
error2f[i]<-mean(error2)
error3f[i]<-mean(error3)
}Tenemos, finalmente, tres distribuciones del error: una para cada modelo
FIG 3: Las distribuciones de los errores.
No obtenemos nada que nos sorprenda: los tres modelos predicen de manera muy similar:
> summary(error1f)
Min. 1st Qu. Median Mean 3rd Qu. Max.
-0.183970 -0.048355 -0.007220 -0.001716 0.033049 0.213168
> summary(error2f)
Min. 1st Qu. Median Mean 3rd Qu. Max.
-0.184470 -0.047643 -0.006076 -0.001551 0.030900 0.212045
> summary(error3f)
Min. 1st Qu. Median Mean 3rd Qu. Max.
-0.180119 -0.043825 -0.005959 -0.001566 0.034216 0.208222 Consejos de utilización:
- Si en su pregunta pretende analziar si un factor (o conjunto de factores) son buenos predictores de otro, le recomendamos utilizar la primera técnica (y realizar, además, un análisis previo descriptivo con la muestra de entrenamiento, no con la muestra completa)
- Si su pregunta consiste en analizar el poder predictivo que tendría un conjunto de modelos preseleccionados, puede usar la segunda técnica para obtener una estimación del error de predicción.
Clase 2. Los límites de la validación de modelos
Con el conjunto de datos: datos_clínicos.csv trata de explorar los mecanismos de validación cruzada. Haz los siguientes ejercicios que te servirán para discutir posibles malas prácticas (o prácticas irreales) de esta técnica.
- Utiliza validación cruzada para analizar el impacto de la edad y la diabetes sobre la probabilidad de desarrollar anemia. Diseñe una metodología sensata para poder responder a esa pregunta (desde definir los modelos, el conjunto de entrenamiento y test y la medida de “error” que va a utilizar)
- Ahora, piense que ganaría mucho dinero si obtiene el menor error predictivo (lo que hacen en los hackatones). Desarrolle primeramente, una manera de trabajar poco higiénica y que, seguramente proporcione resultados sesgados. Use esa técnica para asegurar que su error va a ser bajo (quizás más bajo que el de sus compañeros). Ahora piense, a la manera de los Hackatones, cómo obtener un resultado más realista del error de predicción utilizando validación cruzada (pista: quizás tenga que partir la muestra en más trozos)
Propuesta de solución
En el caso 1 queremos aprovechar la validación cruzada para analizar qué variable tiene mayor poder predictivo sobre la variable de interés que es desarrollar o no anemia. Es una variable dicotómica (toma valor 1 si el individuo desarrolla anemia y cero en caso contrario). Entonces, utilizaremos la partición “entrenamiento + test”. De esta forma, el 70% de la muestra lo utilizaremos para entrenar los modelos candidatos y el 30% restante para ver cuál de las variables tiene mayor poder predictivo. Vamos a entrenar tres modelos logísticos:
- M1: Anemia es explicada por la edad del individuo
- M2: Anemia es explicada por la variable “diabetes”
- M3: Anemia es explicada por la edad del individuo y la variable “diabetes”
- M4: Anemia es explicada por la edad del individuo, la variable “diabetes” y un término de interacción entre ambas variables (indicando que el efecto de la edad y la diabetes sobre la anemia depende de ambos efectos simultaneamente).
clinicos <- read_delim("clinicos_final.csv",
delim = ";", escape_double = FALSE, trim_ws = TRUE)
error1f<-c(0) #Creo un vector para almacenar errores para el modelo 1
error2f<-c(0) #Creo un vector para almacenar errores para el modelo 2
error3f<-c(0) #Creo un vector para almacenar errores para el modelo 3
error4f<-c(0) #Creo un vector para almacenar errores para el modelo 3
sample <- sample.int(n = nrow(clinicos),size = floor(.7*nrow(clinicos)), replace = F) #me da el sorteo de filas aleatorias
train <- clinicos[sample, ] #Obtengo la muestra de entrenamiento
test <- clinicos[-sample, ] #y la muestra de validación
Mtrain1<-glm(anaemia~diabetes,family=binomial, data=train)# Entreno el modelo 1 con la muestra de train
Mtrain2<-glm(anaemia~age,family=binomial, data=train)# Entreno el modelo 2 con la muestra de train
Mtrain3<-glm(anaemia~diabetes+age,family=binomial, data=train)# Entreno el modelo 3 con la muestra de train
Mtrain4<-glm(anaemia~diabetes+age+I(diabetes*age),family=binomial, data=train)# Entreno el modelo 4 con la muestra de train
prediction_1<-predict.glm(Mtrain1, test,type="response") #predigo con el modelo 1 y la muestra de validación
prediction_2<-predict.glm(Mtrain2, test,type="response") #predigo con el modelo 2 y la muestra de validación
prediction_3<-predict.glm(Mtrain3, test,type="response") #predigo con el modelo 3 y la muestra de validación
prediction_4<-predict.glm(Mtrain4, test,type="response") #predigo con el modelo 4 y la muestra de validación
anaemia_pred1<-prediction_1>0.5
anaemia_pred2<-prediction_2>0.5
anaemia_pred3<-prediction_3>0.5
anaemia_pred4<-prediction_4>0.5
error1<-test$anaemia!=as.numeric(anaemia_pred1) #obtenemos el error de predicción 1 para cada elemento de validación
error2<-test$anaemia!=as.numeric(anaemia_pred2) #obtenemos el error de predicción 2 para cada elemento de validación
error3<-test$anaemia!=as.numeric(anaemia_pred3) #obtenemos el error de predicción 3 para cada elemento de validación
error4<-test$anaemia!=as.numeric(anaemia_pred4) #obtenemos el error de predicción 4 para cada elemento de validación
error1f<-mean(error1)
error2f<-mean(error2)
error3f<-mean(error3)
error4f<-mean(error4)La medida del error es una decisión sensible que no se puede tomar a la ligera, ya que condicionará muchos de los resultados de su análisis. En este caso, se ha decidido evaluar el porcentaje de veces que, en la muestra de test, cada modelo no acierta con la predicción (tanto si dice que el individuo tiene anemia y no la tiene y viceversa).
Como se ve en los resultados, no parece que ningún modelo se mejor que otro.
> error1f
[1] 0.4666667
> error2f
[1] 0.5
> error3f
[1] 0.4888889
> error4f
[1] 0.5333333De hecho, cometería usted el mismo error si predijera la anemia de un individuo lanzando una moneda al aire. Como verá: >- No tiene nada que ver esta manera de decidir la relevancia de una variable con mirar el p-valor del modelo (le parece convincente?) >- Estamos sujetos a la elección de la muestra de test y entrenamiento. Si la muestra es pequeña (en nuestro caso no es excesivamente grande) hay que tomar los resultados con cautela >- Se puede seguir indagando en otro conjunto de modelos (que se aprenderán pronto).
Respecto al caso 2, como ya se ha comentado, hay que tener cuidado con el uso indiscriminado de la validación cruzada, puesto que podemos “aprender” modelos que obtengan errores pequeños en base a los modelos que funcionan peor en el proceso de entrenar-validar. Por eso es bueno tener una muestra adicional guardada que no se utilizará para la validación cruzada.
Ahora, en la primera línea, nos quedamos con el 15% de los datos que NO VAMOS A TOCAR hasta el final
clinicos <- read_delim("clinicos_final.csv",
delim = ";", escape_double = FALSE, trim_ws = TRUE)
##### voy a quedarme una muestra que sólo usaré al final, como haría Kaggle
sample <- sample.int(n = nrow(clinicos),size = floor(.15*nrow(clinicos)), replace = F) #me da el sorteo de filas aleatorias
Muestra_validacion_final<-clinicos[sample,]
clinicos2<-clinicos[-sample,] #mi muestra sin los datos que me he guardado para jugar a se Kaggle
N<-100
error1f<-rep(0,N) #Creo un vector para almacenar errores para el modelo 1
error2f<-rep(0,N) #Creo un vector para almacenar errores para el modelo 2
error3f<-rep(0,N) #Creo un vector para almacenar errores para el modelo 3
error4f<-rep(0,N) #Creo un vector para almacenar errores para el modelo 4
for(i in 1:N){ #Inicio el bucle
sample <- sample.int(n = nrow(clinicos2),size = floor(.8*nrow(clinicos2)), replace = F) #me da el sorteo de filas aleatorias
train <- clinicos2[sample, ] #Obtengo la muestra de entrenamiento
test <- clinicos2[-sample, ] #y la muestra de validación
Mtrain1<-glm(anaemia~diabetes,family=binomial, data=train)# Entreno el modelo 1 con la muestra de train
Mtrain2<-glm(anaemia~age,family=binomial, data=train)# Entreno el modelo 2 con la muestra de train
Mtrain3<-glm(anaemia~diabetes+age,family=binomial, data=train)# Entreno el modelo 3 con la muestra de train
Mtrain4<-glm(anaemia~diabetes+age+I(diabetes*age),family=binomial, data=train)# Entreno el modelo 4 con la muestra de train
prediction_1<-predict.glm(Mtrain1, test,type="response") #predigo con el modelo 1 y la muestra de validación
prediction_2<-predict.glm(Mtrain2, test,type="response") #predigo con el modelo 2 y la muestra de validación
prediction_3<-predict.glm(Mtrain3, test,type="response") #predigo con el modelo 3 y la muestra de validación
prediction_4<-predict.glm(Mtrain4, test,type="response") #predigo con el modelo 4 y la muestra de validación
anaemia_pred1<-prediction_1>0.5
anaemia_pred2<-prediction_2>0.5
anaemia_pred3<-prediction_3>0.5
anaemia_pred4<-prediction_4>0.5
error1<-test$anaemia!=as.numeric(anaemia_pred1) #obtenemos el error de predicción 1 para cada elemento de validación
error2<-test$anaemia!=as.numeric(anaemia_pred2) #obtenemos el error de predicción 2 para cada elemento de validación
error3<-test$anaemia!=as.numeric(anaemia_pred3) #obtenemos el error de predicción 3 para cada elemento de validación
error4<-test$anaemia!=as.numeric(anaemia_pred4) #obtenemos el error de predicción 4 para cada elemento de validación
error1f[i]<-mean(error1)
error2f[i]<-mean(error2)
error3f[i]<-mean(error3)
error4f[i]<-mean(error4)
}Los resultados que se obtienen son, en promedio:
> mean(error1f)
[1] 0.4460784
> mean(error2f)
[1] 0.452549
> mean(error3f)
[1] 0.4637255
> mean(error4f)
[1] 0.4482353Ahora, con toda la muestra anterior, se entrenan los modelos y, a continuación, se evalúan los errores cometidos sobre la muestra que se ha guardado al principio del experimento
Mtrain1<-glm(anaemia~diabetes,family=binomial, data=clinicos2)# Entreno el modelo 1 con la muestra de train
Mtrain2<-glm(anaemia~age,family=binomial, data=clinicos2)# Entreno el modelo 2 con la muestra de train
Mtrain3<-glm(anaemia~diabetes+age,family=binomial, data=clinicos2)# Entreno el modelo 3 con la muestra de train
Mtrain4<-glm(anaemia~diabetes+age+I(diabetes*age),family=binomial, data=clinicos2)# Entreno el modelo 4 con la muestra de train
prediction_1<-predict.glm(Mtrain1, Muestra_validacion_final,type="response") #predigo con el modelo 1 y la muestra de validación
prediction_2<-predict.glm(Mtrain2, Muestra_validacion_final,type="response") #predigo con el modelo 2 y la muestra de validación
prediction_3<-predict.glm(Mtrain3, Muestra_validacion_final,type="response") #predigo con el modelo 3 y la muestra de validación
prediction_4<-predict.glm(Mtrain4, Muestra_validacion_final,type="response") #predigo con el modelo 4 y la muestra de validación
anaemia_pred1<-prediction_1>0.5
anaemia_pred2<-prediction_2>0.5
anaemia_pred3<-prediction_3>0.5
anaemia_pred4<-prediction_4>0.5
error1<-Muestra_validacion_final$anaemia!=as.numeric(anaemia_pred1) #obtenemos el error de predicción 1 para cada elemento de validación
error2<-Muestra_validacion_final$anaemia!=as.numeric(anaemia_pred2) #obtenemos el error de predicción 2 para cada elemento de validación
error3<-Muestra_validacion_final$anaemia!=as.numeric(anaemia_pred3) #obtenemos el error de predicción 3 para cada elemento de validación
error4<-Muestra_validacion_final$anaemia!=as.numeric(anaemia_pred4) #obtenemos el error de predicción 4 para cada elemento de validación
error1f_FINAL<-mean(error1)
error2f_FINAL<-mean(error2)
error3f_FINAL<-mean(error3)
error4f_FINAL<-mean(error4)Esto tiene mucho sentido que se haga cuando se quiere tener una medida final y fiable del posible error de predicción que se cometería con el modelo en un caso real.
> error1f_FINAL
[1] 0.4745763
> error2f_FINAL
[1] 0.4915254
> error3f_FINAL
[1] 0.559322
> error4f_FINAL
[1] 0.4915254En este caso puede verse que serían, para cada uno de los modelos, ligeramente superiores a los que se prometerían haciendo validación cruzada únicamente.
Clase 3. Los árboles de clasificación
Vamos ahora a presentar una técnica que permite predecir, de la misma manera que hemos hecho hasta ahora, una variable objetivo (por ejemplo, la puntuación de los episodios de los Simpson, el tener una enfermedad o no, etc…) utilizando un conjunto de variables explicativas. Vamos a entender, primeramente, la filosofía.
variable objetivo cualitativa: árbol de clasificación
Imagine que tiene una base de datos donde cuenta con dos variables explicativas (\(X_{1},X_{2})\) y pretende explicar la variable \(Y\) que, en este caso, sólo presenta dos posibles valores \(Y=\{S\acute{I},NO\}.\) Piense, por ejemplo, en que \(Y\) se corresponde con la devolución (o no) de un crédito y \(X_{1},X_{2}\) son variables continuas que miden dos características de cada individuo. Entonces, podemos visualizar la información disponible en este gráfico:
FIG 1: Los datos de partida.
Si observa detenidamente, es fácil encontrar cierta manera, usando rectángulos, de clasificar en bloques los síes y los noes:
FIG 2: Los datos de partida con una “regla de clasificación”.
Ahora bien, si quiere representar la regla que le permita saber, de un vistazo, si conceder o no el crédito, puede hacer un diagrama de árbol. Consiste en poner la misma información que tiene en la FIG2 de una manera más sistemática, de tal forma que le sea fácil de seguir:
FIG 3: El árbol que se deduce de la figura 2: fíjese que, a la izquierda van los caminos que cumplen la regla que aparece en cada rama y, por tanto, a la derecha, los caminos que no la cumplen.
Sin embargo, en la vida real los datos, en general, tendrán una estructura caótica, nada obvia en muchos casos y, por lo tanto, deberemos dejar a los ordenadores que construyan estos árboles. Veamos los pasos que seguirá un algoritmo que genera árboles de clasificación
PASO 1: Elegir un punto de corte
Lo primero que hace su computadora es “rastrear” entre las \(n\) variables explicativas disponibles. Por ejemplo, empieza por \(X_{1}\) , la discretiza (es decir, la convierte en una variable discreta tomando un tamaño predefinido para el corte, por ejemplo \(X_{1}=\{0,0.1,0.2,0.3,....,12\}\) y se va parando en cada uno de esos valores. Entonces, hace unas cuentas que vamos a ir desarrollando. Para que se vea bien la idea, vamos a suponer que, en su trabajo de rastreo, se para en \(X_{1}=3:\)
FIG 4: El algoritmo se detiene en \(X_{1}=3\).
PASO 2: Plantearse cómo de bueno es ese punto de corte
Necesitamos alguna medida que nos permita analizar si el punto de corte debe ser \(X_{1}=3\) o debemos seguir buscando. Una idea, que parece bastante clara, es contabilizar qué tal discrimina, el corte en \(X_{1}=3\), los síes y los noes. La mejor discriminación sería, obviamente, que a la izquierda (es decir, \(X_{1}<3)\) hubiera sólo síes y a la derecha (\(X_{1}>3)\) hubiera sólo noes. Eso no es así (salvo en contadas ocasiones relacionadas con el problema de predicción perfecta del que hablamos en clases pasadas) y, por tanto, debemos asumir que hay cierta heterogeneidad en los grupos. A esa heterogeneidad la llamaremos caos o entropía. Cuanto más caos o entropía haya en un grupo, peor lo estará haciendo el método. Por ejemplo, en el grupo que se forma si \(X_{1}<3\) vemos que, claramente, los síes ganan a los noes. Ese grupo es poco caótico o, dirá, tendrá una entropía baja. Esto quiere decir que, si \(X_{1}<3\), nos podemos apostar el cuello a que la mejor predicción es SÍ ya que, seguramente, no nos equivocaremos. Pero ¿se atrevería a jugarse todo su dinero para predecir qué ocurrirá con el individuo si \(X_{1}>3?\) Seguramente, no. Y eso es, naturalmente, porque el grupo está más desorganizado o caótico. No sabría, entonces, si apostar al SÍ o al NO.
Entran las matemáticas
Es el momento de plantearse cómo cuantificar el grado de caos o, como hemos dicho, entropía presenta cada grupo. Hay varias expresiones pero nosotros vamos a elegir una clásica:
\[ E\left(p_{1},p_{2},...,p_{n}\right)=\sum_{i=1}^{n}-p_{i}\log_{2}(p_{i}) \]
donde \(n\) es el número de posibles valores de la variable dependiente (en nuestro caso, 2) y \(p_{i}\) representa la frecuencia relativa de cada una de las categorías en el grupo (es decir, la probabilidad que tienen de pertenecer al grupo). Por ejemplo, en este caso, en el grupo generado por \(X_{1}<3\), tendremos:
\[ {\color{green}P(S\acute{I})=\frac{10}{12}=\frac{5}{6}},{\color{red}P(NO)=\frac{2}{12}=\frac{1}{6}} \]
de tal forma que la entropía será: \[ E_{1}=-\frac{5}{6}\log_{2}\frac{5}{6}-\frac{1}{6}\log_{2}\frac{1}{6}=0.35 \]
Por otro lado, en el grupo generado por \(X_{1}>3\), tendremos:
\[ {\color{green}P(S\acute{I})=\frac{15}{28}},{\color{red}P(NO)=\frac{13}{28}} \]
de tal forma que la entropía será: \[ E_{2}=-\frac{15}{28}\log_{2}\frac{15}{28}-\frac{13}{28}\log_{2}\frac{13}{28}=0.64 \]
Finalmente, necesitamos una medida global de caos para la participación que ha realizado el algoritmo. Para ello, obtendremos una media ponderada de la entropía \[ E=\frac{12}{40}(0.35)+\frac{28}{40}(0.64)=0.55 \]
PASO 3: decidir sobre el resultado
¿Qué tal es ese 0.55 que hemos obtenido? Pues no lo sabemos. Deberemos compararlo con otro corte. Lo que hará su ordenador es obtener las entropías asociadas a cada corte y quedarse con la que dé un valor menor. Por ejemplo, si corta en \(X_{1}=6,\) como en la figura:
FIG 5: Un nuevo corte en \(X_{1}=6\).
de nuevo, haciendo los números, tendrá, si \(X_{1}<6\)
\[ {\color{green}P(S\acute{I})=\frac{13}{22}},{\color{red}P(NO)=\frac{9}{22}} \]
de tal forma que la entropía será: \[ E_{1}=-\frac{13}{22}\log_{2}\frac{13}{22}-\frac{9}{22}\log_{2}\frac{9}{22}=0.60 \]
Por otro lado, en el grupo generado por \(X_{1}>6\), tendremos:
\[ {\color{green}P(S\acute{I})=\frac{12}{18}=\frac{2}{3}},{\color{red}P(NO)=\frac{6}{18}=\frac{1}{3}} \]
de tal forma que la entropía será: \[ E_{2}=-\frac{2}{3}\log_{2}\frac{2}{3}-\frac{1}{3}\log_{2}\frac{1}{3}=0.55 \]
Finalmente, necesitamos una medida global de caos. Para ello, obtendremos una media ponderada:
\[ E=\frac{22}{40}(0.60)+\frac{18}{40}(0.55)=0.57 \]
que, como ve, es ligeramente peor que el anterior.
Resumiendo:
el ordenador, entonces, lo que hará es tomar nota de las diferentes entropías asociadas a cada una de las particiones en torno a \(X_{1}\). Elegirá, por tanto, el corte que menor entropía produzca. A continuación, hará lo mismo con \(X_{2}\). El árbol entonces, se empezará por la variable cuyo corte genere menor entropía global es decir, entre las entropías para cada corte de \(X_{1}\)y \(X_{2}\) elegirá el corte de la variable que menor entropía tenga. A partir de ese corte, irá eligiendo cortes sucesivos en ambas variables, de manera alternativa.
Con \(N\) variables, sucede lo mismo: del conjunto \(\{X_{1},...,X_{N}\}\) de variables, elegirá aquella que -al hacer una primera partición- devuelva una menor entropía. A partir de ahí, hará lo mismo: realizará cortes en todas las variables e irá quedándose el resultado que menor entropía proporcione en cada iteración. ¿Hasta cuándo? Necesitaremos un criterio de parada. Ese criterio lo veremos en la siguiente clase.
EJERCICIO
Busque una librería de [R] que entrene árboles de clasificación, y trate de- siguiendo la documentación- ser capaz de entrenar un árbol para predecir si un individuo tendrá anemia dado si tiene diabetes y su edad. Trate de interpretar dicho árbol
propuesta de solución Aquí elegimos la librería rpart pero puede elegir cualquier otra de las disponibles.
install.packages("rpart")
library(rpart)
install.packages("rpart.plot")
library(rpart.plot)
ANEMIA<-as.factor(clinicos$anaemia) #Hacemos que la variable output sea un factor
tree.v1 <-rpart(ANEMIA~age+diabetes,data=clinicos, method="class")
summary(tree.v1)
rpart.plot(tree.v1)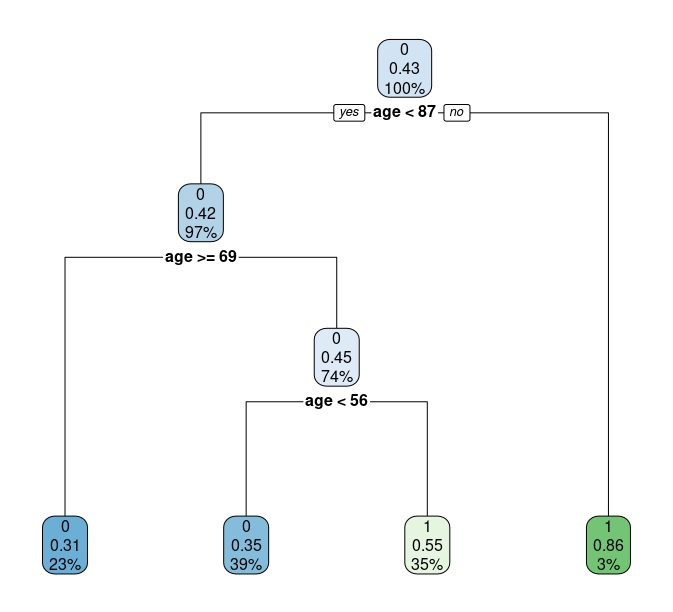
FIG 6: Ejemplo del árbol.
Explicamos lo que significa con detalle:
- El primer nodo tiene el 100% de la muestra (que tiene un 42% de eventos iguales a 1). Si nos quedáramos en ese nodo y no siguiéramos, deberíamos predecir que una persona, elegida al azar de la población de este estudio, tiene un 43% de probabilidades de tener anemia (que es, justo, el porcentaje de individuos con Anemia en la muestra. Nada nuevo. Debemos avanzar)
- Si vamos al siguiente nodo, si una persona tiene más de 87 años directamente predecimos que tiene anemia (además, el 3% de la muestra son individuos mayores de 87 años con anemia). En caso contrario, debemos seguir haciendo el camino:
- como vemos, la probabilidad de tener anemia se concentra entre los individuos entre 56 y 69 años.
Sin embargo, este árbol nos ha servido para entrenarnos parece muy pobre porque sólo utiliza la variable edad para clasificar. En la clase siguiente, verá cómo poder elegir un buen árbol.
Clase 4. Los árboles de clasificación (elección del árbol)
Es el momento de indagar más sobre la construcción de los árboles. En general, diremos que los árboles tienden a sobreajustar la muestra. De hecho, son muy sensibles a la propia muestra, cambiando (bastante) el resultado si se añaden o eliminan observaciones. La razón es sencilla: el procedimiento que hemos contado anteriormente nos puede llevar a hacer árboles que clasifiquen perfectamente la muestra (para ello, sólo se necesita añadir ramas y más ramas). Sin embargo, esto será perjudicial cuando intentemos utilizar el propio árbol para predecir. Es por ello que parece sensato, de nuevo, utilizar validación cruzada para la elección de un árbol óptimo.
Sin embargo, lo normal es que en la propia validación cruzada se decida sobre un conjunto de árboles posibles:
- basados en subconjuntos de variables distintas
- basados en diferentes tamaños del propio árbol
Nos enfocamos en la idea segunda. Tenemos que pensar que cada rama que se añade, esta supone un “coste” puesto que hace el árbol más complejo.
Lo normal es que se utilice una medida de coste:
\[ CP(T)=R(T)+\alpha T \] con \(\alpha>0\). \(R(T)\) es una medida de error de clasificación para el árbol con T ramas. Por otro lado, se penaliza el número de ramas (T) mediante un parámetro \(\alpha\). La librería rpart no permite que el usuario proponga un valor, sino que lo estima internamente.
AL acto de elegir el número de ramas que irán en el árbol se le denomina podado o pruning.
Por ejemplo, aquí puede ver el resultado para el árbol de la FIG6
> summary(tree.v1)
Call:
rpart(formula = ANEMIA ~ age + diabetes, data = clinicos, method = "class",
control = rpart.control(cp = 0.03))
n= 240
CP nsplit rel error xerror xstd
1 0.04854369 0 1.0000000 1.000000 0.07444509
2 0.04368932 1 0.9514563 1.077670 0.07499175
3 0.03000000 3 0.8640777 1.058252 0.07488693Si se fija en la función que ha generado este árbol, en “control” se puso que CP debía llegar a 0.03, como mucho. Al llegar ahí se para. Se ha fijado 0.03 para que el árbol no fuera muy “grande”. Verá que si no pone nada, el árbol sale más grande (R tiene la orden interna de usar 0.01) como parámetro interno.
De esta forma, usted puede jugar con dichos parámetros de control en la validación cruzada. A continuación le mostramos una posible idea:
- Como puntos importantes, creamos un vector con diferentes parámetros de control (es decir, valores umbral para podar el árbol)
- Para ello, tenemos que tener dos bucles: uno para almacenar errores y otro para probar distintos parámetros de control
- Por otro lado, elegimos una medida de error (en este caso, ele elemento 1,2 de la matriz de confusión-como se ve en la última línea de código- es decir, predecir que el individuo tiene anemia y que, en realidad, no la tenga )
clinicos <- read_delim("clinicos_final.csv",
delim = ";", escape_double = FALSE, trim_ws = TRUE)
sample <- sample.int(n = nrow(clinicos),size = floor(.20*nrow(clinicos)), replace = F) #me da el sorteo de filas aleatorias
Muestra_validacion_final<-clinicos[sample,]
clinicos<-clinicos[-sample,] #mi muestra sin los datos que me he guardado para jugar a se Kaggle
error<-mat.or.vec(50, 4) #creamos un vector que almacenará 50 errores
cpv<-c(0.001,0.01,0.05,0.1) #creamos un vector que contiene los diferentes valores del parámetro de control
for(i in 1:50){ #Iniciamos los subíndices: este es el de la iteración
for (j in 1:4){ #este es el subíndice del parámetro de control
random <- sample(1:nrow(clinicos), size = floor(.9*nrow(clinicos))) # Elegimos la muestra de entrenamiento al azar
train<-clinicos[random,]
test <-clinicos[-random, ] #por lo que la muestra de test es la contraria
tree.v1 <-rpart(anaemia~age+diabetes,data=train,control = rpart.control(cp = cpv[j],minsplit=10), method="class") #permitimos que el parámetro de control cp, varíe y minsplit es el número mínimo de observaciones en un nodo
tree.pred <- predict(tree.v1, newdata = test, type = "class") #predecimos con la muestra «test»
xt1<-table(tree.pred, test$anaemia) #obtenemos la tabla de confusión...
error[i,j]<-xt1[1,2]/length(tree.pred) #...y de la tabla seleccionamos la(s) magnitud(es) que nos interesen
}}Atendiendo al gráfico, se puede intuir que el modelo 2 (asociado a un cp=0.01) parece ser el que mejor se comporta en este error especificado
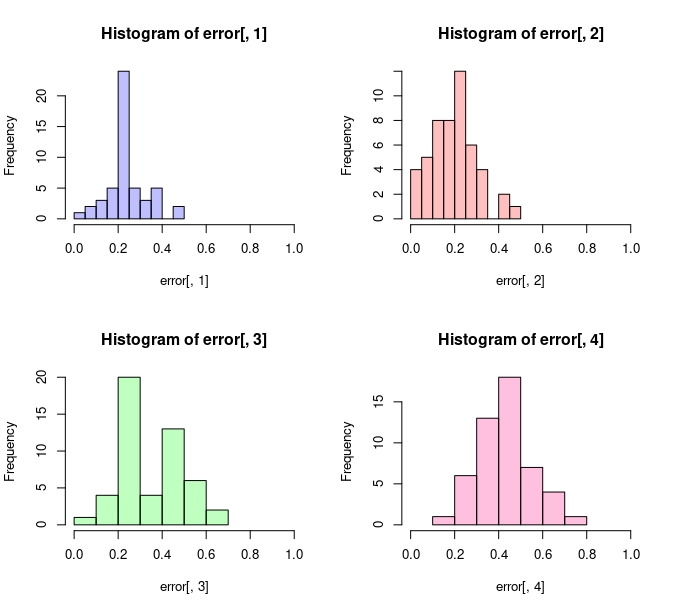
FIG 1: Errores de predicción
Puede generar estos errores usando este script
p1 <- hist(error[,1])
p2 <- hist(error[,2])
p3 <- hist(error[,3])
p4 <- hist(error[,4])
par(mfrow=c(2,2))
plot( p1, col=rgb(0,0,1,1/4), xlim=c(0,1))
plot( p2, col=rgb(1,0,0,1/4), xlim=c(0,1))
plot( p3, col=rgb(0,1,0,1/4), xlim=c(0,1))
plot( p4, col=rgb(1,0,1/2,1/4), xlim=c(0,1)) Y, además, puede obtener los descriptivos asociados a cada error
> summary(error)
V1 V2 V3 V4
Min. :0.04167 Min. :0.04167 Min. :0.08333 Min. :0.1667
1st Qu.:0.20833 1st Qu.:0.12500 1st Qu.:0.20833 1st Qu.:0.3750
Median :0.22917 Median :0.18750 Median :0.31250 Median :0.4167
Mean :0.24167 Mean :0.20333 Mean :0.34833 Mean :0.4400
3rd Qu.:0.29167 3rd Qu.:0.28125 3rd Qu.:0.50000 3rd Qu.:0.5000
Max. :0.45833 Max. :0.50000 Max. :0.62500 Max. :0.7500 Aunque si quiere prometer un error más fiable, deberá haberse guardado otro porcentaje de muestra (como hemos hecho al principio) y probar el segundo modelo sobre ese porcentaje
tree.vf <-rpart(anaemia~age+diabetes,data=clinicos,control = rpart.control(cp = 0.01,minsplit=10), method="class") #permitimos que el parámetro de control cp, varíe y minsplit es el número mínimo de observaciones en un nodo
tree.pred <- predict(tree.vf, newdata = Muestra_validacion_final, type = "class") #predecimos con la muestra «test»
xt1<-table(tree.pred, Muestra_validacion_final$anaemia) #obtenemos la tabla de confusión...
error<-xt1[1,2]/length(tree.pred) #...y de la tabla seleccionamos la(s) magnitud(es) que nos intereseny, pese a que el error mediano no llega al 18% en el modelo 2, en esta muestra nos sale del 25%.
Clase 5. Algunos comentarios sobre la práctica de segmentación de clientes
- No se olvide de guardar un porcentaje de la muestra para hacer una validación final. La muestra, además, es muy grande:
sample <- sample.int(n = nrow(cross_selling),size = floor(.20*nrow(cross_selling)), replace = F) #me da el sorteo de filas aleatorias
Muestra_validacion_final<-cross_selling[sample,]
CS<-cross_selling[-sample,] #mi muestra sin los datos que me he guardado para jugar a se Kagglepuede hacer sus análisis descriptivos sobre la muestra que aquí hemos llamado CS (cross selling). Esta muestra servirá para hacer un estudio previo de los datos y una validación cruzada que le permitirá seleccionar modelos. El análisis descriptivo ni ningún otro modelo se podrán realizar con la Muestra_validación_final esta deberá permanecer intocable.
- Haga diferentes análisis descriptivos de interés. Si quiere analizar una variable discreta (como, en este caso, es Response) frente a una variable continua (como la edad) podrá utilizar box plot.
boxplot(Age ~ Response, data = CS) # box plot con un sólo factor
boxplot(Age ~ Response + Gender, data=CS) # box plot con dos factores (interactuando)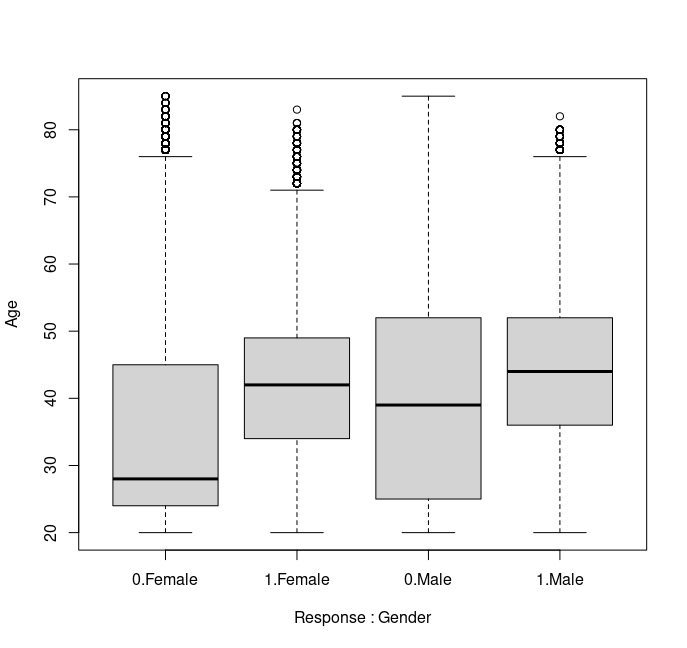
FIG 1: Box plot con interacción
En este, por ejemplo, puede ver cómo hay una gran diferencia entre la edad mediana de la mujer que compra el producto (en torno a 40 años) frente a la que no lo compra (menos de 30 años). En el caso de los hombres, la diferencia es mucho menor. Un primer factor relevante es la edad y el género. Recuerde que los modelos predictivos posteriores vienen a cuantificar lo que usted ya es capaz de detectar analizando los datos de manera descriptiva. No se hacen modelos a “ciegas” sin entender lo que cabe esperar dada la muestra.
- Si quiere analizar variables cualitativas, puede utilizar tablas de contingencia, utilizando-de manera adecuada- los porcentajes por fila o columna
prop.table(table(CS$Vehicle_Age,CS$Response),margin=2 )
0 1
< 1 Year 0.4709 0.1541
> 2 Years 0.0341 0.1009
1-2 Year 0.4950 0.7450En esta tabla, puede ver cómo el 75% de los que deciden comprar tienen un coche entre 1 y 2 años. Por otro lado, entre los que no compran parece dividirse al 50% (aproximadamente) entre los que su coche tiene entre 1 y 2 años y el resto. De aquí puede tomar diferentes opciones: por ejemplo, reducir las clases de la variable “vehicle age” a “1-2 años” y resto.
- Por otro lado, vigile siempre las unidades de medida de las variables (introduzca logaritmos si ve que tiene problemas en las escalas). En este caso, haga un boxplot de la variable “annual premium” frente a la variable “Response” sin logaritmos y utilizando logaritmos ¿Qué observa?
Ahora, un punto importante Si entrena primeros árboles de clasificación, encontrará como resultado:
random <- sample(1:nrow(CS), size = floor(.9*nrow(CS))) # Elegimos la muestra de entrenamiento al azar
train<-CS[random,]
test<-CS[-random]
tree.v1 <-rpart(Response~Age,data=train, method="class") #permitimos que el parámetro de control cp, varíe y minsplit es el número mínimo de observaciones en un nodo
rpart.plot(tree.v1)
FIG 2: Primer árbol de clasificación
Sin duda, no parece muy prometedor. Este árbol dice, simplemente que el 12% le va a comprar el producto. Es decir, el promedio muestral. ¿Por qué?
- Al haber un gran desequilibrio entre 1 y 0 en la variable “Response”, que es la que quiere predecir, el algoritmo de entrenamiento no tendrá ningún problema en predecir la clase mayoritaria (en este caso los ceros). El mecanismo del árbol, entonces, no será capaz de deterctar los 1 (por su escasez) y, por tanto, el algoritmo de clasificación se parará en etapas iniciales (ya que la clasificación no mejorará). Por ello, es común hacer un muestreo de los datos. Esto consiste en-aleatoriamente- eliminar observaciones de la clase predominante (para cada muestra de entrenamiento) y hacer que queden equilibradas. Esto se puede hacer con la librería ROSE
install.packages("ROSE")
library(ROSE)
error1<-mat.or.vec(50, 5) #creamos un vector que almacenará 50 errores
error2<-mat.or.vec(50, 5) #creamos un vector que almacenará 50 errores
error3<-mat.or.vec(50, 5) #creamos un vector que almacenará 50 errores
error4<-mat.or.vec(50, 5) #creamos un vector que almacenará 50 errores
cpv<-c(0.0001,0.001,0.01,0.1) #creamos un vector que contiene los diferentes valores del parámetro de control del árbol
for(i in 1:50){ #Iniciamos los subíndices: este es el de la iteración
for (j in 1:5){ #este es el subíndice del parámetro de control
random <- sample(1:nrow(CS), size = floor(.9*nrow(CS))) # Elegimos la muestra de entrenamiento al azar
train<-CS[random,]
test <-CS[-random, ] #por lo que la muestra de test es la contraria
train_balanced <- ovun.sample(Response ~ ., data = train, method = "both", p=0.5,N=33800, seed=1)$data # Aquí creamos una muestra nueva. Le pedimos que la muestra quede igualada en cantidad de 0 y 1 para la variable "Response"
tree.v1 <-rpart(Response~Age,data=train_balanced,control = rpart.control(cp = cpv[j]), method="class") #permitimos que el parámetro de control cp, varíe. Usamos la nueva muestra balanceada
tree.v2 <-rpart(Response~Gender+Age,data=train_balanced,control = rpart.control(cp = cpv[j]), method="class")
tree.v3 <-rpart(Response~Age+Gender+as.factor(Vehicle_Age),data=train_balanced,control = rpart.control(cp = cpv[j]), method="class")
tree.v4 <-rpart(Response~Age+Gender+vehicle_age_new,data=train_balanced,control = rpart.control(cp = cpv[j]), method="class")
tree.pred.v1 <- predict(tree.v1, newdata = test, type = "class") #predecimos con la muestra «test»
tree.pred.v2 <- predict(tree.v2, newdata = test, type = "class") #predecimos con la muestra «test»
tree.pred.v3 <- predict(tree.v3, newdata = test, type = "class") #predecimos con la muestra «test»
tree.pred.v4 <- predict(tree.v4, newdata = test, type = "class") #predecimos con la muestra «test»
xt1<-table(tree.pred.v1, test$Response) #obtenemos la tabla de confusión...
error1[i,j]<-(xt1[1,2]+xt1[2,1])/length(tree.pred.v1) #...y de la tabla seleccionamos la(s) magnitud(es) que nos interesen
xt2<-table(tree.pred.v2, test$Response) #obtenemos la tabla de confusión...
error2[i,j]<-(xt2[1,2]+xt2[2,1])/length(tree.pred.v2) #...y de la tabla seleccionamos la(s) magnitud(es) que nos interesen
xt3<-table(tree.pred.v3, test$Response) #obtenemos la tabla de confusión...
error3[i,j]<-(xt3[1,2]+xt3[2,1])/length(tree.pred.v3) #...y de la tabla seleccionamos la(s) magnitud(es) que nos interesen
xt4<-table(tree.pred.v4, test$Response) #obtenemos la tabla de confusión...
error4[i,j]<-(xt4[1,2]+xt4[2,1])/length(tree.pred.v4) #...y de la tabla seleccionamos la(s) magnitud(es) que nos interesen
}}
- Como verá, la muestra test no está “remuestreada” o “balanceada”: de eta forma podrá valorar de una manera más fidedigna el error de predicción. Si remuestrea también el test podría tener una idea errónea de la verdadera capacidad predictiva de los modelos.
Los árboles de regresión
Por otra parte, si la variable que quiere predecir es continua (o discreta, pero no cualitativa) podrá ajustar un árbol de regresión. La idea de su mecanismo es similar a lo que acaba de ver. Supongamos que tenemos dos variables candidatas (por ejemplo, \(x_1\),\(x_2\)) para explicar una tercera (\(y\)). Podemos visualizar los datos disponibles en la FIG 3
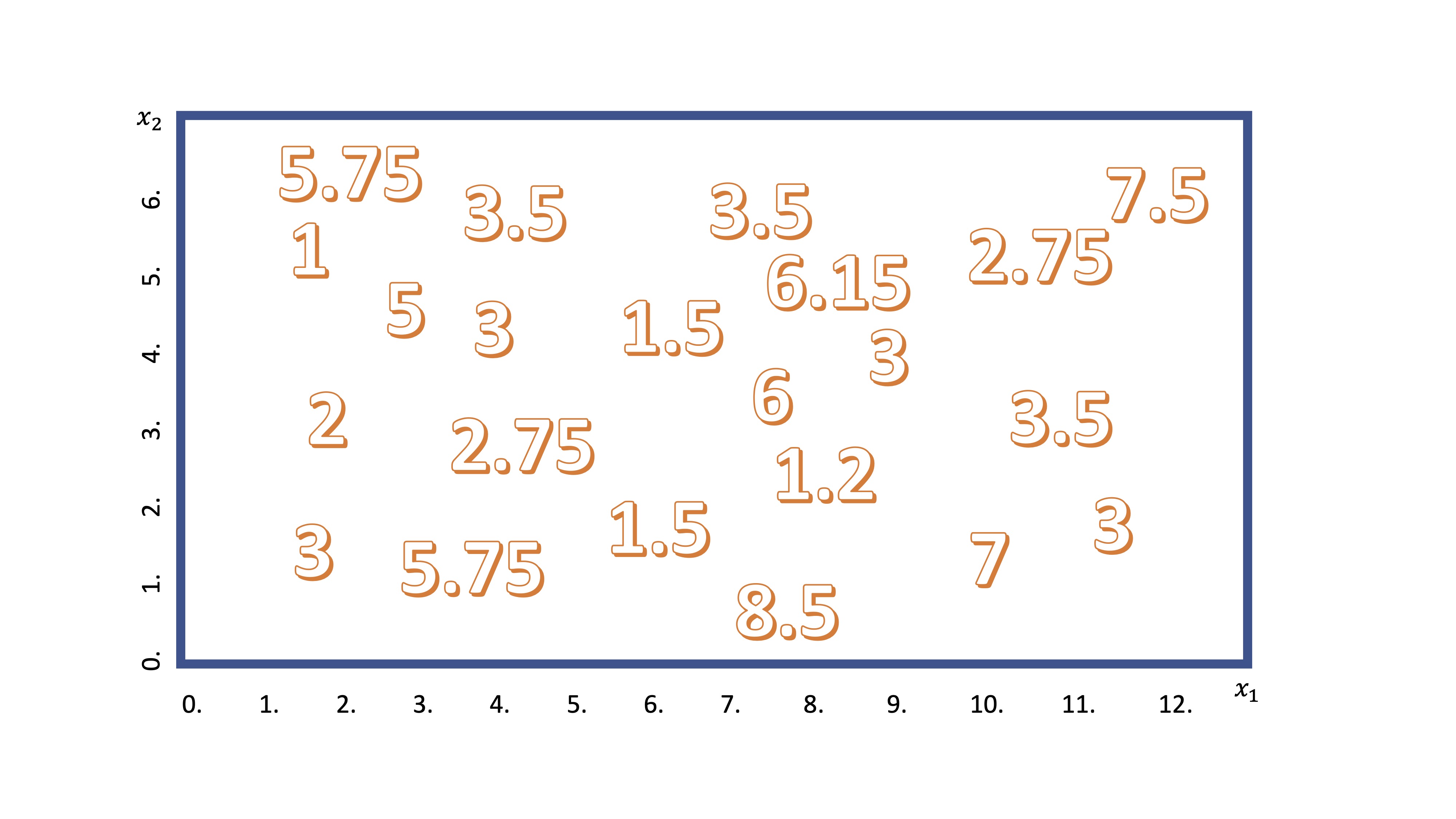
FIG 3: Idea del árbol de regresión
Ahora, como ya sabe, el algoritmo empezará a realizar un corte en una de las variables. De manera secuencial, discretizará la variable \(x_1\), en porciones pequeñas (por ejemplo, \(x_1=[0,0.5,1,1.5,2,....,12])\) y realizará cortes para ver cuál es mejor. Imagine que se para en el corte \(x_1=5\), entonces, tendrá algo así:
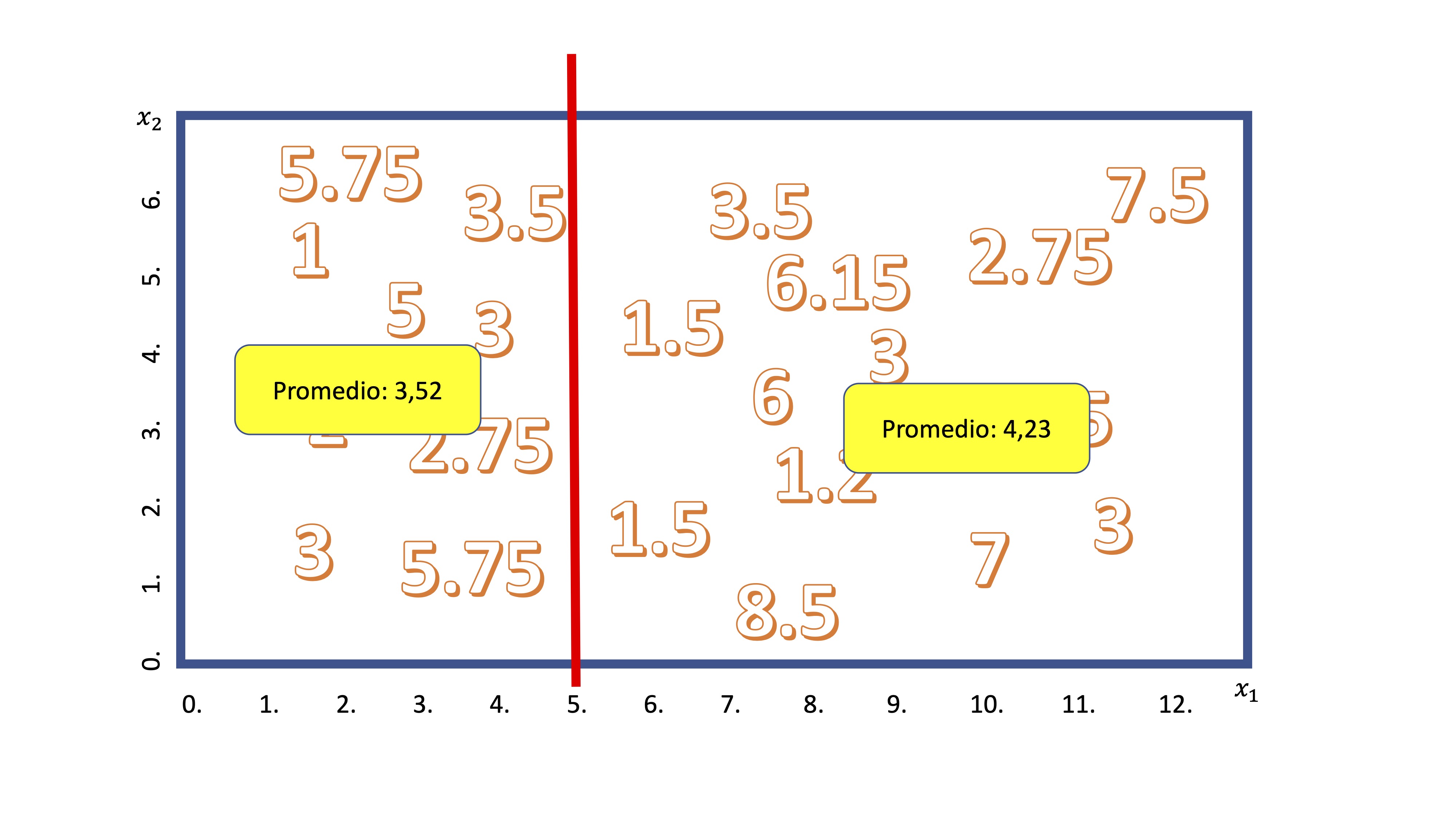 Como ahora el promedio de la variable \(y\) tiene sentido, el algoritmo calcula - para cada grupo- la media condicionada al corte. En este caso, diremos que si \(x_1<5\), entonces el valor esperado para \(y\) es 3.52 y, en caso contrario es 4.23.
Ahora debemos preguntarnos si el corte es “bueno”. Para ello, el algoritmo calcula el Error cuadrático Medio. Para el grupo \(x_1<5\) será \(ECM_1=\frac{(5.75-3,52)^2+(3.5-3.52)^2+....+(5.75-3,52)^2}{9}\) y lo mismo para el otro grupo \(ECM_2=\frac{(3.5-4,23)^2+(6.15-4.23)^2+....+(3-4,23)^2}{13}\). La suma de ambos será el error cuadrático de la partición. Buscará aquella partición que minimice esta suma. Luego, alternatá entre todas las variables explicativas y elegirá empezar a cortar el árbol por aquella que minimice dicha suma. De la misma manera, irá alternando entre las variables de acuerdo con algún criterio de parada, de nuevo, relacionado con el parámetro de control (que regula cuántas ramas tendrá el árbol).
Como ahora el promedio de la variable \(y\) tiene sentido, el algoritmo calcula - para cada grupo- la media condicionada al corte. En este caso, diremos que si \(x_1<5\), entonces el valor esperado para \(y\) es 3.52 y, en caso contrario es 4.23.
Ahora debemos preguntarnos si el corte es “bueno”. Para ello, el algoritmo calcula el Error cuadrático Medio. Para el grupo \(x_1<5\) será \(ECM_1=\frac{(5.75-3,52)^2+(3.5-3.52)^2+....+(5.75-3,52)^2}{9}\) y lo mismo para el otro grupo \(ECM_2=\frac{(3.5-4,23)^2+(6.15-4.23)^2+....+(3-4,23)^2}{13}\). La suma de ambos será el error cuadrático de la partición. Buscará aquella partición que minimice esta suma. Luego, alternatá entre todas las variables explicativas y elegirá empezar a cortar el árbol por aquella que minimice dicha suma. De la misma manera, irá alternando entre las variables de acuerdo con algún criterio de parada, de nuevo, relacionado con el parámetro de control (que regula cuántas ramas tendrá el árbol).
El siguiente esquema le permite ajustar un árbol de regresión. Tendrá que poner en “method” la palabra anova que significa “análisis de varianza”. Esto infica a la librería rpart que ha de entrenar un árbol con variable explicada continua.
rating<-SIMPSONS_FINAL$rating_imdb
BART<-SIMPSONS_FINAL$Bart
HOMER<-SIMPSONS_FINAL$Homer
arbol_simpsons<- rpart(formula = rating ~BART+HOMER, method = "anova", control=list(cp = 0.01, xval = 10))
rpart.plot(arbol_simpsons)Obtendrá algo así,
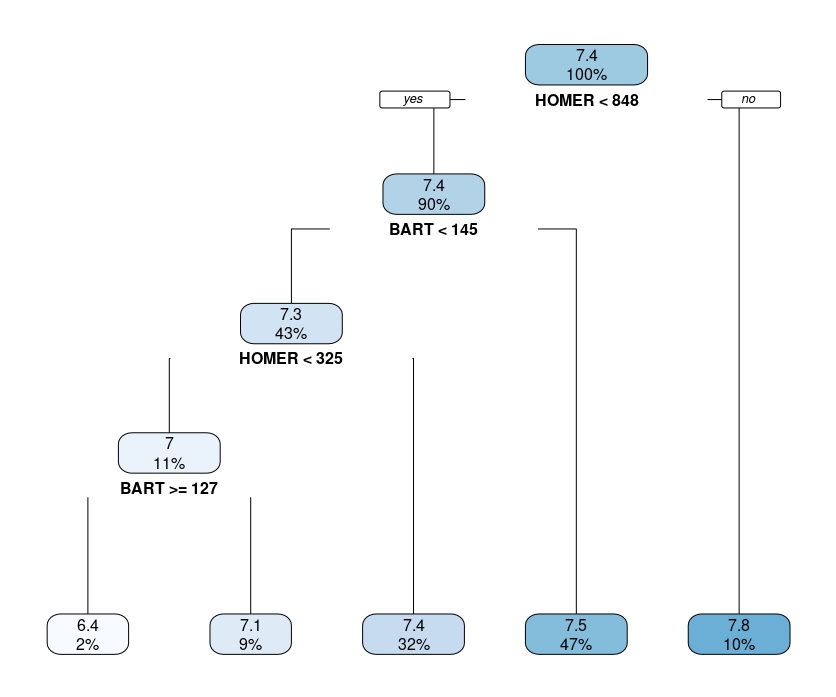
FIG 5: Un árbol de regresión
que, recuerde, deberá interpretar como el promedio condicional. Por ejemplo, el último nodo de la derecha quiere decir que cuando Homer tiene más de 848 líneas de guión los episodios tienen, de media un 7.8. Y eso pasa en el 10% de la muestra.
Clase 6. “K means”
Muchas veces le puede interesar reducir la dimensión asociada al número de variables con el que cuenta. Es posible, por ejemplo, que le interese agrupar la información con la que cuenta para crear una nueva variable sintética. Imagine, por ejemplo, que tiene 10 variables que aluden a la clase social y la formación del individuo: quizás quiera tener una única variable que clasifique a los individuos según la información conjunta contenida en ese bloque de variables. Sin embargo, no va a poder entrenar un modelo y validarlo: usted usa la técnica sin saber, a priori, lo que va a obtener. Por eso, a estas técnicas se las conoce como no supervisadas. No puede realizar comprobaciones cuantitativas mediante test y validación. Sólo podrá usar su lógica para ver si lo que obtiene es sensato o no. Generalmente, estas técnicas son pasos intermedios para tener una base de datos más sintética que le ayude en el proceso de modelar su pregunta de interés.
Este análisis busca realizar agrupaciones de observaciones condicionadas por el valor que tomen ciertas variables disponiles. Imagine que tiene una nube de puntos asociada a dos variables: \(x_1,x_2\). Visualmente, seguro que es capaz de hacer dos grupos ¿no?
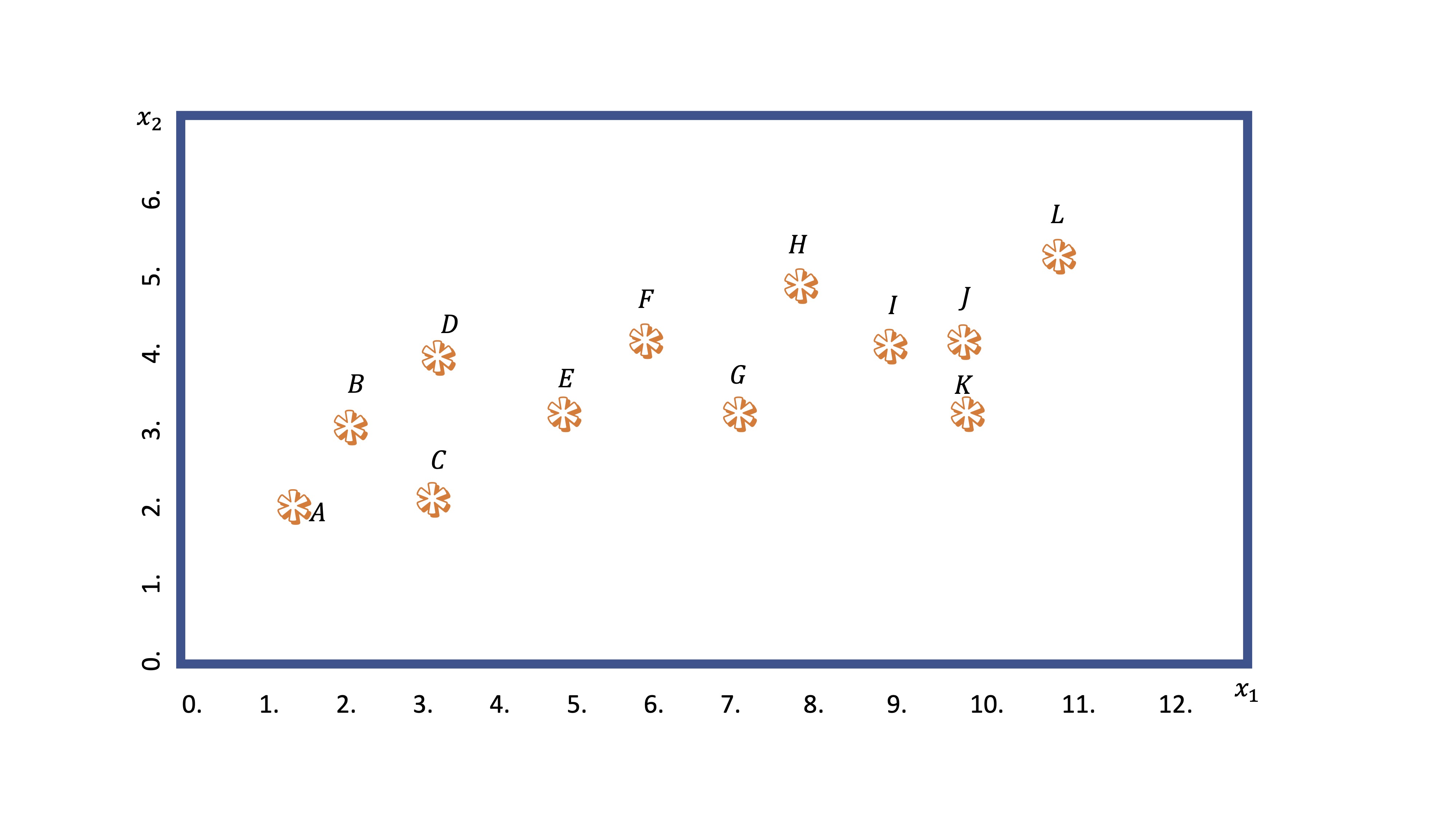
FIG 1: Un ejemplo de variable que queremos agrupar
¿Cómo lo hará la máquina? Veamos el algoritmo en los diferentes pasos:
- Primero: busca dos puntos (generalmente de manera aleatoria). Nosotros elegiremos los más distantes entre sí. Esos puntos se llamarán centroides de los cluster.
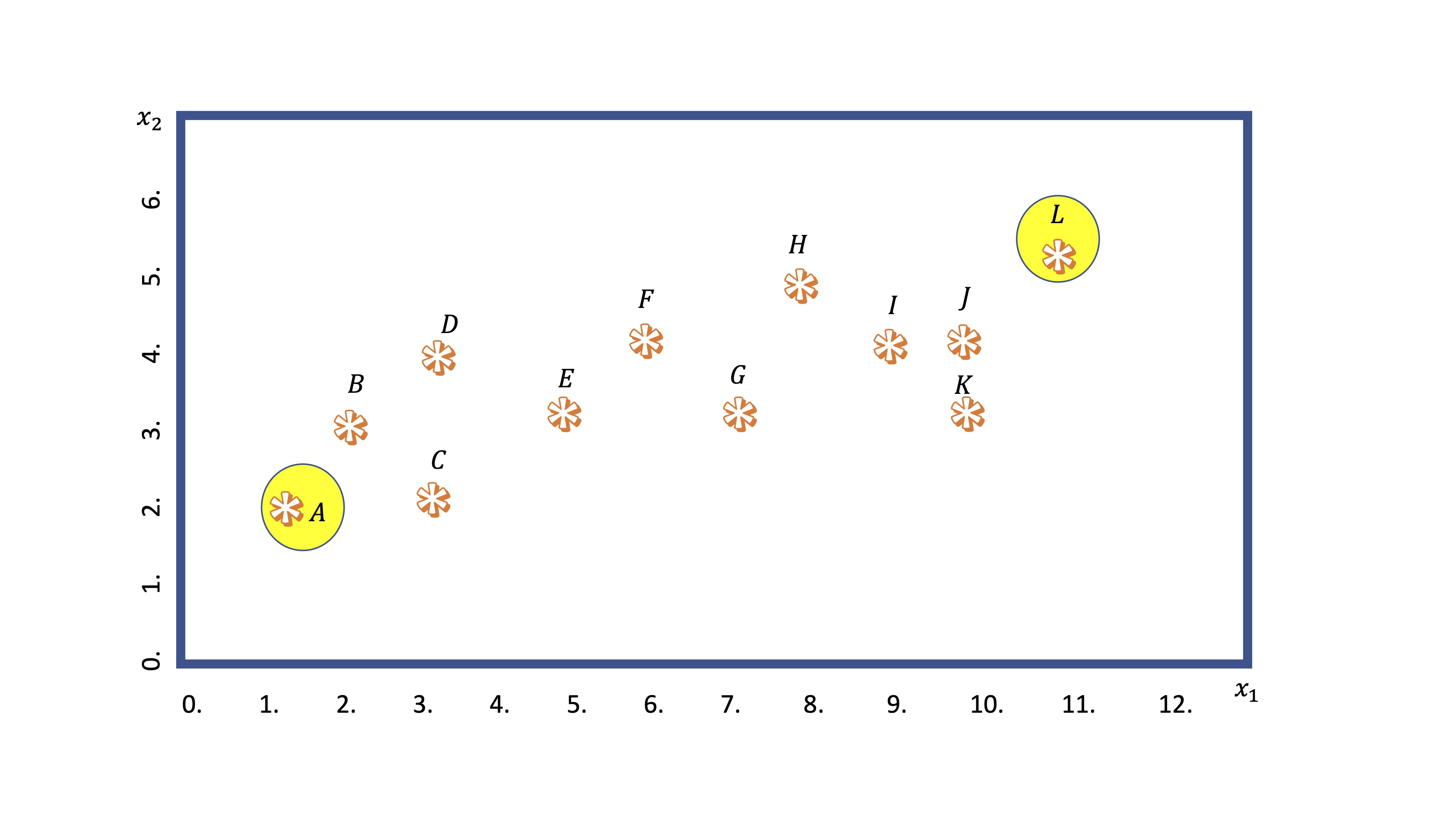
FIG 2: centroides iniciales
El centroide 1, el punto A, tiene coordenadas \(A(1,2)\), mientras que el centroide 2, el punto L, tiene coordenadas \(L(11,5.5)\). Esas son ahora las referencias de los grupos.
- Segundo: Calcula la distancia (mediante alguna medida general, como la norma euclídea) de cada punto a los centroides
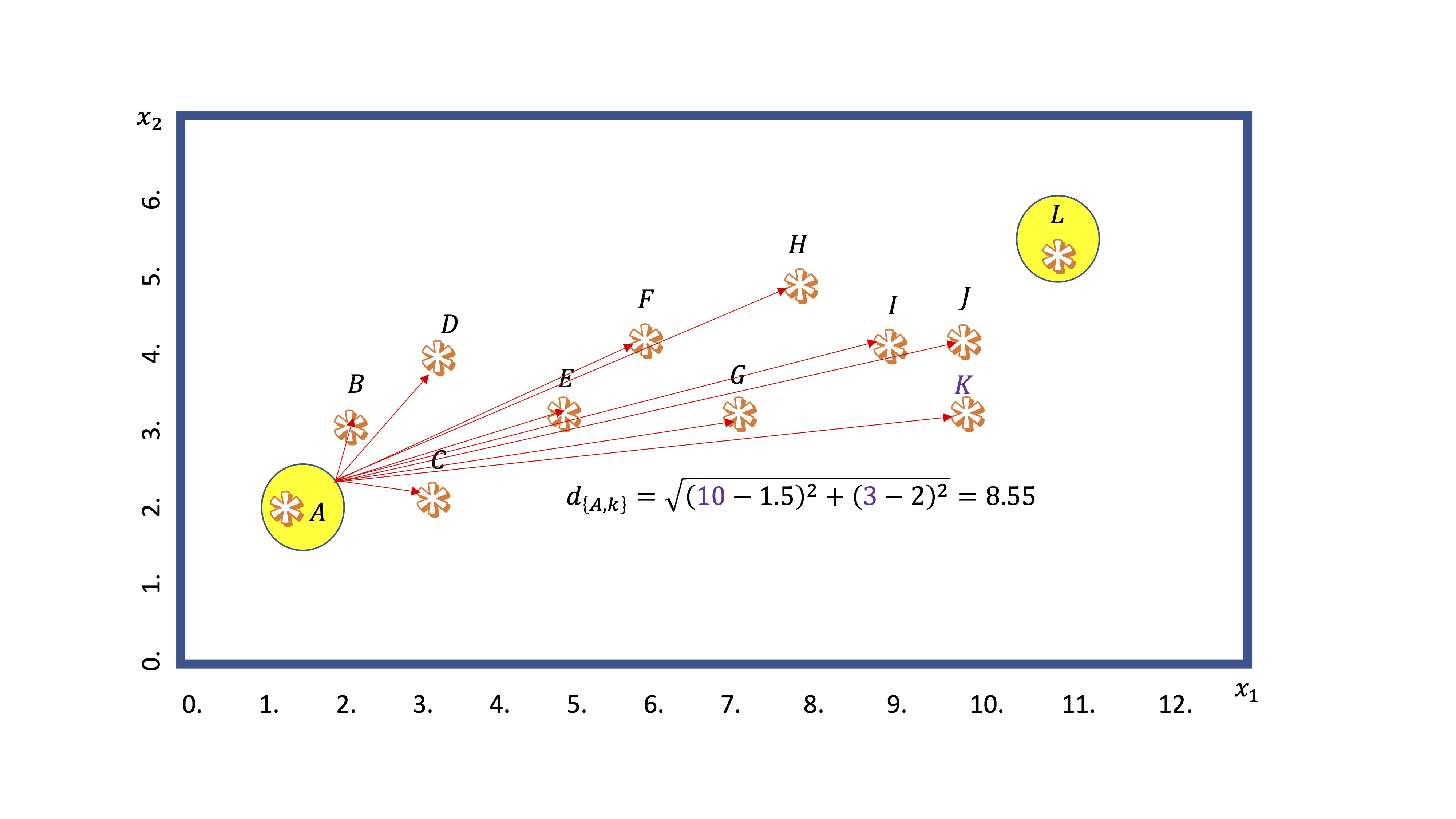
FIG 3: Un ejemplo del cálculo de la distancia
En este ejemplo, calculamos la distancia del centroide A al punto K. Obtenemos \(8.55\). Por otro lado, del centroide L al mismo punto:
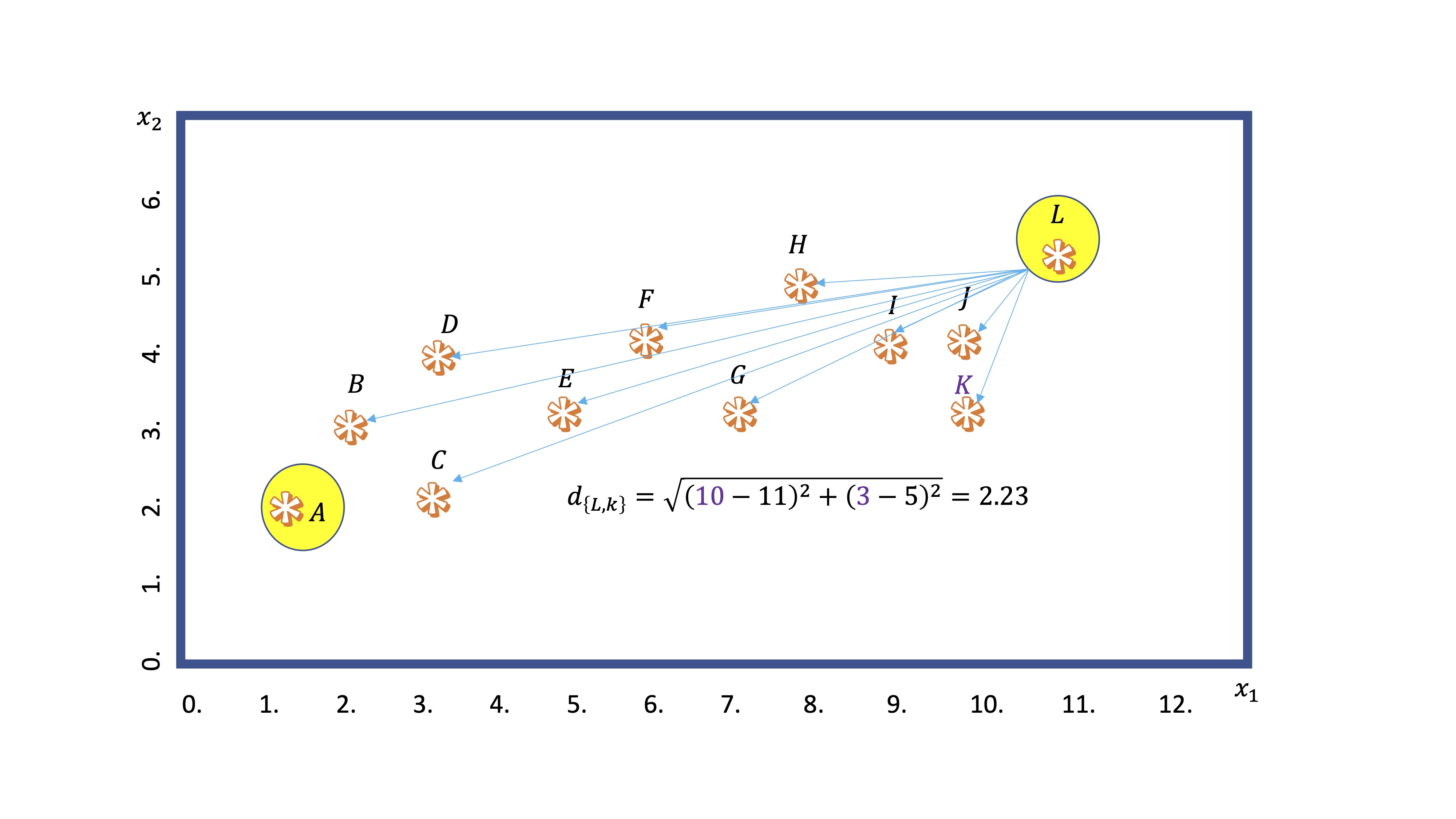
FIG 4: Un ejemplo del cálculo de la distancia
y vemos que es menor. Entonces, el algoritmo dejará al punto K dentro del nodo que “preside” L.
- Tercero: establece una agrupación por clusters en función de la distancia de cada punto a los centroides elegidos
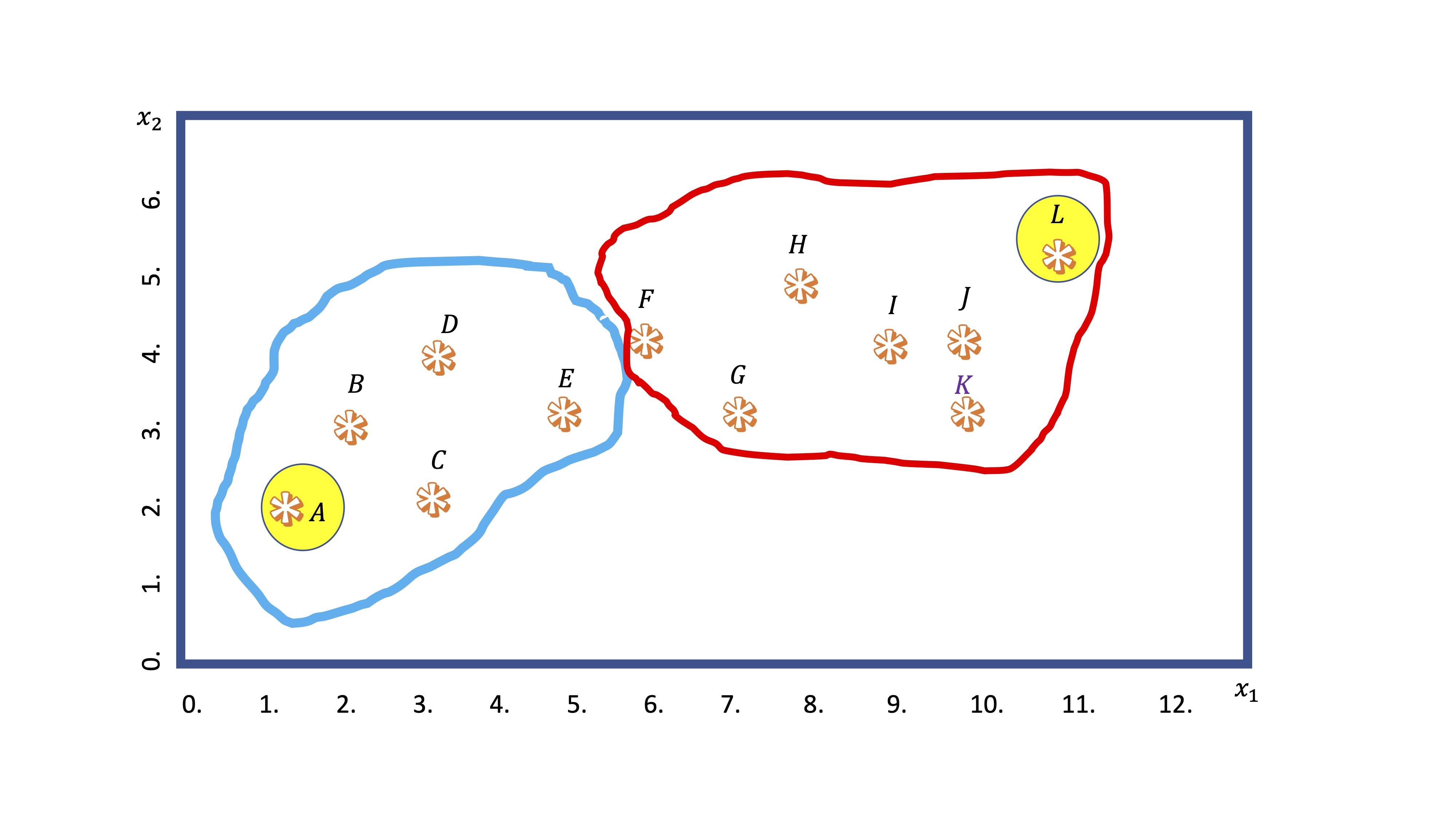
FIG 5: Los primeros clusters!
Sin embargo, con los nuevos grupos hechos, ahora recalcula los centroides (haciendo la media de las coordenadas \(x_1,x_2\) de cada punto dentro del grupo). De hecho, el nuevo centroide puede ya no ser ningún punto de los que se encuentran en el grupo.
- Cuarto: entonces, recalcula la distancia de todos los puntos a los centroides y, en base a ese dato, los grupos van a reorganizarse (observaciones en las fronteras de cada grupo irán cambiándose)
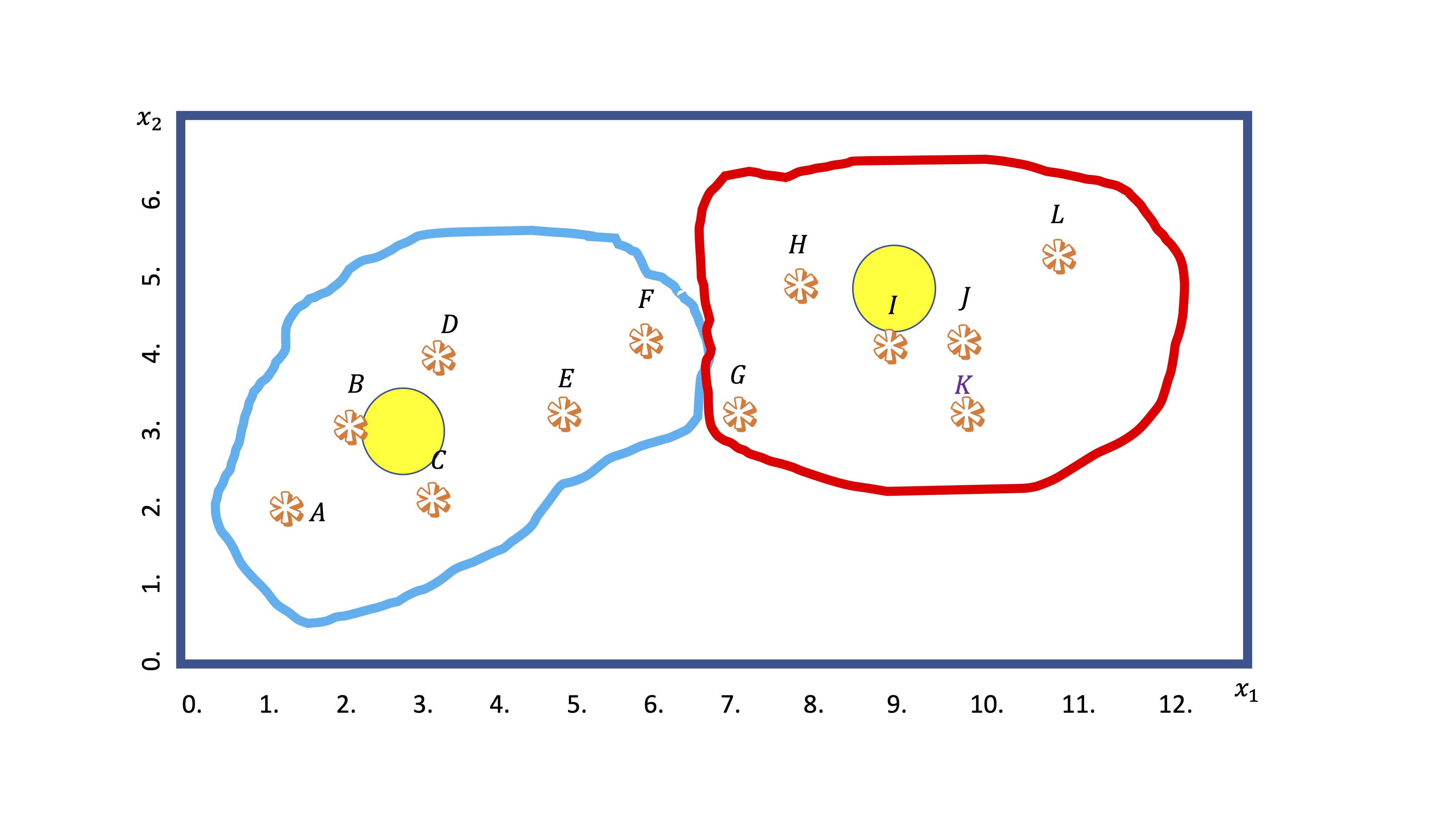
FIG 6: La observación F se cambia de chaqueta
El algoritmo iterará hasta que la clasificación se estabilice. ¿Qué criterio seguiremos para decidir cuántos grupos/cluster hacer?
Elección de número de grupos “k”. Habitualmente, el algoritmo calcula la “suma de cuadrados” de cada uno de los grupos utilizando los centroides como valores de referencia. De esta manera, realiza la suma de cuadrados de las distancias de cada coordenada a su valor central en cada grupo. El objetivo es minimizar la suma total. Esa suma siempre será decreciente, por lo que se elegirá el número de grupos \(k\), donde un incremento adicional de \(k\) haga que el error caiga marginalmente. Lo veremos con un ejemplo:
Introducimos los datos que han generado las figuras anteriores
x1<-c(1.5,2,3,3,5,6,7,8,9,10,10,11)
x2<-c(2,3,2,4,3,4,3,5,4,4,3,6)
dataset<-data.frame(x1,x2)Ahora creamos un bucle que va a ir incrementando la cantidad de clusters (\(k\)) y calculando las sumas de cuadrados (whitinss, en la función empleada)
S <- vector() #inicializo mi vector de dispersiones
for(i in 1:8){
kresult<-kmeans(dataset, i, nstart=100)$withinss
S[i]<-sum(kresult)
} #calculo mediante kmeans la S para cada knote que nstart=100 quiere decir que deberá empezar 100 veces, de manera aleatoria, los centroides. Es una manera de estabilizar el algoritmo y hacer que el resultado no dependa de la selección original de centroides.
ggplot() +
geom_point(aes(x = 1:8, y = S), color = "blue") +
geom_line(aes(x = 1:8, y = S), color = "blue") +
ggtitle("Selección de clusters") +
xlab("Cantidad de Centroides k") +
ylab("S")El gráfico de la FIG7 nos sugiere, de hecho, hacer entre 4 y 5 clusters.
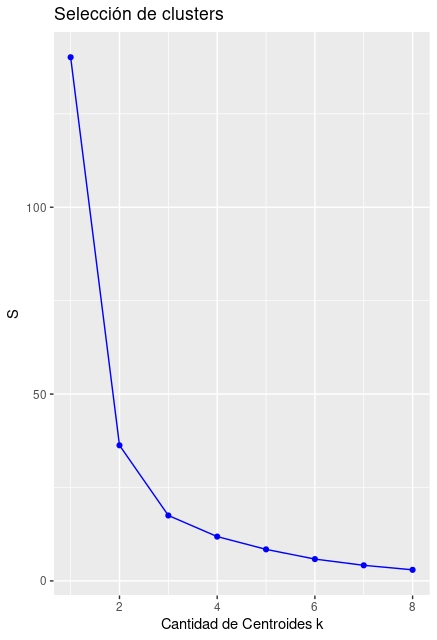
FIG 7: Gráfico para la selección del número de clusters
Aunque, a todas luces, parece demasiado exagerado hacer 4 clusters. Será usted, como usuario, quien deba elegir lo más conveniente (debería, por ejemplo, utilizar algún criterio extramuestral). Con este script puede dibujar los resultados:
install.packages("factoextra")
library(factoextra)
kmeans <- kmeans(dataset, 2, iter.max = 1000, nstart = 25)
fviz_cluster(kmeans, data = dataset)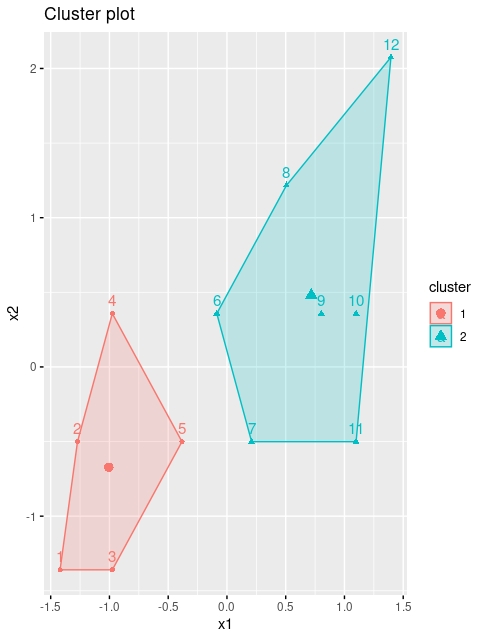
FIG 8: Con \(k=2\)
 Ahora bien, lo normal es no tener acceso a estos gráficos porque usted quiera hacer los cluster con más de dos variables (no olvide que el objetivo es reducir el tamaño del conjunto de variables disponible). Entonces, le interesará acceder a la información que proporciona la función kmeans
Ahora bien, lo normal es no tener acceso a estos gráficos porque usted quiera hacer los cluster con más de dos variables (no olvide que el objetivo es reducir el tamaño del conjunto de variables disponible). Entonces, le interesará acceder a la información que proporciona la función kmeans
K-means clustering with 2 clusters of sizes 5, 7
#por un lado, le proporciona las coordenadas de los centroides. Eso le ayudará a entender el significado de estos, ya que son los promedios de cada variable en el cluster.
Cluster means:
x1 x2
1 2.90 2.80
2 8.71 4.14
#por otro lado, le proporciona la "etiqueta" del cluster al que pertenece cada observación. Esta será una nueva variable para usted que resume las anteriores. Tendrá que darle un significado, basado en los valores de los centroides
Clustering vector:
[1] 1 1 1 1 1 2 2 2 2 2 2 2
#Y estas son las sumas de cuadrados de cada cluster
Within cluster sum of squares by cluster:
[1] 10.0 26.3
(between_SS / total_SS = 74.1 %)Ejercicio Realice un ejercicio de cluster con los datos de audiencia y puntuación de los Simpson de tal forma que trate de ver si encuentra mayor capacidad predictiva de los personajes en el resultado de esa nueva variable caracterizada por los clusters que encuentre. Trate de explicar el significado de los cluster.
Clase 7. “K means (algunos comentarios)”
Si ha tratado de hacer el ejercicio de la clase anterior, habrá visto algo curioso respecto a los clusters. Vamos a ello:
- Ha seleccionado las variables audiencia y rating de los episodios, y ha ejecutado la sentencia que le permite ver cuántos clusters son convenientes:
#Cargo los datos
SIMPSONS<-na.omit(SIMPSONS)
audiencia<-SIMPSONS$millions_audiene
rating<-SIMPSONS$rating_imdb
#creo un data frame y elimino los NAs
dataset<-data.frame(audiencia,rating)
dataset<-na.omit(dataset)
S <- vector() #inicializo mi vector de dispersiones
for(i in 1:8){
kresult<-kmeans(dataset, i, nstart=100)$withinss
S[i]<-sum(kresult)
} #calculo mediante kmeans la S para cada k
ggplot() +
geom_point(aes(x = 1:8, y = S), color = "blue") +
geom_line(aes(x = 1:8, y = S), color = "blue") +
ggtitle("Selección de clusters") +
xlab("Cantidad de Centroides k") +
ylab("S")
FIG 1: Diagrama para la selección de clusters
-Donde, según la FIG 1, parece sensato elegir cuatro clusters (o, como mucho, 5). Seleccionamos cuatro porque seguimos con la intuición de que los episodios se pueden separar en cuatro categorías (muy vistos y mal valorados, poco vistos y mal valorados, muy vistos y bien valorados,poco vistos y bien valorados).
Entonces, solicitamos la separación con \(K=4\), y obtenemos
kmeans <- kmeans(dataset, 4, iter.max = 1000, nstart = 25)
fviz_cluster(kmeans, data = dataset,stand=FALSE)
K-means clustering with 4 clusters of sizes 172, 65, 123, 189
Cluster means:
audiencia rating
1 10.87 7.27
2 24.95 8.07
3 16.76 8.01
4 6.44 6.95
- Si los vemos gráficamente, detectamos un posible problema:
 >-como vemos, parece ser la variable audiencia la que determina los clusters, esto es, la variable rating no parece determinar la separación. De hecho, si vemos la información que nos ofrece [R], la variable rating fluctúa poco (en un grupo es 7.27, en otro 8.07, en otro 8.01 y en el último 6.95).
>-como vemos, parece ser la variable audiencia la que determina los clusters, esto es, la variable rating no parece determinar la separación. De hecho, si vemos la información que nos ofrece [R], la variable rating fluctúa poco (en un grupo es 7.27, en otro 8.07, en otro 8.01 y en el último 6.95).
Recuerde que si ejecuta esto en su ordenador, le saldrá algo parecido pero no igual, debido a la aleatoriedad en los puntos de partida del algoritmo.
Ahora bien, ¿parece sensata dicha separación? Dese cuenta de que, en este algoritmo, se calculan las distancias al centroide utilizando las unidades de medida de las variables. Por lo tanto, si estas son dispares, las distancias las marcará la variable con unidades mayores (en este caso, la audiencia). Por eso, es conveniente tratar de homogeneizar las variables en un caso como esto. Lo que puede hacer es :
Estandarizar las variables
Una variable \(x\) con media \(\bar{x}\) y desviación típica \(\sigma_{x}\) se puede transformar a una variable con media cero y desviación típica 1 mediante la operación \(\frac{(x-\bar{x})}{\sigma_{x}}\). Sin embargo, eso no quiere decir que esté convirtiendo a la variable en Normal (error común), simplemente la está cambiando de escala.
- Ahora, realizamos la operación de estandarización y volvemos a obtener los cluster
audiencia<-(audiencia - mean(audiencia)) / sd(audiencia)
rating<-(rating - mean(rating)) / sd(rating)
dataset<-data.frame(audiencia,rating)
dataset<-na.omit(dataset)
kmeans <- kmeans(dataset, 4, iter.max = 1000, nstart = 25)
fviz_cluster(kmeans, data = dataset,stand=FALSE)
FIG 3: posibles clusters obtenidos con las variables estandarizadas
Ahora podemos separar a los episodios de los simpson en 4 grupos. El morado parecen aquellos con poca audiencia y peor valorados, el verde con mucha audiencia y valoración. El rojo parece que son poca audiencia y muy bien valorados y el grupo 3 parece un intermedio entre el grupo 1 y el 4. Si bien es cierto, esta separación parece que cuenta con más variabildiad en la variable rating.
Esto nos muestra que, en caso de tener unidades de medida distintas, conviene estandarizar las variables que, eso sí, pasan a tener todas la misma varianza (el problema de la estandarización). Podemos ilustrar las bondades de estandarizar
Atendiendo a estos datos simulados con, claramente, distintas unidades de medida
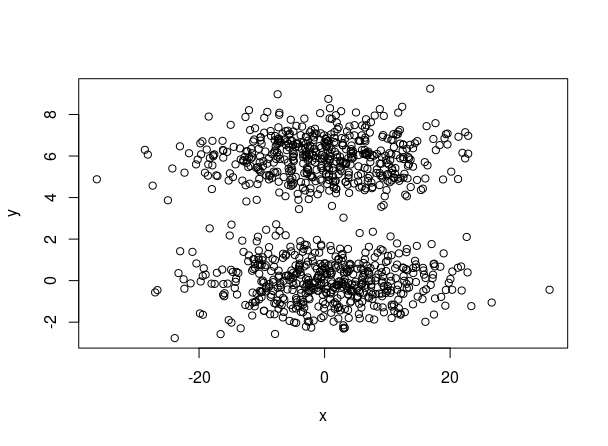
FIG 4: dos clusters simulados
de los que aquí puede disponer del código para simularlos:
x = rnorm(1000) * 10
y = c(rnorm(500), rnorm(500) + 6)
plot(x,y)Se obtienen los siguientes resultados de la función “kmeans” utilizando 2 clusters
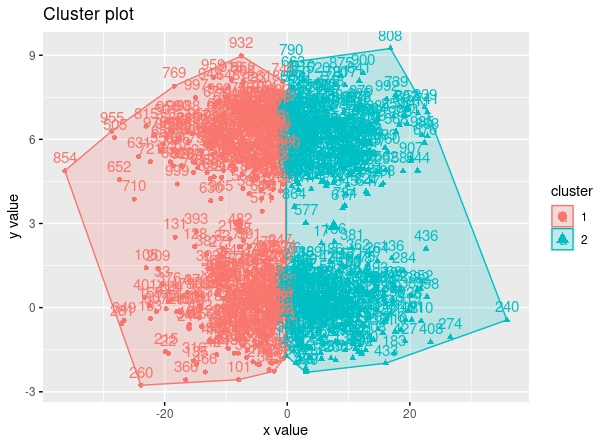
FIG 5: dos clusters simulados, entrenados “malamente” con k means
Sin embargo, tras estandarizarlos, obtenemos una solución más existosa
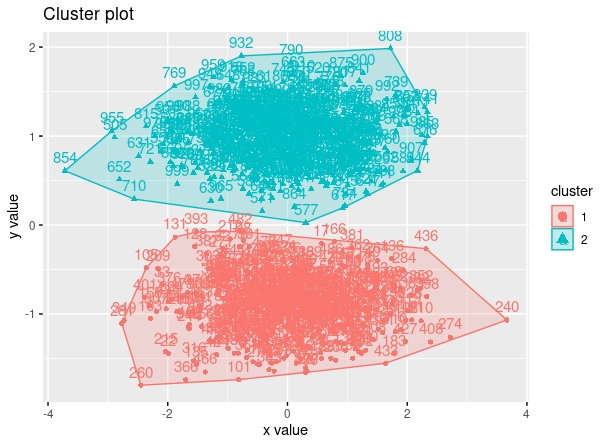
FIG 5: dos clusters simulados, entrenados con k means
Ahora bien, ¿es siempre la solución estandarizar? En el manual FRIEDMAN, Jerome H. The elements of statistical learning: Data mining, inference, and prediction. springer open, 2017.
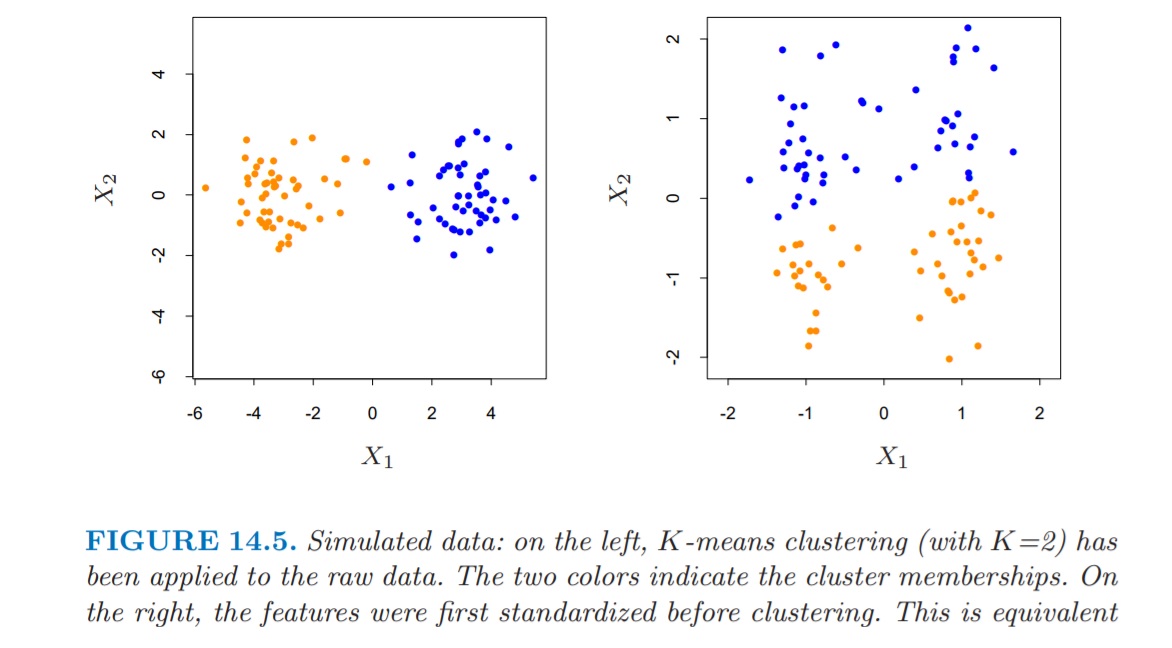
FIG 6: del libro “elements of statistical learning”
donde, como vemos, tras la estandarización, los clusters correspondientes sufren modificaciones al cambiar la dispersión respecto al eje de ordenadas.
- Sin embargo, parece razonable confiar en la estandarización siempre que las unidades de medida sean muy diferentes y nos interese realizar un cluster con dichas variables.
Clase 8. “El dendrograma”
Otra manera de busar clusters es, en vez de en el conjunto de observaciones, en el conjunto de variables (es decir, en vez de por filas, por columnas). Para ello, elegiremos la correlación como una medida de “similaridad” de variables. Aunque, obviamente, la correlación sólo mide similaridad de carácter lineal. Es decir: puede haber relaciones entre variables que este método no capte. Aunque lo usaremos, en general, por su sencillez y flexibilidad.
Suponga que tiene un conjunto de datos, con cinco variables (V1,V2,V3,V4,V5) del que obtiene su matriz de correlaciones, como se ve en la FIG 1, parte 1
FIG 1: De la matriz de correlaciones a la matriz de distancias
- En el punto (1), se muestra la matriz de correlaciones. En este caso, interpretaremos las correlaciones POSITIVAS y ALTAS como variables que están cerca y NEGATIVAS y ALTAS variables que están muy lejos. Como queremos una medida de distancia-positiva- que recoja esta idea, podemos utilizar \(d_{i,j}=1-\rho_{i,j}\), es decir, la distancia entre la variable \(i\) y la variable \(j\) consistirá en 1 menos la correlación.
- En el punto (2) se obtienen dichas distancias.
- Empecemos eligiendo el par de variables que tienen menor distancia que son V4 y V5. Las unimos en un gráfico que llamaremos dendrograma (no “dendograma”) y que se construye indicando, en el eje de ordenadas, dicha distancia. El eje de abcisas es “mudo”.
Ahora, pasemos a la FIG2
- En el punto (4) hemos creado una nueva categoría que es la unión de la variable 4 y la 5 y obtenemos las distancias del resto de variables a esta nueva categoría. Se coge la mayor de entre las dos posibles. Ahora la menor distancia es entre V2 y V3 y hacemos la correspondiente marca en el dendrograma.
- En el punto (5) hemos creado el nuevo grupo y recalculamos distancias hasta que, finalmente, llegamos al último punto
FIG 3: Construimos el dendrograma
Este gráfico nos permite agrupar las variables de forma jerárquica. Por ejemplo, si queremos hacer dos grupos, el gráfico nos dice, claramente, que uno de ellos estará formado por las variables V1,V4 y V5 y el otro por V2 y V3. Si quisiéramos hacer tres grupos, entonces, uno de ellos sería el formado por V1, el otro por V4 y V5 y el último por V2 y V3. Y así sucesivamente, descendiendo de manera imaginaria en el eje de ordenadas.
Es fácil obtener el dendrograma para los datos de los Simpson. Mediante este script y la función “hclust”
Homer<-(SIMPSONS$Homer)
Bart<-(SIMPSONS$Bart)
Lisa<-(SIMPSONS$Lisa)
Marge<-(SIMPSONS$Marge)
Apu<-SIMPSONS$Apu
Flanders<-SIMPSONS$Flanders
Krusty<- SIMPSONS$Krusty
Milhause<-SIMPSONS$Milhause
SIMPSONS_char<-data.frame(Homer,Bart,Lisa,Marge,Apu,Flanders,Krusty,Milhause)
dd <- as.dist((1 - cor(SIMPSONS_char)))
round(1000 * dd)
plot(hclust(dd))llegamos a la FIG 4, donde se pueden ver distintas agrupaciones posibles:
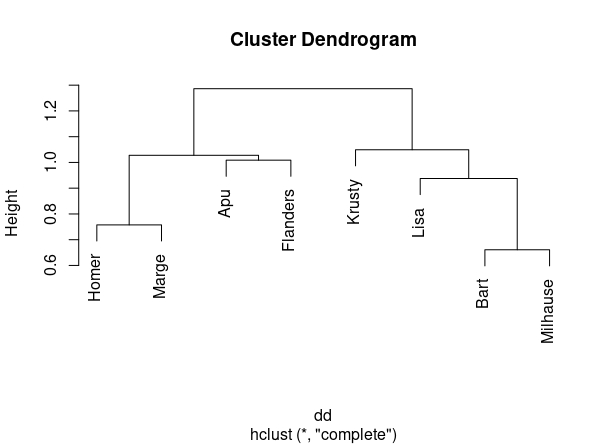 Si queremos:
Si queremos:
- Dos cluster: Homer+Marge+Apu+Flanders y Krusty+Lista+Bart+Milhause
- Tres cluster: Homer+Marge y Apu+Flanders y Krusty+Lista+Bart+Milhause
- Cuatro cluster: Homer+Marge, Apu+Flanders, Krusty, Lisa+Bart+Milhause
y así, sucesivamente.
Ejercicio de interés ¿Pueden estas asociaciones de personajes explicar los resultados de los cluster de episodios?