5 Classificação hierarquica do tipo SAHN
Organização básica
dev.off() #apaga os graficos, se houver algum
rm(list=ls(all=TRUE)) #limpa memoria
cat("\014") #limpa o console Instalando os pacotes necessários
install.packages("vegan")
install.packages("moments")
install.packages("ggplot2")
install.packages("dplyr")
install.packages("tidyr")
install.packages("tibble")
install.packages("tidyverse")
install.packages("forcats")
install.packages("iNEXT")
install.packages("openxlsx")
install.packages('Rcpp')
install.packages("ggplotify")5.1 Importar planilha de trabalho
library(openxlsx)
#shell.exec(dir) #abre o diretorio de trabalho no Windows Explorer
m_trab <- read.csv("zoorebio.csv",
sep = ";",
dec = ",",
header = T,
row.names = 1,
na.strings = NA)
#str(m_trab)
#View(m_trab)5.2 Reset point
m_trab <- m_trabAqui substitui-se uma nova matriz de dados, relativizada e/ou transformada, pela matriz bruta.
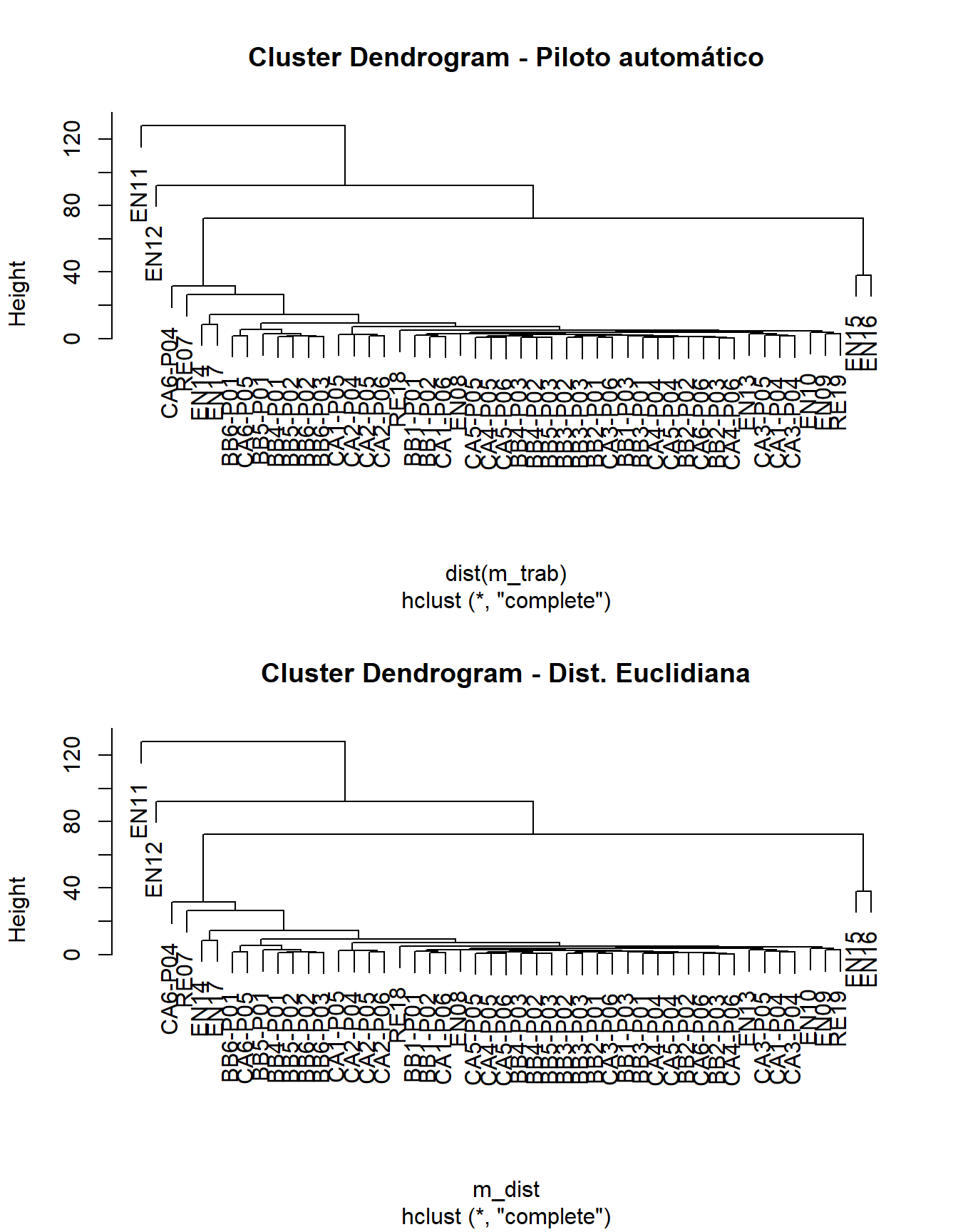
5.3 Cluster 1: Piloto automático
Aqui fazemos uma Análise Cluster no piloto automático. Não decidimos nenhum dos parâmetros importântes para uma Classificação.
cluster1 <- hclust(dist(m_trab))
plot(cluster1, main = "Cluster Dendrogram - Piloto automatico")
#rect.hclust(cluster1, k=3, h = NULL)
#?dist
#?hclust
É possivel definir uma caixa mostrado os principais grupos formados. Isso pode ser feito estabelecendo quantos grupos se quer mostrar, usando a função k = no. de grupos. Ou pode-se definir os grupos formados até uma determinanda distância, usando a função h = altura dos grupos no eixo das distâncias.
5.3.1 Histórico das fusões
library(dplyr)
cluster1$merge #mostra o histórico das fusões
idrow <- mutate(m_trab, id = row_number()) #cria um df com os numeros das linhas
idrow %>% relocate(id)
#?relocate## [,1] [,2]
## [1,] -6 -30
## [2,] -11 -15
## [3,] -28 -31
## [4,] -8 -9
## [5,] -29 -33
## [6,] -36 1
## [7,] -7 3
## [8,] -32 5
## [9,] -4 -27
## [10,] -12 2
## [11,] -5 6
## [12,] 7 11
## [13,] -10 -14
## [14,] -2 -21
## [15,] -17 -18
## [16,] 8 10
## [17,] 4 9
## [18,] -3 12
## [19,] -19 -25
## [20,] 13 15
## [21,] -23 -24
## [22,] -16 -35
## [23,] -26 19
## [24,] 17 18
## [25,] -1 14
## [26,] 16 24
## [27,] -22 21
## [28,] -20 27
## [29,] -37 26
## [30,] -13 20
## [31,] 25 29
## [32,] -38 -47
## [33,] -42 23
## [34,] 31 33
## [35,] -39 32
## [36,] 34 35
## [37,] -48 36
## [38,] 22 30
## [39,] 28 37
## [40,] -45 -46
## [41,] 38 39
## [42,] 40 41
## [43,] -49 42
## [44,] -34 43
## [45,] -43 -44
## [46,] 44 45
## [47,] -41 46
## [48,] -40 47
## id Bra.angularis Lepadella.sp Lecane.sp Lec.leontina Lec.bulla Lec.cornuta Lec.curvicornis Notholca.sp
## BB1-P01 1 0.0000000 0.0000000 0.0000000 0 1.0666667 0.0000000 0.0000000 0.0000000
## BB1-P02 2 0.5333333 0.1777778 0.0000000 0 0.0000000 0.0000000 0.0000000 0.0000000
## BB1-P03 3 0.5333333 0.1777778 0.0000000 0 0.3555556 0.0000000 0.0000000 0.0000000
## BB2-P01 4 0.0000000 0.5333333 0.0000000 0 0.8888889 0.0000000 0.0000000 0.0000000
## BB2-P02 5 0.0000000 0.1777778 0.0000000 0 0.1777778 0.0000000 0.0000000 0.0000000
## BB2-P03 6 0.0000000 0.1777778 0.0000000 0 0.0000000 0.0000000 0.0000000 0.0000000
## BB3-P01 7 0.0000000 0.0000000 0.1777778 0 0.5333333 0.1777778 0.0000000 0.0000000
## BB3-P02 8 0.0000000 0.0000000 0.0000000 0 1.6000000 0.0000000 0.0000000 0.0000000
## BB3-P03 9 0.0000000 0.0000000 0.0000000 0 1.4222222 0.1777778 0.0000000 0.0000000
## BB4-P01 10 0.0000000 0.0000000 0.0000000 0 1.4222222 0.0000000 0.1777778 0.0000000
## BB4-P02 11 0.0000000 0.0000000 0.0000000 0 0.3555556 0.0000000 0.0000000 0.1777778
## Bra.urceolaris Trichocerca.sp Lec.quadridentata Lec.kluchor Lec.lunaris Rotaria.sp Aspelta.sp Lec.furcata
## BB1-P01 0.1777778 0 0 0.1777778 0.3555556 0.0000000 0.1777778 0
## BB1-P02 0.0000000 0 0 0.0000000 0.7111111 1.0666667 0.0000000 0
## BB1-P03 0.0000000 0 0 0.0000000 1.0666667 0.0000000 0.0000000 0
## BB2-P01 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## BB2-P02 0.0000000 0 0 0.0000000 0.1777778 0.0000000 0.0000000 0
## BB2-P03 0.0000000 0 0 0.0000000 0.1777778 0.0000000 0.0000000 0
## BB3-P01 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## BB3-P02 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## BB3-P03 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## BB4-P01 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## BB4-P02 0.0000000 0 0 0.0000000 0.0000000 0.3555556 0.0000000 0
## Col.geophila Lep.dactyliseta Bra.calyciflorus Bra.caudatus Lec.crepida Pla.patulus Lec.ovalis Lec.elasma
## BB1-P01 0 0 0 0 0.0000000 0 0 0
## BB1-P02 0 0 0 0 0.0000000 0 0 0
## BB1-P03 0 0 0 0 0.0000000 0 0 0
## BB2-P01 0 0 0 0 0.0000000 0 0 0
## BB2-P02 0 0 0 0 0.0000000 0 0 0
## BB2-P03 0 0 0 0 0.1777778 0 0 0
## BB3-P01 0 0 0 0 0.0000000 0 0 0
## BB3-P02 0 0 0 0 0.0000000 0 0 0
## BB3-P03 0 0 0 0 0.0000000 0 0 0
## BB4-P01 0 0 0 0 0.0000000 0 0 0
## BB4-P02 0 0 0 0 0.0000000 0 0 0
## Pla.quadricornis Lec.aculeata Ker.tropica Bdelloidea Ker.lenzi Con.unicornis Alo.dadayi Macrothrix.sp
## BB1-P01 0 1.6000000 0 0.0000000 0 0.0000000 0 0.1777778
## BB1-P02 0 0.8888889 0 0.0000000 0 0.0000000 0 0.0000000
## BB1-P03 0 0.0000000 0 0.0000000 0 0.0000000 0 0.0000000
## BB2-P01 0 0.1777778 0 0.0000000 0 0.0000000 0 0.0000000
## BB2-P02 0 0.0000000 0 0.0000000 0 0.0000000 0 0.0000000
## BB2-P03 0 0.0000000 0 0.0000000 0 0.0000000 0 0.0000000
## BB3-P01 0 0.0000000 0 0.1777778 0 0.0000000 0 0.0000000
## BB3-P02 0 0.0000000 0 0.3555556 0 0.5333333 0 0.0000000
## BB3-P03 0 0.0000000 0 0.7111111 0 0.3555556 0 0.0000000
## BB4-P01 0 0.0000000 0 3.5555556 0 0.0000000 0 0.0000000
## BB4-P02 0 0.0000000 0 1.2444444 0 0.0000000 0 0.0000000
## Alonella.sp Nauplii Chy.eurynotus Cyclopoida Mac.collinsi Copepodite Paracyclops.sp Asc.ecaudis Lep.ovalis
## BB1-P01 0 0.7111111 0.3555556 0.0000000 0 0.0000000 0 0 0
## BB1-P02 0 0.8888889 0.0000000 0.0000000 0 0.0000000 0 0 0
## BB1-P03 0 0.1777778 0.0000000 0.0000000 0 0.0000000 0 0 0
## BB2-P01 0 0.8888889 0.0000000 0.0000000 0 0.1777778 0 0 0
## BB2-P02 0 0.7111111 0.0000000 0.0000000 0 0.0000000 0 0 0
## BB2-P03 0 0.3555556 0.0000000 0.0000000 0 0.0000000 0 0 0
## BB3-P01 0 0.0000000 0.0000000 0.0000000 0 0.0000000 0 0 0
## BB3-P02 0 0.3555556 0.0000000 0.1777778 0 0.1777778 0 0 0
## BB3-P03 0 0.3555556 0.0000000 0.0000000 0 0.0000000 0 0 0
## BB4-P01 0 1.4222222 0.0000000 0.0000000 0 0.1777778 0 0 0
## BB4-P02 0 0.1777778 0.0000000 0.0000000 0 0.0000000 0 0 0
## Polyarthra.sp Bra.havanaensis Hexarthra.sp Lec.hastata Tri.tetractis Ker.serrulata Notodiaptomus.sp Bra.falcatus
## BB1-P01 0 0 0 0 0 0 0 0
## BB1-P02 0 0 0 0 0 0 0 0
## BB1-P03 0 0 0 0 0 0 0 0
## BB2-P01 0 0 0 0 0 0 0 0
## BB2-P02 0 0 0 0 0 0 0 0
## BB2-P03 0 0 0 0 0 0 0 0
## BB3-P01 0 0 0 0 0 0 0 0
## BB3-P02 0 0 0 0 0 0 0 0
## BB3-P03 0 0 0 0 0 0 0 0
## BB4-P01 0 0 0 0 0 0 0 0
## BB4-P02 0 0 0 0 0 0 0 0
## Fil.longiseta Myt.crassipes Lep.patella Euchlanis.sp Colurella.sp Mytilina.sp Lec.monostyla Lec.ligona
## BB1-P01 0 0 0 0 0.0000000 0 0 0
## BB1-P02 0 0 0 0 0.0000000 0 0 0
## BB1-P03 0 0 0 0 0.1777778 0 0 0
## BB2-P01 0 0 0 0 0.0000000 0 0 0
## BB2-P02 0 0 0 0 0.0000000 0 0 0
## BB2-P03 0 0 0 0 0.0000000 0 0 0
## BB3-P01 0 0 0 0 0.0000000 0 0 0
## BB3-P02 0 0 0 0 0.0000000 0 0 0
## BB3-P03 0 0 0 0 0.0000000 0 0 0
## BB4-P01 0 0 0 0 0.0000000 0 0 0
## BB4-P02 0 0 0 0 0.0000000 0 0 0
## Ascomorpha.sp Pom.sulcata Harpacticoida Epi.senta Rot.neptunia Dis.aculeata Lec.luna Euc.dilatata
## BB1-P01 0.1777778 0.0000000 0.0000000 0.0000000 0 0 0 0
## BB1-P02 0.0000000 0.0000000 0.0000000 0.0000000 0 0 0 0
## BB1-P03 0.0000000 0.0000000 0.0000000 0.0000000 0 0 0 0
## BB2-P01 0.0000000 0.3555556 0.0000000 0.0000000 0 0 0 0
## BB2-P02 0.0000000 0.3555556 0.0000000 0.0000000 0 0 0 0
## BB2-P03 0.0000000 0.0000000 0.0000000 0.0000000 0 0 0 0
## BB3-P01 0.0000000 0.0000000 0.0000000 0.0000000 0 0 0 0
## BB3-P02 0.0000000 0.0000000 0.1777778 0.0000000 0 0 0 0
## BB3-P03 0.0000000 0.0000000 0.0000000 0.0000000 0 0 0 0
## BB4-P01 0.0000000 0.0000000 0.0000000 0.0000000 0 0 0 0
## BB4-P02 0.0000000 0.0000000 0.0000000 0.1777778 0 0 0 0
## Bea.eudactylota Chy.sphaericus Lec.hornemanni Dia.birgei Mac.subquadratus Bra.quadridentatus Pol.vulgaris
## BB1-P01 0 0 0 0 0 0 0
## BB1-P02 0 0 0 0 0 0 0
## BB1-P03 0 0 0 0 0 0 0
## BB2-P01 0 0 0 0 0 0 0
## BB2-P02 0 0 0 0 0 0 0
## BB2-P03 0 0 0 0 0 0 0
## BB3-P01 0 0 0 0 0 0 0
## BB3-P02 0 0 0 0 0 0 0
## BB3-P03 0 0 0 0 0 0 0
## BB4-P01 0 0 0 0 0 0 0
## BB4-P02 0 0 0 0 0 0 0
## Pol.bicerca Dicranop.sp Asc.saltans Moi.minuta Alo.hamulata Mac.laticornis Mac.mira Pro.similis Alo.pulchella
## BB1-P01 0 0 0 0 0 0 0 0 0
## BB1-P02 0 0 0 0 0 0 0 0 0
## BB1-P03 0 0 0 0 0 0 0 0 0
## BB2-P01 0 0 0 0 0 0 0 0 0
## BB2-P02 0 0 0 0 0 0 0 0 0
## BB2-P03 0 0 0 0 0 0 0 0 0
## BB3-P01 0 0 0 0 0 0 0 0 0
## BB3-P02 0 0 0 0 0 0 0 0 0
## BB3-P03 0 0 0 0 0 0 0 0 0
## BB4-P01 0 0 0 0 0 0 0 0 0
## BB4-P02 0 0 0 0 0 0 0 0 0
## Dia.spinulosum Chydorus.sp Ily.spinifer Dun.odontoplax
## BB1-P01 0 0 0 0
## BB1-P02 0 0 0 0
## BB1-P03 0 0 0 0
## BB2-P01 0 0 0 0
## BB2-P02 0 0 0 0
## BB2-P03 0 0 0 0
## BB3-P01 0 0 0 0
## BB3-P02 0 0 0 0
## BB3-P03 0 0 0 0
## BB4-P01 0 0 0 0
## BB4-P02 0 0 0 0
## [ reached 'max' / getOption("max.print") -- omitted 38 rows ]5.3.1.1 Alguns ajustes para melhorar a visualização
library(tidyverse)
merge <- as.data.frame(cluster1$merge)
merge[nrow(merge)+1,] = c("0","0")
merge
uas <- as.data.frame(rownames_to_column(m_trab,var = "UAs"))
uas
merges <- cbind(uas[c("UAs")], merge)
merges## V1 V2
## 1 -6 -30
## 2 -11 -15
## 3 -28 -31
## 4 -8 -9
## 5 -29 -33
## 6 -36 1
## 7 -7 3
## 8 -32 5
## 9 -4 -27
## 10 -12 2
## 11 -5 6
## 12 7 11
## 13 -10 -14
## 14 -2 -21
## 15 -17 -18
## 16 8 10
## 17 4 9
## 18 -3 12
## 19 -19 -25
## 20 13 15
## 21 -23 -24
## 22 -16 -35
## 23 -26 19
## 24 17 18
## 25 -1 14
## 26 16 24
## 27 -22 21
## 28 -20 27
## 29 -37 26
## 30 -13 20
## 31 25 29
## 32 -38 -47
## 33 -42 23
## 34 31 33
## 35 -39 32
## 36 34 35
## 37 -48 36
## 38 22 30
## 39 28 37
## 40 -45 -46
## 41 38 39
## 42 40 41
## 43 -49 42
## 44 -34 43
## 45 -43 -44
## 46 44 45
## 47 -41 46
## 48 -40 47
## 49 0 0
## UAs Bra.angularis Lepadella.sp Lecane.sp Lec.leontina Lec.bulla Lec.cornuta Lec.curvicornis Notholca.sp
## 1 BB1-P01 0.0000000 0.0000000 0.0000000 0 1.0666667 0.0000000 0.0000000 0.0000000
## 2 BB1-P02 0.5333333 0.1777778 0.0000000 0 0.0000000 0.0000000 0.0000000 0.0000000
## 3 BB1-P03 0.5333333 0.1777778 0.0000000 0 0.3555556 0.0000000 0.0000000 0.0000000
## 4 BB2-P01 0.0000000 0.5333333 0.0000000 0 0.8888889 0.0000000 0.0000000 0.0000000
## 5 BB2-P02 0.0000000 0.1777778 0.0000000 0 0.1777778 0.0000000 0.0000000 0.0000000
## 6 BB2-P03 0.0000000 0.1777778 0.0000000 0 0.0000000 0.0000000 0.0000000 0.0000000
## 7 BB3-P01 0.0000000 0.0000000 0.1777778 0 0.5333333 0.1777778 0.0000000 0.0000000
## 8 BB3-P02 0.0000000 0.0000000 0.0000000 0 1.6000000 0.0000000 0.0000000 0.0000000
## 9 BB3-P03 0.0000000 0.0000000 0.0000000 0 1.4222222 0.1777778 0.0000000 0.0000000
## 10 BB4-P01 0.0000000 0.0000000 0.0000000 0 1.4222222 0.0000000 0.1777778 0.0000000
## 11 BB4-P02 0.0000000 0.0000000 0.0000000 0 0.3555556 0.0000000 0.0000000 0.1777778
## Bra.urceolaris Trichocerca.sp Lec.quadridentata Lec.kluchor Lec.lunaris Rotaria.sp Aspelta.sp Lec.furcata
## 1 0.1777778 0 0 0.1777778 0.3555556 0.0000000 0.1777778 0
## 2 0.0000000 0 0 0.0000000 0.7111111 1.0666667 0.0000000 0
## 3 0.0000000 0 0 0.0000000 1.0666667 0.0000000 0.0000000 0
## 4 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## 5 0.0000000 0 0 0.0000000 0.1777778 0.0000000 0.0000000 0
## 6 0.0000000 0 0 0.0000000 0.1777778 0.0000000 0.0000000 0
## 7 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## 8 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## 9 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## 10 0.0000000 0 0 0.0000000 0.0000000 0.0000000 0.0000000 0
## 11 0.0000000 0 0 0.0000000 0.0000000 0.3555556 0.0000000 0
## Col.geophila Lep.dactyliseta Bra.calyciflorus Bra.caudatus Lec.crepida Pla.patulus Lec.ovalis Lec.elasma
## 1 0 0 0 0 0.0000000 0 0 0
## 2 0 0 0 0 0.0000000 0 0 0
## 3 0 0 0 0 0.0000000 0 0 0
## 4 0 0 0 0 0.0000000 0 0 0
## 5 0 0 0 0 0.0000000 0 0 0
## 6 0 0 0 0 0.1777778 0 0 0
## 7 0 0 0 0 0.0000000 0 0 0
## 8 0 0 0 0 0.0000000 0 0 0
## 9 0 0 0 0 0.0000000 0 0 0
## 10 0 0 0 0 0.0000000 0 0 0
## 11 0 0 0 0 0.0000000 0 0 0
## Pla.quadricornis Lec.aculeata Ker.tropica Bdelloidea Ker.lenzi Con.unicornis Alo.dadayi Macrothrix.sp Alonella.sp
## 1 0 1.6000000 0 0.0000000 0 0.0000000 0 0.1777778 0
## 2 0 0.8888889 0 0.0000000 0 0.0000000 0 0.0000000 0
## 3 0 0.0000000 0 0.0000000 0 0.0000000 0 0.0000000 0
## 4 0 0.1777778 0 0.0000000 0 0.0000000 0 0.0000000 0
## 5 0 0.0000000 0 0.0000000 0 0.0000000 0 0.0000000 0
## 6 0 0.0000000 0 0.0000000 0 0.0000000 0 0.0000000 0
## 7 0 0.0000000 0 0.1777778 0 0.0000000 0 0.0000000 0
## 8 0 0.0000000 0 0.3555556 0 0.5333333 0 0.0000000 0
## 9 0 0.0000000 0 0.7111111 0 0.3555556 0 0.0000000 0
## 10 0 0.0000000 0 3.5555556 0 0.0000000 0 0.0000000 0
## 11 0 0.0000000 0 1.2444444 0 0.0000000 0 0.0000000 0
## Nauplii Chy.eurynotus Cyclopoida Mac.collinsi Copepodite Paracyclops.sp Asc.ecaudis Lep.ovalis Polyarthra.sp
## 1 0.7111111 0.3555556 0.0000000 0 0.0000000 0 0 0 0
## 2 0.8888889 0.0000000 0.0000000 0 0.0000000 0 0 0 0
## 3 0.1777778 0.0000000 0.0000000 0 0.0000000 0 0 0 0
## 4 0.8888889 0.0000000 0.0000000 0 0.1777778 0 0 0 0
## 5 0.7111111 0.0000000 0.0000000 0 0.0000000 0 0 0 0
## 6 0.3555556 0.0000000 0.0000000 0 0.0000000 0 0 0 0
## 7 0.0000000 0.0000000 0.0000000 0 0.0000000 0 0 0 0
## 8 0.3555556 0.0000000 0.1777778 0 0.1777778 0 0 0 0
## 9 0.3555556 0.0000000 0.0000000 0 0.0000000 0 0 0 0
## 10 1.4222222 0.0000000 0.0000000 0 0.1777778 0 0 0 0
## 11 0.1777778 0.0000000 0.0000000 0 0.0000000 0 0 0 0
## Bra.havanaensis Hexarthra.sp Lec.hastata Tri.tetractis Ker.serrulata Notodiaptomus.sp Bra.falcatus Fil.longiseta
## 1 0 0 0 0 0 0 0 0
## 2 0 0 0 0 0 0 0 0
## 3 0 0 0 0 0 0 0 0
## 4 0 0 0 0 0 0 0 0
## 5 0 0 0 0 0 0 0 0
## 6 0 0 0 0 0 0 0 0
## 7 0 0 0 0 0 0 0 0
## 8 0 0 0 0 0 0 0 0
## 9 0 0 0 0 0 0 0 0
## 10 0 0 0 0 0 0 0 0
## 11 0 0 0 0 0 0 0 0
## Myt.crassipes Lep.patella Euchlanis.sp Colurella.sp Mytilina.sp Lec.monostyla Lec.ligona Ascomorpha.sp Pom.sulcata
## 1 0 0 0 0.0000000 0 0 0 0.1777778 0.0000000
## 2 0 0 0 0.0000000 0 0 0 0.0000000 0.0000000
## 3 0 0 0 0.1777778 0 0 0 0.0000000 0.0000000
## 4 0 0 0 0.0000000 0 0 0 0.0000000 0.3555556
## 5 0 0 0 0.0000000 0 0 0 0.0000000 0.3555556
## 6 0 0 0 0.0000000 0 0 0 0.0000000 0.0000000
## 7 0 0 0 0.0000000 0 0 0 0.0000000 0.0000000
## 8 0 0 0 0.0000000 0 0 0 0.0000000 0.0000000
## 9 0 0 0 0.0000000 0 0 0 0.0000000 0.0000000
## 10 0 0 0 0.0000000 0 0 0 0.0000000 0.0000000
## 11 0 0 0 0.0000000 0 0 0 0.0000000 0.0000000
## Harpacticoida Epi.senta Rot.neptunia Dis.aculeata Lec.luna Euc.dilatata Bea.eudactylota Chy.sphaericus Lec.hornemanni
## 1 0.0000000 0.0000000 0 0 0 0 0 0 0
## 2 0.0000000 0.0000000 0 0 0 0 0 0 0
## 3 0.0000000 0.0000000 0 0 0 0 0 0 0
## 4 0.0000000 0.0000000 0 0 0 0 0 0 0
## 5 0.0000000 0.0000000 0 0 0 0 0 0 0
## 6 0.0000000 0.0000000 0 0 0 0 0 0 0
## 7 0.0000000 0.0000000 0 0 0 0 0 0 0
## 8 0.1777778 0.0000000 0 0 0 0 0 0 0
## 9 0.0000000 0.0000000 0 0 0 0 0 0 0
## 10 0.0000000 0.0000000 0 0 0 0 0 0 0
## 11 0.0000000 0.1777778 0 0 0 0 0 0 0
## Dia.birgei Mac.subquadratus Bra.quadridentatus Pol.vulgaris Pol.bicerca Dicranop.sp Asc.saltans Moi.minuta
## 1 0 0 0 0 0 0 0 0
## 2 0 0 0 0 0 0 0 0
## 3 0 0 0 0 0 0 0 0
## 4 0 0 0 0 0 0 0 0
## 5 0 0 0 0 0 0 0 0
## 6 0 0 0 0 0 0 0 0
## 7 0 0 0 0 0 0 0 0
## 8 0 0 0 0 0 0 0 0
## 9 0 0 0 0 0 0 0 0
## 10 0 0 0 0 0 0 0 0
## 11 0 0 0 0 0 0 0 0
## Alo.hamulata Mac.laticornis Mac.mira Pro.similis Alo.pulchella Dia.spinulosum Chydorus.sp Ily.spinifer Dun.odontoplax
## 1 0 0 0 0 0 0 0 0 0
## 2 0 0 0 0 0 0 0 0 0
## 3 0 0 0 0 0 0 0 0 0
## 4 0 0 0 0 0 0 0 0 0
## 5 0 0 0 0 0 0 0 0 0
## 6 0 0 0 0 0 0 0 0 0
## 7 0 0 0 0 0 0 0 0 0
## 8 0 0 0 0 0 0 0 0 0
## 9 0 0 0 0 0 0 0 0 0
## 10 0 0 0 0 0 0 0 0 0
## 11 0 0 0 0 0 0 0 0 0
## [ reached 'max' / getOption("max.print") -- omitted 38 rows ]
## UAs V1 V2
## 1 BB1-P01 -6 -30
## 2 BB1-P02 -11 -15
## 3 BB1-P03 -28 -31
## 4 BB2-P01 -8 -9
## 5 BB2-P02 -29 -33
## 6 BB2-P03 -36 1
## 7 BB3-P01 -7 3
## 8 BB3-P02 -32 5
## 9 BB3-P03 -4 -27
## 10 BB4-P01 -12 2
## 11 BB4-P02 -5 6
## 12 BB4-P03 7 11
## 13 BB5-P01 -10 -14
## 14 BB5-P02 -2 -21
## 15 BB5-P03 -17 -18
## 16 BB6-P01 8 10
## 17 BB6-P02 4 9
## 18 BB6-P03 -3 12
## 19 CA1-P04 -19 -25
## 20 CA1-P05 13 15
## 21 CA1-P06 -23 -24
## 22 CA2-P04 -16 -35
## 23 CA2-P05 -26 19
## 24 CA2-P06 17 18
## 25 CA3-P04 -1 14
## 26 CA3-P05 16 24
## 27 CA3-P06 -22 21
## 28 CA4-P04 -20 27
## 29 CA4-P05 -37 26
## 30 CA4-P06 -13 20
## 31 CA5-P04 25 29
## 32 CA5-P05 -38 -47
## 33 CA5-P06 -42 23
## 34 CA6-P04 31 33
## 35 CA6-P05 -39 32
## 36 CA6-P06 34 35
## 37 EN08 -48 36
## 38 EN09 22 30
## 39 EN10 28 37
## 40 EN11 -45 -46
## 41 EN12 38 39
## 42 EN13 40 41
## 43 EN15 -49 42
## 44 EN16 -34 43
## 45 EN14 -43 -44
## 46 EN17 44 45
## 47 RE19 -41 46
## 48 RE18 -40 47
## 49 RE07 0 05.4 Cluster 2: Criando a matriz de distâncias Euclidiana
m_dist <- dist(m_trab, method = "euclidean",
diag = TRUE,
upper = FALSE)
m_dist[1:6]
cluster2 <- hclust(m_dist, method = "complete") #método de fusão
plot (cluster2, main = "Cluster Dendrogram - Dist.Euclidiana")
#rect.hclust(cluster2, k = 3)
#?dist
#?hclust## [1] 1.881423 2.118467 1.714427 1.955556 2.050233 1.963620
O significado do argumento method = da função dist() usada no códico acima, depende do contexto em que ela é usada.
A função
dist()no R é usada para calcular as distâncias entre as observações em um conjunto de dados. Essa função tem vários métodos de cálculo de distância, que podem ser especificados pelo argumentomethod =, a exemplo de “euclidean”, “manhattan”, “canberra”, entre outras.Quando o argumento
method =é usado em conjunto com a funçãohclust(), como no código acima, para realizar uma análise de classificação (ou cluster) hierárquica, então ela passa a representar um método de fusão, a exemplo de UPGMA, “nearest neighbour”, “furtherst neighbour”, entre outros.
A tabela 5.1 resume os métodos de fusão disponiveis na função hclust().
| Method= | Método de fusão |
|---|---|
| ward.D | Método de Ward com variância mínima |
| ward.D2 | Método de Ward com variância mínima ajustada |
| single | Método de ligação simples (vizinho mais próximo) |
| complete | Método de ligação completa (vizinho mais distante) |
| average | Método de ligação baseado na média, equivale ao UPGMA |
| mcquitty | Método de ligação de McQuitty, equivale ao WPGMA |
| median | Método de ligação baseado na mediana, ou WPGMC |
| centroid | Método de ligação baseado no centróide, ou UPGMC |
5.5 Cluster 3: Relativização pelo total da coluna
library(vegan)
m_relcol <- decostand(m_trab, method = "total", #OUTROS METODOS: total, max, normalize, range, rankm
MARGIN = 2) #1-linha, 2-coluna
#m_relcol5.6 Cluster 4: Transformação pelo arcoseno da raiz quadrada
Agora aplicaremos a transformação do arcoseno da raiz quadrada na matriz relativizada por colunas. Lembre que em ambas as matrizes entramos dados que variam entre 0 e 1, e os resutados retornados também variam entre 0 e 1, mas em um nível de compressão de valores extremos mais desejável. Usaremos a matriz relativizada/transformada para fazermos a proxima classificação.
m_asrq <- asin(sqrt(m_relcol)) #valores de entrada tem que ser entre 0-1
#View(m_asrq)
#m_asrq5.6.1 Gráfico de clustering relativizado/transformado
Aqui fazemos uma Análise Cluster no piloto automático, para comparar com os resultados da primeira classificação, que não havia sido relativizada/transformada.
par(mfrow = c(1,1))
cluster4 <- hclust(dist(m_asrq))
plot(cluster4, main = "Cluster Dendrogram - ArcSin.SQRT")
#rect.hclust(cluster4, k = 3)5.7 Cluster 5: Controle total dos parâmetros
vegdist <- vegdist(m_asrq, method = "bray",
diag = TRUE,
upper = FALSE)
as.matrix(vegdist)[1:7, 1:7]
cluster5 <- hclust(vegdist, method = "average")
plot (cluster5, main = "Cluster Dendrogram - Bray-Curtis")
#rect.hclust(cluster5, k = 3, h = NULL)
#h = 0.8 fornece os grupos formados na altura h
#vegdist
cluster5$merge
cluster5$height
idrow <- mutate(m_asrq, id = row_number())
idrow %>% relocate(id)## BB1-P01 BB1-P02 BB1-P03 BB2-P01 BB2-P02 BB2-P03 BB3-P01
## BB1-P01 0.0000000 0.7689609 0.9112406 0.8853817 0.9238529 0.9368508 0.9764590
## BB1-P02 0.7689609 0.0000000 0.3156620 0.7414775 0.7774095 0.7427936 1.0000000
## BB1-P03 0.9112406 0.3156620 0.0000000 0.8490982 0.7330628 0.7025108 0.9486736
## BB2-P01 0.8853817 0.7414775 0.8490982 0.0000000 0.2237880 0.8439695 0.9423545
## BB2-P02 0.9238529 0.7774095 0.7330628 0.2237880 0.0000000 0.6680542 0.9613441
## BB2-P03 0.9368508 0.7427936 0.7025108 0.8439695 0.6680542 0.0000000 1.0000000
## BB3-P01 0.9764590 1.0000000 0.9486736 0.9423545 0.9613441 1.0000000 0.0000000
## [,1] [,2]
## [1,] -16 -18
## [2,] -4 -5
## [3,] -2 -3
## [4,] -29 -31
## [5,] -14 -32
## [6,] -13 -17
## [7,] -15 -33
## [8,] -12 4
## [9,] -26 -27
## [10,] -47 -48
## [11,] -10 6
## [12,] -8 -9
## [13,] 1 11
## [14,] -38 -39
## [15,] -1 -19
## [16,] -28 8
## [17,] -35 13
## [18,] 5 7
## [19,] -30 16
## [20,] -22 9
## [21,] -46 10
## [22,] -41 -44
## [23,] 17 19
## [24,] -6 3
## [25,] -37 14
## [26,] 18 23
## [27,] -21 15
## [28,] -20 -25
## [29,] 2 24
## [30,] -7 20
## [31,] -45 22
## [32,] -49 21
## [33,] -23 -42
## [34,] 12 26
## [35,] 27 28
## [36,] -36 34
## [37,] -24 35
## [38,] 30 36
## [39,] 25 32
## [40,] -34 31
## [41,] 29 37
## [42,] -43 40
## [43,] 38 41
## [44,] 39 42
## [45,] 33 43
## [46,] 44 45
## [47,] -11 46
## [48,] -40 47
## [1] 0.1420777 0.2237880 0.3156620 0.3360204 0.3411674 0.4110340 0.4628678 0.4677337 0.5212990 0.5450952 0.5461220
## [12] 0.5674691 0.5920172 0.5988653 0.6115249 0.6174625 0.6184685 0.6241690 0.6566281 0.6747656 0.6964157 0.7127419
## [23] 0.7152764 0.7226522 0.7242707 0.7460356 0.7463749 0.7591748 0.7688453 0.7824023 0.7891467 0.7939359 0.8056075
## [34] 0.8114882 0.8220119 0.8225170 0.8335702 0.8509400 0.8566322 0.8698919 0.8824885 0.8867692 0.9045488 0.9071872
## [45] 0.9176239 0.9264934 0.9421947 0.9702455
## id Bra.angularis Lepadella.sp Lecane.sp Lec.leontina Lec.bulla Lec.cornuta Lec.curvicornis Notholca.sp
## BB1-P01 1 0.0000000 0.0000000 0.0000000 0 0.09258224 0.0000000 0.0000000 0.000000
## BB1-P02 2 0.5796397 0.1234045 0.0000000 0 0.00000000 0.0000000 0.0000000 0.000000
## BB1-P03 3 0.5796397 0.1234045 0.0000000 0 0.05340143 0.0000000 0.0000000 0.000000
## BB2-P01 4 0.0000000 0.2148498 0.0000000 0 0.08449545 0.0000000 0.0000000 0.000000
## BB2-P02 5 0.0000000 0.1234045 0.0000000 0 0.03775153 0.0000000 0.0000000 0.000000
## BB2-P03 6 0.0000000 0.1234045 0.0000000 0 0.00000000 0.0000000 0.0000000 0.000000
## BB3-P01 7 0.0000000 0.0000000 0.5235988 0 0.06541870 0.1777106 0.0000000 0.000000
## BB3-P02 8 0.0000000 0.0000000 0.0000000 0 0.11347105 0.0000000 0.0000000 0.000000
## BB3-P03 9 0.0000000 0.0000000 0.0000000 0 0.10695591 0.1777106 0.0000000 0.000000
## BB4-P01 10 0.0000000 0.0000000 0.0000000 0 0.10695591 0.0000000 0.2314774 0.000000
## BB4-P02 11 0.0000000 0.0000000 0.0000000 0 0.05340143 0.0000000 0.0000000 1.570796
## Bra.urceolaris Trichocerca.sp Lec.quadridentata Lec.kluchor Lec.lunaris Rotaria.sp Aspelta.sp Lec.furcata
## BB1-P01 0.6154797 0 0 0.3875967 0.1851713 0.00000000 0.3062774 0
## BB1-P02 0.0000000 0 0 0.0000000 0.2634135 0.12009695 0.0000000 0
## BB1-P03 0.0000000 0 0 0.0000000 0.3245649 0.00000000 0.0000000 0
## BB2-P01 0.0000000 0 0 0.0000000 0.0000000 0.00000000 0.0000000 0
## BB2-P02 0.0000000 0 0 0.0000000 0.1305595 0.00000000 0.0000000 0
## BB2-P03 0.0000000 0 0 0.0000000 0.1305595 0.00000000 0.0000000 0
## BB3-P01 0.0000000 0 0 0.0000000 0.0000000 0.00000000 0.0000000 0
## BB3-P02 0.0000000 0 0 0.0000000 0.0000000 0.00000000 0.0000000 0
## BB3-P03 0.0000000 0 0 0.0000000 0.0000000 0.00000000 0.0000000 0
## BB4-P01 0.0000000 0 0 0.0000000 0.0000000 0.00000000 0.0000000 0
## BB4-P02 0.0000000 0 0 0.0000000 0.0000000 0.06922673 0.0000000 0
## Col.geophila Lep.dactyliseta Bra.calyciflorus Bra.caudatus Lec.crepida Pla.patulus Lec.ovalis Lec.elasma
## BB1-P01 0 0 0 0 0.0000000 0 0 0
## BB1-P02 0 0 0 0 0.0000000 0 0 0
## BB1-P03 0 0 0 0 0.0000000 0 0 0
## BB2-P01 0 0 0 0 0.0000000 0 0 0
## BB2-P02 0 0 0 0 0.0000000 0 0 0
## BB2-P03 0 0 0 0 0.3398369 0 0 0
## BB3-P01 0 0 0 0 0.0000000 0 0 0
## BB3-P02 0 0 0 0 0.0000000 0 0 0
## BB3-P03 0 0 0 0 0.0000000 0 0 0
## BB4-P01 0 0 0 0 0.0000000 0 0 0
## BB4-P02 0 0 0 0 0.0000000 0 0 0
## Pla.quadricornis Lec.aculeata Ker.tropica Bdelloidea Ker.lenzi Con.unicornis Alo.dadayi Macrothrix.sp
## BB1-P01 0 0.6939806 0 0.00000000 0 0.0000000 0 0.3062774
## BB1-P02 0 0.4969325 0 0.00000000 0 0.0000000 0 0.0000000
## BB1-P03 0 0.0000000 0 0.00000000 0 0.0000000 0 0.0000000
## BB2-P01 0 0.2148498 0 0.00000000 0 0.0000000 0 0.0000000
## BB2-P02 0 0.0000000 0 0.00000000 0 0.0000000 0 0.0000000
## BB2-P03 0 0.0000000 0 0.00000000 0 0.0000000 0 0.0000000
## BB3-P01 0 0.0000000 0 0.05421934 0 0.0000000 0 0.0000000
## BB3-P02 0 0.0000000 0 0.07671539 0 0.7853982 0 0.0000000
## BB3-P03 0 0.0000000 0 0.10859890 0 0.6154797 0 0.0000000
## BB4-P01 0 0.0000000 0 0.24479505 0 0.0000000 0 0.0000000
## BB4-P02 0 0.0000000 0 0.14387649 0 0.0000000 0 0.0000000
## Alonella.sp Nauplii Chy.eurynotus Cyclopoida Mac.collinsi Copepodite Paracyclops.sp Asc.ecaudis Lep.ovalis
## BB1-P01 0 0.05514927 0.5235988 0.0000000 0 0.00000000 0 0 0
## BB1-P02 0 0.06166658 0.0000000 0.0000000 0 0.00000000 0 0 0
## BB1-P03 0 0.02756415 0.0000000 0.0000000 0 0.00000000 0 0 0
## BB2-P01 0 0.06166658 0.0000000 0.0000000 0 0.08748195 0 0 0
## BB2-P02 0 0.05514927 0.0000000 0.0000000 0 0.00000000 0 0 0
## BB2-P03 0 0.03898653 0.0000000 0.0000000 0 0.00000000 0 0 0
## BB3-P01 0 0.00000000 0.0000000 0.0000000 0 0.00000000 0 0 0
## BB3-P02 0 0.03898653 0.0000000 0.1549223 0 0.08748195 0 0 0
## BB3-P03 0 0.03898653 0.0000000 0.0000000 0 0.00000000 0 0 0
## BB4-P01 0 0.07803248 0.0000000 0.0000000 0 0.08748195 0 0 0
## BB4-P02 0 0.02756415 0.0000000 0.0000000 0 0.00000000 0 0 0
## Polyarthra.sp Bra.havanaensis Hexarthra.sp Lec.hastata Tri.tetractis Ker.serrulata Notodiaptomus.sp Bra.falcatus
## BB1-P01 0 0 0 0 0 0 0 0
## BB1-P02 0 0 0 0 0 0 0 0
## BB1-P03 0 0 0 0 0 0 0 0
## BB2-P01 0 0 0 0 0 0 0 0
## BB2-P02 0 0 0 0 0 0 0 0
## BB2-P03 0 0 0 0 0 0 0 0
## BB3-P01 0 0 0 0 0 0 0 0
## BB3-P02 0 0 0 0 0 0 0 0
## BB3-P03 0 0 0 0 0 0 0 0
## BB4-P01 0 0 0 0 0 0 0 0
## BB4-P02 0 0 0 0 0 0 0 0
## Fil.longiseta Myt.crassipes Lep.patella Euchlanis.sp Colurella.sp Mytilina.sp Lec.monostyla Lec.ligona
## BB1-P01 0 0 0 0 0.0000000 0 0 0
## BB1-P02 0 0 0 0 0.0000000 0 0 0
## BB1-P03 0 0 0 0 0.1513326 0 0 0
## BB2-P01 0 0 0 0 0.0000000 0 0 0
## BB2-P02 0 0 0 0 0.0000000 0 0 0
## BB2-P03 0 0 0 0 0.0000000 0 0 0
## BB3-P01 0 0 0 0 0.0000000 0 0 0
## BB3-P02 0 0 0 0 0.0000000 0 0 0
## BB3-P03 0 0 0 0 0.0000000 0 0 0
## BB4-P01 0 0 0 0 0.0000000 0 0 0
## BB4-P02 0 0 0 0 0.0000000 0 0 0
## Ascomorpha.sp Pom.sulcata Harpacticoida Epi.senta Rot.neptunia Dis.aculeata Lec.luna Euc.dilatata
## BB1-P01 1.570796 0.0000000 0.000000 0.000000 0 0 0 0
## BB1-P02 0.000000 0.0000000 0.000000 0.000000 0 0 0 0
## BB1-P03 0.000000 0.0000000 0.000000 0.000000 0 0 0 0
## BB2-P01 0.000000 0.7853982 0.000000 0.000000 0 0 0 0
## BB2-P02 0.000000 0.7853982 0.000000 0.000000 0 0 0 0
## BB2-P03 0.000000 0.0000000 0.000000 0.000000 0 0 0 0
## BB3-P01 0.000000 0.0000000 0.000000 0.000000 0 0 0 0
## BB3-P02 0.000000 0.0000000 1.570796 0.000000 0 0 0 0
## BB3-P03 0.000000 0.0000000 0.000000 0.000000 0 0 0 0
## BB4-P01 0.000000 0.0000000 0.000000 0.000000 0 0 0 0
## BB4-P02 0.000000 0.0000000 0.000000 1.570796 0 0 0 0
## Bea.eudactylota Chy.sphaericus Lec.hornemanni Dia.birgei Mac.subquadratus Bra.quadridentatus Pol.vulgaris
## BB1-P01 0 0 0 0 0 0 0
## BB1-P02 0 0 0 0 0 0 0
## BB1-P03 0 0 0 0 0 0 0
## BB2-P01 0 0 0 0 0 0 0
## BB2-P02 0 0 0 0 0 0 0
## BB2-P03 0 0 0 0 0 0 0
## BB3-P01 0 0 0 0 0 0 0
## BB3-P02 0 0 0 0 0 0 0
## BB3-P03 0 0 0 0 0 0 0
## BB4-P01 0 0 0 0 0 0 0
## BB4-P02 0 0 0 0 0 0 0
## Pol.bicerca Dicranop.sp Asc.saltans Moi.minuta Alo.hamulata Mac.laticornis Mac.mira Pro.similis Alo.pulchella
## BB1-P01 0 0 0 0 0 0 0 0 0
## BB1-P02 0 0 0 0 0 0 0 0 0
## BB1-P03 0 0 0 0 0 0 0 0 0
## BB2-P01 0 0 0 0 0 0 0 0 0
## BB2-P02 0 0 0 0 0 0 0 0 0
## BB2-P03 0 0 0 0 0 0 0 0 0
## BB3-P01 0 0 0 0 0 0 0 0 0
## BB3-P02 0 0 0 0 0 0 0 0 0
## BB3-P03 0 0 0 0 0 0 0 0 0
## BB4-P01 0 0 0 0 0 0 0 0 0
## BB4-P02 0 0 0 0 0 0 0 0 0
## Dia.spinulosum Chydorus.sp Ily.spinifer Dun.odontoplax
## BB1-P01 0 0 0 0
## BB1-P02 0 0 0 0
## BB1-P03 0 0 0 0
## BB2-P01 0 0 0 0
## BB2-P02 0 0 0 0
## BB2-P03 0 0 0 0
## BB3-P01 0 0 0 0
## BB3-P02 0 0 0 0
## BB3-P03 0 0 0 0
## BB4-P01 0 0 0 0
## BB4-P02 0 0 0 0
## [ reached 'max' / getOption("max.print") -- omitted 38 rows ]No código acima, h = 0.8 fornece os grupos formados na altura h do eixos das distâncias do dendrograma. Ou seja, no dendrograma, o eixo y (HEIGHT, “h”) representa o valor da distancia escolhida entre os objetos ou grupos de objetos. Portanto, se dois objetos ou grupos de objetos foram agrupados num dado valor (0.8, por exemplo) no eixo height, isso significa que a distancia entre esses objetos é 0.8.
5.8 Construindo uma prancha de gráficos comparativos
#png("fig-clusters1-5.png", width = 15, height = 9, units = "in", res = 300)
par (mfrow = c (2,2))
plot(cluster1, main = "Dist.Euclidiana")
plot(cluster3, main = "Rel.Coluna")
plot(cluster4, main = "ArcSin.SQRT")
plot(cluster5, main = "Bray-Curtis")
5.9 Construindo heatmaps
Agora que vimos de forma comparativa a importância de se relativizar e transformar nossos dados, e também de escolher de forma consciente a medida de distância e o método de fusão, podemos avançar um pouco mais na interpretação dos nossos dados.
Um “heatmap”, ou mapa de calor, é uma imagem em cores (basicamente image(t(x))) com um dendrograma adicionado ao lado esquerdo e/ou ao topo. Normalmente, é realizado o reordenamento das linhas e colunas de acordo com algum conjunto de valores (médias de linha ou coluna) dentro das restrições impostas pelo dendrograma.
Primeiro vamos rever o dendrograma final, usando a Distância de Bray-Curtis e o Método de Fusão UPGMA (ou “average”).
library("RColorBrewer")
library("gplots")
par(mfrow = c(1,1))
vegdist <- vegdist(m_asrq, method = "bray",
diag = TRUE,
upper = FALSE)
cluster5 <- hclust(vegdist, method = "average")
plot (cluster5, main = "Cluster Dendrogram - Bray-Curtis")
heatdist <- as.matrix(vegdist)
#heatdist
Na sequência fazemos um heatmap objetos x objetos, ou seja a comparação bidimensional das clustereds entre unidades amostrais.
AQUI
library(ggplotify)
col <- rev(heat.colors(999)) #rev() reverte as cores do heatmap
p <- as.ggplot(~heatmap.2(x=(as.matrix(vegdist)), #objetos x objetos
Rowv = as.dendrogram(cluster5),
Colv = as.dendrogram(cluster5),
key = T, tracecol = NA, revC = T,
col = heat.colors, #dissimilaridade = 1 - similaridade
density.info = "none",
xlab = "UA´s", ylab = "UA´s",
mar = c(6, 6) + 0.2)
)
p <- p + theme_bw() +
theme(axis.line = element_blank(),
axis.text = element_blank(),
axis.title = element_blank(),
axis.ticks = element_blank(),
panel.grid.major = element_blank(),
panel.grid.minor = element_blank(),
panel.border = element_blank(),
panel.background = element_blank())
ggsave("fig-heatmap1.png", plot = p, dpi = 300)## Saving 8 x 9 in imageAgora vamos criar um novo dendrograma, para as espécies, mostrando sua similaridade em termos de em quais UA´s elas são mais comuns.
cluster6 <- hclust((vegdist(t(m_asrq), method = "bray",
diag = TRUE,
upper = FALSE)), method = "average")
plot (cluster6, main = "Dendrograma dos atributos")
Na sequencia, fazemos um novo heatmap objetos x atributos, com a comparação bidimensional das clustereds formadas entre unidades amostrais em relação as espécies mais comuns que ocorreram em cada unidade amostral.
#png("fig-heatmap2.png", width = 10.8, height = 9.15, units = "in", res = 300)
heatmap.2(t(as.matrix(m_asrq)), #objetos x atributos
Colv = as.dendrogram(cluster5),
Rowv = as.dendrogram(cluster6),
key = T, tracecol = NA, revC = T,
col = col,
density.info = "none",
xlab = "Unidades amostrais", ylab = "Espécies",
mar = c(6, 8) + 0.1) #adjust margin size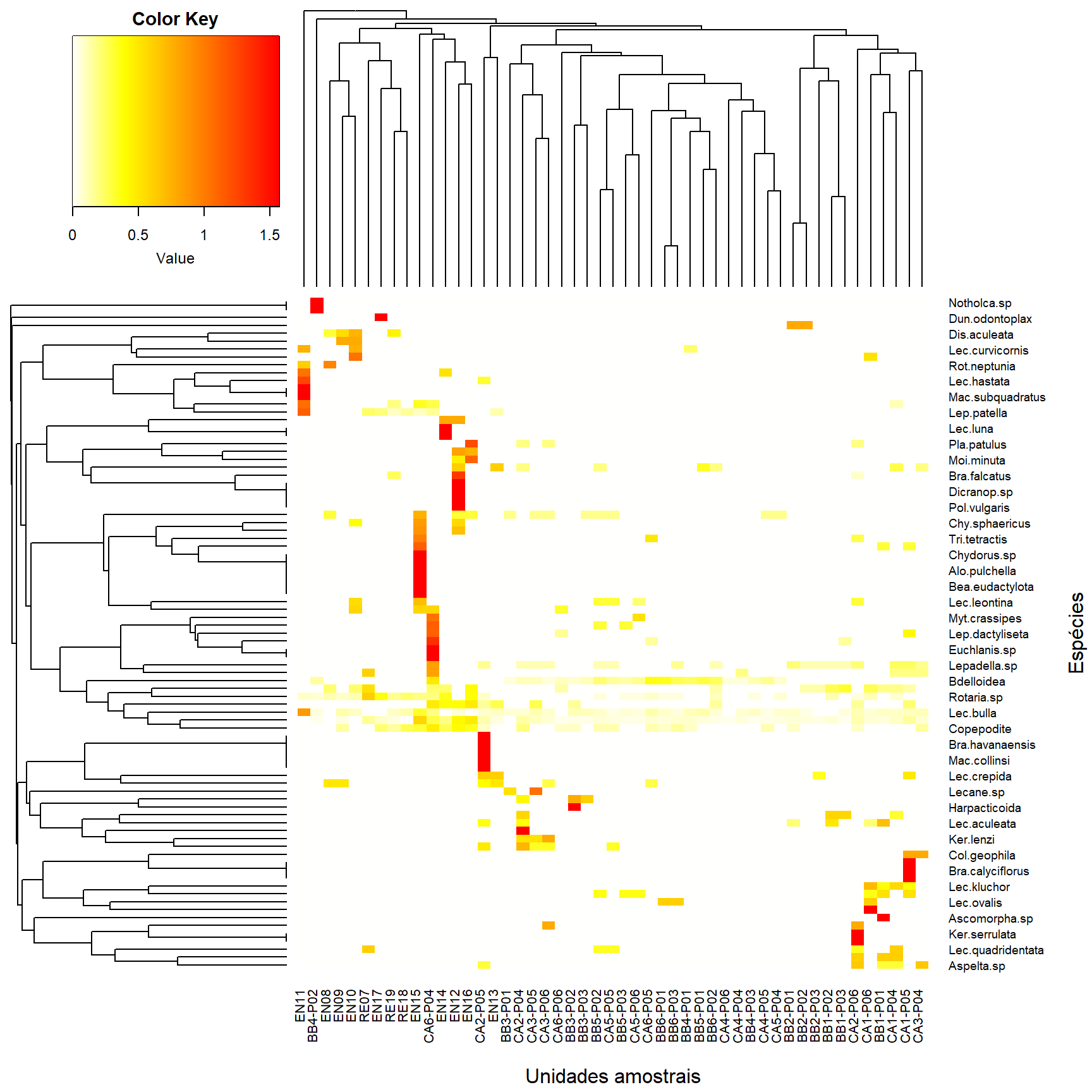
5.9.1 Percentual de encadeamento (% chaining)
Dendrogram Chaining (ou encadeamento) é um termo usado em análises de cluster hierárquico para se referir ao processo de adição sequencial de pequenos grupos a um ou poucos grandes grupos. É possível usar a porcentagem de encadeamento para ajudar a avaliar a qualidade do dendrograma. Análises altamente encadeadas provavelmente não valem a pena serem exploradas em maiores detalhes, provavelmente há alguma falha nos passos anteriores da análise. Por outro lado, pequenas diferenças na porcentagem de encadeamento na extremidade inferior da escala são geralmente um critério pobre para selecionar um algoritmo de ligação em relação a outro (McCune and Grace 2002).
O método para calcular o grau de encadeamento compara o comprimento médio do caminho do dendrograma com o comprimento mínimo possível (sem encadeamento) e o comprimento máximo possível (encadeamento completo). O comprimento do caminho para cada item no dendrograma é o número de nós que o conectam ao nível mais alto do dendrograma. O encadeamento completo ocorre quando cada fusão envolve a adição de um único item a um único grupo, cada vez mais grande (McCune and Grace 2002).
Com esse código podemos calcular o percentual de encadeamento para cada fusão em cluster1 e fazer um gráfico simples para observação.
# calculate the percent chaining for each merge
par(mfrow = c(1,1))
h <- cluster1$height
n <- length(h)
pc <- rep(NA, n-1)
for (i in 1:(n-1)) {
pc[i] <- abs(h[i] - h[i+1]) / h[1] * 100
}
# plot the percent chaining
plot(pc, type = "h", xlab = "Merge", ylab = "Percent chaining", main = "cluster1$height")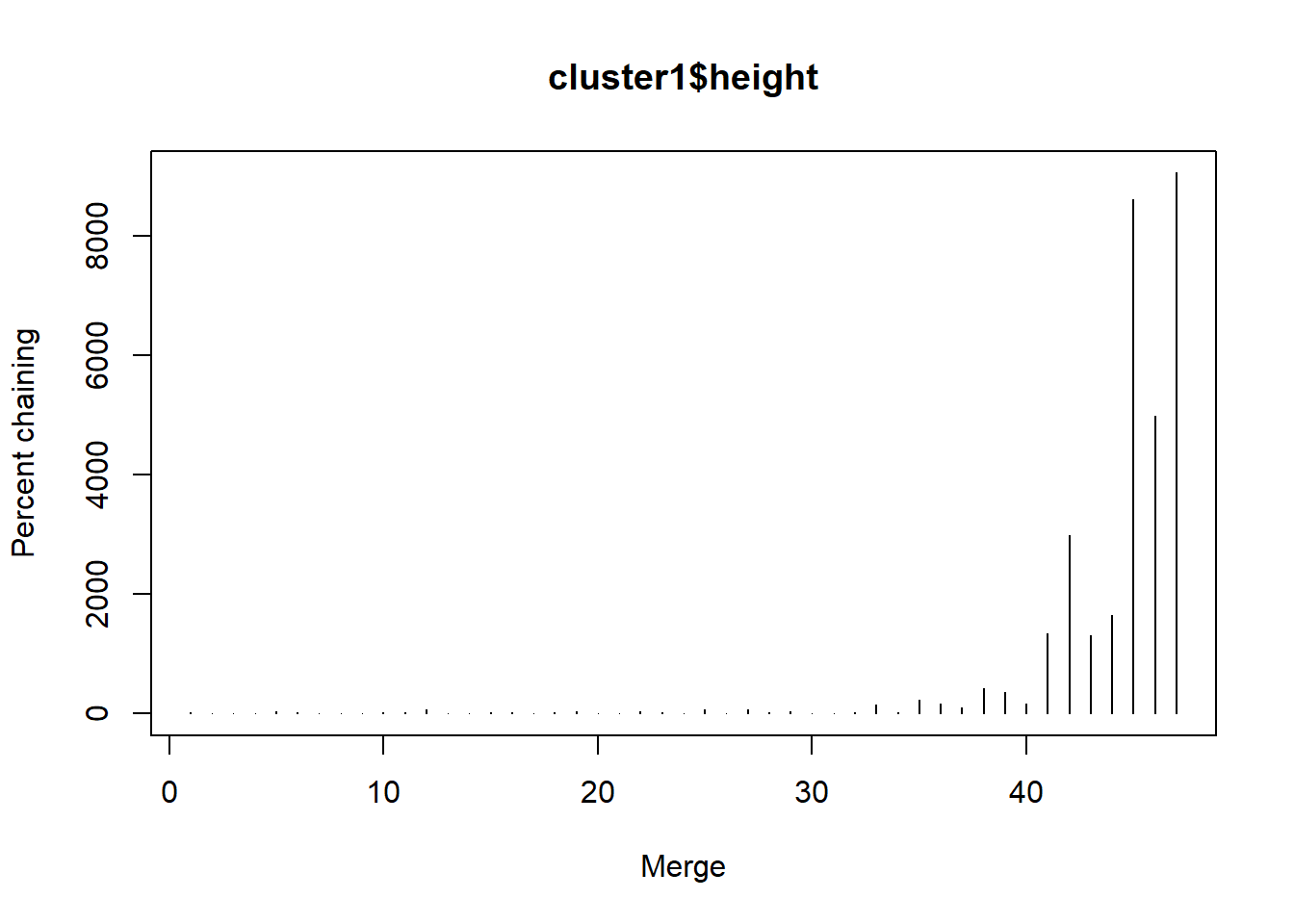
Para calcular o percentual geral de encadeamento em um dendrograma, é necessário calcular a diferença na altura entre cada nó e seus nós filhos e, em seguida, somar essas diferenças. Essa soma representa o comprimento total do dendrograma. Em seguida, é necessário calcular a soma das diferenças na altura entre cada nó que representa uma fusão e a altura de seus dois nós filhos. Essa soma representa o encadeamento total no dendrograma.
Finalmente, você pode calcular o percentual geral de encadeamento como o encadeamento total dividido pelo comprimento total do dendrograma, multiplicado por 100.
O eixo Y do gráfico mostra a percentagem de encadeamento, que é a diferença na altura entre fusões sucessivas dividida pela altura total do dendrograma, multiplicada por 100. É possível que a percentagem de encadeamento seja superior a 100 se a altura das duas fusões comparadas for maior que a altura total do dendrograma. Nesse caso, a percentagem de encadeamento é truncada em 100.
Isso pode acontecer, por exemplo, se a altura de uma fusão for negativa, o que pode ocorrer se a distância entre dois grupos for menor que zero, ou se o dendrograma for escalado ou truncado de alguma forma.
5.9.2 Função loop para comparar os encadeamentos
#create a list of the four cluster analyses
cluster_list <- list(cluster1, cluster2, cluster3, cluster4, cluster5)
par (mfrow = c (4,2)) #posicionamento dos gráficos
#loop through the list and calculate percent chaining for each
for (i in 1:5) {
h <- cluster_list[[i]]$height
n <- length(h)
pc <- rep(NA, n-1)
for (j in 1:(n-1)) {
pc[j] <- abs(h[j] - h[j+1]) / h[1] * 100
}
#plot the percent chaining for each cluster analysis
plot(pc, type = "h", xlab = "Merge", ylab = "Percent chaining", main = paste0("Cluster ", i))
}
## Análise de Classificação - K-means
system(paste("open", shQuote("D:/Elvio/OneDrive/MSS/_Zoo-Rebio/R_ZooRebio/kmeans.qmd")))