Capitulo 7 Apuntes de clase
7.1 21/02/20
Debemos instalar los siguientes paquetes: arules y devtools.
https://github.com/neuroimaginador/fcaR
## [1] "ts"## Time-Series [1:144] from 1949 to 1961: 112 118 132 129 121 135 148 148 136 119 ...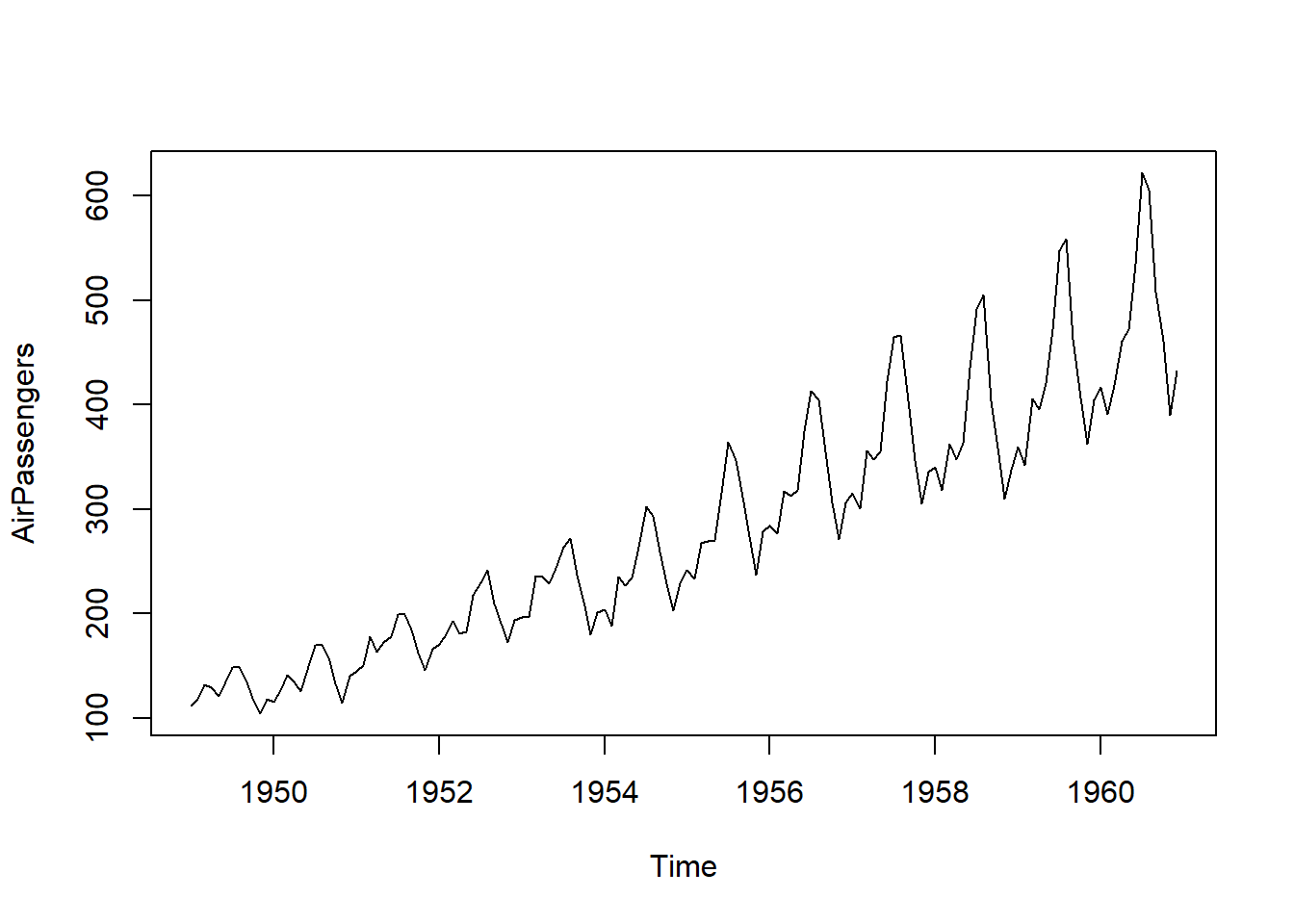
summary(AirPassengers) # La funcion summary devuelve el valor mínimo, los cuartiles, la media y el valor máximo de los datos## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 104.0 180.0 265.5 280.3 360.5 622.0Podemos usar source para ejecutar todo el script: source(“C1 21-02.R”).
Los comandos dir() y ls() nos permiten consultar los archivos o directorios que se encuentran disponibles en nuestro workspace.
## [1] 20Si desconocemos un dato o valor (NA) no podemos realizar operaciones que dependan de él:
## [1] 2.5## [1] NA## [1] 2El operador combine c() puede realizar la conversion de los parametros a un solo tipo, pero hay que tener cuidado a la hora de introducir los datos:
v1 <- c(1,3,5,6,"7") # El vector resultante será de tipo character, pues tiene mayor prioridad que el tipo numérico.
class(v1)## [1] "character"Podemos crear vectores con c() para realizar consultas en los indices que queramos:
## [1] 1 4 5 1v3 <- 1:10
v3 [-c(1, length(v3))] # Eliminamos el primer y ultimo elemento del vector en la consulta## [1] 2 3 4 5 6 7 8 9## [1] 3 6 9 12 157.2 04/03/20 Bookdown
Debemos instalar el paquete bookdown para poder crear nuestro propio book.
Una vez instalado, para crear nuestro book haremos: File -> New Project -> New Directory -> Book Project using Bookdown.
Podemos etiquetar secciones haciendo uso de {#etiqueta}.
Tambien tenemos que instalar Miktex, una distribucion de LateX para Windows. Tambien instalaremos la herramienta PANDOC que sera de utilidad.
Resolucion de ejercicios sobre Data Frames 3.1.3
7.3 06/03/20 DPLYR
Intalamos dplyr y readr, que están dentro de tidyverse.
Para usar las funciones del paquete podemos hacer: dplyr::filter, o podemos situarnos en la libreria del paquete: library(dplyr).
Este contenido es de importancia de cara a los examenes.
Existen 5 funciones clave en dplyr:
filter() # Escoger observaciones (filas) según sus valores
arrange() # Para reordenar las filas
select() # Para seleccionar variables segun sus nombres
mutate() # Crear nuevas variables como función de variables ya existentes
summarise() # Encontrar valores representativos de cada variableTodas funcionan de la misma manera:
- El primer argumento es un dataframe.
- Los demás argumentos describen qué hacer con el dataframe, usando los nombres de las variables (columnas) sin necesidad de utilizar comillas.
- El resultado es un nuevo dataframe.
A continuacion algunos ejemplos del uso del paquete dplyr:
7.3.1 Ejercicios de filter
## # A tibble: 6 x 13
## name height mass hair_color skin_color eye_color birth_year gender homeworld
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Chew~ 228 112 brown unknown blue 200 male Kashyyyk
## 2 Han ~ 180 80 brown fair brown 29 male Corellia
## 3 Jek ~ 180 110 brown fair blue NA male Bestine ~
## 4 Qui-~ 193 89 brown fair blue 92 male <NA>
## 5 Tarf~ 234 136 brown brown blue NA male Kashyyyk
## 6 Raym~ 188 79 brown light brown NA male Alderaan
## # ... with 4 more variables: species <chr>, films <list>, vehicles <list>,
## # starships <list>## # A tibble: 6 x 13
## name height mass hair_color skin_color eye_color birth_year gender homeworld
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 Chew~ 228 112 brown unknown blue 200 male Kashyyyk
## 2 Han ~ 180 80 brown fair brown 29 male Corellia
## 3 Jek ~ 180 110 brown fair blue NA male Bestine ~
## 4 Qui-~ 193 89 brown fair blue 92 male <NA>
## 5 Tarf~ 234 136 brown brown blue NA male Kashyyyk
## 6 Raym~ 188 79 brown light brown NA male Alderaan
## # ... with 4 more variables: species <chr>, films <list>, vehicles <list>,
## # starships <list>## # A tibble: 44 x 13
## name height mass hair_color skin_color eye_color birth_year gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Dart~ 202 136 none white yellow 41.9 male
## 2 Leia~ 150 49 brown light brown 19 female
## 3 Owen~ 178 120 brown, gr~ light blue 52 male
## 4 Beru~ 165 75 brown light blue 47 female
## 5 Bigg~ 183 84 black light brown 24 male
## 6 Anak~ 188 84 blond fair blue 41.9 male
## 7 Chew~ 228 112 brown unknown blue 200 male
## 8 Han ~ 180 80 brown fair brown 29 male
## 9 Jabb~ 175 1358 <NA> green-tan~ orange 600 herma~
## 10 Wedg~ 170 77 brown fair hazel 21 male
## # ... with 34 more rows, and 5 more variables: homeworld <chr>, species <chr>,
## # films <list>, vehicles <list>, starships <list>## # A tibble: 73 x 13
## name height mass hair_color skin_color eye_color birth_year gender
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
## 1 Luke~ 172 77 blond fair blue 19 male
## 2 C-3PO 167 75 <NA> gold yellow 112 <NA>
## 3 R2-D2 96 32 <NA> white, bl~ red 33 <NA>
## 4 Dart~ 202 136 none white yellow 41.9 male
## 5 Leia~ 150 49 brown light brown 19 female
## 6 Owen~ 178 120 brown, gr~ light blue 52 male
## 7 Beru~ 165 75 brown light blue 47 female
## 8 R5-D4 97 32 <NA> white, red red NA <NA>
## 9 Bigg~ 183 84 black light brown 24 male
## 10 Obi-~ 182 77 auburn, w~ fair blue-gray 57 male
## # ... with 63 more rows, and 5 more variables: homeworld <chr>, species <chr>,
## # films <list>, vehicles <list>, starships <list>7.3.2 Uso del operador %>%
Viene de los pipelines de linux, sirve para interconectar funciones entre sí. En el apartado de Visualizacion trabajaremos así.
Ejemplos del uso del operador %>%:
d1 <- starwars %>%
filter(!(mass > 78 | hair_color == "brown"))
# Si falla el comando %>%: library(magrittr)
# Ahora combinamos select
d1 <- starwars %>%
filter(!(mass > 78 | hair_color == "brown")) %>%
select(name, height, mass)
paged_table(d1)d1 <- starwars %>%
filter(!(mass > 78 | hair_color == "brown")) %>%
select(name:skin_color) # Seleccion desde la columna 'name' hasta la columna 'skin_color'
paged_table(d1)Para seleccionar columnas que empiecen de una forma determinada haremos uso de starts_with():
Ejemplos de mutate y transmute:
Ejemplos de uso de arrange():
dplyr devuelve un objeto de tipo tibble (filas x cols), que es un derivado de dataframes, por lo que hay que tener cuidado, puede haber funciones sobre data frames que no se apliquen o den resultados equívocos sobre tibble. Este formato aparte de mostrarte los datos, en cada columna encontramos el tipo de dato que almacena.
Resolucion de ejercicios sobre dplyr 3.2
7.4 11/03/20 Familia Apply
Programacion funcional - Apply.
Veremos en profundidad lapply y sapply para listas.
Instalar el paquete profvis para consultar el rendimiento de nuestro codigo. A continuacion un ejemplo de como usar profvis:
Ejemplos de apply, lapply y sapply
## [,1] [,2] [,3]
## [1,] 1 4 7
## [2,] 2 5 8
## [3,] 3 6 9## [1] 6 7 8# Normalmente encontraremos el uso de funciones anonimas.
r2 <- apply(m1, 1, function(x){mean(x)+2})
r2## [1] 6 7 8# ------ lapply() y sapply() -------
rating <- 1:10
# Va realizando la media de cada numero,
# y los va devolviendo en una lista
result <- lapply(rating, mean)
result## [[1]]
## [1] 1
##
## [[2]]
## [1] 2
##
## [[3]]
## [1] 3
##
## [[4]]
## [1] 4
##
## [[5]]
## [1] 5
##
## [[6]]
## [1] 6
##
## [[7]]
## [1] 7
##
## [[8]]
## [1] 8
##
## [[9]]
## [1] 9
##
## [[10]]
## [1] 10## [1] "list"# Util cuando tengamos listas de listas
calificaciones <- list(as1 = c(3,7,9,10), as2 = c(4,7,8,6))
result <- lapply(calificaciones, mean)
result## $as1
## [1] 7.25
##
## $as2
## [1] 6.25## [1] "list"# La diferencia entre sapply y lapply es lo que devuelve
result <- sapply(calificaciones, mean)
result## as1 as2
## 7.25 6.25## [1] "numeric"7.5 18/03/20 Introduccion a la visualizacion
Filosofia para realización de graficos: http://motioninsocial.com/tufte/
Enlace para observar graficas de R sobre el COVID 19 https://elpais.com/sociedad/2020/03/17/actualidad/1584436648_230452.html
7.5.1 Visualizacion
Buscar ‘Boxplot lenguaje R’, sirve para mostrar distriucion de valores, así como la media y valores anómalos. Tambien podemos realizar directamente summary().
Como analizar los valores: repetidos, como se comportan, si se distribuyen con una normal - EXAMEN
Funcion curve() para poder representar funciones matematicas.
R por defecto trae el paquete plot para representaciones graficas, pero nosotros usaremos el paquete ggplot2. A continuacion un ejemplo de ggplot:
library(gcookbook)
library(ggplot2)
g1 <- ggplot(BOD, aes(x=Time, y=demand)) + geom_point(aes(color = demand))
g1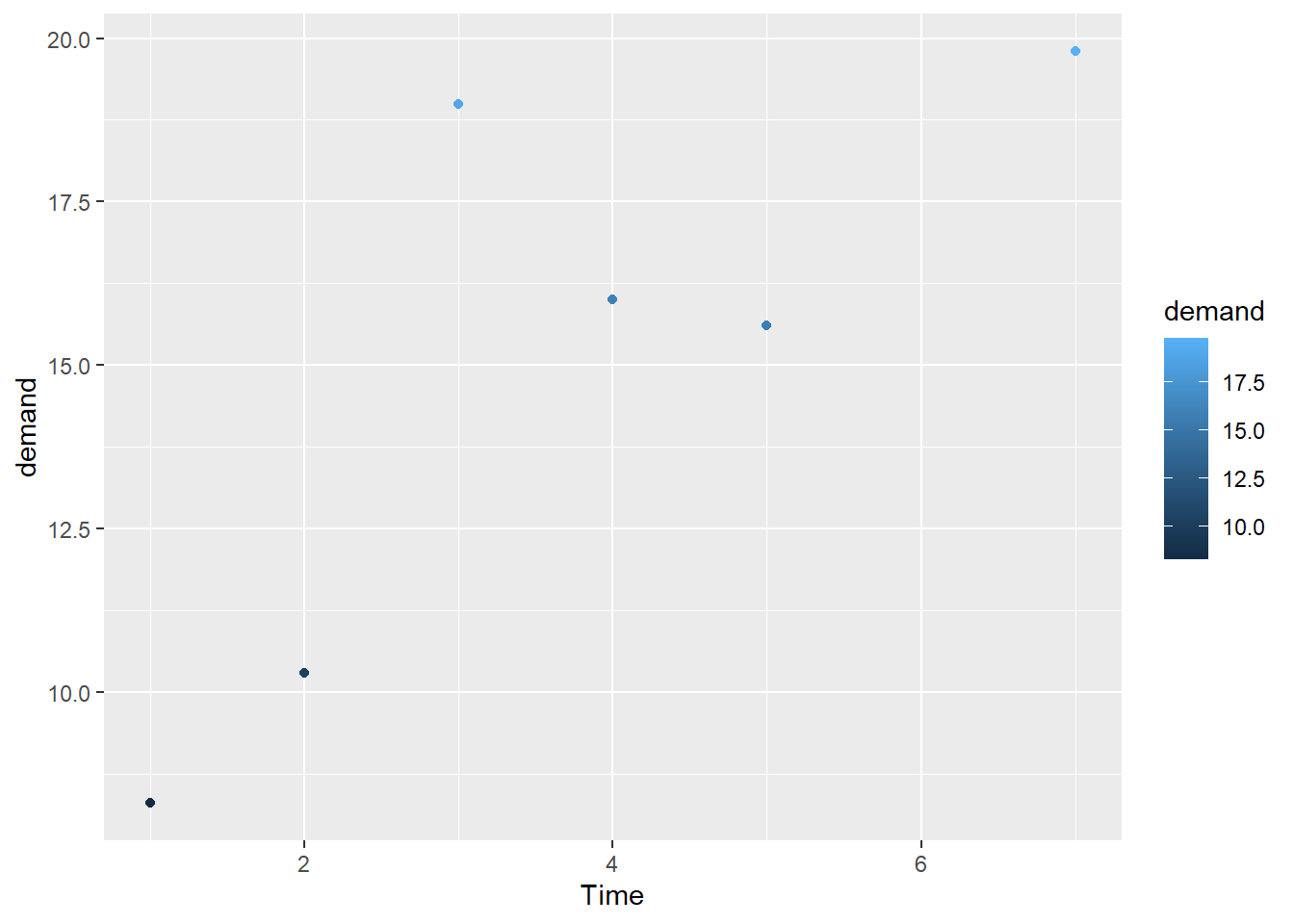
Con la funcion aes() defines lo que puedes ver, es decir, lo que va en el eje X, el eje Y, la forma, el tamaño, el color…
library(magrittr)
library(dplyr)
landdata_states <- read.csv("landdata-states.csv")
hp2001Q1 <- landdata_states %>%
filter(Date == 2001.25)
p1 <- ggplot(hp2001Q1, aes(x = log(Land.Value), y = Structure.Cost))
p1 + geom_point(aes(color=Home.Value)) + geom_smooth()## `geom_smooth()` using method = 'loess' and formula 'y ~ x'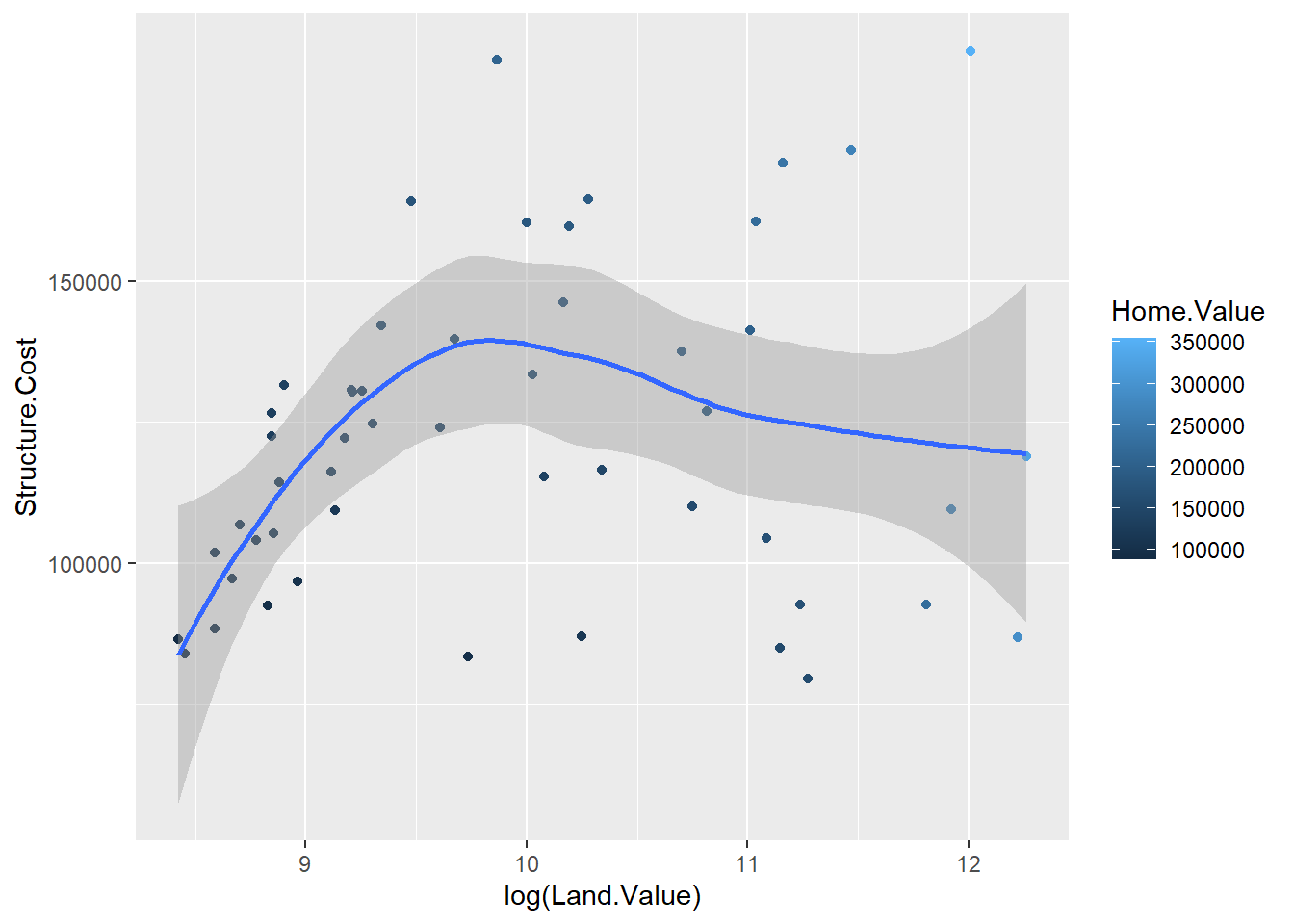
## Warning: Removed 1 rows containing missing values (geom_point).
log sirve para linealizar los valores.
Importante: Hay ciertos problemas con los graficos de barra si la variable no es continua, por ejemplo, una vairable de numeros reales, porque podria haber infinitos valores, por lo que hay que convertir dichas variables en Factor, y luego usar stat=“identity” dentro de geom_bar: x=factor(Time) y geom_bar(stat=“identity”). Transparencia 22.
Aplicacion shiny con R detras: https://scitilab.shinyapps.io/Covid19/
aggregate permite la agregacion de una variable respecto de otra aplicando una funcion determinada.
Ejercicios de Visualizacion: 3.4
7.6 20/03/20 Reglas de Asociacion
En el paquete arules tenemos dos algoritmos sobre conjuntos frecuentes: Apriori y e-clat.
Minimizar los accesos a disco es fundamental en data mining.
Para visualizar reglas tenemos que ejecutar inspect(mis_reglas), no podemos usar View.
Ejemplo de uso de apriori:
# El paquete arules genera un objeto de tipo rules
reglas <- apriori(Groceries, paramete = list(sup = 0.0001, conf= 0.5))7.6.1 Ejercicio voluntario en grupo
Currarse el trabajo de kaggle, realizar analisis de los rmarkdown existentes para profundizar sobre visualizacion de datos, ir añadiendo explicacion a los rmds y probar a variar algunas representaciones.
Analisis de datos -> fichero malaga_covid19.MD y .HTML
7.7 06/05/20
Implicacion es una regla de asociacion con confianza 1.
a1a3 -> a4 siempre que tenga a1 y a3 se da a4.
¿¿a1,a4 –> a5, a5 –> a3?? Podemos deducir a1,a4 –> a3
Simpificacion a1a3 –> (a2a3 - a1a3)= a1a3 –> a2, es decir, eliminar los elementos de la derecha que existan en la izq.
La barrita \(\backslash\) se corresponde con la diferencia de conjuntos: \(A \backslash B\)
Apriori solo saca un atributo a la derecha
En fcaR, podemos hacer uso de $assign(“atributo” = 1/0) y aplicarle el cierre.
En el paquete, sobre el manejo de implicaciones, se ha imitado el comportamiento de arules
Ejercicios sobre simplification logic 4.2.3
7.8 08/05/20
## Parsed with column specification:
## cols(
## State = col_character(),
## Lat = col_double(),
## Mort = col_double(),
## Ocean = col_double(),
## Long = col_double()
## )
# attach, sirve para no tener que usar el data$variable, y usar la variable directamente
# Intentar relacionar la latitud con la mortalidad
# Colocamos la mortalidad dependiente de la latitud
f1 <- lm(Mort ~ Lat, data = skincancer)
f1##
## Call:
## lm(formula = Mort ~ Lat, data = skincancer)
##
## Coefficients:
## (Intercept) Lat
## 389.189 -5.978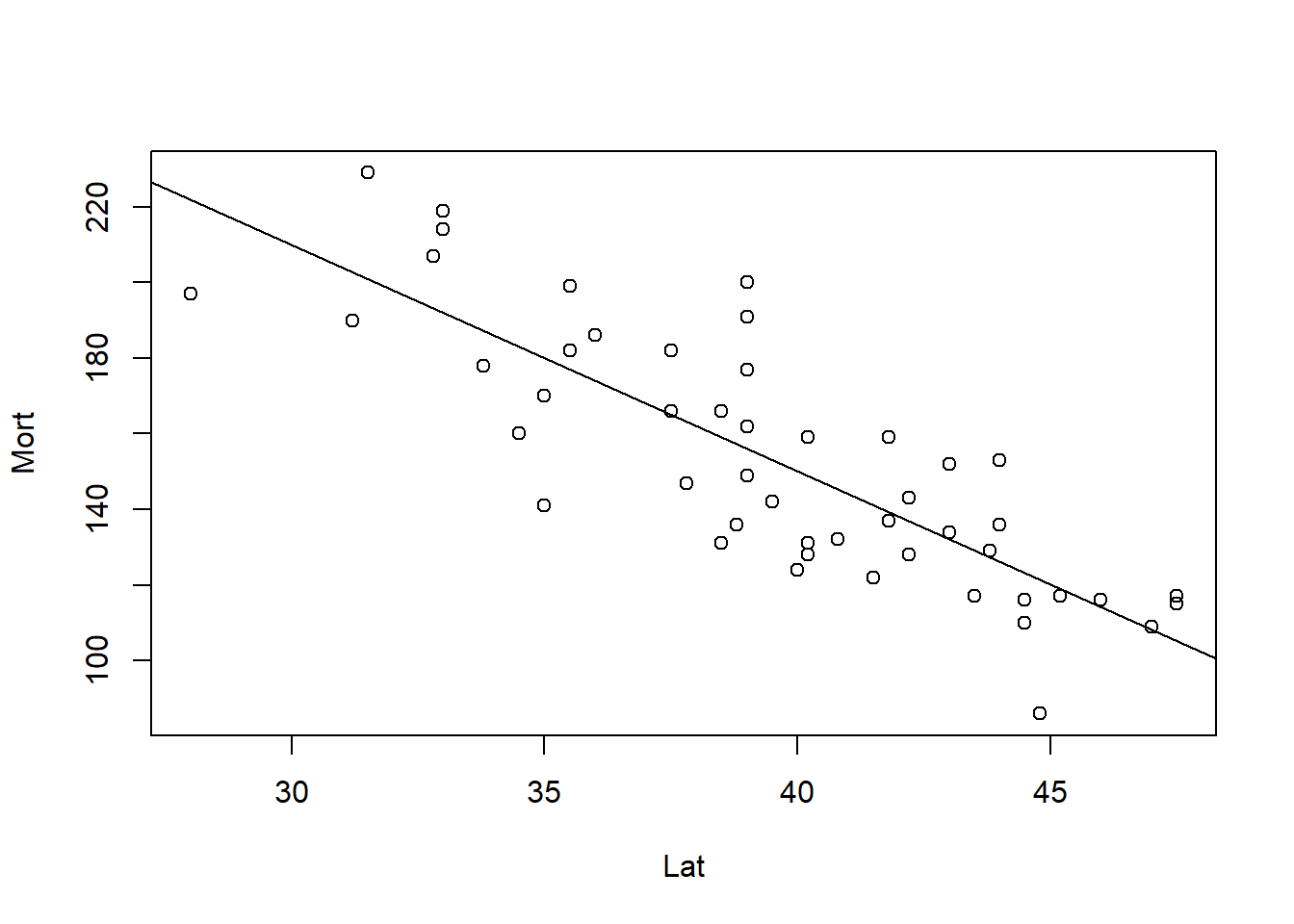
##
## Call:
## lm(formula = Mort ~ Lat + Long, data = skincancer)
##
## Coefficients:
## (Intercept) Lat Long
## 400.6755 -5.9308 -0.1467
7.9 20/05/20
Este dia se realizo un ejercicio completo de repaso de Regresion 4.3.2
7.10 22/05/20
Un ejemplo en el que debemos usar modelos diferentes segun los datos que poseamos. No siempre se puede usar el mismo modelo con los datos que se nos proporcionen.
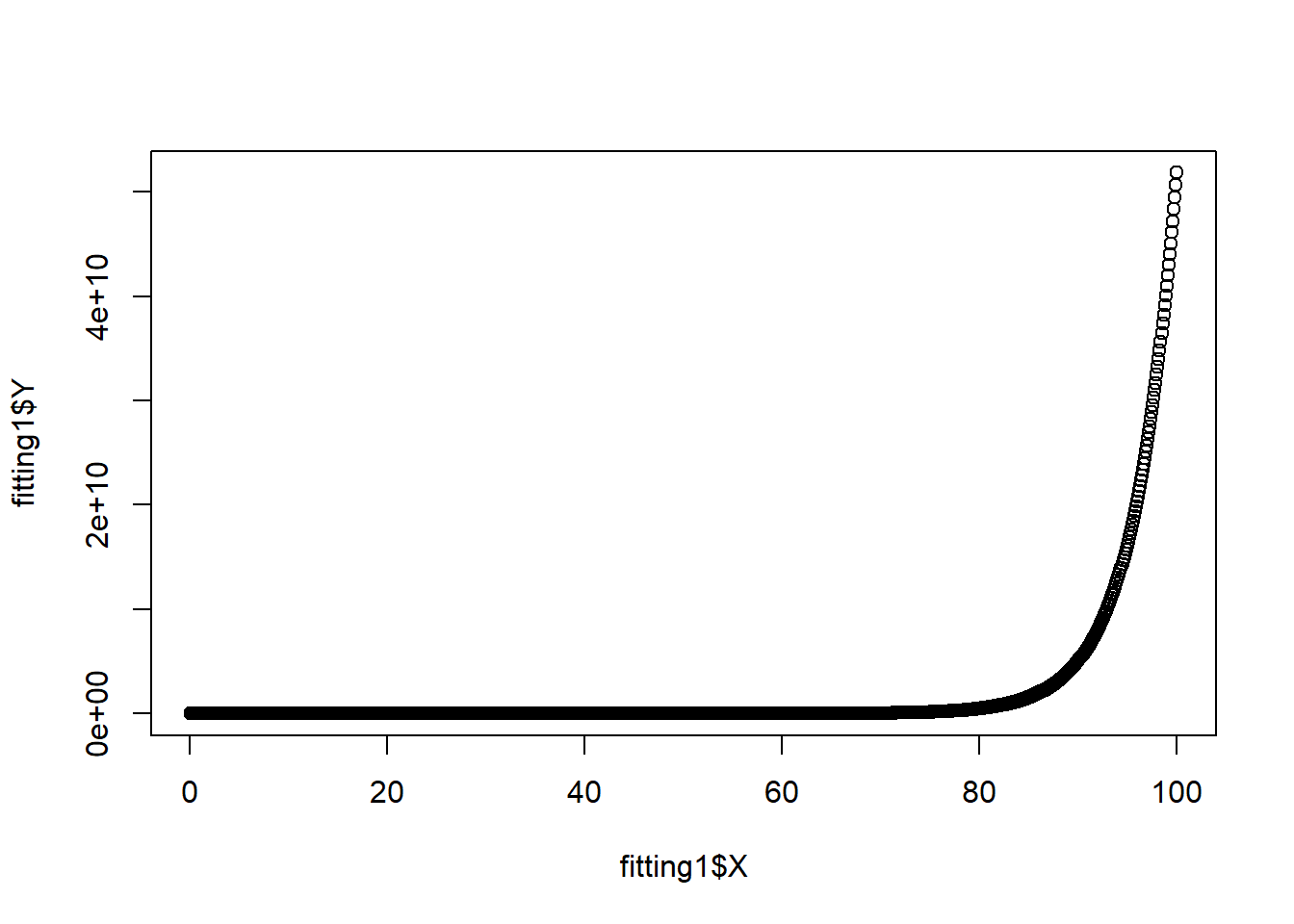
# El problema de este dataset esque el valor Y son exponenciales por lo que tendriamos que hacer es probar con diferentes modelos al lineal.
f1 <- lm(Y~X, data = fitting1)
f1##
## Call:
## lm(formula = Y ~ X, data = fitting1)
##
## Coefficients:
## (Intercept) X
## -3.913e+09 1.229e+08##
## Call:
## lm(formula = Y ~ X, data = fitting1)
##
## Residuals:
## Min 1Q Median 3Q Max
## -5.444e+09 -3.814e+09 -1.109e+09 1.740e+09 4.344e+10
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -3.913e+09 4.062e+08 -9.631 <2e-16 ***
## X 1.229e+08 7.031e+06 17.485 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 6.418e+09 on 998 degrees of freedom
## Multiple R-squared: 0.2345, Adjusted R-squared: 0.2337
## F-statistic: 305.7 on 1 and 998 DF, p-value: < 2.2e-16##
## Call:
## lm(formula = log(Y) ~ X, data = fitting1)
##
## Coefficients:
## (Intercept) X
## 1.271 0.234## Warning in summary.lm(f1a): essentially perfect fit: summary may be unreliable##
## Call:
## lm(formula = log(Y) ~ X, data = fitting1)
##
## Residuals:
## Min 1Q Median 3Q Max
## -1.857e-13 -4.970e-16 4.320e-16 1.168e-15 3.683e-15
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.271e+00 4.660e-16 2.728e+15 <2e-16 ***
## X 2.340e-01 8.065e-18 2.901e+16 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 7.362e-15 on 998 degrees of freedom
## Multiple R-squared: 1, Adjusted R-squared: 1
## F-statistic: 8.418e+32 on 1 and 998 DF, p-value: < 2.2e-16# Si nos fijamos el R cuadrado nos sale 1, es un ajuste perfecto
# Recta perfecta
plot(fitting1$X, log(fitting1$Y))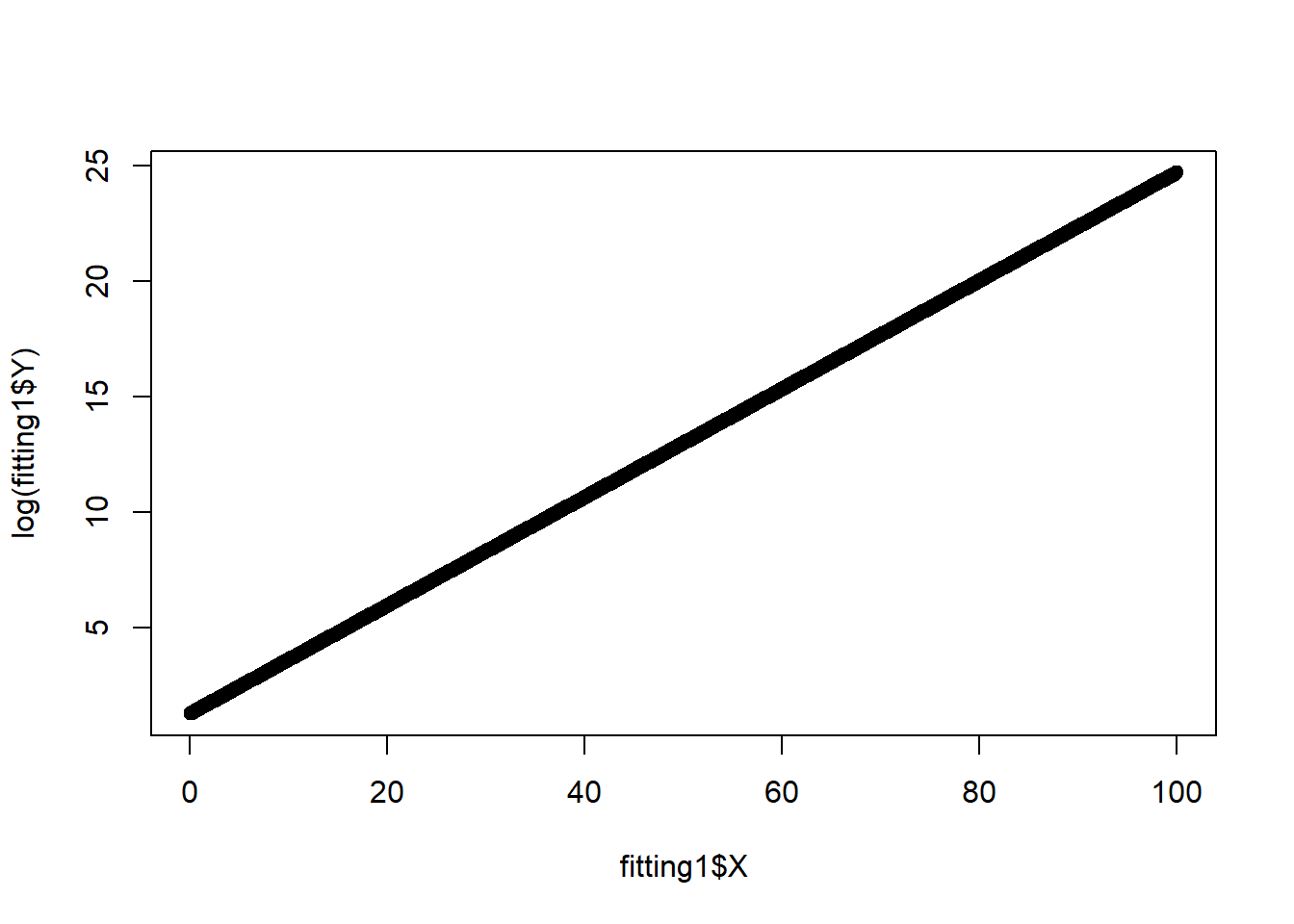
Text mining
Todo el trabajo de text mining se realizará en RStudio Cloud con el fin de evitar problemas de compatibilidad.
Preparar un proyecto, elegir algun texto en el cual realizar la tarea propuesta
gsup –> para transformaciones individuales
stemming –> hace uso de las raices de las palabras, para evitar tener que hacer comprobaciones para cada palabra
En el material del campus tenemos resoluciones a multiples problemas que nos pueden aparecer a la hora de realizar text mining.