Capitulo 3 Lenguaje R
3.1 Ejercicios de Introduccion a R
3.1.1 Ejercicios Introduccion - Vectores I
- Importar el dataset UKgas. Almacenar la columna del dataset en un vector llamado consumo.de.gas. Preparar un fichero .R con las órdenes necesarias para realizar:
- Acceder a los 10 primeros elementos del vector consumo.de.gas
## [1] 160.1 129.7 84.8 120.1 160.1 124.9 84.8 116.9 169.7 140.9- Acceder a los elementos impares del vector consumo.de.gas
## [1] 160.1 84.8 160.1 84.8 169.7 89.7 187.3 92.9 176.1 89.7
## [11] 185.7 99.3 200.1 102.5 204.9 112.1 227.3 115.3 244.9 118.5
## [21] 244.9 188.9 301.0 136.1 317.0 152.1 371.4 158.5 449.9 179.3
## [31] 491.5 177.7 593.9 176.1 584.3 187.3 669.2 216.1 827.7 209.7
## [41] 840.5 217.7 848.5 209.7 925.3 214.5 917.3 224.1 989.4 233.7
## [51] 1087.0 281.8 1163.9 347.4- Acceder a las posiciones 1,4,7,10,… del vector consumo.de.gas
## [1] 160.1 120.1 84.8 140.9 187.3 120.1 89.7 155.3 200.1 136.1 112.1 195.3
## [13] 244.9 153.7 188.9 196.9 317.0 336.2 158.5 286.6 491.5 409.8 176.1 395.4
## [25] 669.2 509.1 209.7 414.6 848.5 701.2 214.5 515.5 989.4 730.0 281.8 613.1- Acceder al vector consumo.de.gas en orden inverso
## [1] 782.8 347.4 613.1 1163.9 787.6 281.8 534.7 1087.0 730.0 233.7
## [11] 477.1 989.4 694.8 224.1 515.5 917.3 683.6 214.5 443.4 925.3
## [21] 701.2 209.7 437.0 848.5 670.8 217.7 414.6 840.5 542.7 209.7
## [31] 467.5 827.7 509.1 216.1 421.0 669.2 485.1 187.3 395.4 584.3
## [41] 483.5 176.1 329.8 593.9 409.8 177.7 321.8 491.5 403.4 179.3
## [51] 286.6 449.9 355.4 158.5 240.1 371.4 336.2 152.1 230.5 317.0
## [61] 267.3 136.1 196.9 301.0 142.5 188.9 216.1 244.9 153.7 118.5
## [71] 214.5 244.9 142.5 115.3 195.3 227.3 140.9 112.1 176.1 204.9
## [81] 136.1 102.5 161.7 200.1 131.3 99.3 155.3 185.7 123.3 89.7
## [91] 147.3 176.1 120.1 92.9 144.1 187.3 123.3 89.7 140.9 169.7
## [101] 116.9 84.8 124.9 160.1 120.1 84.8 129.7 160.1- Acceder a los 50 primeros elementos del vector consumo.de.gas excepto la posición 1, 3 y 5.
## [1] 129.7 120.1 124.9 84.8 116.9 169.7 140.9 89.7 123.3 187.3 144.1 92.9
## [13] 120.1 176.1 147.3 89.7 123.3 185.7 155.3 99.3 131.3 200.1 161.7 102.5
## [25] 136.1 204.9 176.1 112.1 140.9 227.3 195.3 115.3 142.5 244.9 214.5 118.5
## [37] 153.7 244.9 216.1 188.9 142.5 301.0 196.9 136.1 267.3 317.0 230.5- Sumarle 1 a los elementos del vector consumo.de.gas (reciclaje)
## [1] 161.1 130.7 85.8 121.1 161.1 125.9 85.8 117.9 170.7 141.9
## [11] 90.7 124.3 188.3 145.1 93.9 121.1 177.1 148.3 90.7 124.3
## [21] 186.7 156.3 100.3 132.3 201.1 162.7 103.5 137.1 205.9 177.1
## [31] 113.1 141.9 228.3 196.3 116.3 143.5 245.9 215.5 119.5 154.7
## [41] 245.9 217.1 189.9 143.5 302.0 197.9 137.1 268.3 318.0 231.5
## [51] 153.1 337.2 372.4 241.1 159.5 356.4 450.9 287.6 180.3 404.4
## [61] 492.5 322.8 178.7 410.8 594.9 330.8 177.1 484.5 585.3 396.4
## [71] 188.3 486.1 670.2 422.0 217.1 510.1 828.7 468.5 210.7 543.7
## [81] 841.5 415.6 218.7 671.8 849.5 438.0 210.7 702.2 926.3 444.4
## [91] 215.5 684.6 918.3 516.5 225.1 695.8 990.4 478.1 234.7 731.0
## [101] 1088.0 535.7 282.8 788.6 1164.9 614.1 348.4 783.8- Sumar los 10 primeros elementos del vector consumo.de.gas con el vector v2=(1,2,3)
# Como son tamaños diferentes, cogeré un vector multiplo del vector v2 y luego seleccionare los 10 elementos requeridos
v2 <- c(1,2,3)
v7 <- consumo.de.gas[1:12]
v7 <- v7+v2
v7 <- v7[1:10]
v7## [1] 161.1 131.7 87.8 121.1 162.1 127.9 85.8 118.9 172.7 141.93.1.2 Ejercicios Funciones
- Programar una función que devuelva dado un vector cuantos números impares tiene. AYUDA: Usar %% para calcular el resto.
n_impares <- function(vector){
sum <- length(which(vector%%2 != 0))
return(sum)
}
v1 <- 1:10
n_impares(v1)## [1] 5- Programar una función que calcule el volumen de una esfera dado el radio r de dicha esfera. La ecuacion para hallar el volumen de una esfera es \(V = \frac{4}{3}\pi r^3\)
## [1] 33.51032- Programar una función que calcule el volumen de esferas con radio r =1,2,…,20 y devuelva un data.frame con las dos columnas (radio, volumen).
volumenes <- function(vector){
vols <- vol_esfera(vector)
return(data.frame("radio"=vector, "volumen"=vols))
}
v <- 1:20
paged_table(volumenes(v))3.1.3 Manipular Data Frames - Ejercicio 2.1
Importar el dataset HairEyeColor y preparar un fichero .R con los comandos requeridos para lo siguiente:
- Si el dataset importado no es un dataframe convertirlo a dataframe
## [1] "data.frame"- Analizar la estructura del dataset, tipo de datos, estructura, clase, etc.
## 'data.frame': 32 obs. of 4 variables:
## $ Hair: Factor w/ 4 levels "Black","Brown",..: 1 2 3 4 1 2 3 4 1 2 ...
## $ Eye : Factor w/ 4 levels "Brown","Blue",..: 1 1 1 1 2 2 2 2 3 3 ...
## $ Sex : Factor w/ 2 levels "Male","Female": 1 1 1 1 1 1 1 1 1 1 ...
## $ Freq: num 32 53 10 3 11 50 10 30 10 25 ...- Obtener número de columnas, número de filas. Guardar en un vector el nombre de las columnas.
## [1] 32 4## [1] 32## [1] 4- Cambiar el nombre de la primera columna.
- Averiguar el tipo de datos de la primera columna.
## [1] "factor"- Comando para averiguar si hay valores NA en todo el dataframe, y lo mismo en la columna 1.
## [1] FALSE## [1] FALSE- Practicar con el dataset para extraer un conjunto de filas y/o columnas.
- Guardar cinco primeras filas en una variable d1 y las cinco últimas en una variable d2 y combinarlas en un nuevo dataframe llamado d12.
## Hair Eye Sex Freq
## 1 Black Brown Male 32
## 2 Brown Brown Male 53
## 3 Red Brown Male 10
## 4 Blond Brown Male 3
## 5 Black Blue Male 11## Hair Eye Sex Freq
## 28 Blond Hazel Female 5
## 29 Black Green Female 2
## 30 Brown Green Female 14
## 31 Red Green Female 7
## 32 Blond Green Female 8## Hair Eye Sex Freq
## 1 Black Brown Male 32
## 2 Brown Brown Male 53
## 3 Red Brown Male 10
## 4 Blond Brown Male 3
## 5 Black Blue Male 11
## 28 Blond Hazel Female 5
## 29 Black Green Female 2
## 30 Brown Green Female 14
## 31 Red Green Female 7
## 32 Blond Green Female 8- Utilizar el comando subset para extraer, eliminar, etc. un conjunto de columnas de un data set.
- Usar subset para guardar en una variable las filas de dataset con valor para eye de Blue.
## Hair Eye Sex Freq
## 5 Black Blue Male 11
## 6 Brown Blue Male 50
## 7 Red Blue Male 10
## 8 Blond Blue Male 30
## 21 Black Blue Female 9
## 22 Brown Blue Female 34
## 23 Red Blue Female 7
## 24 Blond Blue Female 64- Guardar la primera columna en variable d y utilizar los comandos unique, table. ¿Para qué sirven estos comandos?.
## [1] Black Brown Red Blond Black Brown Red Blond Black Brown Red Blond
## [13] Black Brown Red Blond Black Brown Red Blond Black Brown Red Blond
## [25] Black Brown Red Blond Black Brown Red Blond
## Levels: Black Brown Red Blond## [1] Black Brown Red Blond
## Levels: Black Brown Red Blondtable(d) # 'table' utiliza los factores de clasificación cruzada para construir una tabla de contingencia de los recuentos en cada combinación de niveles de factores.## d
## Black Brown Red Blond
## 8 8 8 8- Con la variable d usar colSums, colMeans. Explicar con ejemplos para qué sirven estos comandos.
- Explicar con ejemplos para qué sirven los comandos rowSums, rowMeans.
# No podemos usar esos comandos con la variable d, pues se trata de una estructura unidimensional
# Usare un data frame compuesto por los valores 1:10 para probar las funciones
df <- data.frame(matrix(1:9,3,3))
df## X1 X2 X3
## 1 1 4 7
## 2 2 5 8
## 3 3 6 9## X1 X2 X3
## 6 15 24## X1 X2 X3
## 2 5 8## [1] 12 15 18## [1] 4 5 63.1.4 Errores comunes - Ejercicio 2.2
- Explica por qué los siguientes comandos dan error o no hacen lo que podría ser previsible con la lectura del comando. Arreglalo.
# usa mtcars de paquete datasets
library(datasets)
data("mtcars")
mtcars[mtcars$cyl = 4, ] # La comparacion se realiza con ==
mtcars[-1:4, ] # Los indices no estan seleccionados correctamente
mtcars[mtcars$cyl <= 5] # No se han especificado las columnas
mtcars[mtcars$cyl == 4 | 6, ] # Se evalua primero 4|6 por lo que siempre será true (suma lógica) y selecciona todos los valores
mtcars[1:20] # No se han especificado las columnas
# Errores??
x <- 1:5; x[NA] # Esta transformando todos los valores en NAA continuacion se muestra la correccion de los errores
## mpg cyl disp hp drat wt qsec vs am gear carb
## Datsun 710 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1
## Merc 240D 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
## Merc 230 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
## Fiat 128 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
## Honda Civic 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
## Toyota Corolla 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1
## Toyota Corona 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1
## Fiat X1-9 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1
## Porsche 914-2 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2
## Lotus Europa 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2
## Volvo 142E 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2## mpg cyl disp hp drat wt qsec vs am gear carb
## Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
## Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
## Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
## Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1## mpg cyl disp hp drat wt qsec vs am gear carb
## Hornet Sportabout 18.7 8 360.0 175 3.15 3.440 17.02 0 0 3 2
## Valiant 18.1 6 225.0 105 2.76 3.460 20.22 1 0 3 1
## Duster 360 14.3 8 360.0 245 3.21 3.570 15.84 0 0 3 4
## Merc 240D 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
## Merc 230 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
## Merc 280 19.2 6 167.6 123 3.92 3.440 18.30 1 0 4 4
## Merc 280C 17.8 6 167.6 123 3.92 3.440 18.90 1 0 4 4
## Merc 450SE 16.4 8 275.8 180 3.07 4.070 17.40 0 0 3 3
## Merc 450SL 17.3 8 275.8 180 3.07 3.730 17.60 0 0 3 3
## Merc 450SLC 15.2 8 275.8 180 3.07 3.780 18.00 0 0 3 3
## Cadillac Fleetwood 10.4 8 472.0 205 2.93 5.250 17.98 0 0 3 4
## Lincoln Continental 10.4 8 460.0 215 3.00 5.424 17.82 0 0 3 4
## Chrysler Imperial 14.7 8 440.0 230 3.23 5.345 17.42 0 0 3 4
## Fiat 128 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
## Honda Civic 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
## Toyota Corolla 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1
## Toyota Corona 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1
## Dodge Challenger 15.5 8 318.0 150 2.76 3.520 16.87 0 0 3 2
## AMC Javelin 15.2 8 304.0 150 3.15 3.435 17.30 0 0 3 2
## Camaro Z28 13.3 8 350.0 245 3.73 3.840 15.41 0 0 3 4
## Pontiac Firebird 19.2 8 400.0 175 3.08 3.845 17.05 0 0 3 2
## Fiat X1-9 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1
## Porsche 914-2 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2
## Lotus Europa 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2
## Ford Pantera L 15.8 8 351.0 264 4.22 3.170 14.50 0 1 5 4
## Ferrari Dino 19.7 6 145.0 175 3.62 2.770 15.50 0 1 5 6
## Maserati Bora 15.0 8 301.0 335 3.54 3.570 14.60 0 1 5 8
## Volvo 142E 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2## mpg cyl disp hp drat wt qsec vs am gear carb
## Datsun 710 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1
## Merc 240D 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
## Merc 230 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
## Fiat 128 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
## Honda Civic 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
## Toyota Corolla 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1
## Toyota Corona 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1
## Fiat X1-9 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1
## Porsche 914-2 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2
## Lotus Europa 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2
## Volvo 142E 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2mtcars[mtcars$cyl == 4 | mtcars$cyl == 6, ] # Separamos las comparaciones y luego realizamos la operacion OR## mpg cyl disp hp drat wt qsec vs am gear carb
## Mazda RX4 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4
## Mazda RX4 Wag 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4
## Datsun 710 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1
## Hornet 4 Drive 21.4 6 258.0 110 3.08 3.215 19.44 1 0 3 1
## Valiant 18.1 6 225.0 105 2.76 3.460 20.22 1 0 3 1
## Merc 240D 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
## Merc 230 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
## Merc 280 19.2 6 167.6 123 3.92 3.440 18.30 1 0 4 4
## Merc 280C 17.8 6 167.6 123 3.92 3.440 18.90 1 0 4 4
## Fiat 128 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
## Honda Civic 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
## Toyota Corolla 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1
## Toyota Corona 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1
## Fiat X1-9 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1
## Porsche 914-2 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2
## Lotus Europa 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2
## Ferrari Dino 19.7 6 145.0 175 3.62 2.770 15.50 0 1 5 6
## Volvo 142E 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2## mpg cyl disp hp drat wt qsec vs am gear carb
## Mazda RX4 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4
## Mazda RX4 Wag 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4
## Datsun 710 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1
## Hornet 4 Drive 21.4 6 258.0 110 3.08 3.215 19.44 1 0 3 1
## Hornet Sportabout 18.7 8 360.0 175 3.15 3.440 17.02 0 0 3 2
## Valiant 18.1 6 225.0 105 2.76 3.460 20.22 1 0 3 1
## Duster 360 14.3 8 360.0 245 3.21 3.570 15.84 0 0 3 4
## Merc 240D 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
## Merc 230 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
## Merc 280 19.2 6 167.6 123 3.92 3.440 18.30 1 0 4 4
## Merc 280C 17.8 6 167.6 123 3.92 3.440 18.90 1 0 4 4
## Merc 450SE 16.4 8 275.8 180 3.07 4.070 17.40 0 0 3 3
## Merc 450SL 17.3 8 275.8 180 3.07 3.730 17.60 0 0 3 3
## Merc 450SLC 15.2 8 275.8 180 3.07 3.780 18.00 0 0 3 3
## Cadillac Fleetwood 10.4 8 472.0 205 2.93 5.250 17.98 0 0 3 4
## Lincoln Continental 10.4 8 460.0 215 3.00 5.424 17.82 0 0 3 4
## Chrysler Imperial 14.7 8 440.0 230 3.23 5.345 17.42 0 0 3 4
## Fiat 128 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
## Honda Civic 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
## Toyota Corolla 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1## integer(0)3.1.5 Vectores y Listas - Ejercicio 2.3
Dado la siguiente pareja de vectores mu y nu, realizar una función que haga lo siguiente:
Vaya cogiendo los elementos de mu y nu que estén en la misma posición y los reuna en vectores.
Todas las parejas se reunirán en una lista y está es la salida de la función.
iset <- function(mu,nu){
mi.iset <- mapply(c, mu, nu, SIMPLIFY = FALSE)
return(mi.iset)
}#end function
mu <- c(0.1, 0.3, 1)
nu <- c(0.7, 0.5, 0)
memb <- iset(mu,nu)
str(memb)## List of 3
## $ : num [1:2] 0.1 0.7
## $ : num [1:2] 0.3 0.5
## $ : num [1:2] 1 03.1.6 Vectores y Listas - Ejercicio 2.4
Implementa una función que reciba una matriz y devuelva la diagonal de la matriz D, la submatriz inferior debajo de la diagonal R y la la submatriz superior encima de la diagonal R.
divido.matriz <- function(M){
return(list(D=diag(M),R=M[upper.tri(M)], L=M[lower.tri(M)]))
}# fin función
matriz <- matrix(1:9, 3, 3)
divido.matriz(matriz)## $D
## [1] 1 5 9
##
## $R
## [1] 4 7 8
##
## $L
## [1] 2 3 63.1.7 Laboratorio C1 - Ejercicio 1
# 1. Crea un vector v1 con los siguientes 10 valores (3, 12, 6, -5, 0, NA, 15, 1, -10, 7).
v1 <- c(3, 12, 6, -5, 0, NA, 15, 1, -10, 7)
# 2. Crea un vector v2 con valores de 0 a 10 de 0.1 en 0.1
v2 <- seq(from = 0, to = 10, by = 0.1)
# 3. Comando para ver el tipo de datos de v1.
class(v1)## [1] "numeric"# 4. Resta 2 a los elementos pares de v1 (con una sola orden y de forma vectorial). Define dos funciones que realicen esta tarea.
v1[c(FALSE,TRUE)]-2## [1] 10 -7 NA -1 5# 5. Calcula la suma y la media de v2. tem Con los vectores v2 y v3 crea un data.frame de nombre mi.df1.
sumV2 <- sum(v2)
sumV2## [1] 505## [1] 5# 6. Cambia el nombre de columnas de data.frame anterior con las etiquetas x y seno(x).
names(mi.df1) <- c("x", "seno(x)")
# 7. Invierte el orden de los elementos de v2 y lo guardas en v4.
v4 <- v2[(length(v2)):1]
v4## [1] 10.0 9.9 9.8 9.7 9.6 9.5 9.4 9.3 9.2 9.1 9.0 8.9 8.8 8.7 8.6
## [16] 8.5 8.4 8.3 8.2 8.1 8.0 7.9 7.8 7.7 7.6 7.5 7.4 7.3 7.2 7.1
## [31] 7.0 6.9 6.8 6.7 6.6 6.5 6.4 6.3 6.2 6.1 6.0 5.9 5.8 5.7 5.6
## [46] 5.5 5.4 5.3 5.2 5.1 5.0 4.9 4.8 4.7 4.6 4.5 4.4 4.3 4.2 4.1
## [61] 4.0 3.9 3.8 3.7 3.6 3.5 3.4 3.3 3.2 3.1 3.0 2.9 2.8 2.7 2.6
## [76] 2.5 2.4 2.3 2.2 2.1 2.0 1.9 1.8 1.7 1.6 1.5 1.4 1.3 1.2 1.1
## [91] 1.0 0.9 0.8 0.7 0.6 0.5 0.4 0.3 0.2 0.1 0.0# 8. Guarda en un vector v5 los elementos de v1 que no son negativos ni NA.
v5 <- v1[(v1>0) & (!is.na(v1))]
v5## [1] 3 12 6 15 1 7# 9. Borra de v1 los elementos negativos.
v1 <- v1[v1 > 0]
# 10. Calcula la longitud de v1 y mira la estructura del vector.
length(v1)## [1] 7## num [1:7] 3 12 6 NA 15 1 7# 11. Elimina v1 del Workspace de R.
rm(v1)
# 12. Calcula la media de aquellos valores de v2 que sean menores de 1.
media <- mean(v2[v2 < 1])
# 13. Elimina de v2 aquellos elementos mayores de 1 y menores de 3.
v2 <- v2[(v2 <= 1)|(v2 >= 3)]
# 14. Calcula la suma acumulada de los valores de v2.
suma <- sum(v2)
# 15. De varias formas distintas crea una matriz M1 con los valores de 1 a 25 con 5 filas (almacenados por filas).
M1 <- matrix(1:25,5,5,byrow = TRUE) # 1 Forma
M1 <- rbind(1:5,6:10,11:15,16:20,21:25) # 2 Forma
M1 <- 1:25 # 3 Forma
dim(M1) <- c(5,5)
M1 <- t(M1)
M1## [,1] [,2] [,3] [,4] [,5]
## [1,] 1 2 3 4 5
## [2,] 6 7 8 9 10
## [3,] 11 12 13 14 15
## [4,] 16 17 18 19 20
## [5,] 21 22 23 24 25## [1] 11 16 21 12 17 22 13 18 23 14 19 24 15 20 25# 17. Extrae de la matriz M1 la primera y la última columna y guarda el resultado en matriz M2.
M2 <- cbind(M1[,1], M1[,ncol(M1)])
# 18. Calcula el determinante de M1.
det(M1)## [1] 0# 19. Calcula la inversa de M1.
# solve(M1) Su determinante es 0, no se puede calcular
# 20. Calcula los autovectores y autovalores de M1. Define una función que dada una matriz devuelvan una lista su radio espectral que se define como el mayor autovalor en módulo y su autovector correspondiente.
eigen(M1)## eigen() decomposition
## $values
## [1] 6.864208e+01+0.000000e+00i -3.642081e+00+0.000000e+00i
## [3] -2.153465e-15+2.015276e-15i -2.153465e-15-2.015276e-15i
## [5] 7.219523e-16+0.000000e+00i
##
## $vectors
## [,1] [,2] [,3] [,4]
## [1,] -0.1079750+0i 0.67495283+0i 0.0381046+0.2345472i 0.0381046-0.2345472i
## [2,] -0.2527750+0i 0.36038970+0i 0.2645195-0.2955559i 0.2645195+0.2955559i
## [3,] -0.3975750+0i 0.04582657+0i -0.1224010-0.0257606i -0.1224010+0.0257606i
## [4,] -0.5423751+0i -0.26873656+0i -0.7011750+0.0000000i -0.7011750+0.0000000i
## [5,] -0.6871751+0i -0.58329969+0i 0.5209519+0.0867693i 0.5209519-0.0867693i
## [,5]
## [1,] -0.11703805+0i
## [2,] -0.22725312+0i
## [3,] 0.82596595+0i
## [4,] -0.50202033+0i
## [5,] 0.02034555+0if <- function(m){
eigens <- eigen(m)
modulos <- unlist(lapply(eigens$values, Mod))
indice <- which.max(modulos)
radEsp <- list(autovalor = (max(modulos[1])), autovector = eigens$vectors[, indice])
return(radEsp)
}
f(M1)## $autovalor
## [1] 68.64208
##
## $autovector
## [1] -0.1079750+0i -0.2527750+0i -0.3975750+0i -0.5423751+0i -0.6871751+0i3.1.8 Laboratorio C2 - Ejercicio 2
Primero importa el dataset landdata-states.csv del CV
landdata_states <- read.csv("landdata-states.csv")
# 1. Calcula el máximo, mínimo y media de la columna Home.Value.
maximo <- max(landdata_states$Home.Value)
minimo <- min(landdata_states$Home.Value)
media <- mean(landdata_states$Home.Value)
# 2. Elimina la última columna. Elimina la segunda fila del dataset.
landdata_states2 <- landdata_states[, -ncol(landdata_states)]
landdata_states2 <- landdata_states[-2,]
# 3. Explica con ejemplos la diferencia entre usar [] o [[]] para acceder a elementos del data frame. Mira la clase y la estructura de la segunda y tercera columna. Di de que tipo es cada una y su longitud.
# Con [] accedemos a las columnas que indiquemos entre corchetes, pero devuelve un data frame. Si queremos acceder al vector que está dentro del data frame usaremos [[]].
class(landdata_states[[2]])## [1] "factor"## [1] "numeric"## Factor w/ 4 levels "Midwest","N. East",..: 4 4 4 4 4 4 4 4 4 4 ...## num [1:7803] 2010 2010 2010 2010 2008 ...## [1] 7803## [1] 7803# 4. De la columna Year cambia todos los años menores del 2000 por NA.
landdata_states$Year[landdata_states$Year < 2000] <- NA
# 5. Cambia - traduciendo el nombre de las columnas del dataset (si alguna columna tiene nombre extraño que no sabemos significado - simplemente coloca primera letra del nombre de inglés y un número).
names(landdata_states) <- c("Estado", "Region", "Fecha", "Valor.Casa", "Estructura.Coste", "Coste.Terreno", "LS1", "Indice.Precio.Casa", "Indice.Precio.Terreno", "Año", "Cuatrimestre")
names(landdata_states)## [1] "Estado" "Region" "Fecha"
## [4] "Valor.Casa" "Estructura.Coste" "Coste.Terreno"
## [7] "LS1" "Indice.Precio.Casa" "Indice.Precio.Terreno"
## [10] "Año" "Cuatrimestre"3.2 Ejercicios DPLYR
Los siguientes ejercicios se encuentran en el documento dplyr_breast-cancer guion.Rmd del campus.
Descargar a local desde directorio Datasets de CV, un dataset descargado del repositorio UCI: breast-cancer.data, y breast-cancer.names1.csv (nombres de las columnas)
Usar el paquete
dplyr, otro de los paquetes centrales del denominado tidyverse.En
tidyverse-dplyrInstalar
tidyverseCargar paquete
tidyverse
3.2.1 Importación de datos
- Importa en R el fichero breast-cancer.data.
- Colocar los nombres al dataset que están en breast-cancer.names1.csv.
cancer <- read.csv("breast-cancer.data", header=FALSE)
nombres <- read.csv("breast-cancer.names1.csv", header=FALSE)
names(cancer) <- nombres[, 1]
names(cancer) # Comprobamos que se hayan añadido los nombres correctamente## [1] "Class" "age" "menopause" "tumor_size" "inv_nodes"
## [6] "node_caps" "deg_malig" "breast" "breast_quad" "irradiat"3.2.2 dplyr para Manipulación de Datos
Fundamentos de dplyr
5 funciones clave de dplyr:
- Escoger observaciones (filas) según sus valores (
filter()). - Reordenar las filas (
arrange()). - Seleccionar variables por su nombre (
select()). - Crear nuevas variables como función de variables ya existentes (mutate()).
- Encontrar valores representativos de cada variable (
summarise()).
Todos estos verbos funcionan de la misma manera:
- El primer argumento es un dataframe.
- Los demás argumentos describen qué hacer con el dataframe, usando los nombres de las variables (columnas) sin necesidad de utilizar comillas.
- El resultado es un nuevo dataframe.
Filtrado de filas con filter()
filter() extrae un subconjunto de las observaciones (filas), basándose en los valores de una o más columnas.
Argumentos: - nombre del dataframe - expresiones (lógicas) para filtrar el dataframe
Ejercicios:
- Extraer las filas del dataset correspondientes al año 1952.
- Extraer las filas del dataset correspondientes al año 1952 y que tienen cancer en el pecho izquierdo.
- Extraer las filas del dataset que tienen cancer en el pecho izquierdo, no radiadas y de edad 40 a 49.
- Reordenar el dataset según el tamaño del tumor.
# Primero reordenare los levels, ya que no se encuentran ordenados por lo que la funcion arrange no realizara una correcta ordenacion.
cancer$tumor_size <- factor(cancer$tumor_size, ordered = TRUE, levels = c("0-4", "5-9", "10-14", "15-19", "20-24", "25-29", "30-34", "35-39", "40-44", "45-49", "50-54"))
paged_table(cancer %>%
arrange(tumor_size))- Reordenar el dataset según el tamaño del tumor y el grado que tiene el tumor.
- Reordenar el dataset según el tamaño del tumor (pero descendiente) y el grado de maligno que tiene el tumor - ayuda - desc(columna).
- Extraer los 10 pacientes con pre-menopausia, con mayor tamaño de tumor y menor número de nodos invasores y ordenados por edad.
# Al igual que la columna tumor_size, reordenare primero los levels de la columna inv_nodes
cancer$inv_nodes <- factor(cancer$inv_nodes, ordered=TRUE, levels = c("0-2", "3-5", "6-8", "9-11", "12-14", "15-17", "24-26"))
consulta <- cancer %>%
filter(menopause=="premeno") %>%
arrange(desc(tumor_size), inv_nodes, age)
consulta <- consulta[1:10,]
paged_table(consulta)top_n()
re-ordenación por una variable y sólo tomar los n valores más altos o más bajos:
- nombre del dataframe,
n, el número de elementos que recuperar (n negativo los n más bajos)- el nombre de la variable por la que ordenar
Ejercicios:
- Usando
top_n()extraer los 10 pacientes con pre-menopausia, con mayor tamaño de tumor y menor número de nodos invasores y ordenados por edad.
consulta <- cancer %>%
filter(menopause=="premeno") %>%
top_n(10, tumor_size) %>%
top_n(-10, inv_nodes) %>%
arrange(age)
consulta <- consulta[1:10,]
paged_table(consulta)Selección de columnas select()
- seleccionar únicamente aquellas variables en las que estamos interesados - ayuda - select(dataset, col1, col2, col3)
Ejercicios:
- Seleccionar del dataset las columnas clase, tamaño de tumor y grado de tumor
Podemos seleccionar intervalos de columnas (de col1 a col4):
- ‘deseleccionar’ columnas
Otra utilidad de select(), conjuntamente con la función everything(), es mover algunas columnas o variables al principio del dataframe.
select()
Otras funciones auxiliares, además del mencionado everything(), que nos pueden ser de ayuda a la hora de seleccionar columnas:
starts_with('abc'): encuentra todas las columnas cuyo nombre comienza por “abc”.ends_with('xyz'): encuentra todas las columnas cuyo nombre termina en “xyz”.contains('ijk'): para seleccionar las columnas cuyo nombre contenga la cadena de caracteres “ijk”.
Y otras funciones más complejas (que filtran las columnas por expresiones regulares), que se pueden ver al hacer ?select.
Añadir o renombrar variables con mutate()
El verbo mutate() se usa para añadir nuevas columnas al final del dataframe.
Por ejemplo, añadimos columna dist_grado_peor que es 4.
Ejercicio
- añadir una columna media_tumor que tenga el valor medio de los valores almacenados en variable tumor_size
# Creo una funcion para poder hacer la media del tamaño del tumor.
f <- function(data){
res <- strsplit(as.character(cancer$tumor_size), "-") # Al ser tipo factor, debo convertirlo en character para separar los valores
res <- lapply(res, as.integer) # convierto a integer los char resultantes
res <- unlist(lapply(res,mean)) # Hago la media a cada par y aplano la lista
return(res)
}
paged_table(cancer %>%
mutate(media_tumor = f(tumor_size)))transmute()
Si sólo se desea guardar las nuevas variables, se puede usar la función transmute():
rename()
- función
rename(), que, internamente, se comporta comoselect(), pero guardando todas las variables que no se mencionan explícitamente:
Uso del operador %>%
%>%utiliza la salida del término que hay a la izquierda del símbolo%>%como primer argumento de la función que está a la derecha de dicho símbolo.x %>% f(y)es igual que hacerf(x, y)
br1 <- brcan_data %>%
filter(breast == "left" & age == "30-39") %>%
select(deg_malig, tumor_size, everything()) %>%
arrange(desc(deg_malig),tumor_size)
br1[1:5,]brcan_data %>%
filter(breast == "left" & age == "30-39") %>%
select(age,tumor_size,deg_malig) %>%
top_n(5,desc(deg_malig))3.3 Ejercicios Apply Functions
- Dada la siguiente lista con nombre lista1 que debes introducir en R, resolver los siguientes ejercicios:
lista1 <- list(notas_grupo1 = c(1,2,3,4,5,7,6,5,4,3), notas_grupo2_3 = matrix(c(2,3,2,9,8,8), nrow = 2))
lista1## $notas_grupo1
## [1] 1 2 3 4 5 7 6 5 4 3
##
## $notas_grupo2_3
## [,1] [,2] [,3]
## [1,] 2 2 8
## [2,] 3 9 8- Usa lapply() para encontrar la longitud de las notas de todos los grupos que están en lista1.
## $notas_grupo1
## [1] 10
##
## $notas_grupo2_3
## [1] 6- Usa sapply() para encontrar la media de las notas de todos los grupos que están en lista1.
## notas_grupo1 notas_grupo2_3
## 4.000000 5.333333- Usa lapply() para encontrar los quantiles de las notas de todos los grupos que están en lista1.
## $notas_grupo1
## 0% 25% 50% 75% 100%
## 1 3 4 5 7
##
## $notas_grupo2_3
## 0% 25% 50% 75% 100%
## 2.00 2.25 5.50 8.00 9.00- Dada la siguiente función f1(x)=(3x)/10, aplica f1(x) a todas las notas para cambiar las puntuaciones a la escala de 1 a 3.
## $notas_grupo1
## [1] 0.3 0.6 0.9 1.2 1.5 2.1 1.8 1.5 1.2 0.9
##
## $notas_grupo2_3
## [,1] [,2] [,3]
## [1,] 0.6 0.6 2.4
## [2,] 0.9 2.7 2.4- Repite el ejercicio 4 pero haciendo que f1(x) sea una función anónima dentro de lapply().
## $notas_grupo1
## [1] 0.3 0.6 0.9 1.2 1.5 2.1 1.8 1.5 1.2 0.9
##
## $notas_grupo2_3
## [,1] [,2] [,3]
## [1,] 0.6 0.6 2.4
## [2,] 0.9 2.7 2.4- Encuentra los valores no repetidos de las notas.
## [[1]]
## [1] 2 8
##
## [[2]]
## [1] 3 9 8## [1] 2 8 3 9 8## [[1]]
## [1] 1 2 3 4 5 7 6 5 4 3
##
## [[2]]
## [1] 2 8 3 9 8## [1] 1 2 3 4 5 7 6 8 9- Convierte todas las notas de la lista a un vector (aplana los valores).
# Hacemos uso de unlist y covertimos a vector
vector <- as.vector(unlist(lista1,use.names = FALSE))
vector## [1] 1 2 3 4 5 7 6 5 4 3 2 3 2 9 8 83.4 Visualizacion
Paquetes necesarios
Para poder usar el comando ‘include_graphics’ necesitais que vuestros ordenadores tengan:
- Latex: En Windows instalar MikTeX, en Mac MacTex.
- Pandoc: universal document converter - (https://pandoc.org). Instalar para el S.O. de tu ordenador.
3.4.1 Incrustar gráficos
El siguiente chunk podéis usarlo como plantilla para que sepáis como incrustar gráficos en RMarkdown utilizando el comando ‘include_graphics’ por si os hace falta en algún trabajo. Permite controlar tamaños, etc. Podéis ver en el .Rmd, los parámetros que he puesto en el chunk :
- eval=FALSE. No lo evalua
- echo=TRUE. Si muestra el código en el documento generado.
- fig.width, fig.height, out.width para controlar los tamaños de la salida y del gráfico
- fig.cap: poner una leyenda al gráfico
3.4.2 Ejercicio 1
Introducir en R el siguiente data frame:
mis.Plantas <- data.frame(Plantas = c("Planta1", "Planta1", "Planta1", "Planta2",
"Planta2", "Planta2"), Tipo = c(1, 2, 3, 1, 2, 3),
Eje1 = c(0.2, -0.4, 0.8, -0.2, -0.7, 0.1),
Eje2 = c(0.5, 0.3, -0.1, -0.3, -0.1, -0.8))- Utilizando los comandos básicos de R realizar un gráfico lo más similar posible al que aparece en ModeloDeVisualizacion_ejercicio1_2.html.
# Primer Grafico con 'Plot'
# Defino los colores
colors <- c("#00DCFF", "#007CFF", "#1B00FF")
colors <- colors[mis.Plantas$Tipo]
# Defino las formas
shapes <- c(1,2)
shapes <- shapes[as.numeric(mis.Plantas$Plantas)]
plot(mis.Plantas$Eje1, mis.Plantas$Eje2,
main = "Sin ggplot", xlab = "Eje 1", ylab = "Eje 2",
pch = shapes, col = colors
)
legend("topright", legend = c("Planta 1", "Planta 2"), pch = c(1,2))
legend("bottomright", c("Tipo 1", "Tipo 2", "Tipo 3"), col = c("#00DCFF", "#007CFF", "#1B00FF"), pch = 19)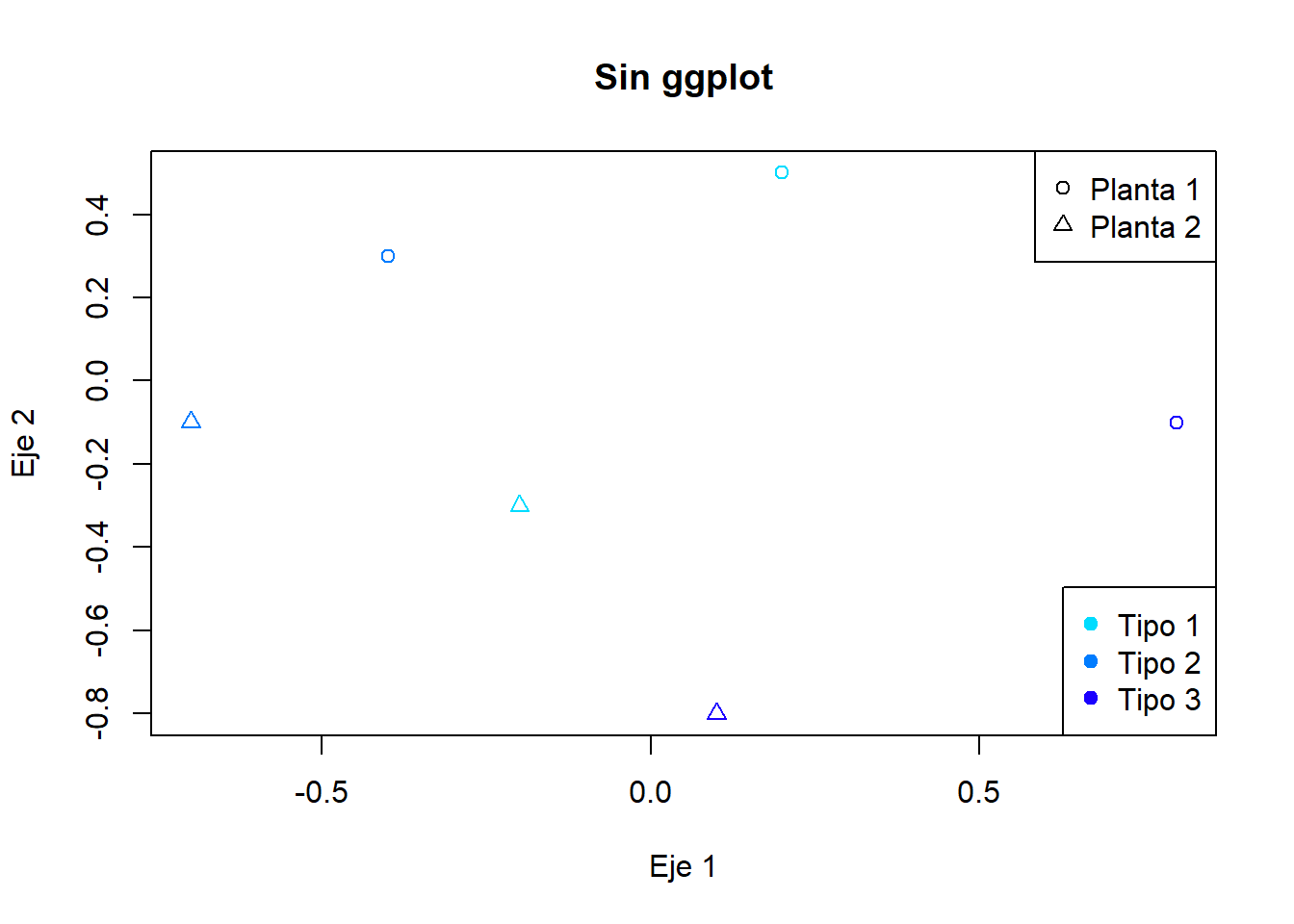
# Segundo grafico con 'ggplot2'
library(ggplot2)
p1 <- ggplot(mis.Plantas, aes(Eje1, Eje2)) +
ggtitle("Con ggplot") +
geom_point(aes(color = Tipo, shape = Plantas))
p1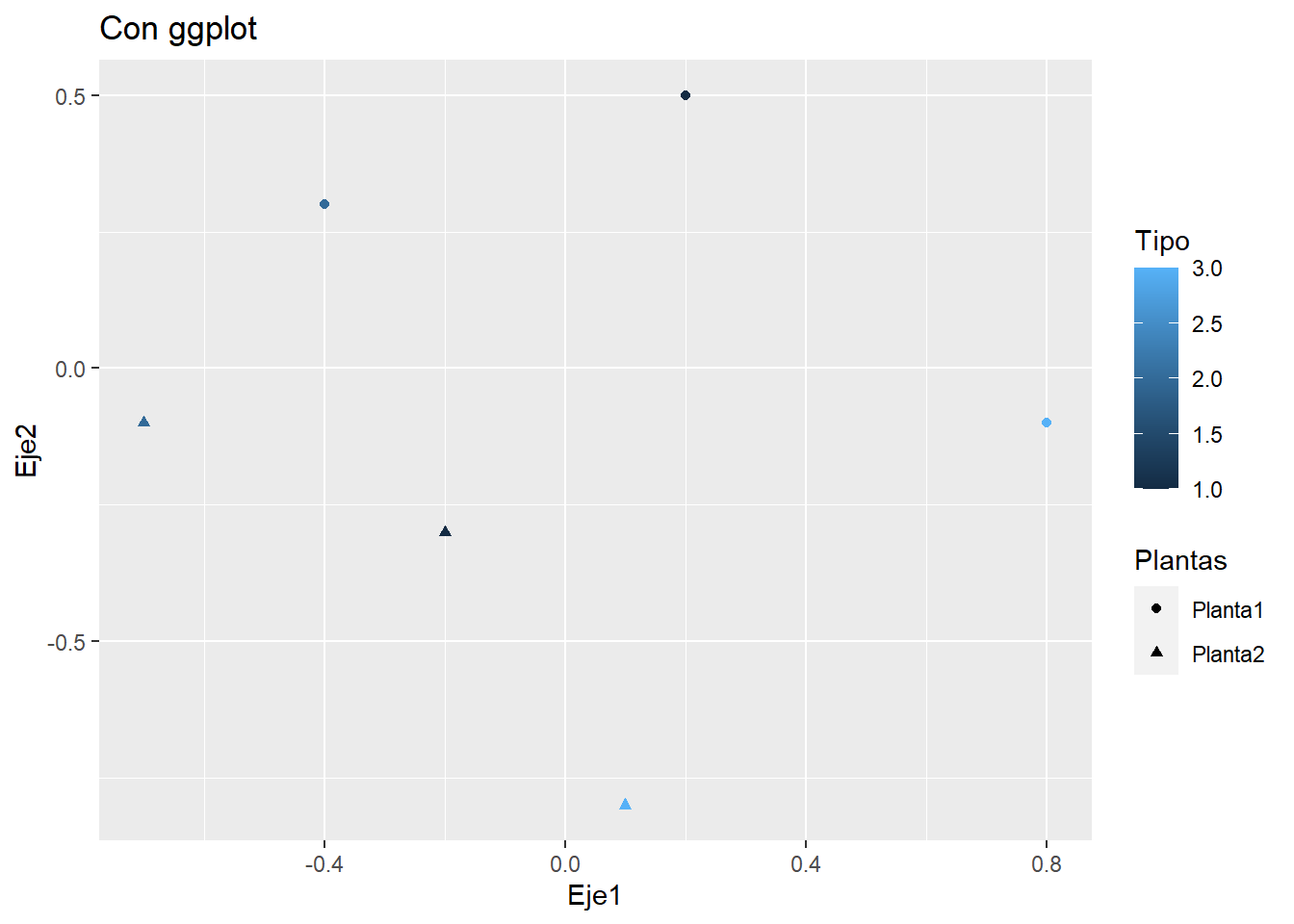
3.4.3 Ejercicio 2
Usaremos el dataset msleep de R (ver las primeras filas del dataset para comprobar la estructura).
- Aplicar ggplot2 para realizar gráficos lo más similares posibles al que aparece en ModeloDeVisualizacion_ejercicio1_2.html.
# Gráfica 3
data("msleep")
sleep <- as.data.frame(msleep)
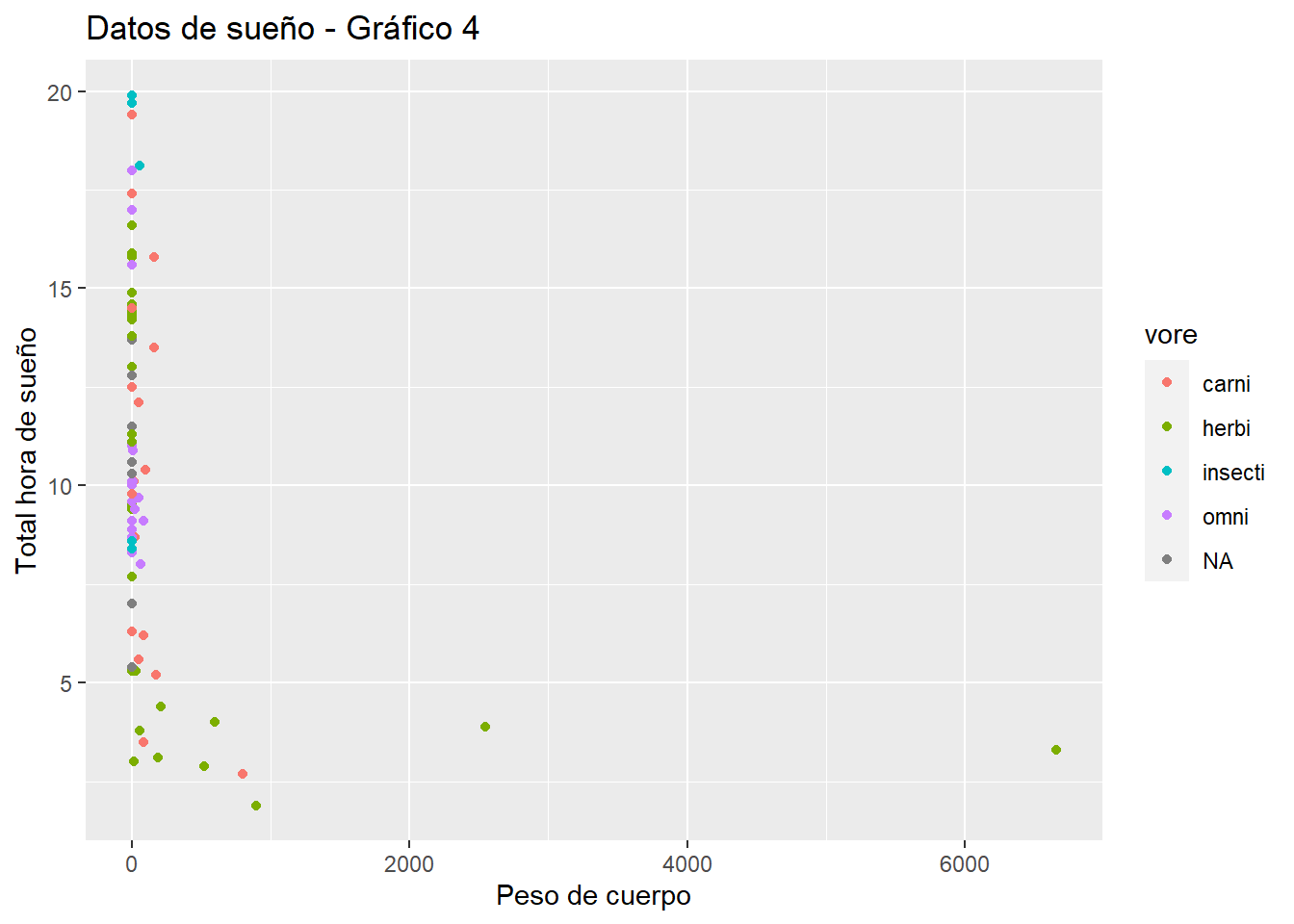
p2 <- ggplot(sleep, aes(bodywt, sleep_total)) + xlab("Peso de cuerpo") + ylab("Total hora de sueño") +
ggtitle("Datos de sueño - Gráfico 3") +
geom_point()
p2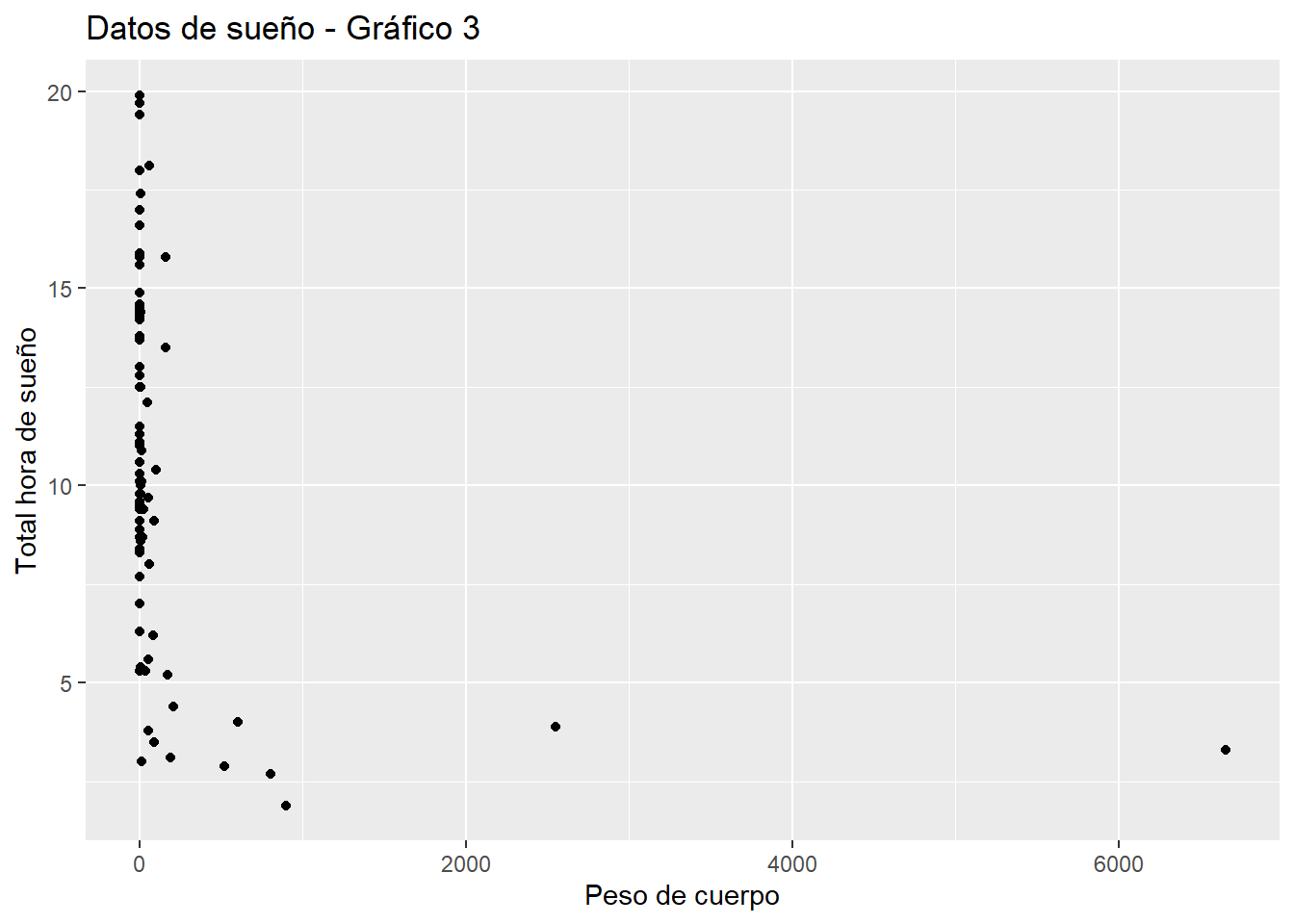
3.4.4 Ejercicio 3
Con el mismo dataset del Ejercicio 2, aplicar ggplot2 para realizar gráficos lo más similares posibles al que aparece en ModeloDeVisualizacion_ejercicio3_4.html.
Ayuda: En el eje X usaremos la función logaritmo, en el eje Y se mostrará la fracción de sueño que es REM respecto al total para cada fila. Modificar posteriormente para la segunda figura.
# Grafica 5
p5 <- ggplot(sleep, aes(log(bodywt), sleep_rem/sleep_total)) + # Habra filas que se eliminan por tener el valor NA
xlab("Peso de cuerpo") + ylab("Fracción REM respecto al total de horas de sueño") +
ggtitle("Datos de sueño - Gráfico 5") +
geom_point(aes(color = vore))
p5## Warning: Removed 22 rows containing missing values (geom_point).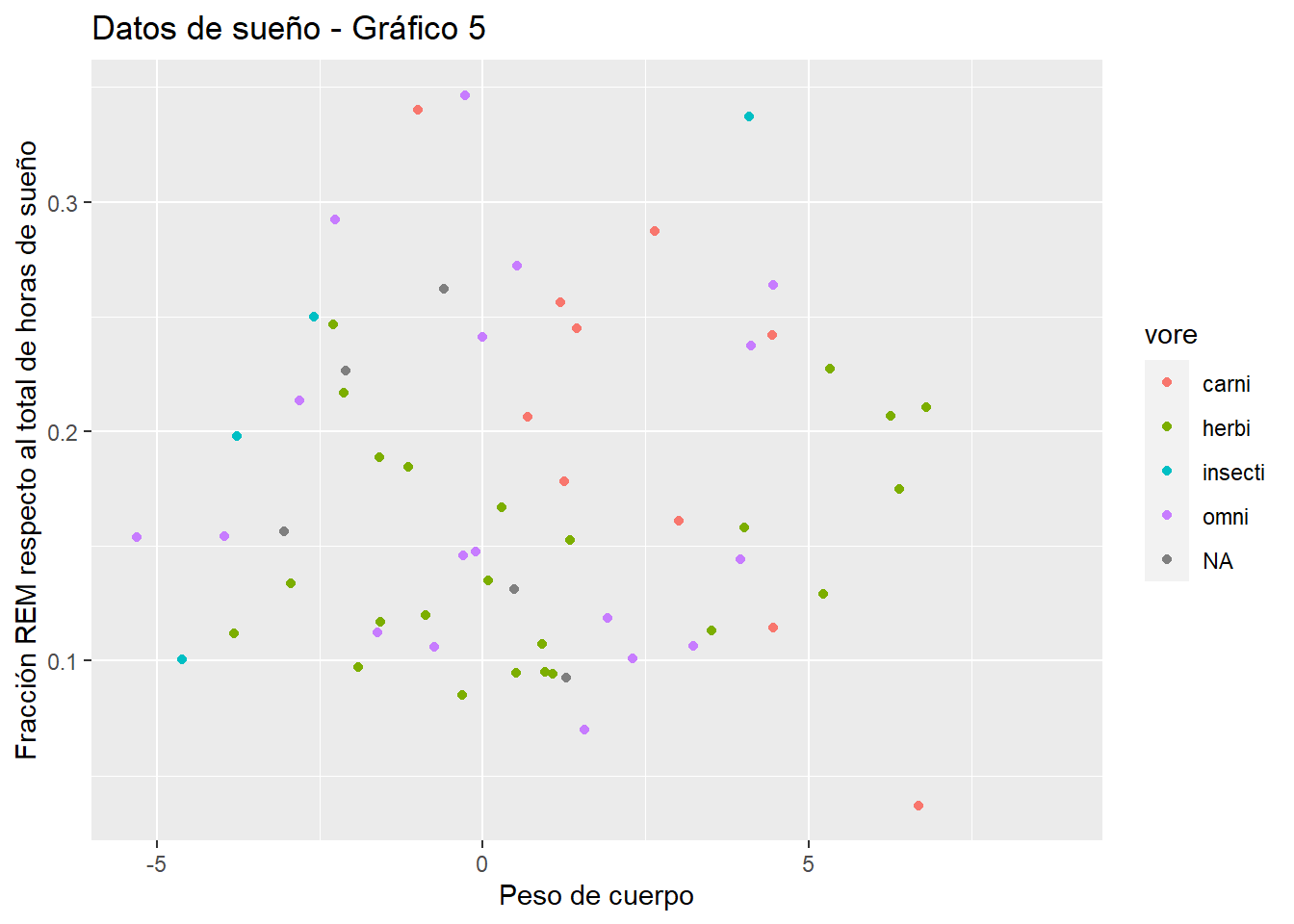
# Grafica 6
p6 <- p5 + ggtitle("Datos de sueño - Gráfico 6") + geom_point(aes(color = vore), size = 4)
p6## Warning: Removed 22 rows containing missing values (geom_point).
## Warning: Removed 22 rows containing missing values (geom_point).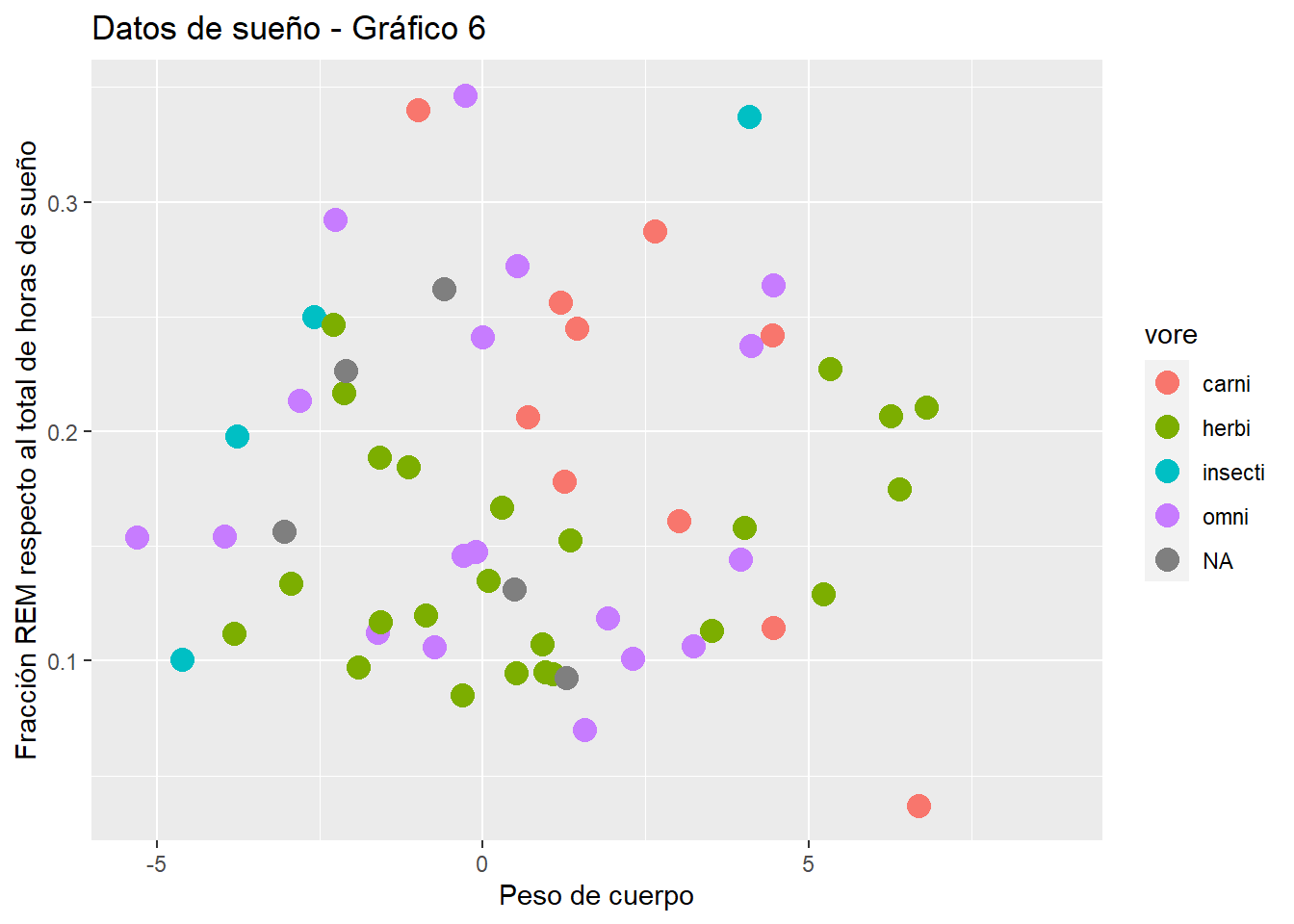
3.4.5 Ejercicio 4
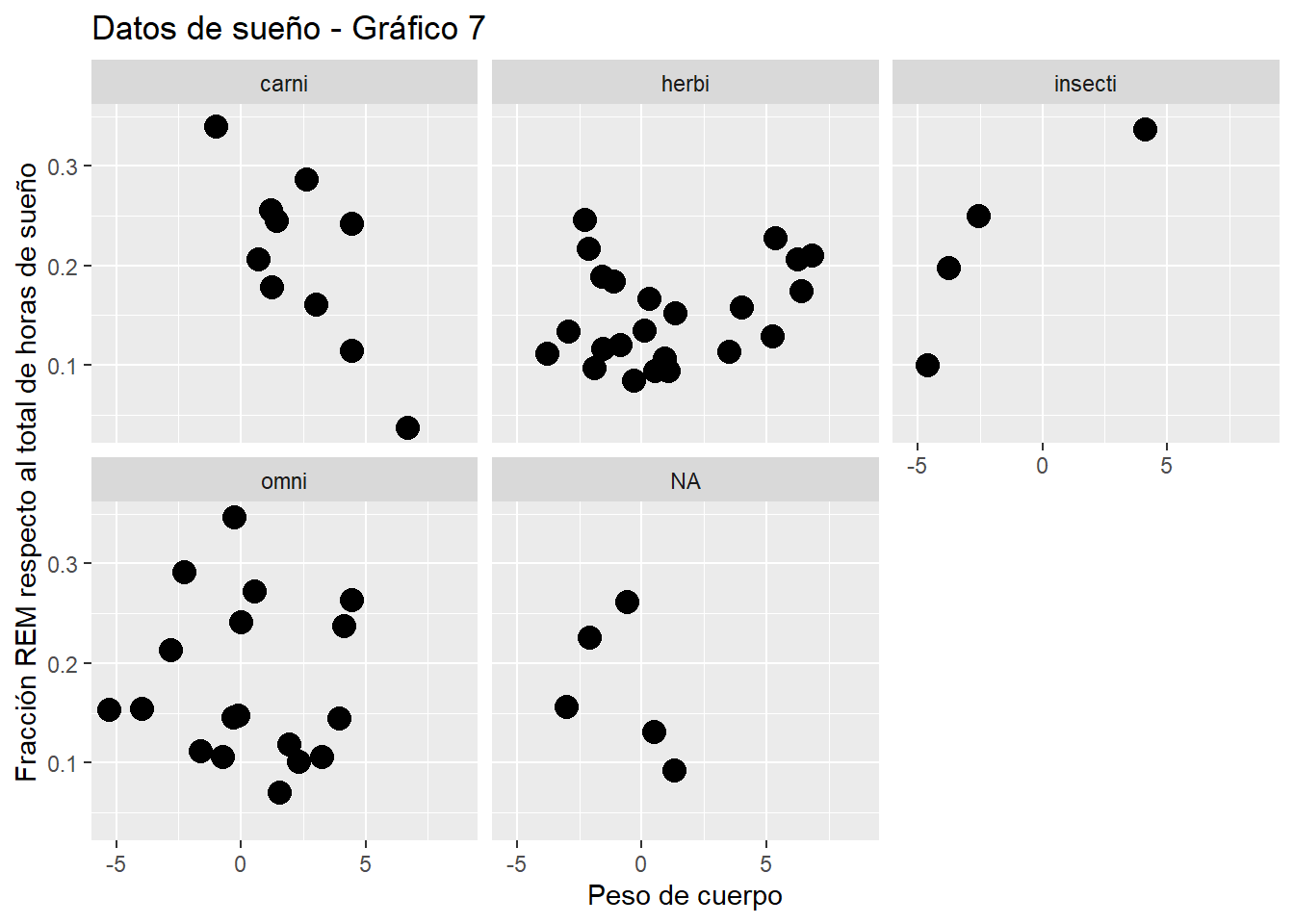
Con el mismo dataset del Ejercicio 2, usar comando facet para dividir los gráficos anteriores en múltiples gráficos.
Realizar gráfico lo más similar posible al que aparece en ModeloDeVisualizacion_ejercicio3_4.html.
# Grafica 7
p7 <- ggplot(sleep, aes(log(bodywt), sleep_rem/sleep_total)) + # Habra filas que se eliminan por tener el valor NA
xlab("Peso de cuerpo") + ylab("Fracción REM respecto al total de horas de sueño") +
ggtitle("Datos de sueño - Gráfico 7") +
geom_point(size = 4) + facet_wrap(~sleep$vore)
p7## Warning: Removed 22 rows containing missing values (geom_point).