第5章 ctDNA检测的终极武器
第5期,我们将对ctDNA的关键技术细节进行介绍,期望使您深刻理解ctDNA在液体活检中占据重要地位的原因。
ctDNA中的肿瘤信息就像是宝藏吸引着科学家们的眼光,然而要想探索宝藏,首先要得到绝世神兵,今天就给大家介绍下这把终极武器的妙用吧!
5.1 ctDNA检测为什么需要更高灵敏度的技术?
ctDNA的检测,相比于组织活检,有更高的灵敏度要求。这其中最重要的原因,就是我们目前没有办法单独获取来自于肿瘤的游离DNA!所谓ctDNA检测,是检测所有cfDNA后分析肿瘤特异性的变异,并将携带这些变异的DNA片段视作ctDNA。
Figure 5.1: 从一堆游离DNA中挑出来自肿瘤的游离DNA,可不是一件容易的事儿。
然而,机体的肿瘤细胞相比于正常细胞的数量,简直是九牛一毛。当然这也跟肿瘤的大小、分期、转移等是相关的,不同患者间ctDNA占cfDNA的比例变化范围很大,可从0.01%-90%,大多在千分之几的级别。所以,ctDNA的突变频率明显低于组织活检,有研究表明,有半数以上的突变频率低于0.4%!
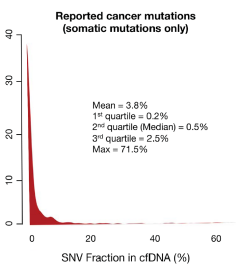
Figure 5.2: 半数以上的突变频率低于0.4%。突变如此低频,技术须更灵敏。
5.2 常用检测技术为什么难以满足需求?
目前常用的ctDNA检测技术主要有以下几种:
| 技术 | 适用性 | 灵敏度 |
|---|---|---|
| ARMS-qPCR | 组织突变检测 | 1% |
| NGS | 多基因扫描 | 1% |
| dPCR | 位点监测 | 0.01% |
| BEAMing | 位点监测 | 0.01% |
ARMS-qPCR操作简便,且有成熟试剂盒。NGS的检测范围广,能同时检测突变、扩增、融合。然而这两种技术都存在灵敏度低的问题,特别是NGS,更需要降低背景噪音的技术。
dPCR和BEAMing的灵敏度足够高,但两者都存在检测位点数目少的局限性,因而不适合用于用药指导。
ARMS-qPCR (Amplification Refractory Mutation System)方法是临床上常用的组织活检突变检测方法,简便易行,大多数医院都可以自行操作。然而在液体活检中,这1%的灵敏度可就不够看了,会导致大量假阴性的出现。《非小细胞肺癌(NSCLC)血液EGFR基因突变检测中国专家共识》中提到,与肿瘤组织相比,基于ARMS方法的ctDNA检测敏感性相对较低,在IPASS、IFUM和IGNITE三项研究中分别为43. 1%、65. 7%和49. 6%,这种敏感性显然是不能满足需求的!
dPCR(digital PCR)和BEAMing的检测方式类似,通过将反应体系细分来提高灵敏度。我们可以理解成一个集成的qPCR反应,将反应体系分成成千上万个,每个反应中含有一个模板DNA分子,通过识别阳性反应数来实现模板分子的绝对定量。反应体系越多,能达到的灵敏度就越高。这两种方法的优势自然是灵敏度,高达0.01%甚至0.001%。然而,我们也能看出,灵敏度跟模板分子数量相关,所以对样本量要求较大,10ml血液往往仅能检测有限几个位点。目前,主要应用在于特定位点的疗效跟踪和耐药监测,对于用药指导、早期辅助诊断等则不适用。
NGS由于存在大量背景噪音,会淹没频率在1%以下的突变,也因此限制了测序的准确度。也就是说,普通的NGS用于ctDNA检测,其结果将会与ARMS-qPCR半斤八两,但为什么NGS技术在ctDNA研究中反而应用越来越广泛呢?这就要归功于今天的主角——分子标签技术了!
5.3 分子标签技术是什么?它是如何提高NGS检测灵敏度的?
要理解分子标签技术的作用,首先要理解NGS的背景噪音来源。NGS有另外一个名字,叫深度测序,也就是说,对于一个位点,需要很多个DNA片段覆盖它,才能够进行准确分析。在这过程中,需要经过模板的PCR扩增,而DNA聚合酶可能会连接上一个错误的碱基,导致错误;另外,测序系统最后的信号识别也会发生错误。当然,常规情况下,我们不知道这些是真突变还是系统错误,而系统错误又极多,所以只能根据突变频率将背景噪音全部过滤,同时低频的真突变也会被殃及,这就是为什么背景噪音是限制灵敏度的原因了。
2012年5月,Forshew T等人率先发表文章,开发了基于分子标签的TAm-Seq方法。2012年9月,Michael W等人也发表文章,利用双链分子标签,将错误率显著降低,其中双链匹配的数据中,错误率可降低至∼0.001% mutations/bp!当然,实际应用中很难达到这么高,但也足够将灵敏度提高到0.1%以上了。
分子标签技术是在DNA模板上连接一段随机序列,相当于给每一个DNA模板加上一个独特的分子标签,从而可区分不同来源的模板。这样在进行数据分析时,可以根据标签序列识别同一DNA模板扩增出的片段,把它们统一分析,从而过滤掉PCR错误及测序错误,提高检测灵敏度和准确性。
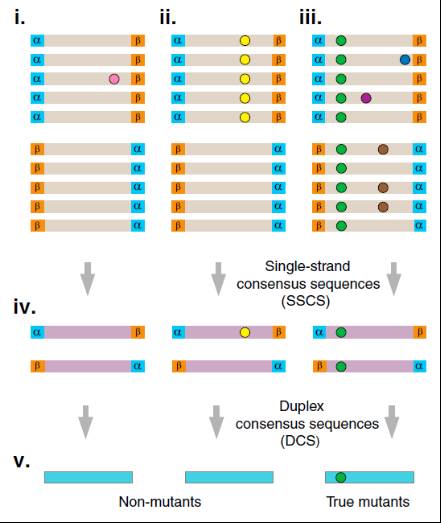
Figure 5.3: 双链分子标签原理图。
如图中显示的就是双链分子标签的分析原理,α和β就是一段随机的DNA序列,不同的DNA模板连接的α和β是不一样的。我们可以通过α和β序列来识别同一模板来源的片段,同时根据α和β互补识别同一模板来源的正反义链(单链分子标签相当于只有α,就无法实现这个功能了)。
左侧显示的是一个PCR扩增后期引入的错误或一个测序错误,仅影响少数DNA序列,由于同一模板来源的片段理应序列相同,因此利用少数服从多数的原则就可以过滤掉;中间部分显示的是一个PCR扩增早期引入的错误,影响了正链所有序列,但反链无突变,而碱基互补是DNA双链的基本原则,因此这种错误也可以得到校正(仅双链分子标签可以实现)。
通过分子标签技术,可以大幅降低NGS的背景噪音,从而提高测序准确度,显然双链标签比单链标签的过滤能力更强!然而,虽然原理上容易理解,科研上也逐渐得到广泛应用,但实现应用起来却存在接头连接效率低、双链互补数据比例少等问题,需要不断优化,还要开发配套的信息分析系统。因此能够实现其应用的公司就寥寥可数了!这些公司的NGS液体活检技术会有所差别,但背景噪音过滤的原理都基于分子标签。
海普洛斯通过不断优化,开发出专利的CUBE-ctDNA测序技术,基于双链分子标签的原理,将检测灵敏度提高至0.05%。
基于分子标签技术的NGS技术,集合了检测范围广和高灵敏度的优势,在肿瘤用药指导、早期诊断、早期筛查、预后判断、耐药机制分析等方面,势必会得到越来越广泛的应用!后面的文章会陆续介绍给大家。
最后要吐一个槽:一切脱离技术原理吹嘘灵敏度的行为都是耍流氓!
参考文献
- Lanman RB, et al. Analytical and Clinical Validation of a Digital Sequencing Panel for Quantitative, Highly Accurate Evaluation of Cell-Free Circulating Tumor DNA. PLoS ONE 10(10):e0140712.doi:10.1371/journal. pone.0140712.
- Sorber L, et al. Circulating cell-free nucleic acids and platelets as a liquid biopsy in the provision of personalized therapy for lung cancer patients. Lung Cancer. 2016 May 4. pii: S0169-5002(16)30312-9.
- Forshew, T. et al. Noninvasive identification and monitoring of cancer mutations by targeted deep sequencing of plasma DNA. Sci. Transl Med. 4, 136ra68 (2012).
- Schmitt MW. et al. Detection of ultra-rare mutations by next-generation sequencing. Proc Natl Acad Sci U S A. 2012 Sep 4;109(36):14508-13.