第4章 ctDNA的成长之路:从孟德尔的发现到NCCN指南
第4期,我们将对ctDNA的发展史进行介绍,期望使您理解ctDNA在液体活检中占据重要地位的原因。
ctDNA作为现在液体活检最重要的标志物,从其发现到进入临床应用都经历了哪些过程呢?今天就跟大家分享一下ctDNA研究和应用的历史。
首先为大家展现一个timeline,简要地囊括了ctDNA发展史上的重要事件。
1948年:Mandel和Metais发现血浆中存在游离的核酸分子。
1977年:Leon等人发现,肿瘤患者的血浆游离DNA水平要明显高于健康人群,据此推测游离DNA与肿瘤相关。
1989年:Stroun和colleagues报道肿瘤患者的cfDNA中部分来源于肿瘤细胞。
1994年:第一次利用PCR方法在胰腺癌患者血液cfDNA中检测到了KRAS突变,该突变与肿瘤组织中检测到的一致。
2008年:Diehl F.等对18名肠癌患者的ctDNA进行了跟踪,利用BEAMing技术检测APC、KRAS、TP53、PIK3CA等基因热点突变,发现ctDNA突变率随着治疗过程变化,变化趋势与肿瘤负荷及CEA浓度呈正相关。
2012年:Forshew T等人与Michael W等人分别发表文章,利用分子标签的原理过滤NGS中的PCR错误和测序错误,能够显著降低背景噪音,从而提高NGS检测灵敏度,使之适用于cfDNA中超低频突变的检测。
2014年:欧盟EMA批准利用ctDNA检测EGFR突变用于易瑞沙的伴随诊断,标志着ctDNA正式临床应用。
2015年:《非小细胞肺癌(NSCLC)血液EGFR基因突变检测中国专家共识》于《中华医学杂志》上发表,补充了如果肿瘤标本不可评估EGFR基因状态,则可使用从血液(血浆)标本中获得的ctDNA进行评估。
2016年:非小细胞肺癌NCCN指南中添加了液体活检推荐。
通过这条时间线我们可以看到,ctDNA有着数十年的历史。接下来就为大家详细解说,它是如何从孟德尔的发现一路走进NCCN指南的。
4.1 cfDNA的发现
血中游离DNA简称cfDNA(Cell-Free DNA),是指循环血中游离于细胞外的部分降解了的机体内源性DNA。
早在1948年,比DNA双螺旋结构发现还早6年的时候,Mandel和Metais就发现血浆中存在游离的核酸分子;1977年,Leon等人发现,肿瘤患者的血浆游离DNA水平要明显高于健康人群,据此推测游离DNA与肿瘤相关。但由于缺乏高灵敏性和高特异性的实验方法,导致有关血中游离DNA与疾病相关性的研究在较长时期内进展缓慢。直到有效分离游离DNA技术的出现,和特殊荧光染料与PCR技术相结合的检测技术的应用,才使这一领域的研究得到了较迅速发展。
4.2 ctDNA作为肿瘤标志物地位的确立
1989年,Stroun和colleagues报道肿瘤患者的cfDNA中部分来源于肿瘤细胞;1991年, Sidransky等人研究表明,膀胱癌患者尿沉渣中DNA携带有TP53突变,提示了可以利用基因分析来检测cfDNA;之后陆续在结直肠癌、胰腺癌及肺癌等患者的粪便和痰样本中检测到了KRAS突变。1994年, 第一次利用PCR方法在胰腺癌患者血液cfDNA中检测到了KRAS突变,该突变与肿瘤组织中检测到的一致。也就是说,cfDNA中携带肿瘤特有突变的那一小部分DNA,确确实实是由肿瘤细胞释放出来的。
此后,人们认识到cfDNA中的肿瘤相关突变是肿瘤特异性的标志物,并称这些携带肿瘤特征的cfDNA片段为循环肿瘤DNA(Circulating Tumor DNA,ctDNA)。
4.3 技术革新促进ctDNA研究爆发
测序技术限制着ctDNA的研究发展。直到2000年前后,高灵敏度技术开始出现,ctDNA的研究开始爆发。
随着更高灵敏度的数字PCR(digital PCR,dPCR于1999年出现)和BEAMing技术(于2006年出现)的应用,ctDNA的研究逐渐增多。2008年,Diehl F.等对18名肠癌患者的ctDNA进行了跟踪,利用BEAMing技术检测APC、KRAS、TP53、PIK3CA等基因热点突变,发现ctDNA突变率随着治疗过程变化,变化趋势与肿瘤负荷及CEA浓度呈正相关。
然而,dPCR和BEAMing虽然检测灵敏度高达0.01%,但其检测范围仅局限于少许位点,往往需要先对肿瘤组织进行测序挑选特定位点,再进行后续监测,严重限制了ctDNA研究的广度。
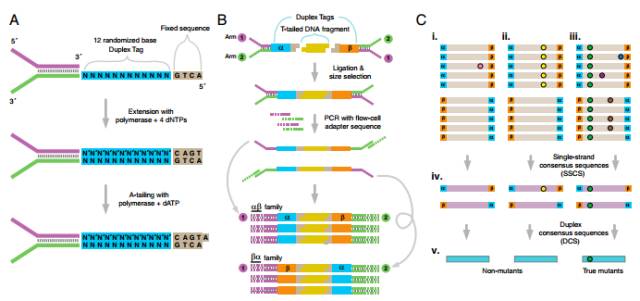
Figure 4.1: 单分子编码技术的建立是ctDNA发展史上的一个里程碑。
2012 年,Forshew T等人与Michael W等人分别发表文章,利用分子标签的原理过滤NGS中的PCR错误和测序错误,能够显著降低背景噪音,从而提高NGS检测灵敏度,使之适用于cfDNA中超低频突变的检测(关于这一技术我们会在下一期详细说明)。从此,基于分子标签原理的NGS检测成为ctDNA研究最强大的工具。由于NGS检测范围广的特点,拓展了ctDNA的应用场景,使之可以不局限于特定位点突变频率的检测,还可以进行数十上百个基因的广泛筛查,从而可以应用于药物靶点检测、耐药机制分析、早期肿瘤筛查等方向,ctDNA研究也迎来了爆发期。
2016年ASCO上,美国Guardant Health发布了史上最大规模的ctDNA检测研究结果,其Guardant360平台检测了15191例病人的17628份血液标本和398位病人的组织标本,结果发现,在83%病人的血液中能够检测到ctDNA,血检结果对肿瘤的诊断准确率可高达87% (336/386),若血液检查和组织活检相差六个月内,血检和活检的一致性可高达98%。
4.4 ctDNA逐渐走入临床应用

Figure 4.2: 若组织活检不可重复实行,应考虑进行血浆活检。”——NCCN指南,2017年第4版,非小细胞肺癌。
随着ctDNA研究的逐渐成熟,利用ctDNA进行临床检测也逐渐受到了认可。2014年9月,欧盟EMA批准利用ctDNA检测EGFR突变用于易瑞沙的伴随诊断,标志着ctDNA正式临床应用。2015年12月,《非小细胞肺癌(NSCLC)血液EGFR基因突变检测中国专家共识》于《中华医学杂志》上发表,补充了如果肿瘤标本不可评估EGFR基因状态,则可使用从血液(血浆)标本中获得的ctDNA进行评估。2016年6月,美国FDA批准罗氏cobas EGFR Mutation Test V2试剂盒作为特罗凯伴随诊断试剂盒,用于血液ctDNA评估EGFR突变情况。2016年10月,非小细胞肺癌NCCN指南也添加了液体活检推荐。
相信随着研究的成熟和样本的积累,ctDNA检测将越来越广泛的临床应用。
参考文献
- Mandel, P. & Métais, P. Les acides nucléiques du plasma sanguin chez l’homme. C. R. Seances Soc. Biol. Fil. 142, 241–243 (in French) (1948).
- Leon, S. A., Shapiro, B., Sklaroff, D. M. & Yaros, M. J. Free DNA in the serum of cancer patients and the effect of therapy. Cancer Res. 37, 646–650 (1977).
- Stroun, M. et al. Neoplastic characteristics of the DNA found in the plasma of cancer patients. Oncology 46, 318–322 (1989).
- Sidransky, D. et al. Identification of p53 gene mutations in bladder cancers and urine samples. Science 252, 706–709 (1991).
- Sidransky, D. et al. Identification of ras oncogene mutations in the stool of patients with curable colorectal tumors. Science 256, 102–105 (1992).
- Caldas, C. et al. Detection of K‑ras mutations in the stool of patients with pancreatic adenocarcinoma and pancreatic ductal hyperplasia. Cancer Res. 54, 3568–3573 (1994).
- Mao, L., Hruban, R. H., Boyle, J. O., Tockman, M. & Sidransky, D. Detection of oncogene mutations in sputum precedes diagnosis of lung cancer. Cancer Res. 54, 1634–1637 (1994).
- Takeda, S., Ichii, S. & Nakamura, Y. Detection of K‑ras mutation in sputum by mutant-allele-specific amplification (MASA). Hum. Mutat. 2, 112–117 (1993).
- Sorenson, G. D. et al. Soluble normal and mutated DNA-sequences from single-copy genes in human blood. Cancer Epidemiol. Biomarkers Prev. 3, 67–71 (1994).
- Diehl, F. et al. Circulating mutant DNA to assess tumor dynamics. Nat. Med. 14, 985–990 (2008).
- Forshew, T. et al. Noninvasive identification and monitoring of cancer mutations by targeted deep sequencing of plasma DNA. Sci. Transl Med. 4, 136ra68 (2012).
- Schmitt MW. et al. Detection of ultra-rare mutations by next-generation sequencing. Proc Natl Acad Sci U S A. 2012 Sep 4;109(36):14508-13.
- Large Study Gives Boost to Liquid Biopsy Testing for Mutations. 2016 ASCO Annual Meeting.