4 Tópicos avanzados
En esta sección se presentan algunos casos de uso específicos, que en general requieren un uso más avanzado de las herramientas introducidas en las secciones 2 y 3.
4.1 Relativización
Cada vez que queremos hacer comparaciones entre cantidades que tienen distintas escalas es importante relativizar, es decir expresarlas de manera relativa a alguna referencia. Esta referencia puede ser un total, el máximo o mínimo de un conjunto de datos, un promedio, entre otros.
Para ejemplificar esta idea, consideremos los siguientes datos de la población urbana y rural de países de Sudamérica12:
pob_sudam <- read_csv("https://raw.githubusercontent.com/rocarvaj/rcd1-uai/main/data/poblacion-sudam.csv")## Rows: 26 Columns: 3
## ── Column specification ───────────────────────────
## Delimiter: ","
## chr (2): Pais, Tipo
## dbl (1): Poblacion
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.## # A tibble: 26 × 3
## Pais Tipo Poblacion
## <chr> <chr> <dbl>
## 1 Brazil Urbana 186782772
## 2 Brazil Rural 25470378
## 3 Colombia Urbana 40617502
## 4 Colombia Rural 10154376
## 5 Argentina Urbana 41953443
## 6 Argentina Rural 3157786
## 7 Peru Urbana 25972819
## 8 Peru Rural 6904167
## 9 Venezuela Urbana 25037609
## 10 Venezuela Rural 3414219
## # ℹ 16 more rowsNotemos que esta base de datos está en formato tidy (ver Sección 2.1.1), es decir cada fila representa una observación (una población) y cada columna representa a una variable (país al que pertenece la población, tipo de población y número de personas en dicha población).
Si quisiéramos comparar la población rural y urbana entre estos países, podríamos elaborar el siguiente gráfico de columnas:
pob_sudam %>%
ggplot(aes(x = Pais, y = Poblacion, fill = Tipo)) +
geom_col(position = "dodge") +
coord_flip() +
scale_y_continuous(labels = scales::label_comma()) +
labs(title = "Población en Sudamérica",
subtitle = "Población Rural y Urbana Reportada Durante 2020",
x = "País",
y = "Población")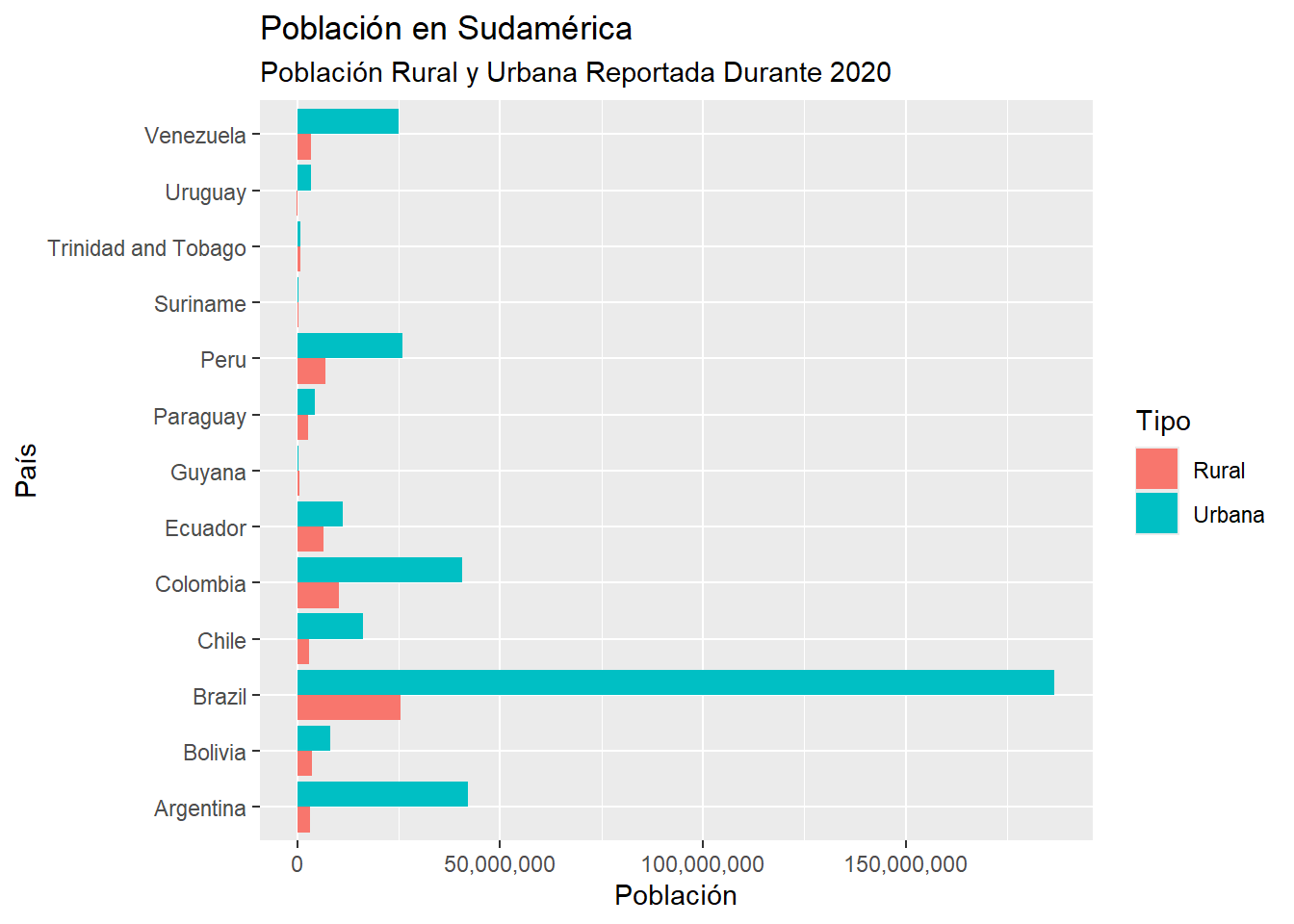
Este gráfico no es muy efectivo para comparar, por ejemplo, la población rural y urbana de Brasil con la de Guyana. Esto es debido a que las escalas de la población en ambos países es muy distinta.
Para resolver este preoblema, podemos relativizar los datos de población. Acá tenemos dos maneras distintas de relativizar:
- Con respecto al total de personas en el país.
- Con respecto al total de personas que pertenecen al tipo de población respectiva.
Primero relativicemos con respecto al total del país:
pob_sudam <- pob_sudam %>%
group_by(Pais) %>%
mutate(Poblacion_RelPais = Poblacion/sum(Poblacion))
pob_sudam## # A tibble: 26 × 4
## # Groups: Pais [13]
## Pais Tipo Poblacion Poblacion_RelPais
## <chr> <chr> <dbl> <dbl>
## 1 Brazil Urbana 186782772 0.88
## 2 Brazil Rural 25470378 0.12
## 3 Colombia Urbana 40617502 0.800
## 4 Colombia Rural 10154376 0.200
## 5 Argentina Urbana 41953443 0.930
## 6 Argentina Rural 3157786 0.0700
## 7 Peru Urbana 25972819 0.790
## 8 Peru Rural 6904167 0.210
## 9 Venezuela Urbana 25037609 0.880
## 10 Venezuela Rural 3414219 0.120
## # ℹ 16 more rowsGrafiquemos estos datos:
pob_sudam %>%
ggplot(aes(x = Pais, y = Poblacion_RelPais, fill = Tipo)) +
geom_col(position = "dodge") +
coord_flip() +
labs(title = "Población en Sudamérica",
subtitle = "Población Rural y Urbana Reportada Durante 2020",
x = "País",
y = "Porcentaje de la Población del País")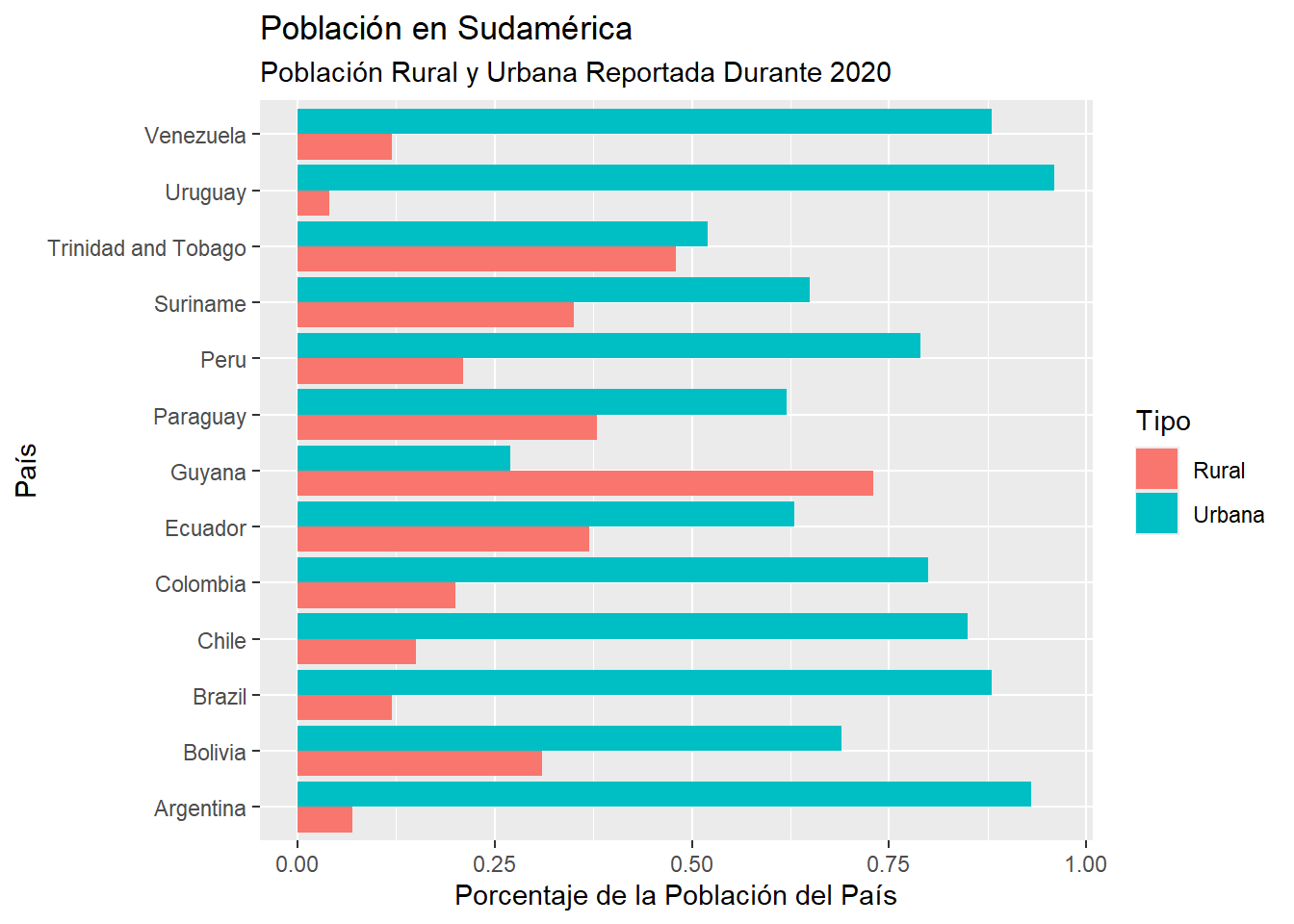
Este gráfico nos permite comparar de mejor manera las proporciones de población urbana y rural entre países. Es decir, responde a la pregunta: de la población total de cada país ¿qué porcentaje es urbana y qué porcentaje es rural?
En particular podemos observar que, a diferencia del resto de países, Guyana tiene una mayoría de población rural. Además, Uruguay y Argentina son los países con una mayor proporción de población urbana en Sudamérica.
Nota: La relativización hecha en el gráfico anterior es equivalente a haber usado
directamente la variable Población y el parámetro position = 'fill' en geom_col():
pob_sudam %>%
ggplot(aes(x = Pais, y = Poblacion, fill = Tipo)) +
geom_col(position = "fill") +
coord_flip() +
labs(title = "Población en Sudamérica",
subtitle = "Población Rural y Urbana Reportada Durante 2020",
x = "País",
y = "Porcentaje de la Población del País")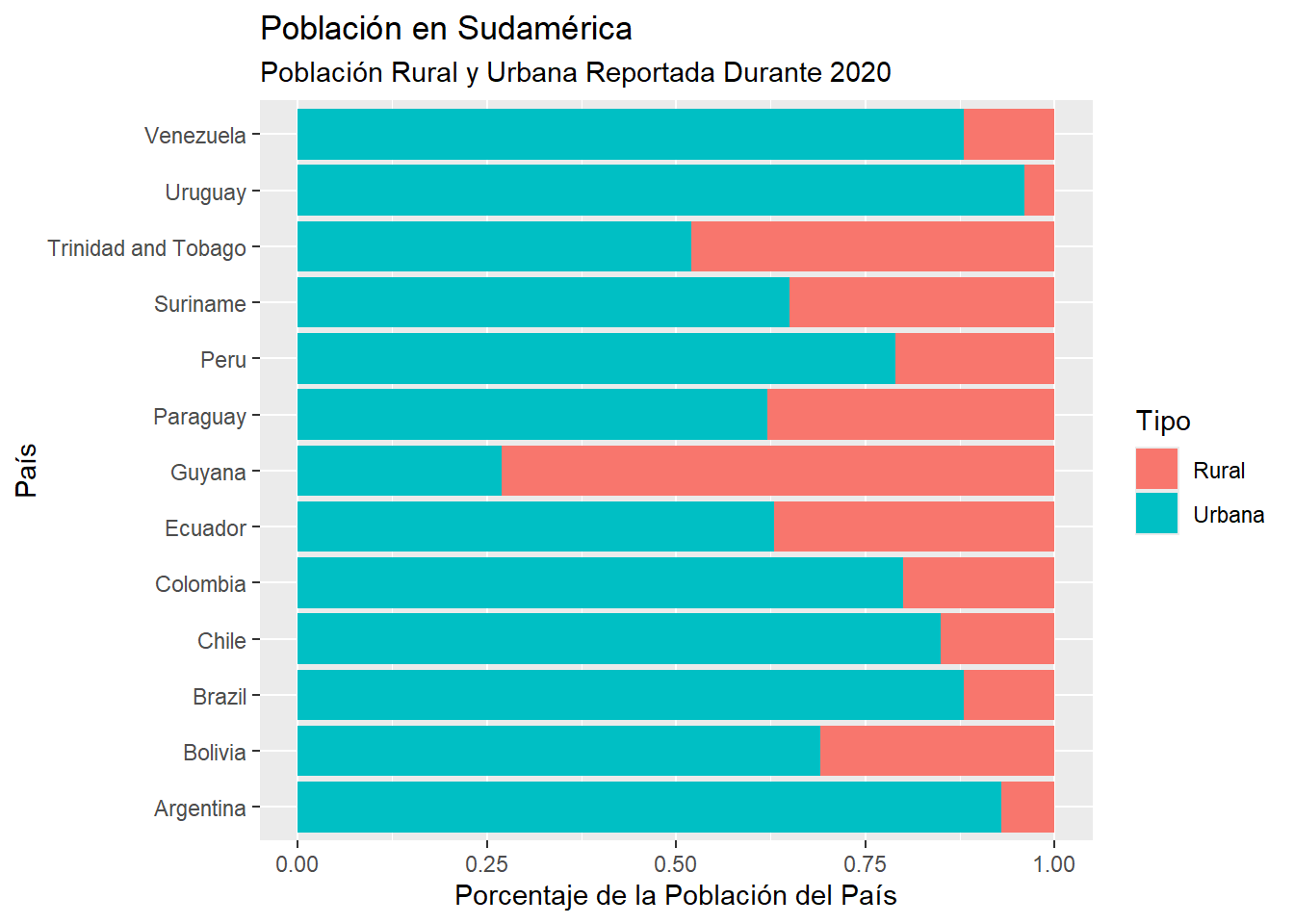
A continuación relativicemos con respecto al total de personas en Sudamérica que corresponden a cada tipo de población (urbana y rural).
pob_sudam <- pob_sudam %>%
group_by(Tipo) %>%
mutate(Poblacion_RelTipo = Poblacion/sum(Poblacion))
pob_sudam## # A tibble: 26 × 5
## # Groups: Tipo [2]
## Pais Tipo Poblacion Poblacion_RelPais Poblacion_RelTipo
## <chr> <chr> <dbl> <dbl> <dbl>
## 1 Brazil Urbana 186782772 0.88 0.512
## 2 Brazil Rural 25470378 0.12 0.384
## 3 Colombia Urbana 40617502 0.800 0.111
## 4 Colombia Rural 10154376 0.200 0.153
## 5 Argentina Urbana 41953443 0.930 0.115
## 6 Argentina Rural 3157786 0.0700 0.0476
## 7 Peru Urbana 25972819 0.790 0.0712
## 8 Peru Rural 6904167 0.210 0.104
## 9 Venezuela Urbana 25037609 0.880 0.0686
## 10 Venezuela Rural 3414219 0.120 0.0514
## # ℹ 16 more rowsGrafiquemos los datos obtenidos:
pob_sudam %>%
ggplot(aes(x = Pais, y = Poblacion_RelTipo, fill = Tipo)) +
geom_col(position = "dodge") +
coord_flip() +
labs(title = "Población en Sudamérica",
subtitle = "Población Rural y Urbana Reportada Durante 2020",
x = "País",
y = "Porcentaje del Total de Población de Cada Tipo")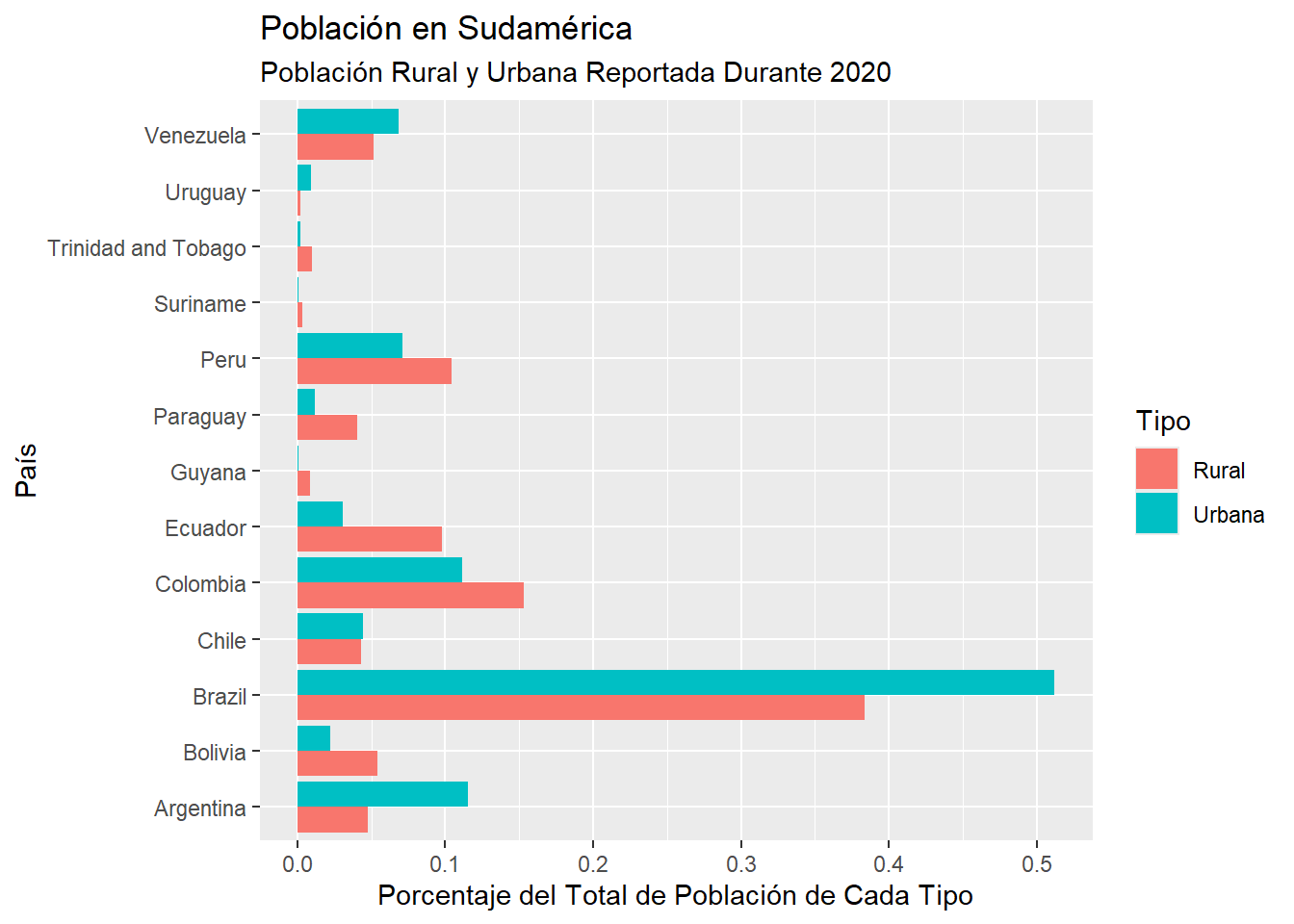
Este gráfico responde otra pregunta: ¿Cómo se reparte el total de población urbana (y rural) de Sudamérica entre los distintos países?
Aquí podemos ver que los países que concentran el mayor porcentaje de población urbana en Sudamérica son Brasil, Argentina y Colombia. Por otra parte los países que concentran un mayor porcentaje de población rural en Sudamérica son Brasil, Colombia y Perú.
Nota: La relativización hecha en el gráfico anterior también podría conseguirse
a través del parámetro position = 'fill' en geom_col(), pero cambiando el
rol de las variables Pais y Tipo:
pob_sudam %>%
ggplot(aes(x = Tipo, y = Poblacion, fill = Pais)) +
geom_col(position = "fill") +
coord_flip() +
labs(title = "Población en Sudamérica",
subtitle = "Población Rural y Urbana Reportada Durante 2020",
x = "País",
y = "Porcentaje del Total de Población de Cada Tipo")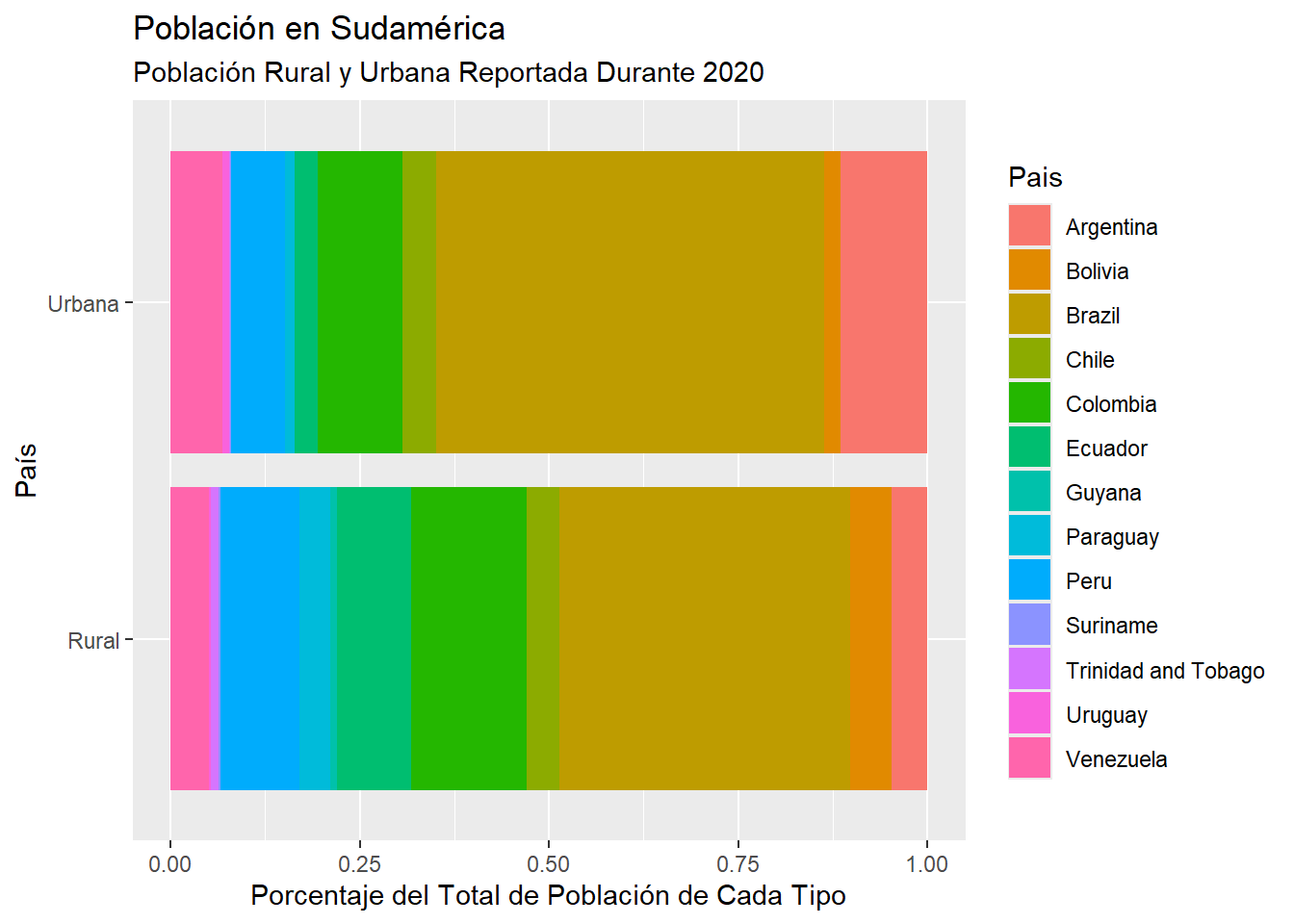
Sin embargo, vemos que esta visualización no es muy efectiva ya que el lector tiene que hacer el esfuerzo adicional de identificar a qué color corresponde cada país.
Como comentario final de esta sección, es importante notar el rol que juega
la variable que se utiliza en el comando group_by() para determinar qué
relativización se está haciendo de los datos.
4.2 Limpieza de bases de datos
Una de las dificultades que surgen al usar datos es que las bases de datos a las que tenemos acceso no siempre están listas para realizar exploración
A continuación describiremos los pasos que recomendados al limpiar una base de datos. Esto no pretende ser una lista exhaustiva de pasos para limpiar una base de datos, ya que existen otras complejidades que salen del alcance de este texto. Sin embargo creemos que contempla varios de los casos más comunes.
Comparación de tipos de variables y su significado.
Limpieza de variables según qué representan:
- Variables que representan cadenas de caracteres
- Cambiar capitalización
- Reemplazos en strings
- Variables que representan numéros
- Convertir strings con distintos formatos de número a tipo numérico
- Chequear rangos para identificar valores anormales
- Procurar consistencia en las unidades de la variable
- Convertir strings con distintos formatos de número a tipo numérico
- Variables que representan categorías
- Revisar valores únicos
- Recodificar categorías
- Convertir variables categóricas a tipo factor
- Variables que representan fechas
- Convertir strings con diversos formatos de fecha a tipo fecha.
- Variables que representan cadenas de caracteres
Ejemplificaremos todos estos pasos con la siguiente base de datos:
sucio <- read_csv("https://raw.githubusercontent.com/rocarvaj/rcd1-uai/main/data/ejemplo-limpieza-datos.csv")## Rows: 9 Columns: 5
## ── Column specification ───────────────────────────
## Delimiter: ","
## chr (5): Nombre, Sexo, Región, Nacimiento, Sueldo Mensual
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.## # A tibble: 9 × 5
## Nombre Sexo Región Nacimiento `Sueldo Mensual`
## <chr> <chr> <chr> <chr> <chr>
## 1 Rodolfo MAsculino 1 15-03-78 1304000,2
## 2 viviana Mujer 10 27-11-80 CLP 304500,3
## 3 CLAUDIA femenino 3 10/30/2001 1.201,4
## 4 Marcos Hombre 8 15-11-1970 3,300,321.4
## 5 Antonia Mujer 6 30-01-1922 5000
## 6 Tomás Hombre RM 15/10/81 1.400.000
## 7 Alfonsina Mujer RM 10-10-80 1.500.000
## 8 Felipe Hombrre 7 01-02-90 500456
## 9 Hortensia Mujer 24 13-01-78 -14499994.2.1 Comparación de tipos de variables y su significado
Primero hagamos un rápido análisis de cómo se compara el tipo asignado a cada variable por R con lo que la variable representa:
Nombre: Vemos que esta variable representa a nombres de personas. La variable ha sido identificada por R con el tipo chr, lo cual es correcto. Hay algunos aspectos de formato, como capitalización, que revisaremos más adelante.Sexo: Esta variable representa a una categoría, el sexo de cada persona. La variable fue identificada por R con el tipo chr. Esto es correcto, aunque más adelante trataremos a este tipo de variables como “factores” (un tipo especial de R para variables categóricas).Región: Esta variable representa una categoría (la región a la cual pertenece la persona). R identificó esta columna con el tipo chr, debido a la mezcla de números y strings que hay en esta columna. Más adelante decidiremos cómo tratar esta columna de manera más clara.Nacimiento: Esta variable representa la fecha de nacimiento de cada persona. Ha sido ha sido identificada como chr por R. Podemos ver que las fechas han sido ingresadas de distintas formas en esta base de datos, por lo que utilizaremos funcionalidades especiales de tidyverse para tratar este caso.Sueldo Mensual: Esta variable representa el sueldo mensual de cada una de las personas, por lo que naturalmente debería ser tratada como una variable numérica. Sin embargo, R ha identificado a esta variable con el tipo char. Analizaremos con más detención por qué pasa esto más adelante.
4.2.2 Limpieza de variables según qué representan
Variables que representan cadenas de caracteres
Consideremos la variable Nombre y veamos sus valores únicos:
## # A tibble: 9 × 1
## Nombre
## <chr>
## 1 Rodolfo
## 2 viviana
## 3 CLAUDIA
## 4 Marcos
## 5 Antonia
## 6 Tomás
## 7 Alfonsina
## 8 Felipe
## 9 HortensiaObservamos que hay algunas inconsistencias en la capitalización de los valores. Corregiremos la capitalización usando comandos del paquete especializado de tidyverse para la manipulación de cadenas de caracteres llamado stringr. Este paquete es parte del tidyverse y no debe ser cargado de forma separada.
Para transformar una palabra a capitalización de nombre propio (primera letra mayúscula), usamos
el comando str_to_title() en conjunto con mutate():
sucio <- sucio %>%
mutate(Nombre_Limpio = str_to_title(Nombre))
sucio %>%
select(Nombre, Nombre_Limpio)## # A tibble: 9 × 2
## Nombre Nombre_Limpio
## <chr> <chr>
## 1 Rodolfo Rodolfo
## 2 viviana Viviana
## 3 CLAUDIA Claudia
## 4 Marcos Marcos
## 5 Antonia Antonia
## 6 Tomás Tomás
## 7 Alfonsina Alfonsina
## 8 Felipe Felipe
## 9 Hortensia HortensiaTe instamos a revisar la documentación de stringr para más comandos de manipulación de strings.
Variables que representan numéros
Analicemos la variable Sueldo Mensual. Dado que R identificó esta variable como
string (chr), podemos usar el método parse_number()13 para intentar convertir sus valores a numéricos:
sucio %>%
mutate(Sueldo_Num = parse_number(`Sueldo Mensual`)) %>%
select(`Sueldo Mensual`, Sueldo_Num)## # A tibble: 9 × 2
## `Sueldo Mensual` Sueldo_Num
## <chr> <dbl>
## 1 1304000,2 13040002
## 2 CLP 304500,3 3045003
## 3 1.201,4 1.20
## 4 3,300,321.4 3300321.
## 5 5000 5000
## 6 1.400.000 1.4
## 7 1.500.000 1.5
## 8 500456 500456
## 9 -1449999 -1449999Notemos algunas cosas:
Los números en la tabla de datos pueden venir expresados usando distintas convenciones para los separadores de miles o decimales. Por defecto,
parse_number()utilizará las convenciones de Estados Unidos (“,” para separador de miles y “.” para decimales).Para cambiar el separador de decimales, usamos el argumento
locale = locale(decimal_mark = ",").parse_number()ignorará cualquier caracter no numérico antes o después del primer número. Es importante tener mucho cuidado con algunos símbolos que dan contexto al número en cuestion, por ejemplo el tipo de moneda (“CLP 300”) o un símbolo de porcetaje (“23%”).En el caso en que el valor de la variable no pueda ser convertido a un número,
parse_number()retornará un valor faltanteNA.
Volvamos a nuestra base de datos.
Al usar el comando parse_number() nos damos cuenta de que el separador de
decimales no es el correcto, lo corregiremos:
sucio <- sucio %>%
mutate(Sueldo_Num = parse_number(`Sueldo Mensual`,
locale = locale(decimal_mark = ",")))
sucio %>%
select(`Sueldo Mensual`, Sueldo_Num)## # A tibble: 9 × 2
## `Sueldo Mensual` Sueldo_Num
## <chr> <dbl>
## 1 1304000,2 1304000.
## 2 CLP 304500,3 304500.
## 3 1.201,4 1201.
## 4 3,300,321.4 3.3
## 5 5000 5000
## 6 1.400.000 1400000
## 7 1.500.000 1500000
## 8 500456 500456
## 9 -1449999 -1449999Ahora que hemos convertido la variable a numérica, hagamos un chequeo de su rango y la distribución de las observaciones:
| Name | Piped data |
| Number of rows | 9 |
| Number of columns | 7 |
| _______________________ | |
| Column type frequency: | |
| numeric | 1 |
| ________________________ | |
| Group variables | None |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| Sueldo_Num | 0 | 1 | 396129.1 | 931655.2 | -1449999 | 1201.4 | 304500.3 | 1304000 | 1500000 | ▂▁▇▂▆ |
Existen sueldos negativos, veamos cuáles son:
## # A tibble: 1 × 7
## Nombre Sexo Región Nacimiento `Sueldo Mensual` Nombre_Limpio Sueldo_Num
## <chr> <chr> <chr> <chr> <chr> <chr> <dbl>
## 1 Hortensia Mujer 24 13-01-78 -1449999 Hortensia -1449999Parece ser debido a un error de tipeo. Supondremos que quien ingresó los datos en esta base de datos puso un signo “-” por error. Cambiaremos ese signo:
sucio <- sucio %>%
mutate(Sueldo_Num = abs(Sueldo_Num))
sucio %>%
select(`Sueldo Mensual`, Sueldo_Num)## # A tibble: 9 × 2
## `Sueldo Mensual` Sueldo_Num
## <chr> <dbl>
## 1 1304000,2 1304000.
## 2 CLP 304500,3 304500.
## 3 1.201,4 1201.
## 4 3,300,321.4 3.3
## 5 5000 5000
## 6 1.400.000 1400000
## 7 1.500.000 1500000
## 8 500456 500456
## 9 -1449999 1449999Volvamos a revisar la distribución de esta variable:
| Name | Piped data |
| Number of rows | 9 |
| Number of columns | 7 |
| _______________________ | |
| Column type frequency: | |
| numeric | 1 |
| ________________________ | |
| Group variables | None |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| Sueldo_Num | 0 | 1 | 718351.1 | 681162.8 | 3.3 | 5000 | 500456 | 1400000 | 1500000 | ▆▃▁▁▇ |
Vemos que alguien tiene un sueldo mensual de 3.3. ¿Qué significa esto? Miremos este caso con más detención:
## # A tibble: 1 × 2
## `Sueldo Mensual` Sueldo_Num
## <chr> <dbl>
## 1 3,300,321.4 3.3Al parecer este sueldo fue escrito usando “,” como separador de miles y “.” como
separador de decimales (mientras que
el resto de los datos tenía “,” como separador de decimal). Modifiquemos esta
observación, usando una combinación de if_else() y parse_number():
sucio <- sucio %>%
mutate(Sueldo_Num = if_else(Sueldo_Num == 3.3,
parse_number(`Sueldo Mensual`,
locale = locale(decimal_mark = ".")),
Sueldo_Num))
sucio %>%
select(`Sueldo Mensual`, Sueldo_Num)## # A tibble: 9 × 2
## `Sueldo Mensual` Sueldo_Num
## <chr> <dbl>
## 1 1304000,2 1304000.
## 2 CLP 304500,3 304500.
## 3 1.201,4 1201.
## 4 3,300,321.4 3300321.
## 5 5000 5000
## 6 1.400.000 1400000
## 7 1.500.000 1500000
## 8 500456 500456
## 9 -1449999 1449999Nuevamente, miramos la distribución de los sueldos:
| Name | Piped data |
| Number of rows | 9 |
| Number of columns | 7 |
| _______________________ | |
| Column type frequency: | |
| numeric | 1 |
| ________________________ | |
| Group variables | None |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| Sueldo_Num | 0 | 1 | 1085053 | 1039962 | 1201.4 | 304500.3 | 1304000 | 1449999 | 3300321 | ▇▂▆▁▂ |
Vemos que el mínimo sueldo es 1201 y nos surge una duda, ¿cuál es la moneda en la cual están estos datos? Suponemos que los datos vienen en pesos.
Filtremos todos aquellos sueldos que están bajo el percentil 25%:
## # A tibble: 2 × 2
## `Sueldo Mensual` Sueldo_Num
## <chr> <dbl>
## 1 1.201,4 1201.
## 2 5000 5000Al parecer estas dos personas tienen sus sueldos escritos en dólares. Usemos una tasa de cambio de 1 USD = 780 CLP para convertir estos sueldos a pesos chilenos:
sucio <- sucio %>%
mutate(Sueldo_Num = if_else(Sueldo_Num < 304500,
Sueldo_Num*780,
Sueldo_Num))
sucio %>%
select(`Sueldo Mensual`, Sueldo_Num)## # A tibble: 9 × 2
## `Sueldo Mensual` Sueldo_Num
## <chr> <dbl>
## 1 1304000,2 1304000.
## 2 CLP 304500,3 304500.
## 3 1.201,4 937092
## 4 3,300,321.4 3300321.
## 5 5000 3900000
## 6 1.400.000 1400000
## 7 1.500.000 1500000
## 8 500456 500456
## 9 -1449999 1449999Importante: Nota que parte del análisis y los criterios que hemos usado en la limpieza de esta variable son basados en supuestos acerca de información externa a lo que vemos en la base de datos (por qué hay un sueldo negativo, que los sueldos deben ser expresados en CLP, etc.). En la práctica, la limpieza y manejo de una base de datos requiere tener claridad del origen de los datos y su contexto. Limpiar una base de datos sin tener claridad de qué representan las variables y sus valores puede significar seguir ensuciándola.
Realicemos una última revisión de la variable de sueldos:
| Name | Piped data |
| Number of rows | 9 |
| Number of columns | 7 |
| _______________________ | |
| Column type frequency: | |
| numeric | 1 |
| ________________________ | |
| Group variables | None |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| Sueldo_Num | 0 | 1 | 1621819 | 1207461 | 304500.3 | 937092 | 1400000 | 1500000 | 3900000 | ▆▇▁▁▃ |
Estos valores parecen ser razonables.
Variables que representan categorías
Veamos los valores únicos de la variable Sexo:
## # A tibble: 5 × 1
## Sexo
## <chr>
## 1 MAsculino
## 2 Mujer
## 3 femenino
## 4 Hombre
## 5 HombrrePodemos ver que hay varios valores incorrectos. Usaremos como etiquetas de categoría
a “Mujer” y “Hombre”. Usemos el comando str_replace() del paquete stringr para
reemplazar los valores incorrectos:
sucio <- sucio %>%
mutate(Sexo_Limpio = str_replace(Sexo, "MAsculino", "Hombre")) %>%
mutate(Sexo_Limpio = str_replace(Sexo_Limpio, "femenino", "Mujer")) %>%
mutate(Sexo_Limpio = str_replace(Sexo_Limpio, "Hombrre", "Hombre"))Volvemos a chequear los valores únicos:
## # A tibble: 2 × 1
## Sexo_Limpio
## <chr>
## 1 Hombre
## 2 MujerHasta ahora hemos tratado las variables categóricas como variables de tipo chr (cadena de caracteres), sin embargo R provee de un tipo especial para variables categóricas llamado “factor”.
Transformemos la variable de sexo al tipo factor, usando el comando factor():
## # A tibble: 9 × 2
## Sexo Sexo_Limpio
## <chr> <fct>
## 1 MAsculino Hombre
## 2 Mujer Mujer
## 3 femenino Mujer
## 4 Hombre Hombre
## 5 Mujer Mujer
## 6 Hombre Hombre
## 7 Mujer Mujer
## 8 Hombrre Hombre
## 9 Mujer MujerEste tipo es conveniente porque permite definir cuáles son los posibles valores que tomará una variable (llamados niveles) y ordenarlos de maneras que tengan sentido en un determinado contexto.
Para ejemplificar esto, veamos la variable Región y sus valores únicos:
## # A tibble: 8 × 1
## Región
## <chr>
## 1 1
## 2 10
## 3 3
## 4 8
## 5 6
## 6 RM
## 7 7
## 8 24Aquí los valores numéricos son la numeración tradicional que se entrega a las regiones de Chile, salvo por la Región Metropolitana que recibe el código “RM”.
Es común que algunas bases de datos tengan los valores de sus variables codificados, este es por ejemplo el caso de datos provenientes de algunas encuestas. Una manera de mejorar la legibilidad y el análisis de estos datos es recodificando variables.
Para recodificar variables en R, usamos el comando recode(). Primero debemos
definir un vector que cumple el rol de diccionario, al traducir los valores
de la variable de un códgio a otro.
En el caso de las regiones, usamos el siguiente diccionario:
dicc_regiones <- c(
"1" ="Tarapacá",
"2" ="Antofagasta",
"3" ="Atacama",
"4" ="Coquimbo",
"5" ="Valparaíso",
"6" ="Libertador General Bernardo O’Higgins",
"7" ="Maule",
"8" ="Biobío",
"9" ="La Araucanía",
"10" ="Los Lagos",
"11" ="Aysén del General Carlos Ibáñez del Campo",
"12" ="Magallanes y Antártica Chilena",
"RM" ="Metropolitana",
"14" ="Los Ríos",
"15" ="Arica y Parinacota",
"16" ="Ñuble"
)Luego realizando la recodificación con mutate() y recode():
sucio <- sucio %>%
mutate(Region_Nombre = recode(Región, !!!dicc_regiones))
sucio %>%
select(Región, Region_Nombre)## # A tibble: 9 × 2
## Región Region_Nombre
## <chr> <chr>
## 1 1 Tarapacá
## 2 10 Los Lagos
## 3 3 Atacama
## 4 8 Biobío
## 5 6 Libertador General Bernardo O’Higgins
## 6 RM Metropolitana
## 7 RM Metropolitana
## 8 7 Maule
## 9 24 24El uso de “!!!” cumple la función
de “desarmar” el vector diccionario y entregar cada uno de sus componentes como
un argumento distinto al comando recode().
Veamos los valores únicos de la variable recodificada:
## # A tibble: 8 × 1
## Region_Nombre
## <chr>
## 1 Tarapacá
## 2 Los Lagos
## 3 Atacama
## 4 Biobío
## 5 Libertador General Bernardo O’Higgins
## 6 Metropolitana
## 7 Maule
## 8 24Hay un valor que no fue recodificado, ya que no existe la región “24” en el diccionario.
Como no sabemos a qué región pertenece esta persona, podemos especificar cuál es el valor
que se asignará a los códigos no encontrados en el diccionario, usando el argumento .default =
en el comando recode().
Por ejemplo, asignemos un valor faltante NA:
sucio <- sucio %>%
mutate(Region_Nombre = recode(Región, !!!dicc_regiones, .default = NA_character_))
sucio %>%
select(Región, Region_Nombre)## # A tibble: 9 × 2
## Región Region_Nombre
## <chr> <chr>
## 1 1 Tarapacá
## 2 10 Los Lagos
## 3 3 Atacama
## 4 8 Biobío
## 5 6 Libertador General Bernardo O’Higgins
## 6 RM Metropolitana
## 7 RM Metropolitana
## 8 7 Maule
## 9 24 <NA>Transformemos ahora la variable Region_Nombre al tipo factor:
sucio <- sucio %>%
mutate(Region_Nombre = factor(Region_Nombre))
sucio %>%
select(Región, Region_Nombre)## # A tibble: 9 × 2
## Región Region_Nombre
## <chr> <fct>
## 1 1 Tarapacá
## 2 10 Los Lagos
## 3 3 Atacama
## 4 8 Biobío
## 5 6 Libertador General Bernardo O’Higgins
## 6 RM Metropolitana
## 7 RM Metropolitana
## 8 7 Maule
## 9 24 <NA>Una de las ventajas del tipo factor es que permite definir un orden para los posibles
valores de la variable.
Podemos especificar un orden deseado a través del argumento levels en el comando factor, el cual recibe un
vector con los niveles.
Creemos un vector con las regiones ordenadas de norte a sur:
niveles_regiones <- c(
"Arica y Parinacota",
"Tarapacá",
"Antofagasta",
"Atacama",
"Coquimbo",
"Valparaíso",
"Metropolitana",
"Libertador General Bernardo O’Higgins",
"Maule",
"Ñuble",
"Biobío",
"La Araucanía",
"Los Ríos",
"Los Lagos",
"Aysén del General Carlos Ibáñez del Campo",
"Magallanes y Antártica Chilena"
)Ahora, repetimos la transformación a factor:
sucio <- sucio %>%
mutate(Region_Nombre = factor(Region_Nombre,
levels = niveles_regiones))
sucio %>%
select(Región, Region_Nombre)## # A tibble: 9 × 2
## Región Region_Nombre
## <chr> <fct>
## 1 1 Tarapacá
## 2 10 Los Lagos
## 3 3 Atacama
## 4 8 Biobío
## 5 6 Libertador General Bernardo O’Higgins
## 6 RM Metropolitana
## 7 RM Metropolitana
## 8 7 Maule
## 9 24 <NA>Podemos usar la función count() para ver el orden en el que son definidos los
niveles de este factor:
## # A tibble: 8 × 2
## Region_Nombre n
## <fct> <int>
## 1 Tarapacá 1
## 2 Atacama 1
## 3 Metropolitana 2
## 4 Libertador General Bernardo O’Higgins 1
## 5 Maule 1
## 6 Biobío 1
## 7 Los Lagos 1
## 8 <NA> 1¿Pero cuál es la ventaja de usar un factor? Algunas de estas ventajas son:
La salida de comandos como
count(), vendrá ordenada usando el orden en el que fueron ingresados los niveles.Gráficos en ggplot2 que tengan a la variable factor en un eje, la presentarán en el orden en el que fueron ingresados los niveles.
Si además agregamos el argumento
ordered = TRUEal comandofactor(), podemos usar los comparadores de orden<,<=,>y>=para filtrar datos según el orden de la variable factor.
Por ejemplo, si queremos todos los
datos de personas que estén al norte de la región de O’Higgins. Definamos
la variable Region_Nombre como ordenada:
sucio <- sucio %>%
mutate(Region_Nombre = factor(Region_Nombre,
levels = niveles_regiones,
ordered = TRUE))
sucio %>%
select(Región, Region_Nombre)## # A tibble: 9 × 2
## Región Region_Nombre
## <chr> <ord>
## 1 1 Tarapacá
## 2 10 Los Lagos
## 3 3 Atacama
## 4 8 Biobío
## 5 6 Libertador General Bernardo O’Higgins
## 6 RM Metropolitana
## 7 RM Metropolitana
## 8 7 Maule
## 9 24 <NA>Y filtramos de la siguiente manera:
## # A tibble: 4 × 9
## Nombre Sexo Región Nacimiento `Sueldo Mensual` Nombre_Limpio Sueldo_Num
## <chr> <chr> <chr> <chr> <chr> <chr> <dbl>
## 1 Rodolfo MAsculi… 1 15-03-78 1304000,2 Rodolfo 1304000.
## 2 CLAUDIA femenino 3 10/30/2001 1.201,4 Claudia 937092
## 3 Tomás Hombre RM 15/10/81 1.400.000 Tomás 1400000
## 4 Alfonsina Mujer RM 10-10-80 1.500.000 Alfonsina 1500000
## # ℹ 2 more variables: Sexo_Limpio <fct>, Region_Nombre <ord>Nota: También es posible recodificar una variable y directamente transformarla
a factor, usando el comando [recode_factor()](https://dplyr.tidyverse.org/reference/recode.html).
En este caso, el orden de los niveles estará dado por el orden de los elementos del
diccionario utilizado para recodificar.
Variables que representan fechas
Transformamos una columna con fechas, usando el paquete lubridate (parte de tidyverse).
Cargamos lubridate con:
Lubridate incluye un conjunto de comandos que permiten procesar cadenas de caracteres o números que contienen fechas en algún formato y las convierten en datos de tipo fecha (date) en R.
Para usar estas funciones, debemos reconocer primero en qué formato vienen las fechas en la tabla de datos que estamos analizando, identificando el día (d), mes (m) y año (y). Luego, seleccionamos la función que corresponda de esta lista:
dmy(): La fecha viene en formato día, mes, año.mdy(): La fecha viene en formato mes, día, año.ymd(): La fecha viene en formato año, mes, día.
Estas son sólo algunos de los comandos que lubridate provee para transformar fechas escritas como números o cadenas de caracteres a datos de tipo fecha. Puedes ver otras funciones incluyendo algunas para transformar fechas con horas, minuto y segundos, en la documentación de lubridate.
Veamos cómo aplicar esto a nuestra base de datos “sucia”. A partir de las fechas
en la columna Nacimiento, construiremos una nueva columna llamada Nacimiento_Date
con estas fechas convertidas al tipo date:
## Warning: There was 1 warning in `mutate()`.
## ℹ In argument: `Nacimiento_Date =
## dmy(Nacimiento)`.
## Caused by warning:
## ! 1 failed to parse.## # A tibble: 9 × 2
## Nacimiento Nacimiento_Date
## <chr> <date>
## 1 15-03-78 1978-03-15
## 2 27-11-80 1980-11-27
## 3 10/30/2001 NA
## 4 15-11-1970 1970-11-15
## 5 30-01-1922 1922-01-30
## 6 15/10/81 1981-10-15
## 7 10-10-80 1980-10-10
## 8 01-02-90 1990-02-01
## 9 13-01-78 1978-01-13Algunas cosas importantes que notar:
Lubridate nos advierte que una observación no puso ser convertido correctamente. Esto se debe a que no está en el formato que especificamos al llamar al comando
dym()(día, mes, año) o que simplemente o que simplemente no es una fecha válida. Lubridate dejará como resultado un dato faltanteNApara esta fila.No existe una manera totalmente automática de reconocer en qué formato vienen las fechas. Necesariamente tenemos que reconocer el formato de las fechas, usando una inspección visual.
Independientemente del orden en el que vengas los componentes de una fecha (día, mes, año), lubridate es bastante flexible al momento de reconocer distintos formatos de fechas. Por ejemplo, todos los comandos a continuación generarán la fecha 1 de enero de 1970:
dmy("01-01-1970")dmy("01/01/1970")dmy(01011970)dmy("1 January, 1970")dmy("1 Jan, 1970")
4.3 Cálculos con factor de expansión en encuestas
Las encuestas son herramientas usadas para recopilar información acerca de un grupo grande de personas. A diferencia de un censo, en donde se busca entrevistar a toda una población, en una encuesta se entrevista a una muestra (subconjunto de personas en la población).
Con el fin de inferir información acerca de la población, se calcula un factor de expansión para cada observación, el cual indica el número de personas en la población a las cuales esta representa. Se calcula como el número de personas en la población, dividido por el número de observaciones en la muestra.
En el caso de encuestas estratificadas, en donde se definen estratos o categorías de personas y se realiza un muestreo dentro de cada uno ellos, el factor de expansión se calcula relativo al estrato al cual la observación pertenece.
Al momento de hacer ciertos cálculos con los datos de una encuesta, debemos tomar en cuenta estos factores de expansión.
Usaremos a modo de ejemplo, una encuesta ficticia realizada a 20 odontólogos de un país ficticio. A cada uno de ellos se le preguntó cuántos años de experiencia tienen, cuántos tratamientos de conducto realizaron en el último mes y cuál es su sueldo mensual (en USD).
encuesta <- read_csv("https://raw.githubusercontent.com/rocarvaj/rcd1-uai/main/data/ejemplo-encuesta.csv")## # A tibble: 20 × 4
## Años_Experiencia Trat_Conducto_Mes Sueldo Factor_Exp
## <dbl> <dbl> <dbl> <dbl>
## 1 5 1 1950 2500
## 2 3 6 1825 962
## 3 1 4 1369 750
## 4 1 3 1486 600
## 5 6 11 2275 840
## 6 4 12 1884 405
## 7 6 8 2170 620
## 8 1 6 1601 1000
## 9 6 12 1989 2500
## 10 6 11 2256 330
## 11 2 1 1736 405
## 12 5 5 2162 962
## 13 4 6 2076 415
## 14 2 4 1736 330
## 15 2 4 1736 750
## 16 6 4 2530 405
## 17 5 7 2069 1200
## 18 5 12 2168 840
## 19 3 5 1543 620
## 20 6 9 2170 330El factor de expansión de esta encuesta está contenido en la variable Factor_Exp.
A continuación se muestra cómo realizar algunas cálculos usando factores de expansión, de modo de inferir ciertas cantidades respecto de la población.
4.3.1 Conteos
Para inferir el número de personas de la población a partir de las observaciones en la muestra, podemos usar dos maneras:
- Sumando la variable que contiene el factor de expansión con los comandos
summarise()ysum(). - Usando el comando
count()y el argumentowt = factor_expansion, en dondefactor_expansiones el nombre de la variable que contiene el factor de expansión.
4.3.2 Totales o Sumas
En el caso de inferir la suma de una variable sobre toda la población, debemos sumar dicha variable para cada observación en la muestra pero ponderándola por el factor de expansión respectivo.
4.3.3 Promedios
En el caso de inferir el promedio de una variable sobre toda la problación, debemos calcular un promedio ponderado de dicha variable por el factor de expansión.
Esto podemos hacerlo de dos maneras:
- Utilizando la función
summarise()en conjunto conweighted.mean(). - Utilizando la función
summarise(), multiplicando la variable por el factor de expansión y luego diviendo por la suma total de los factores de expansión.
4.3.4 Percentiles
El cálculo del percentil \(p\) para una variable se realiza de la siguiente manera:
- Se ordenan los datos según la variable.
- Para cada valor de la variable, se calcula la suma de los factores de expansión de todos los datos que están por debajo de este.
- Se identifica el mínimo valor de la variable tal que la suma anterior sea al menos \(p\)% de la suma total del factor de expansión.
Ejemplo
Calculemos el percentil 0.75 de la variable Sueldo:
encuesta %>%
arrange(Sueldo) %>%
mutate(cum_fact_cal = cumsum(Factor_Exp)) %>%
filter(cum_fact_cal >= 0.75 * sum(Factor_Exp)) %>%
head(1) %>%
select(Sueldo)## # A tibble: 1 × 1
## Sueldo
## <dbl>
## 1 2162Es decir, un 75% de los odontólogos del país ficticio gana menos que este sueldo.
4.3.5 Cálculos por grupo
Notemos que todos estos cálculos es posible realizarlos para cada uno de los
grupos determinados por alguna variable (usando el comando group_by()).
Ejemplos
Calculemos el número total de odontólogos para cada número de años de experiencia:
## # A tibble: 6 × 2
## # Groups: Años_Experiencia [6]
## Años_Experiencia n
## <dbl> <dbl>
## 1 1 2350
## 2 2 1485
## 3 3 1582
## 4 4 820
## 5 5 5502
## 6 6 5025Calculemos el sueldo promedio para cada número de años de experiencia:
encuesta %>%
group_by(Años_Experiencia) %>%
summarise(Sueldo_Promedio = weighted.mean(Sueldo, Factor_Exp))## # A tibble: 6 × 2
## Años_Experiencia Sueldo_Promedio
## <dbl> <dbl>
## 1 1 1498.
## 2 2 1736
## 3 3 1714.
## 4 4 1981.
## 5 5 2046.
## 6 6 2132.Ejercicios
Usando los datos de la encuesta a odontólogos, realiza los siguientes ejercicios:
Calcula el número de odontólogos en la población que tienen un sueldo mensual de menos de $1600.
Calcula el promedio y el percentil 95% de número de tratamientos de conducto al mes que realizan los odontólogos.
Calcula la mediana de sueldo para cada número de años de experiencia de los odontólogos.