Capítulo 6 Trabajar con datos
6.1 Resumen
En este capítulo, vamos a revisar algunas de las principales operaciones que tenemos que realizar con bases de datos para ordenarlas y para hacer algunos análisis. Entre estas se encuentran hacer subconjuntos de datos, lidiar con datos no disponibles, agrupar observaciones y unir bases de datos. Las herramientas que vamos a utilizar para hacer estas operaciones están principalmente contenidas en la metalibrería tidyverse, en particular las librerías dplyr y tidyr.
- Principales conceptos: hacer subconjuntos de datos; agrupar observaciones; unir bases de datos relacionales.
- Principales funciones:
filter();select();group_by();summarize();left_join().
6.1.1 Librerías
Vamos a utilizar las siguientes librerías:
library(tidyverse)## Warning: package 'tidyverse' was built under R version 4.0.3## -- Attaching packages --------------------------------------- tidyverse 1.3.0 --## v ggplot2 3.3.2 v purrr 0.3.4
## v tibble 3.0.4 v dplyr 1.0.2
## v tidyr 1.1.2 v stringr 1.4.0
## v readr 1.3.1 v forcats 0.5.0## Warning: package 'tibble' was built under R version 4.0.3## -- Conflicts ------------------------------------------ tidyverse_conflicts() --
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()library(gapminder) # datos
library(readxl) # cargar .xlsx
library(writexl) # guardar .xlsx
library(knitr) # tablas## Warning: package 'knitr' was built under R version 4.0.3library(haven) # cargar .dta
library(janitor) # limpiar nombres de variables##
## Attaching package: 'janitor'## The following objects are masked from 'package:stats':
##
## chisq.test, fisher.testlibrary(countrycode) # codigos para combinar bases de datos6.1.2 Datos
Debemos descargar los siguientes archivos de datos y guardarlos en la carpeta /data de nuestro proyecto:
- Polity IV: link. Para descargar, hacer click en “Download”.
- Database of Political Institutions, 2017: link. Para descargar, hacer click en “Download”.
- Homicidios en Medellín: link. Para descargar, hacer click derecho, “Guardar como…”.
- Comunas de Medellín: link. Para descargar, hacer click derecho, “Guardar como…”.
6.2 Cargar bases de datos
Para efectos de nuestra discusión, una base de datos es una recopilación de información en formato rectangular, con filas (que representan casos u observaciones) y columnas (que representan variables o características de esos casos). Las celdas contienen información sobre características de cada caso. La mayor parte de los análisis estadísticos se realizan al hacer operaciones o aplicar funciones a bases de datos de este tipo.
Como mencionamos anteriormente, vamos a trabajar con distintas librerías del tidyverse, así que empecemos por cargarlas a la sesión. Estaremos utilizando varias funciones de dplyr y tidyr para transformar bases de datos y organizarlos de tal manera que podamos realizar análisis exploratorios.
6.2.1 Datos incluidos en R
R incluye unas cuantas bases de datos que podemos usar sin descargar primero - ya están incluidas y disponibles. La lista es extensa e incluye:
AirPassengers: viajeros mensuales en aerolíneas americanas, 1949-1960.Titanic: sobrevivientes del hundimiento del RMS Titanic.mtcars: tests de carros de la revista MotorTrend.airquality: calidad del aire en New York.iris: especies de plantas iris.
Otras vienen en librerías adicionales. Como cargamos tidyverse, podemos mirar economics, una base de datos macroeconómicos básicos de Estados Unidos:
economics## # A tibble: 574 x 6
## date pce pop psavert uempmed unemploy
## <date> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1967-07-01 507. 198712 12.6 4.5 2944
## 2 1967-08-01 510. 198911 12.6 4.7 2945
## 3 1967-09-01 516. 199113 11.9 4.6 2958
## 4 1967-10-01 512. 199311 12.9 4.9 3143
## 5 1967-11-01 517. 199498 12.8 4.7 3066
## 6 1967-12-01 525. 199657 11.8 4.8 3018
## 7 1968-01-01 531. 199808 11.7 5.1 2878
## 8 1968-02-01 534. 199920 12.3 4.5 3001
## 9 1968-03-01 544. 200056 11.7 4.1 2877
## 10 1968-04-01 544 200208 12.3 4.6 2709
## # ... with 564 more rowsEsta base de datos contiene datos en serie de tiempo -mes a mes- del desempleo en Estados Unidos, de julio de 1967 a abril de 2015 (la columna unemploy). Son en total 574 observaciones. ¿Cuál ha sido el comportamiento de este indicador en este periodo? Utilicemos ggplot2 para visualizar esta serie de tiempo.
Calculamos la tasa de desempleo dentro de la misma función (unemploy/pop) y graficamos el resultado a través del tiempo. Podemos observar claramente un pico durante la “Gran Recesión” (2007-2009).
# datos y variables a usar
ggplot(data = economics, aes(x = date, y = unemploy/pop)) +
# tipo de gráfica: línea
geom_line(color = "darkred") 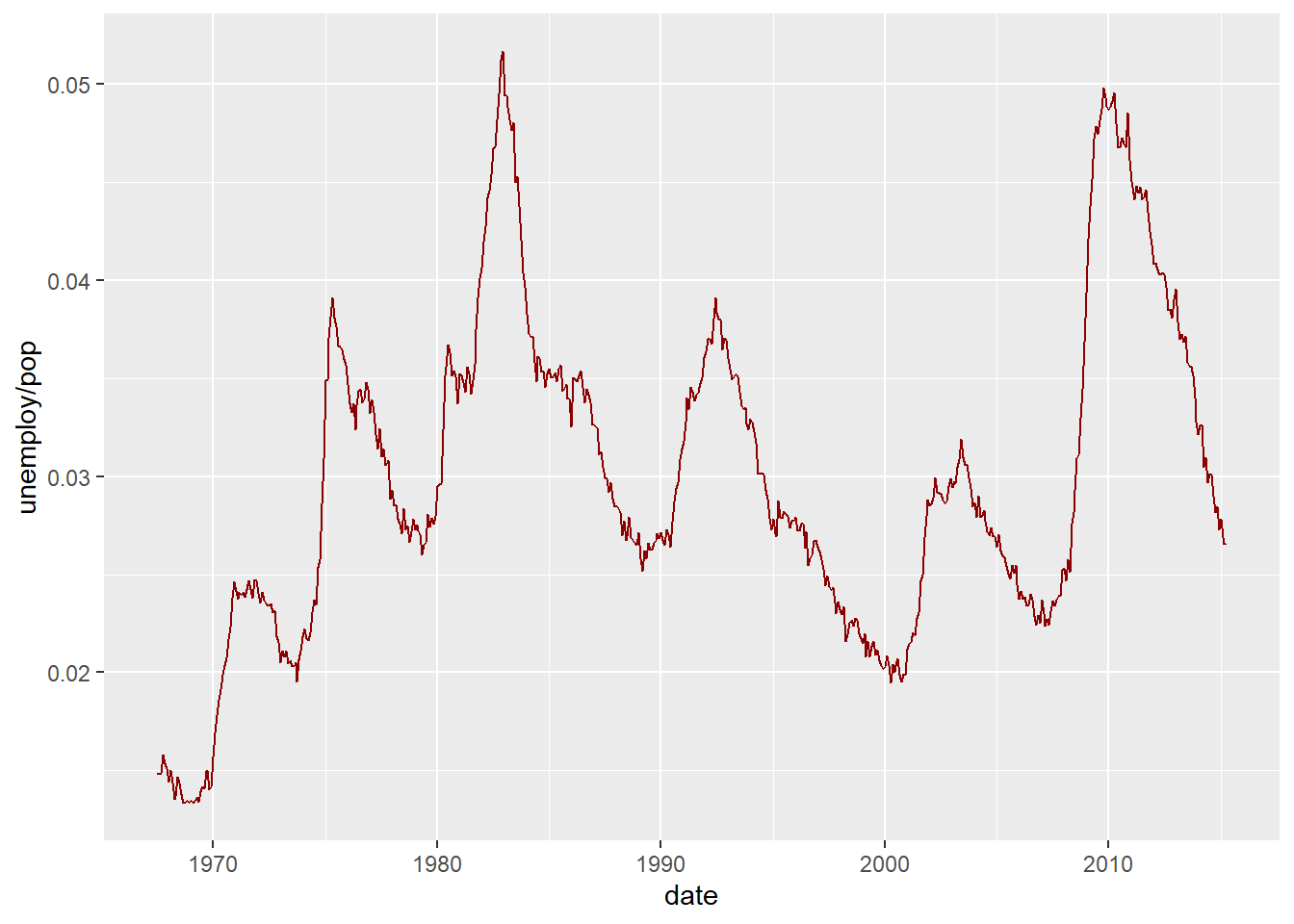
Otras librerías vienen con sus propias bases de datos. Aquí cargamos datos de la librería gapminder, que se deriva del trabajo educativo de la Gapminder Foundation. Esta librería contiene una base de datos llamada gapminder también. Veamos 10 observaciones aleatorias de esta base de datos, usando la función sample_n().
sample_n(gapminder, size = 10)## # A tibble: 10 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Singapore Asia 2002 78.8 4197776 36023.
## 2 Mauritania Africa 1957 42.3 1076852 846.
## 3 Finland Europe 1992 75.7 5041039 20647.
## 4 Kuwait Asia 1972 67.7 841934 109348.
## 5 Argentina Americas 1992 71.9 33958947 9308.
## 6 Colombia Americas 2007 72.9 44227550 7007.
## 7 Japan Asia 1977 75.4 113872473 16610.
## 8 Bolivia Americas 1992 60.0 6893451 2962.
## 9 Botswana Africa 1967 53.3 553541 1215.
## 10 India Asia 2007 64.7 1110396331 2452.Vemos que hay información sobre expectativa de vida al nacer (lifeExp) y el PIB per cápita (gdpPercap) para 142 países en intervalos de 5 años. ¿Cuál es la relación entre estas dos variables? Podemos hacer una gráfica de dispersión:
# datos y variables
ggplot(data = gapminder, aes(x = gdpPercap, y = lifeExp)) +
# tipo de gráfica, puntos con con opacidad 50%
geom_point(alpha = 0.5, color = "darkblue") 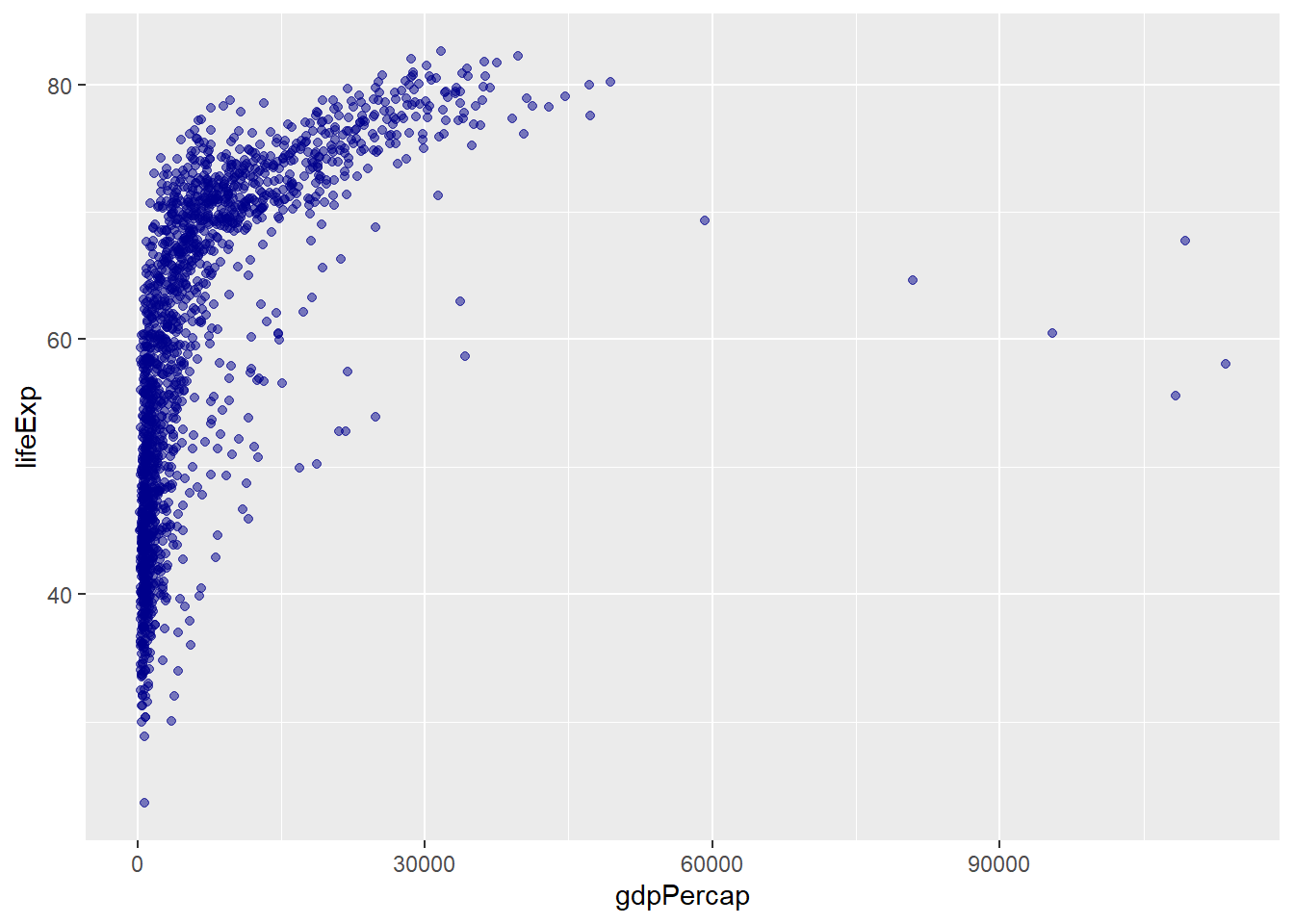
Parece que hay una relación positiva, pero no estrictamente lineal. Más bien, puede ser de tipo logarítmico, indicando rendimientos decrecientes. Nuevamente, podemos hacer la transformación de la variable gdpPercap directamente en la función ggplot() usando log():
# datos y variables
ggplot(data = gapminder, aes(x = log(gdpPercap), y = lifeExp)) +
# tipo de gráfica, puntos con con opacidad 50%
geom_point(alpha = 0.5, color = "darkgreen")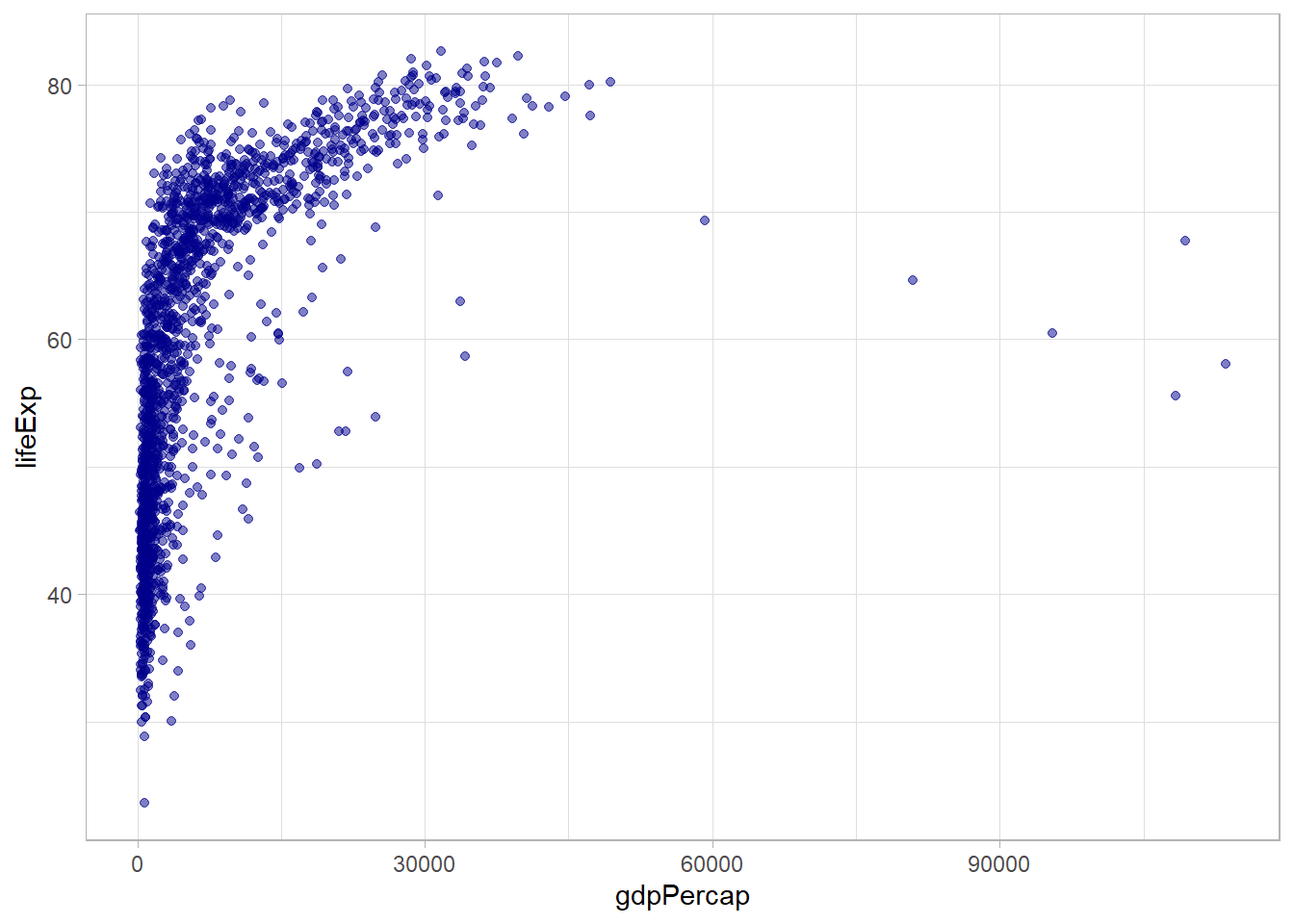
Por otro lado, si nos interesa ver la frecuencia de una variable categórica, podríamos contar cuántas observaciones (países-año) hay por continente usando la función count(), algo similar a lo que hicimos con table() en capítulos anteriores:
count(gapminder, continent)## # A tibble: 5 x 2
## continent n
## <fct> <int>
## 1 Africa 624
## 2 Americas 300
## 3 Asia 396
## 4 Europe 360
## 5 Oceania 246.2.2 Archivos locales
Es más frecuente que estemos interesados en trabajar con otros datos que recogimos o descargamos de otras fuentes. Para esto, debemos tener el archivo de datos ubicado en una carpeta en nuestro equipo. Si estamos en RStudio Cloud, debemos empezar por subirlos y ubicarlos en una carpeta dentro del proyecto.9 Si queremos ver qué archivos ya hay en una carpeta, podemos usar la función list.files(), dándole la dirección de la carpeta:
list.files("data/")## [1] "cede-agro.csv" "cede-conf.csv"
## [3] "cede-edu.csv" "cede-gen.csv"
## [5] "cede-gob.csv" "cede-salud.csv"
## [7] "codebooks" "DahlDims.sav"
## [9] "data.zip" "datos_ingreso.csv"
## [11] "datos_polity_dpi.csv" "datos_taller1.csv"
## [13] "DPI2017.dta" "gapminder_america.xlsx"
## [15] "gp0070_cronograma_20211.xlsx" "mde_df.csv"
## [17] "mde_homicidio.csv" "nafta.dta"
## [19] "nes_2004_data.csv" "OECD_country_data.csv"
## [21] "OECD_country_data.xls" "p4v2017.xls"
## [23] "p5v2018.xls" "riverside_final.csv"
## [25] "wb_tidy.csv"Vemos que en esta carpeta ya hay varios archivos. Dependiendo del tipo de archivo (Excel, CSV, de Stata, etc.) utilizamos una función distinta. Las librerías readr, readxl y haven del tidyverse son nuestras amigas.
Carguemos los datos del proyecto Polity IV sobre democracia institucional. Como están en formato Excel 97-03 (.xls), usamos read_excel() de la librería readxl y le asignamos un nombre al objeto (los datos) que estamos creando.10
polity4 <- read_excel("data/p4v2017.xls")Para confirmar que todo está bien, revisemos las primeras filas del objeto que creamos:
polity4## # A tibble: 17,395 x 36
## cyear ccode scode country year flag fragment democ autoc polity polity2
## <dbl> <dbl> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 21800 2 USA United~ 1800 0 NA 7 3 4 4
## 2 21801 2 USA United~ 1801 0 NA 7 3 4 4
## 3 21802 2 USA United~ 1802 0 NA 7 3 4 4
## 4 21803 2 USA United~ 1803 0 NA 7 3 4 4
## 5 21804 2 USA United~ 1804 0 NA 7 3 4 4
## 6 21805 2 USA United~ 1805 0 NA 7 3 4 4
## 7 21806 2 USA United~ 1806 0 NA 7 3 4 4
## 8 21807 2 USA United~ 1807 0 NA 7 3 4 4
## 9 21808 2 USA United~ 1808 0 NA 7 3 4 4
## 10 21809 2 USA United~ 1809 0 NA 9 0 9 9
## # ... with 17,385 more rows, and 25 more variables: durable <dbl>, xrreg <dbl>,
## # xrcomp <dbl>, xropen <dbl>, xconst <dbl>, parreg <dbl>, parcomp <dbl>,
## # exrec <dbl>, exconst <dbl>, polcomp <dbl>, prior <dbl>, emonth <dbl>,
## # eday <dbl>, eyear <dbl>, eprec <dbl>, interim <dbl>, bmonth <dbl>,
## # bday <dbl>, byear <dbl>, bprec <dbl>, post <dbl>, change <dbl>, d4 <dbl>,
## # sf <dbl>, regtrans <dbl>Parece que todo está bien. Miremos la distribución de la variable polity2, el popular indicador combinado de democracia de Polity IV. Aquí volvemos a usar la librería ggplot2 en vez de las funciones de base, como plot() y barplot():
# datos y variables
ggplot(data = polity4, aes(x = polity2)) +
# tipo de gráfica y ancho de las barras
geom_histogram(binwidth = 1)## Warning: Removed 236 rows containing non-finite values (stat_bin).
Vemos que hay más casos de democracia (valores altos del indicador) y de autocracia (valores bajos) que anocracias (valores medios).
6.2.3 Descarga web
Por último, carguemos unos datos directamente de la web, sin tener que descargarlos primero. El Uppsala Conflict Data Program (UCDP) tiene una de las bases de datos más completas sobre conflicto armado y está fácilmente disponible para su uso. Con read_csv() podemos darle a R el URL del archivo y cargarlo como un objeto nuevo:
ucdp <- read_csv("http://ucdp.uu.se/downloads/ucdpprio/ucdp-prio-acd-191.csv")UCDP distingue entre cuatro tipos de conflicto armado: interestatal, extraestatal, interno e interno internacionalizado. ¿Cuántos casos hay de cada tipo en la base de datos? Si leemos el libro de códigos de la base de datos, vemos que la variable type_of_conflict toma cuatro valores: 1 (inter-), 2 (extra-), 3 (intra-) y 4 (intra- internacionalizado). La prevalencia global de los conflictos armados internos es clara:
# datos y variables
ggplot(ucdp, aes(type_of_conflict)) +
# tipo de gráfica
geom_bar()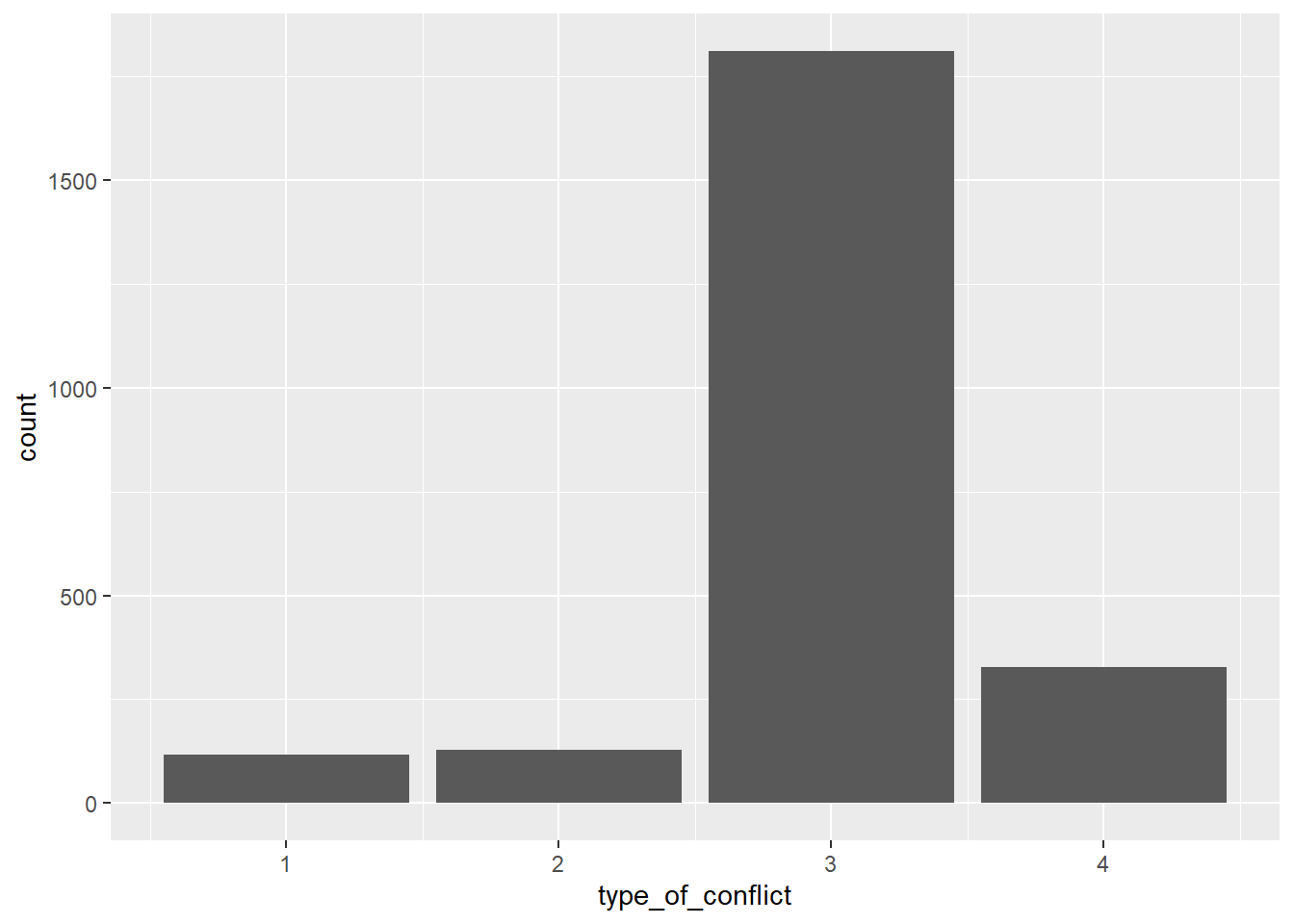
6.3 Transformar datos
Una vez tenemos una base de datos cargada en R (o sea, que es un objeto que aparece en Environment, con nombre y todo) queremos hacer algo con esos datos. Usualmente, ese algo implica transformarlos. Hay muchas formas de transformar una base de datos en R:
- Seleccionar filas (observaciones o casos).
- Organizar y ordenar estas observaciones.
- Seleccionar columnas (variables).
- Computar nuevas variables o transformar las existentes.
- Resumir datos por grupos.
- Trabajar con datos no disponibles (
NA).
Miremos cada una de estas operaciones. Al final, veremos que, en conjunto, ofrecen una caja de herramientas potente –y necesaria– para el analista de datos políticos.
6.4 Seleccionar filas: filter()
A veces no necesitamos todas las filas en una base de datos, sino que queremos concentrarnos en algunas observaciones o casos que creemos son relevantes. Por ejemplo, puede que solo queramos estudiar los países más pobres según los datos de gapminder() o solo las guerras civiles de UCDP. Para esto, la principal función que usamos es filter().
6.4.1 Seleccionar filas por condiciones
El tipo de selección más sencilla consiste en seleccionar condicionalmente filas usando evaluaciones lógicas. Por ejemplo, puede que nos interese ver solamente las observaciones para el año 2007 de la base de datos de gapminder, para lo cual usamos filter() en conjunción con el operador ==, el cual nos ayuda a seleccionar las filas que tienen exactamente el valor 2007 en la variable year:
filter(gapminder, year == 2007)## # A tibble: 142 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afghanistan Asia 2007 43.8 31889923 975.
## 2 Albania Europe 2007 76.4 3600523 5937.
## 3 Algeria Africa 2007 72.3 33333216 6223.
## 4 Angola Africa 2007 42.7 12420476 4797.
## 5 Argentina Americas 2007 75.3 40301927 12779.
## 6 Australia Oceania 2007 81.2 20434176 34435.
## 7 Austria Europe 2007 79.8 8199783 36126.
## 8 Bahrain Asia 2007 75.6 708573 29796.
## 9 Bangladesh Asia 2007 64.1 150448339 1391.
## 10 Belgium Europe 2007 79.4 10392226 33693.
## # ... with 132 more rowsAsí mismo, podemos hacer una selección según valores de más de una variable. Nos valemos de otros operadores, como & y |. El operador & es el booleano “y” (AND). Con & podemos seleccionar observaciones que cumplan dos condiciones simultáneamente (observaciones del año 2007 y expectativa de vida por encima de los 60 años):
head(filter(gapminder, year == 2007 & lifeExp > 60))## # A tibble: 6 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Albania Europe 2007 76.4 3600523 5937.
## 2 Algeria Africa 2007 72.3 33333216 6223.
## 3 Argentina Americas 2007 75.3 40301927 12779.
## 4 Australia Oceania 2007 81.2 20434176 34435.
## 5 Austria Europe 2007 79.8 8199783 36126.
## 6 Bahrain Asia 2007 75.6 708573 29796.Simplificando, filter() (y dplyr en general) nos permite reemplazar los & por ,:
filter(gapminder, year == 2007, lifeExp > 60)## # A tibble: 99 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Albania Europe 2007 76.4 3600523 5937.
## 2 Algeria Africa 2007 72.3 33333216 6223.
## 3 Argentina Americas 2007 75.3 40301927 12779.
## 4 Australia Oceania 2007 81.2 20434176 34435.
## 5 Austria Europe 2007 79.8 8199783 36126.
## 6 Bahrain Asia 2007 75.6 708573 29796.
## 7 Bangladesh Asia 2007 64.1 150448339 1391.
## 8 Belgium Europe 2007 79.4 10392226 33693.
## 9 Bolivia Americas 2007 65.6 9119152 3822.
## 10 Bosnia and Herzegovina Europe 2007 74.9 4552198 7446.
## # ... with 89 more rowsMientras, el operador | es el booleano “o” (OR): seleccionamos observaciones que cumplan una condición o la otra (el año 2007 o el año 1997):
filter(gapminder, year == 2007 | year == 1997)## # A tibble: 284 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afghanistan Asia 1997 41.8 22227415 635.
## 2 Afghanistan Asia 2007 43.8 31889923 975.
## 3 Albania Europe 1997 73.0 3428038 3193.
## 4 Albania Europe 2007 76.4 3600523 5937.
## 5 Algeria Africa 1997 69.2 29072015 4797.
## 6 Algeria Africa 2007 72.3 33333216 6223.
## 7 Angola Africa 1997 41.0 9875024 2277.
## 8 Angola Africa 2007 42.7 12420476 4797.
## 9 Argentina Americas 1997 73.3 36203463 10967.
## 10 Argentina Americas 2007 75.3 40301927 12779.
## # ... with 274 more rowsSi tenemos muchas condiciones de tipo OR (|) usamos el operador %in% seguido de una lista de condiciones concatenadas con c() para simplificar el código, así:
# miramos 5 observaciones aleatorias
sample_n(
filter(gapminder, continent %in% c("Europe", "Americas", "Oceania")), 5
) ## # A tibble: 5 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Ecuador Americas 1987 67.2 9545158 6482.
## 2 Switzerland Europe 1977 75.4 6316424 26982.
## 3 Austria Europe 1997 77.5 8069876 29096.
## 4 Brazil Americas 1997 69.4 168546719 7958.
## 5 Albania Europe 1982 70.4 2780097 3631.También podemos usar funciones dentro de filter(). Además, podemos utilizar otros operadores como <, <=, > y >=. Por ejemplo, a continuación seleccionamos solo los casos que tengan una expectativa de vida mayor o igual a la media global de esa variable.
filter(gapminder, lifeExp >= mean(lifeExp, na.rm = TRUE))## # A tibble: 895 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Albania Europe 1962 64.8 1728137 2313.
## 2 Albania Europe 1967 66.2 1984060 2760.
## 3 Albania Europe 1972 67.7 2263554 3313.
## 4 Albania Europe 1977 68.9 2509048 3533.
## 5 Albania Europe 1982 70.4 2780097 3631.
## 6 Albania Europe 1987 72 3075321 3739.
## 7 Albania Europe 1992 71.6 3326498 2497.
## 8 Albania Europe 1997 73.0 3428038 3193.
## 9 Albania Europe 2002 75.7 3508512 4604.
## 10 Albania Europe 2007 76.4 3600523 5937.
## # ... with 885 more rowsA veces, es más fácil pedirle a R que seleccione los datos que no cumplen una condición. Para eso está el operador != (no es igual a):
sample_n(
filter(gapminder, continent != "Asia"), 5
)## # A tibble: 5 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Guinea-Bissau Africa 1957 33.5 601095 432.
## 2 Belgium Europe 1957 69.2 8989111 9715.
## 3 Congo, Rep. Africa 1987 57.5 2064095 4201.
## 4 Djibouti Africa 1957 37.3 71851 2865.
## 5 Australia Oceania 1982 74.7 15184200 19477.Dos funciones -top_n() y top_frac- nos permiten seleccionar las primeras observaciones cuando ordenamos los datos según una variable (como cuando hacemos “Sort” u “Ordenar” en un programa como Excel). Por ejemplo, aquí seleccionamos el “top 5” de países según PIB per cápita:
top_n(gapminder, 5, gdpPercap)## # A tibble: 5 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Kuwait Asia 1952 55.6 160000 108382.
## 2 Kuwait Asia 1957 58.0 212846 113523.
## 3 Kuwait Asia 1962 60.5 358266 95458.
## 4 Kuwait Asia 1967 64.6 575003 80895.
## 5 Kuwait Asia 1972 67.7 841934 109348.Y aquí los 5 peores:
top_n(gapminder, -5, gdpPercap)## # A tibble: 5 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Congo, Dem. Rep. Africa 1997 42.6 47798986 312.
## 2 Congo, Dem. Rep. Africa 2002 45.0 55379852 241.
## 3 Congo, Dem. Rep. Africa 2007 46.5 64606759 278.
## 4 Guinea-Bissau Africa 1952 32.5 580653 300.
## 5 Lesotho Africa 1952 42.1 748747 299.Veamos el top 99.5% de los países con mayor expectativa de vida al nacer:
top_frac(gapminder, 0.005, lifeExp)## # A tibble: 8 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Australia Oceania 2007 81.2 20434176 34435.
## 2 Hong Kong, China Asia 2002 81.5 6762476 30209.
## 3 Hong Kong, China Asia 2007 82.2 6980412 39725.
## 4 Iceland Europe 2007 81.8 301931 36181.
## 5 Japan Asia 2002 82 127065841 28605.
## 6 Japan Asia 2007 82.6 127467972 31656.
## 7 Spain Europe 2007 80.9 40448191 28821.
## 8 Switzerland Europe 2007 81.7 7554661 37506.Finalmente, podemos guardar los resultados de esta selección como un objeto, para seguir trabajando con este subconjunto de los datos. Esto es altamente recomendable, para no tener que repetir el mismo código cada vez que queremos trabajar con un subconjunto de datos.
gapminder_reciente <- filter(gapminder, year %in% c(1997, 2002, 2007))
gapminder_america <- filter(gapminder, continent == "Americas")Además, si guardamos el objeto como un archivo .csv o .xlsx por ejemplo y lo cargamos después, podemos “saltarnos” muchas líneas de código. Si queremos guardar un archivo de Excel, usamos la función write_xlsx() de la librería writexl, especificando la carpeta de destino y el nombre del nuevo archivo:
write_xlsx(gapminder_america, "data/gapminder_america.xlsx")6.4.2 Seleccionar filas por posición
Si con filter() podemos usar valores de variables como criterios de selección, con slice() podemos seleccionar un número arbitrario de observaciones según su posición en la base de datos. Por ejemplo, si queremos solo la observación número 100:
slice(gapminder, 100)## # A tibble: 1 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Bangladesh Asia 1967 43.5 62821884 721.O las observaciones de la 55 a la 65 de una base de datos:
slice(gapminder, 55:65)## # A tibble: 11 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Argentina Americas 1982 69.9 29341374 8998.
## 2 Argentina Americas 1987 70.8 31620918 9140.
## 3 Argentina Americas 1992 71.9 33958947 9308.
## 4 Argentina Americas 1997 73.3 36203463 10967.
## 5 Argentina Americas 2002 74.3 38331121 8798.
## 6 Argentina Americas 2007 75.3 40301927 12779.
## 7 Australia Oceania 1952 69.1 8691212 10040.
## 8 Australia Oceania 1957 70.3 9712569 10950.
## 9 Australia Oceania 1962 70.9 10794968 12217.
## 10 Australia Oceania 1967 71.1 11872264 14526.
## 11 Australia Oceania 1972 71.9 13177000 16789.Quizás la última observación, usando n():
slice(gapminder, n())## # A tibble: 1 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Zimbabwe Africa 2007 43.5 12311143 470.O todas, menos de la 8 hasta la última:
slice(gapminder, -8:-n())## # A tibble: 7 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afghanistan Asia 1952 28.8 8425333 779.
## 2 Afghanistan Asia 1957 30.3 9240934 821.
## 3 Afghanistan Asia 1962 32.0 10267083 853.
## 4 Afghanistan Asia 1967 34.0 11537966 836.
## 5 Afghanistan Asia 1972 36.1 13079460 740.
## 6 Afghanistan Asia 1977 38.4 14880372 786.
## 7 Afghanistan Asia 1982 39.9 12881816 978.En cierto sentido, slice() es similar a head() y tail(), pero estas dos últimas funciones no pueden seleccionar filas en la mitad de una base de datos.
6.5 Reordenar filas: arrange()
Todos conocemos la opción “Ordernar” (“Sort”) en Excel y programas similares: nos permite ordenar datos de mayor a menor (o menor a mayor) según los valores de una variables. En el tidyverse, tenemos arrange(), una función que cambia el orden de las filas. En vez del orden alfabético por defecto de gapminder, organicemos la base de datos por PIB per cápita, de menor a mayor:
arrange(gapminder, gdpPercap)## # A tibble: 1,704 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Congo, Dem. Rep. Africa 2002 45.0 55379852 241.
## 2 Congo, Dem. Rep. Africa 2007 46.5 64606759 278.
## 3 Lesotho Africa 1952 42.1 748747 299.
## 4 Guinea-Bissau Africa 1952 32.5 580653 300.
## 5 Congo, Dem. Rep. Africa 1997 42.6 47798986 312.
## 6 Eritrea Africa 1952 35.9 1438760 329.
## 7 Myanmar Asia 1952 36.3 20092996 331
## 8 Lesotho Africa 1957 45.0 813338 336.
## 9 Burundi Africa 1952 39.0 2445618 339.
## 10 Eritrea Africa 1957 38.0 1542611 344.
## # ... with 1,694 more rowsAsí mismo, podemos ordenar los datos de manera descendente usando el operador -:
arrange(gapminder, -gdpPercap)## # A tibble: 1,704 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Kuwait Asia 1957 58.0 212846 113523.
## 2 Kuwait Asia 1972 67.7 841934 109348.
## 3 Kuwait Asia 1952 55.6 160000 108382.
## 4 Kuwait Asia 1962 60.5 358266 95458.
## 5 Kuwait Asia 1967 64.6 575003 80895.
## 6 Kuwait Asia 1977 69.3 1140357 59265.
## 7 Norway Europe 2007 80.2 4627926 49357.
## 8 Kuwait Asia 2007 77.6 2505559 47307.
## 9 Singapore Asia 2007 80.0 4553009 47143.
## 10 Norway Europe 2002 79.0 4535591 44684.
## # ... with 1,694 more rows6.6 Seleccionar variables: select()
Ya vimos cómo seleccionar filas u observaciones (casos). En algunas ocasiones, no necesitamos todas las columnas en una base de datos: solo nos interesan las variables económicas o queremos eliminar unas columnas y mantener otras. Para esto, usamos la función select(). Por ejemplo, a continuación seleccionamos solo tres columnas de una base de datos:
select(gapminder, country, year, gdpPercap)## # A tibble: 1,704 x 3
## country year gdpPercap
## <fct> <int> <dbl>
## 1 Afghanistan 1952 779.
## 2 Afghanistan 1957 821.
## 3 Afghanistan 1962 853.
## 4 Afghanistan 1967 836.
## 5 Afghanistan 1972 740.
## 6 Afghanistan 1977 786.
## 7 Afghanistan 1982 978.
## 8 Afghanistan 1987 852.
## 9 Afghanistan 1992 649.
## 10 Afghanistan 1997 635.
## # ... with 1,694 more rowsPodemos usar el operador : para seleccionar un rango de variables, según el orden en que aparecen en la base de datos (desde year hasta pop):
select(gapminder, year:pop)## # A tibble: 1,704 x 3
## year lifeExp pop
## <int> <dbl> <int>
## 1 1952 28.8 8425333
## 2 1957 30.3 9240934
## 3 1962 32.0 10267083
## 4 1967 34.0 11537966
## 5 1972 36.1 13079460
## 6 1977 38.4 14880372
## 7 1982 39.9 12881816
## 8 1987 40.8 13867957
## 9 1992 41.7 16317921
## 10 1997 41.8 22227415
## # ... with 1,694 more rowsDe manera análoga a filter(), podemos seleccionar todo excepto ciertas variables. Aquí, seleccionamos todas menos continent y pop:
select(gapminder, -c(continent, pop))## # A tibble: 1,704 x 4
## country year lifeExp gdpPercap
## <fct> <int> <dbl> <dbl>
## 1 Afghanistan 1952 28.8 779.
## 2 Afghanistan 1957 30.3 821.
## 3 Afghanistan 1962 32.0 853.
## 4 Afghanistan 1967 34.0 836.
## 5 Afghanistan 1972 36.1 740.
## 6 Afghanistan 1977 38.4 786.
## 7 Afghanistan 1982 39.9 978.
## 8 Afghanistan 1987 40.8 852.
## 9 Afghanistan 1992 41.7 649.
## 10 Afghanistan 1997 41.8 635.
## # ... with 1,694 more rowsLa función select() tiene unas funciones hermanas que le agregan flexibilidad y expanden lo que podemos hacer con ella. Por ejemplo, con select_if() podemos seleccionar variables que cumplen una condición - en este caso, solo las columnas numéricas (según su clase en R):
select_if(gapminder, is.numeric)## # A tibble: 1,704 x 4
## year lifeExp pop gdpPercap
## <int> <dbl> <int> <dbl>
## 1 1952 28.8 8425333 779.
## 2 1957 30.3 9240934 821.
## 3 1962 32.0 10267083 853.
## 4 1967 34.0 11537966 836.
## 5 1972 36.1 13079460 740.
## 6 1977 38.4 14880372 786.
## 7 1982 39.9 12881816 978.
## 8 1987 40.8 13867957 852.
## 9 1992 41.7 16317921 649.
## 10 1997 41.8 22227415 635.
## # ... with 1,694 more rowsUsando starts_with, podemos quedarnos solo con las columnas cuyo nombre comienza por una letra o una expresión en específico. Esto puede ser útil cuando tenemos columnas con nombres como indicador_a, indicador_b, indicador_c, etc. y queremos seleccionarlas. Por ejemplo, columnas que empiezan por “c”:
select(gapminder, starts_with("c"))## # A tibble: 1,704 x 2
## country continent
## <fct> <fct>
## 1 Afghanistan Asia
## 2 Afghanistan Asia
## 3 Afghanistan Asia
## 4 Afghanistan Asia
## 5 Afghanistan Asia
## 6 Afghanistan Asia
## 7 Afghanistan Asia
## 8 Afghanistan Asia
## 9 Afghanistan Asia
## 10 Afghanistan Asia
## # ... with 1,694 more rowsLa función ends_with() hace lo opuesto:
select(gapminder, c(country, ends_with("p")))## # A tibble: 1,704 x 4
## country lifeExp pop gdpPercap
## <fct> <dbl> <int> <dbl>
## 1 Afghanistan 28.8 8425333 779.
## 2 Afghanistan 30.3 9240934 821.
## 3 Afghanistan 32.0 10267083 853.
## 4 Afghanistan 34.0 11537966 836.
## 5 Afghanistan 36.1 13079460 740.
## 6 Afghanistan 38.4 14880372 786.
## 7 Afghanistan 39.9 12881816 978.
## 8 Afghanistan 40.8 13867957 852.
## 9 Afghanistan 41.7 16317921 649.
## 10 Afghanistan 41.8 22227415 635.
## # ... with 1,694 more rowsMientras, contains() busca columnas que tengan ciertos caracteres, sea al principio, final o en la mitad:
select(gapminder, contains("Exp"))## # A tibble: 1,704 x 1
## lifeExp
## <dbl>
## 1 28.8
## 2 30.3
## 3 32.0
## 4 34.0
## 5 36.1
## 6 38.4
## 7 39.9
## 8 40.8
## 9 41.7
## 10 41.8
## # ... with 1,694 more rows6.6.1 Renombrar columnas: rename()
Podemos renombrar columnas dentro de select(), pero esto elimina todas las variables no renombradas explícitamente:
select(gapminder, pib_percap = gdpPercap)## # A tibble: 1,704 x 1
## pib_percap
## <dbl>
## 1 779.
## 2 821.
## 3 853.
## 4 836.
## 5 740.
## 6 786.
## 7 978.
## 8 852.
## 9 649.
## 10 635.
## # ... with 1,694 more rowsEn cambio, rename() nos permite renombrar y además mantener las demás:
rename(
gapminder,
# nombre_nuevo = nombre_viejo
continente = continent, ano = year, exp_vida = lifeExp,
pob = pop, pib_percap = gdpPercap
)## # A tibble: 1,704 x 6
## country continente ano exp_vida pob pib_percap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afghanistan Asia 1952 28.8 8425333 779.
## 2 Afghanistan Asia 1957 30.3 9240934 821.
## 3 Afghanistan Asia 1962 32.0 10267083 853.
## 4 Afghanistan Asia 1967 34.0 11537966 836.
## 5 Afghanistan Asia 1972 36.1 13079460 740.
## 6 Afghanistan Asia 1977 38.4 14880372 786.
## 7 Afghanistan Asia 1982 39.9 12881816 978.
## 8 Afghanistan Asia 1987 40.8 13867957 852.
## 9 Afghanistan Asia 1992 41.7 16317921 649.
## 10 Afghanistan Asia 1997 41.8 22227415 635.
## # ... with 1,694 more rows6.7 Crear y transformar variables: mutate()
Las bases de datos no siempre tienen todas las variables que necesitamos. Pero pueden tener la información necesaria para que las creemos nosotros mismos. mutate() es la principal función que usamos para crear variables o modificar variables existentes. mutate() siempre adiciona columnas nuevas a la base de datos, pero -como veremos- si queremos que queden grabadas al objeto, debemos usar el operador de asignación <-.
Como ejemplo, tomemos gapminder. Tenemos información sobre PIB per cápita (gdpPercap), pero en realidad queremos el PIB. Esa columna no existe en los datos, pero tenemos la información suficiente para construirle. El PIB per cápita se define como \(\frac{PIB}{población}\), así que si utilizamos la variable pop tenemos lo necesario para calcular el PIB:
mutate(gapminder, gdp = gdpPercap*pop)## # A tibble: 1,704 x 7
## country continent year lifeExp pop gdpPercap gdp
## <fct> <fct> <int> <dbl> <int> <dbl> <dbl>
## 1 Afghanistan Asia 1952 28.8 8425333 779. 6567086330.
## 2 Afghanistan Asia 1957 30.3 9240934 821. 7585448670.
## 3 Afghanistan Asia 1962 32.0 10267083 853. 8758855797.
## 4 Afghanistan Asia 1967 34.0 11537966 836. 9648014150.
## 5 Afghanistan Asia 1972 36.1 13079460 740. 9678553274.
## 6 Afghanistan Asia 1977 38.4 14880372 786. 11697659231.
## 7 Afghanistan Asia 1982 39.9 12881816 978. 12598563401.
## 8 Afghanistan Asia 1987 40.8 13867957 852. 11820990309.
## 9 Afghanistan Asia 1992 41.7 16317921 649. 10595901589.
## 10 Afghanistan Asia 1997 41.8 22227415 635. 14121995875.
## # ... with 1,694 more rowsSi solo queremos mantener las variables creadas (y descartar las originales), usamos transmute(), pero rara vez queremos deshacernos de todo:
transmute(gapminder, gdp = gdpPercap*pop)## # A tibble: 1,704 x 1
## gdp
## <dbl>
## 1 6567086330.
## 2 7585448670.
## 3 8758855797.
## 4 9648014150.
## 5 9678553274.
## 6 11697659231.
## 7 12598563401.
## 8 11820990309.
## 9 10595901589.
## 10 14121995875.
## # ... with 1,694 more rowsAdemás, podemos usar muchas otras funciones, como log(), para crear nuevas variables. Por ejemplo, si queremos el logaritmo del PIB per cápita:
mutate(gapminder, gdpPercap_log = log(gdpPercap))## # A tibble: 1,704 x 7
## country continent year lifeExp pop gdpPercap gdpPercap_log
## <fct> <fct> <int> <dbl> <int> <dbl> <dbl>
## 1 Afghanistan Asia 1952 28.8 8425333 779. 6.66
## 2 Afghanistan Asia 1957 30.3 9240934 821. 6.71
## 3 Afghanistan Asia 1962 32.0 10267083 853. 6.75
## 4 Afghanistan Asia 1967 34.0 11537966 836. 6.73
## 5 Afghanistan Asia 1972 36.1 13079460 740. 6.61
## 6 Afghanistan Asia 1977 38.4 14880372 786. 6.67
## 7 Afghanistan Asia 1982 39.9 12881816 978. 6.89
## 8 Afghanistan Asia 1987 40.8 13867957 852. 6.75
## 9 Afghanistan Asia 1992 41.7 16317921 649. 6.48
## 10 Afghanistan Asia 1997 41.8 22227415 635. 6.45
## # ... with 1,694 more rowsSi queremos que las variables creadas permanezcan y se vuelvan parte de la base de datos en R, debemos reescribir el objeto o crear uno nuevo, usando el operador de asignación <-. Si usamos el mismo nombre del objeto original, lo reescribimos. Si usamos un nuevo nombre, creamos un objeto adicional en el Environment:
gapminder <- mutate(
gapminder,
gdp = gdpPercap*pop,
gdp_log = log(gdp),
gdpPercap_log = log(gdpPercap)
)Veamos el resultado para confirmar que las nuevas columnas ahora sí quedaron guardadas en el objeto:
select(gapminder, country, year, gdp_log, gdpPercap_log)## # A tibble: 1,704 x 4
## country year gdp_log gdpPercap_log
## <fct> <int> <dbl> <dbl>
## 1 Afghanistan 1952 22.6 6.66
## 2 Afghanistan 1957 22.7 6.71
## 3 Afghanistan 1962 22.9 6.75
## 4 Afghanistan 1967 23.0 6.73
## 5 Afghanistan 1972 23.0 6.61
## 6 Afghanistan 1977 23.2 6.67
## 7 Afghanistan 1982 23.3 6.89
## 8 Afghanistan 1987 23.2 6.75
## 9 Afghanistan 1992 23.1 6.48
## 10 Afghanistan 1997 23.4 6.45
## # ... with 1,694 more rowsOtro ejemplo útil: podemos calcular valores acumulados, rezagados y adelantados de una variable. Para ilustrar, hagamos un pequeño subconjunto de datos:
gapminder_colombia <- filter(gapminder, country == "Colombia")Ahora, creemos cuatro nuevas variables: para cada observación, queremos ver el PIB per cápita más alto de la serie (cummax()), el valor de la observación anterior (lag()), el valor de hace 2 observaciones (con el argumento n =), y el del año siguiente (lead())
gapminder_colombia <- mutate(
gapminder_colombia,
gdpPercap_max = cummax(gdpPercap),
gdpPercap_lag = lag(gdpPercap),
gdpPercap_lag2 = lag(gdpPercap, n = 2),
gdpPercap_lead = lead(gdpPercap)
)
select(gapminder_colombia, year, gdpPercap, gdpPercap_max, gdpPercap_lag, gdpPercap_lag2, gdpPercap_lead)## # A tibble: 12 x 6
## year gdpPercap gdpPercap_max gdpPercap_lag gdpPercap_lag2 gdpPercap_lead
## <int> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1952 2144. 2144. NA NA 2324.
## 2 1957 2324. 2324. 2144. NA 2492.
## 3 1962 2492. 2492. 2324. 2144. 2679.
## 4 1967 2679. 2679. 2492. 2324. 3265.
## 5 1972 3265. 3265. 2679. 2492. 3816.
## 6 1977 3816. 3816. 3265. 2679. 4398.
## 7 1982 4398. 4398. 3816. 3265. 4903.
## 8 1987 4903. 4903. 4398. 3816. 5445.
## 9 1992 5445. 5445. 4903. 4398. 6117.
## 10 1997 6117. 6117. 5445. 4903. 5755.
## 11 2002 5755. 6117. 6117. 5445. 7007.
## 12 2007 7007. 7007. 5755. 6117. NAPodemos usar lag() y lead() aquí porque solo hay datos de un país y están ordenados cronológicamente por año. En paneles de datos con muchos países y años, necesitaríamos agrupar los datos por país primero, como veremos más adelante.
6.7.1 Cambiar clases
Todos los objetos en R tienen una clase, incluyendo las columnas de una base de datos. Las principales clases de variables en R son: numeric, integer, date, factor, character y logical. A veces, tenemos una variable tipo caracter o texto que queremos como categórica (factor). O sucede que cargamos los datos y hay una variable numérica que R interpreta como texto. Miremos cómo cambiar tipos de variables usando los datos de PolityIV.
polity4 <- select(polity4, ccode, country, year, polity2, parreg, parcomp, exconst)
polity4## # A tibble: 17,395 x 7
## ccode country year polity2 parreg parcomp exconst
## <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2 United States 1800 4 4 2 7
## 2 2 United States 1801 4 4 2 7
## 3 2 United States 1802 4 4 2 7
## 4 2 United States 1803 4 4 2 7
## 5 2 United States 1804 4 4 2 7
## 6 2 United States 1805 4 4 2 7
## 7 2 United States 1806 4 4 2 7
## 8 2 United States 1807 4 4 2 7
## 9 2 United States 1808 4 4 2 7
## 10 2 United States 1809 9 2 4 7
## # ... with 17,385 more rowsHay varias funciones que nos permite hacer cambios de tipo fácilmente en conjunción con mutate(): as.numeric(), as.factor(), as.integer(), as.character()… Convirtamos el código de país y el año a factor (variable categórica) y a (número) entero, respectivamente.
mutate(polity4, ccode = as.factor(ccode), year = as.integer(year))## # A tibble: 17,395 x 7
## ccode country year polity2 parreg parcomp exconst
## <fct> <chr> <int> <dbl> <dbl> <dbl> <dbl>
## 1 2 United States 1800 4 4 2 7
## 2 2 United States 1801 4 4 2 7
## 3 2 United States 1802 4 4 2 7
## 4 2 United States 1803 4 4 2 7
## 5 2 United States 1804 4 4 2 7
## 6 2 United States 1805 4 4 2 7
## 7 2 United States 1806 4 4 2 7
## 8 2 United States 1807 4 4 2 7
## 9 2 United States 1808 4 4 2 7
## 10 2 United States 1809 9 2 4 7
## # ... with 17,385 more rowsSi tenemos muchas variables que aparecen con la clase incorrecta, podemos cambiarlas masivamente usando mutate_if(). Esta función nos permite imponer una condición para seleccionar qué variables vamos a transformar y seleccionar una función (as.factor() en este ejemplo) para aplicarles:
mutate_if( # cambiar si
polity4,
is.character, # característica que tienen las variables que queremos cambiar
as.factor # función que queremos aplicarles
)## # A tibble: 17,395 x 7
## ccode country year polity2 parreg parcomp exconst
## <dbl> <fct> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2 United States 1800 4 4 2 7
## 2 2 United States 1801 4 4 2 7
## 3 2 United States 1802 4 4 2 7
## 4 2 United States 1803 4 4 2 7
## 5 2 United States 1804 4 4 2 7
## 6 2 United States 1805 4 4 2 7
## 7 2 United States 1806 4 4 2 7
## 8 2 United States 1807 4 4 2 7
## 9 2 United States 1808 4 4 2 7
## 10 2 United States 1809 9 2 4 7
## # ... with 17,385 more rows6.7.2 Variables categóricas
Ya hemos visto cómo crear nuevas variables numéricas aplicando funciones en el contexto de mutate(). Pero también podemos crear variables categóricas o cualitativas. En R, estas son llamadas “factores” (o factors).
6.7.2.1 Numérica a categórica
Primero, utilicemos los valores de una variable numérica para crear una categórica. Por ejemplo, clasifiquemos como “democracias” a los países que tienen un valor mayor de 5 en el indicador polity2. Para esto, la función if_else() nos permite evaluar con expresiones lógicas si se cumplen condiciones y reemplazar valores. A su vez, la función factor() le dice a R que la variable creada es categórica - es importante seguir bien los paréntesis. Usamos <- para asegurarnos que los resultados queden guardados en polity4:
polity4 <- mutate(
polity4,
democracia = factor( # creamos una nueva variable categórica o factor llamada democracia
if_else(
condition = polity2 > 5, # qué condición se tiene que cumplir
true = "democracia", # qué hacer si se cumple
false = "otro" # qué hacer si no se cumple
),
ordered = FALSE # especificamos que la variable no es ordenada
)
)Revisamos el resultado:
count(polity4, democracia)## # A tibble: 3 x 2
## democracia n
## <fct> <int>
## 1 democracia 5050
## 2 otro 12109
## 3 <NA> 236Podemos crear mas de una categoría a la vez usando case_when(), la cual evalúa si cumple con unas condiciones y crea una variable acordemente:
polity4 <- mutate(
polity4,
regimen = factor( # crear una nueva variable tipo factor
case_when(
polity2 > 5 ~ "democracia", # condición ~ resultado
polity2 < -5 ~ "autocracia",
TRUE ~ "anocracia" # para los demás casos ~ resultado
),
ordered = FALSE # el factor no es ordenado
)
)
count(polity4, regimen)## # A tibble: 3 x 2
## regimen n
## <fct> <int>
## 1 anocracia 6133
## 2 autocracia 6212
## 3 democracia 5050Otra forma de hacer esto es con la función cut_number(). A continuación, la utilizamos para dividir la variable polity2 en 5 grupos o categorías con aproximadamente el mismo número de observaciones en cada una:
polity4 <- mutate(
polity4,
regimen_cut = cut_number(polity2, 5)
)
count(polity4, regimen_cut)## # A tibble: 6 x 2
## regimen_cut n
## <fct> <int>
## 1 [-10,-7] 4913
## 2 (-7,-4] 2607
## 3 (-4,1] 2844
## 4 (1,8] 3657
## 5 (8,10] 3138
## 6 <NA> 236Podemos darle nombre a las categorías con el argumento labels =:
polity4 <- mutate(
polity4,
regimen_cut = cut_number(
polity2, 5, labels = c("bajo", "med-bajo", "medio", "med-alto", "alto")
)
)
count(polity4, regimen_cut)## # A tibble: 6 x 2
## regimen_cut n
## <fct> <int>
## 1 bajo 4913
## 2 med-bajo 2607
## 3 medio 2844
## 4 med-alto 3657
## 5 alto 3138
## 6 <NA> 2366.7.2.1.1 Reordenar factores
Tenemos varias maneras de reordenar o recodificar factores; algunos de estos son funciones de la librería forcats del tidyverse.
Si queremos especificar los niveles de un factor al crealo, usamos el argumento "levels =" en factor(). El orden de un factor importa a la hora de saber cuál es la categoría base para comparar a la hora de hacer gráficas o estimar modelos estadísticos:
polity4 <- mutate(
polity4,
regimen = factor(regimen,
levels = c("democracia", "anocracia", "autocracia"))
)
count(polity4, regimen)## # A tibble: 3 x 2
## regimen n
## <fct> <int>
## 1 democracia 5050
## 2 anocracia 6133
## 3 autocracia 6212En forcats podemos hacer algo similar -un reordenamiento manual- con fct_relevel() cuando tenemos una variable categórica ya existente.
polity4 <- mutate(
polity4,
regimen = fct_relevel(regimen,
c("autocracia", "anocracia", "democracia"))
)
count(polity4, regimen)## # A tibble: 3 x 2
## regimen n
## <fct> <int>
## 1 autocracia 6212
## 2 anocracia 6133
## 3 democracia 5050Igualmente, podemos reordenar por frecuencias, trayendo al frente a la categoría con más observaciones. Hacemos esto con fct_infreq() y es útil cuando queremos construir una gráfica:
ggplot(polity4, aes(fct_infreq(regimen_cut))) +
geom_bar() +
coord_flip()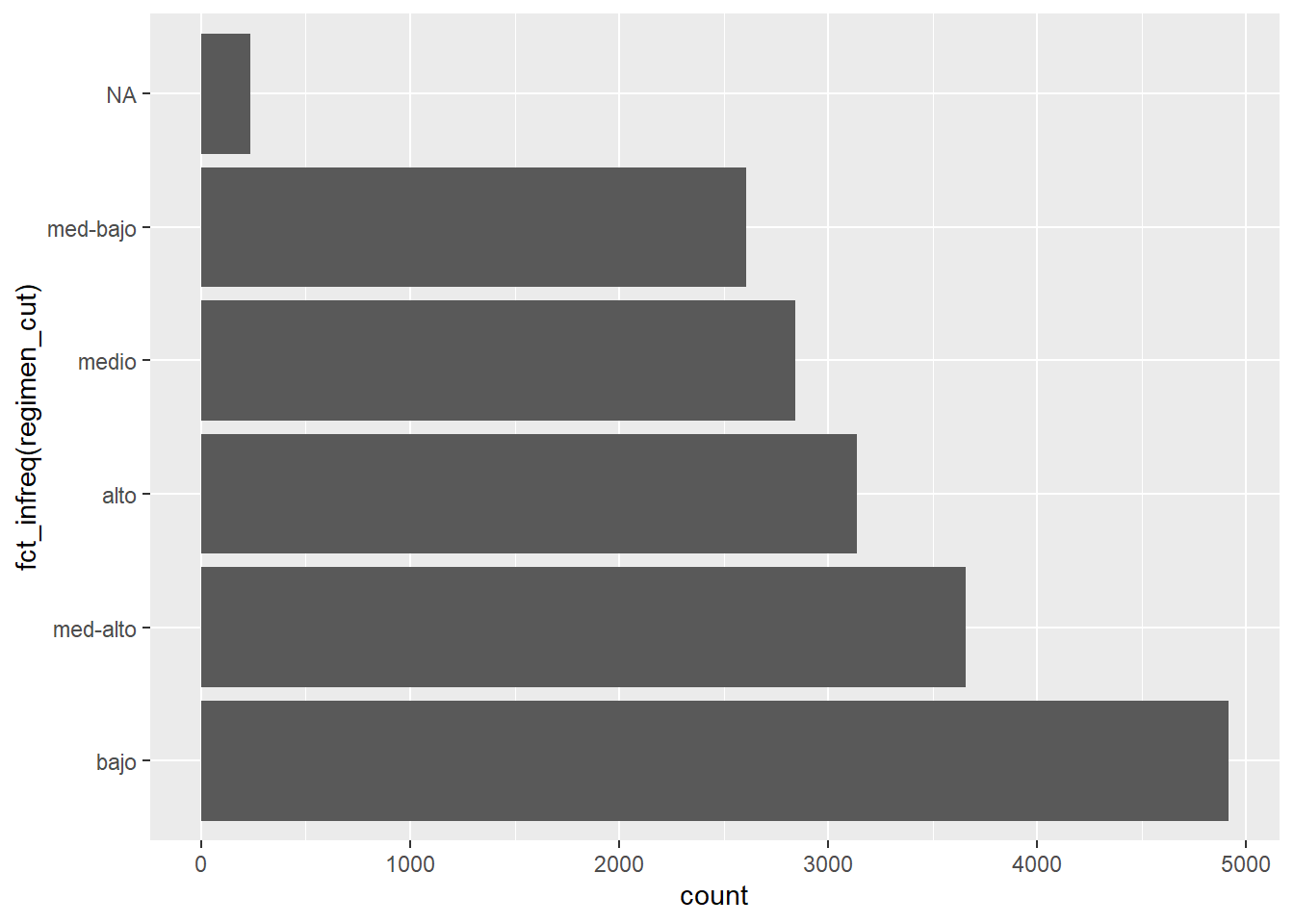
También podemos ordenar los niveles de un factor según los valores de otra variable con fct_reorder(). En la siguiente gráfica, vemos el PIB per cápita de cuatro países suramericanos para el año 2007, pero el orden de los países en la gráfica corresponde a la expectativa de vida promedio en cada país:
gapminder_sub <- filter(gapminder, year == max(year), country %in% c("Colombia", "Argentina", "Peru", "Venezuela"))
ggplot(gapminder_sub, aes(fct_reorder(country, lifeExp), gdpPercap)) +
geom_col()
Finalmente, podemos combinar o colapsar categorías en una categoría de “otros” usando la función fct_lump() con el argumento opcional other_level = para especificar el nombre de la categoría residual (por defecto, es “Other”):
polity4 <- mutate(
polity4,
regimen_bin = fct_lump(regimen_cut, n = 2, other_level = "otros")
)
count(polity4, regimen_bin)## # A tibble: 4 x 2
## regimen_bin n
## <fct> <int>
## 1 bajo 4913
## 2 med-alto 3657
## 3 otros 8589
## 4 <NA> 2366.7.2.2 Categórica a numérica
Finalmente, utilizamos los valores de una variable categórica para crear una numérica discreta. Ojo: debemos asegurarnos que esto tenga sentido: por ejemplo, en términos de democratización, ¿es igual pasar de autocracia a anocracia, que pasar anocracia a democracia? El truco que usamos es la función recode() que nos permite asignar nuevos valores:
polity4 <- mutate(
polity4,
regimen_num = recode(
regimen,
"autocracia" = -1, # valor original = valor nuevo
"anocracia" = 0,
"democracia" = 1
)
)
count(polity4, regimen_num)## # A tibble: 3 x 2
## regimen_num n
## <dbl> <int>
## 1 -1 6212
## 2 0 6133
## 3 1 50506.8 Resumir: count(), group_by() y summarize()
Con frecuencia, nuestros datos están agrupados: las observaciones pertenecen a grupos, indicador por una variable categórica. Por ejemplo, los países del mundo están ubicados en distintos continentes:
count(gapminder, continent)## # A tibble: 5 x 2
## continent n
## <fct> <int>
## 1 Africa 624
## 2 Americas 300
## 3 Asia 396
## 4 Europe 360
## 5 Oceania 24Si queremos una nueva variable que nos diga, para cada observación, cuántas observaciones hay en su grupo, usamos add_count().
sample_n(select(add_count(gapminder, continent), country, continent, n), 5)## # A tibble: 5 x 3
## country continent n
## <fct> <fct> <int>
## 1 Switzerland Europe 360
## 2 Turkey Europe 360
## 3 Korea, Rep. Asia 396
## 4 Congo, Rep. Africa 624
## 5 South Africa Africa 6246.8.1 Tablas cruzadas
La función count() sirve para hacer tablas cruzadas con dos variables categóricas. Primero, creamos una nueva variable para niveles altos de PIB per cápita (por encima de la media global):
gapminder <- mutate(
gapminder,
pib_dummy = if_else(
gdpPercap >= mean(gdpPercap, na.rm = TRUE), "alto", "bajo"
)
)Ahora, tabulamos:
count(gapminder, continent, pib_dummy)## # A tibble: 9 x 3
## continent pib_dummy n
## <fct> <chr> <int>
## 1 Africa alto 36
## 2 Africa bajo 588
## 3 Americas alto 92
## 4 Americas bajo 208
## 5 Asia alto 112
## 6 Asia bajo 284
## 7 Europe alto 270
## 8 Europe bajo 90
## 9 Oceania alto 24Si queremos tener una tabla para importar a un documento Word, usamos la libreria knitr() y la funcion kable(). Primero, creamos la tabla:
library(knitr)
tabla <- kable(
count(gapminder, continent, pib_dummy), # datos
format = "html", # formato .html
col.names = c("Continente", "Nivel PIB", "Casos"), # nombres de columnas
caption = "Tabla cruzada" # titulo
)Creamos una nueva carpeta para guardar la tabla:
#dir.create("/cloud/project/output") # ya existe en el proyecto, pero así lo haríamosY luego guardamos la tabla con write_file() de readr.
write_file(tabla, "output/tabla.doc")6.8.2 Agrupar y resumir
Aprovechamos que los datos son agrupados para aplicar operaciones que los resumen. Además, estas agrupaciones nos permiten hacer comparaciones interesantes. Por si solo, summarize() resume variables y el resultado siempre es un solo valor.
summarize(gapminder, lifeExp_media = mean(lifeExp, na.rm = TRUE))## # A tibble: 1 x 1
## lifeExp_media
## <dbl>
## 1 59.5Por si solo, group_by() aparentemente no hace mucho
group_by(gapminder, country)## # A tibble: 1,704 x 10
## # Groups: country [142]
## country continent year lifeExp pop gdpPercap gdp gdp_log
## <fct> <fct> <int> <dbl> <int> <dbl> <dbl> <dbl>
## 1 Afghan~ Asia 1952 28.8 8.43e6 779. 6.57e 9 22.6
## 2 Afghan~ Asia 1957 30.3 9.24e6 821. 7.59e 9 22.7
## 3 Afghan~ Asia 1962 32.0 1.03e7 853. 8.76e 9 22.9
## 4 Afghan~ Asia 1967 34.0 1.15e7 836. 9.65e 9 23.0
## 5 Afghan~ Asia 1972 36.1 1.31e7 740. 9.68e 9 23.0
## 6 Afghan~ Asia 1977 38.4 1.49e7 786. 1.17e10 23.2
## 7 Afghan~ Asia 1982 39.9 1.29e7 978. 1.26e10 23.3
## 8 Afghan~ Asia 1987 40.8 1.39e7 852. 1.18e10 23.2
## 9 Afghan~ Asia 1992 41.7 1.63e7 649. 1.06e10 23.1
## 10 Afghan~ Asia 1997 41.8 2.22e7 635. 1.41e10 23.4
## # ... with 1,694 more rows, and 2 more variables: gdpPercap_log <dbl>,
## # pib_dummy <chr>Aunque sí nos permite hallar valores razagados y adelantadas por grupo.
head(mutate(
group_by(gapminder, country),
gdp_lag = lag(gdpPercap),
gdp_lead = lead(gdpPercap)
))## # A tibble: 6 x 12
## # Groups: country [1]
## country continent year lifeExp pop gdpPercap gdp gdp_log gdpPercap_log
## <fct> <fct> <int> <dbl> <int> <dbl> <dbl> <dbl> <dbl>
## 1 Afghan~ Asia 1952 28.8 8.43e6 779. 6.57e 9 22.6 6.66
## 2 Afghan~ Asia 1957 30.3 9.24e6 821. 7.59e 9 22.7 6.71
## 3 Afghan~ Asia 1962 32.0 1.03e7 853. 8.76e 9 22.9 6.75
## 4 Afghan~ Asia 1967 34.0 1.15e7 836. 9.65e 9 23.0 6.73
## 5 Afghan~ Asia 1972 36.1 1.31e7 740. 9.68e 9 23.0 6.61
## 6 Afghan~ Asia 1977 38.4 1.49e7 786. 1.17e10 23.2 6.67
## # ... with 3 more variables: pib_dummy <chr>, gdp_lag <dbl>, gdp_lead <dbl>Por sus poderes combinados… Uno de los “combos” mas potentes: group_by() junto a summarize(). Agrupamos los datos (con una variable categórica) y resumimos. Así, calculamos la media de la expectativa de vida de cada ano en los datos, por ejemplo:
summarize(
group_by(gapminder, year), # agrupamos
lifeExp_media = mean(lifeExp, na.rm = TRUE) # creamos una variable que resume cada grupo
)## `summarise()` ungrouping output (override with `.groups` argument)## # A tibble: 12 x 2
## year lifeExp_media
## <int> <dbl>
## 1 1952 49.1
## 2 1957 51.5
## 3 1962 53.6
## 4 1967 55.7
## 5 1972 57.6
## 6 1977 59.6
## 7 1982 61.5
## 8 1987 63.2
## 9 1992 64.2
## 10 1997 65.0
## 11 2002 65.7
## 12 2007 67.0Cuántas observaciones hay en cada grupo (usando n()):
summarize(
group_by(gapminder, continent),
num_obs = n()
)## `summarise()` ungrouping output (override with `.groups` argument)## # A tibble: 5 x 2
## continent num_obs
## <fct> <int>
## 1 Africa 624
## 2 Americas 300
## 3 Asia 396
## 4 Europe 360
## 5 Oceania 24O cuál ha sido el nivel más alto del PIB per cápita para cada país en la muestra:
summarize(
group_by(gapminder, country),
gdpPercap_max = max(gdpPercap, na.rm = TRUE)
)## `summarise()` ungrouping output (override with `.groups` argument)## # A tibble: 142 x 2
## country gdpPercap_max
## <fct> <dbl>
## 1 Afghanistan 978.
## 2 Albania 5937.
## 3 Algeria 6223.
## 4 Angola 5523.
## 5 Argentina 12779.
## 6 Australia 34435.
## 7 Austria 36126.
## 8 Bahrain 29796.
## 9 Bangladesh 1391.
## 10 Belgium 33693.
## # ... with 132 more rowsEl combo group_by() + summarize() es clave porque permite empezar a explorar relaciones entre variables. Por ejemplo, continente y PIB per cápita:
summarize(
group_by(gapminder, continent),
gdpPercap_media = mean(gdpPercap, na.rm = TRUE)
)## `summarise()` ungrouping output (override with `.groups` argument)## # A tibble: 5 x 2
## continent gdpPercap_media
## <fct> <dbl>
## 1 Africa 2194.
## 2 Americas 7136.
## 3 Asia 7902.
## 4 Europe 14469.
## 5 Oceania 18622.6.9 Datos no disponibles: na_if(), replace_na() y drop_na()
En R, las celdas con datos disponibles deben aparecer como NA. Este valor es distinto al texto “NA” o cosas como “N/A”, “No disponible” y “-”. NaN es similar, pero distinto: “Not a Number”. En R, los datos no disponibles deben aparecer como NA para que poder tratarlos correctamente. Con dplyr, contamos con tres funciones para lidiar con valores NA: na_if(), replace_na() y drop_na().
6.9.1 Convertir a NA
Algunas bases de datos especifican en el libro de códigos como vienen los NA. En el caso de Polity IV, los valores -66 pueden ser interpretados como valores no disponibles. Si miramos la variables exconst podemos entender un poco mejor a qué nos referimos:
polity4 %>% count(exconst)## # A tibble: 10 x 2
## exconst n
## <dbl> <int>
## 1 -88 328
## 2 -77 214
## 3 -66 234
## 4 1 4777
## 5 2 952
## 6 3 3798
## 7 4 363
## 8 5 1316
## 9 6 758
## 10 7 4655Podemos cambiar estos valores a NA en una variable con na_if() dentro de un mutate().
polity4 <- polity4 %>%
mutate(
exconst_mod = na_if(exconst, "-66") # variable nueva; podríamos reescribir la original
)
count(polity4, exconst_mod) # revisamos que los convertimos a NA## # A tibble: 10 x 2
## exconst_mod n
## <dbl> <int>
## 1 -88 328
## 2 -77 214
## 3 1 4777
## 4 2 952
## 5 3 3798
## 6 4 363
## 7 5 1316
## 8 6 758
## 9 7 4655
## 10 NA 234Este cambio a NA l podemos realizar para una variable solamente o para toda la base de datos.
polity4 <- na_if(polity4, "-66") # reemplazamos los valores -66 con NA en la totalidad de la base de datosComo “-77” y “-88” también pueden ser vistos como NA, repetimos la operaciones:
polity4 <- na_if(polity4, "-77")
polity4 <- na_if(polity4, "-88")
count(polity4, exconst)## # A tibble: 8 x 2
## exconst n
## <dbl> <int>
## 1 1 4777
## 2 2 952
## 3 3 3798
## 4 4 363
## 5 5 1316
## 6 6 758
## 7 7 4655
## 8 NA 7766.9.2 Reemplazar NA
Puede suceder que hay valores que aparecen como NA pero que sabemos que no lo son. Por ejemplo, puede que sean igual a 0 y no indiquen una falta de datos. Para tratar con estas situaciones, usamos replace_na():
polity4 %>%
mutate(
exconst_mod2 = replace_na(exconst_mod, "-999")
) %>%
count(exconst_mod2)## # A tibble: 8 x 2
## exconst_mod2 n
## <chr> <int>
## 1 -999 776
## 2 1 4777
## 3 2 952
## 4 3 3798
## 5 4 363
## 6 5 1316
## 7 6 758
## 8 7 46556.9.3 Descartar NA
Por último, si queremos descartar las observaciones que tienen valores NA, usamos drop_na(). Nuevamente, lo podemos usar para una variable -descartar observaciones con NA en esa columna en particular- o para toda la base de datos -descartar observaciones con NA en cualquier variable. Comparemos el número de observaciones cuando descartamos filas con NA en la variable polity2:
drop_na(polity4, polity2)## # A tibble: 17,159 x 13
## ccode country year polity2 parreg parcomp exconst democracia regimen
## <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <fct> <fct>
## 1 2 United~ 1800 4 4 2 7 otro anocra~
## 2 2 United~ 1801 4 4 2 7 otro anocra~
## 3 2 United~ 1802 4 4 2 7 otro anocra~
## 4 2 United~ 1803 4 4 2 7 otro anocra~
## 5 2 United~ 1804 4 4 2 7 otro anocra~
## 6 2 United~ 1805 4 4 2 7 otro anocra~
## 7 2 United~ 1806 4 4 2 7 otro anocra~
## 8 2 United~ 1807 4 4 2 7 otro anocra~
## 9 2 United~ 1808 4 4 2 7 otro anocra~
## 10 2 United~ 1809 9 2 4 7 democracia democr~
## # ... with 17,149 more rows, and 4 more variables: regimen_cut <fct>,
## # regimen_bin <fct>, regimen_num <dbl>, exconst_mod <dbl>Con el número de filas restantes cuando descartamos filas con NA en cualquier columna:
drop_na(polity4)## # A tibble: 16,619 x 13
## ccode country year polity2 parreg parcomp exconst democracia regimen
## <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <fct> <fct>
## 1 2 United~ 1800 4 4 2 7 otro anocra~
## 2 2 United~ 1801 4 4 2 7 otro anocra~
## 3 2 United~ 1802 4 4 2 7 otro anocra~
## 4 2 United~ 1803 4 4 2 7 otro anocra~
## 5 2 United~ 1804 4 4 2 7 otro anocra~
## 6 2 United~ 1805 4 4 2 7 otro anocra~
## 7 2 United~ 1806 4 4 2 7 otro anocra~
## 8 2 United~ 1807 4 4 2 7 otro anocra~
## 9 2 United~ 1808 4 4 2 7 otro anocra~
## 10 2 United~ 1809 9 2 4 7 democracia democr~
## # ... with 16,609 more rows, and 4 more variables: regimen_cut <fct>,
## # regimen_bin <fct>, regimen_num <dbl>, exconst_mod <dbl>6.10 Simplificar código: tuberías %>%
Ya que dominamos las principales formas de trabajar con datos, nos damos cuenta que el código a veces se vuelve engorroso y largo. En particular, se vuelve difícil hacerle seguimiento a todos los paréntesis incluidos. En esta sección, damos un paso adelante hacia la simplificación de nuestro código a través del uso del operador %>%.
El operador %>% (pipe, tubo o o tubería) sirve para simplificar nuestro código. Viene de la librería magrittr (¿sí agarran la referencia?) y es usado extensamente en el tidyverse – si cargamos esta librería, podemos usar %>%. Para entender la utilidad de los pipes, comparemos distintas formas de usar varias funciones al mismo tiempo:
- Anidadas: se vuelve confuso tener tantos paréntesis.
head(arrange(select(filter(gapminder, year == 2007, continent == "Americas"), country, gdpPercap), desc(gdpPercap)))## # A tibble: 6 x 2
## country gdpPercap
## <fct> <dbl>
## 1 United States 42952.
## 2 Canada 36319.
## 3 Puerto Rico 19329.
## 4 Trinidad and Tobago 18009.
## 5 Chile 13172.
## 6 Argentina 12779.Romper el código en líneas -como hemos hecho hasta ahora- ayuda un poco a entender qué está pasando, pero sigue exigiendo jugar una ronda de “veo, veo”:
head(
arrange(
select(
filter(gapminder, year == 2007, continent == "Americas"), country, gdpPercap
),
desc(gdpPercap)
)
)## # A tibble: 6 x 2
## country gdpPercap
## <fct> <dbl>
## 1 United States 42952.
## 2 Canada 36319.
## 3 Puerto Rico 19329.
## 4 Trinidad and Tobago 18009.
## 5 Chile 13172.
## 6 Argentina 12779.- Paso a paso: ineficiente, pues estamos crenado nuevos objetos “intermedios” o temporales que luego debemos eliminar (para eso está
rm()).
gapminder_2007 <- filter(gapminder, year == 2007, continent == "Americas")
gapminder_2007 <- select(gapminder_2007, country, gdpPercap)
gapminder_2007 <- arrange(gapminder_2007, desc(gdpPercap))
head(gapminder_2007)## # A tibble: 6 x 2
## country gdpPercap
## <fct> <dbl>
## 1 United States 42952.
## 2 Canada 36319.
## 3 Puerto Rico 19329.
## 4 Trinidad and Tobago 18009.
## 5 Chile 13172.
## 6 Argentina 12779.rm(gapminder_2007)- Pipes: “entonces”. Se leen izquierda-derecha o de arriba-abajo, si partimos el código en líneas:
# tomar un vector numérico
c(1, 1, 2, 3, 5, 8, 13, 21) %>%
# encontrar la media
mean() %>%
# redondear
round() ## [1] 7Pueden usarse como “tuberías” que conectan varias funciones, como filter(), select() y arrange(), cada una construyendo sobre los resultados que arroja la anterior, para analizar una base de datos:
gapminder %>%
# filtrar por valores de year y continent
filter(year == 2007, continent == "Americas") %>%
# seleccionar solo las variables country y gdpPercap
select(country, gdpPercap) %>%
# ordenar descendente segun gdpPercap
arrange(desc(gdpPercap)) %>%
# ver las primeras filas
head() ## # A tibble: 6 x 2
## country gdpPercap
## <fct> <dbl>
## 1 United States 42952.
## 2 Canada 36319.
## 3 Puerto Rico 19329.
## 4 Trinidad and Tobago 18009.
## 5 Chile 13172.
## 6 Argentina 12779.Con tuberías más largas, podemos responder a preguntas interesantes y comparaciones relevantes de forma más eficiente. Por ejemplo, digamos que queremos encontrar la media continental del PIB per cápita en el último año para el que tenemos información, porque estamos interesados en explorar la variación espacial en la riqueza de las naciones. ¿Cómo lo hacemos? ¡Pues armamos una tubería con group_by() y summarize()! Comparar medidas entre dos grupos nos permite ver la relación entre una variable numérica y una categórica:
gapminder %>%
filter(year == max(year, na.rm = TRUE)) %>% # el ultimo año presente en los datos
group_by(continent) %>% # agrupar por continente
summarize( # hacemos un resumen
pib_media = mean(gdpPercap, na.rm = TRUE),
pib_mediana = median(gdpPercap, na.rm = TRUE),
pib_max = max(gdpPercap, na.rm = TRUE),
pib_min = min(gdpPercap, na.rm = TRUE),
pib_de = sd(gdpPercap, na.rm = TRUE),
num_casos = n() # n() cuenta las observaciones por grupo
)## `summarise()` ungrouping output (override with `.groups` argument)## # A tibble: 5 x 7
## continent pib_media pib_mediana pib_max pib_min pib_de num_casos
## <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <int>
## 1 Africa 3089. 1452. 13206. 278. 3618. 52
## 2 Americas 11003. 8948. 42952. 1202. 9713. 25
## 3 Asia 12473. 4471. 47307. 944 14155. 33
## 4 Europe 25054. 28054. 49357. 5937. 11800. 30
## 5 Oceania 29810. 29810. 34435. 25185. 6541. 2O nos interesa saber cuál es el promedio de varios indicadores de la base de datos de Polity IV para cada tipo de régimen (usando la variable categórica que creamos anteriormente):
polity4 %>%
filter(year %in% c(1997:2017)) %>% # solo unos años
group_by(regimen) %>% # agrupamos por tipo de régimen (demo-ano-auto)
summarize( # hacemos un resumen
media_polity2 = mean(polity2, na.rm = TRUE),
media_parreg = mean(parreg, na.rm = TRUE),
media_parcomp = mean(parcomp, na.rm = TRUE)
)## `summarise()` ungrouping output (override with `.groups` argument)## # A tibble: 3 x 4
## regimen media_polity2 media_parreg media_parcomp
## <fct> <dbl> <dbl> <dbl>
## 1 autocracia -7.61 3.90 1.43
## 2 anocracia 0.309 2.80 2.75
## 3 democracia 8.51 3.29 4.23La versión summarize_if() nos permite seleccionar variables a resumir si cumplen alguna condición:
polity4 %>%
filter(year %in% c(1997:2017)) %>% # solo unos años
group_by(regimen) %>% # agrupamos por tipo de régimen (demo-ano-auto)
summarize_if( # hacemos un resumen
is.numeric, # condicion
mean, na.rm = TRUE
)## # A tibble: 3 x 9
## regimen ccode year polity2 parreg parcomp exconst regimen_num exconst_mod
## <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 autocracia 616. 2006. -7.61 3.90 1.43 1.94 -1 1.94
## 2 anocracia 540. 2007. 0.309 2.80 2.75 3.52 0 3.52
## 3 democracia 382. 2007. 8.51 3.29 4.23 6.53 1 6.53summarize_at() nos permite usar otro tipo de condiciones, como solo resumir variables que empiezan por “pa”:
polity4 %>%
filter(year %in% c(1997:2017)) %>% # solo unos años
group_by(regimen) %>% # agrupamos por tipo de régimen (demo-ano-auto)
summarize_at( # hacemos un resumen
vars(starts_with("pa")),
mean, na.rm = TRUE
)## # A tibble: 3 x 3
## regimen parreg parcomp
## <fct> <dbl> <dbl>
## 1 autocracia 3.90 1.43
## 2 anocracia 2.80 2.75
## 3 democracia 3.29 4.23Las tablas cruzados que hicimos arriba también se benfician del uso de pipes. Hagamos una tabla que tabule y muestre la relación entre la variable continente (continent) y la variable binaria de nivel de ingreso alto o bajo que construimos anteriormente (pib_dummy). Usando prop.table() podemos hacer esta tabla con proporciones en vez de cuentas. Al final, guardamos como archivo de Word.
Noten que aquí agregamos una función más: ungroup() - si queremos seguir la tubería sin hacer operaciones por grupo, esta función nos permite hacerlo, de lo contrario todas las operaciones se harían por grupos.
gapminder %>%
count(continent, pib_dummy) %>%
group_by(continent) %>%
mutate(prop = round(prop.table(n), 2)) %>%
ungroup() %>%
kable(
format = "html",
col.names = c("Continente", "Nivel PIB", "Casos", "Prop."),
caption = "Tabla cruzada",
format.args = list(decimal.mark = ",")
) %>%
write_file("output/tabla2.doc")Otro uso común de group_by() seguido de summarize() es agregar datos. Al agrupar y resumir podemos cambiar el nivel de análisis. Por ejemplo, en vez de país-año podemos pasar a continente-año: cada fila sería un continente en un año y las columnas son resúmenes (la media en este caso) de las variables para cada continente. Por cierto, tanto summarize() como summarise() son válidas.
gapminder_cont <- gapminder %>%
group_by(continent, year) %>%
summarise_at(vars(lifeExp:gdp), mean, na.rm = TRUE) %>%
ungroup()
gapminder_cont## # A tibble: 60 x 6
## continent year lifeExp pop gdpPercap gdp
## <fct> <int> <dbl> <dbl> <dbl> <dbl>
## 1 Africa 1952 39.1 4570010. 1253. 5992294608.
## 2 Africa 1957 41.3 5093033. 1385. 7359188796.
## 3 Africa 1962 43.3 5702247. 1598. 8784876958.
## 4 Africa 1967 45.3 6447875. 2050. 11443994101.
## 5 Africa 1972 47.5 7305376. 2340. 15072241974.
## 6 Africa 1977 49.6 8328097. 2586. 18694898732.
## 7 Africa 1982 51.6 9602857. 2482. 22040401045.
## 8 Africa 1987 53.3 11054502. 2283. 24107264108.
## 9 Africa 1992 53.6 12674645. 2282. 26256977719.
## 10 Africa 1997 53.6 14304480. 2379. 30023173824.
## # ... with 50 more rowsPor último, estos pipes y los verbos que hemos utilizado sirven para hacer gráficas de manera eficiente y clara.
gapminder_cont %>% # tomar datos
ggplot(mapping = aes(x = log(gdpPercap), y = lifeExp)) + # seleccionar variables
geom_point(color = "darkblue") + # gráfica de dispersion
labs(x = "PIB per cápita (USD, log.)", y = "Expectativa de vida al nacer (años)") # titulos de los ejes
Construyamos una gráfica similar, pero trabajando con los datos a nivel-país año:
gapminder %>% # tomar datos
filter(year %in% c(1962, 1972, 1982, 1992, 2002)) %>% # filtrar para incluir ciertos anos
ggplot(mapping = aes(x = log(gdpPercap), y = lifeExp)) + # seleccionar variables
geom_point(color = "darkblue", alpha = 0.5) + # gráfica de dispersion, transparencia 50%
labs(x = "PIB per cápita (USD, log.)", y = "Expectativa de vida al nacer (años)") # titulos de los ejes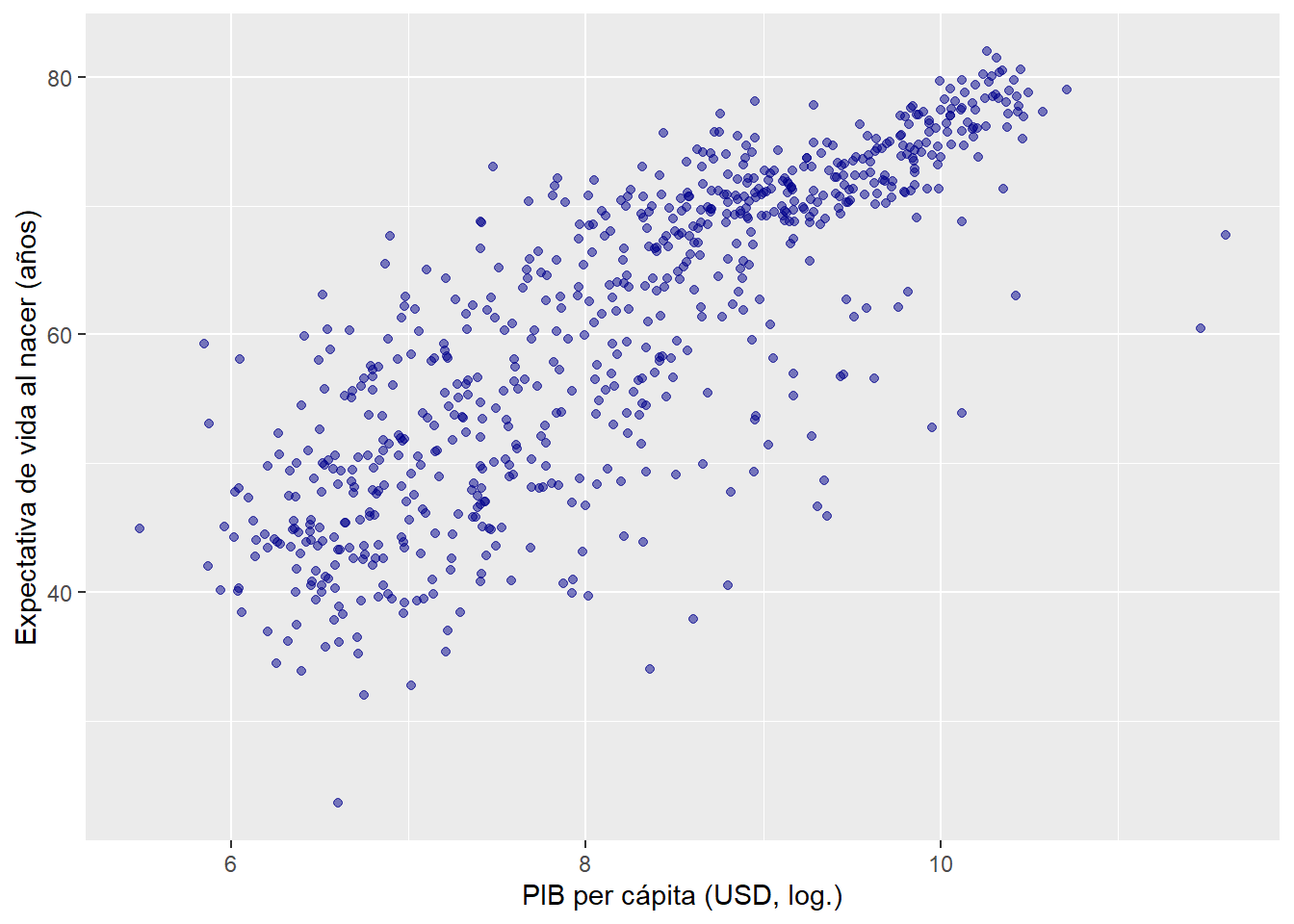
Finalmente, si queremos ver las trayectorias de los distintos continentes, usamos el agumento color = de ggplot() para graficar una línea de color distinto para cada grupo:
gapminder_cont %>%
ggplot(aes(year, log(gdpPercap), color = continent)) +
geom_line() +
labs(x = "Año", y = "PIB per cápita (USD, log.)", color = "Continente")
6.11 Ordenar y reformatear: pivot_()
Muy frecuentemente, los datos que encontramos están desordenados. Por ejemplo, usualmente debemos corregir los NA.
Otro paso habitual es “limpiar” los nombres de las variables. No es recomendable tener espacios o caracteres especiales en los nombres de las variables y en general de los objetos en R. La librería janitor facilita hacer estos cambios con la función clean_names(), la cual automáticamente cambia el formato del nombre de las variables a snake_case.
library(janitor)Veamos esto que hace con gapminder:
gapminder %>% clean_names() %>% head()## # A tibble: 6 x 10
## country continent year life_exp pop gdp_percap gdp gdp_log
## <fct> <fct> <int> <dbl> <int> <dbl> <dbl> <dbl>
## 1 Afghan~ Asia 1952 28.8 8.43e6 779. 6.57e 9 22.6
## 2 Afghan~ Asia 1957 30.3 9.24e6 821. 7.59e 9 22.7
## 3 Afghan~ Asia 1962 32.0 1.03e7 853. 8.76e 9 22.9
## 4 Afghan~ Asia 1967 34.0 1.15e7 836. 9.65e 9 23.0
## 5 Afghan~ Asia 1972 36.1 1.31e7 740. 9.68e 9 23.0
## 6 Afghan~ Asia 1977 38.4 1.49e7 786. 1.17e10 23.2
## # ... with 2 more variables: gdp_percap_log <dbl>, pib_dummy <chr>Lidiar con NA y nombres de variables es sencillo. Más complejo es asegurarse que una base de datos estén ordenadas (“tidy data”). Es el principio central del tidyverse(). Según este principio, cada fila debe ser una observacion y cada columna una variable. A veces es conocido como formato “largo” (el otro es “ancho”). gapminder está en formato largo y tidy. Cada fila es una observación, cada columna una variable.
sample_n(gapminder, 10) ## ver 15 filas aleatorias de los datos## # A tibble: 10 x 10
## country continent year lifeExp pop gdpPercap gdp gdp_log
## <fct> <fct> <int> <dbl> <int> <dbl> <dbl> <dbl>
## 1 El Sal~ Americas 2002 70.7 6.35e6 5352. 3.40e10 24.2
## 2 Guinea Africa 1967 37.2 3.45e6 709. 2.45e 9 21.6
## 3 Puerto~ Americas 1987 74.6 3.44e6 12281. 4.23e10 24.5
## 4 Djibou~ Africa 2007 54.8 4.96e5 2082. 1.03e 9 20.8
## 5 Sudan Africa 1952 38.6 8.50e6 1616. 1.37e10 23.3
## 6 Hungary Europe 1982 69.4 1.07e7 12546. 1.34e11 25.6
## 7 Bulgar~ Europe 1997 70.3 8.07e6 5970. 4.82e10 24.6
## 8 Bahrain Asia 1997 73.9 5.99e5 20292. 1.21e10 23.2
## 9 Slovak~ Europe 1977 70.4 4.83e6 10923. 5.27e10 24.7
## 10 Albania Europe 1957 59.3 1.48e6 1942. 2.87e 9 21.8
## # ... with 2 more variables: gdpPercap_log <dbl>, pib_dummy <chr>A continuación, veamos un ejemplo sencillo de datos desordenados, o en formato ancho.
ancho <- tibble(
pais = c("Colombia", "Argentina", "Brazil"),
indicador_2000 = rnorm(3, 2),
indicador_2010 = rnorm(3, 3),
indicador_2020 = rnorm(3, 4)
)
ancho## # A tibble: 3 x 4
## pais indicador_2000 indicador_2010 indicador_2020
## <chr> <dbl> <dbl> <dbl>
## 1 Colombia 3.42 4.26 3.67
## 2 Argentina 3.68 3.54 4.89
## 3 Brazil 0.531 2.77 3.66Según el principio tidy, debería haber tres columnas: pais, indicador y ano. Aquí, en vez de hacerlo completamente “a mano”, usamos expand_grid() para repetir los nombres de país y años, para luego agregar el indicador.
largo <- expand_grid(
pais = c("Colombia", "Argentina", "Brazil"),
ano = c(2000, 2010, 2020)
) %>%
mutate(indicador = rnorm(9, 3))
largo## # A tibble: 9 x 3
## pais ano indicador
## <chr> <dbl> <dbl>
## 1 Colombia 2000 4.82
## 2 Colombia 2010 1.51
## 3 Colombia 2020 3.20
## 4 Argentina 2000 3.57
## 5 Argentina 2010 3.02
## 6 Argentina 2020 3.67
## 7 Brazil 2000 2.32
## 8 Brazil 2010 5.57
## 9 Brazil 2020 1.87Pero, ¿cómo hacemos para pasar fácilmente de un formato al otro? Para reformatear datos de esta manera hacemos uso de funciones de la librería tidyr. Usamos pivot_wider() para pasar de largo a ancho y pivot_longer() para hacer lo contrario.
6.11.1 Largo a ancho
Ocasionalmente, tenemos datos en formato largo y queremos pasarlos a un forma más ancho. Esto puede ser porque queremos hacer una tabla para presentar la información (una tabla muy larga es difícil de leer) o porque queremos cambiar el nivel de análisis de los datos (“agregarlos”) para analizarlos o graficarlos.
gapminder_ancho <- gapminder %>%
pivot_wider(
id_cols = country,
names_from = year,
names_prefix = "year_",
values_from = lifeExp,
)
gapminder_ancho## # A tibble: 142 x 13
## country year_1952 year_1957 year_1962 year_1967 year_1972 year_1977 year_1982
## <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Afghan~ 28.8 30.3 32.0 34.0 36.1 38.4 39.9
## 2 Albania 55.2 59.3 64.8 66.2 67.7 68.9 70.4
## 3 Algeria 43.1 45.7 48.3 51.4 54.5 58.0 61.4
## 4 Angola 30.0 32.0 34 36.0 37.9 39.5 39.9
## 5 Argent~ 62.5 64.4 65.1 65.6 67.1 68.5 69.9
## 6 Austra~ 69.1 70.3 70.9 71.1 71.9 73.5 74.7
## 7 Austria 66.8 67.5 69.5 70.1 70.6 72.2 73.2
## 8 Bahrain 50.9 53.8 56.9 59.9 63.3 65.6 69.1
## 9 Bangla~ 37.5 39.3 41.2 43.5 45.3 46.9 50.0
## 10 Belgium 68 69.2 70.2 70.9 71.4 72.8 73.9
## # ... with 132 more rows, and 5 more variables: year_1987 <dbl>,
## # year_1992 <dbl>, year_1997 <dbl>, year_2002 <dbl>, year_2007 <dbl>6.11.2 Ancho a largo:
Es más común tener que pasar de ancho (desordenado) a largo (ordenado). Usamos pivot_longer() para realizar este reformateo:
gapminder_largo <- gapminder_ancho %>% # creamos un objeto nuevo usando los datos "anchos"
pivot_longer(
-country,
names_to = "year",
names_prefix = "year_",
values_to = "lifeExp"
) %>%
arrange(country, year)
gapminder_largo## # A tibble: 1,704 x 3
## country year lifeExp
## <fct> <chr> <dbl>
## 1 Afghanistan 1952 28.8
## 2 Afghanistan 1957 30.3
## 3 Afghanistan 1962 32.0
## 4 Afghanistan 1967 34.0
## 5 Afghanistan 1972 36.1
## 6 Afghanistan 1977 38.4
## 7 Afghanistan 1982 39.9
## 8 Afghanistan 1987 40.8
## 9 Afghanistan 1992 41.7
## 10 Afghanistan 1997 41.8
## # ... with 1,694 more rowsConvertimos el ejemplo que construimos arriba:
ancho %>%
pivot_longer(
-pais,
names_to = "ano",
names_prefix = "indicador_",
values_to = "indicador"
)## # A tibble: 9 x 3
## pais ano indicador
## <chr> <chr> <dbl>
## 1 Colombia 2000 3.42
## 2 Colombia 2010 4.26
## 3 Colombia 2020 3.67
## 4 Argentina 2000 3.68
## 5 Argentina 2010 3.54
## 6 Argentina 2020 4.89
## 7 Brazil 2000 0.531
## 8 Brazil 2010 2.77
## 9 Brazil 2020 3.666.11.3 Datos del Banco Mundial
La librería tidyr incluye unos datos del Banco Mundial.
world_bank_pop## # A tibble: 1,056 x 20
## country indicator `2000` `2001` `2002` `2003` `2004` `2005` `2006`
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 ABW SP.URB.T~ 4.24e4 4.30e4 4.37e4 4.42e4 4.47e+4 4.49e+4 4.49e+4
## 2 ABW SP.URB.G~ 1.18e0 1.41e0 1.43e0 1.31e0 9.51e-1 4.91e-1 -1.78e-2
## 3 ABW SP.POP.T~ 9.09e4 9.29e4 9.50e4 9.70e4 9.87e+4 1.00e+5 1.01e+5
## 4 ABW SP.POP.G~ 2.06e0 2.23e0 2.23e0 2.11e0 1.76e+0 1.30e+0 7.98e-1
## 5 AFG SP.URB.T~ 4.44e6 4.65e6 4.89e6 5.16e6 5.43e+6 5.69e+6 5.93e+6
## 6 AFG SP.URB.G~ 3.91e0 4.66e0 5.13e0 5.23e0 5.12e+0 4.77e+0 4.12e+0
## 7 AFG SP.POP.T~ 2.01e7 2.10e7 2.20e7 2.31e7 2.41e+7 2.51e+7 2.59e+7
## 8 AFG SP.POP.G~ 3.49e0 4.25e0 4.72e0 4.82e0 4.47e+0 3.87e+0 3.23e+0
## 9 AGO SP.URB.T~ 8.23e6 8.71e6 9.22e6 9.77e6 1.03e+7 1.09e+7 1.15e+7
## 10 AGO SP.URB.G~ 5.44e0 5.59e0 5.70e0 5.76e0 5.75e+0 5.69e+0 4.92e+0
## # ... with 1,046 more rows, and 11 more variables: `2007` <dbl>, `2008` <dbl>,
## # `2009` <dbl>, `2010` <dbl>, `2011` <dbl>, `2012` <dbl>, `2013` <dbl>,
## # `2014` <dbl>, `2015` <dbl>, `2016` <dbl>, `2017` <dbl>Los datos está en formato ancho. Nuestro objetivo es tener una base de datos donde cada variable esté en una columna y cada fila sea una observación. Nuestro problema es que el año está en multiples columnas.
pop2 <- world_bank_pop %>%
pivot_longer(
`2000`:`2017`, # comillas para que R entienda que son nombres de columnas, no valores
names_to = "year",
values_to = "value"
)
pop2## # A tibble: 19,008 x 4
## country indicator year value
## <chr> <chr> <chr> <dbl>
## 1 ABW SP.URB.TOTL 2000 42444
## 2 ABW SP.URB.TOTL 2001 43048
## 3 ABW SP.URB.TOTL 2002 43670
## 4 ABW SP.URB.TOTL 2003 44246
## 5 ABW SP.URB.TOTL 2004 44669
## 6 ABW SP.URB.TOTL 2005 44889
## 7 ABW SP.URB.TOTL 2006 44881
## 8 ABW SP.URB.TOTL 2007 44686
## 9 ABW SP.URB.TOTL 2008 44375
## 10 ABW SP.URB.TOTL 2009 44052
## # ... with 18,998 more rowsAun tenemos que considerar la variable indicator:
pop2 %>% count(indicator)## # A tibble: 4 x 2
## indicator n
## <chr> <int>
## 1 SP.POP.GROW 4752
## 2 SP.POP.TOTL 4752
## 3 SP.URB.GROW 4752
## 4 SP.URB.TOTL 4752Aquí:
SP.POP.GROWes la tasa de crecimiento poblacional.SP.POP.TOTLes la población total.SP.URB.*es lo mismo, pero para áreas urbanas
Dividamos indicator dos variables, área (total o urbana) y
la variable en si (población o crecimiento). Usamos la función separate():
pop3 <- pop2 %>%
separate(indicator, c(NA, "area", "variable"))
pop3## # A tibble: 19,008 x 5
## country area variable year value
## <chr> <chr> <chr> <chr> <dbl>
## 1 ABW URB TOTL 2000 42444
## 2 ABW URB TOTL 2001 43048
## 3 ABW URB TOTL 2002 43670
## 4 ABW URB TOTL 2003 44246
## 5 ABW URB TOTL 2004 44669
## 6 ABW URB TOTL 2005 44889
## 7 ABW URB TOTL 2006 44881
## 8 ABW URB TOTL 2007 44686
## 9 ABW URB TOTL 2008 44375
## 10 ABW URB TOTL 2009 44052
## # ... with 18,998 more rowsAhora podemos terminar de limpiar los datos: convertir los valores TOTAL y GROW en variables:
wb_tidy <- pop3 %>%
pivot_wider(names_from = variable, values_from = value)
wb_tidy## # A tibble: 9,504 x 5
## country area year TOTL GROW
## <chr> <chr> <chr> <dbl> <dbl>
## 1 ABW URB 2000 42444 1.18
## 2 ABW URB 2001 43048 1.41
## 3 ABW URB 2002 43670 1.43
## 4 ABW URB 2003 44246 1.31
## 5 ABW URB 2004 44669 0.951
## 6 ABW URB 2005 44889 0.491
## 7 ABW URB 2006 44881 -0.0178
## 8 ABW URB 2007 44686 -0.435
## 9 ABW URB 2008 44375 -0.698
## 10 ABW URB 2009 44052 -0.731
## # ... with 9,494 more rowsGuardamos los datos como archivo tipo .csv:
write_csv(wb_tidy, "data/wb_tidy.csv")6.12 Combinar/unir: _join()
Tenemos dos bases de datos relacionadas, de pronto porque ambas incluyen información sobre los mismos casos, y queremos combinarlas. Volvamos a los datos de Polity IV:
polity4## # A tibble: 17,395 x 13
## ccode country year polity2 parreg parcomp exconst democracia regimen
## <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <fct> <fct>
## 1 2 United~ 1800 4 4 2 7 otro anocra~
## 2 2 United~ 1801 4 4 2 7 otro anocra~
## 3 2 United~ 1802 4 4 2 7 otro anocra~
## 4 2 United~ 1803 4 4 2 7 otro anocra~
## 5 2 United~ 1804 4 4 2 7 otro anocra~
## 6 2 United~ 1805 4 4 2 7 otro anocra~
## 7 2 United~ 1806 4 4 2 7 otro anocra~
## 8 2 United~ 1807 4 4 2 7 otro anocra~
## 9 2 United~ 1808 4 4 2 7 otro anocra~
## 10 2 United~ 1809 9 2 4 7 democracia democr~
## # ... with 17,385 more rows, and 4 more variables: regimen_cut <fct>,
## # regimen_bin <fct>, regimen_num <dbl>, exconst_mod <dbl>Pero esta base de datos solo tiene informacion sobre algunas características institucionales de la democracia. ¿Qué pasa si queremos explorar la relación de estas medidas con otros factores? Necesitaríamos unir otra base de datos que tenga información sobre los mismos casos (o un subconjunto de estos). Por ejemplo, miremos la _base de datos Database of Political Institutions. Cargamos el archivo (en formato .dta de Stata), seleccionamos unas variables y hacemos un poco de limpieza.
library(haven)
dpi <- read_dta("data/DPI2017.dta") %>%
select(countryname, ifs, year, system, pr, numopp) %>%
zap_labels() %>% # quita las etiquetas de variable de Stata
na_if("-999")
sample_n(dpi, 10)## # A tibble: 10 x 6
## countryname ifs year system pr numopp
## <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 UK GBR 1991 2 0 270
## 2 Kuwait KWT 1977 0 NA 0
## 3 Belarus BLR 1975 NA NA 0
## 4 Poland POL 2006 0 1 215
## 5 Mali MLI 2003 0 0 119
## 6 Congo (DRC) ZAR 2015 0 1 322
## 7 Cent. Af. Rep. CAF 2010 0 0 20
## 8 Thailand THA 2007 2 0 0
## 9 Kyrgyzstan KGZ 1992 0 NA 0
## 10 Somalia SOM 2011 0 NA 0Ahora tenemos dos bases de datos y queremos unirlas o combinarlas. Seguimos tres pasos para alcanzar este objetivo.
- Encontrar variables en común.
- Combinar las dos bases de datos.
- Guardar el resultado como un nuevo objeto en R.
6.12.1 Variables en común
Debe haber variables en común para poder unir dos bases de datos. También debemos pensar en la unidad de análisis: ¿a qué corresponde cada fila en las bases de datos?
En este ejemplo, ambas bases de datos están en un formato largo de país-año: cada fila es un país observado en un año (repetidamente, o sea, es un panel). Entonces, podemos usar el año y el nombre de los de países para unir las bases de datos pues son elementos en común. Por un lado, polity4 y dpi ambas tienen la columna year que representa el año de la observación.
Por otro lado, es recomendable usar códigos de país, en vez de nombres, ya que los códigos tienen estándares y los nombres no tanto (y dependen del idioma también). Los códigos de país de dpi, que están en la columna ifs, siguen el estándar del Fondo Monetario Internacional (FMI). Por tanto, convertimos los codigos de polity4 a ese formato, apoyándonos en la libreria countrycode. Creamos una nueva variable que aloja los códigos de país según el estándar del FMI, usando los códigos de país que ya había en polity4 (la columna ccode, que sigue el estándar numérico del Correlates of War, COW) como fuente.
library(countrycode)
polity4 <- polity4 %>%
mutate(
ifs = as.character(countrycode(
sourcevar = ccode, # variable de origen
origin = "cown", # formato de origen: COW numerico
destination = "iso3c" # formato de destino: ISO 3 caracteres
))
) %>%
select(ifs, ccode, year, polity2, regimen)## Warning: Problem with `mutate()` input `ifs`.
## i Some values were not matched unambiguously: 89, 99, 245, 260, 265, 267, 269, 271, 315, 324, 329, 332, 335, 337, 342, 345, 347, 348, 364, 525, 529, 564, 678, 680, 730, 769, 817, 818
##
## i Input `ifs` is `as.character(...)`.## Warning in countrycode(sourcevar = ccode, origin = "cown", destination = "iso3c"): Some values were not matched unambiguously: 89, 99, 245, 260, 265, 267, 269, 271, 315, 324, 329, 332, 335, 337, 342, 345, 347, 348, 364, 525, 529, 564, 678, 680, 730, 769, 817, 818polity4## # A tibble: 17,395 x 5
## ifs ccode year polity2 regimen
## <chr> <dbl> <dbl> <dbl> <fct>
## 1 USA 2 1800 4 anocracia
## 2 USA 2 1801 4 anocracia
## 3 USA 2 1802 4 anocracia
## 4 USA 2 1803 4 anocracia
## 5 USA 2 1804 4 anocracia
## 6 USA 2 1805 4 anocracia
## 7 USA 2 1806 4 anocracia
## 8 USA 2 1807 4 anocracia
## 9 USA 2 1808 4 anocracia
## 10 USA 2 1809 9 democracia
## # ... with 17,385 more rows6.12.2 Combinar bases de datos
Ya tenemos la primera parte del proceso: encontramos variables en común. Para unir o combinar las bases de datos, usamos las funciones join() de dplyr.
Existen varios tipos de “joins”. Podemos ejecutar ?dplyr::join para ver todas las opciones. Hay dos grandes categorías, cada una con varios tipos de “joins”. Asumiendo que x y y son dos bases de datos relacionadas con variables en común:
- Joins para “mutar” datos: todas toman dos bases de datos (X y Y) y las combinan Combinar variables de X y de Y
- left_join(x, y): El mas comun es left_join(X, Y) Mantiene todas las filas de X y todas las columnas de X y Y Filas de X sin pareja en Y tienen NA en las nuevas columnas
- inner_join(x, y): Mantiene solo las filas de X que tienen pareja en Y y todas las columnas de X y Y
Si hay multiples parejas, mantiene todas las combinaciones
- right_join(x, y): Complemento de left_join()
Mantiene todas las filas de Y y todas las columnas de X y Y
Filas de Y sin pareja en X tienen NA en las nuevas columnas
- full_join(x, y): Combinacion completa Mantiene todas las filas y columnas de X y Y. Si no hay parejas, da NA en esas columnas
- Joins para “filtrar” datos: unir para mantener las filas de X
- semi_join(x, y): Todas las filas de X con pareja en Y
Solo columnas de X
- anti_join(x, y): Todas las filas de X sin pareja en Y Solo columnas de X
Usualmente usamos left_join(), la cual es la más común y cubre la mayoría de nuestras necesidades. Esta función une las dos bases de datos, agregando las columnas de una (aquí, dpi) a la otra (polity4). Este “join” mantiene todas las filas de la primera base de datos (polity4), las filas de ambas que coinciden (según las variables en común que especificamos) y todas las columnas de las dos bases de datos. Las filas de la primera base de datos que no tienen una “pareja” en la otra, aparecen con valores NA en las nuevas columnas (las de dpi que se agregan a polity4). Revisamos mirando algunos países:
datos <- polity4 %>%
left_join(
dpi,
by = c("ifs", "year") # variables para combinar; mismo nombre y tipo
)
datos %>% select(ifs, year, polity2, regimen, pr, numopp) %>% sample_n(10)## # A tibble: 10 x 6
## ifs year polity2 regimen pr numopp
## <chr> <dbl> <dbl> <fct> <dbl> <dbl>
## 1 MWI 1981 -9 autocracia NA 0
## 2 URY 1926 3 anocracia NA NA
## 3 BRA 1902 -3 anocracia NA NA
## 4 NZL 1961 10 democracia NA NA
## 5 NIC 1841 -5 anocracia NA NA
## 6 NIC 1991 6 democracia 1 41
## 7 FRA 1808 -8 autocracia NA NA
## 8 <NA> 1891 1 anocracia NA NA
## 9 FIN 1984 10 democracia 1 77
## 10 RUS 2008 4 anocracia 1 135Podemos guardar o exportar este nuevo objeto en diversos formatos. Por ejemplo, write_excel_csv() crea archivos .csv para Excel.
write_excel_csv(datos, path = "data/datos_polity_dpi.csv")Unir estas dos bases relacionadas nos permite evaluar más hipótesis. Podríamos sugerir que la presencia de una oposición fuerte (en términos de su presencia en el legislativo) lleva a un nivel de democracia más alto en términos de límites al ejecutivo. O al revés: cuando hay niveles de democracia liberal altos en los que se limita el poder del ejecutivo, es más probable que la oposición creza. Pero ¿hay alguna asociación empírica entre el número de curules que tiene la oposición y el nivel de democracia de un país?
datos %>%
ggplot(aes(x = factor(polity2), y = numopp)) +
geom_boxplot() +
labs(x = "Indicador de democracia", y = "Número de curules de la oposición")
6.12.3 Homicidios en Medellin
Un último ejemplo para entender cómo unir bases de datos y lo que esto nos permite hacer. Supongamos que estamos interesados en analizar los homicidios de Medellín por zona a través del tiempo. Sin embargo, solo tenemos datos sobre homicidios en Medellin (tomados del SISC) en formato comuna-año. ¡No podemos hacer el análisis por zona si no tenemos una columna que nos indique la zona a la que pertenece cada observación!
homicidio <- read_csv("data/mde_homicidio.csv")## Parsed with column specification:
## cols(
## cod_comuna = col_character(),
## ano = col_double(),
## homicidios_total = col_double(),
## homicidios_jovenes = col_double()
## )homicidio## # A tibble: 335 x 4
## cod_comuna ano homicidios_total homicidios_jovenes
## <chr> <dbl> <dbl> <dbl>
## 1 01 2003 119 83
## 2 01 2004 36 26
## 3 01 2005 24 16
## 4 01 2006 23 16
## 5 01 2007 24 15
## 6 01 2008 39 20
## 7 01 2009 180 113
## 8 01 2010 133 82
## 9 01 2011 25 18
## 10 01 2012 37 22
## # ... with 325 more rowsAfortunadamente, encontramos un archivo .csv que contiene el listado comunas y corregimientos de Medellín y las zonas a las que pertenecen. Está en formato comuna-año para preparar un panel de datos y podemos usarlo para añadir a los datos sobre homicidios las variables cod_zona y nom_zona. Esto nos ahorra tener que clasificar las observaciones a mano.
mde <- read_csv("data/mde_df.csv")## Parsed with column specification:
## cols(
## cod_comuna = col_character(),
## ano = col_double(),
## nom_comuna = col_character(),
## cod_zona = col_double(),
## nom_zona = col_character()
## )mde## # A tibble: 420 x 5
## cod_comuna ano nom_comuna cod_zona nom_zona
## <chr> <dbl> <chr> <dbl> <chr>
## 1 01 2000 Popular 1 Zona 1 - Nororiental
## 2 02 2000 Santa Cruz 1 Zona 1 - Nororiental
## 3 03 2000 Manrique 1 Zona 1 - Nororiental
## 4 04 2000 Aranjuez 1 Zona 1 - Nororiental
## 5 05 2000 Castilla 2 Zona 2 - Noroccidental
## 6 06 2000 Doce de Octubre 2 Zona 2 - Noroccidental
## 7 07 2000 Robledo 2 Zona 2 - Noroccidental
## 8 08 2000 Villa Hermosa 3 Zona 3 - Centroriental
## 9 09 2000 Buenos Aires 3 Zona 3 - Centroriental
## 10 10 2000 La Candelaria 3 Zona 3 - Centroriental
## # ... with 410 more rowsEn ambas bases de datos hay codigos de comuna y años en común. Y las variables tienen la misma clase (caracter y numérico, respectivamente). Están relacionadas y podemos unirlas; queremos unirlas porque nos interesan los homicidios por zona, no por comuna. Como ya habíamos mencionado, podemos especificar las columnas que queremos usar para combinar las bases de datos (las columnas que identifican a cada observación). Si tienen nombres distintos en las dos bases de datos, podemos ser más explícitos usando "nombre_en_x" = "nombre_en_y".
mde_right <- left_join(
homicidio, mde,
by = c("cod_comuna" = "cod_comuna", "ano" = "ano")
)
mde_right## # A tibble: 335 x 7
## cod_comuna ano homicidios_total homicidios_jove~ nom_comuna cod_zona
## <chr> <dbl> <dbl> <dbl> <chr> <dbl>
## 1 01 2003 119 83 Popular 1
## 2 01 2004 36 26 Popular 1
## 3 01 2005 24 16 Popular 1
## 4 01 2006 23 16 Popular 1
## 5 01 2007 24 15 Popular 1
## 6 01 2008 39 20 Popular 1
## 7 01 2009 180 113 Popular 1
## 8 01 2010 133 82 Popular 1
## 9 01 2011 25 18 Popular 1
## 10 01 2012 37 22 Popular 1
## # ... with 325 more rows, and 1 more variable: nom_zona <chr>Ahora tenemos la información necesaria para nuestro análisis. Empezamos usando group_by() y summarize() para agregar los datos a nivel de zona-año: ahora, tenemos el número de homicidios por zona en cada año.
mde_zonas <- mde_right %>%
group_by(nom_zona, ano) %>% # agrupar por zona y ano
summarize(
homicidios_total = sum(homicidios_total, na.rm = TRUE) # homicidios por comuna y ano
) %>%
ungroup() %>%
drop_na() # eliminar NA## `summarise()` regrouping output by 'nom_zona' (override with `.groups` argument)mde_zonas## # A tibble: 112 x 3
## nom_zona ano homicidios_total
## <chr> <dbl> <dbl>
## 1 Corregimientos 2003 57
## 2 Corregimientos 2004 14
## 3 Corregimientos 2005 20
## 4 Corregimientos 2006 15
## 5 Corregimientos 2007 14
## 6 Corregimientos 2008 17
## 7 Corregimientos 2009 106
## 8 Corregimientos 2010 150
## 9 Corregimientos 2011 253
## 10 Corregimientos 2012 205
## # ... with 102 more rowsPor último, construimos una gráfica con funciones de ggplot2, capa por capa: una serie de tiempo anual de los homicidios en la ciudad, zona a zona (color = nom_zona crea una línea por zona de la ciudad). Agregamos títulos, cambiamos colores, etc. y guardamos la gráfica como un archivo .png con ggsave().
mde_zonas %>%
ggplot(aes(x = ano, y = homicidios_total, color = nom_zona)) + # asignar variables
geom_line(size = 1, alpha = 0.75) + # gráfica de lineas
scale_x_continuous(breaks = seq(min(mde_right$ano), max(mde_right$ano))) + # etiquetas de eje X
scale_color_brewer(palette = "Dark2") + # paleta de colores
labs(
title = "Heterogeneidad temporal y espacial del homicidio en Medellín", # título
subtitle = "Homicidios por comuna, Medellín, 2003-2018", # subtítulo
caption = "Fuente: Elaboración propia con datos del Sistema de Información para la Seguridad y Convivencia (SISC).", # pie de imagen
x = NULL, y = "Número de homicidios", color = NULL # títulos de ejes y leyenda
) +
theme_classic(base_size = 12) + # fondo, tema y tamaño de letra
theme(legend.position = "bottom") + # mover leyenda
ggsave("output/homicidios_mde.png") # guardar## Saving 7 x 5 in image
6.13 Repaso
Trabajar con bases de datos:
- Subir datos a la nube usando la opción
Uploadde las pestañaFiles. - Cargar datos: usamos las funciones incluidas en
readr,readxlohaven. - Limpiar:
na_if()para convertir aNA. - Seleccionar y realizar subconjuntos de datos:
- Columnas/variables/propiedades con
select(). - Filas/observaciones/casos con
filter().
- Columnas/variables/propiedades con
- Crear/modificar variables:
%>%(“pipes”) para pasar objetos a las funciones.mutate()para crear nuevas variables usando variables existentes, por ejemplo, para crear proporciones y porcentajes.mutate_if()para crear/cambiar si cumple condiciones.as.numeric(),as.factor(),as.integer(),as.character()para cambiar la clase de una variable.if_else()crea una variable dummy/binaria.case_when()crea una variable nominal u ordinal.factor()y funcionesfct_()para crear un factor o cambiar su orden y categorías.
- Resumir y agregar datos con
group_by()ysummarize(). - Modificar la estructura de bases de datos:
- Cambiar de formato largo a ancho con
pivot_longer()ypivot_wider(). - Combinar dos bases de datos relacionadas con
left_join().
- Cambiar de formato largo a ancho con
6.14 Taller: funciones y datos
6.14.1 Crear una base de datos
Construir, guardar e imprimir un objeto tibble llamado datos_pib que contenga la siguiente información en filas y columnas:
- Colombia es un país democrático en América con un PIB per cápita de 6,651 USD y una expectativa de vida de 77 años.
- En el régimen dictatorial de Corea del Norte, ubicado en Asia, la expectativa de vida es de unos 67 años.
- En España, una democracia europea, la expectativa de vida es 83 años y el PIB per cápita es de 30,524 dólares.
- El PIB per cápita de Arabia Saudí, una dictadura en Asia donde la expectativa de vida es 73 años, es de 23,219 USD.
- En Sudán, el PIB per cápita es de 977 dólares y la expectativa de vida es 61 años; este país es una dictadura africana.
Usando datos_pib, encontrar (1.0 punto):
- El promedio del PIB per cápita para la muestra.
- La correlación de esta variable con la expectativa de vida al nacer.
6.14.2 Mostrar relaciones
Usando la función apropiada, cargar el archivo de datos datos_taller1.csv (pueden descargarlo de aquí a la carpeta "data/) como un objeto tipo tibble.
La base de datos incluye las siguientes variables tomadas de Varieties of Democracy, v. 10:
vdem_country_name: nombre del país.year: año de la observación.v2x_polyarchy: indicador (numérico) de democracia electoral.v2elparlel: tipo de sistema electoral; puede tomar cuatro valores: mayoritario, representación proporcional y “otros” (incluye mixtos).v2psoppaut: indicador (numérico) de grado de autonomía de partidos en la oposición.v2elmulpar: indicador (binario) de multipartidismo; puede tomar dos valores: “no/limitado” y “sí”.
Usando estos datos y las funciones apropiadas en R (1.0 punto):
- Construir una tabla cruzada que cuente cuántos países con sistemas mayoritarios tienen sistemas multipartidistas.
- Construir e interpretar una gráfica sencilla que muestre la relación entre el grado de autonomía de la oposición y el nivel de democracia electoral.
6.14.3 Seleccionar observaciones
Cargar a la sesión de R la base de datos que piensan utilizar en el proyecto de investigación del curso. Utilizar las funciones y operadores apropiados para seleccionar las filas (casos) potencialmente interesantes o relevantes para la investigación. Guardar este subconjunto de datos (un “subset”) como un objeto nuevo tipo data.frame o tibble nuevo (1.0 punto).
- Bono: Convertir todos los nombres de columna a
snake_case(usando funciones, no a mano). Esto les ahorrará dolores de cabeza a largo plazo. - Bono: mostrar que los valores
NAde las variables de interés -si los hay- están codificados apropiadamente (no como-999,"N/A"o similares). Esto les ahorrará dolores de cabeza a largo plazo.
6.14.4 Seleccionar variables
Usando el objeto creado en el punto anterior (el subconjunto de datos), utilizar las funciones y operadores apropiados para seleccionar las variables que tengan más sentido para su investigación. Guardar este subconjunto de datos como un objeto tipo data.frame o tibble. A su vez, deben guardar este objeto como un archivo de datos en "data/"; este archivo puede ser .rds, .csv, .xlsx, .dta, etc. (1.0 punto).
6.14.5 Medias de grupos
Estimar la media de una variable numérica de interés (puede ser una variable independiente o dependiente) y comparar la media de esta variable para por lo menos dos grupos o categorías de interés teórico. En otras palabras, estimar la media de una variable para varios grupos o categorías. Interpretar los resultados (1.0 punto).
- Bono: realizar e interpretar una prueba \(t\) de Student para evaluar si estas dos medias son diferentes en términos estadísticos. Pista: la librería
inferofrece una función para hacerlo, pero R ya incluye una. - Bono: realizar e interpretar una tabla cruzada de dos variables y utilizar una prueba \(\chi^{2}\) para evaluar si las dos variables son independientes. Pista: la librería
inferofrece una función para hacerlo, pero R ya incluye una.
En RStudio Cloud, vamos al panel inferior derecho, pestaña
Files, click enUploady seguimos las indicaciones.↩︎También los podemos cargar usando los menús del programa; el asistente para cargar datos en RStudio (pestaña
Environment, opciónImport Dataset) es bastante bueno y nos arroja código que después debemos copiar en un Rmarkdown o R script para replicar nuestros análisis.↩︎