Chapter 8 資料基本處理
tidyverse 套件系統 包含許多不同套件, 提供資料科學一些實用的函式. 包含
- tidyverse 核心
ggplot2視覺化分析.purrr執行迴圈.tibble增強資料框架.dplyr資料處裡.tidyr精簡資料,stringr字串整理.readr資料輸入.forcats處理類別變數 (factors).
tidyverse 套件系統的其他關聯套件包含
- 資料輸入 import
readxl輸入 excel 關聯檔案.
haven輸入 SPSS, Stata 與 SAS 關聯檔案.jsonlite輸入 JSON 關聯檔案.xml2輸入 XML 關聯檔案.httr輸入 web APIs 關聯檔案.rvest輸入 web scraping 關聯檔案.
- DBI 輸入 關聯資料庫, 協同套件有
RSQLite,RPostgres或odbc. - 資料處理 tidy/wrangle
stringr處理字串.lubridate處理日期與時間.forcats處理類別變數 (factors).hms處理日期與時間.blob處理二元資料儲存檔案.
- 程式寫作 program
rlang提供寫作核心語言與 tidyverse.magrittr提供資料流動通道%>%.glue提供字串合併.
- 建模 model
broom處理建模結果.modelr偕同執行建模族.
8.1 資料輸入套件 readr
tidyverse 套件系統中的 readr 套件提供一些方便資料輸入的函式.
包含
read_csv() 輸入 .csv 資料檔,
read_excel 輸入 excel 資料檔,
read_delim() 輸入不同分隔符號資料檔.
其中引數如下 (help(read_delim)).
read_delim(
file,
delim,
quote = "\"",
escape_backslash = FALSE,
escape_double = TRUE,
col_names = TRUE,
col_types = NULL,
locale = default_locale(),
na = c("", "NA"),
quoted_na = TRUE,
comment = "",
trim_ws = FALSE,
skip = 0,
n_max = Inf,
guess_max = min(1000, n_max),
progress = show_progress(),
skip_empty_rows = TRUE
)file= 路徑與檔名delim= 分隔符號quote= 視同分隔符號 (資料的文字變數值常放在雙引號中)escape_backslash= 預設 FALSE, 是否有逃脫符號escape_double= 預設TRUE, 是否用引號符號作為逃脫符號col_names= 設定變數名 (T或F)col_types= 設定變數的類型na= 設定 NA 符號comment= 設定注釋符號, 在注釋符號之後的文字不會被讀入trim_ws= 去除變數值得空白skip= 要跳過幾行 (row) 才開始讀入資料n_max= 最大輸入行數
糖尿病與人工關節術後感染臨床試驗 (糖尿病與人工全膝關節置換手術研究)
退化性膝關節炎是人類關節疾病中最常見的一種,
主要病變的地方是膝關節的的關節軟骨受到傷害與磨損,
造成關節腔變窄以及產生骨刺, 變化與年紀有高度相關.
年長者患有嚴重退化性膝關節炎, 常須進行人工全膝關節置換手術 (TKR),
但是若是術後感染, 則有可能照造成截肢或因敗血症而死亡,
且糖尿病 (DM) 患者是此全膝關節置換手術後感染的高危險群.
因此一位骨科醫師進行一預防性臨床試驗,
對患有嚴重退化性膝關節炎的糖尿病患者,
隨機分配 2 組, 一組在全膝關節置換手術中, 使用的骨水泥內,
加入預防性抗生素藥劑, 另一組為對照組.
研究的主要目的是探討全膝關節置換手術中,
在骨水泥內加入預防性抗生素藥劑, 是否可以預防術後感染.
另外一個研究目的是在骨水泥內加入預防性抗生素藥劑,
研究共收集了 78 位病患的資料,
變數名稱列在表 1.
資料檔案為 DMTKRcsv.csv, DMTKAInfMo.csv,
DMTKRtabsep.txt 與 DMTKRblanksep.txt.
| 變數 | 說明 |
|---|---|
| NO | 病患辨識碼 |
| AGE | 年紀歲數 (單位: 年) (age) |
| SEX | 性別: 0 = F = 女 (female), 1 = M = 男 (Male). |
| DM | 糖尿病形態: 0 = Type II (NIDDM); 1 = Type I (IDDM) |
| DMYR | 手術前糖尿病病史 (單位: 年) |
| PREAC | 手術前空腹血糖 (mg/dL) |
| PREPC | 手術前飯後血糖 (mg/dL) |
| POSTAC | 手術後空腹血糖 (mg/dL) |
| POSTPC | 手術後飯後血糖 (mg/dL) |
| MEDICATION | 糖尿病治療方式: |
| 0 = 口服降血糖藥劑; 1 = 胰島素注射; 2 = 飲食控制 | |
| SIDE | 患側: 0 = 左側; 1 = 右側 |
| PREKS | 手術前膝功能分數 |
| POSTKS | 手術後膝功能分數 |
| ABS | 骨水泥是否加抗生素 1 = Yes; 0 = No |
| INFECT | 術後感染: 0 = No; 1 = Yes, 發現術後感染 |
| INFMO | 發現術後感染的時間 (單位: 月) |
| MISC | 註記 |
Table 1. 糖尿病與人工關節術後感染臨床試驗: 變數說明
# .csv
library(tidyverse)
## Warning: package 'tibble' was built under R version 4.0.3
## Warning: package 'readr' was built under R version 4.0.3
library(readr)
dd <- readr::read_csv("./Data/DMTKAInfMo.csv")
print(dd, n = 5, width = Inf)
## # A tibble: 78 x 16
## No age sex DM DMyr preAC prePC postAC postPC medication SIDE PREKS POSTKS
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 67 0 0 10 120 160 140 180 0 0 56 92
## 2 2 67 0 0 11 100 150 150 220 0 1 62 62
## 3 3 72 1 0 4 150 200 120 150 2 0 60 94
## 4 4 82 1 0 8 150 200 160 250 0 1 47 90
## 5 5 73 1 0 3 85 110 140 200 0 0 44 88
## ABS INFECT INFMO
## <dbl> <dbl> <dbl>
## 1 1 0 0
## 2 0 1 2
## 3 1 0 0
## 4 1 0 0
## 5 0 0 0
## # ... with 73 more rows
# .xls
library(readxl)
dd <- readxl::read_excel("C:/RData/DMTKAInfMo.xls")
print(dd, n = 5, width = Inf)
## # A tibble: 78 x 16
## No age sex DM DMyr preAC prePC postAC postPC medication SIDE PREKS POSTKS
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 67 0 0 10 120 160 140 180 0 0 56 92
## 2 2 67 0 0 11 100 150 150 220 0 1 62 62
## 3 3 72 1 0 4 150 200 120 150 2 0 60 94
## 4 4 82 1 0 8 150 200 160 250 0 1 47 90
## 5 5 73 1 0 3 85 110 140 200 0 0 44 88
## ABS INFECT I0FECTMO
## <dbl> <dbl> <dbl>
## 1 1 0 0
## 2 0 1 2
## 3 1 0 0
## 4 1 0 0
## 5 0 0 0
## # ... with 73 more rows8.2 整潔資料 Tidy Data
統計計算可以進行分析的資料,
通常有一個的簡單的基本架構,
在 {R} 稱作
資料框架
(data frame).
資料框架是類似於在 SAS, STATA 等的 dataset 架構.
資料框架通常類似矩陣,
資料框架也類似矩形的
交叉列聯表
(cross table),
稱為
資料表
(** data table).
套件 tidverse 原始套件 tidy` 強調外部資料檔案必須符合
整潔資料** (tidy data) 方便操作.
整潔資料的基本要求特徵是
每個變數各自形成 1 欄 (縱行, row),
每個列 (橫列, row) 各自為一個觀測時間的測量.
例如, 每位個體只有觀測一次,
則每個列為一位個體的觀測值.
若每位個體有多次觀測時間點,
則一位個體一次觀測時間點的觀測值為一列,
一位個體或有多列的觀測值.
一個整潔資料的資料表包含以下重點.
- 一個檔案只用一張資料表
- 一張資料表 (EXCEL 的 sheet).
- 一個欄位 (縱行, Column) 只有一個變數, 同時有清楚的變數名.
- 若完整資料包含不同資料表, 則不同資料表要有索引 (inxex) 或指標變數 (id) 可進行關聯與串聯.

Tidy Data: R for Data Science, Figure 12.1
整潔資料 指引主要提供研究者在輸入資料檔案時能夠遵循.
例如, 一些者常將 EXCEL 一張 sheel 內混合著原始資料, 分析結果與圖形.
也經常將二個變數放在同一個欄位, 二個變數簡單以 / 分隔.
例如檔案 DMTKAORI.xls.
若資料檔案不是整潔資料, 則需浩費學多時間清理, 甚至必須重新輸入到資料檔案.
8.3 Tibble 與 Data Frame
tidyverse 系統中的 readr 套件輸入資料後,
tidyverse 會儲存成 (tibble) 資料物件,
成為tbl_df, tbl 類型的資料物件,
主要是 tidyverse 系統會使用 tibble 套件進行操作.
tibbles 資料物件與原有資料框架 data.frame 物件有相同的屬性外,
另外多了些讓 tidyverse 系統容易操作的屬性.
對初學者而言, 二者幾呼無任何差異.
若有 {R} base 函式無法操作 tibble 資料物件,
可使用 as.data.frame()
轉成 {R} base 的 data.frame 物件.
同樣, 若 tidyverse 系統函式無法操作的 data.frame 物件,
可用 ‵as_tibble()將data.frame物件 轉成tibbles` 資料物件.
以資料在檔案第 5 章的 survVATrial.csv 為例.
## data frame object
dd <- read.table("./Data/survVATrial.csv",
header = TRUE,
sep = ",",
quote = "\"'",
dec = ".",
row.names = NULL,
# col.names,
as.is = TRUE,
# as.is = !stringsAsFactors,
na.strings = c(".", "NA"))
class(dd)
## [1] "data.frame"
library(tibble)
dd <- as_tibble(dd)
class(dd)
## [1] "tbl_df" "tbl" "data.frame"tibble 套件的 tibble() 函式的使用如同 {R} base data.frame().
tibble() 函式可以使用 {R} base 不准使用的變數名.
tibble 物件與 data.frame 物件的主要差別有 2 種情境.
tibble物件的顯示,print()只用 10 rows, 變數 (columns) 只有納入符合視窗寬度的數目, 這對大數據檢視較為方便. 且顯示str()函式可呈現變數基本模式的類別. 可使用n = k顯示 \(k\) rows,width = Inf顯示所有變數. 也可設定整區選項為 options(tibble.print_min = Inf), 顯示所有 rows, 以及options(tibble.width = Inf)顯示所有 columns.{R} base 多數容許使用部分物件名 (變數名), 但
tibble物件無法使用部分變數名. 可能會對資深程式寫作人員些許困擾.
8.4 資料流動管道運算指令 Pipe
tidyverse 系統中的 magrittr 套件提供一個非常實用的運算指令
%>% 稱為 pipe, 管線, 管道, 導管.
此運算符號, 可將函式與運算串聯,
%>% 左側通常是資料物件, 資料框架, 矩陣, 向量等,
%>% 右側通常是函式 fun_name(),
而 %>% 左側資料自動為函式 fun_name() 內的第一個引數.
在 Unix/Linux 系統的各種指令可以運用 pipe 的方式串連執行,
magrittr 套件提供類似的思維,
且資料流動從 %>% 左測的資料送到 %>%右側的函式執行運算, 可讓程式寫作符合文字書寫由左到右的思考邏輯. 使用pipe` 並不會改變程式的執行順序,
主要目的在於讓程式更容易寫作與閱讀,
降低程式寫作的錯誤與負擔.
## short and clean
log(mean(c(1:10)))
## [1] 1.705
## easily read
x <- c(1:10)
x.mean <- mean(x)
log.mean <- log(x.mean)
log.mean
## [1] 1.705
## pipe %>%
library(magrittr)
c(1:10) %>% mean() %>% log()
## [1] 1.7058.5 資料檢視函式 glimpse()
資料輸入到 {R} 後, 必須先檢視資料內容, 比對原始資料檔案是否正確讀入資料. 包含觀測值數目, 變數數目, 那些變數為辨識指標, 變數屬性, 那些變數為連續變數或類別變數, 類別變數屬性, 名目變數或是順序變數, 整理類別變數的類別水準名稱. 每個變數的缺失數目與頻率, 每個個體的缺失數目與頻率. 以資料在檔案第 5 章的 survVATrial.csv 為例.
dd = dd %>% as_tibble()
print(dd, n = 5, width = Inf)
## # A tibble: 137 x 8
## treat cellcode time censor diagtime kps age prior
## <int> <int> <int> <int> <int> <int> <int> <int>
## 1 0 1 72 1 60 7 69 0
## 2 0 1 411 1 70 5 64 10
## 3 0 1 228 1 60 3 38 0
## 4 0 1 126 1 60 9 63 10
## 5 0 1 118 1 70 11 65 10
## # ... with 132 more rows可以看到資料有 137 位個體, 8 個變數.
其中time, diagtime, kps, age 為連續變數.
treat, cellcode, censor, prior 等為類別變數,
但類別變數以數字輸入, 需先設定成為類別變數.
以資料在檔案第 5 章的 survVATrial.csv 為例.
dd$treat <- factor(dd$treat, labels = c("placebo", "test"))
dd$cellcode <- factor(dd$cellcode,
labels = c("squamous", "small", "adeno", "large"))
dd$censor <- factor(dd$censor, labels = c("survival", "dead"))
dd$prior <- factor(dd$prior, labels = c("no", "yes"))
print(dd, n = 5, width = Inf)
## # A tibble: 137 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo squamous 72 dead 60 7 69 no
## 2 placebo squamous 411 dead 70 5 64 yes
## 3 placebo squamous 228 dead 60 3 38 no
## 4 placebo squamous 126 dead 60 9 63 yes
## 5 placebo squamous 118 dead 70 11 65 yes
## # ... with 132 more rowstidyverse 系統中的 tibble 套件提供一個檢視資料函式 `glimpse(),
類似 {R} base 的 str() 函式.
## R base
str(dd)
## tibble [137 x 8] (S3: tbl_df/tbl/data.frame)
## $ treat : Factor w/ 2 levels "placebo","test": 1 1 1 1 1 1 1 1 1 1 ...
## $ cellcode: Factor w/ 4 levels "squamous","small",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ time : int [1:137] 72 411 228 126 118 10 82 110 314 100 ...
## $ censor : Factor w/ 2 levels "survival","dead": 2 2 2 2 2 2 2 2 2 1 ...
## $ diagtime: int [1:137] 60 70 60 60 70 20 40 80 50 70 ...
## $ kps : int [1:137] 7 5 3 9 11 5 10 29 18 6 ...
## $ age : int [1:137] 69 64 38 63 65 49 69 68 43 70 ...
## $ prior : Factor w/ 2 levels "no","yes": 1 2 1 2 2 1 2 1 1 1 ...
## glimpse()
glimpse(dd)
## Rows: 137
## Columns: 8
## $ treat <fct> placebo, placebo, placebo, placebo, placebo, placebo, placebo, pla...
## $ cellcode <fct> squamous, squamous, squamous, squamous, squamous, squamous, squamo...
## $ time <int> 72, 411, 228, 126, 118, 10, 82, 110, 314, 100, 42, 8, 144, 25, 11,...
## $ censor <fct> dead, dead, dead, dead, dead, dead, dead, dead, dead, survival, de...
## $ diagtime <int> 60, 70, 60, 60, 70, 20, 40, 80, 50, 70, 60, 40, 30, 80, 70, 60, 60...
## $ kps <int> 7, 5, 3, 9, 11, 5, 10, 29, 18, 6, 4, 58, 4, 9, 11, 3, 9, 2, 4, 4, ...
## $ age <int> 69, 64, 38, 63, 65, 49, 69, 68, 43, 70, 81, 63, 63, 52, 48, 61, 42...
## $ prior <fct> no, yes, no, yes, yes, no, yes, no, no, no, no, yes, no, yes, yes,...8.6 資料處裡 dplyr 套件
tidyverse 系統中的資料處裡 dplyr 套件,
有一些資料資料處裡常用函式.
%>%=pipe指令rename()= 變數 (column) 重新命名filter()= 選擇個體或行位子集 (rows)arrange()= 依據變數值排序select()= 選擇變數 (variables) 或欄位子集 (columns)mutate()= 變數轉換sample_n()與sample_frac()= 隨機抽樣函式distinct()與n_distinct()= 選出明顯不同個體slice()= 利用橫列指標 (row index) 選出個體 (row)summarise()= 計算常見統計量group_by()= 資料分組操作
這些函式的第 1 個引數為資料物件,
後續只用為變數名 (不加雙引號),
可以合併使用 group_by() 函式與 %>% 指令.
8.6.1 選擇個體函式 filter()
資料常常需要依據納入與排除條件,
此時會依據變數值進行篩選.
filter() 函式可協助選擇個體.
以資料在檔案第 5 章的 survVATrial.csv 為例,
選擇個體 treat 為 placebo, cellcode 為 large.
## filter()
dd.a <- dd %>%
filter(treat == 'placebo', cellcode == 'large')
dd.a
## # A tibble: 15 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo large 177 dead 50 16 66 yes
## 2 placebo large 162 dead 80 5 62 no
## 3 placebo large 216 dead 50 15 52 no
## 4 placebo large 553 dead 70 2 47 no
## 5 placebo large 278 dead 60 12 63 no
## 6 placebo large 12 dead 40 12 68 yes
## 7 placebo large 260 dead 80 5 45 no
## 8 placebo large 200 dead 80 12 41 yes
## 9 placebo large 156 dead 70 2 66 no
## 10 placebo large 182 survival 90 2 62 no
## 11 placebo large 143 dead 90 8 60 no
## 12 placebo large 105 dead 80 11 66 no
## 13 placebo large 103 dead 80 5 38 no
## 14 placebo large 250 dead 70 8 53 yes
## 15 placebo large 100 dead 60 13 37 yes選擇個體 treat 為試驗藥組, age 大於 50, kps 小於或等於 7
dd.b <- dd %>%
filter(treat == 'test', age > 50, kps <= 7)
dd.b
## # A tibble: 35 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 test squamous 112 dead 80 6 60 no
## 2 test squamous 242 dead 50 1 70 no
## 3 test squamous 111 dead 70 3 62 no
## 4 test squamous 587 dead 60 3 58 no
## 5 test squamous 389 dead 90 2 62 no
## 6 test squamous 33 dead 30 6 64 no
## 7 test squamous 467 dead 90 2 64 no
## 8 test squamous 283 dead 90 2 51 no
## 9 test small 25 dead 30 2 69 no
## 10 test small 21 dead 20 4 71 no
## # ... with 25 more rows8.6.2 依據變數值排序函式 arrange()
資料常常需要依據變數值排序,
arrange() 函式內設依照變數在函式出現順序以及變數值由小到大排序.
若要變數值從由大到小排序, 可使用 desc() 函式.
例如, 以資料在檔案第 5 章的 survVATrial.csv 為例,
依照 age 由小到大排序,
time 由大到小排序.
無論如何排序, 缺失值總是排在最後,
這須與 {R} base 的 order(), sort() 或 rank() 進行比較.
## arrange()
dd.s <- dd %>%
arrange(age, desc(time))
dd.s
## # A tibble: 137 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo adeno 95 dead 80 4 34 no
## 2 placebo small 4 dead 40 2 35 no
## 3 test squamous 1 dead 50 7 35 no
## 4 test small 103 survival 70 22 36 yes
## 5 placebo large 100 dead 60 13 37 yes
## 6 test large 49 dead 30 3 37 no
## 7 placebo squamous 228 dead 60 3 38 no
## 8 placebo adeno 117 dead 80 2 38 no
## 9 placebo large 103 dead 80 5 38 no
## 10 test adeno 31 dead 80 3 39 no
## # ... with 127 more rows8.6.3 選擇變數或欄位子集函式 select()
面對大數據時, 可能有成千上萬的變數,
但通常部不會使用到所有的變數,
選擇所需要的變數另組成分析子集,
可減少 RAM 的負擔並加速分析執行速度.
以資料在檔案第 5 章的 survVATrial.csv 為例,
選擇 treat, cellcode, censor 等變數.
dd.c <- dd %>%
select(treat, cellcode, censor)
dd.c
## # A tibble: 137 x 3
## treat cellcode censor
## <fct> <fct> <fct>
## 1 placebo squamous dead
## 2 placebo squamous dead
## 3 placebo squamous dead
## 4 placebo squamous dead
## 5 placebo squamous dead
## 6 placebo squamous dead
## 7 placebo squamous dead
## 8 placebo squamous dead
## 9 placebo squamous dead
## 10 placebo squamous survival
## # ... with 127 more rows8.6.4 變數轉換函式 mutate()
資料分析前常常需要變數進行轉換, 例如取對數轉換,
標準化, 也常將二個以上不同變數進行計算, 轉換成新變數,
例如計算 BMI (生體質量指數).
以資料在檔案第 5 章的 survVATrial.csv 為例,
對 time 取對數轉換, diagtime * age / 100.
## mutate()
dd.a <- dd %>%
mutate(
log_age = log(age),
diag_age = diagtime * age / 100
)
dd.a %>%
select(age, diagtime, log_age, diag_age) %>%
print(n = 5, width = Inf)
## # A tibble: 137 x 4
## age diagtime log_age diag_age
## <int> <int> <dbl> <dbl>
## 1 69 60 4.23 41.4
## 2 64 70 4.16 44.8
## 3 38 60 3.64 22.8
## 4 63 60 4.14 37.8
## 5 65 70 4.17 45.5
## # ... with 132 more rows8.6.5 向量 if_else()
dplyr 套件中的函式 if_else() 與 {R} base 函式 ifelse() 有類似功能,
執行特殊的變數轉換.
if_else(condition, true, false, missing = NULL)引數 condition 為邏輯向量,
true 與 false 為相同模式且相同長度的向量或相同模式的純量.
當 condition = TRUE, 則回傳 true,
反之, 當 condition = FALSE, 則回傳 false.
x <- c(NA, -2:2, NA)
x
## [1] NA -2 -1 0 1 2 NA
## R base
ifelse(x > 0, 1, 0)
## [1] NA 0 0 0 1 1 NA
## if_else
if_else(x > 0, 1, 0)
## [1] NA 0 0 0 1 1 NA
if_else(x < 0, "negative", "positive", missing = "missing value")
## [1] "missing value" "negative" "negative" "positive" "positive"
## [6] "positive" "missing value"
if_else(is.na(x), 1, 0) %>% mean()
## [1] 0.28578.6.6 變數重新命名 rename()
dplyr 套件中的函式 rename() 可將變數重新命名.
rename(new_name = old_name)以資料在檔案第 5 章的 survVATrial.csv 為例,
將變數 treat 重新命名為 drug.
## original
print(dd, n = 5, width = Inf)
## # A tibble: 137 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo squamous 72 dead 60 7 69 no
## 2 placebo squamous 411 dead 70 5 64 yes
## 3 placebo squamous 228 dead 60 3 38 no
## 4 placebo squamous 126 dead 60 9 63 yes
## 5 placebo squamous 118 dead 70 11 65 yes
## # ... with 132 more rows
## change
dd.new <- dd %>% rename(drug = treat)
print(dd.new, n = 5, width = Inf)
## # A tibble: 137 x 8
## drug cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo squamous 72 dead 60 7 69 no
## 2 placebo squamous 411 dead 70 5 64 yes
## 3 placebo squamous 228 dead 60 3 38 no
## 4 placebo squamous 126 dead 60 9 63 yes
## 5 placebo squamous 118 dead 70 11 65 yes
## # ... with 132 more rows8.6.7 移除缺失資料 drop_na()
tidyr 套件中的函式 drop_na() 可將資料內具有缺失值得個體移除,
但這樣處理資料必須非常小心解讀.
dd.mis <- tibble(age = c(30, 40, NA, 60),
gender = c("M", "M", "F", NA))
dd.mis
## # A tibble: 4 x 2
## age gender
## <dbl> <chr>
## 1 30 M
## 2 40 M
## 3 NA F
## 4 60 <NA>
dd.mis %>% drop_na()
## # A tibble: 2 x 2
## age gender
## <dbl> <chr>
## 1 30 M
## 2 40 M
dd.mis %>% drop_na(age)
## # A tibble: 3 x 2
## age gender
## <dbl> <chr>
## 1 30 M
## 2 40 M
## 3 60 <NA>
dd.mis %>% drop_na(gender)
## # A tibble: 3 x 2
## age gender
## <dbl> <chr>
## 1 30 M
## 2 40 M
## 3 NA F8.6.8 隨機抽樣函式 sample_n() 與 sample_frac()
函式 sample_n() 與 sample_frac()
可以對資料進行隨機抽樣.
引數為
size = k設定所要抽出之樣本數或分率.weight抽取之相對應權重.- 若無設定值, 則每一個個體被抽取之相對應權重相等.
replace = FALSE邏輯指令, 設定是否可重複抽取.
以資料在檔案第 5 章的 survVATrial.csv 為例, 由資料抽樣 5 位個體.
## sample_n()
set.seed(1)
dd %>% sample_n(size = 5, replace = FALSE)
## # A tibble: 5 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo large 250 dead 70 8 53 yes
## 2 test large 53 dead 60 12 66 no
## 3 placebo small 63 dead 50 11 48 no
## 4 placebo squamous 25 survival 80 9 52 yes
## 5 placebo adeno 12 dead 50 4 63 yes
set.seed(10)
dd %>% sample_n(size = 5, replace = TRUE)
## # A tibble: 5 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 test large 49 dead 30 3 37 no
## 2 test squamous 242 dead 50 1 70 no
## 3 test adeno 51 dead 60 5 62 no
## 4 test squamous 87 survival 80 3 48 no
## 5 test squamous 283 dead 90 2 51 no
set.seed(100)
dd %>% sample_frac(size = 0.1)
## # A tibble: 14 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 test small 61 dead 70 2 71 no
## 2 test adeno 51 dead 60 5 62 no
## 3 placebo squamous 126 dead 60 9 63 yes
## 4 placebo large 177 dead 50 16 66 yes
## 5 test squamous 999 dead 90 12 54 yes
## 6 test small 24 dead 60 8 49 no
## 7 placebo squamous 82 dead 40 10 69 yes
## 8 placebo small 63 dead 50 11 48 no
## 9 placebo adeno 12 dead 50 4 63 yes
## 10 test squamous 87 survival 80 3 48 no
## 11 placebo small 4 dead 40 2 35 no
## 12 placebo small 117 dead 80 3 46 no
## 13 placebo squamous 411 dead 70 5 64 yes
## 14 test large 53 dead 60 12 66 no8.6.9 選出明顯不同個體函式 distinct() 與 n_distinct()
資料常常重覆輸入的個體, 或是個體有重覆測量,
distinct() 函式可以查詢多少明顯不同的個體,
使用引數 .keep_all = TRUE 可以保留所有變數.
函式 n_distinct() 計算明顯不同的個體數目,
使用引數 na.rm = FALSE 決定是否納入缺失值.
## distinct()
set.seed(1)
df <- tibble(
x = sample(5, 100, rep = TRUE),
y = sample(5, 100, rep = TRUE)
)
print(df, n = 20)
## # A tibble: 100 x 2
## x y
## <int> <int>
## 1 1 1
## 2 4 3
## 3 1 3
## 4 2 3
## 5 5 3
## 6 3 4
## 7 2 1
## 8 3 1
## 9 3 4
## 10 1 2
## 11 5 1
## 12 5 2
## 13 2 3
## 14 2 4
## 15 1 1
## 16 5 3
## 17 5 5
## 18 1 3
## 19 1 4
## 20 5 2
## # ... with 80 more rows
nrow(df)
## [1] 100
nrow(distinct(df))
## [1] 24
nrow(distinct(df, x, y))
## [1] 24
distinct(df, x)
## # A tibble: 5 x 1
## x
## <int>
## 1 1
## 2 4
## 3 2
## 4 5
## 5 3
distinct(df, y)
## # A tibble: 5 x 1
## y
## <int>
## 1 1
## 2 3
## 3 4
## 4 2
## 5 5
distinct(df, x, .keep_all = TRUE)
## # A tibble: 5 x 2
## x y
## <int> <int>
## 1 1 1
## 2 4 3
## 3 2 3
## 4 5 3
## 5 3 4
##
set.seed(1)
df <- tibble(
x = sample(10, 100, rep = TRUE),
y = sample(10, 100, rep = TRUE)
)
df
## # A tibble: 100 x 2
## x y
## <int> <int>
## 1 9 3
## 2 4 10
## 3 7 3
## 4 1 1
## 5 2 6
## 6 7 6
## 7 2 4
## 8 3 9
## 9 1 5
## 10 5 1
## # ... with 90 more rows
nrow(df)
## [1] 100
nrow(distinct(df))
## [1] 65
nrow(distinct(df, x, y))
## [1] 65
distinct(df, x)
## # A tibble: 10 x 1
## x
## <int>
## 1 9
## 2 4
## 3 7
## 4 1
## 5 2
## 6 3
## 7 5
## 8 10
## 9 6
## 10 8
distinct(df, y)
## # A tibble: 10 x 1
## y
## <int>
## 1 3
## 2 10
## 3 1
## 4 6
## 5 4
## 6 9
## 7 5
## 8 7
## 9 2
## 10 8
distinct(df, x, .keep_all = TRUE)
## # A tibble: 10 x 2
## x y
## <int> <int>
## 1 9 3
## 2 4 10
## 3 7 3
## 4 1 1
## 5 2 6
## 6 3 9
## 7 5 1
## 8 10 6
## 9 6 3
## 10 8 2
#
set.seed(1)
x <- sample(1:10, 1e5, rep = TRUE)
length(x)
## [1] 100000
length(unique(x))
## [1] 10
n_distinct(x)
## [1] 108.6.10 利用橫列指標選出個體函式 slice()
slice() 為一系列函式可以利用橫列指標 (row index)
選出個體 (row). 包含
slice()
slice_head()選出資料最前端的個體slice_last()選出資料最末端的個體slice_min()選出資料變數值最小的個體slice_max()選出資料變數值最大的個體slice_sample()隨機選出個體
## slice()
set.seed(1)
dd %>% slice(1)
## # A tibble: 1 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo squamous 72 dead 60 7 69 no
dd %>% slice(1:3)
## # A tibble: 3 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo squamous 72 dead 60 7 69 no
## 2 placebo squamous 411 dead 70 5 64 yes
## 3 placebo squamous 228 dead 60 3 38 no
dd %>% slice(101:n())
## # A tibble: 37 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 test small 99 dead 85 4 62 no
## 2 test small 61 dead 70 2 71 no
## 3 test small 25 dead 70 2 70 no
## 4 test small 95 dead 70 1 61 no
## 5 test small 80 dead 50 17 71 no
## 6 test small 51 dead 30 87 59 yes
## 7 test small 29 dead 40 8 67 no
## 8 test adeno 24 dead 40 2 60 no
## 9 test adeno 18 dead 40 5 69 yes
## 10 test adeno 83 survival 99 3 57 no
## # ... with 27 more rows
dd %>% slice(-c(1:100))
## # A tibble: 37 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 test small 99 dead 85 4 62 no
## 2 test small 61 dead 70 2 71 no
## 3 test small 25 dead 70 2 70 no
## 4 test small 95 dead 70 1 61 no
## 5 test small 80 dead 50 17 71 no
## 6 test small 51 dead 30 87 59 yes
## 7 test small 29 dead 40 8 67 no
## 8 test adeno 24 dead 40 2 60 no
## 9 test adeno 18 dead 40 5 69 yes
## 10 test adeno 83 survival 99 3 57 no
## # ... with 27 more rows
dd %>% slice_head(n = 3)
## # A tibble: 3 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo squamous 72 dead 60 7 69 no
## 2 placebo squamous 411 dead 70 5 64 yes
## 3 placebo squamous 228 dead 60 3 38 no
dd %>% slice_tail(n = 3)
## # A tibble: 3 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 test large 231 dead 70 18 67 yes
## 2 test large 378 dead 80 4 65 no
## 3 test large 49 dead 30 3 37 no
dd %>% slice_min(time, n = 3)
## # A tibble: 3 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 test squamous 1 dead 20 21 65 yes
## 2 test squamous 1 dead 50 7 35 no
## 3 test small 2 dead 40 36 44 yes
dd %>% slice_max(time, n = 3)
## # A tibble: 3 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 test squamous 999 dead 90 12 54 yes
## 2 test squamous 991 dead 70 7 50 yes
## 3 test squamous 587 dead 60 3 58 no
set.seed(1)
dd %>% slice_sample(n = 3)
## # A tibble: 3 x 8
## treat cellcode time censor diagtime kps age prior
## <fct> <fct> <int> <fct> <int> <int> <int> <fct>
## 1 placebo large 250 dead 70 8 53 yes
## 2 test large 53 dead 60 12 66 no
## 3 placebo small 63 dead 50 11 48 no8.6.11 計算常見統計量函式 summarise()
資料分析常常需要變數基本統計量,
例如, 計算個數, 平均值, 變異數等等.
summarise() 函式可計算常見統計量,
期常用引數有
- Center:
mean(),median() - Spread:
var(),sd(),IQR(),mad(),range() - Range:
min(),max(),quantile() - Position:
first(),last(),nth() - Count:
n(),n_distinct() - Logical:
any(),all()
以資料在檔案第 5 章的 survVATrial.csv 為例,
對資料計算個數 n(),
對 age 計算平均值與標準差.
## summarise
dd %>%
summarise(
count = n(),
age_mean = mean(age, na.rm = TRUE),
age_sd = sd(age, na.rm = TRUE)
)
## # A tibble: 1 x 3
## count age_mean age_sd
## <int> <dbl> <dbl>
## 1 137 58.3 10.58.6.12 資料分組操作函式 group_by()
資料分析常常需要類別變數分組, 個別操作資料或進行計算統計量.
函式 group_by() 引數可放入類別變數, 然後分組進行相同資料分析.
以資料在檔案第 5 章的 survVATrial.csv 為例,
對試驗藥組與安慰劑組分別對 diagtime 計算計算平均值與標準差..
## group_by()
dd %>%
group_by(treat) %>%
summarise(
diagtime_mean = mean(diagtime, na.rm = TRUE),
diagtime_sd = sd(diagtime, na.rm = TRUE)
)
## # A tibble: 2 x 3
## treat diagtime_mean diagtime_sd
## <fct> <dbl> <dbl>
## 1 placebo 59.2 18.7
## 2 test 57.9 21.4
dd %>%
group_by(treat, cellcode) %>%
summarise(
diagtime_mean = mean(diagtime, na.rm = TRUE),
diagtime_sd = sd(diagtime, na.rm = TRUE)
)
## # A tibble: 8 x 4
## # Groups: treat [2]
## treat cellcode diagtime_mean diagtime_sd
## <fct> <fct> <dbl> <dbl>
## 1 placebo squamous 57.3 17.9
## 2 placebo small 54.8 17.7
## 3 placebo adeno 58.9 24.2
## 4 placebo large 70 15.1
## 5 test squamous 63.5 22.3
## 6 test small 51.4 21.5
## 7 test adeno 57.7 21.7
## 8 test large 58.8 18.88.6.13 多變數計算統計量函式 summarise_all()
summarise() 函式只能分別對當一變數進行計算,
若要同時對許多變數進行相同操作,
可使用以下函式.
summarise_all()對每一個變數進行相同操作summarise_each()對每一個變數進行相同操作, 需加變數名summarise_at()對選出的變數進行相同操作 需加變數名summarise_if()對符合特定條件的變數進行相同操作
## summarise_all()
con.mean <- dd %>%
select(time, diagtime, kps, age) %>%
summarise_all(mean, na.rm = TRUE)
con.mean
## # A tibble: 1 x 4
## time diagtime kps age
## <dbl> <dbl> <dbl> <dbl>
## 1 122. 58.6 8.77 58.3
#
con.sd <- dd %>%
select(time, diagtime, kps, age) %>%
summarise_all(sd, na.rm = TRUE)
con.sd
## # A tibble: 1 x 4
## time diagtime kps age
## <dbl> <dbl> <dbl> <dbl>
## 1 158. 20.0 10.6 10.5
id <- c("mean", "sd")
comb <- rbind(con.mean, con.sd)
comb <- cbind(id, comb)
comb
## id time diagtime kps age
## 1 mean 121.6 58.57 8.774 58.31
## 2 sd 157.8 20.04 10.612 10.54
#
dd %>% select(time, diagtime, kps, age) %>%
summarise_all(list(mean, sd), na.rm = TRUE)
## # A tibble: 1 x 8
## time_fn1 diagtime_fn1 kps_fn1 age_fn1 time_fn2 diagtime_fn2 kps_fn2 age_fn2
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 122. 58.6 8.77 58.3 158. 20.0 10.6 10.5
dd %>% select(time, diagtime, kps, age) %>%
summarise_all(lst(mean, sd), na.rm = TRUE)
## # A tibble: 1 x 8
## time_mean diagtime_mean kps_mean age_mean time_sd diagtime_sd kps_sd age_sd
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 122. 58.6 8.77 58.3 158. 20.0 10.6 10.5
dd %>%
summarise_each(list(mean, sd), time, age) # not so useful
## Warning: `summarise_each_()` is deprecated as of dplyr 0.7.0.
## Please use `across()` instead.
## This warning is displayed once every 8 hours.
## Call `lifecycle::last_warnings()` to see where this warning was generated.
## # A tibble: 1 x 4
## time_fn1 age_fn1 time_fn2 age_fn2
## <dbl> <dbl> <dbl> <dbl>
## 1 122. 58.3 158. 10.5
dd %>%
summarise_each(lst(mean, sd), time, age) # not so useful
## # A tibble: 1 x 4
## time_mean age_mean time_sd age_sd
## <dbl> <dbl> <dbl> <dbl>
## 1 122. 58.3 158. 10.5
dd %>%
summarise_at(c("time", "age"), mean, na.rm = TRUE)
## # A tibble: 1 x 2
## time age
## <dbl> <dbl>
## 1 122. 58.3
dd %>%
summarise_at(.vars = vars(time, age), mean, na.rm = TRUE)
## # A tibble: 1 x 2
## time age
## <dbl> <dbl>
## 1 122. 58.3
dd %>%
summarise_at(.vars = vars(time, age),
.funs = c(Mean = "mean", SD = "sd"), na.rm = TRUE)
## # A tibble: 1 x 4
## time_Mean age_Mean time_SD age_SD
## <dbl> <dbl> <dbl> <dbl>
## 1 122. 58.3 158. 10.5
dd %>%
summarise_if(is.numeric, list(mean, sd), na.rm = TRUE)
## # A tibble: 1 x 8
## time_fn1 diagtime_fn1 kps_fn1 age_fn1 time_fn2 diagtime_fn2 kps_fn2 age_fn2
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 122. 58.6 8.77 58.3 158. 20.0 10.6 10.5
dd %>%
summarise_if(is.numeric, lst(mean, sd), na.rm = TRUE)
## # A tibble: 1 x 8
## time_mean diagtime_mean kps_mean age_mean time_sd diagtime_sd kps_sd age_sd
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 122. 58.6 8.77 58.3 158. 20.0 10.6 10.58.7 資料聯集與交集函式
若二組資料有相同的變數, 但可能僅有部分相同的個體, 可以利用二組資料聯集與交集操作選出個體.
intersect(x, y)選出x與y都存在的不同個體.union(x, y)選出x與y存在的不同個體.setdiff(x, y)選出存在x但不存在y的不同個體.
以資料在檔案第 5 章的 survVATrial.csv 為例, 隨機從前 10 位個體重覆抽取 7 位, 進行 2 次, 得到 2 組資料.
## set operation
df <- dd %>%
select(treat, cellcode, time, censor, age) %>%
mutate(id = 1:n()) %>%
filter(id <= 10)
df
## # A tibble: 10 x 6
## treat cellcode time censor age id
## <fct> <fct> <int> <fct> <int> <int>
## 1 placebo squamous 72 dead 69 1
## 2 placebo squamous 411 dead 64 2
## 3 placebo squamous 228 dead 38 3
## 4 placebo squamous 126 dead 63 4
## 5 placebo squamous 118 dead 65 5
## 6 placebo squamous 10 dead 49 6
## 7 placebo squamous 82 dead 69 7
## 8 placebo squamous 110 dead 68 8
## 9 placebo squamous 314 dead 43 9
## 10 placebo squamous 100 survival 70 10
set.seed(1)
x <- df %>% sample_n(size = 7, replace = FALSE)
y <- df %>% sample_n(size = 7, replace = FALSE)
x
## # A tibble: 7 x 6
## treat cellcode time censor age id
## <fct> <fct> <int> <fct> <int> <int>
## 1 placebo squamous 314 dead 43 9
## 2 placebo squamous 126 dead 63 4
## 3 placebo squamous 82 dead 69 7
## 4 placebo squamous 72 dead 69 1
## 5 placebo squamous 411 dead 64 2
## 6 placebo squamous 118 dead 65 5
## 7 placebo squamous 228 dead 38 3
y
## # A tibble: 7 x 6
## treat cellcode time censor age id
## <fct> <fct> <int> <fct> <int> <int>
## 1 placebo squamous 411 dead 64 2
## 2 placebo squamous 228 dead 38 3
## 3 placebo squamous 72 dead 69 1
## 4 placebo squamous 118 dead 65 5
## 5 placebo squamous 82 dead 69 7
## 6 placebo squamous 100 survival 70 10
## 7 placebo squamous 10 dead 49 6
intersect(x, y)
## # A tibble: 1 x 2
## treat treat
## <fct> <fct>
## 1 placebo placebo
union(x, y)
## [[1]]
## [1] placebo placebo placebo placebo placebo placebo placebo
## Levels: placebo test
##
## [[2]]
## [1] squamous squamous squamous squamous squamous squamous squamous
## Levels: squamous small adeno large
##
## [[3]]
## [1] 314 126 82 72 411 118 228
##
## [[4]]
## [1] dead dead dead dead dead dead dead
## Levels: survival dead
##
## [[5]]
## [1] 43 63 69 69 64 65 38
##
## [[6]]
## [1] 9 4 7 1 2 5 3
##
## [[7]]
## [1] 411 228 72 118 82 100 10
##
## [[8]]
## [1] dead dead dead dead dead survival dead
## Levels: survival dead
##
## [[9]]
## [1] 64 38 69 65 69 70 49
##
## [[10]]
## [1] 2 3 1 5 7 10 6
setdiff(x, y)
## # A tibble: 7 x 4
## time censor age id
## <int> <fct> <int> <int>
## 1 314 dead 43 9
## 2 126 dead 63 4
## 3 82 dead 69 7
## 4 72 dead 69 1
## 5 411 dead 64 2
## 6 118 dead 65 5
## 7 228 dead 38 38.8 資料合併函式
資料經常儲存再不同檔案,
例如門診檔, 住院檔, 實驗室檔,
同一位個體常須使用個體辨識碼 (id) 或姓名 (names)
進行合併或清理.
用來連結不同資料的個體辨識碼或變數稱為
關鍵碼 或 所引鍵
(key),
多數為個體辨識碼, 當有時為時間, 日期, 文件編號等,
可能同時須使用 2 個變數才能成為單一辨識碼,
例如, 同時須使用醫院與院內病歷號,
避免不同個體在不同醫院卻有有相同的病歷號.
tidyverse 系統的有許多 _join_(x, y) 函式可進行各種資料合併.
若 2 組資料包含一些相同變數, 盪不同觀測值, 此時必須小心處理.
inner_join(x, y)包函x與y都配對存在的x與y個體與變數left_join(x, y)包函所有x個體與變數且在y有配對存在的y個體與變數right_join(x, y)包函所有y個體與變數且在x有配對存在的x個體與變數full_join(x, y)包函所有x與y的個體與變數資料semi_join(x, y)包函x在y有配對存在的x個體與變數anti_join(x, y)包函x在y無配對存在的x個體與變數
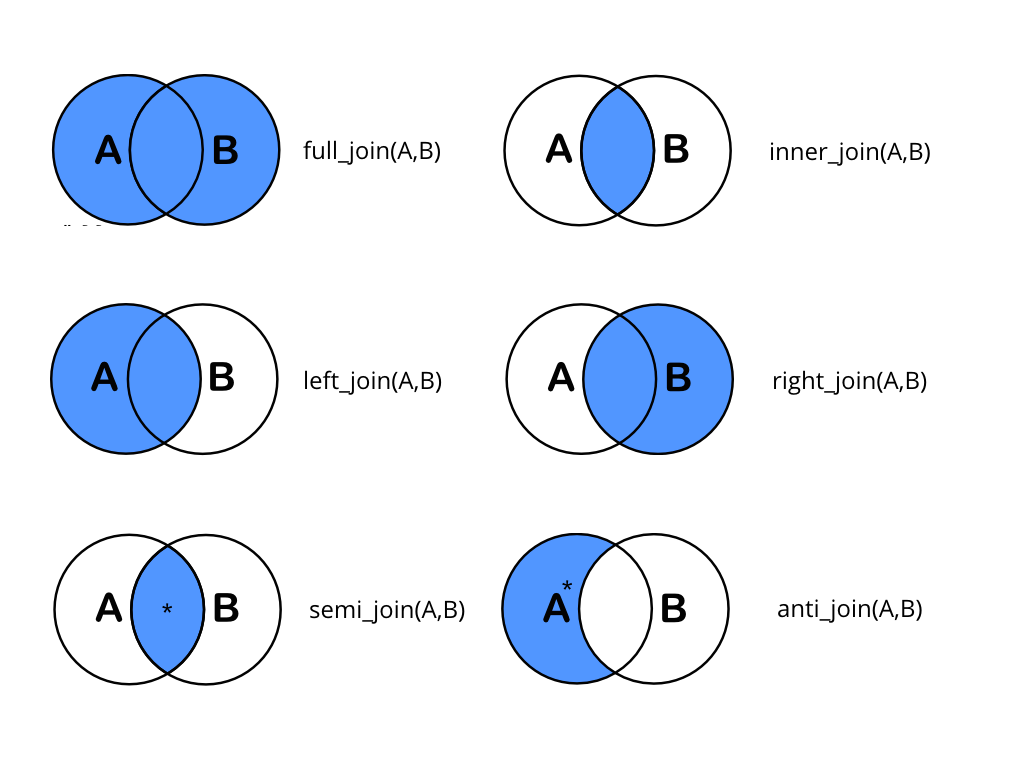
dplyr::_join
以資料在檔案第 5 章的 survVATrial.csv 為例,
隨機從前 10 位個體抽取 7 位, 進行 2 次, 得到 2 組資料 x 與 y.
x 資料包函 id, treat, time,age.y資料包函id,cellcode,ceosor,age`.
## _join()
set.seed(1)
df <- dd %>%
select(treat, cellcode, time, censor, age) %>%
mutate(id = 1:n()) %>%
filter(id <= 10)
x <- df %>%
select(id, treat, time, age) %>%
sample_n(size = 7, replace = FALSE) %>%
arrange(id)
y <- df %>%
select(id, cellcode, censor, age) %>%
sample_n(size = 7, replace = FALSE) %>%
arrange(id)
x
## # A tibble: 7 x 4
## id treat time age
## <int> <fct> <int> <int>
## 1 1 placebo 72 69
## 2 2 placebo 411 64
## 3 3 placebo 228 38
## 4 4 placebo 126 63
## 5 5 placebo 118 65
## 6 7 placebo 82 69
## 7 9 placebo 314 43
y
## # A tibble: 7 x 4
## id cellcode censor age
## <int> <fct> <fct> <int>
## 1 1 squamous dead 69
## 2 2 squamous dead 64
## 3 3 squamous dead 38
## 4 5 squamous dead 65
## 5 6 squamous dead 49
## 6 7 squamous dead 69
## 7 10 squamous survival 70
inner_join(x, y)
## # A tibble: 5 x 6
## id treat time age cellcode censor
## <int> <fct> <int> <int> <fct> <fct>
## 1 1 placebo 72 69 squamous dead
## 2 2 placebo 411 64 squamous dead
## 3 3 placebo 228 38 squamous dead
## 4 5 placebo 118 65 squamous dead
## 5 7 placebo 82 69 squamous dead
left_join(x, y)
## # A tibble: 7 x 6
## id treat time age cellcode censor
## <int> <fct> <int> <int> <fct> <fct>
## 1 1 placebo 72 69 squamous dead
## 2 2 placebo 411 64 squamous dead
## 3 3 placebo 228 38 squamous dead
## 4 4 placebo 126 63 <NA> <NA>
## 5 5 placebo 118 65 squamous dead
## 6 7 placebo 82 69 squamous dead
## 7 9 placebo 314 43 <NA> <NA>
right_join(x, y)
## # A tibble: 7 x 6
## id treat time age cellcode censor
## <int> <fct> <int> <int> <fct> <fct>
## 1 1 placebo 72 69 squamous dead
## 2 2 placebo 411 64 squamous dead
## 3 3 placebo 228 38 squamous dead
## 4 5 placebo 118 65 squamous dead
## 5 7 placebo 82 69 squamous dead
## 6 6 <NA> NA 49 squamous dead
## 7 10 <NA> NA 70 squamous survival
full_join(x, y)
## # A tibble: 9 x 6
## id treat time age cellcode censor
## <int> <fct> <int> <int> <fct> <fct>
## 1 1 placebo 72 69 squamous dead
## 2 2 placebo 411 64 squamous dead
## 3 3 placebo 228 38 squamous dead
## 4 4 placebo 126 63 <NA> <NA>
## 5 5 placebo 118 65 squamous dead
## 6 7 placebo 82 69 squamous dead
## 7 9 placebo 314 43 <NA> <NA>
## 8 6 <NA> NA 49 squamous dead
## 9 10 <NA> NA 70 squamous survival
semi_join(x, y)
## # A tibble: 5 x 4
## id treat time age
## <int> <fct> <int> <int>
## 1 1 placebo 72 69
## 2 2 placebo 411 64
## 3 3 placebo 228 38
## 4 5 placebo 118 65
## 5 7 placebo 82 69
anti_join(x, y)
## # A tibble: 2 x 4
## id treat time age
## <int> <fct> <int> <int>
## 1 4 placebo 126 63
## 2 9 placebo 314 43