Chapter 7 第七讲:描述性统计与数据可视化基础
本节课将引入一个新的知识点:描述性统计和数据可视化。根据课程大纲,数据可视化将分为两课,今天我们将初步了解并实践数据可视化,而后续课程会进一步讲如何对可视化结果进行精细调整,以达到可出版的标准。 本次课程增加了一些新的工具包。如果是第一次运行这些代码,可能需要执行一些安装命令,以确保所有相关的工具包都能正确安装。安装命令代码:
if (!requireNamespace('pacman', quietly = TRUE)) { install.packages('pacman')}
pacman::p_load(here,skimr,quartets,GGally,showtext,bruceR,tidyverse,DataExplorer)7.1 回顾
上节课我们介绍了数据预处理、如何批量读取数据、如何获取一个文件夹中特定类型文件的名字、如何定义函数进行转换数据类型以确保每一列数据的类型都是我们期望的、如何通过for循环来批量读取数据、介绍了for循环和if语句在R语言数据处理中的重要性并演示了如何使用for循环读取数据、合并数据,并将数据保存为CSV格式,这样可以避免每次都重新进行批量读取,节省时间。
7.1.1 批量导入数据
获取地址
# 所有数据路径
files <- list.files(
## <- & =
here::here("Book", "data", "match"),
pattern = "data_exp7_rep_match_.*.out$",
full.names = TRUE)数据类型转换
convert_data_types <- function(df) {
df <- df %>%
dplyr::mutate(Date = as.character(Date),Prac = as.character(Prac),
Sub = as.numeric(Sub),Age = as.numeric(Age),
Sex = as.character(Sex),Hand = as.character(Hand),
Block = as.numeric(Block),Bin = as.numeric(Bin),
Trial = as.numeric(Trial),Shape = as.character(Shape),
Label = as.character(Label),Match = as.character(Match),
CorrResp = as.character(CorrResp),Resp = as.character(Resp),
ACC = as.numeric(ACC),RT = as.numeric(RT))
return(df)
}for循环批量读取数据,将数据进行批量合并批量合并
df3 <- data.frame()
for (i in seq_along(files)) {
# 读取
df <- read.table(files[i], header = TRUE) %>%
dplyr::filter(Date != "Date") %>%
convert_data_types()
# 合并
df3 <- dplyr::bind_rows(df3, df)
}
# 删除临时变量
rm(df, files, i)通过export将数据保存为csv 保存数据
## NOT RUN
## 上节课介绍了write.csv,也可使用bruceR::export
bruceR::export(
df3,
file = here::here("Book","data", "match","match_raw.csv"))
## 当然,export 不仅可以保存数据,也可以输出模型结果当出现列名和变量名不同,可以通过rename将列名进行修改 修改列名-rename
## [1] "date" "Prac" "Sub" "Age" "Sex" "Hand" "Block" "Bin" "Trial"
## [10] "Shape" "Label" "Match" "CorrResp" "Resp" "ACC" "RT"## 将全部列名都变成小写
df3 %>% dplyr::rename_with(
## 将字符向量全部变成小写; ~ 声明这是一个函数,.代表前面的数据(df3)传到.所在的位置
~tolower(.) #<<
## 即使用 tolower()对所有列名进行批量处理
##
) %>% colnames()## [1] "date" "prac" "sub" "age" "sex" "hand" "block" "bin" "trial"
## [10] "shape" "label" "match" "corrresp" "resp" "acc" "rt"7.1.2 代码书写规范
为了使代码看起来更整洁,我们建议每个预处理步骤占用一行,并在管道符后换行,这样每一步的操作都清晰可见。
## 以下代码看起来如何?
iris %>% group_by(Species) %>%
summarize_all(mean) %>% ungroup %>% gather(measure, value, -Species)%>%
arrange(value) ## # A tibble: 12 × 3
## Species measure value
## <fct> <chr> <dbl>
## 1 setosa Petal.Width 0.246
## 2 versicolor Petal.Width 1.33
## 3 setosa Petal.Length 1.46
## 4 virginica Petal.Width 2.03
## 5 versicolor Sepal.Width 2.77
## 6 virginica Sepal.Width 2.97
## 7 setosa Sepal.Width 3.43
## 8 versicolor Petal.Length 4.26
## 9 setosa Sepal.Length 5.01
## 10 virginica Petal.Length 5.55
## 11 versicolor Sepal.Length 5.94
## 12 virginica Sepal.Length 6.59### 管道操作的代码看上去更加清晰,整洁
iris %>%
dplyr::group_by(Species) %>%
dplyr::summarize_if(is.numeric, mean) %>%
dplyr::ungroup() %>%
tidyr::gather(measure, value, -Species) %>%
dplyr::arrange(value)## # A tibble: 12 × 3
## Species measure value
## <fct> <chr> <dbl>
## 1 setosa Petal.Width 0.246
## 2 versicolor Petal.Width 1.33
## 3 setosa Petal.Length 1.46
## 4 virginica Petal.Width 2.03
## 5 versicolor Sepal.Width 2.77
## 6 virginica Sepal.Width 2.97
## 7 setosa Sepal.Width 3.43
## 8 versicolor Petal.Length 4.26
## 9 setosa Sepal.Length 5.01
## 10 virginica Petal.Length 5.55
## 11 versicolor Sepal.Length 5.94
## 12 virginica Sepal.Length 6.59如果下载了notebook,能够获取到一个参考链接参考链接:tidyverse style guide,该链接可提供有助于记忆和提高数据分析思路的编码风格,供大家参考。 另外,值得一提的是,R Studio 有时会自动帮你整理代码。比如,我们可以采用 R Studio 中的一个自动格式调整功能,它会呈现给你一个更规整的代码样式,但这个快捷键可能在部分电脑上适用,而在其他电脑上可能有不同的快捷键组合,有时可能会发生冲突。但如果不冲突,你可以直接按 Ctrl + Shift + A 来实现格式化。 我们可以通过点击 R Studio 上面的“Code”来找到并使用这个功能。你可以看到,它自动地把代码调整得非常整齐,基本上是以逗号分隔,每一个逗号后面都分割成一行,前面的缩进也完全对齐。这样,你就可以清晰地看到这一行代码从哪里开始,到哪里结束,以及它完成了几个任务或输入了几个参数。
7.2 探索性数据分析
了解原始数据的特点,做到心中有数,属于一个更广泛的概念:探索性数据分析(Exploratory Data Analysis,EDA),在传统的心理学中,我们通常会清楚地知道要进行什么样的分析,但是在数据科学中,可能面临的是一个未知的数据集,我们不知道其中的规律和数据的结构。因此,探索性数据分析非常重要。 探索性分析通常通过可视化的方式总结数据特征,在大数据时代被广泛推崇。 进行EDA是为了更加了解自己的数据,从而做出基本的判断,但每一次探索背后都对应着特定的问题,进行EDA时,需要了解数据的基本信息,比如有哪些变量?变量的类型?变量的分布?变量之间的关系?
# 读取数据
pg_raw <- bruceR::import(here::here(
"Book","data", "penguin","penguin_rawdata.csv"))
mt_raw <- bruceR::import(here::here(
"Book","data", "match","match_raw.csv")) 7.2.1 常用函数介绍
summary
summary(mt_raw) %>%
knitr::kable() # 注:kable函数只为了输出 skim函数能够实现数据的快速预览,可以根据每一列的数据类型进行数据预览,会显示字符型数据的分布特点| Date | Prac | Sub | Age | Sex | Hand | Block | Bin | Trial | Shape | Label | Match | CorrResp | Resp | ACC | RT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Length:25920 | Length:25920 | Min. : 7302 | Min. :18.0 | Length:25920 | Length:25920 | Min. :1.00 | Min. :1.00 | Min. : 1.00 | Length:25920 | Length:25920 | Length:25920 | Length:25920 | Length:25920 | Min. :-1.000 | Min. :0.106 | |
| Class :character | Class :character | 1st Qu.: 7313 | 1st Qu.:19.0 | Class :character | Class :character | 1st Qu.:1.00 | 1st Qu.:1.00 | 1st Qu.: 6.75 | Class :character | Class :character | Class :character | Class :character | Class :character | 1st Qu.: 1.000 | 1st Qu.:0.610 | |
| Mode :character | Mode :character | Median : 7324 | Median :20.0 | Mode :character | Mode :character | Median :1.00 | Median :2.00 | Median :12.50 | Mode :character | Mode :character | Mode :character | Mode :character | Mode :character | Median : 1.000 | Median :0.702 | |
| NA | NA | Mean : 8853 | Mean :20.8 | NA | NA | Mean :1.61 | Mean :2.42 | Mean :12.50 | NA | NA | NA | NA | NA | Mean : 0.796 | Mean :0.715 | |
| NA | NA | 3rd Qu.: 7336 | 3rd Qu.:22.0 | NA | NA | 3rd Qu.:2.00 | 3rd Qu.:3.00 | 3rd Qu.:18.25 | NA | NA | NA | NA | NA | 3rd Qu.: 1.000 | 3rd Qu.:0.805 | |
| NA | NA | Max. :73370 | Max. :28.0 | NA | NA | Max. :3.00 | Max. :5.00 | Max. :24.00 | NA | NA | NA | NA | NA | Max. : 2.000 | Max. :1.183 |
skimr::skim()–1
## [1] "── Data Summary ────────────────────────" " Values"
## [3] "Name mt_raw" "Number of rows 25920 "
## [5] "Number of columns 16 " "_______________________ "
## [7] "Column type frequency: " " character 9 "
## [9] " numeric 7 " "________________________ "
## [11] "Group variables None " ""skimr::skim()–2
## [1] "── Variable type: character ───────────────────────────────────────────────────────────────────────────────────"
## [2] " skim_variable n_missing complete_rate min max empty n_unique whitespace"
## [3] "1 Date 0 1 20 20 0 24362 0"
## [4] "2 Prac 0 1 3 3 0 1 0"
## [5] "3 Sex 0 1 1 6 0 4 0"
## [6] "4 Hand 0 1 1 1 0 2 0"
## [7] "5 Shape 0 1 9 12 0 4 0"
## [8] "6 Label 0 1 9 12 0 4 0"
## [9] "7 Match 0 1 5 8 0 2 0"
## [10] "8 CorrResp 0 1 1 1 0 2 0"
## [11] "9 Resp 658 0.975 1 5 0 9 0"
## [12] ""skimr::skim()–3
## [1] "── Variable type: numeric ─────────────────────────────────────────────────────────────────────────────────────"
## [2] " skim_variable n_missing complete_rate mean sd p0 p25 p50 p75 p100 hist "
## [3] "1 Sub 0 1 8853. 9932. 7302 7313 7324 7336 73370 ▇▁▁▁▁"
## [4] "2 Age 0 1 20.8 2.48 18 19 20 22 28 ▇▂▁▁▁"
## [5] "3 Block 0 1 1.61 0.803 1 1 1 2 3 ▇▁▃▁▃"
## [6] "4 Bin 0 1 2.42 1.36 1 1 2 3 5 ▇▇▃▃▃"
## [7] "5 Trial 0 1 12.5 6.92 1 6.75 12.5 18.2 24 ▇▇▆▇▇"
## [8] "6 ACC 0 1 0.796 0.464 -1 1 1 1 2 ▁▂▁▇▁"
## [9] "7 RT 0 1 0.715 0.151 0.106 0.610 0.702 0.805 1.18 ▁▂▇▅▁"
## [10] NA
## [11] NA
## [12] NA
## [13] NA
## [14] NA
## [15] NA
## [16] NA
## [17] NAbruceR::Describe()
## [1] "────────────────────────────────────────────────────────────────────────────────────"
## [2] " N (NA) Mean SD | Median Min Max Skewness Kurtosis"
## [3] "────────────────────────────────────────────────────────────────────────────────────"
## [4] "Date* 25920 12218.64 6999.30 | 12220.50 1.00 24362.00 0.00 -1.19"
## [5] "Prac* 25920 1.00 0.00 | 1.00 1.00 1.00 NaN NaN"
## [6] "Sub 25920 8852.59 9931.83 | 7324.00 7302.00 73370.00 6.34 38.22"
## [7] "Age 25920 20.83 2.48 | 20.00 18.00 28.00 1.09 0.35"
## [8] "Sex* 25920 3.27 0.65 | 3.00 1.00 4.00 -0.84 1.58"
## [9] "Hand* 25920 1.98 0.15 | 2.00 1.00 2.00 -6.34 38.22"
## [10] "Block 25920 1.61 0.80 | 1.00 1.00 3.00 0.82 -0.97"
## [11] "Bin 25920 2.42 1.36 | 2.00 1.00 5.00 0.67 -0.82"
## [12] "Trial 25920 12.50 6.92 | 12.50 1.00 24.00 0.00 -1.20"
## [13] "Shape* 25920 2.50 1.12 | 2.50 1.00 4.00 0.00 -1.36"
## [14] "Label* 25920 2.49 1.11 | 2.00 1.00 4.00 0.02 -1.35"
## [15] "Match* 25920 1.50 0.50 | 1.50 1.00 2.00 0.00 -2.00"
## [16] "CorrResp* 25920 1.50 0.50 | 1.50 1.00 2.00 0.00 -2.00"Describe函数适用于我们已经知道要对哪些数据进行描述性统计的情况,可输出心理学所需的三线表,但如果不清楚需要对哪些数据进行描述性统计,那么describe函数不适用。
7.3 数据可视化
7.3.1 可视化的重要性
可视化有利于我们检查数据是否有意义,同时其也是一个诚实展现我们数据特征的方法。R绘图的方式有许多:Base graphics,grid,lattice,plotly……但在心理学研究中我们一般选择ggplot2。 7.2.2 为什么选择使用ggplot2? ggplot2是一种图形语法,它的核心是用图层来描述和构建图形。可以将数据映射到不同的图层中,然后将这些图层叠加起来形成最终的图形。所谓gg源于“grammar of graphics”,即图形语法。 化繁为简:ggplot2有大量的默认值,适合新手 精准定制:所有元素均可控,有利于文章的发表 易于重叠:ggplot2作出的图包含不同的图层,不同的图层包含不同的信息,叠加起来信息丰富且美观 日益丰富的生态系统:http://r-graph-gallery.com/
7.3.2 可视化的逻辑
在我们的R语言环境中,我们通常处理的是数据框,这是最常见的数据结构。数据框可以简单理解为一张表格,例如有三列数据,每列数据有不同的行,每一行代表一个数据点。 当我们在R中使用ggplot2这个包来画图时,首先需要建立数据与可视化元素之间的映射关系。我们要思考的是如何把数据转换成可视化的元素。比如,我们有一列数字1、2、3,我们会自然地想到将这些数字放在坐标轴上,按照大小顺序排列。
ggplot2在画图时的第一步也是类似的,它将数字沿着坐标轴排列,然后将这些数字映射成几何图形,也就是将数字转换成视觉上的效果,并将其放置在一个特定的坐标系统中。通常我们看到的坐标系统就是笛卡尔坐标系,即我们常说的x轴和y轴。
在将数据映射到坐标轴上之后,它就变成了一个基本的图表。这就是ggplot2所做的最基本的事情:将数字映射到一个空间中,然后放置到坐标轴上,形成一个基本的图表。这里涉及到的一个重要概念是映射关系,比如我们设置x等于f,这里的f对应的是数据框中的一个列,y等于a,这里的a对应的是数据框中的另一个列。如果我们处理的是二维数据,我们就在二维空间中将每个数值进行映射,从而形成图表上的点。每个点在f和a上都有一个值,这两个值在二维坐标体系中确定了一个独特的位置。
除了映射位置之外,我们还可以通过数值来改变可视化元素的其他特性,比如颜色和大小。例如,我们可以用a来表示大小,用f来表示颜色。数字越小,颜色越浅,数字越大,颜色越深;同样,大小也是根据数字的大小来表示。这样,我们不仅可以在xy轴上从左到右、从下到上地映射数据,还可以根据它们的数值来赋予不同的颜色深度和大小,从而增加了图表的信息量和可读性。通过这种方式,我们可以通过映射到不同的空间位置和改变可视化元素的特性来创建丰富多样的图表。
一旦我们在不同的图层上完成了数据映射,我们可以将它们叠加在一起,创建一个复合图表。例如,我们可以在基本的笛卡尔坐标系上,首先叠加一个图层,将一列数据映射为条形图,然后再叠加另一个图层,将另一列数据映射为线图。通过这样的多层叠加,我们可以把想要表达的信息全部呈现在图表上。 在ggplot2中,我们首先进行数据的映射,这通过aes函数完成,它定义了数据如何映射到图表的视觉元素上。
例如,我们可以将时间点一的温度映射到x轴上,将时间点二的温度映射到y轴上。这样,尽管我们一开始看到的是一个空的图表,但实际上R已经读取了温度信息,并给了我们一个默认的坐标轴范围。然而,仅仅映射数据还不够,我们还需要指定数据的可视化形式,即选择合适的几何对象来表示数据。在ggplot2中,这通过添加各种以geom开头的图层来实现,比如geom_point用于绘制点图。我们还可以继续添加其他图层,比如通过geom_smooth添加回归线,来观察两个变量之间的关系。
ggplot2的一个特点是它提供了很多默认值,使得我们可以用很少的代码生成复杂的图表。例如,默认情况下,点图是以黑色圆点表示的,大小和颜色都是根据默认比例来设置的。但我们也完全可以自定义这些元素,比如用不同颜色表示不同性别的体温数据。此外,我们还可以对图表的各个元素进行精细控制,包括坐标轴和图例。我们可以调整坐标轴的标签、名称,以及图例的内容和样式。这些元素的每一个细节都是可以修改的,以适应我们的具体需求。
最后,我们可以在图层上应用美学映射(aes),这不仅仅是关于颜色和形状,还包括如何处理数据,比如将性别转换为因子,并据此生成不同的可视化元素。 总结一下,我们首先进行数据映射,然后选择合适的几何对象来表示数据,最后将不同的图层叠加起来,形成一个完整的图表。通过这种方式,我们可以创建出既丰富又易于理解的视觉表示。
数据映射
# 以penguin问卷中前后体温为例
p1 <- pg_raw %>%
ggplot(aes(x = Temperature_t1, # 确定映射到xy轴的变量
y = Temperature_t2))
p1 ## 坐标轴名称已对应,虽然图片为空 添加图层-散点
添加图层-散点
 添加图层-拟合曲线
添加图层-拟合曲线
## `geom_smooth()` using formula = 'y ~ x'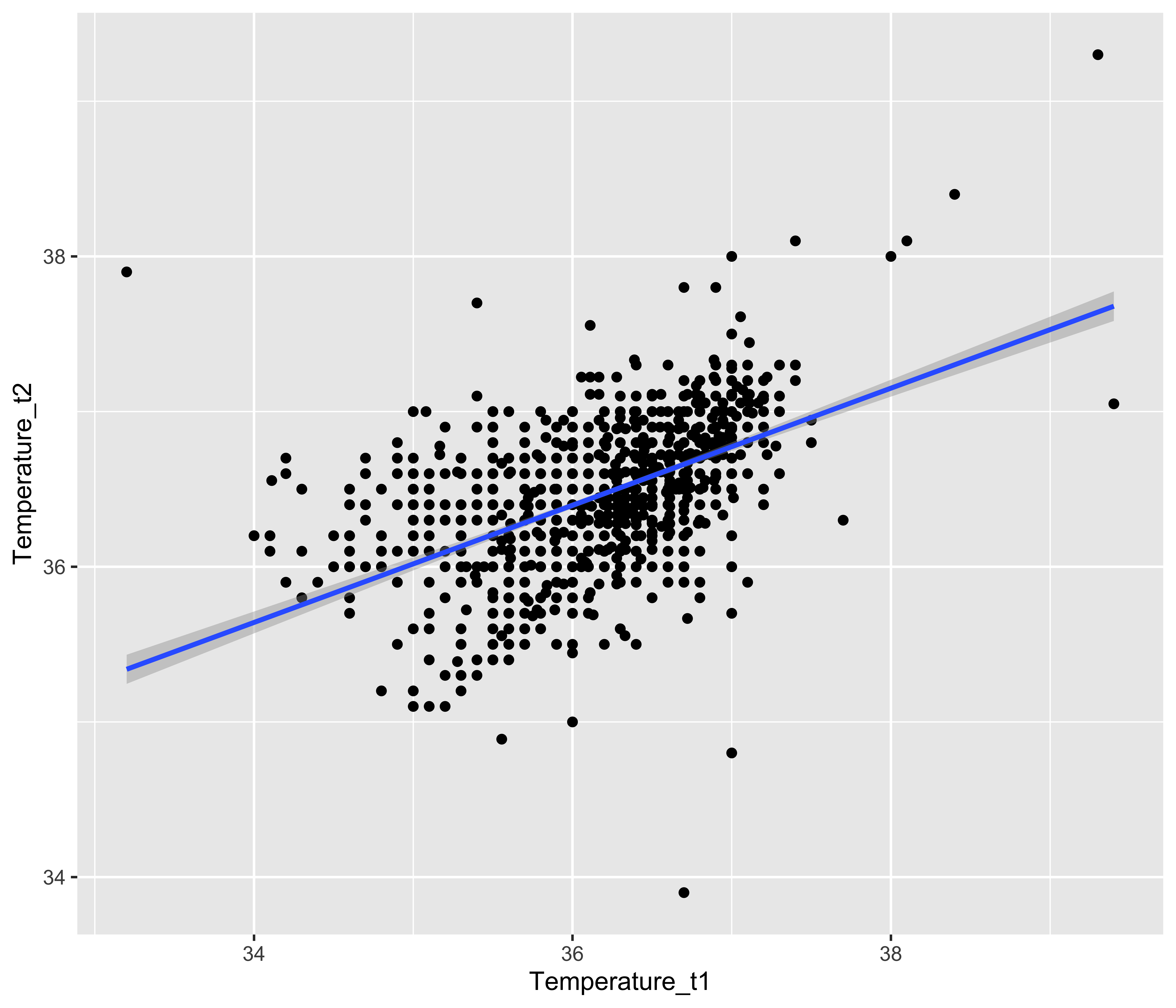 改变映射
改变映射
pg_raw %>%
drop_na(Temperature_t1,Temperature_t2,sex) %>%
ggplot(aes(x = Temperature_t1,
y = Temperature_t2,
color = factor(sex))) +
geom_point() + geom_smooth() ## `geom_smooth()` using method = 'gam' and formula = 'y ~ s(x, bs = "cs")'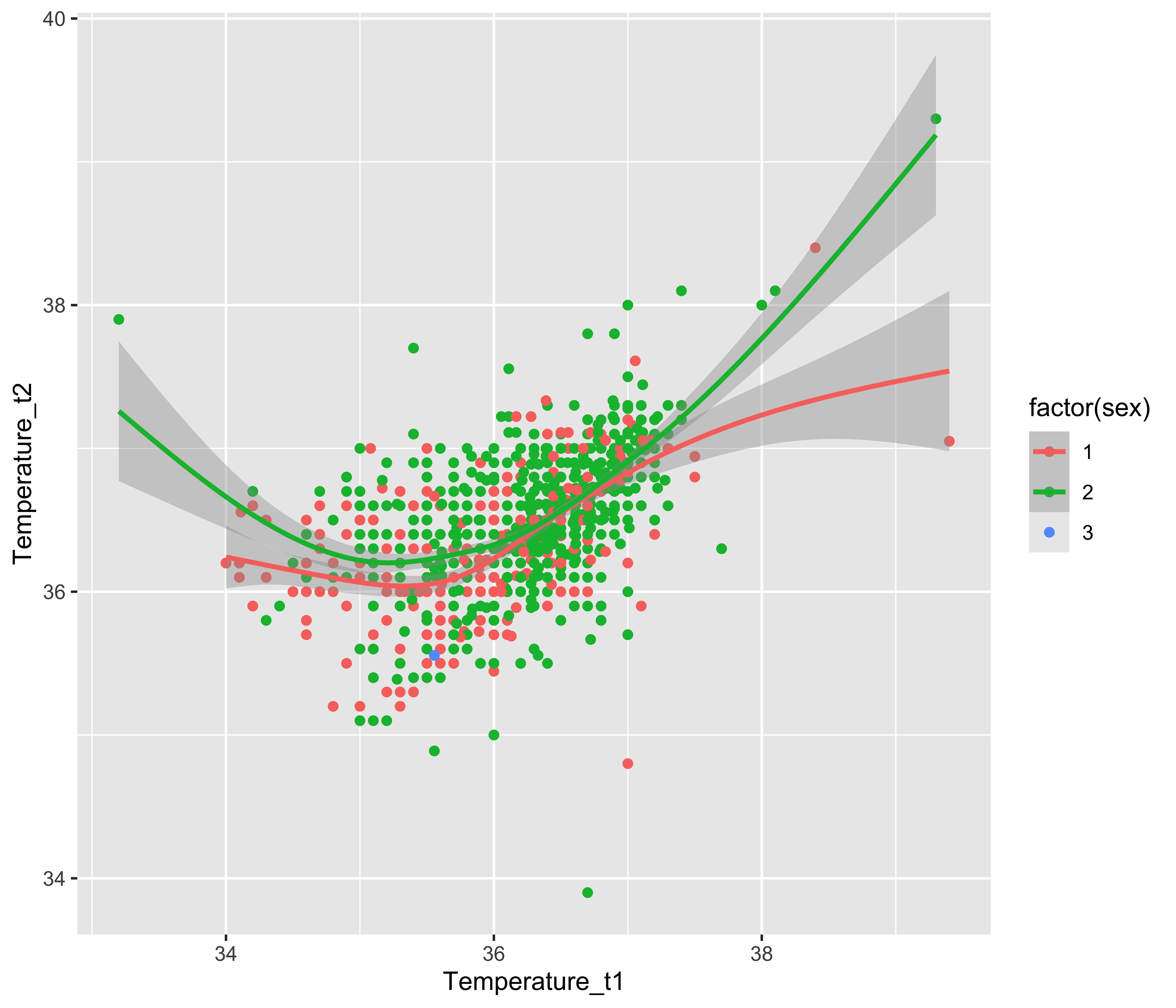
7.3.3 单个图片的组成
图表中实际上有许多元素是可以被操控的。这里举一个例子,比如坐标轴和图例。坐标轴,就像我们这里下面框出来的部分,它包括轴本身和框出的整个区域。在坐标轴中,我们可以进一步细分,比如轴的标签、名称,以及轴的尾部名称,这些都是可以调节的元素。此外,图表中的线条,比如颜色和粗细,也都是可以协调一致的。 另一个我们可以精细操控的元素是图例。图例的整个区域,包括名称、取值范围,以及每个取值范围对应的可视化元素,都是可以自定义的。例如,我们之前使用了性别变量,并将其转换为一个因子,图例中直接显示了因子的名称。这些名称当然是可以修改的,而且每个因子都有自己对应的可视化元素。这意味着图表中的每一个小元素都可以进行精细的修改,以满足我们的具体需求。
7.4 常用图形
7.4.1 直方图
对于连续变量,一般通过直方图对它进行可视化。以认知实验中被试的反应时为例,反应时的单位一般到秒,如何对它进行可视化呢?首先,我们需要进行映射,目的是为了对数据进行可视化。我们关注的是数据的频次分布,因此只需映射一个值,即反应时间,代码如下:
pic_his <- mt_raw %>%
# 确定映射到x轴的变量
ggplot(aes(x = RT)) +
geom_histogram(bins = 40) +
theme_classic()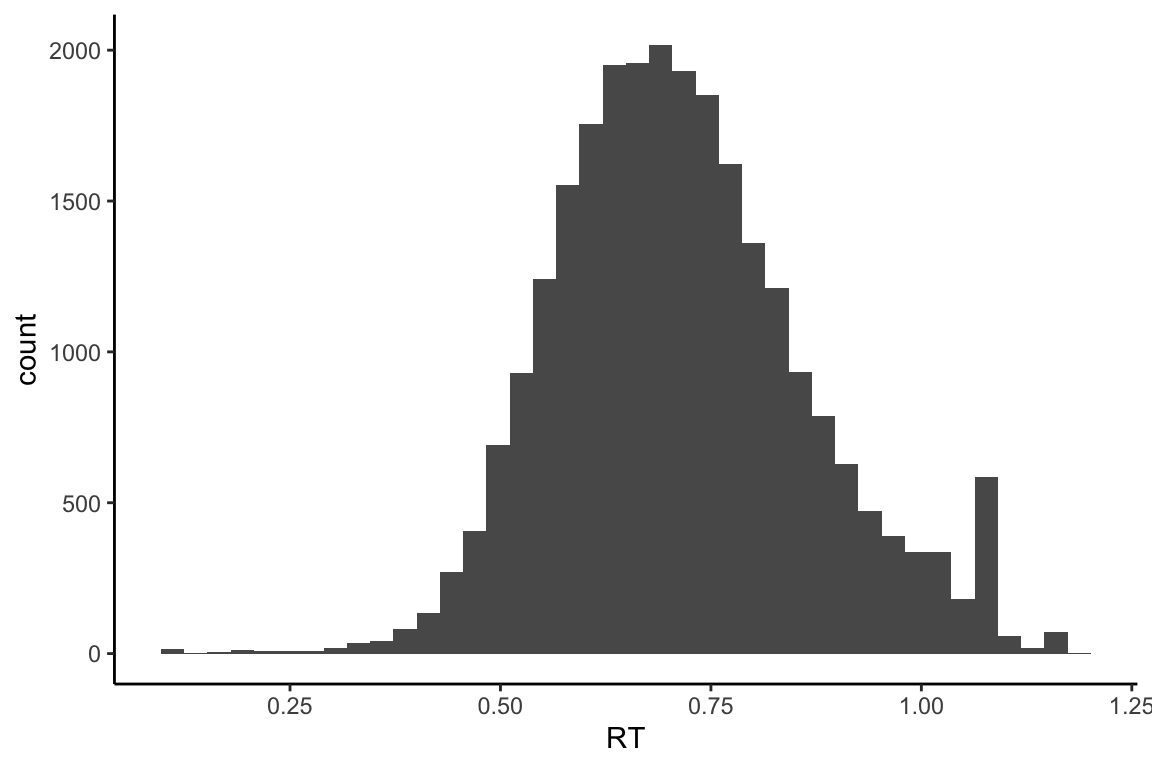 在代码中,我们使用了前面提到的管道操作,将数据传入一个函数,并作为其输入。这个函数就是ggplot。ggplot的第一个参数是数据框,我们默认使用前面的数据框作为输入。aes进行数据映射,x是什么?是我们用数据框中的哪一个变量来做x的映射值。映射完成后,我们对映射的数据进行可视化,使用的是以goem开头的函数,它是直方图。如果我们不输入bins,它也会出图,但我们可以选择一个bins的数量,即直方图中的间隔数量。通过这样的处理,我们可以看到图像变得更加干净。而
theme_classic()会使X轴和Y轴在图中更加清晰,使图的整体风格相比默认模式发生变化。
### 密度图
我们也可以使用密度图来描述反应时的分布情况。在直方图中,我们使用高度来表示数据的多少,而在密度图中,我们使用平滑曲线来表示数据的分布情况。在绘制图形时,我们可以使用“geom”命令来选择几何图形,如条形图或密度图。
在代码中,我们使用了前面提到的管道操作,将数据传入一个函数,并作为其输入。这个函数就是ggplot。ggplot的第一个参数是数据框,我们默认使用前面的数据框作为输入。aes进行数据映射,x是什么?是我们用数据框中的哪一个变量来做x的映射值。映射完成后,我们对映射的数据进行可视化,使用的是以goem开头的函数,它是直方图。如果我们不输入bins,它也会出图,但我们可以选择一个bins的数量,即直方图中的间隔数量。通过这样的处理,我们可以看到图像变得更加干净。而
theme_classic()会使X轴和Y轴在图中更加清晰,使图的整体风格相比默认模式发生变化。
### 密度图
我们也可以使用密度图来描述反应时的分布情况。在直方图中,我们使用高度来表示数据的多少,而在密度图中,我们使用平滑曲线来表示数据的分布情况。在绘制图形时,我们可以使用“geom”命令来选择几何图形,如条形图或密度图。
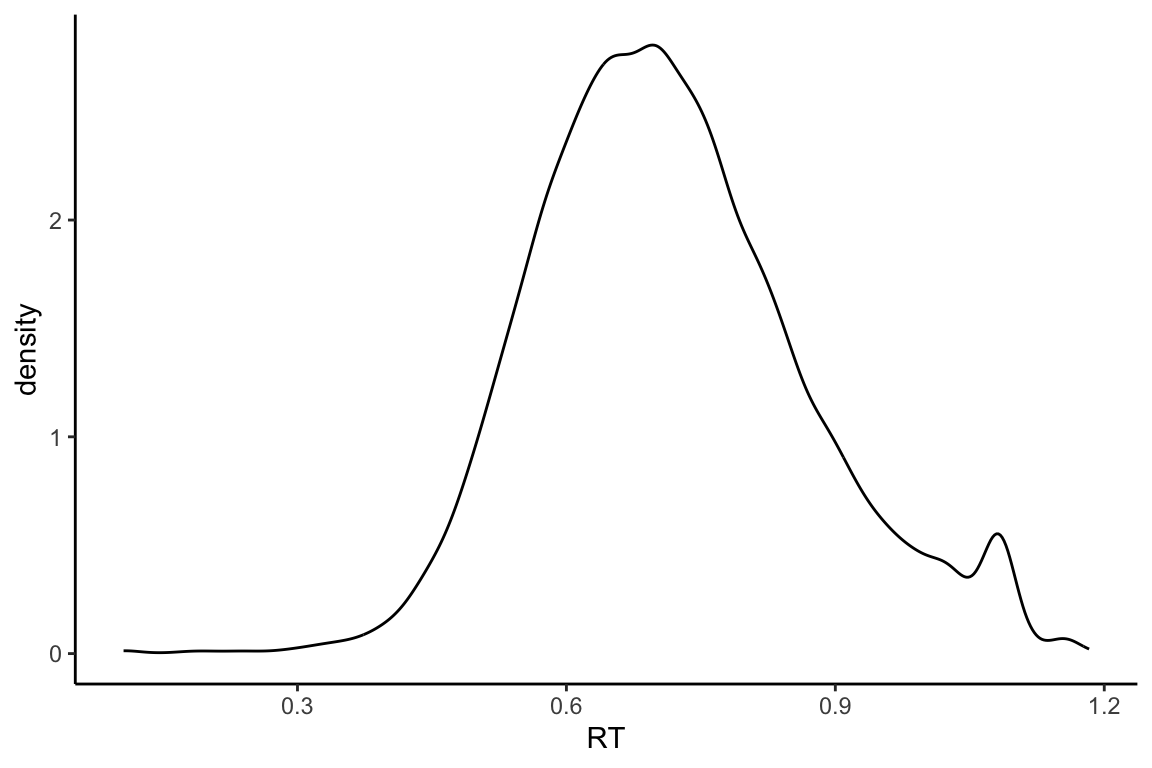 大家可以看到,这里的代码与之前有一点变化。我们同样将整个数据框,包括反应时数据,作为输入,并直接使用ggplot函数。在这里,我们没有添加任何额外的参数。接下来,我们通过叠加图层的方式来增加一个密度曲线。在这个密度曲线的函数中,我们补充了数据映射信息,这与之前的做法是一样的,即只映射x轴。我们将rt(反应时间)映射到x轴上,然后函数就会生成一个曲线图。我们使用了class口的一个功能来实现这一点。大家可以看到,这两个图的信息非常相似:曲线的高度与频次相对应,频次越高的地方,曲线的高度也越高。这里的y轴代表density,即密度。
大家可以看到,这里的代码与之前有一点变化。我们同样将整个数据框,包括反应时数据,作为输入,并直接使用ggplot函数。在这里,我们没有添加任何额外的参数。接下来,我们通过叠加图层的方式来增加一个密度曲线。在这个密度曲线的函数中,我们补充了数据映射信息,这与之前的做法是一样的,即只映射x轴。我们将rt(反应时间)映射到x轴上,然后函数就会生成一个曲线图。我们使用了class口的一个功能来实现这一点。大家可以看到,这两个图的信息非常相似:曲线的高度与频次相对应,频次越高的地方,曲线的高度也越高。这里的y轴代表density,即密度。
7.4.2 直方图+密度图
我们也可以将直方图和密度图尝试叠加在一起
## 尝试将两个图层叠加在一起
mt_raw %>% ggplot(aes(x = RT))+
geom_histogram(bins = 40) +
geom_density() +
theme_classic()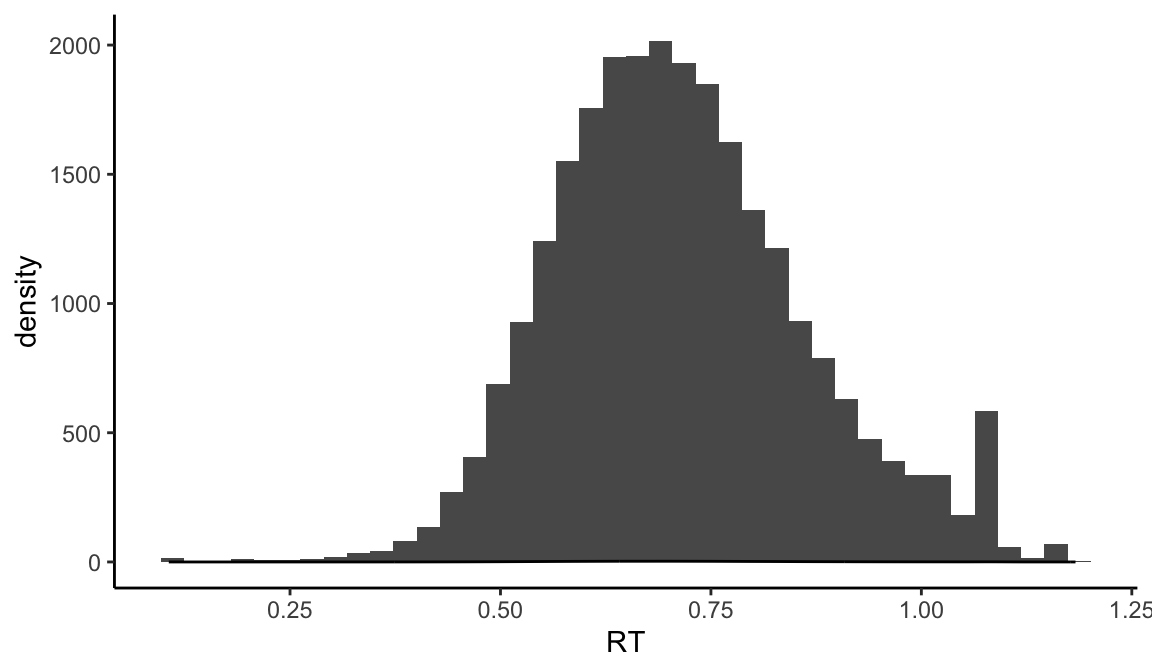 我们想要直接在这个频次图上叠加图层,首先将rt(反应时间)映射到x轴上,并使用histogram函数来绘制直方图。画完直方图后,我们考虑到ggplot2可以直接进行图层的叠加,所以我们可以尝试直接在直方图上叠加一层密度图。我们期望的是,如果图绘制正确,我们应该能看到一条沿着直方图边缘的曲线,因为这两者实际上表示的是同一种信息。
但是,我们实际上看不到这条曲线,这可能是因为y轴的信息差异,单位没有保持一致,直方图的单位可能是500或1000,而密度图的可能只有1或2,这导致密度图可能已经叠加在上面,但由于其规模较小,我们无法直观地看到。这是因为直方图和密度图在y轴上的单位没有保持一致。
为了解决这个问题,我们可以将直方图的y轴从计数转换为密度,这样无论是绘制直方图还是密度图,我们都是以密度作为绘图的数据映射,这样,我们就能在直方图上清晰地看到叠加的密度曲线,它们表示的是类似的信息,代码如下:
我们想要直接在这个频次图上叠加图层,首先将rt(反应时间)映射到x轴上,并使用histogram函数来绘制直方图。画完直方图后,我们考虑到ggplot2可以直接进行图层的叠加,所以我们可以尝试直接在直方图上叠加一层密度图。我们期望的是,如果图绘制正确,我们应该能看到一条沿着直方图边缘的曲线,因为这两者实际上表示的是同一种信息。
但是,我们实际上看不到这条曲线,这可能是因为y轴的信息差异,单位没有保持一致,直方图的单位可能是500或1000,而密度图的可能只有1或2,这导致密度图可能已经叠加在上面,但由于其规模较小,我们无法直观地看到。这是因为直方图和密度图在y轴上的单位没有保持一致。
为了解决这个问题,我们可以将直方图的y轴从计数转换为密度,这样无论是绘制直方图还是密度图,我们都是以密度作为绘图的数据映射,这样,我们就能在直方图上清晰地看到叠加的密度曲线,它们表示的是类似的信息,代码如下:
pic_mix <- mt_raw %>%
ggplot(aes(x = RT,
## 直方图的统计结果通过after_stat(density)传递给了密度图
y = after_stat(density))) + #<<
geom_histogram() +
geom_density() +
theme_classic()
# 设定绘图风格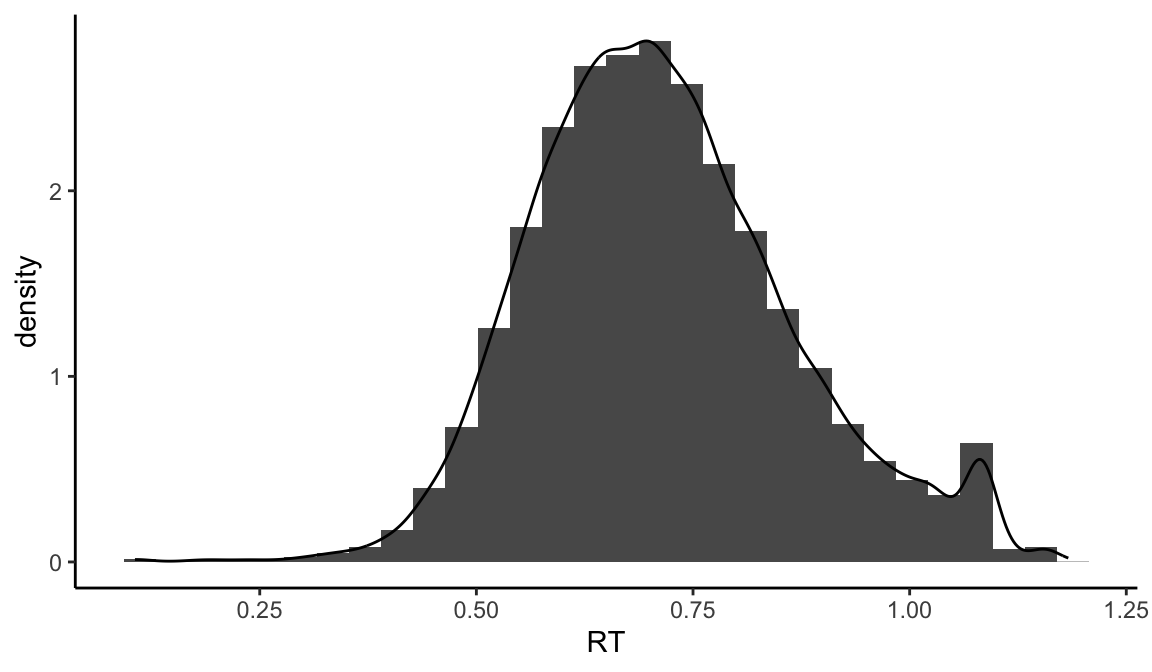 在代码中,我们需要做的是将rt映射到x轴上,并显式地指定y轴的处理方式。默认情况下,当只绘制直方图时,y轴会自动设置为计数。但我们可以显式地将y轴设置为对rt进行density处理,这样y轴就会显示rt在不同区间的密度。通过这种方式,无论是使用histogram还是density,它们都将使用一致的y轴范围,从而使得两种信息能够在同一张图上进行呈现。
在代码中,我们需要做的是将rt映射到x轴上,并显式地指定y轴的处理方式。默认情况下,当只绘制直方图时,y轴会自动设置为计数。但我们可以显式地将y轴设置为对rt进行density处理,这样y轴就会显示rt在不同区间的密度。通过这种方式,无论是使用histogram还是density,它们都将使用一致的y轴范围,从而使得两种信息能够在同一张图上进行呈现。
7.4.3 箱线图
箱型图也是我们常用的一种图形,除了将单个变量可视化,我们可以尝试将两个变量的关系可视化,在这种情况下,我们可以考虑使用箱线图(box plot)。大家可能还记得,我们的数据根据不同的条件(比如图形的好与坏,自己与他人,以及匹配与不匹配)分成了几种情况。如果我们想查看这四种条件下,无论匹配与否,反应时间是否有显著差异,我们可以通过箱线图进行快速可视化,代码如下:
pic_box <- mt_raw %>% ggplot(aes(
x = Label,
y = RT)) +
geom_boxplot(staplewidth = 1) +
# 绘制箱线图并添加上下边缘线
theme_classic()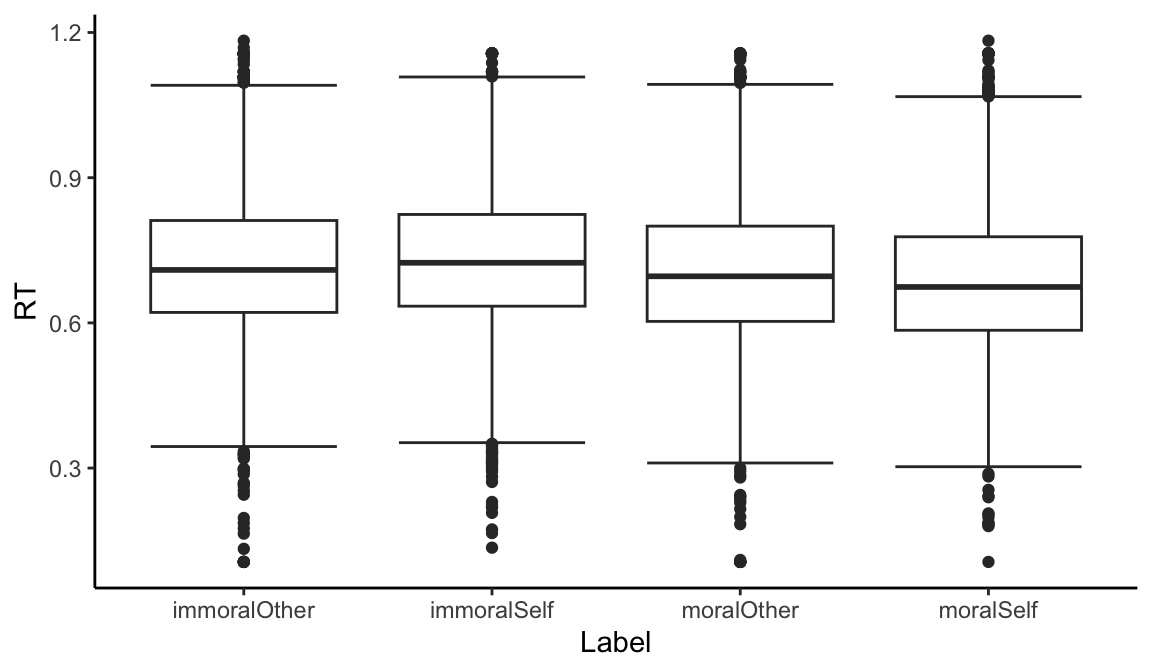 在映射数据时,我们将条件(label)映射到x轴,由于这是字符型数据,R语言会将其视为离散数据,并在x轴上表示为四个不同的点,每个点代表一个条件。R语言默认按照字母顺序对字符型数据进行排序。在y轴上,我们映射的是rt(反应时间),这是一个连续型数值。
完成数据映射后,我们可以选择多种方式来展示结果。对于初步查看不同条件之间是否有区别,我们通常使用均值或中位数来表示数据的整体情况,使用四分位距、全距或标准差来表示数据的离散情况。箱线图能够将这些元素综合展示,让我们能够同时看到数据的集中趋势和离散程度。
在这里,我们使用了ggplot2的箱线图功能,它会自动计算每个条件下反应时间的中位数和四分位距,并将离群值标记出来。箱线图的每个框代表一个条件,框中的横线表示中位数,上下边界表示四分位距。R语言默认使用1.5倍四分位距作为离群值的判定标准。
ggplot2的优势在于,它简化了绘制复杂图形的过程,通过集成的默认值和简单的命令,就能生成箱线图这样的数据可视化图形。
箱型图矩形中间线为中位数,上下两条线分别为上四分位数和下四分位数;1.5个四分位距(Q3-Q1)以外的值为离群值;goem_boxplot默认使用1.5IQR。
在映射数据时,我们将条件(label)映射到x轴,由于这是字符型数据,R语言会将其视为离散数据,并在x轴上表示为四个不同的点,每个点代表一个条件。R语言默认按照字母顺序对字符型数据进行排序。在y轴上,我们映射的是rt(反应时间),这是一个连续型数值。
完成数据映射后,我们可以选择多种方式来展示结果。对于初步查看不同条件之间是否有区别,我们通常使用均值或中位数来表示数据的整体情况,使用四分位距、全距或标准差来表示数据的离散情况。箱线图能够将这些元素综合展示,让我们能够同时看到数据的集中趋势和离散程度。
在这里,我们使用了ggplot2的箱线图功能,它会自动计算每个条件下反应时间的中位数和四分位距,并将离群值标记出来。箱线图的每个框代表一个条件,框中的横线表示中位数,上下边界表示四分位距。R语言默认使用1.5倍四分位距作为离群值的判定标准。
ggplot2的优势在于,它简化了绘制复杂图形的过程,通过集成的默认值和简单的命令,就能生成箱线图这样的数据可视化图形。
箱型图矩形中间线为中位数,上下两条线分别为上四分位数和下四分位数;1.5个四分位距(Q3-Q1)以外的值为离群值;goem_boxplot默认使用1.5IQR。
7.5 Data Explorer
7.5.1 Data Explorer
Explorer也是一个很不错的数据探索工具,可以帮助我们快速探索数据。我们可以使用安装工具包来实现可视化,比如plot_string,它可以将DataFrame中的所有列名以可视化的形式表达出来,类似于思维导图中的树形图。

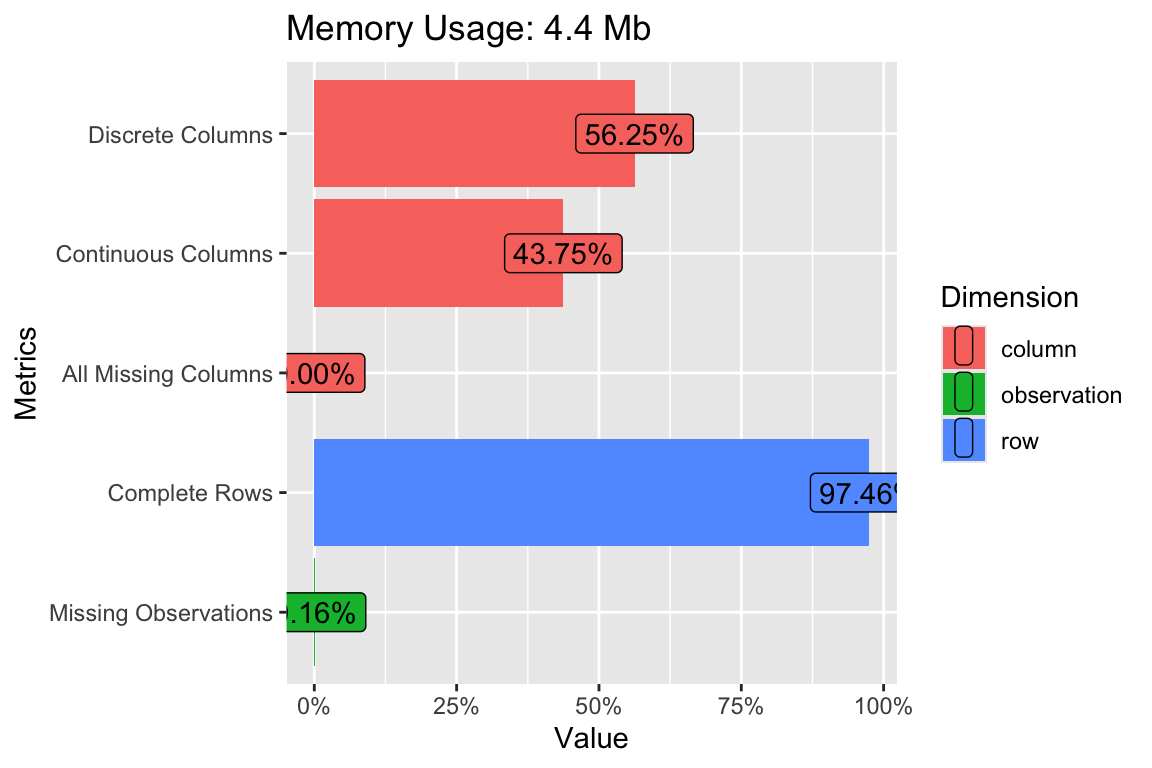
另一个是plot_intro,它可以显示一些信息,比如有多少个离散数据列,有多少个连续数据列等等。我们可以看到,对于我们的匹配数据,离散列占56%,连续列占43%,所有列都是缺失值的占0%。每个数据至少都有一些值,完整的行占97.46%。缺失观测值的数量也可以通过可视化方式快速了解。
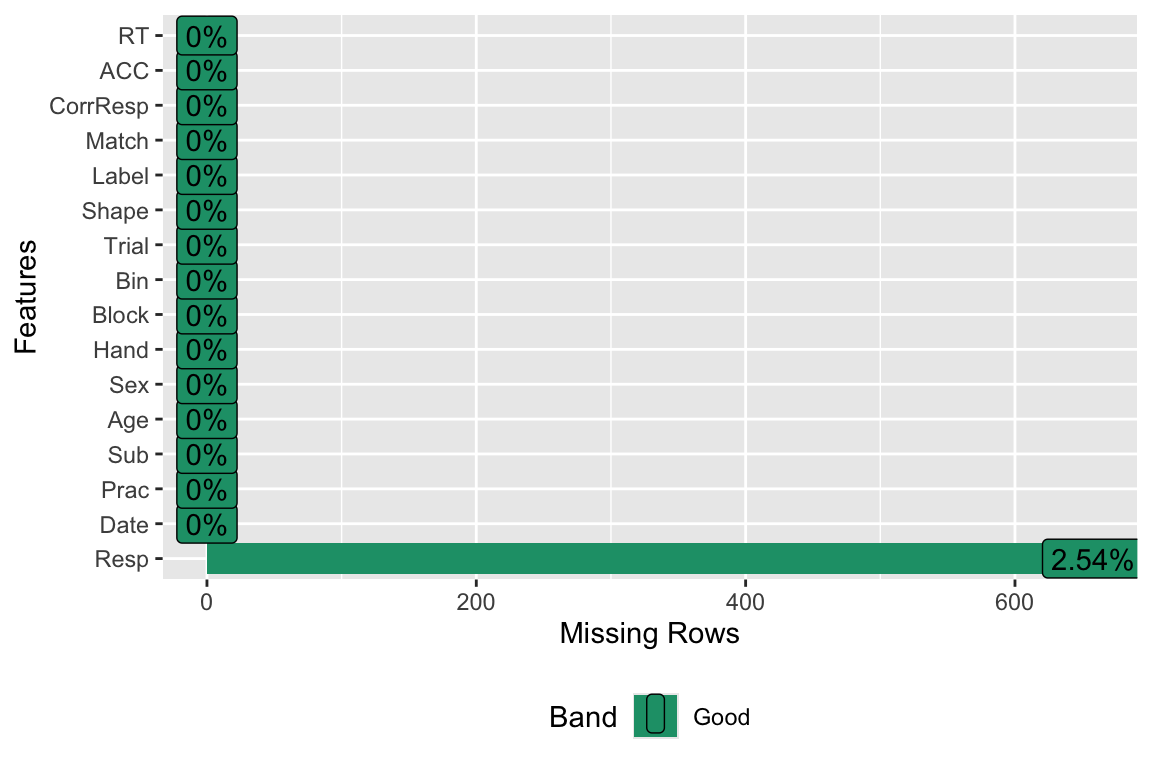
据探索包的一个独特特点,它可以帮助我们快速可视化数据。关于缺失值,我们可以使用plot_missing命令将具有缺失值的列可视化。大多数列都没有缺失值,只有一个响应列有2.5%的缺失值。
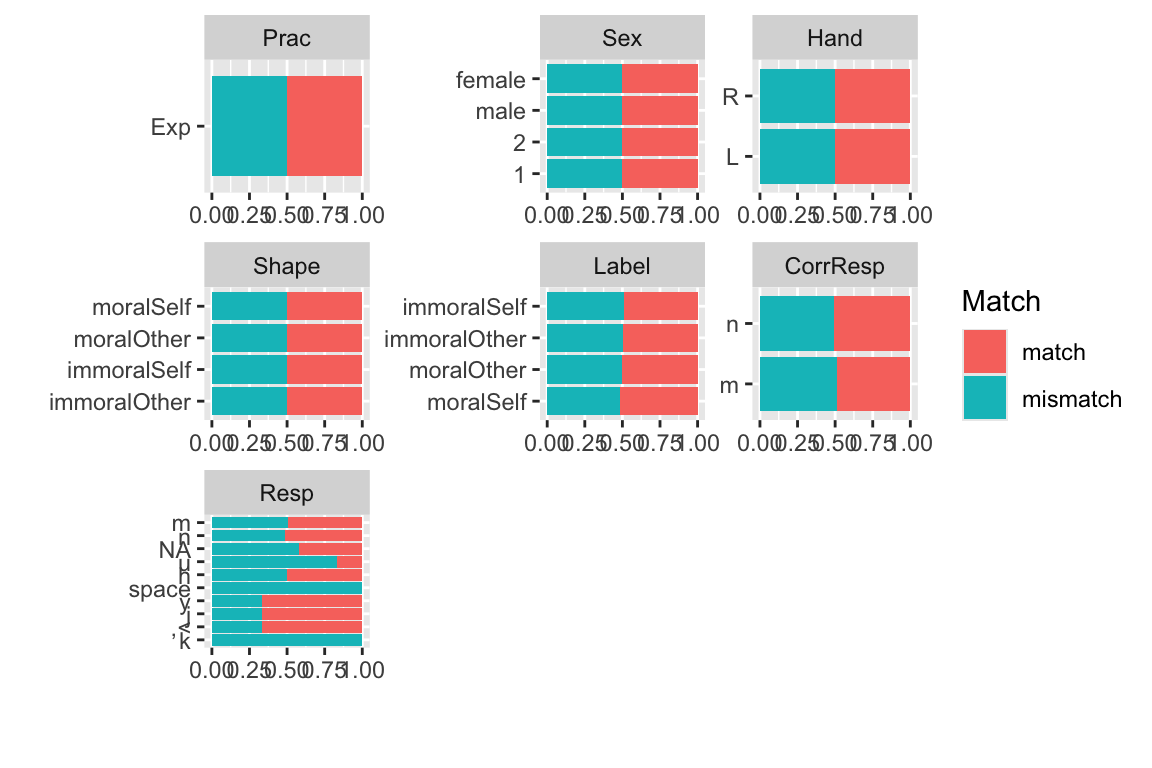
我们可以看到,几乎所有数字化变量的计数都可以用条形图表示。例如,性别可以用female,male,2和1表示。我们可以看到,大多数人是右撇子,而匹配和不匹配的比例是一致的。如果我们在匹配条件下看到匹配比不匹配多或不匹配更多,那么可能存在问题,因为我们的实验设计是一致的。同样,我们的实验条件应该是平衡的,因此看起来应该是一模一样的。
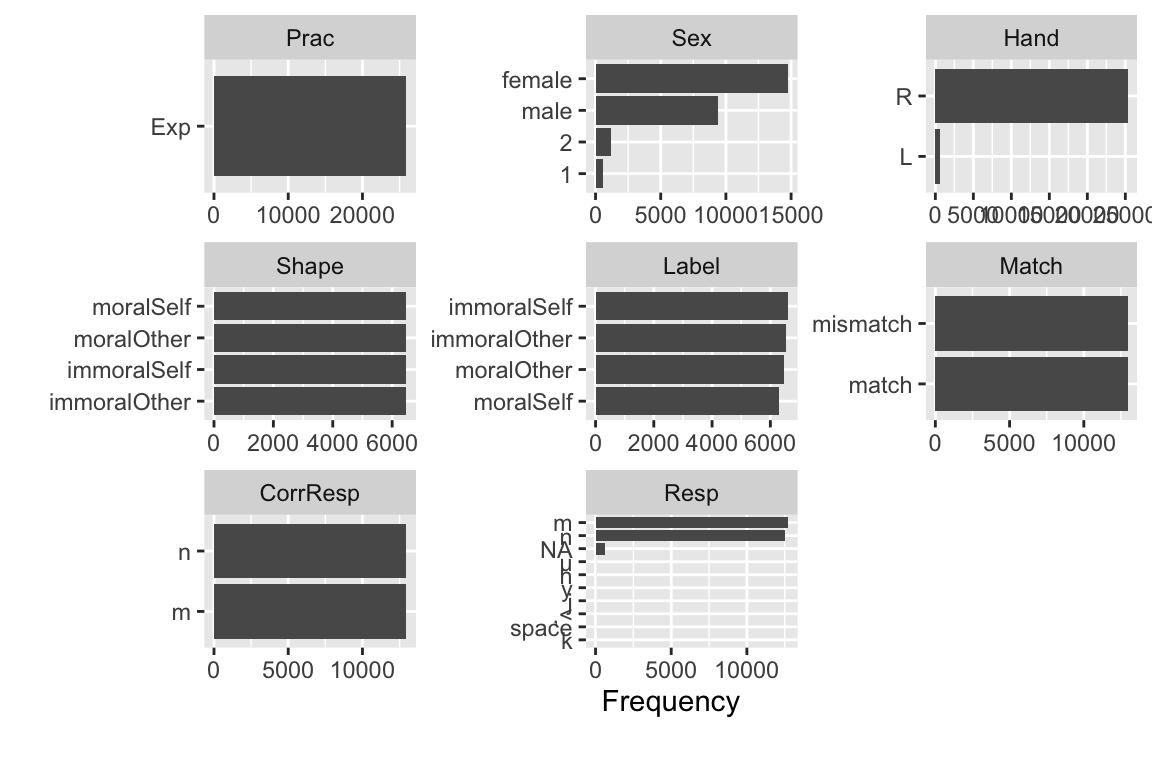 我们可以使用plot_bar将所有变量以bar图的形式呈现出来。我们还可以根据match条件将数据分成matched和mismatched两部分,并用bar图表示每个部分的比例。在大多数情况下,matched和mismatched是平衡的。我们还可以使用histogram来快速绘制所有变量的分布情况,特别是连续变量的分布情况。我们可以使用ggplot来检验数据是否符合正态分布。
我们还可以通过选择一个特定的列作为分组条件,来绘制不同组别的比例。当然,我们这里选择的比例基本上都是相同的,这是因为我们的条件设置在各部分都是一致的。例如,在练习部分,无论是左利手还是右利手完成,图形的呈现都是一样的。这说明了什么呢?这表明大多数情况下,匹配和未匹配的比例是1:1,这符合我们的实验设计。但是,在反应方面,比例可能不是1:1,这意味着被试的反应可能存在一些差异。
我们可以使用plot_bar将所有变量以bar图的形式呈现出来。我们还可以根据match条件将数据分成matched和mismatched两部分,并用bar图表示每个部分的比例。在大多数情况下,matched和mismatched是平衡的。我们还可以使用histogram来快速绘制所有变量的分布情况,特别是连续变量的分布情况。我们可以使用ggplot来检验数据是否符合正态分布。
我们还可以通过选择一个特定的列作为分组条件,来绘制不同组别的比例。当然,我们这里选择的比例基本上都是相同的,这是因为我们的条件设置在各部分都是一致的。例如,在练习部分,无论是左利手还是右利手完成,图形的呈现都是一样的。这说明了什么呢?这表明大多数情况下,匹配和未匹配的比例是1:1,这符合我们的实验设计。但是,在反应方面,比例可能不是1:1,这意味着被试的反应可能存在一些差异。
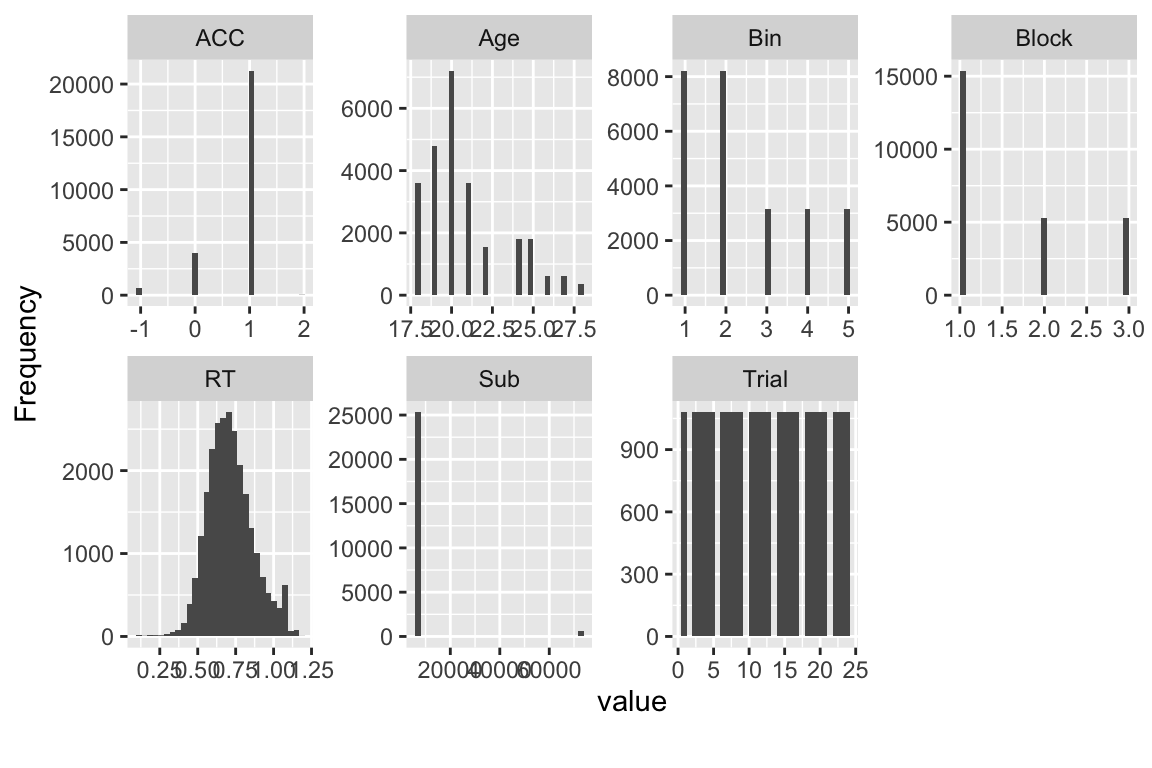
 对于直方图,它能够快速地为数字型数据生成可视化图形。我们之前已经关注了反应时间(rt),如果你有许多这样的数字型数据,多列的频数图可以一下子全部展示出来。这样,我们就可以看到每个列、每个数据列的整体情况。
对于直方图,它能够快速地为数字型数据生成可视化图形。我们之前已经关注了反应时间(rt),如果你有许多这样的数字型数据,多列的频数图可以一下子全部展示出来。这样,我们就可以看到每个列、每个数据列的整体情况。
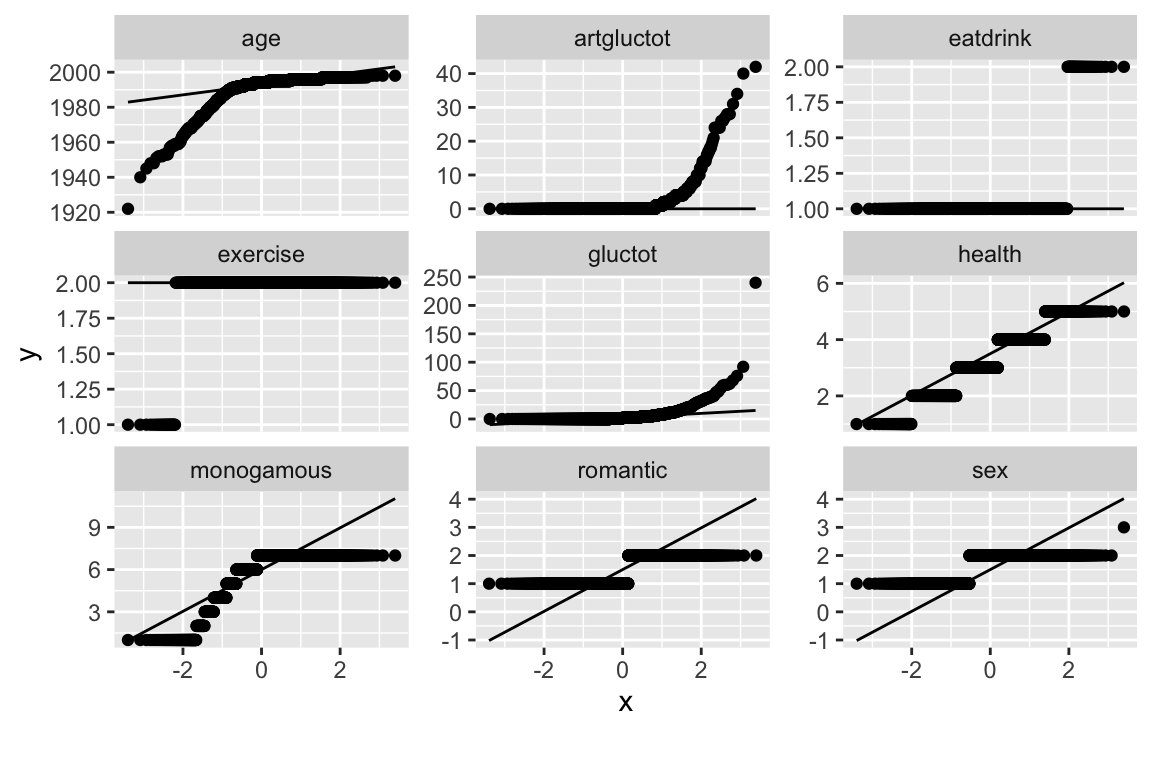
 还可以画qq图来看数据的正态性
还可以画qq图来看数据的正态性
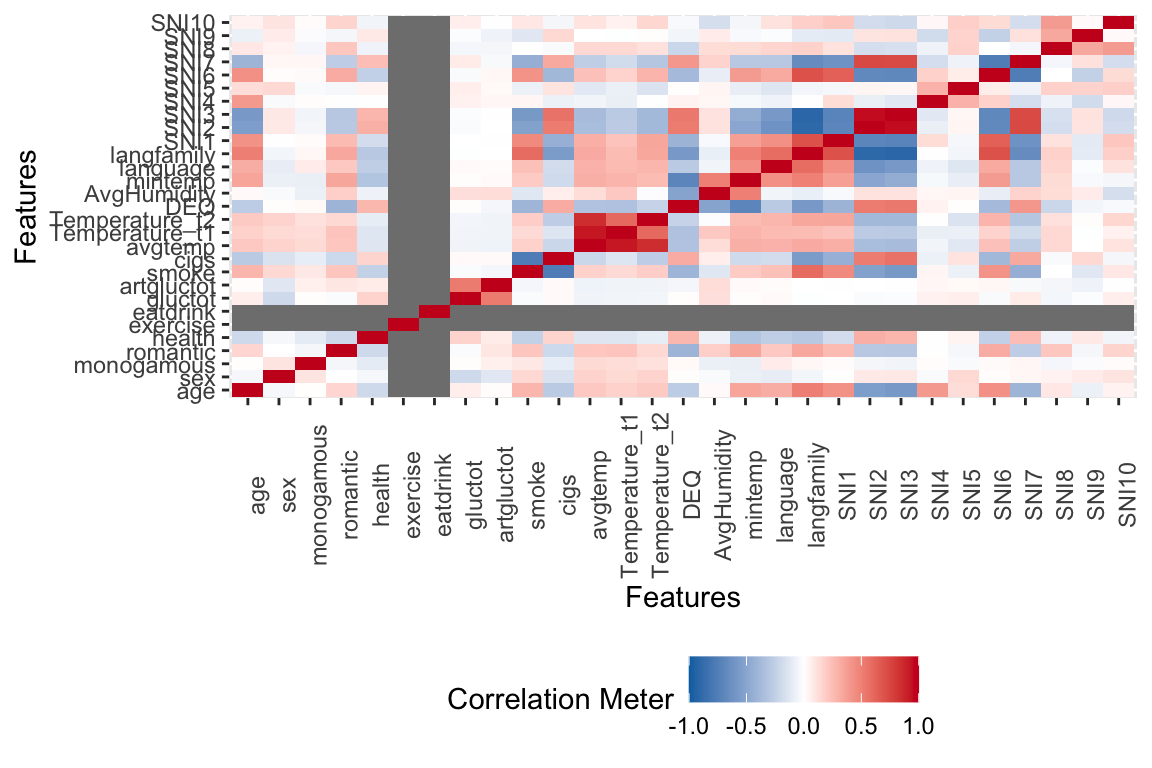
 这个包是为了快速探索数据框中数据的情况而专门开发的。它提供了许多实用且快速的功能,例如相关系数(correlation)分析。如果你进行问卷分析,结构图会非常友好,因为它能让你看到不同条目之间的得分,以及它们是否显示出预期的高相关或低相关。
这个包是为了快速探索数据框中数据的情况而专门开发的。它提供了许多实用且快速的功能,例如相关系数(correlation)分析。如果你进行问卷分析,结构图会非常友好,因为它能让你看到不同条目之间的得分,以及它们是否显示出预期的高相关或低相关。
 在ggplot2体系中,还开发了一些符合其规范的附加包,比如“ggpairs”。这个包可以在数据组合后直接绘制它们之间的关系图,图中会展示相关系数和每个数字的分布。此外,还有散点图,它能在探索相关性时快速提供原始数据、统计值以及每个数据自身的分布情况。这些功能都帮助我们快速地进行数据探索。
在ggplot2体系中,还开发了一些符合其规范的附加包,比如“ggpairs”。这个包可以在数据组合后直接绘制它们之间的关系图,图中会展示相关系数和每个数字的分布。此外,还有散点图,它能在探索相关性时快速提供原始数据、统计值以及每个数据自身的分布情况。这些功能都帮助我们快速地进行数据探索。
7.5.2 使用ggpairs
code
## 以 penguin project 数据中 ALEX, stress和 ECR 为例
pg_raw %>%
mutate(
# 计算均值
m_ALEX = bruceR::MEAN(.,
var = 'ALEX',items = 1:16,rev = c(4,12,14,16)),
m_stress = bruceR::MEAN(.,
var = 'stress',items = 1:14,rev = c(4:7,9,10,13)
),
m_ECR = bruceR::MEAN(.,
var = 'ECR',items = 1:36
)
) %>%
select(contains('m_')) %>%
GGally::ggpairs()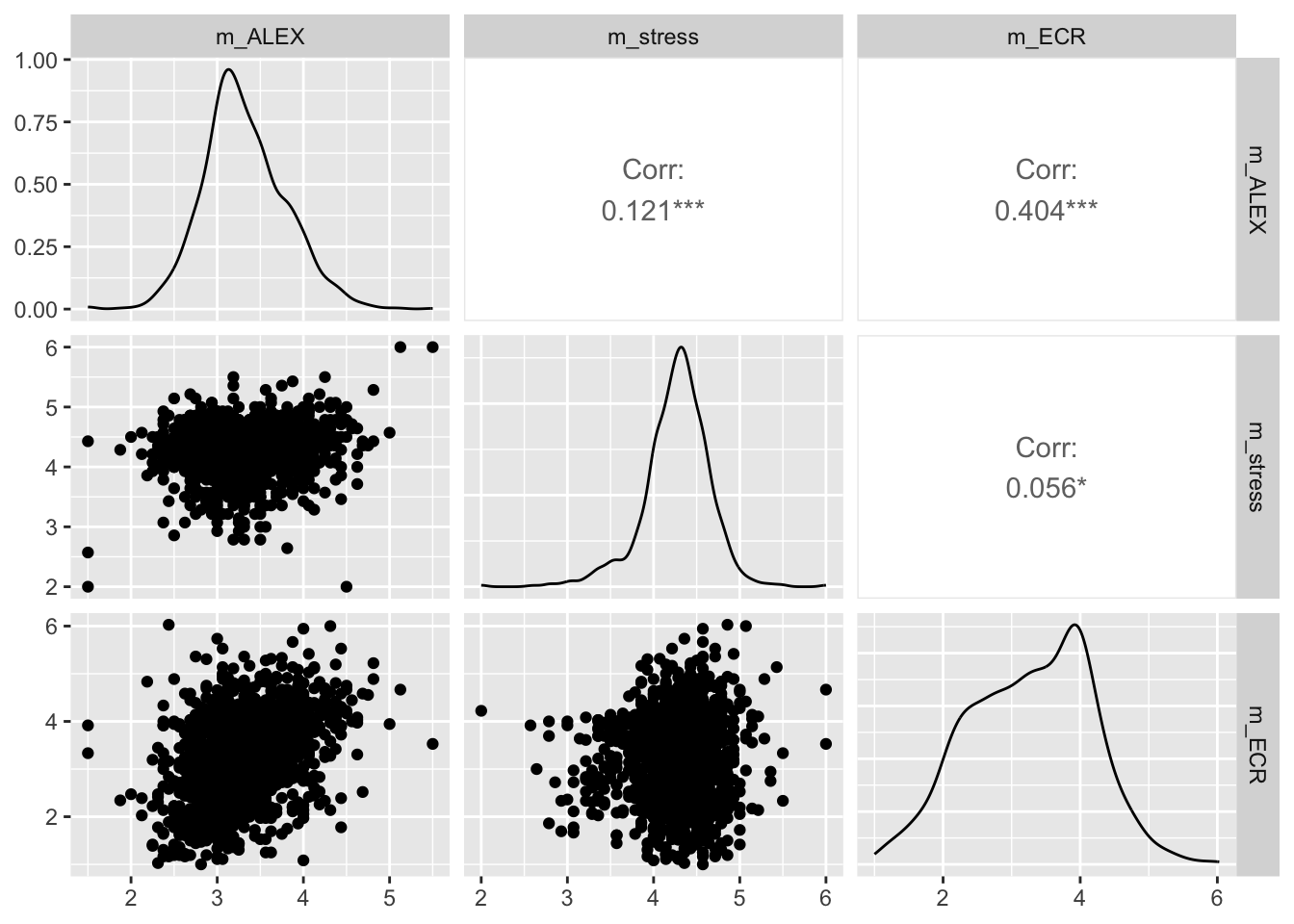
本节课重点强调了数据探索的重要性,尤其是通过可视化方式进行探索。我们已经开始使用ggplot2,虽然我只是展示了ggplot2的一些功能,并没有深入讲解每个元素的操控细节。这是因为我们的数据分析课程有一个渐进的流程:从数据导入、清洗,到初步了解数据,接下来自然是进行统计分析。在接下来的几节课中,我们将学习如何使用R进行统计分析。 这就要求大家通过练习来提高自己操作数据和探索数据的能力,将前面学到的内容整合起来。例如,通过选择数据框中的数据来进行绘图,以及对数据类型进行转换,改变图形中元素的呈现顺序。我建议大家针对不同图形的击中率进行分组绘图,并使用箱线图来展示。

