Capítulo 4 Visualización de datos
4.1 Paquetes necesarios para este capítulo
Para este capítulo necesitas tener instalado el paquete tidyverse.
En este capítulo se explicará qué es el paquete ggplot2 (Wickham 2016) y cómo utilizarlo para visualizar datos.
Dado que este libro es un apoyo para el curso BIO4022, esta clase puede también ser seguida en este link. El video de la clase se encontrará disponible en este link
4.2 El esqueleto
El esqueleto de una visualización usando ggplot2 es la siguiente
Como ejemplo para discutir usaremos el siguiente código que genera la figura 4.1:
library(tidyverse)
data("diamonds")
ggplot(diamonds, aes(x = carat, y = price)) + geom_point(aes(color = cut)) +
theme_classic()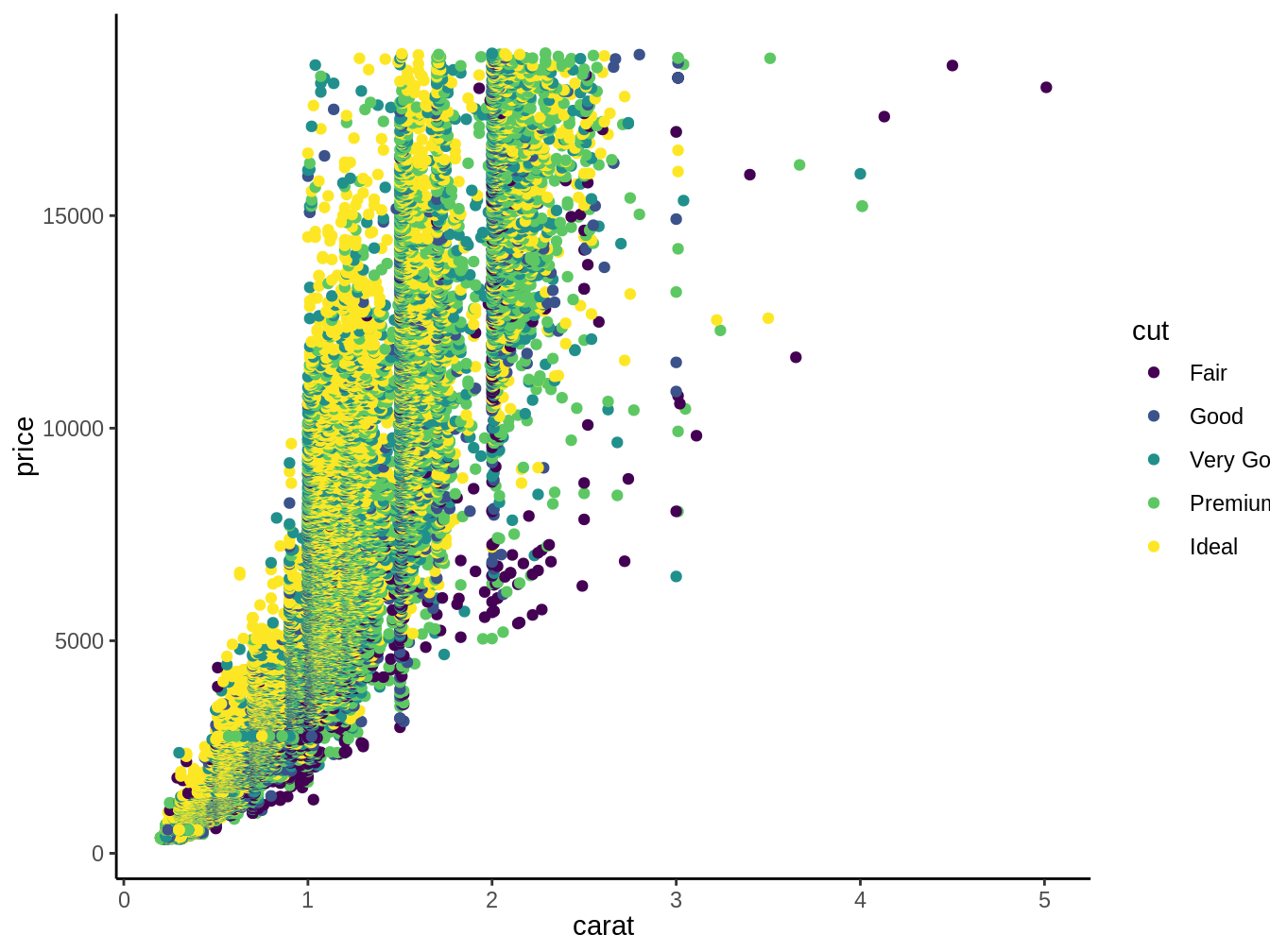
Figura 4.1: Gráfico en el cual gráficamos los quilates de diamantes versus su precio, con el corte del diamante representado por el color
En este caso general, lo primero que ponemos después de ggplot es el data.frame desde el cual graficaremos algo. En el ejemplo de la figura 4.1 usamos la base de datos diamonds del paquete ggplot2 (Wickham 2016), luego dentro de aes ponemos las columnas que graficaremos como x y/o y. En nuestro ejemplo dentro de aes ponemos como eje x los quilates de los diamantes (caret) y como y el precio de los mismos (price). Ojo que existe la necesidad de poner aes en ggplot2 (algo que no había sido necesario cuando usamos dplyr o tidyr).
4.3 Por que usamos aes() y +
Al ser el primer paquete creado en el tidyverse, ggplot2 tiene un par de convenciones distintas. Por un lado, cada vez que usamos el nombre de una columna que está en un data frame debemos usarlo dentro de la función aes. Además, cuando se creó el paquete ggplot2 no existia el pipeline (%>%), por lo que se utilizaba el signo + con la misma función.
4.4 geom_algo
Luego de especificar una base de datos, se debe continuar con un geom_algo, esto nos indicará que tipo de gráfico usaremos. Los gráficos pueden ser combinados como veremos en ejemplos futuros.
4.4.1 Una variable categórica una continua
Primero veremos algunos de los geom que podemos utilizar con una variable categórica y una continua
4.4.1.1 geom_boxplot
En la figura 4.2, generado a partir del código a continuación con la base de datos iris presente en R (Anderson 1935).
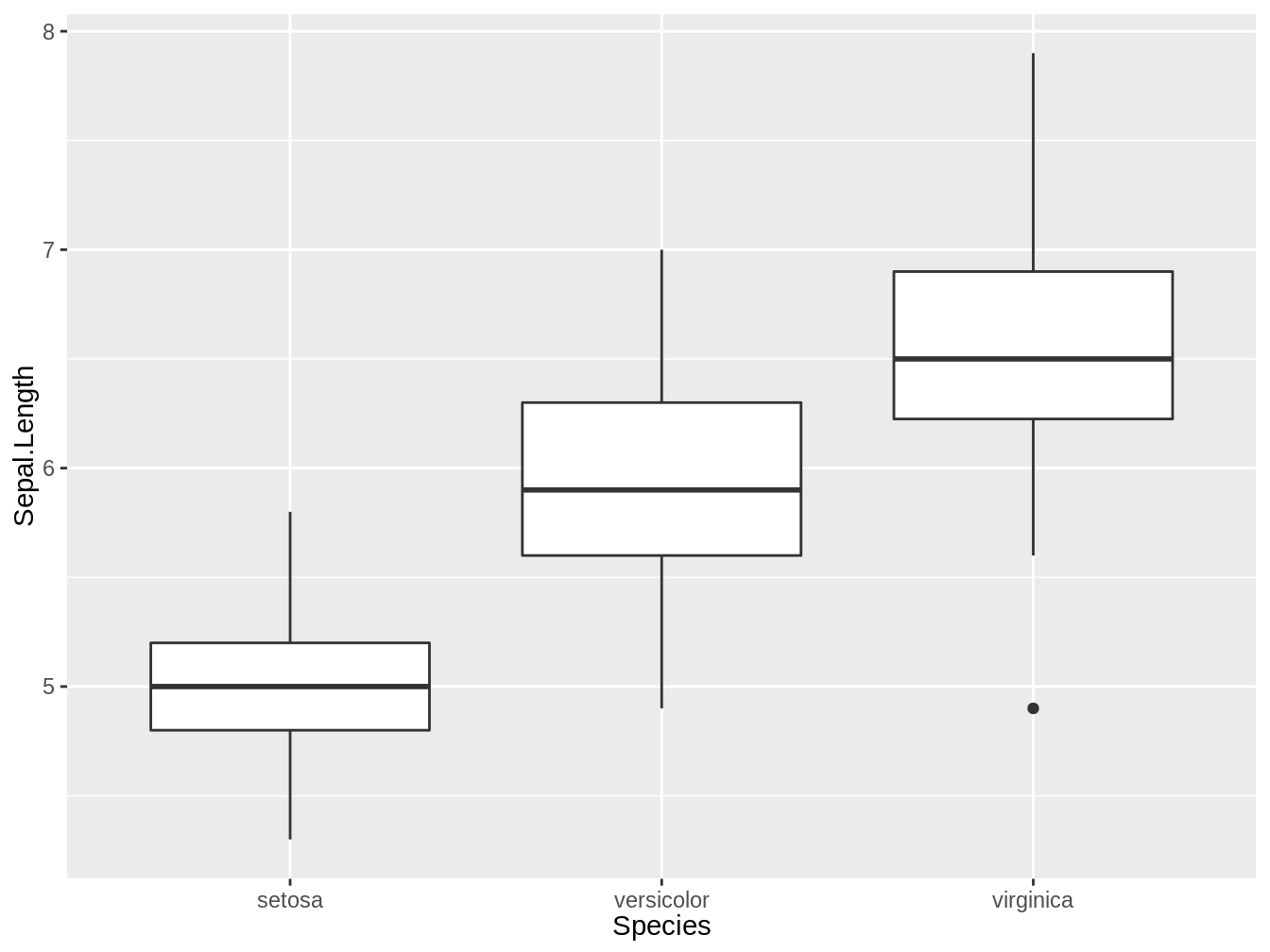
Figura 4.2: Boxplot que representa los largos del sépalo de tres especies del género Iris
Los boxplots muestran una línea gruesa central (la mediana), una caja, que delimita el primer y tercer cuartil y los bigotes, los cuales se extienden hasta los valores extremos. En el caso que estos valores estén por sobre 1.5 veces la distancia entre el primer y tercer cuartil, estos serán representados por puntos (siendo considerados outlyers). En la figura 4.2, sólo Iris virginica presenta un outlayer en cuanto a las medidas del largo del sépalo.
Los boxplots, como todos los gráficos pueden ser personalizados usando otros argumentos, los que mostraremos en esta sección los iremos introduciendo de a poco. Si quisieramos por ejemplo que el color de las cajas del boxplot fueran dea cuerdo a la especie, cambiamos el llenado (fill) de la caja, como vemos en el siguiente ejemplo y figura 4.3
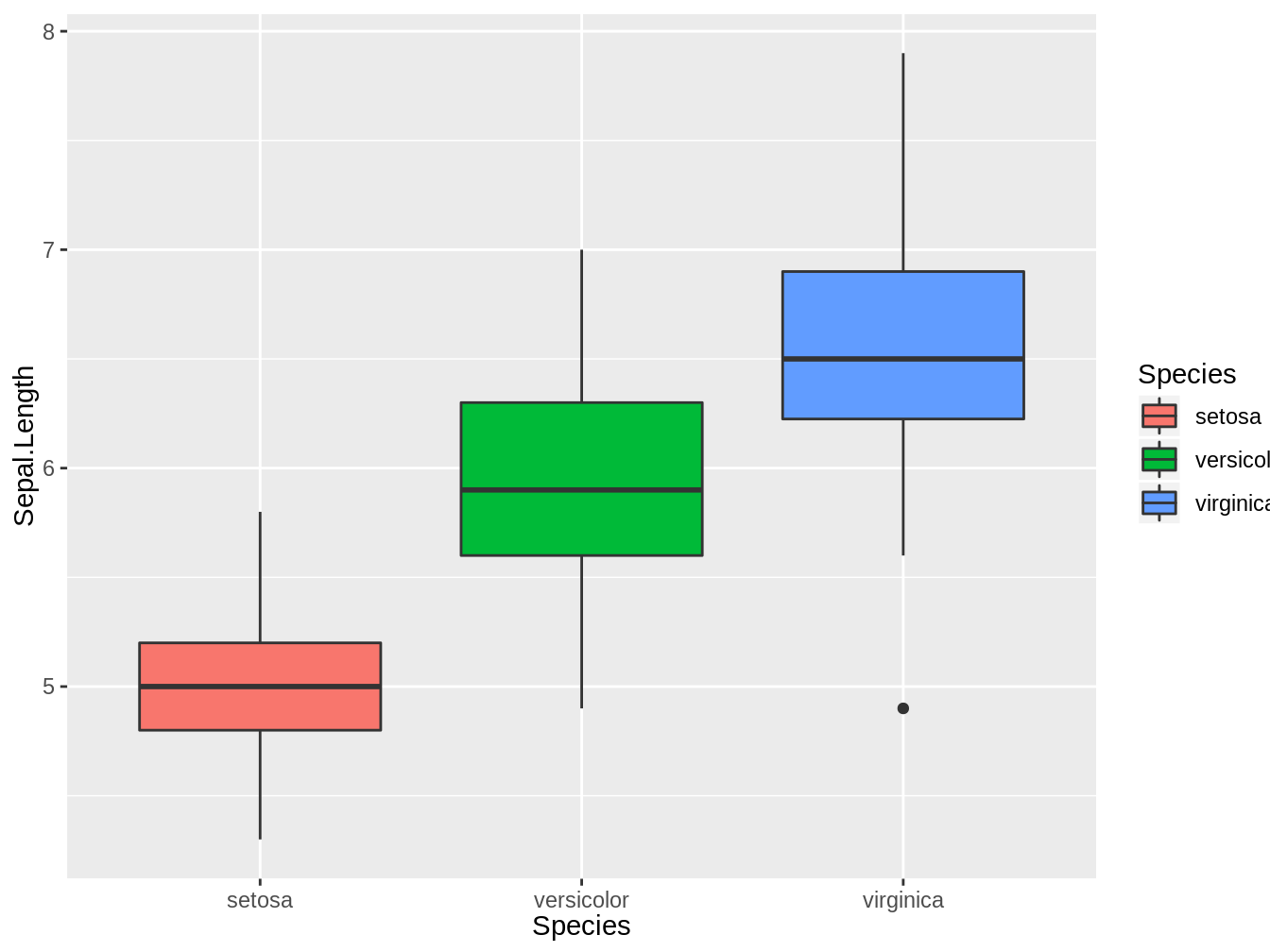
Figura 4.3: Boxplot que representa los largos del sépalo de tres especies del género Iris, en este caso el color de la caja representa la especie
Dos cosas a notar en este ejemplo, por un lado la leyenda se genera de forma automática, y por otro lado, vemos que es necesario poner Species dentro de aes, esto es debido a que Species es una columna y como se explicó al principio de este capítulo, todas las columnas deben ser incuidas dentro de la función aes para poder ser referenciadas.
4.4.1.2 geom_jitter
Utilizando la misma base de datos, podemos crear un gráfico del tipo jitter. En este caso hay un punto por cada observación, lo cual puede ayudar a entender mejor los datos que tenemos.
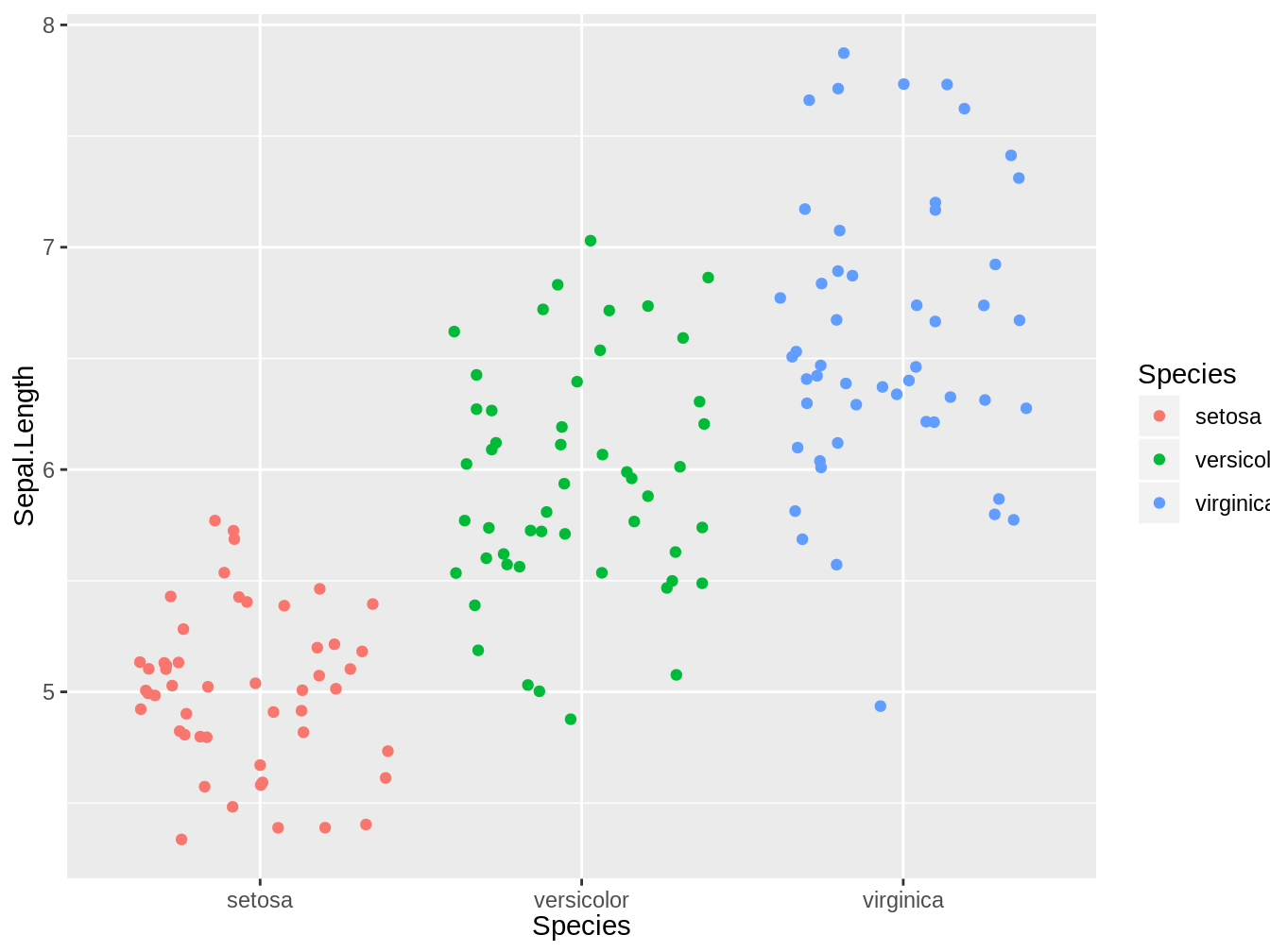
Figura 4.4: jitter plot que representa los largos del sépalo de tres especies del género Iris, en este caso el color de los puntos representan la especie
En la figura 4.4 vemos los mismos datos que en la figura 4.2, el agregar el color = Species dentro del aes nos permite que el color de cada punto este determinado por la especie a la que pertenece.
4.4.1.3 Otros geom categóricos
Otros geom categóricos que podemos explorar con esta base de datos son:
- geom_violin
- geom_bar
- geom_col
4.5 Combinando geoms
Uno puede escribir varios geoms para formar un gráfico combinado. Por ejemplo, podríamos generar un gráfico con un boxplot y un jitter plot, como vemos en la figura 4.5
ggplot(iris, aes(x = Species, y = Sepal.Length)) + geom_boxplot() +
geom_jitter(aes(color = Species))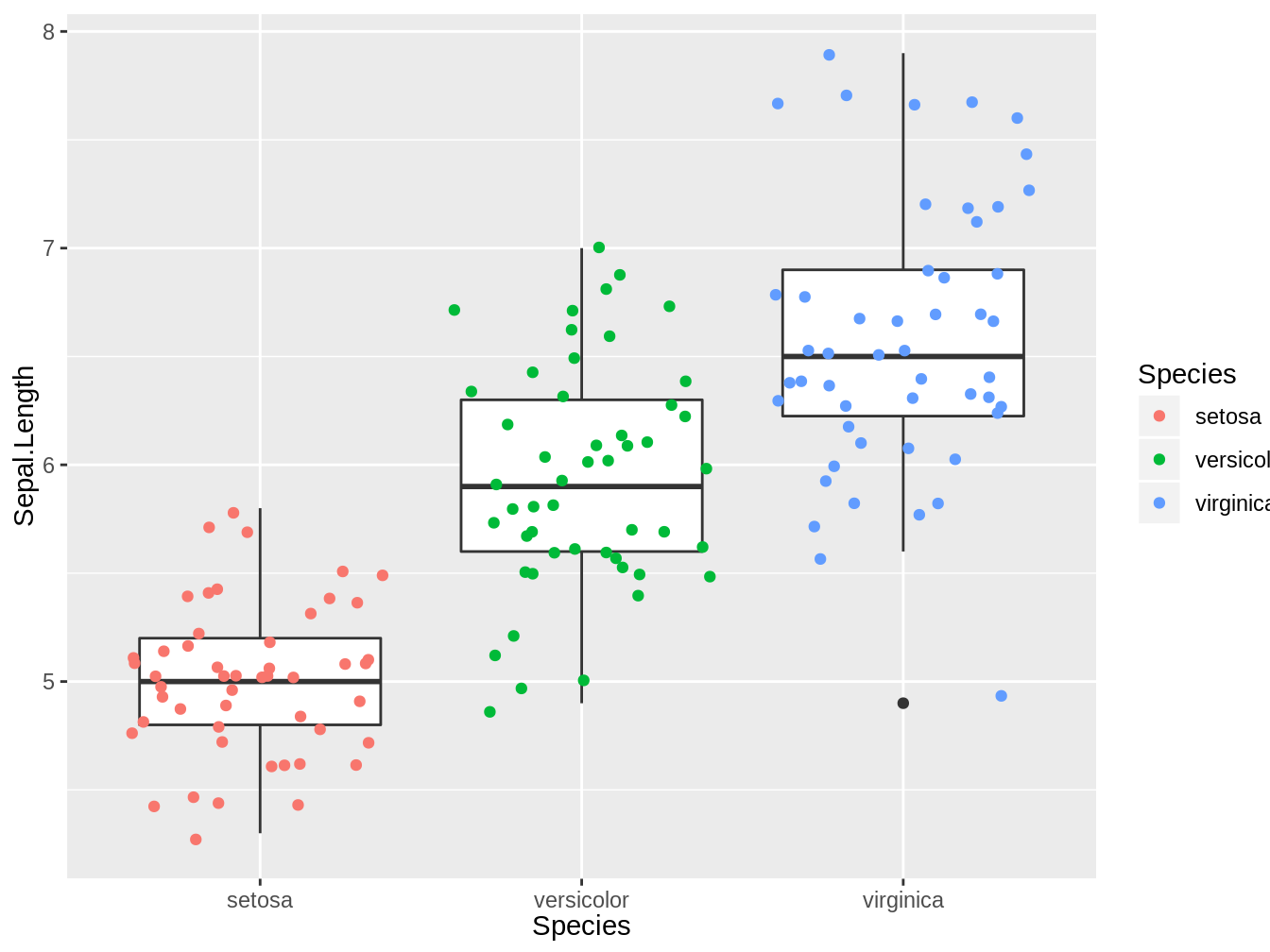
Figura 4.5: Boxplot y jitter plot combinados que representa los largos del sépalo de tres especies del género Iris.
4.5.1 El orden importa
Si bien se pueden combinar los geom, el orden de estos importa, ya que ggplot2 genera las figuras por capas. Esto es ilustrado en la figura 4.6, en la cual al crear primero el jitter y luego el boxplot, las cajas del boxplot tapan los puntos, a diferencia de la figura 4.5
ggplot(iris, aes(x = Species, y = Sepal.Length)) + geom_jitter(aes(color = Species)) +
geom_boxplot()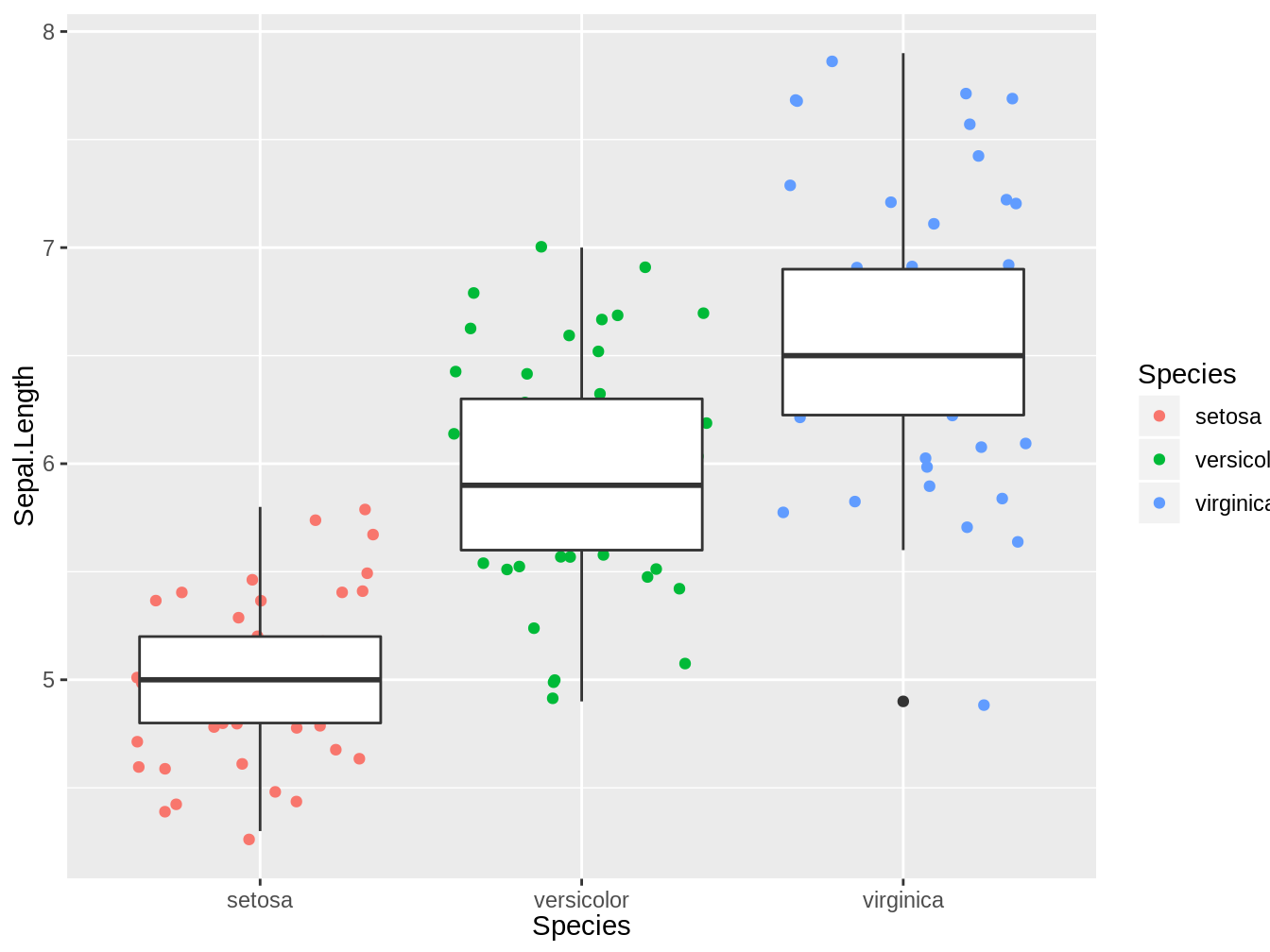
Figura 4.6: Boxplot y jitter plot combinados que representa los largos del sépalo de tres especies del género Iris, en este caso al llamar al jitter antes del boxplot, las cajas tapan los puntos.
4.5.2 Dos variables continuas
Algunos de los geoms que podemos usar para dos variables continuas son:
- geom_point
- geom_smooth
- geom_line
- geom_hex
- geom_rug
Ahora veremos algunos de ellos:
4.5.2.1 geom_point
Este geom es el que nos permite hacer un gráfico de dispersión en R. Para esto tenemos que poner variables continuas en x e y en ggplot y agregar la función geom_point, como vemos en el siguiente código y en la figura 4.7.
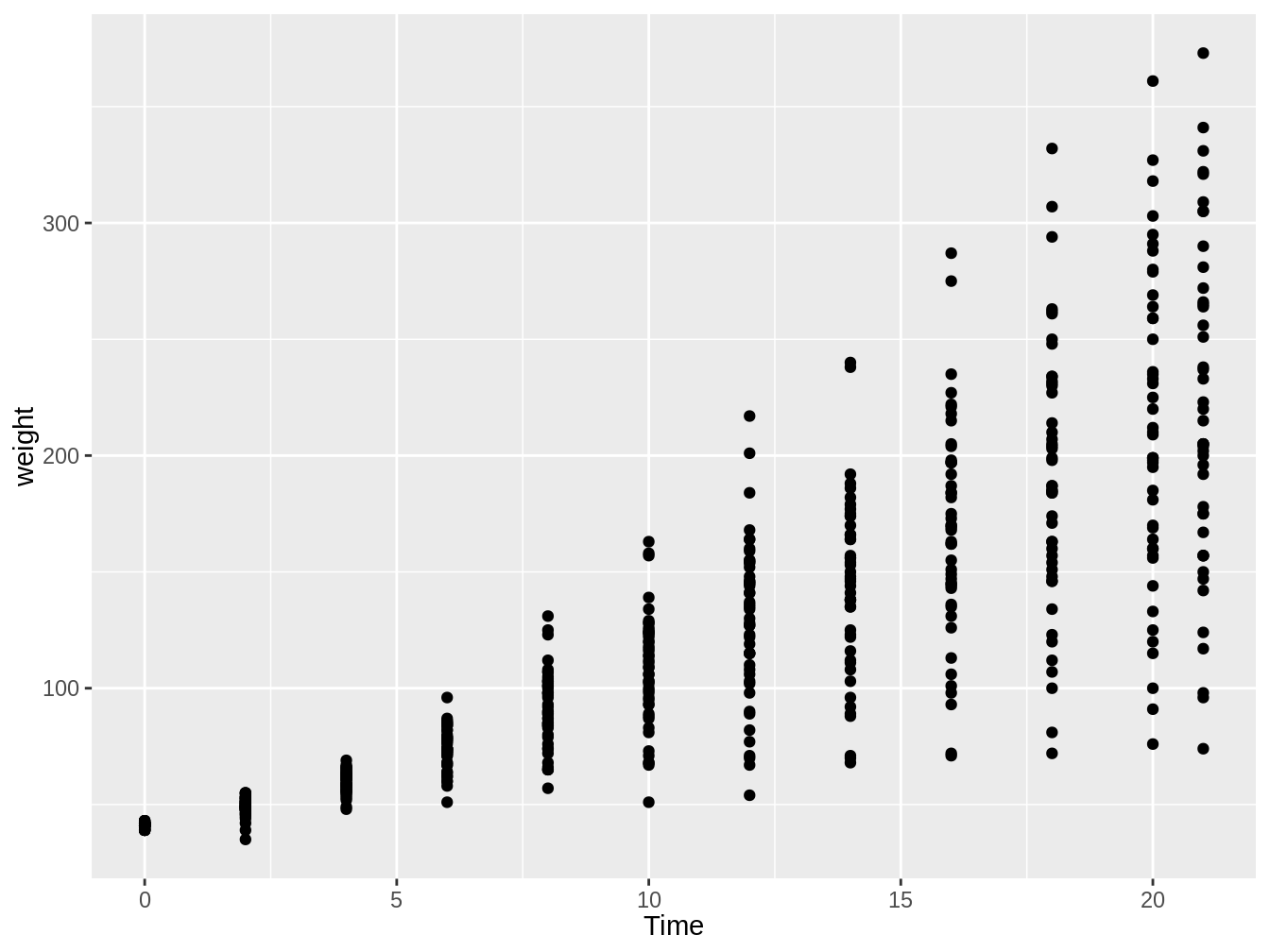
Figura 4.7: Gráfico en el cual vemos el peso de pollos en el tiempo
Si quisieramos que el color de cada punto estuviera separado por dieta, podríamos agregarle aes(color = Diet) a geom_point. Sin embargo, deberíamos transformar Diet en factor, ya sea antes de usar ggplot o dentro de ggplot tal como vemos en el siguiente código y en la figura 4.8.
data("ChickWeight")
ggplot(ChickWeight, aes(x = Time, y = weight)) + geom_point(aes(color = factor(Diet)))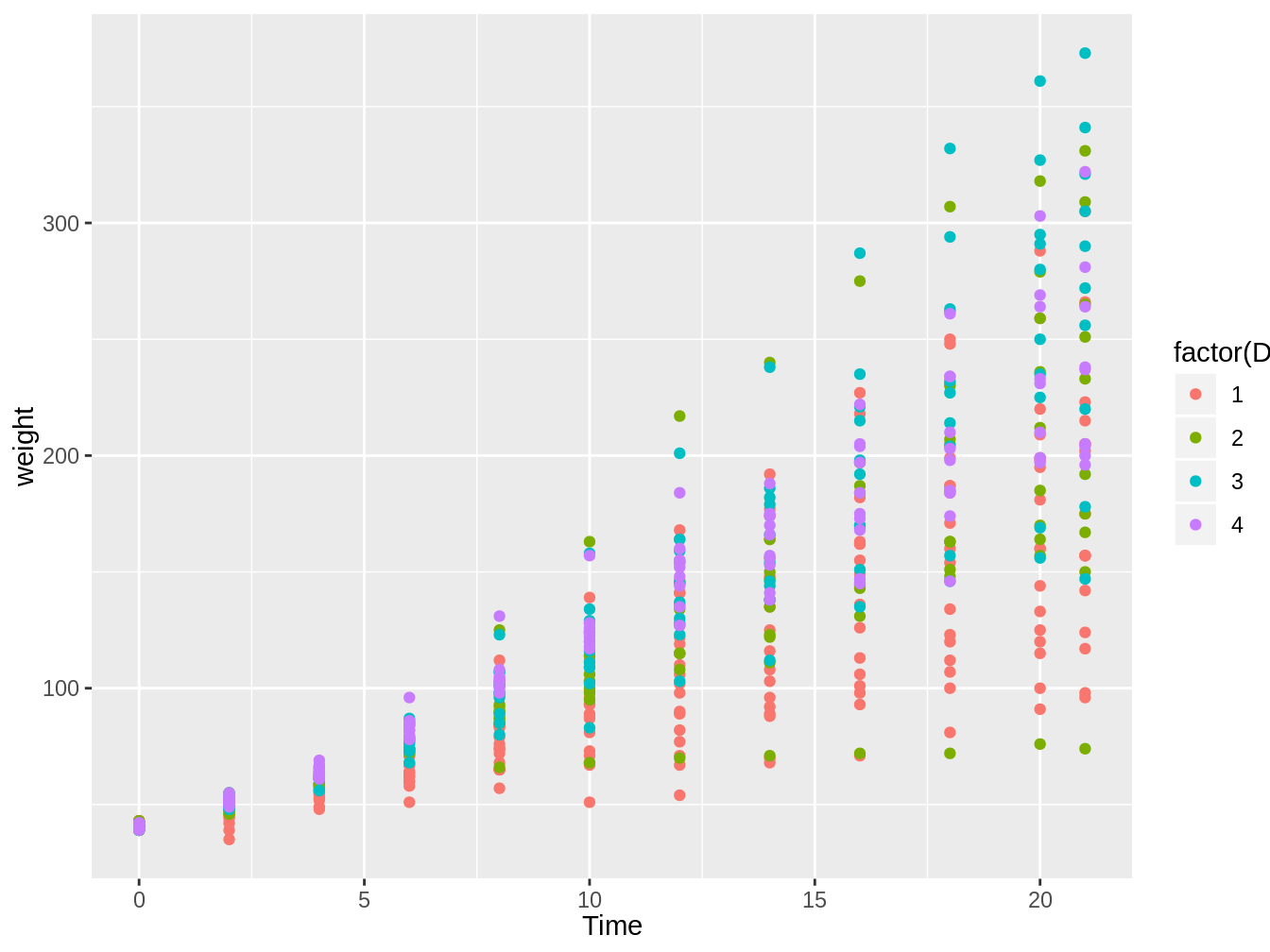
Figura 4.8: Gráfico en el cual vemos el peso de pollos en el tiempo, con colores distintos según el tipo de dieta
4.5.2.2 geom_smooth y stat_smooth
4.5.2.2.1 geom_smooth
Estas funciones nos permiten generar líneas de tendencias con intervalos de confianza. Así si quisieramos ver líneas de tendencias para nuestro scatterplot, dependiendo de la dieta, usaríamos el siguiente código, el cual nos da la figura 4.9.
ggplot(ChickWeight, aes(x = Time, y = weight)) + geom_point(aes(color = factor(Diet))) +
geom_smooth(aes(fill = factor(Diet)))## `geom_smooth()` using method = 'loess' and formula 'y ~ x'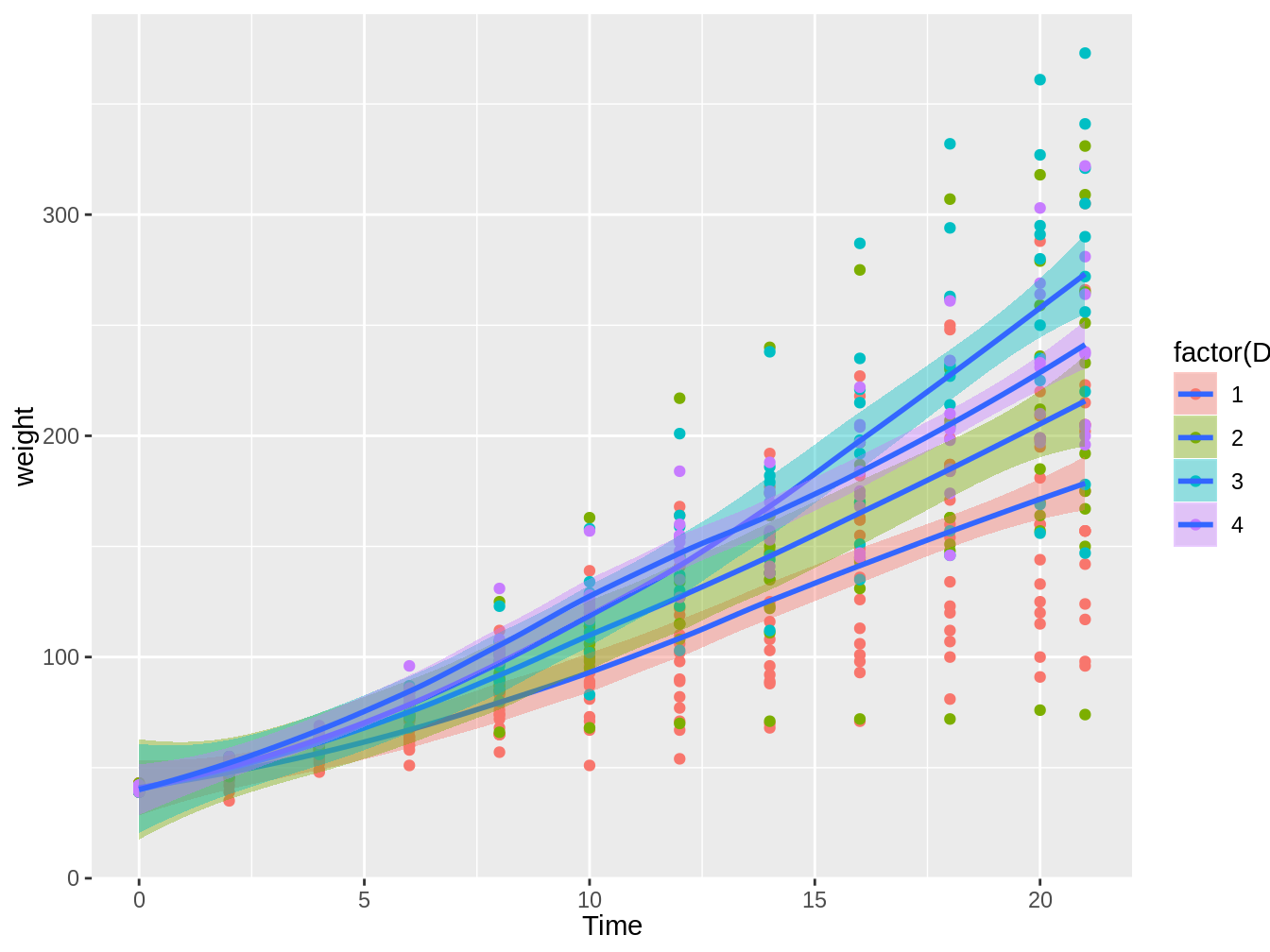
Figura 4.9: Gráfico en el cual vemos el peso de pollos en el tiempo, con colores distintos según el tipo de dieta, con líneas de tendencia e intervalos de confianza basados en el método loess
Por defecto, la función geom_smooth generará una tendencia basada en loess, lo cual es una correlación local. En general, es mejor hacer una línea de tendencia basado en modelos que uno puede explicar mejor como un modelo lineal. Para esto, cambiamos el argumento method a lm como en el siguiente código y la figura 4.10.
ggplot(ChickWeight, aes(x = Time, y = weight)) + geom_point(aes(color = factor(Diet))) +
geom_smooth(aes(fill = factor(Diet)), method = "lm")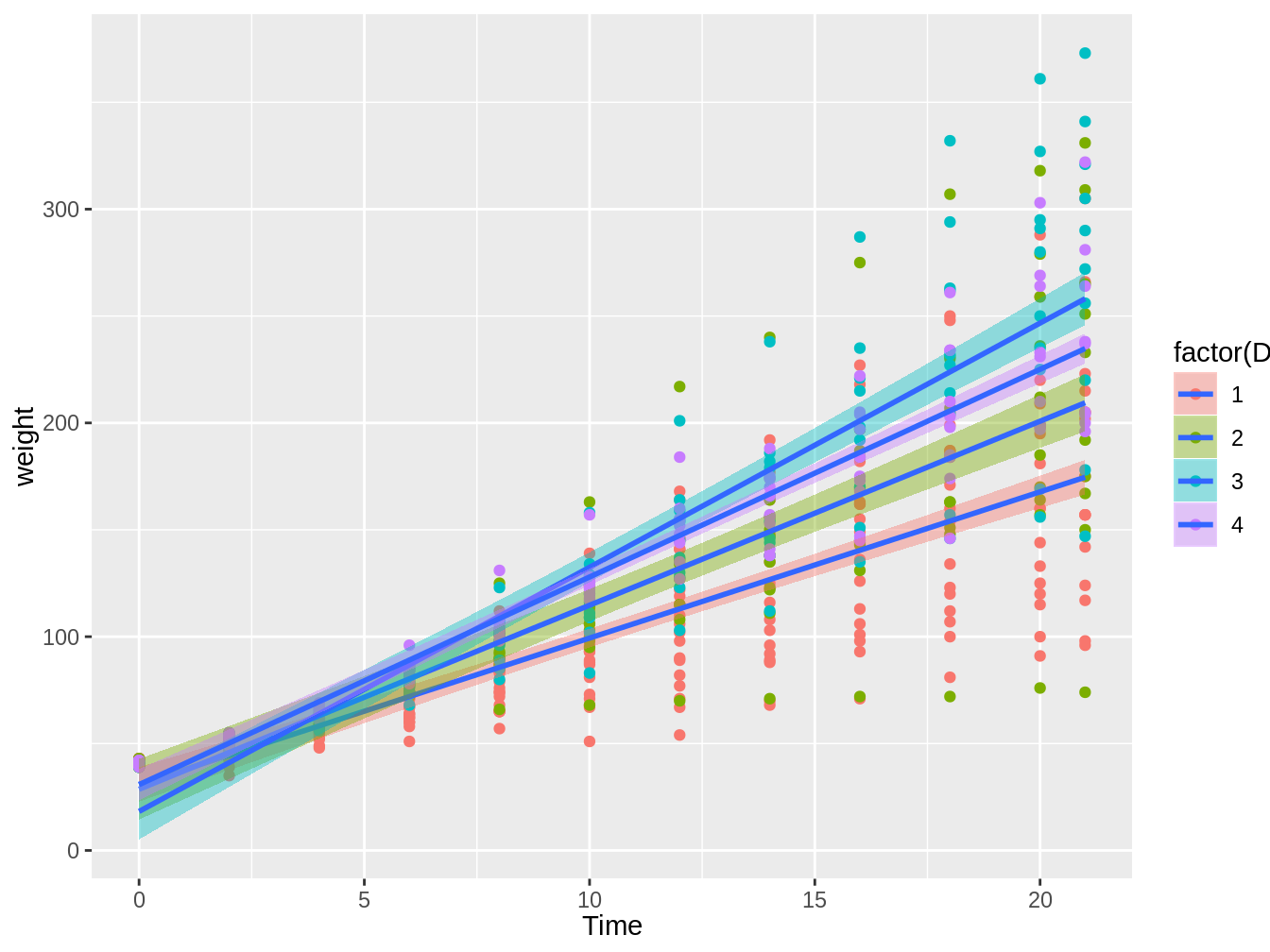
Figura 4.10: Gráfico en el cual vemos el peso de pollos en el tiempo, con colores distintos según el tipo de dieta, con líneas de tendencia e intervalos de confianza basados en modelos lineales
4.5.2.2.2 stat_smooth
La función stat_smooth es más flexible que geom_smooth. La gran diferencia es que nos permite incluir una fórmula para expresar la relación entre \(x\) e \(y\). Por ejemplo, si pensaramos que en el caso de la base de datos ChickWeight la relación entre el peso y el tiempo se expresa mejor con una ecuación cuadrática (ver ecuación (4.1)) tendríamos el siguiente código que genera la figura 4.11.
ggplot(ChickWeight, aes(x = Time, y = weight)) + geom_point(aes(color = factor(Diet))) +
stat_smooth(aes(fill = factor(Diet)), method = "lm", formula = y ~
x + I(x^2))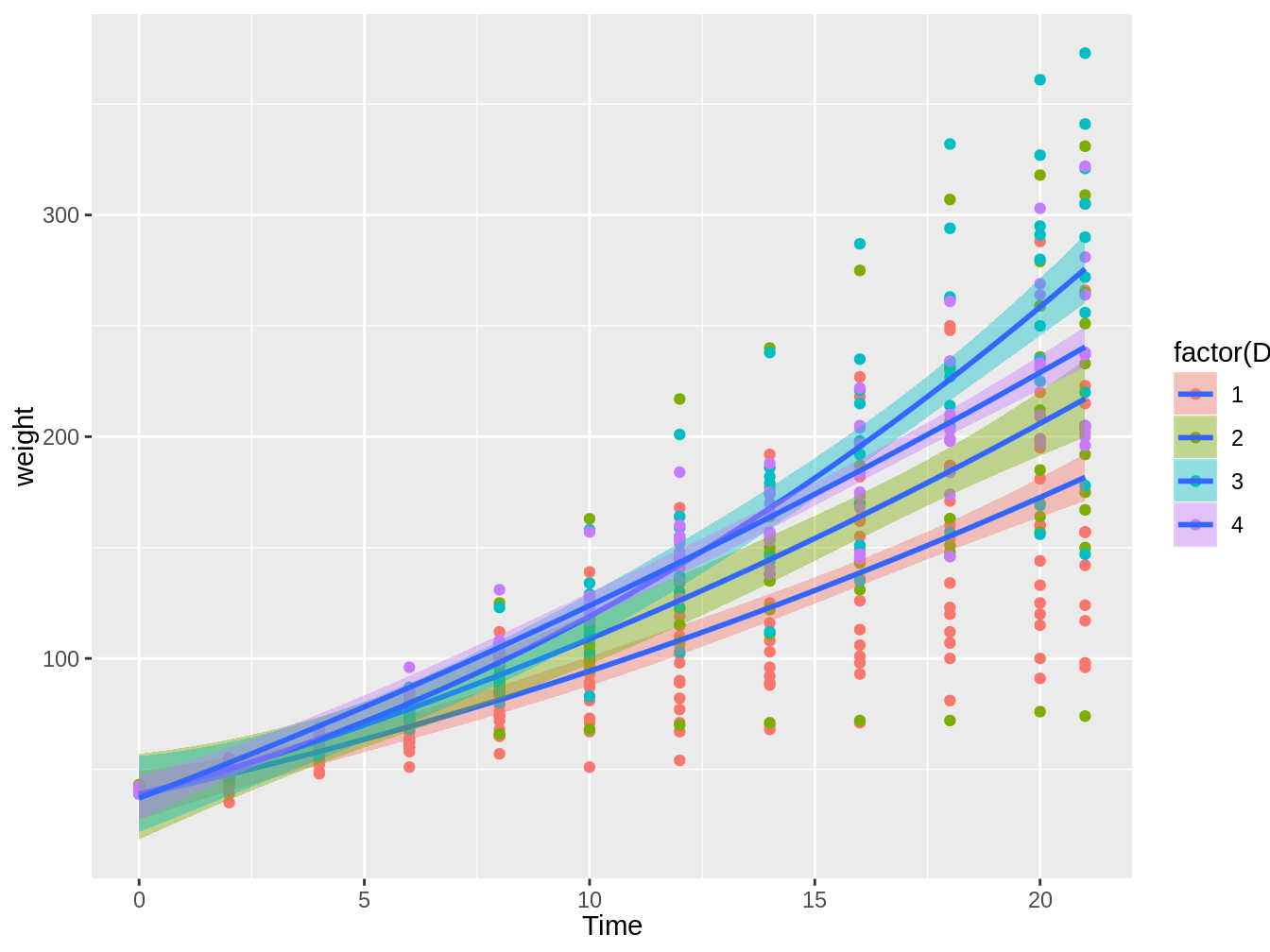
Figura 4.11: Gráfico en el cual vemos el peso de pollos en el tiempo, con colores distintos según el tipo de dieta, con líneas de tendencia e intervalos de confianza basados en modelos lineales con una relación cuadrática
\[\begin{equation} y = \beta_2 x^2 + \beta_1 x + c \tag{4.1} \end{equation}\]
4.5.3 Combinando varios gráficos con facet_wrap
Algunas veces, en particular si tenemos muchas variables categóricas, no es recomendable generar una línea o punto de color distinto para cada variable. Por ejemplo, si seguimos con el crecimiento de los pollos de la base de datos ChickWeight, vemos que la variable Chick representa cada pollo. Dado que hay varios pollos por dieta se vuelve confuso y poco informativo como se ve en la figura 4.12 generada con el siguiente código.
ggplot(ChickWeight, aes(x = Time, y = weight)) + geom_point(aes(color = Diet)) +
geom_line(aes(color = Diet, group = Chick))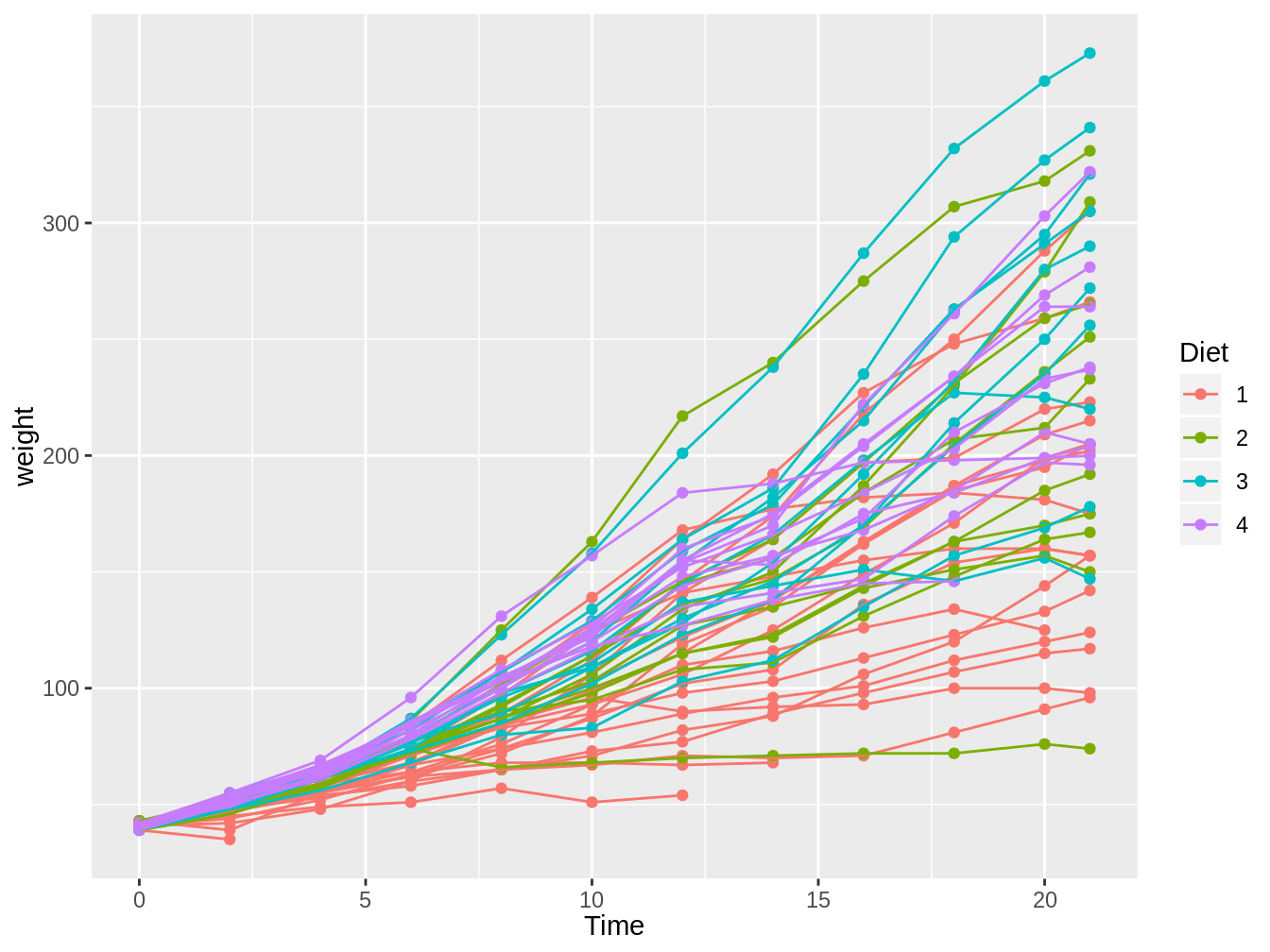
Figura 4.12: Gráfico en el cual vemos el peso de pollos en el tiempo, con colores distintos según el tipo de dieta y con líneas para cada pollo individual.
Para aclarar este enredo, es mejor el generar un gráfico para cada dieta, y es ahí donde entra la función facet_wrap. Esta función nos permite generar el gráfico deseado al agregar como argumento dentro de la función el simbolo ~ seguido del nombre de la variable a utilizar para separar los gráficos, tal como en la figura 4.13 y su código correspondiente.
ggplot(ChickWeight, aes(x = Time, y = weight)) + geom_point(aes(color = Diet)) +
geom_line(aes(color = Diet, group = Chick)) + facet_wrap(~Diet)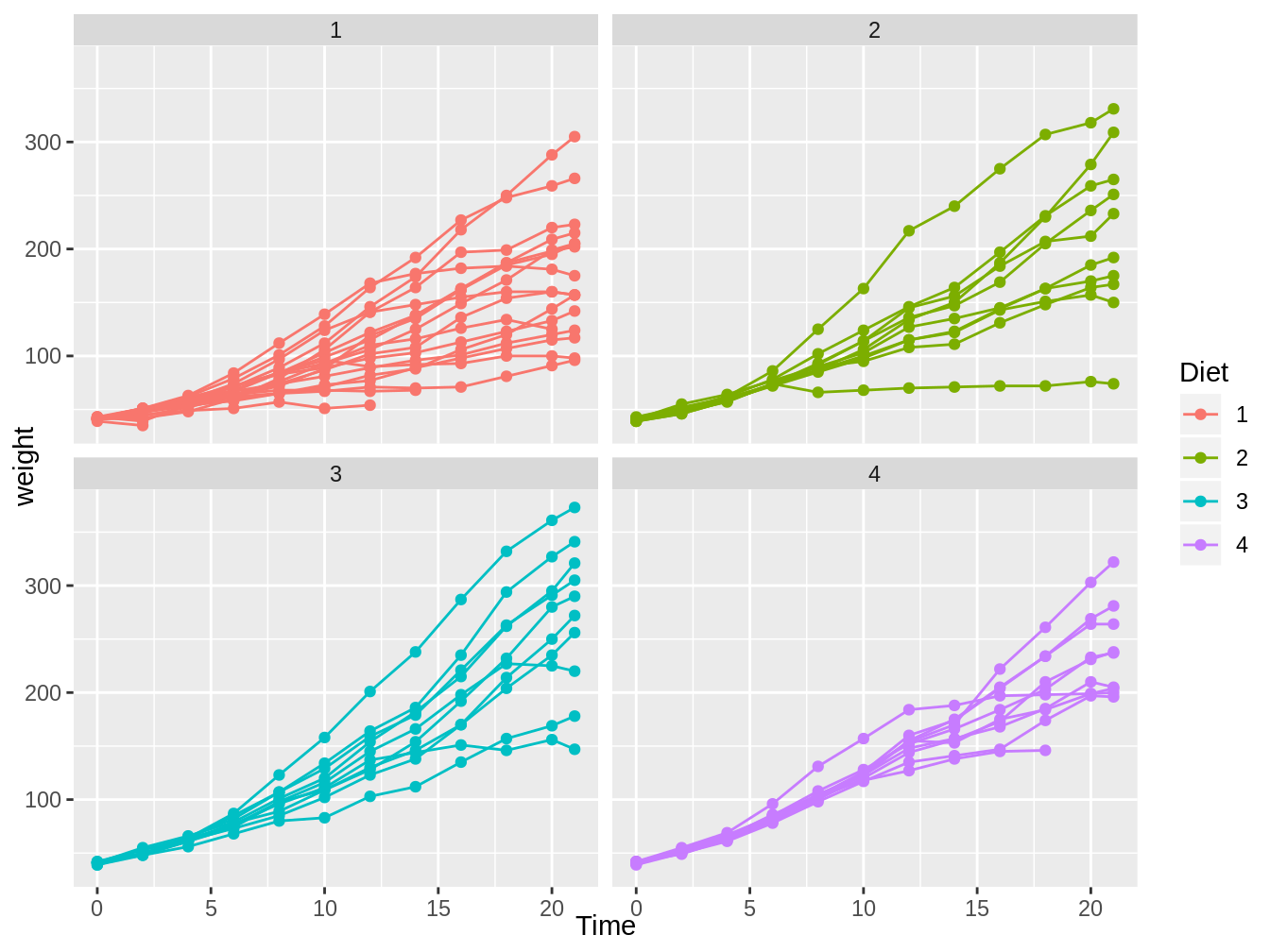
Figura 4.13: Gráfico en el cual vemos el peso de pollos en el tiempo, con colores y gráficos distintos según el tipo de dieta y con líneas para cada pollo individual.
Esta función siempre tendrá los mismos ejes y escala para todos los gráficos. Además, intentará siempre dejar la disposición de los gráficos de la forma más cuadrada posible, pero esto puede ser modificado agregando el argumento ncol y un número de columnas, así como vemos en la figura 4.14 y su código correspondiente.
ggplot(ChickWeight, aes(x = Time, y = weight)) + geom_point(aes(color = Diet)) +
geom_line(aes(color = Diet, group = Chick)) + facet_wrap(~Diet,
ncol = 3)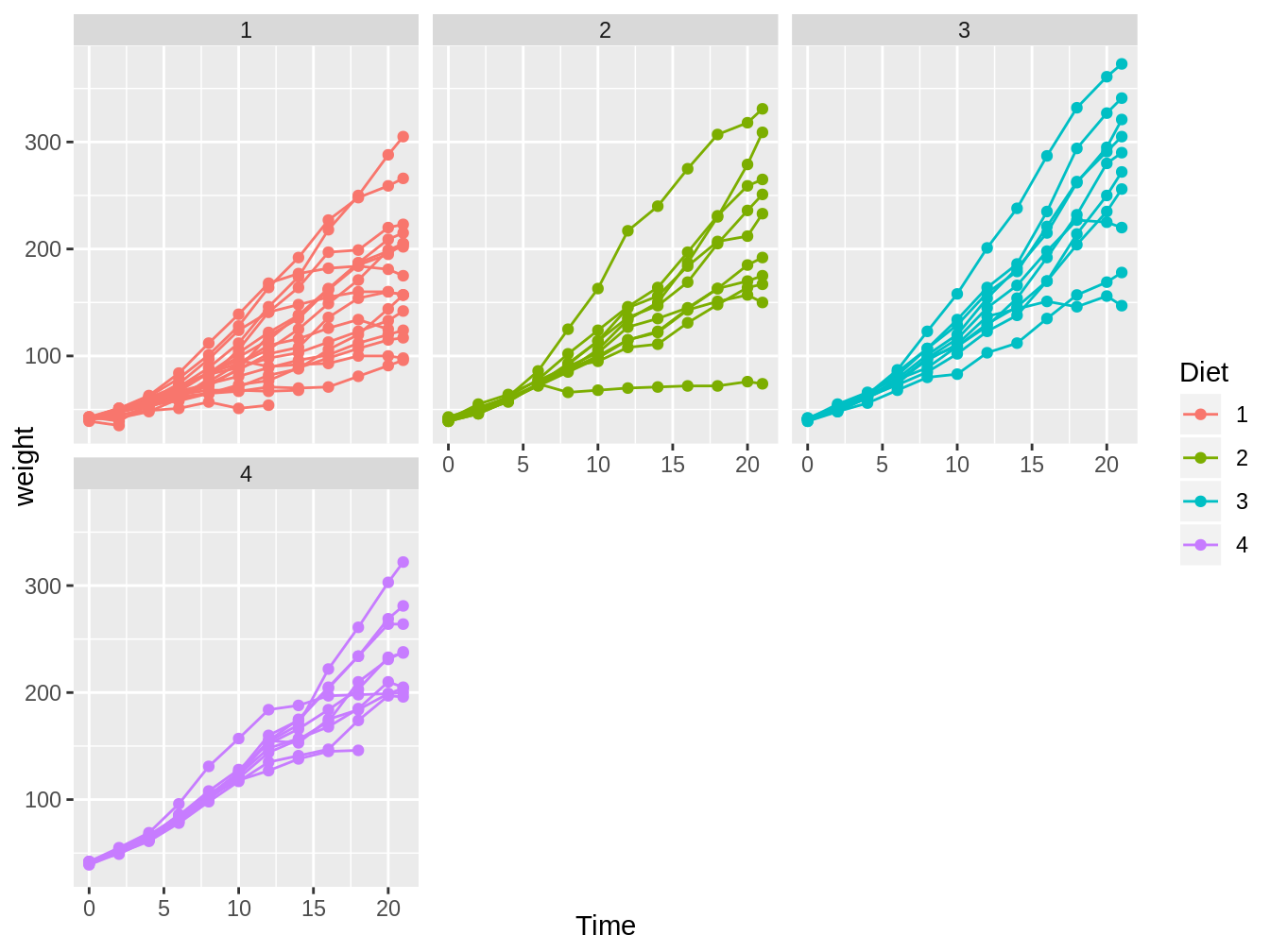
Figura 4.14: Gráfico en el cual vemos el peso de pollos en el tiempo, con colores y gráficos distintos según el tipo de dieta y con líneas para cada pollo individual.
Referencias
Anderson, Edgar. 1935. “The Irises of the Gaspe Peninsula.” Bulletin of the American Iris Society 59: 2–5.
Wickham, Hadley. 2016. Ggplot2: Elegant Graphics for Data Analysis. Springer-Verlag New York. http://ggplot2.org.