Capítulo 2 Investigación reproducible
2.1 Paquetes necesarios para este capítulo
Para este capítulo se necesita tener instalado los paquetes rmarkdown, knitr y stargazer
En este capítulo se explicará qué es investigación reproducible, cómo aplicarla usando github más los paquetes rmarkdown (Allaire et al. 2018) y knitr (Xie 2015). Además, se aprenderá a usar tablas usando knitr (Xie 2015) y stargazer (Hlavac 2018)
Recuerda que este libro es un apoyo para el curso BIO4022, puedes seguir la clase de este curso en este link, y en cuanto el video de la clase encontrarás un link aca.
2.2 Investigación reproducible
La investigación reproducible no es lo mismo que la investigación replicable. La replicabilidad implica que experimentos o estudios llevados a cabo en condiciones similares nos llevarán a conclusiones similares. La investigación reproducible implica que desde los mismos datos y/o el mismo código se generarán los mismos resultados.

Figura 2.1: Continuo de reproducibilidad (extraido de Peng 2011)
En la figura 2.1 vemos el continuo de reproducibilidad (Peng 2011). En este continuo tenemos el ejemplo de no reproducibilidad como una publicación sin código. Se pasa de menos a más reproducible por la publicación y el código que generó los resultados y gráficos; seguido por la publicación, el código y los datos que generan los resultados y gráficos; y por último código, datos y texto entrelazados de forma tal que al correr el código obtenemos exactamente la mismma publicación que leímos.
Esto tiene muchas ventajas, incluyendo el que es más fácil aplicar exactamente los mismos métodos a otra base de datos. Basta poner la nueva base de datos en el formato que tenía el autor de la primera publicación y podremos comparar los resultados.
Además en un momento en que la ciencia está basada cada vez más en bases de datos, se puede poner en el código la recolección y/o muestreo de datos.
2.3 Guardando nuestro proyecto en github
2.3.1 Que es github?
Github es una suerte de dropbox o google drive pensado para la investigación reproducible, en donde cada proyecto es un repositorio. La mayoría de los investigadores que trabajan en investigación reproducible dejan todo su trabajo documentado en sus repositorios, lo cual permite interactuar con otros autores.
2.3.2 creando un proyecto de github en RStudio
Para crear un proyecto en github presionamos start a project en la página inicial de nuestra cuenta, como vemos en la figura 2.2
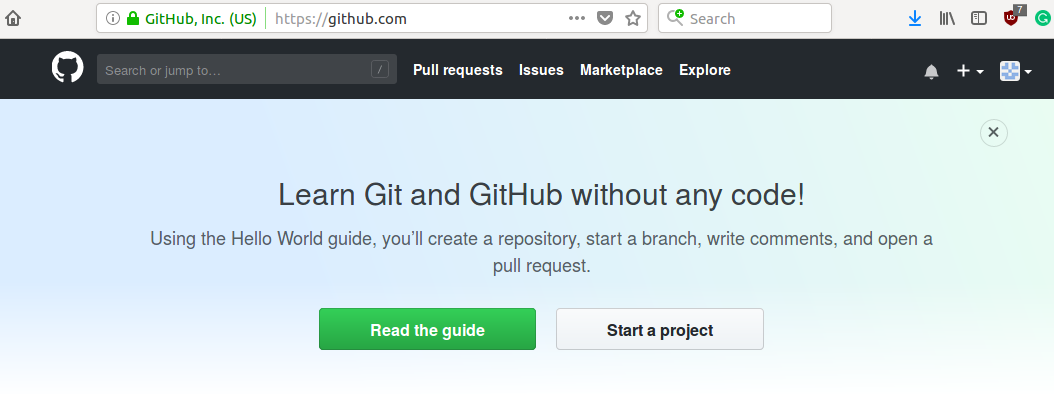
Figura 2.2: Para empezar un projecto en github, debes presionar Start a project en tu página de inicio
Luego se debe crear un nombre único, y sin cambiar nada más presiona create repository en el botón verde como vemos en la figura 2.3.
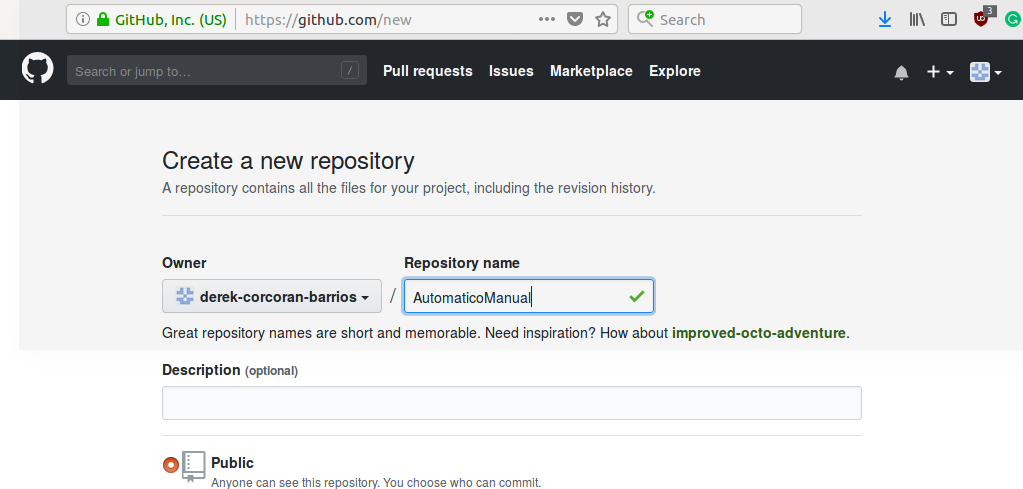
Figura 2.3: Crea el nombre de tu repositorio y apreta el boton create repository
Esto te llevará a una página donde aparecerá una url de tu nuevo repositorio como en la figura 2.4
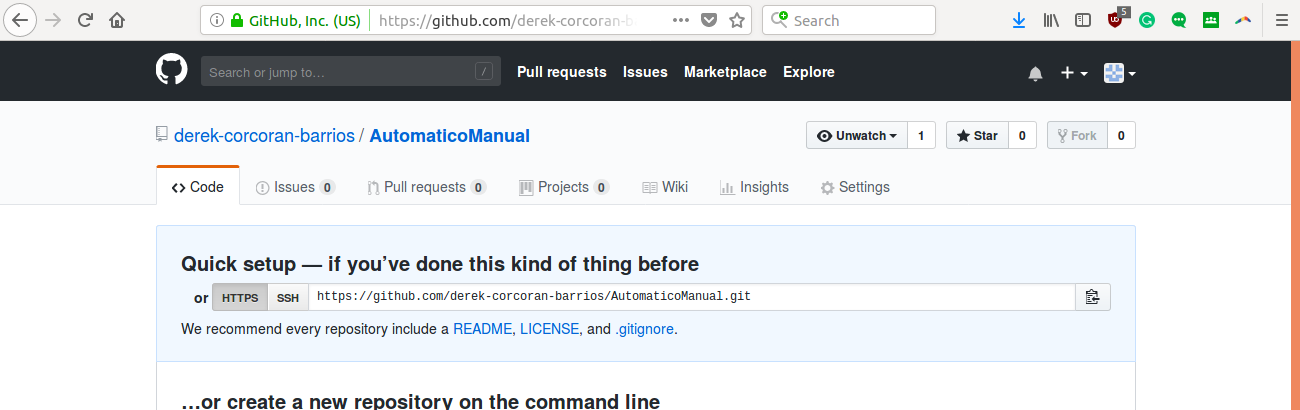
Figura 2.4: El contenido del cuadro en el cual dice ssh es la url de tu repisitorio
Para incorporar tu proyecto en tu repositorio, lo primero que debes hacer es generar un proyecto en RStudio. Para esto debes ir en el menú superior de Rstudio a File > New Project > Git como se ve en las figuras 2.5 y 2.5.
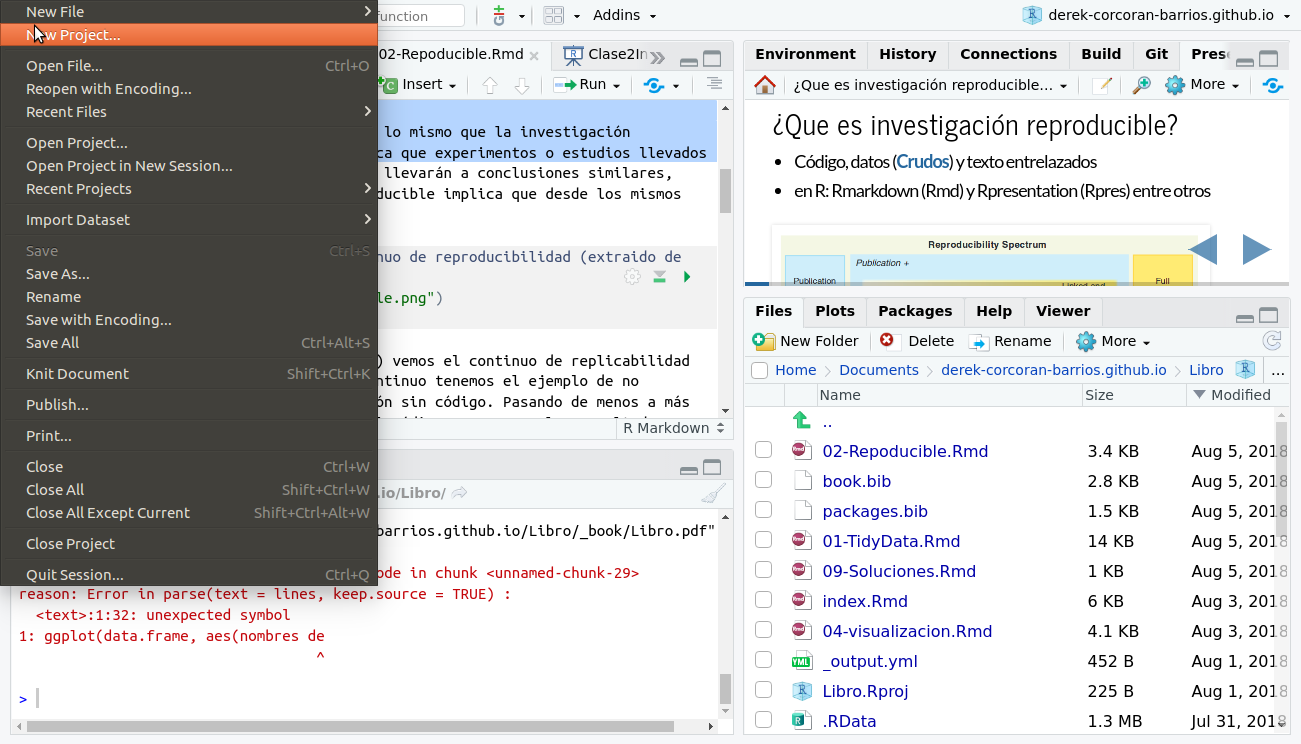
Figura 2.5: Menú para crear un proyecto nuevo
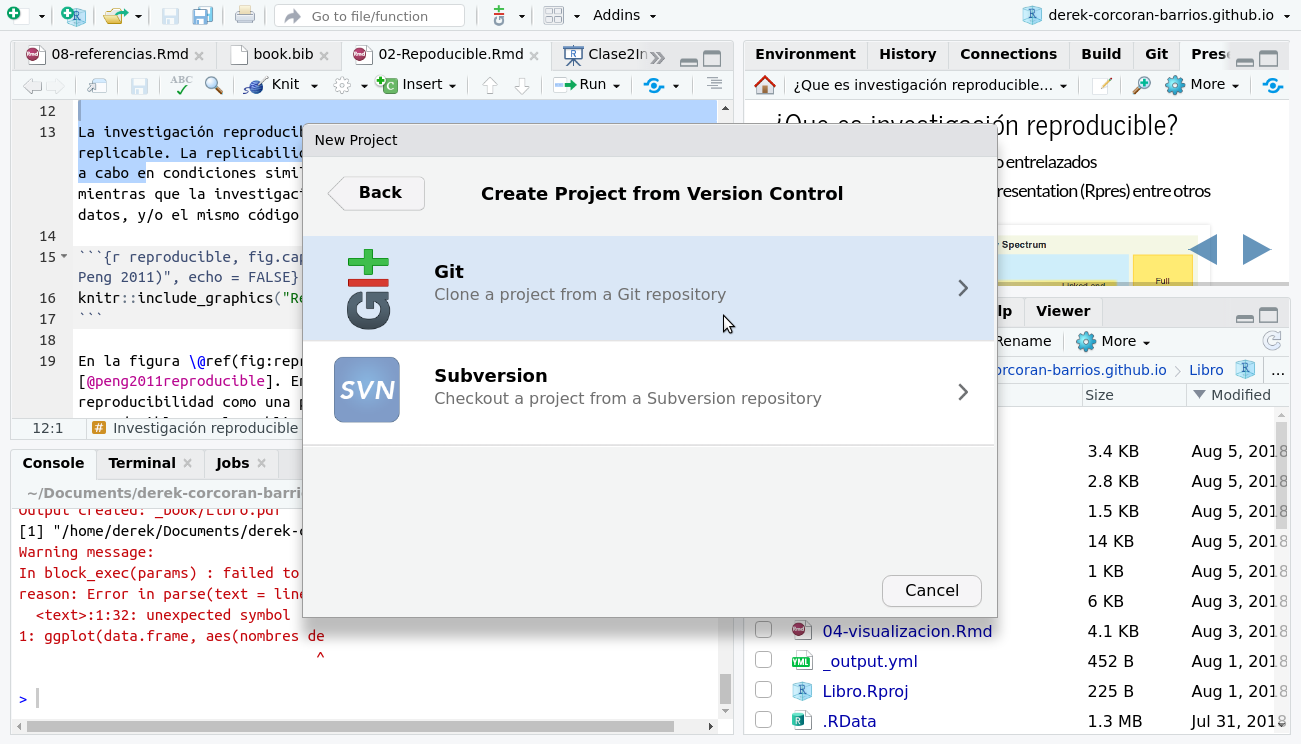
Figura 2.6: Seleccionar git dentro de las opciones
Luego seleccionar la ubicación del proyecto nuevo y pegar el url que aparece en la figura 2.4 en el espacio que dice Repository URL:, como muestra en la figura 2.7.
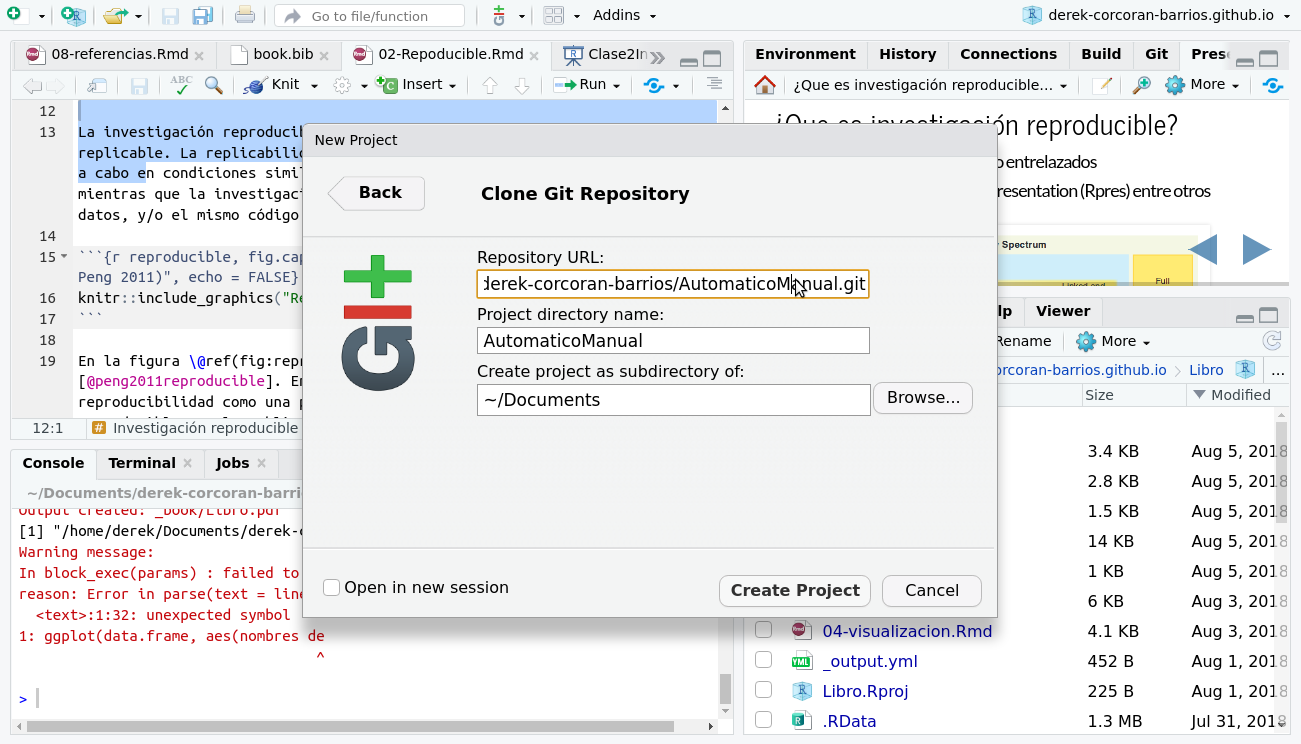
Figura 2.7: Pegar el url del repositorio en el cuadro de dialogo Repository URL:
Cuando tu proyecto de R ya este siguiendo los cambios en github, te aparecerá una pestaña git dentro de la ventana superior derecha de tu sesión de RStudio, tal como vemos en la figura 2.8
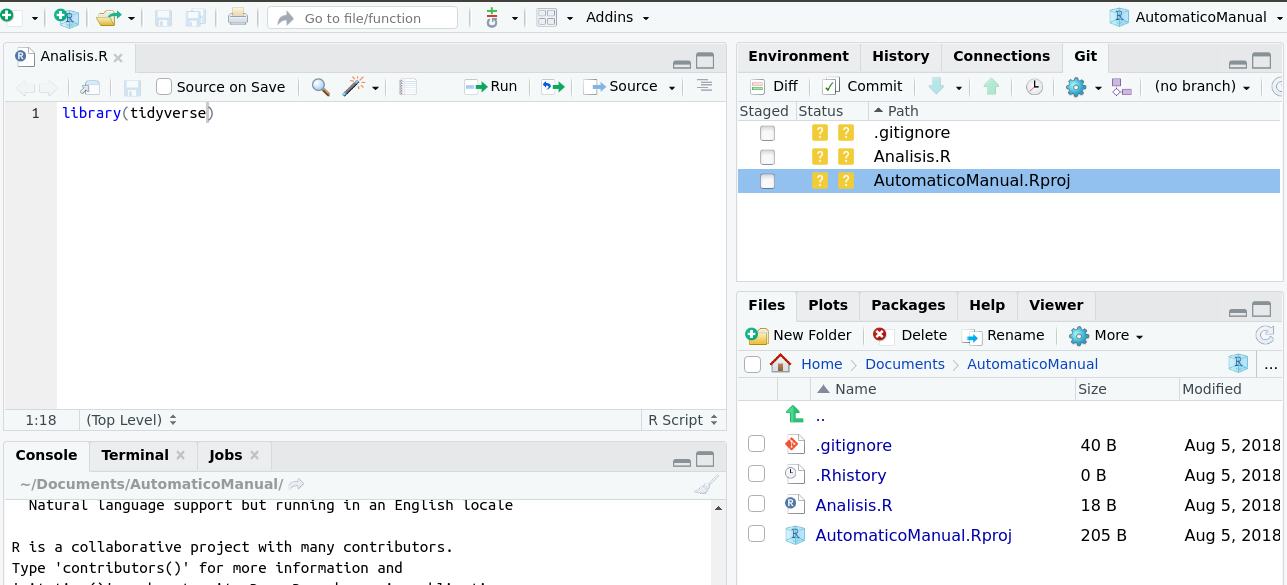
Figura 2.8: Al incluir tu repositorio en tu sesión de Rstudio, aparecera la pestaña git en la ventana superior derecha
2.3.3 Los tres principales pasos de un repositorio
Github es todo un mundo, existen muchas funciones y hay expertos en el uso de github. En este curso, nos enfocaremos en los 3 pasos principales de un repositorio: add, commit y push. Para entender bien qué significa cada uno de estos pasos, tenemos que entender que existen dos repositorios en todo momento: uno local (en tu computador) y otro remoto (en github.com). Los dos primeros pasos add y commit, solo generan cambios en tu repositorio local. Mientras que push, salva los cambios al repositorio remoto.
2.3.3.1 git add
Esta función es la que agrega archivos a tu repositorio local. Solo estos archivos serán guardados en github. Github tienen un límite de tamaño de repositorio de 1 GB y de archivos de 100 MB, ya que si bien te dan repositorios ilimitados, el espacio de cada uno no lo es, en particular en cuanto a bases de datos. Para adicionar un archivo a tu repositorio tan solo debes selecionar los archivos en la pestaña git. Al hacer eso una letra A verde aparecerá en vez de los dos signos de interrogación amarillos, como vemos en la figura 2.9. En este caso solo adicionamos al repositorio el archivo Analisis.r pero no el resto.
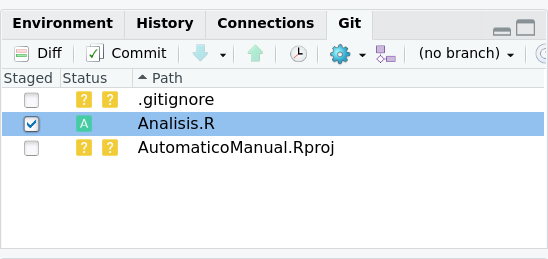
Figura 2.9: Al incluir tu repositorio en tu sesión de Rstudio, aparecera la pestaña git en la ventana superior derecha
2.3.3.2 git commit
Cuando ocupas el comando commit estas guardando los cambios de los archivos que adicionaste en tu repositorio local. Para hacer esto en Rstudio, en la misma pestaña de git, debes presionar el botón commit como vemos en la figura 2.10.
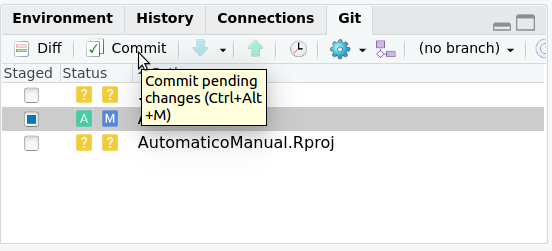
Figura 2.10: Para guardar los cambios en tu repositorio apretar commit en la pestaña git de la ventana superior derecha
Al presionar commit, se abrirá una ventana emergente, donde deberás escribir un mensaje que describa lo que guardarás. Una vez echo eso, presiona commit nuevamente en la ventana emergente como aparece en la figura 2.11.
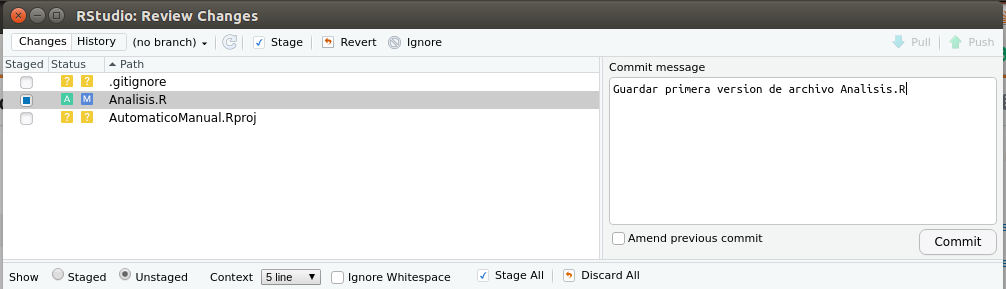
Figura 2.11: Escribir un mensaje que recuerde los cambios que hiciste en la ventana emergente
2.3.3.3 git push
Finalmente, push te permitirá guardar los cambios en tu repositorio remoto, lo cual asegura tus datos en la nube y además lo hace disponible a otros investigadores. Luego de apretar commit en la ventana emergente (figura 2.11), podemos presionar push en la flecha verde de la ventana emergente como se ve el a figura 2.12. Luego se nos pedirá nuestro nombre de usuario y contraseña, y ya podemos revisar que nuestro repositorio esta online entrando a nuestra sesión de github.
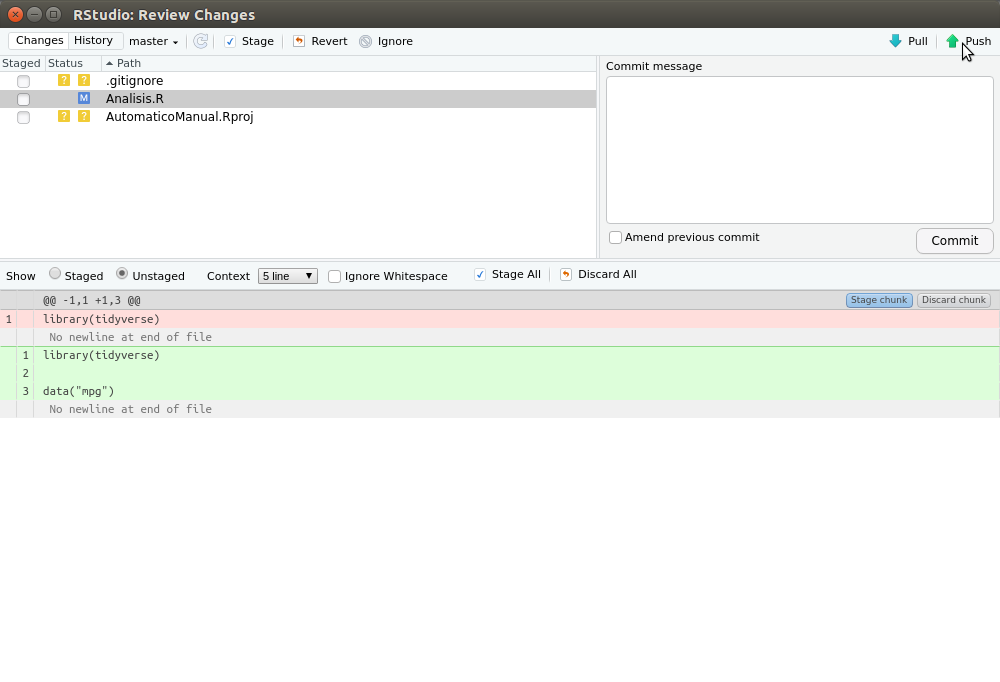
Figura 2.12: Para guardar en el repositorio remoto apretar push en la ventana emergente
2.4 Reproducibilidad en R
Existen varios paquetes que permiten que hagamos investigación reproducible en R, pero sin duda los más relevantes son rmarkdown y knitr. Ambos paquetes funcionan en conjunto cuando generamos un archivo Rmd (Rmarkdown), en el cual ocupamos al mismo tiempo texto, código de R y otros elementos para generar un documento word, pdf, página web, presentación y/o aplicación web (fig 2.13).

Figura 2.13: El objetivo de Rmarkdown es el unir código de r con texto y datos para generar un documento reproducible
2.4.1 Creando un Rmarkdown
Para crear un archivo Rmarkdown, simplemente ve a el menu File > New file > Rmarkdown y con eso habrás creado un nuevo archivo Rmd. Veremos algunos de los elementos más típicos de un archivo Rmarkdown.
2.4.1.1 Markdown
El markdown es la parte del archivo en que simplemente escribimos texto, aunque tiene algunos detalles para el formato como generar texto en negrita, cursiva, títulos y subtitulos.
Para hacer que un texto este en negrita, se debe poner entre dos asteriscos **negrita**, para que un texto aparezca en cursiva debe estar entre asteriscos *cursiva*. Otros ejemplos son los títulos de distintos niveles, los cuales se denotan con distintos números de #, así los siguientes 4 títulos o subtítulos:
subtitulo 1
subtítulo 2
subtítulo 3
subtítulo 4
se vería de la siguiente manera en el código
2.4.1.2 Chunks
Los chunks son una de las partes más importantes del un Rmarkdown. En estos es donde se agrega el código de R (u otros lenguajes de programación). Lo cual permíte que el producto de nuestro código no sea sólo un escrito con resultados pegados, sino que efectivamente generados en el mismo documento que nuestro escrito. La forma más fácil de agregar un chunk es apretando el botón de insert chunk en Rstudio, este boton se encuentra en la ventana superior izquierda de nuestra sesión de RStudio, tal como se muestra en la figura 2.14
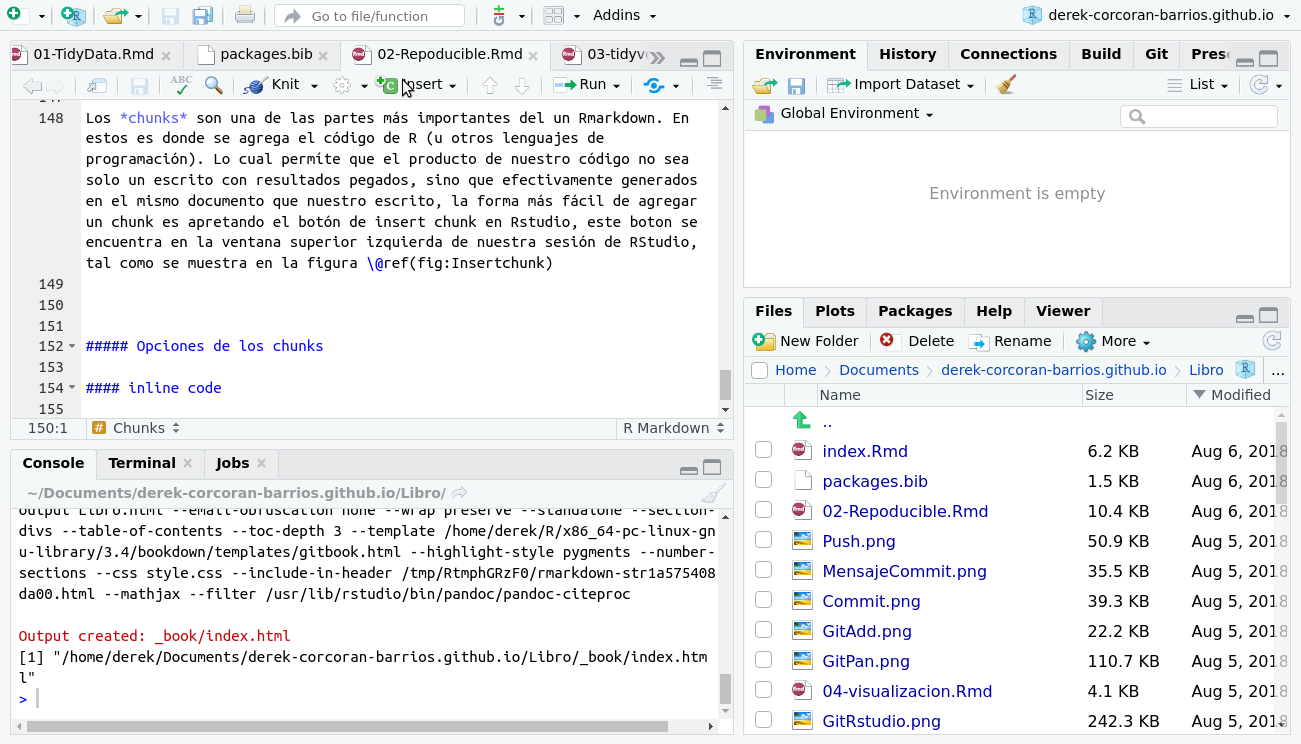
Figura 2.14: Al apretar el botón insert chunk, aparecera un espacio en el cuál insertar código
Al apretar este botón aparecera un espacio, ahí se puede agregar un código como el que aparece a continuación, y ver a continuación los resultados.
```{r}
library(tidyverse)
iris %>% group_by(Species) %>% summarize(Petal.Length = mean(Petal.length))
```## # A tibble: 3 x 2
## Species Petal.Length
## <fct> <dbl>
## 1 setosa 1.46
## 2 versicolor 4.26
## 3 virginica 5.552.4.1.2.1 Opciones de los chunks
Existen muchas opciones para los chunks, una documentación completa podemos encontrarle en el siguiente link, pero acá mostraremos los más comunes:
- echo = T o F muestro o no el código, respectivamente
- message = T o F muestra mensajes de paquetes, respectivamente
- warning = T o F muestra advertencias, respectivamente
- eval = T o F evaluar o no el código, respectivamente
- cache = T o F guarda o no el resultado, respectivamente
2.4.1.3 inline code
Los inline codes son útiles para agregar algún valor en el texto, como por ejemplo el valor de p o la media. Para usarlo, se debe poner un backtick (comilla simple hacia atrás), r, el código en cuestion y otro backtick como se ve a continuación `r R_código`. No podemos poner cualquier cosa en un inline code, ya que sólo puede generar vectores, lo cuál muchas veces requiere de mucha creatividad para lograr lo que queremos. Por ejemplo si se quisiera poner el promedio del largo del sépalo de la base da dato iris en un inline code pondríamos `r mean(iris$Sepal.Length)`, lo cual resultaría en 5.8433333. Como en un texto se vería extraño un número con 7 cifras significativas, querríamos usar ademas la función round, para que tenga 2 cifras significativas, para eso ponemos el siguiente inline code `r round(mean(iris$Sepal.Length),2)` que da como resultado 5.84. Esto se puede complejizar más aún si se quiere trabajar con una tabla resumen. Por ejemplo, si quisieramos listar el promedio del tamaño de sépalo usaríamos summarize de dplyr, pero esto nos daría como resultado un data.frame, el cual no aparece si intentamos hacer un inline code. Partamos por ver como se vería el código donde obtuvieramos la media del tamaño del sépalo.
El resultado de ese código lo veríamos 2.1
| Species | Mean |
|---|---|
| setosa | 5.006 |
| versicolor | 5.936 |
| virginica | 6.588 |
Para sacar de este data frame el vector de la media podríamos subsetearlo con el signo $. Entonces si queremos sacar como vector la columna Mean del data frame que creamos, haríamos lo siguiente `r (iris %>% group_by(Species) %>% summarize(Mean = mean(Sepal.Length)))$Mean`. Esto daría como resultado 5.006, 5.936, 6.588.
2.4.2 Ejercicios
2.4.2.1 Ejercicio 1
Usando la base de datos iris, crea un inline code que diga cuál es la media del largo del pétalo de la especie Iris virginica
La solución a este ejercicio se encuentra en el capítulo 8
2.4.2.2 Tablas en Rmarkdown
La función más típica para generar tablas en un archivo rmd es kable del paquete knitr, que en su forma más simple se incluye un dataframe como único argumento. Además de esto, podemos agregar algunos parámetros como caption, que nos permite poner un título a la tabla o row.names, que si se pone como se ve en el código (FALSE) no mostrará en la tabla los nombres de las filas, tal como se ve en la tabla 2.2.
DF <- iris %>% group_by(Species) %>% summarize_all(mean)
kable(DF, caption = "Promedio por especie de todas las variables de la base de datos iris.",
row.names = FALSE)| Species | Sepal.Length | Sepal.Width | Petal.Length | Petal.Width |
|---|---|---|---|---|
| setosa | 5.006 | 3.428 | 1.462 | 0.246 |
| versicolor | 5.936 | 2.770 | 4.260 | 1.326 |
| virginica | 6.588 | 2.974 | 5.552 | 2.026 |
Referencias
Allaire, JJ, Yihui Xie, Jonathan McPherson, Javier Luraschi, Kevin Ushey, Aron Atkins, Hadley Wickham, Joe Cheng, and Winston Chang. 2018. Rmarkdown: Dynamic Documents for R. https://CRAN.R-project.org/package=rmarkdown.
Hlavac, Marek. 2018. Stargazer: Well-Formatted Regression and Summary Statistics Tables. Bratislava, Slovakia: Central European Labour Studies Institute (CELSI). https://CRAN.R-project.org/package=stargazer.
Peng, Roger D. 2011. “Reproducible Research in Computational Science.” Science 334 (6060). American Association for the Advancement of Science: 1226–7.
Xie, Yihui. 2015. Dynamic Documents with R and Knitr. 2nd ed. Boca Raton, Florida: Chapman; Hall/CRC. http://yihui.name/knitr/.