Capitulo 5 Procesando los datos con tidyverse
Habitualmente tendremos que trabajar los datos para arreglarlos. Este proceso, que en castellano podría llamarse “limpieza” o procesado de datos, se conoce en inglés como data munging or data wrangling.
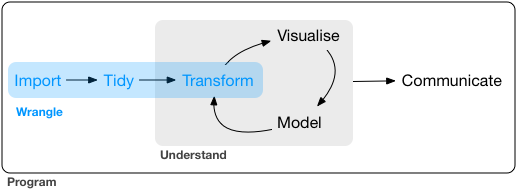
Procesar datos. Fuente: https://r4ds.had.co.nz/wrangle-intro.html
Se suele decir que el procesado/limpieza de los datos suele ocupar un 80% del tiempo de un análisis de datos. Quizás sea una cifra un poco exagerada, pero, en cualquier caso, es una tarea que ocupa tiempo y que puede llegar a ser tediosa y frustante si no se dispone de las herramientas adecuadas.
Aprenderemos a limpiar y transformar datos en R. Priorizaremos la nueva forma de hacer las cosas en R (o workflow) conocido como tidyverse. Es mucho más eficiente que R-Base.
5.1 Tidyverse
Con la palabra tidyverse se hace referencia a una nueva forma de afrontar el análisis de datos en R. Se hace uso de un grupo de paquetes que trabajan en armonía porque comparten ciertos principios, como por ejemplo, la forma de estructurar los datos. La mayoría de estos paquetes han sido desarrollados por (o al menos con la colaboración de Hadley Wickham, un “dios” de R).
5.2 The pipe (%>%)
El operador %>% es básico en el tidyverse, ya que permite encadenar llamadas a funciones para así realizar de forma sencilla transformaciones de datos complejas. En palabras, lo que hace este operador es pasar el elemento que está a su izquierda como un argumento de la función que tiene a la derecha.
Se entiende mejor con un ejemplo sencillo. Las siguientes dos instrucciones de R hacen exactamente lo mismo: permiten ver las 7 primeras filas de la base de datos iris.
Esto está bien, pero no supone ninguna ventaja, sólo es una forma distinta de ejecutar o llamar a una función. Lo importante para nosotros es que las pipes se pueden encadenar.
El operador pipe podemos leerlo como “entonces” y permite encadenar sucesivas llamadas a funciones. Por ejemplo:
La anterior línea de código R hace:
- coge los datos del dataframe
dfy selecciona (o filtra) las filas que cumplen que el valor deX1es mayor que 100, entonces (o después) - agrupa los datos por la variable
X2, entonces - calcula la media de
X3
En conjunto, encadenando las 3 funciones hemos seleccionado las filas de las personas que tienen un salario (X1) mayor de 100, agrupado las filas que cumplen esta condición por genero (X2) y calculado la media para cada uno de los grupos; es decir, hemos calculado la media salarial para hombres y mujeres, teniendo en cuenta sólo a los individuos con salario superior a 100.
5.3 Principales pkgs del tidyverse
Los principales packages del tidyverse son:
- readr: para importar datos
- tidyr: para convertir los datos a tidy data
- dplyr: para manipular datos
- ggplot2: para hacer gráficos
- purrr: para functional programming ( no nos interesa)
Todos estos pkgs se han agrupado en un solo package, el tidyverse package. por lo tanto solamente debemos instalar este último. ¿Recordamos cómo se hace esto?
5.4 Tidy data (tidyr)
5.4.1 ¿Qué son los tidy data?
De forma sencilla, tidy data son simplemente datos organizados de una determinada manera y es justo la manera a la nos estamos familiarizando.
La mayoría de datos de trabajo se ajustan a la categoría de datos tabulares; es decir, están organizados en filas y columnas. En R este tipo de datos se almacenan en data.frames (o tibbles). En esencia, un data.frame será tidy si cada columna es una variable y cada fila es una unidad de análisis (persona, país, región etc…); es decir, cada celda contiene el valor de una variable para una unidad de análisis.

Tidy data. Fuente: http://r4ds.had.co.nz/tidy-data.html
5.4.1.1 Un ejemplo de datos (no tidy)
| Sepal.Length | 5.1 | 4.9 | 4.7 | 4.6 | 5.0 | 5.4 |
| Sepal.Width | 3.5 | 3.0 | 3.2 | 3.1 | 3.6 | 3.9 |
| Petal.Length | 1.4 | 1.4 | 1.3 | 1.5 | 1.4 | 1.7 |
| Petal.Width | 0.2 | 0.2 | 0.2 | 0.2 | 0.2 | 0.4 |
| Species | setosa | setosa | setosa | setosa | setosa | setosa |
5.4.1.2 Un ejemplo de datos (tidy pero wide)
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|
| 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| 5.4 | 3.9 | 1.7 | 0.4 | setosa |
También es un formato fácil de entender por nosotros, pero ¿son tidy? Si, pero…
Es el formato al que estamos más acostumbrados (individuos o registros en filas y “variables” en columnas).
En jerga del tidyverse este formato de datos es wide (o ancho).
Podemos trabajar tranquilamente con el anterior formato, pero, si queremos “sacar todo el provecho” a tidyverse es mejor tener los datos en formato “long”.
| Species | flower_att | measurement |
|---|---|---|
| setosa | Sepal.Length | 5.1 |
| setosa | Sepal.Length | 4.9 |
| setosa | Sepal.Length | 4.7 |
| setosa | Sepal.Width | 3.5 |
| setosa | Sepal.Width | 3.0 |
| setosa | Sepal.Width | 3.2 |
| setosa | Petal.Length | 1.4 |
| setosa | Petal.Length | 1.4 |
| setosa | Petal.Length | 1.3 |
| setosa | Petal.Width | 0.2 |
| setosa | Petal.Width | 0.2 |
| setosa | Petal.Width | 0.2 |
Siendo sincero tampoco es muy necesario esto. Pero sí que es importante saber crear formatos “long” para la visualización de datos usando el potente paquete ggplot2 y demás paquetes contruidos sobre él.
5.4.2 gather() y spread()
Estas son funciones para pasar de “wide” a “long” y viceversa. El formato “wide”, como hemos visto, es el formato típico (filas idividuos y columnas variables). Personalmente el formato “long” solo lo uso para la visualización de datos con ggplot2 y similares, pero bien es cierto que es el formato más eficiente para tidyverse. Para pasar de uno a otro usaremos las funciones gather() y spread() del paquete tidyr.
El siguiente diagrama ilustra el uso de gather() y spread().
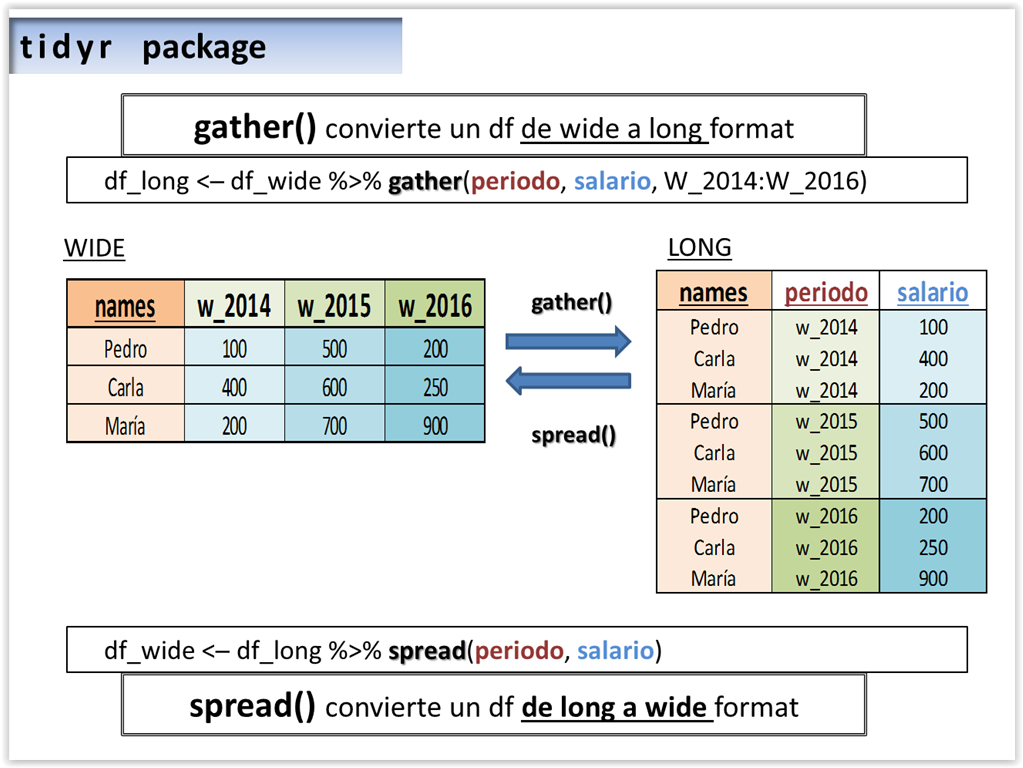
Fuente: https://tinyurl.com/yuf8k5dm
5.4.2.1 De wide a long format con gather()
La función gather() convierte dataframes de wide a long format.
library(tidyr)
head(iris)
## # A tibble: 6 x 5
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## <dbl> <dbl> <dbl> <dbl> <chr>
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosa
iris$ID = 1:nrow(iris)
#- la función gather() transforma los datos de formato ancho(wide) a formato largo(long)
iris_long <- iris %>% gather(key = "atributo", value = "medida", -Species,-ID)
head(iris_long)
## # A tibble: 6 x 4
## Species ID atributo medida
## <chr> <int> <chr> <dbl>
## 1 setosa 1 Sepal.Length 5.1
## 2 setosa 2 Sepal.Length 4.9
## 3 setosa 3 Sepal.Length 4.7
## 4 setosa 4 Sepal.Length 4.6
## 5 setosa 5 Sepal.Length 5
## 6 setosa 6 Sepal.Length 5.4
### o lo mismo
iris %>% gather(key = "atributo", value = "medida", Sepal.Length,Sepal.Width,Petal.Length,Petal.Width) %>%head()
## # A tibble: 6 x 4
## Species ID atributo medida
## <chr> <int> <chr> <dbl>
## 1 setosa 1 Sepal.Length 5.1
## 2 setosa 2 Sepal.Length 4.9
## 3 setosa 3 Sepal.Length 4.7
## 4 setosa 4 Sepal.Length 4.6
## 5 setosa 5 Sepal.Length 5
## 6 setosa 6 Sepal.Length 5.45.4.2.2 De long a wide format con spread()
Pasar pasar de “long” a “wide”, tenemos la función spread().
iris_wide <- iris_long %>% spread(key=atributo,value = medida)
head(iris_wide)
## # A tibble: 6 x 6
## Species ID Petal.Length Petal.Width Sepal.Length Sepal.Width
## <chr> <int> <dbl> <dbl> <dbl> <dbl>
## 1 setosa 1 1.4 0.2 5.1 3.5
## 2 setosa 2 1.4 0.2 4.9 3
## 3 setosa 3 1.3 0.2 4.7 3.2
## 4 setosa 4 1.5 0.2 4.6 3.1
## 5 setosa 5 1.4 0.2 5 3.6
## 6 setosa 6 1.7 0.4 5.4 3.95.5 package dplyr
dplyr es un package que permite manipular datos de forma intuitiva y super efectiva.
Tiene 7 funciones o verbos principales.
filter(): permite seleccionar filas (que cumplen una o varias condiciones).arrange(): reordena las filas.rename(): cambia los nombres de las columnas (variables).select(): selecciona columnas (variables).mutate(): crea nuevas variables.summarise(): resume (colapsa) unos cuantos valores a uno sólo. Por ejemplo, calcula la media, moda, etc… de un conjunto de valores.group_by(): permite agrupar filas en función de una o varias condiciones.
Cada uno de ellos hace “una sola cosa”, así que para realizar transformaciones complejas hay que ir concatenando instrucciones sencillas. Esto se hace con el operador pipe %>%.
Veamoslas una a una. Veremos sólo algunos ejemplos. Y vamos practicando.
5.5.1 filter()
Esta función se utiliza para seleccionar filas de un dataframe (df). Se seleccionan las filas que cumplen una determinada condición o criterio lógico. Por ejemplo:
#- vamos a trabajar con los datos del package gapminder. He seguido el curso de Jenny Bryan. Fuente: https://stat545.com/dplyr-intro.html
if(!require(gapminder)) {
install.packages("gapminder")
library(gapminder)}
df <- gapminderEchemos un vistazo a la base de datos.
Y empezamos seleccionando las filas que cumplen determinados criterios:
df <- gapminder
#- Observaciones de España (country == "Spain")
a <- df %>% filter(country == "Spain")
#- filas con valores de "lifeExp" < 29
a <- df %>% filter(lifeExp < 29)
#- filas con valores de "lifeExp" entre [29, 32]
a <- df %>% filter(lifeExp >= 29 , lifeExp <= 32)
a <- df %>% filter(lifeExp >= 29 & lifeExp <= 32)
a <- df %>% filter(between(lifeExp, 29, 32))
#- observaciones de países de Africa con lifeExp > 32
a <- df %>% filter(lifeExp > 72 & continent == "Africa")
#- observaciones de países de Africa o Asia con lifeExp > 32
a <- df %>% filter(lifeExp > 72 & continent %in% c("Africa", "Asia") )
a <- df %>% filter(lifeExp > 72 & ! continent %in% c("Africa", "Asia") )
a <- df %>% filter(lifeExp > 72 & (continent == "Africa" | continent == "Asia") ) La función filter() tiene muchas más posibilidades. Ya las iremos viendo. Y google es la hostia para esto :).
5.5.1.1 slice() y top_n() tambien pueden ser muy útiles para seleccionar filas
## Selecting by gdpPercap#- selecciona las observaciones de la 12 a 13 Y de la 44 a 46, Y las 4 últimas
aa <- df %>% slice( c(12:14, 44:46, (n()-4):n()) )
## Pero, qué hemos hecho?
#- Pista: igual os ayuda crear una columna con el índice de rows
aa <- df %>% mutate(index = 1:n()) # fijaros que para ello hemos usado una nueva función `mutate()`
aa <- aa %>% slice( c(12:14, 44:46, (n()-4):n()) )5.5.2 arrange()
Esta función se utiliza para reordenar las filas de un dataframe.
df <- gapminder
#- ordena las filas de MENOR a mayor según los valores de la variable lifeExp
a <- df %>% arrange(lifeExp)
#- ordena las filas de MAYOR a menor según los valores de la variable lifeExp
a <- df %>% arrange(desc(lifeExp))
#- ordena las filas de MENOR a mayor según los valores de la variable lifeExp.
#- Si hay empates se resuelve con la variable "pop"
a <- df %>% arrange(lifeExp, pop)5.5.3 rename()
Esta función permite cambiar los nombres de las columnas (o variables si los datos son tidy).
#- cambia los nombres de lifeExp y gdpPercap a life_exp y gdp_percap
df %>% rename(life_exp = lifeExp, gdp_percap = gdpPercap)
## # A tibble: 1,704 x 6
## country continent year life_exp pop gdp_percap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afghanistan Asia 1952 28.8 8425333 779.
## 2 Afghanistan Asia 1957 30.3 9240934 821.
## 3 Afghanistan Asia 1962 32.0 10267083 853.
## 4 Afghanistan Asia 1967 34.0 11537966 836.
## 5 Afghanistan Asia 1972 36.1 13079460 740.
## 6 Afghanistan Asia 1977 38.4 14880372 786.
## 7 Afghanistan Asia 1982 39.9 12881816 978.
## 8 Afghanistan Asia 1987 40.8 13867957 852.
## 9 Afghanistan Asia 1992 41.7 16317921 649.
## 10 Afghanistan Asia 1997 41.8 22227415 635.
## # ... with 1,694 more rows
#-(!!) la función names() es muy útil. Tb setNames() y set_names()
aa <- df
names(aa) <- names(aa) %>% toupper
names(aa) <- names(aa) %>% tolower
names(aa) <- c("var_01", "var_02", "var_03", "var_04", "var_05" , "var_06")
names(aa) <- paste0("Var_", 1:6)
names(aa) <- paste0("Lag_", formatC(1:6, width = 2, flag = "0")) La función rename() es útil pero enseguida veremos que la siguiente función select() también permite renombrar las columnas, e incluso reordenar la posición de estas.
5.5.4 select()
Esta función sirve para seleccionar columnas (o variables si el fichero es tidy).
Seleccionar variables por nombre:
df <- gapminder
#- Se lee como: “coge el df gapminder, y selecciona las variables year y lifeExp”
aa <- df %>% select(year, lifeExp)
aa <- df %>% select(c(year, lifeExp))5.5.4.1 quitar variables
Seleccionamos todas las variables del df gapminder excepto “year”:
5.5.4.2 seleccionar por posicion
Seleccionamos las variables del df gapminder siguientes: de la primera a la tercera y también la quinta (mejor seleccionarlas por nombre!).
5.5.4.3 renombrando y reordenando columnas con select()
Lo que sí vamos a ver son 2 posibilidades de select() que son muy útiles. Con select() podemos: renombrar y reordenar las columnas:
5.5.5 mutate()
Esta función sirve para crear nuevas variables (columnas). Es muy útil en análisis de datos.
Creamos por ejemplo la variable: GDP = pop*gdpperCap
5.5.6 summarise()
Esta función sirve para resumir (o “colapsar filas”). Coge un grupo de valores como input y devuelve un solo valor; por ejemplo, haya la media aritmética (o el mínimo, o el máximo …) de un grupo de valores.
Obtengamos determinados estadísticos de una variable. Para esto no nos hace falta dplyr pero conviene ir habituándose a su sintaxis.
df <- gapminder
#- retornará un único valor: la media global de la variable "lifeExp"
aa <- df %>% summarise(media = mean(lifeExp))
#- retornará un único valor: el número de filas
aa <- df %>% summarise(NN = n()) #- retornará un único valor: el número de filas
#- retornará un único valor: la desviación típica de la variable "lifeExp"
aa <- df %>% summarise(desviacion_tipica = sd(lifeExp))
#- retornará un único valor: el máximo de la variable "pop"
aa <- df %>% summarise(max(pop))
#- retornará 2 valores: la media y desviación típica de la variable "lifeExp"
aa <- df %>% summarise(mean(lifeExp), sd(lifeExp))
#- retornará 2 valores: las medias de "lifeExp" y "gdpPercap", respectivamente
aa <- df %>% summarise(mean(lifeExp), mean(gdpPercap)) También podemos calcular determinados estadísticos de todas las variables del df con summarise_all()
#- media de cada una de las 6 variables. las 2 primeras son texto (en realidad son factores, lo puedes ver con str(df))
aa <- df %>% summarise_all(mean) ## Warning in mean.default(country): argument is not numeric or logical: returning NA## Warning in mean.default(continent): argument is not numeric or logical: returning NA5.5.7 group_by()
Con esta función ya empezaremos a ver la potencia de dplyr :). En análisis de datos, muchas operaciones (media etc..) queremos calcularlas para distintos grupos (hombre, mujer,…). group_by() permite hacerlo.
group_by() coge un data.frame y lo convierte en un data.frame agrupado. En ese nuevo data.frame agrupado, las operaciones que hagamos con summarise() se harán por separado para cada uno de los grupos que hayamos definido. Ahora lo vemos.
Si, por ejemplo, agrupamos un data.frame por países, al ejecutar summarise(), nos retornará una fila con el resultado para cada país.
#- cogemos df y lo agrupamos por grupos definidos por la variable "continent"; o sea, habrá 5 grupos
#- después con summarise() calcularemos el nº de observaciones en cada continente o grupo; es decir, nos retornará un df con una fila por cada continente
aa <- df %>%
group_by(continent) %>%
summarise(NN = n())
aa
## # A tibble: 5 x 2
## continent NN
## <fct> <int>
## 1 Africa 624
## 2 Americas 300
## 3 Asia 396
## 4 Europe 360
## 5 Oceania 24¿Y cuantos países hay en la base de datos?
#- cogemos df y lo agrupamos por "continent",
#- después calculamos 2 cosas: el número de observaciones o rows
#- y el número de países en cada continente (NN_countries)
aa <- df %>% group_by(continent) %>%
summarize(NN = n(), NN_countries = n_distinct(country))
aa
## # A tibble: 5 x 3
## continent NN NN_countries
## <fct> <int> <int>
## 1 Africa 624 52
## 2 Americas 300 25
## 3 Asia 396 33
## 4 Europe 360 30
## 5 Oceania 24 2Calculemos la esperanza de vida media por continente
#- cogemos df y lo agrupamos por "continent", después calculamos la media de "lifeExp"
aa <- df %>% group_by(continent) %>%
summarize(mean(lifeExp))
aa
## # A tibble: 5 x 2
## continent `mean(lifeExp)`
## <fct> <dbl>
## 1 Africa 48.9
## 2 Americas 64.7
## 3 Asia 60.1
## 4 Europe 71.9
## 5 Oceania 74.3Calculemos la esperanza de vida media por continente en el primer periodo (1952)
#- cogemos df y filtramos para quedarnos con las observaciones de 1952
#- después lo agrupamos por "continent",
#- después calculamos la media de "lifeExp"
aa <- df %>% filter(year == "1952") %>% ## también podemos usar min(year) en vez de 1952
group_by(continent) %>%
summarize(mean(lifeExp))
aa
## # A tibble: 5 x 2
## continent `mean(lifeExp)`
## <fct> <dbl>
## 1 Africa 39.1
## 2 Americas 53.3
## 3 Asia 46.3
## 4 Europe 64.4
## 5 Oceania 69.3Se pueden calcular varios estadísticos a la vez
aa <- df %>% filter(year %in% c(1952, 2007)) %>%
group_by(continent, year) %>%
summarize(mean(lifeExp), mean(gdpPercap))
aa
## # A tibble: 10 x 4
## # Groups: continent [5]
## continent year `mean(lifeExp)` `mean(gdpPercap)`
## <fct> <int> <dbl> <dbl>
## 1 Africa 1952 39.1 1253.
## 2 Africa 2007 54.8 3089.
## 3 Americas 1952 53.3 4079.
## 4 Americas 2007 73.6 11003.
## 5 Asia 1952 46.3 5195.
## 6 Asia 2007 70.7 12473.
## 7 Europe 1952 64.4 5661.
## 8 Europe 2007 77.6 25054.
## 9 Oceania 1952 69.3 10298.
## 10 Oceania 2007 80.7 29810.5.5.7.1 summarise_at()
Y un paso más allá con summarise_at()
#- cogemos df y lo agrupamos por "continent" y "year",
# después calculamos la media y mediana de "lifeExp" y de "gdpPercap"
aa <- df %>%
filter(year %in% c(1952, 2007)) %>%
group_by(continent, year) %>%
summarise_at(vars(lifeExp, gdpPercap), funs(mean, median) )
aa
## # A tibble: 10 x 6
## # Groups: continent [5]
## continent year lifeExp_mean gdpPercap_mean lifeExp_median gdpPercap_median
## <fct> <int> <dbl> <dbl> <dbl> <dbl>
## 1 Africa 1952 39.1 1253. 38.8 987.
## 2 Africa 2007 54.8 3089. 52.9 1452.
## 3 Americas 1952 53.3 4079. 54.7 3048.
## 4 Americas 2007 73.6 11003. 72.9 8948.
## 5 Asia 1952 46.3 5195. 44.9 1207.
## 6 Asia 2007 70.7 12473. 72.4 4471.
## 7 Europe 1952 64.4 5661. 65.9 5142.
## 8 Europe 2007 77.6 25054. 78.6 28054.
## 9 Oceania 1952 69.3 10298. 69.3 10298.
## 10 Oceania 2007 80.7 29810. 80.7 29810.5.5.7.2 summarise_if()
Y más:
#- cogemos df y lo agrupamos por "continent" y "year",
# después calculamos la media y mediana de las variables que sean numéricas
aa <- df %>%
filter(year %in% c(1952, 2007)) %>%
group_by(continent, year) %>%
summarise_if(is.numeric, funs(mean, median) )
aa
## # A tibble: 10 x 8
## # Groups: continent [5]
## continent year lifeExp_mean pop_mean gdpPercap_mean lifeExp_median pop_median gdpPercap_median
## <fct> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Africa 1952 39.1 4570010. 1253. 38.8 2668124. 987.
## 2 Africa 2007 54.8 17875763. 3089. 52.9 10093310. 1452.
## 3 Americas 1952 53.3 13806098. 4079. 54.7 3146381 3048.
## 4 Americas 2007 73.6 35954847. 11003. 72.9 9319622 8948.
## 5 Asia 1952 46.3 42283556. 5195. 44.9 7982342 1207.
## 6 Asia 2007 70.7 115513752. 12473. 72.4 24821286 4471.
## 7 Europe 1952 64.4 13937362. 5661. 65.9 7199786. 5142.
## 8 Europe 2007 77.6 19536618. 25054. 78.6 9493598 28054.
## 9 Oceania 1952 69.3 5343003 10298. 69.3 5343003 10298.
## 10 Oceania 2007 80.7 12274974. 29810. 80.7 12274974. 29810.5.5.7.3 relocate
Para cambiar la posición de las columnas, podemos usar la función relocate
df %>% relocate(pop, .after = continent) %>% slice(1)
## # A tibble: 1 x 6
## country continent pop year lifeExp gdpPercap
## <fct> <fct> <int> <int> <dbl> <dbl>
## 1 Afghanistan Asia 8425333 1952 28.8 779.
df %>% relocate(pop, .before = continent) %>% slice(1)
## # A tibble: 1 x 6
## country pop continent year lifeExp gdpPercap
## <fct> <int> <fct> <int> <dbl> <dbl>
## 1 Afghanistan 8425333 Asia 1952 28.8 779.5.5.7.4 case_when
Una función auxiliar que es muy útil al utilizarla junto a mutate y crear nuevas variables en base a unas condiciones es: case_when().
Podemos concatenar condiciones con una , y ~ asigna el valor que queremos darle en cada caso. En caso que queramos decir un “y en todo el resto hazme tal”, usamos un TRUE ~ ...
## [1] "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "medio" "medio"
## [18] "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio"
## [35] "medio" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande"## [1] "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "txiki" "medio" "medio"
## [18] "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio" "medio"
## [35] "medio" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande" "grande"5.5.8 Unos ejercicios
¿Qué continente ha tenido la esperanza de vida más alta en 1952 y en 2007?
aa <- df %>%
filter(year %in% c(1952, 2007)) %>%
group_by(continent, year) %>%
summarize(mean(lifeExp)) %>% ungroup() ## con este argumento ungroup() deshacemos los grupos creados
aa
## # A tibble: 10 x 3
## continent year `mean(lifeExp)`
## <fct> <int> <dbl>
## 1 Africa 1952 39.1
## 2 Africa 2007 54.8
## 3 Americas 1952 53.3
## 4 Americas 2007 73.6
## 5 Asia 1952 46.3
## 6 Asia 2007 70.7
## 7 Europe 1952 64.4
## 8 Europe 2007 77.6
## 9 Oceania 1952 69.3
## 10 Oceania 2007 80.7¿Cómo ha evolucionado la esperanza de vida en Spain lustro a lustro?
#- variación de lifeExp en Spain lustro a lustro
aa <- df %>%
filter(country == "Spain" ) %>%
select(year, lifeExp) %>%
mutate(lifeExp_gain_cada_lustro = lifeExp - lag(lifeExp))
aa
## # A tibble: 12 x 3
## year lifeExp lifeExp_gain_cada_lustro
## <int> <dbl> <dbl>
## 1 1952 64.9 NA
## 2 1957 66.7 1.72
## 3 1962 69.7 3.03
## 4 1967 71.4 1.75
## 5 1972 73.1 1.62
## 6 1977 74.4 1.33
## 7 1982 76.3 1.91
## 8 1987 76.9 0.6
## 9 1992 77.6 0.670
## 10 1997 78.8 1.2
## 11 2002 79.8 1.01
## 12 2007 80.9 1.16¿Y la variación acumulada?
#- ganancia acumulada
aa <- df %>%
filter(country == "Spain") %>%
select(year, lifeExp) %>%
mutate(lifeExp_gain_cada_lustro = lifeExp - lag(lifeExp)) %>%
#--- 2 filas nuevas
mutate(lifeExp_gain_cada_lustro2 = if_else(is.na(lifeExp_gain_cada_lustro), 0, lifeExp_gain_cada_lustro)) %>%
mutate(lifeExp_gain_acumulado = cumsum(lifeExp_gain_cada_lustro)) Me han hecho falta 2 lineas extra, porque la primera observación de “lifeExp_gain_cada_lustro” es un NA y eso hacía que la función cumsum() no funcionase.
Otra forma de hacer lo mismo sería, y más fácil:
#- ganancia acumulada (otra forma de hacer lo mismo)
aa <- df %>%
group_by(country) %>%
select(country, year, lifeExp) %>%
mutate(lifeExp_gain_acumulada = lifeExp - lifeExp[1]) %>%
filter(country == "Spain")Obtener, para cada periodo, los (3) países de Asia con mayor lifeExp.
aa <- df %>%
filter(continent == "Asia") %>%
select(year, country, lifeExp) %>%
group_by(year) %>%
top_n(3, lifeExp) %>%
arrange(year)Para obtener los 3 países con menor lifeExp sólo tendríamos que sustituir la quinta linea por top_n(-3, lifeExp).
Una función auxiliar que es muy útil al utilizarla junto a mutate, y que puede veniros bien para buscar buenos clientes o malos clientes es: case_when().
A ver si entendéis este ejemplo:
aa <- df %>%
group_by(continent, year) %>%
mutate (media_lifeExp = mean(lifeExp)) %>%
mutate (media_gdpPercap = mean(gdpPercap)) %>%
mutate(GOOD_or_BAD = case_when(
lifeExp > mean(lifeExp) & gdpPercap > mean(gdpPercap) ~ "good",
lifeExp < mean(lifeExp) & gdpPercap < mean(gdpPercap) ~ "bad" ,
TRUE ~ "medium"
)) %>%
filter(country == "Spain")5.5.9 Y ahora entre todos
El paquete nycflights13, disponible en CRAN, contiene datos sobre 336.776 vuelos que despegaron de alguno de los tres aeropuertos que dan servicio a la ciudad de Nueva York (EE.UU.) en 2013.
El conjunto principal de datos sobre los vuelos está disponible en el data.frame “flights”, dentro de este paquete. Adicionalmente, su autor (Hadley Wickham) también ha incluido datos sobre los propios aeropuertos, condiciones meteorológicas, etc. Para más detalles, ver archivo de descripción del paquete con el comando ?nycflights13.
Utiliza las funciones incluidas en el paquete dplyr, para responder a las siguientes preguntas:
- ¿Cuantos vuelos se realizan en total cada mes?
- ¿Qué aeropuerto acumula el mayor número de salidas de vuelos en todo el año?
- ¿Qué compañía acumula el mayor número de salida de vuelos en los meses de verano (jun-sep.)?
5.6 Combinar data.frames con dplyr
Ya sabemos manejar, filtrar, poner condiciones, etc. a un conjunto de datos, pero muchas veces lo que queremos hacer es unir o combinar varios conjuntos de datos (Joinings en inglés).
En dplyr hay 3 tipos de funciones (verbos) que se ocupan de diferentes operaciones para unir datasets:
Mutating joins, añade nuevas variables (o columnas) a un dataframe (df1). Estas nuevas columnas vienen de un segundo df2 (hay varias mutating joins, dependiendo del criterio para seleccionar las filas).
Filtering joins, filtra las filas (observaciones) de un dataframe (df1) basándose en si las filas de df1 coinciden (match) o no con una observación del segundo df2
Set operations, combina las observaciones de los dos datasets (df1 y df2) como si fueran elementos.
Todas estas funciones tienen una estructura similar: sus dos primeros argumentos son 2 dfs (en realidad tablas de datos): df1 y df2. El output de la función es siempre una nueva tabla (del mismo tipo que df1).
5.6.1 Mutating joins
Hay 4 tipos de mutating joins. Su sintaxis es idéntica, sólo se diferencian en que las filas que se seleccionan dependen del criterio para hacer el match:
inner_join(df1,df2): Retorna todas las columnas de df1 y también las de df2, pero solo retorna las filas de df1 que tienen una equivalencia en df2. (la equivalencia se define en función del valor de una variable o variables comunes en df1 y df2)left_join(df1,df2): Retorna todas las columnas de df1 y también las de df2; en cuanto a las filas, retorna todas las filas de df1. (Si hubiesen varios matches entre df1 y df2 se retornan todas las combinaciones!!!!)rigth_join(df1,df2): Retorna todas las columnas de df1 y también las de df2; en cuanto a las filas, retorna TODAS las filas de df2. De df2!! (Si hubiesen varios matches entre df1 y df2 se retornan todas las combinaciones!!!!)full_join(df1,df2): Retorna todas las columnas de df1 y también las de df2; en cuanto a las filas, retorna TODAS las filas de df1 y de df2. O sea, retorna todas las filas y TODAS las columnas de las 2 tablas. (Donde no hay matches retorna NA’s)
5.6.1.1 Ejemplos (de Haley)
## # A tibble: 2 x 2
## x y
## <dbl> <int>
## 1 1 2
## 2 2 1## # A tibble: 2 x 3
## x a b
## <dbl> <dbl> <chr>
## 1 1 10 a
## 2 3 10 aInner Join: inner_join() nos da las observaciones que hay en ambas df1 y df2.
df_inner <- inner_join(df1, df2)
## Joining, by = "x"
df_inner
## # A tibble: 1 x 4
## x y a b
## <dbl> <int> <dbl> <chr>
## 1 1 2 10 aLeft Join: left_join() incluye todas las observaciones de df1. Esta es la forma más común de juntar tablas ya que no perdemos ningún dato en la tabla primaria (en este caso (df1).
df_left_join <- left_join(df1, df2)
## Joining, by = "x"
df_left_join
## # A tibble: 2 x 4
## x y a b
## <dbl> <int> <dbl> <chr>
## 1 1 2 10 a
## 2 2 1 NA <NA>Right Join: right_join() incluye todas las observaciones de df2.
df_right_join <- right_join(df1, df2)
## Joining, by = "x"
df_right_join
## # A tibble: 2 x 4
## x y a b
## <dbl> <int> <dbl> <chr>
## 1 1 2 10 a
## 2 3 NA 10 aFull Joint: full_join() todas las observaciones de df1 y df2
5.6.1.2 Cosa IMPORTANTE ha tener en cuenta con “mutating joins”:
Las (left, right and full) joins se llaman colectivamente como “outer joins”. Cuando una fila no tiene match en una outer join, las nuevas variables que se crean se llenan con NA’s.
Las mutating joins se usan principalmente para añadir columnas, pero en el proceso pueden generarse nuevas filas: si un match no es único, se añadirán todas las combinaciones posibles (el producto cartesiano) de las matching observations. Veamos un ejemplo con una left_join:
(df1 <- data_frame(x = c(1, 1, 2), y = 1:3))
## # A tibble: 3 x 2
## x y
## <dbl> <int>
## 1 1 1
## 2 1 2
## 3 2 3
(df2 <- data_frame(x = c(1, 1, 2), z = c("a", "b", "a")))
## # A tibble: 3 x 2
## x z
## <dbl> <chr>
## 1 1 a
## 2 1 b
## 3 2 aleft_join() crea nuevas filas:
5.6.1.3 ¿Como elegir la columna (o columnas) que se usaran para hacer los matching?
Podemos(DEBEMOS) elegir las columnas (o variables) que nos servirán para unir los 2 dfs. Estas columnas que se usan para para hallar los matchings y que por tanto nos permiten fusionar los 2 dfs se llaman “keys”.
La opción de las funciones para seleccionar estas columnas “keys” es by =.
Si ponemos
left_join(df1, df2, by = "X1")se hará una left_join siendo la variable “X1” la que hará de key. Si las variables key no se llamasen igual en los 2 dfs siempre podemos renombrarlas o hacerleft_join(df1, df2, by = c("X1" = "D4").Si ponemos
left_join(df1, df2,by = c("X1", "X2")hará falta que una row de df1 tenga los valores tanto de X1 como de X2 iguales a los de esas mismas variables en df2. Si no se llamasen igual las variables en df1 y df2 haríamosby = c("X1" = "D4", "X2" = "D7").
d1 <- tibble(
x = letters[1:3],
y = LETTERS[1:3],
a = rnorm(3)
)
d2 <- tibble(
x = letters[3:1],
y2 = LETTERS[3:1],
b = rnorm(3)
)
d1 ; d2
## # A tibble: 3 x 3
## x y a
## <chr> <chr> <dbl>
## 1 a A -0.595
## 2 b B 0.244
## 3 c C 1.02
## # A tibble: 3 x 3
## x y2 b
## <chr> <chr> <dbl>
## 1 c C -0.403
## 2 b B -1.78
## 3 a A 0.0416
left_join(d1, d2, by = c("x"))
## # A tibble: 3 x 5
## x y a y2 b
## <chr> <chr> <dbl> <chr> <dbl>
## 1 a A -0.595 A 0.0416
## 2 b B 0.244 B -1.78
## 3 c C 1.02 C -0.403
left_join(d1, d2, by = c("x", "y" = "y2"))
## # A tibble: 3 x 4
## x y a b
## <chr> <chr> <dbl> <dbl>
## 1 a A -0.595 0.0416
## 2 b B 0.244 -1.78
## 3 c C 1.02 -0.4035.6.2 Filtering joins
Filtering joins son similares a los anteriores (Mutating joins); o sea, hacen machting con las filas de la misma manera, PERO afectan a las filas, NO a las columnas. Hay filtering joins de 2 tipos:
semi_join(df1,df2): retorna las observaciones de df1 que tienen un match en df2. En cuanto a las columnas sólo retorna las columnas de df1anti_join(df1,df2): retorna las observaciones de df1 que NO tienen un match en df2; osea, quita las observaciones con match. En cuanto a las columnas sólo retorna las columnas de df1
La semi_join se diferencia de la inner_join en que la inner_join solo retorna una fila de df1 por cada matching, mientras que la semi_join NUNCA duplica filas de df1
Las filtering joins son útiles para diagnosticar disparidades. Si quieres saber sobre los datos que SI estan emparejados, haz una semi_join() or anti_join(). semi_join() y anti_join() NUNCA duplican filas; solo pueden quitar filas.
(df1 <- data_frame(V1 = c(1, 1, 3, 4), V2 = 1:4))
## # A tibble: 4 x 2
## V1 V2
## <dbl> <int>
## 1 1 1
## 2 1 2
## 3 3 3
## 4 4 4
(df2 <- data_frame(V1 = c(1, 1, 2), V3 = c("a", "b", "a")))
## # A tibble: 3 x 2
## V1 V3
## <dbl> <chr>
## 1 1 a
## 2 1 b
## 3 2 asemi_join:
df_semi_join <- semi_join(df1, df2, by = "V1")
df_semi_join
## # A tibble: 2 x 2
## V1 V2
## <dbl> <int>
## 1 1 1
## 2 1 2anti_join:
df_anti_join <- anti_join(df1, df2, by = "V1")
df_anti_join
## # A tibble: 2 x 2
## V1 V2
## <dbl> <int>
## 1 3 3
## 2 4 45.6.2.1 Comparemos la semi_join con la inner_join
## # A tibble: 4 x 3
## V1 V2 V3
## <dbl> <int> <chr>
## 1 1 1 a
## 2 1 1 b
## 3 1 2 a
## 4 1 2 b## # A tibble: 2 x 2
## V1 V2
## <dbl> <int>
## 1 1 1
## 2 1 2La inner_join
- Añade variables de df2 a df1 (en este caso V3).
- Sólo retiene las rows de df1 que tienen un match en df2 (en este caso las 2 primeras filas de df1).
- Pero en este ejemplo además duplica rows. La razón es que hay matching duplicados, hay 2 unos en df1 y otros 2 unos en df2 (con distintos valores de V3) (así que salen 4 rows).
5.6.3 Set operations
Este tipo de joins son más estrictos: hace falta que los 2 dfs tengan las mismas variables (o columnas). Los 2 dfs pueden tener observaciones (filas) diferentes, PERO es necesario que tengan las mismas variables (o columnas).
Como los 2 dfs tienen las mismas columnas, entonces es como si se tratasen los dfs como conjuntos:
intersect(df1, df2): devuelve un df con las observaciones comunes en df1 y df2union(df1, df2): devuelve la unión; o sea, las observaciones de df1 y de df2 (quitando las posibles filas duplicadas)union_all(df1, df2): devuelve la unión (sin quitar los duplicados)setdiff(df1, df2): devuelve las filas en df1 que no están en df2setequal(df1,df2): retorna TRUE si df y df2 tienen exactamente las mismas filas (da igual el orden en el que estén las filas)
5.6.4 bind_rows y bind_cols
Juntar data frames for filas o columnas.
(one <- starwars[1:2, ])
## # A tibble: 2 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender homeworld species films vehicles starships
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> <chr> <chr> <list> <list> <list>
## 1 Luke Skywalker 172 77 blond fair blue 19 male masculine Tatooine Human <chr [5]> <chr [2]> <chr [2]>
## 2 C-3PO 167 75 <NA> gold yellow 112 none masculine Tatooine Droid <chr [6]> <chr [0]> <chr [0]>
(two <- starwars[11:12, ])
## # A tibble: 2 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender homeworld species films vehicles starships
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> <chr> <chr> <list> <list> <list>
## 1 Anakin Skywalker 188 84 blond fair blue 41.9 male masculine Tatooine Human <chr [3]> <chr [2]> <chr [3]>
## 2 Wilhuff Tarkin 180 NA auburn, grey fair blue 64 male masculine Eriadu Human <chr [2]> <chr [0]> <chr [0]>
bind_rows(one, two)
## # A tibble: 4 x 14
## name height mass hair_color skin_color eye_color birth_year sex gender homeworld species films vehicles starships
## <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> <chr> <chr> <list> <list> <list>
## 1 Luke Skywalker 172 77 blond fair blue 19 male masculine Tatooine Human <chr [5]> <chr [2]> <chr [2]>
## 2 C-3PO 167 75 <NA> gold yellow 112 none masculine Tatooine Droid <chr [6]> <chr [0]> <chr [0]>
## 3 Anakin Skywalker 188 84 blond fair blue 41.9 male masculine Tatooine Human <chr [3]> <chr [2]> <chr [3]>
## 4 Wilhuff Tarkin 180 NA auburn, grey fair blue 64 male masculine Eriadu Human <chr [2]> <chr [0]> <chr [0]>(one <- starwars[1:3,1:2 ])
## # A tibble: 3 x 2
## name height
## <chr> <int>
## 1 Luke Skywalker 172
## 2 C-3PO 167
## 3 R2-D2 96
(two <- starwars[1:3,c(4:6) ])
## # A tibble: 3 x 3
## hair_color skin_color eye_color
## <chr> <chr> <chr>
## 1 blond fair blue
## 2 <NA> gold yellow
## 3 <NA> white, blue red
bind_cols(one, two )
## # A tibble: 3 x 5
## name height hair_color skin_color eye_color
## <chr> <int> <chr> <chr> <chr>
## 1 Luke Skywalker 172 blond fair blue
## 2 C-3PO 167 <NA> gold yellow
## 3 R2-D2 96 <NA> white, blue red5.6.5 Usando el %>% para concatenar acciones
df1 <- data_frame(V1 = c(1, 1, 3, 4), V2 = 1:4)
df2 <- data_frame(V1 = c(1, 1, 2), V3 = c("a", "b", "a"))
df1 %>%
left_join(df2)## Joining, by = "V1"## # A tibble: 6 x 3
## V1 V2 V3
## <dbl> <int> <chr>
## 1 1 1 a
## 2 1 1 b
## 3 1 2 a
## 4 1 2 b
## 5 3 3 <NA>
## 6 4 4 <NA>## Joining, by = "V1"## # A tibble: 4 x 3
## V1 V2 V3
## <dbl> <int> <chr>
## 1 1 1 a
## 2 1 1 b
## 3 1 2 a
## 4 1 2 b5.7 Y mucho más (importante)
Esta página contiene la mayoría de funciones de dplyr con las que podremos hacer casi todo lo que queramos https://dplyr.tidyverse.org/reference/index.html
5.8 Ejercicios
Vamos a cargar las bases de datos “Siniestros” e “Historicos_siniestros” y vamos a escribir ecomn el script las líneas necesarias para cargar bien los datos.
Vamos a pensar qué cosas queremos hacer con estas bases de datos, y hagámoslas!!