Capítulo 4 Modelos con respuesta normal
En este capítulo se describirán los métodos y modelos estadísticos para analizar medidas respetidas cuando la variable respuesta sigue una distribución normal o gaussiana. Se pueden probar algunas transformaciones, como el logaritmo, para normalizar la distribución de la variable.
4.1 Técnica de la suma de cuadrados
Este método o técnica se basa en la suma de cuadrados. Es la más simple des de el punto de vista estadístico y computacional. Por contra, sólo permite analizar diseños balanceados, sin variables independientes cuantitativas (covariables), sólo cualitativas o factores y con un número limitado de factores que tienen que estar cruzados (no anidados).
A continuación se presentan los dos diseños más simples de medidas repetidas que se pueden analizar con esta técnica.
4.1.1 Diseño 1W+1B
Cuando el diseño es balanceado (mismo número de individuos por grupo), las medidas son las mismas para todos los individuos y no hay covariables, se puede usar la técnica de suma de cuadrados o tabla ANOVA.
La notación que se usa para la ecuación del modelo en el contexto de suma de cuadrados es:
\[y_{ijk} = \mu + \alpha_i + \beta_j + \alpha\beta_{ij} + \pi_{k(i)} + e_{ijk}\]
Donde
- \(\mu\) es la constante del modelo,
- \(\alpha_i\), son los efectos del grupo o tratamiento
- \(\beta_j\), son los efectos del tiempo
- \(\alpha\beta_{ij}\) es la interacción del tiempo con el grupo
- \(\pi_{k(i)}\) es el efecto aleatorio del individuo que está anidado al grupo
- \(e_{ijk}\) son los errores
\(\sum_{i=1}^a \alpha_i=0\), \(\sum_{j=1}^b \beta_j=0\), \(\sum_{i=1}^a \alpha\beta_{ij}=0, \forall j\), \(\sum_{j=1}^b \alpha\beta_{ij}=0, \forall i\),
\(\pi_{k(i)} \sim N(0, \sigma_{ind})\)
En este contexto se dice que el tiempo y la interacción tratamiento:tiempo son términos o componentes “intra sujeto” (within subject). Mientras que el grupo es un componente “entre sujeto” (between subject). Por lo tanto, se trata de un diseño 1W+1B.
Las técnicas clásicas de la tabla ANOVA y su inferencia són válidas siempre y cuando se cumpla la condició de esfericidad: la variancia de la diferencia entre dos medidas es constante. Para comprobar la condición de esfericidad se puede aplicar el test de Mauchly. Si no se cumple hay que corregir los grados de libertad de los términos “intra sujetos” de la tabla ANOVA y se recalculan sus p-valores. Hay dos métodos para corregir los grados de libertad: método “Huynh and Feldt” (H-F) y el método “Greenhouse-Geisser” (G-G) .
4.1.2 Diseño 1W
Si en el diseño no hay grupos, luego el modelo se simplifica a un diseño de un solo factor “intra sujeto” (1W)
\[y_{ij} = \mu + \pi_i + \beta_j + e_{ij}\]
En ambos casos, tanto en el diseño en que tenemos grupos (1W+1B) como en el que no (1W), no nos interesa evaluar el efecto del individuo; ya sabemos que hay variabilidad entre ellos. Veremos en un ejemplo como el paquete ez que se usará para esta técnica de suma de cuadrados omite los resultados sobre el factor aleatorio individuo.
4.1.3 Función ezANOVA
Para ajustar los modelos de medidas repetidas balanceados mediante la técnica de suma de cuadrados existe la función ezANOVA del paquete ez (Lawrence 2016)
Tanto la corrección por H-F o G-G, como el test de esfericidad de Mauchly estan implementados en el package ez de R.
Para visualizar gráficamente los resultados, se usará la función ezPlot. Más adelante en esta sección se verá en un ejemplo.
Los datos deben estar en formato vertical. Es obligatorio que las variables estén en una base de datos (“data.frame”).
data: base de datos donde se encuentran las variablesdv: variable respuesta o variable dependientewid: variable individuowithin: factor o factores “intra sujeto”. Típicamente en este argumento se espedificará el tiempo. Si se especifica más de un factor, éstos deben estar cruzados y se escribirá.(var1,var2).between: factor o factores “entre sujetos”. Si no hay ningún factor “intra-sujeto” se deja aNULL. Como en el argumentowithin, si hay más de un factor “entre sujetos”, éstos deben estar cruzados y se escribirá.(var1,var2).
Observaciones:
- La variable respuesta y los factores deben escribirse sin comillas.
- Los factores “intra”, “entre” y el sujeto deben estar en format
factor. - El factor individuo debe tener tantos niveles como individuos.
- Aunque en teoría la función permite covariables (variables independientes contínuas), esta opción está en versión “beta”.
- Todos los factores, excepto el individuo, deben ser de efectos fijos.
4.2 Respuesta Multivariante
\[\vec{y}_i = \vec{\mu}_i + \vec{e}_i\]
Donde
\(\vec{y_i}=(y_{i1},\ldots,y_{iT})\) es el vector de medidas para el individuo \(i\).
\(\vec{\mu}_i=(\mu_{i1},\ldots,\mu_{iT})\) es el vector con las medias de cada momento y para cada individuo. Las medias pueden depender de las variables independientes \(x_k\). Fíjate que el coeficiente \(\beta_{kj}\) puede ser diferente para cada momento.
\[\mu_{ij} = \sum_{k=1}^K \beta_{jk} x_{ik}, \quad j=1,...,T\]
\(\vec{e_i} \sim N(\vec{0},\Sigma)\), donde \(\Sigma\) es la matrix de covarianzas de los errores y tiene que ser la misma para todos los individuos. Su estructura, pero, puede ser cualquiera.
\(x_{ik}\) valor de la variable independiente \(k\) del individuo \(i\).
Observaciones
Para ajustar este modelo los datos se disponen de forma horizontal.
En este modelo los tiempos en que se toman las \(T\) medidas tienen que ser los mismos para todos los individuos.
Para estudiar la evolución en el tiempo se puede realizar un contraste polinómico en el vector de medias \(\vec{\mu}\).
Para comparar grupos de medidas, por ejemplo si se tienen cinco medidas, las dos primeras corresponden al tratamiento A y las otras tres al tratamiento B, se puede realizar un contraste lineal para comparar los dos tratamientos.
Cuando hay un valor faltante en alguna medida, toda la fila del individuo se tiene que eliminar.
Cada variable independiente, \(x_{ki}\) es un único valor por individuo. O sea, que este modelo no contempla que las variables independientes sean de medidas repetidas. Si tuviéramos una variable que cambiara en el tiempo, se tienen que poner como variables diferentes (una para cada momento).
Los factores contribuyen con tantas dummy variables como categorías menos uno en los términos \(x_{ik}\).
Los términos \(x_{ik}\) pueden ser también interacciones entre variables, como el producto de sus términos.
| indiv | edad | fumador | sexo | fumadorEx | fumadorNunca | edad | sexomujer | fumadorEx:edad | fumadorNunca:edad |
|---|---|---|---|---|---|---|---|---|---|
| 1 | 50 | Ex | mujer | 1 | 0 | 50 | 1 | 50 | 0 |
| 2 | 55 | Actual | mujer | 0 | 0 | 55 | 1 | 0 | 0 |
| 3 | 60 | Actual | hombre | 0 | 0 | 60 | 0 | 0 | 0 |
| 4 | 65 | Nunca | mujer | 0 | 1 | 65 | 1 | 0 | 65 |
| 5 | 62 | Ex | hombre | 1 | 0 | 62 | 0 | 62 | 0 |
4.3 Modelos Lineales Mixtos (LMM)
Esta técnica es la más potente para analizar datos longitudinales ya que permite introducir efectos aleatorios y especificar la estructura de correlaciones de los residuos dentro de un mismo individuo.
Además, a diferencia de las dos técnicas anteriores, permite trabajar con missings.
4.3.1 Ecuación
\[ y_{ij} = \beta_{0i} + \sum_{k=1}^K \beta_{ki} x_{ijk} + e_{ij} \]
Donde \(i\) representa al individuo, \(j\) representa el momento (de uno hasta hasta el número de observaciones del individuo \(i\)),
\(x_{ijk}\) valor de la \(k\)-ésima variable independiente del individuo \(i\) en el momento \(j\).
\(\beta_{0i} \sim N\left(\beta_0, \sigma_{\beta_0}^2\right)\) es la constante del modelo aunque en general se supone aleatoria, o sea que tiene cierta varianza entre individuos y está centrada en la contaste \(\mu\).
\(\beta_{ki} \sim N\left(\beta_k, \sigma_{\beta_k}^2\right)\): pendientes o coeficientes de las variables del modelo. Pueden ser aleatorias, o sea, variar entre individuos.
En general puede haber correlación entre la contante \(\beta_{0i}\) y las pendientes \(\beta_{ki}\).
El vector formado por la constante y por los coeficientes aleatorios, son los efectos aleatorios y se supone que sigue una distribución normal multivariada:
\[\vec{\beta}_i = (\beta_{0i},\beta_{1i},\ldots,\beta_{Ki})^t \sim N\left(\vec{\beta}, \Omega) \right)\]
- El vector formado por los errores de un individuo \(\vec{e_i} \sim N(\vec{0},\Sigma_i)\), sigue una distribuión normal multivariante con una cierta matriz de covarianzas \(\Sigma_i\) que no tiene porqué ser la misma ni del mismo tamaño para todos los individuos ya que no todos los individuos tendrán el mismo número de observaciones. Los errores son independientes de la constante aleatoria y de los coeficientes aleatorios.
Observaciones
Para ajustar este modelo los datos se disponen de forma vertical.
El modelo LMM es muy flexible y potente. No sólo permite especificar efectos aleatorios con lo que evaluar la variabilidad de ciertos efectos o variables entre individuos sinó también la correlación residual entre las distintas medidas repetidas en un mismo individuo.
Cuando hay missings en una observación no hace falta eliminar las otras del mismo individuo, ya que cada fila aquí es una observación y no un individuo.
La esperanza de la constante y coeficientes aleatorios \(\vec{\beta}_i\) es la misma para todos los individuos, \(\vec{\beta}\), y la matriz de covarianzas, \(\Omega\), también (homocedesticidad).
Si un coeficiente no es aleatorio, se puede notar como \(\beta_{k'i} = \beta_{k'}\) en lugar de suponer que sigue una distribución normal. También se podría pensar que sigue una distribución “normal” con varianza cero.
Los efectos fijos son la esperanza de los efectos aleatorios (\(\beta_0, \beta_1, \ldots, \ldots, \beta_k\)). Además, cuando un coeficiente no es aleatorio (tiene varianza cero) se denomina fijo directamente.
Hay un número limitado de efectos aleatorios que se pueden incorporar en el modelo, que no puede exceder el número de medidas por individuo.
La presencia de efectos aleatorios inducen correlación entre medidas de un mismo individuo. Sin embargo, según que estructura de correlación sólo se puede conseguir definiendo también una estructura de correlación entre residuos no nula (no diagonal).
Considerando los coeficientes del tiempo como aleatorios se induce correlación distinta según los tiempos que se toman las medidas.
Considerando el coeficiente de una variable no cambiante en el tiempo se induce heterocedesticidad (varianza diferente) entre los individuos.
Por ejemplo, supongamos un modelo con efecto lineal del tiempo y una covariable no cambiante del tiempo (\(x_i\)). Y tomamos la constante y el coeficiente de \(x_i\) aleatorios y ambos no correlacionados.
\[y_{ij} = \beta_{0i} + \beta_1 t_{ij} + \beta_{2i} x_i\]
Luego la varianza de \(y_{ij}\) es \(\sigma_{\beta_{0}}^2 + \sigma_{2i}^2 x_i^2\). Si \(x_i\) vale 0 para el grupo placebo y 1 para los tratados, entonces la varianza del grupo placebo será \(\sigma_{\beta_{0}}^2\) y para los tratados \(\sigma_{\beta_{0}}^2 + \sigma_{\beta_{2}}^2\)
- A diferencia de las técnicas de sumas de cuadrados y de respuesta multivariante, en que la variable tiempo se trata como a un factor, con los LMM se tratar también como variable continua.
4.3.2 Casos particulares
4.3.2.1 Modelo con constante aleatoria
\[y_{ij} = \beta_{0i} + \beta_{1} t_{ij} + e_{ij}\]
Donde \(\beta_{0i} \sim N(\beta_0, \sigma_{\beta_0})\), y \(\beta_1\) es el coeficiente fijo del tiempo. En este caso se supone que el tiempo tiene un efecto lineal.
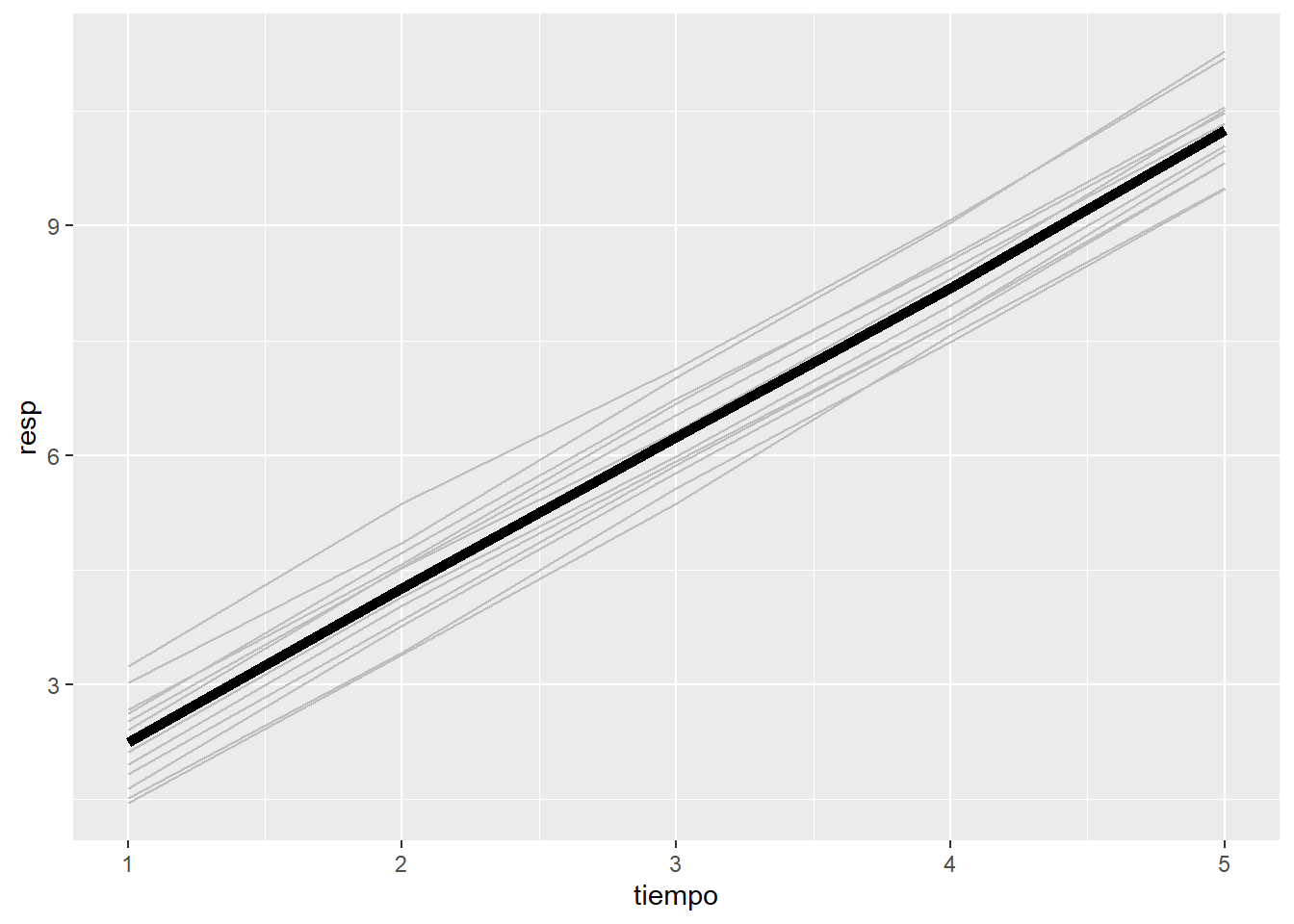
Podríamos añadir un término cuadrático, cúbico, etc. si el efecto no fuera lineal y añadiéramos un término cuadrático:
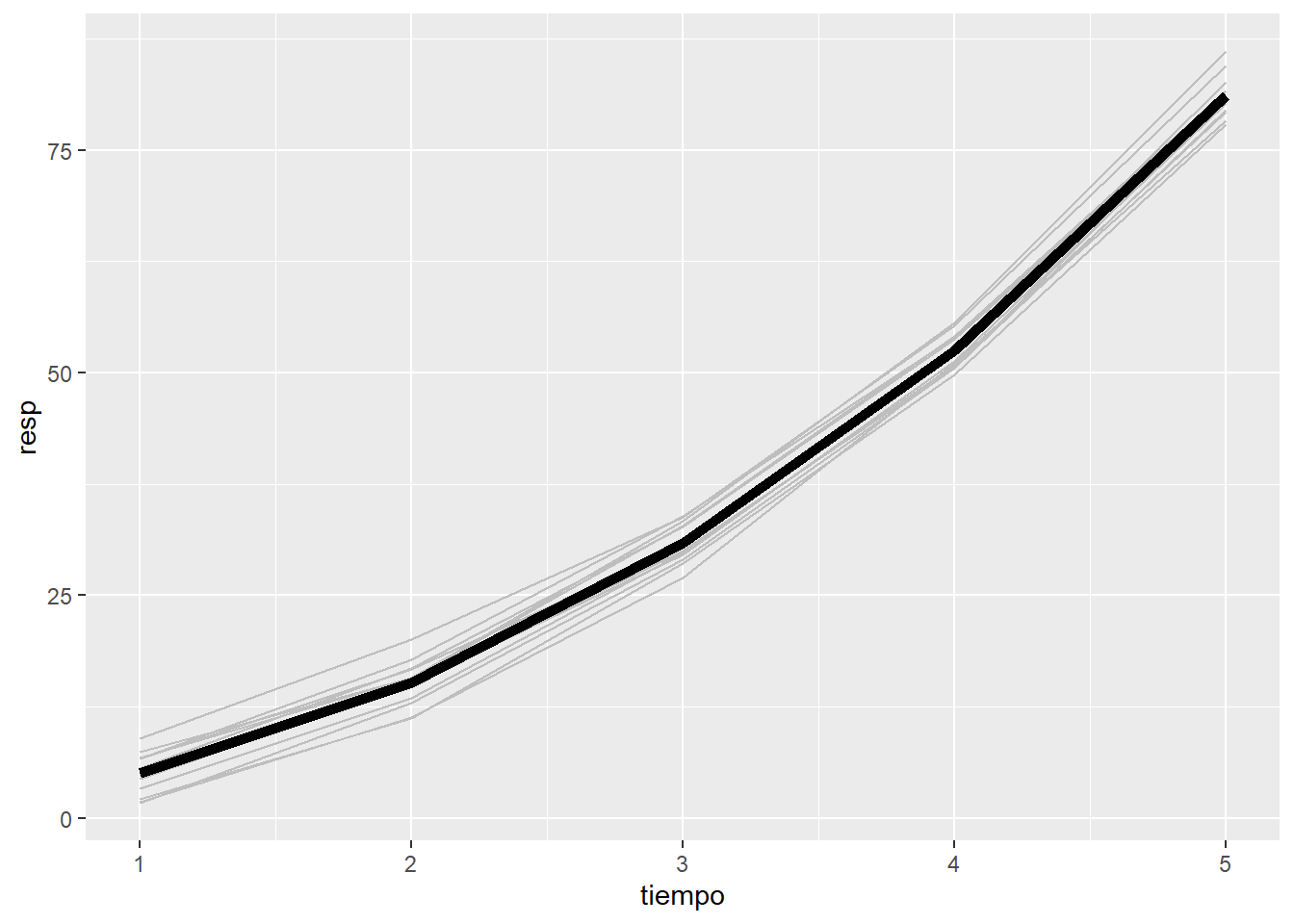
\[y_{ij} = \beta_{0i} + \beta_{1} t_{ij} + \beta_{2} t_{ij}^2 + e_{ij}\] en este caso, \(x_{ij1}=t_{ij}\) y \(x_{ij2}=t_{ij}^2\).
Correlacion entre observaciones
Si los errores son independientes, las observaciones de la variable respuesta de un mismo individuo están correlacionadas. Y esta correlación es constante: no depende de la distancia entre las medidas.
\[\text{corr}(y_{i1},y_{i2}) = \text{corr}(y_{i1},y_{i3}) = \ldots = \frac{\sigma_{\beta_{0}}^2}{\sigma_{e}^2}\]
A esta correlación también se la conoce como coeficiente de correlación intraclase (ICC)
4.3.2.2 Modelo con pendiente y constante aleatoria
\[y_{ij} = \beta_{0i} + \beta_{1i} t_{ij} + e_{ij}\]
\(\vec{\beta}_i = (\beta_{0i}, \beta_{1i})^t \sim N\left((\beta_0,\beta_1)^t, \Omega\right)\), donde
\[\Omega= \begin{pmatrix} \sigma_{\beta_0}^2 & \sigma_{\beta_0,\beta_1} \\ \sigma_{\beta_0,\beta_1} & \sigma_{\beta_1}^2 \end{pmatrix} \]
El término \(\sigma_{\beta_0,\beta_1}\) es la covarianza entre la constante y la pendiente. Ésta en general puede no ser cero.
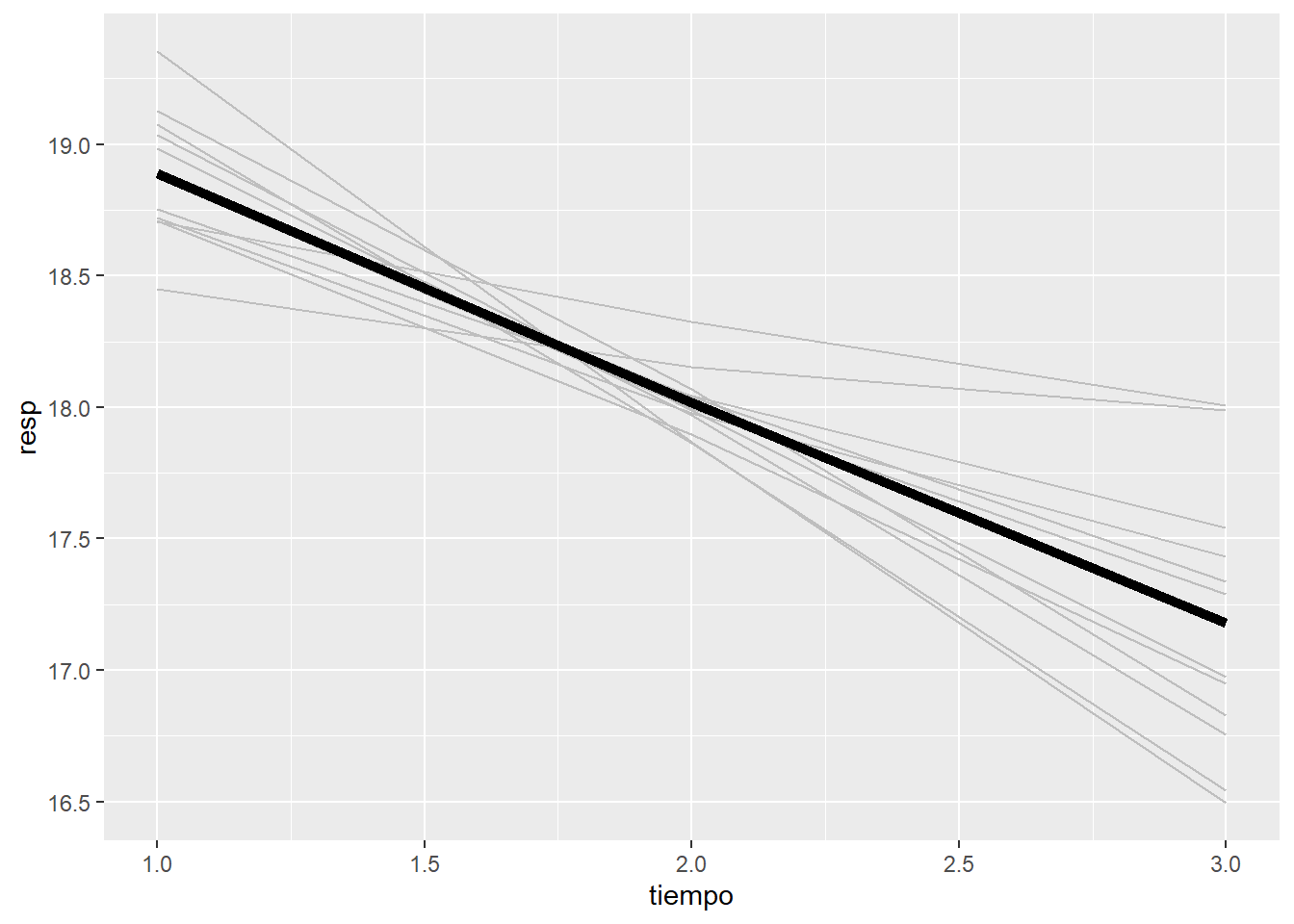
En este gráfico se observa primero que las pendientes son diferentes entre los individuos. Y además, que los individuos que empiezan de más arriba bajan más rápido y viceversa. Así pues, la correlación entre la constante y la pendiente es negativa.
Correlacion entre observaciones
En el modelo con constante y pendientes aleatorios, si asumimos que los errores son independientes, las observaciones de la variable respuesta de un mismo individuo están correlacionadas. Y esta correlación depende de los momentos:
\[\text{corr}(y_{i1},y_{i2}) = \frac{\sigma_{\beta_{0}}^2+\sigma_{\beta_{1i}^2 \cdot t_1 \cdot t_2}}{\sqrt{\sigma_{\beta_{0}}^2+\sigma_{\beta_{1}}^2 \cdot t_1^2 + \sigma_e^2}\sqrt{\sigma_{\beta_{0}}^2+\sigma_{\beta_{1}}^2 \cdot t_2^2 + \sigma_e^2}}\]
Por lo tanto depende tanto de \(t_1\) como de \(t_2\) y no sólo de la distancia entre las medidas.
Si lo comparamos con el AR(1):
Ejemplo con \(t_1=1\), \(\sigma_{\beta_{0}}^2=0.5^2\), \(\sigma_{\beta_{1}}^2=0.3^2\) y \(\sigma_{e}^2=0.1^2\), con \(t_1=1\)
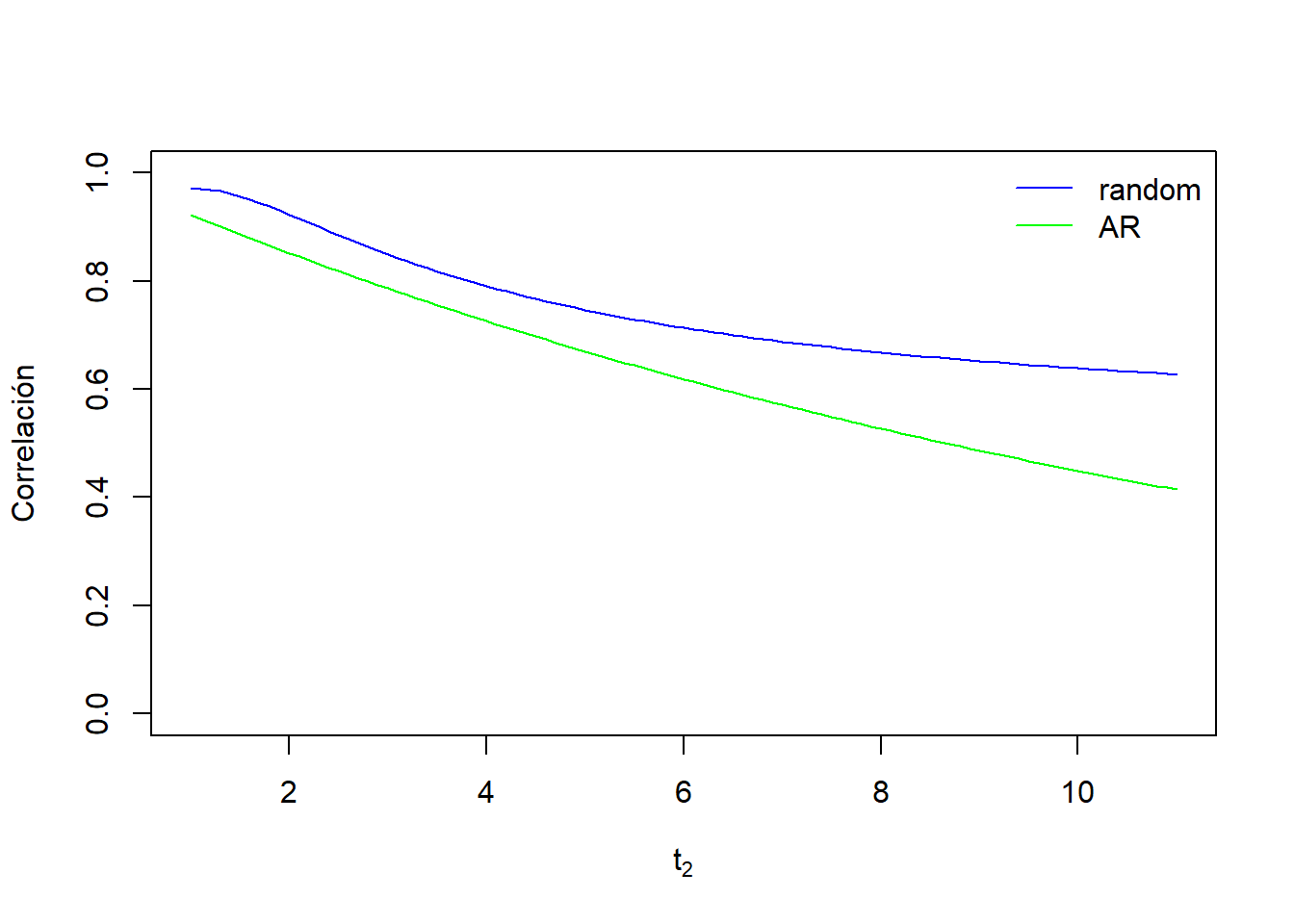
En este ejemplo, vemos como especificando el AR y la pendiente fija, la correlación entre observaciones baja más rápidamente a medida que las observaciones se alejan (\(t_2\)) que lo que se consigue especificando la pendiente aleatoria y los errores incorrelacionados.
4.3.3 Simplificación del modelo
Empezaremos con el modelo más general, o sea, sin asumir independencia de los residuos, con efectos aleatorios (todos los que se admitan) correlacionados.
En cuanto a los efectos fijos, se incluyrán también los máximos que se puedan, interacciones si es pertinente, terminos cuadráticos (cúbicos), …
A partir de aquí se simplificará el modelo en el siguiente orden:
4.3.3.1 Significación de los efectos aleatorios
La hipótesis nula para contrastar los factores de efectos aleatorios es que su varianza es igual a cero. Por ejemplo para la constante aleatoria:
\[\left\{\begin{array}{l} \text{H}_0: \sigma_{\beta_0}^2 = 0 \\ \text{H}_1: \sigma_{\beta_0}^2 > 0 \end{array}\right. \]
Hay diferentes técnicas estadísticas para contrastar estos tests, pero no son estándard. El problema es que la varianza de una distribución normal no puede ser cero, por lo tanto la hipótesis nula está fuera del espacio parametrico (“beyond boundary”). Existen, pero, algunas herramientas en R que lo realizan mediante técnicas de remuestreo (“bootstrap”). Éstas son complejas desde el punto de visto teórico y no se explicarán en este curso (véase el paquete de R pbkrtest Halekoh and Højsgaard (2014)).
Otra alternativa es usar índices como el AIC o BIC (cuanto más bajo mejor), que proporciona la función anova en la comparación de dos modelos: uno considerando el coeficiente como aleatorio (\(\beta_{ik}\)) el otro considerando el coeficiente como fijo (\(\beta_{k}\)).
4.3.3.2 Elección matriz covarianzas de los efectos aleatorios
Si en el paso anterior, hay más de un efecto aleatorio significativo, seguidamente hay que contrastar si la correlación entre ellos es cero o no. Es decir, H\(_0\) postula que la matriz \(\Omega\) es diagonal, mientras que la H\(_1\) se asume que las correlaciones pueden ser no nulas.
\[\left\{\begin{array}{l} \text{H}_0: \Omega= \begin{pmatrix} \sigma_{\beta_0}^2 & 0 \\ 0 & \sigma_{\beta_1}^2 \end{pmatrix}\\ \text{H}_1: \Omega= \begin{pmatrix} \sigma_{\beta_0}^2 & \sigma_{\beta_0\beta_1} \\ \sigma_{\beta_0\beta_1} & \sigma_{\beta_1}^2 \end{pmatrix} \end{array}\right. \]
Como la matriz diagonal es un caso particular de la matriz general, en que las correlaciones son cero se puede aplicar el test de razón de verosimilitudes.
4.3.3.3 Estructura de correlación de los errores
Mediante el test de razón de verosimilitudes (LRT), se comparan las verosimilitudes de dos modelos.
Hay que ajustar el modelo mediante el criterio de máxima verosimilutud.
Los modelos tienen que estar anidados: la matriz de covarianzas de los errores de un modelo se pueda expresar como un caso particular de la del otro modelo. Por ejemplo, la matriz sin estructura sería la más general de todas, y la matriz de simetría compuesta sería un caso particular en que todas las correlación son iguales. No están anidadas las matrices con estructura MA(1) y una AR(1).
La simetría compuesta es un caso particular de matriz sin estructura.
\[ \begin{pmatrix} 1 & \rho_{12} & \rho_{13} \\ \rho_{12} & 1 & \rho_{23} \\ \rho_{13} & \rho_{23} & 1 \\ \end{pmatrix} \Rightarrow \color{blue}{(\rho_{12}=\rho_{13}=\rho_{23} =\rho)} \Rightarrow \begin{pmatrix} 1 & \rho & \rho \\ \rho & 1 & \rho \\ \rho & \rho & 1 \\ \end{pmatrix} \]
La matriz que supone independencia entre los residuos es un caso particular de matriz de simetría compuesta.
\[ \begin{pmatrix} 1 & \rho & \rho \\ \rho & 1 & \rho \\ \rho & \rho & 1 \\ \end{pmatrix} \Rightarrow \color{blue}{(\rho=0)} \Rightarrow \begin{pmatrix} 1 & 0 & 0 \\ 0 & 1 & 0 \\ 0 & 0 & 1 \\ \end{pmatrix} \]
Pero no se puede pasar de una AR(1) a una MA(1) ni viceversa. En este caso el test LRT no es válido pero sí el criterio AIC o BIC.
\[ \begin{pmatrix} 1 & \rho & \rho^2 \\ \rho & 1 & \rho \\ \rho^2 & \rho & 1 \\ \end{pmatrix} \Rightarrow \color{blue}{(????)} \Rightarrow \begin{pmatrix} 1 & \frac{\theta}{1+\theta^2} & 0 \\ \frac{\theta}{1+\theta^2} & 1 & \frac{\theta}{1+\theta^2} \\ 0 & \frac{\theta}{1+\theta^2} & 1 \\ \end{pmatrix} \]
Una matriz AR(p’) está anidada a AR(p) si p’ < p, o sea un AR de orden menor está anidado a una de orden mayor, y por lo tanto, se puede aplicar un LRT para decidir el valor de p. Por ejemplo, un AR de orden 3 se especificaría como correlation = corARMA(p=3, q=0). Lo mismo sucede para decidir el orden de una MA. Por ejemplo, para una MA de orden 4, correlation = corARMA(p=0, q=4).
Heterocedesticidad:
La heterocedesticidad se produce cuando los parámetros de la matriz de covarianzas \(\Sigma\) dependen de variables. Por ejemplo, del sexo o de la edad, etc, o de una combinación lineal de las variables (valor esperado).
Por ejemplo, que la varianza sea distinta según el sexo, mientras que la correlación sea la misma:
para hombres
\[ \Sigma_{\text{H}} = \sigma_{\text{H}}^2 \begin{pmatrix} 1 & \rho & \rho^2 \\ \rho & 1 & \rho \\ \rho^2 & \rho & 1 \\ \end{pmatrix} \],
y para las mujeres
\[ \Sigma_{\text{M}} = \sigma_{\text{M}}^2 \begin{pmatrix} 1 & \rho & \rho^2 \\ \rho & 1 & \rho \\ \rho^2 & \rho & 1 \\ \end{pmatrix} \]
También podríamos definir las varianzas (diagonal de \(\Sigma\)), en función del tiempo.
Veremos como es posible modelizar diferentes varianzas distintas entre grupos de individuos con la función lme de R que se describirá en esta sección.
4.3.3.4 Efectos fijos
Una vez escogida la estructura de covarianzas de los efectos aleatorios, de los errores, y qué efectos son aleatorios (contraste sobre sus varianzas), vamos a contrastar la significación de los efectos fijos:
Para ello, se puede usar el test de Wald para testar un único parámetro:
\[ \left\{\begin{array}{l} \text{H}_0: \beta_1 = 0\\ \text{H}_1: \beta_1 \neq 0 \end{array}\right. \]
o LRT para testar más de un parámetro a la vez, por ejemplo las dummies de un factor de más de dos categorías:
\[ \left\{\begin{array}{l} \text{H}_0: \beta_1 = \beta_2 = 0\\ \text{H}_1: \text{alguno diferente de 0} \end{array}\right. \]
4.3.4 Validación del modelo
Una vez simplificado y seleccionado el modelo, hay que validarlo.
De todas las premisas a comprobar en este curso nos limitaremos a las asumciones sobre los residuos.
Para ellos se realizarán dos gráficos:
Residuos estandarizados vs valores predichos: en este gráficos debería aparecer una nube de puntos uniformemente distribuida sin ninguna tendencia. Ésto nos indicaría que no nos hemos dejado ninguna variable, o ningún término cuadrático o cúbico del tiempo.
QQ-plot: éste gráfico está pensado para comprobar la normalidad. Si los puntos se encuentran alrededor de la diagonal sin seguir ningún patrón, dará evidencia de que los residuos siguen una distribución normal
Hay otras premisas que se deberían comprobar, como por ejemplo la normalidad de los efectos aleatorios. Pero, por su complejidad, no se verá en este curso.
La valicación de los efectos aleatorios es más compleja. Una posibilidad “naive” es considerar que sus estimaciones siguen una distribución normal y que su distribución no depende de ninguna covariable a nivel de individuo. Veremos como las funciones de R para estimar los LMM proporcionan las estimaciones de los efectos aleatorios (“Empirical Bayes Estimates”). Aunque los efectos aleatorios se suponen normalmente distribuidos, los “Empirical Bayes Estimates” no tienen porqué.
4.3.5 Predicciones
4.3.5.1 Efectos marginales
Los efectos marginales representan el valor esperado de la variable respuesta. Para calcularlos hay que especificar los valores de las variables predictoras (condicionar):
\[E(Y_{ij} | x_{ij1},\ldots,x_{ijK}) = \beta_0 + \sum_{k=i}^K \beta_k x_{ijk}\]
Una vez ajustado el modelo con los datos de la muestra, se estiman los valores de los parámetros para estimar los efectos marginales o predicciones.
\[\hat{E}(Y_{ij} | x_{ij1},\ldots,x_{ijK}) = \hat{\beta}_0 + \sum_{k=i}^K \hat{\beta}_k x_{ijk}\]
4.3.5.2 Estimación de los efectos aleatorios
También podemos condicionar al individuo:
\[\hat{E}(Y_{ij} | x_{ij1},\ldots,x_{ijK}, i) = \hat{\beta}_{0i} + \sum_{k=i}^K \hat{\beta}_{ki} x_{ijk}\]
Donde \(\hat{\beta}_{0i}\), \(\hat{\beta}_{ki}\) son los “Empirical Bayes Estimates”.
4.3.6 Función lme
Para ajustar los modelos lineales mixtos usaremos la función lme del paquete nlme (Pinheiro, Bates, and R-core 2020).
Esta función permite incorporar efectos aleatorios, así como especificar la estructura de la matriz de correlaciones de los residuos.
El método se basa en el criterio de máxima verosimilidud (“Maximum Likelihood” - ML), que busca el valor de los parámetros que maximizan la función de verosimilitud. Generalmente, la solución no es una fórmula cerrada y se necesitan métodos iterativos numéricos para encontrar el óptimo. También se calculan mediante métodos numéricos la primera y segunda derivada para acelerar el proceso de estimación y para obtener los errores estándard de las estimaciones.
Para usar la función lme, los datos deben estar en formato horizontal. No hace falta que haya el mismo número de medidas para cada individuo, ni que las medidas se hayan producido en los mismos tiempos.
Los argumentos más importantes de la función lme
fixed: Fórmula de la forma
respuesta ~ var1 + var2 + var3
La constante se presupone que está y no hace falta escribir 1+.
La sintaxis es la misma que para el “formula environment” de otras funciones estándard como lm para regresión lineal ordinaria (los términos van separados con +, las interacciones se especifican con :, etc.).
A la izquierda de ~ se especifica la variable respuesta.
random: fórmula de la forma
~ var1 + var2 + ... + varK | indivsin ninguna variable a la izquierda de ~, donde indiv es la variable sujeto y var1, var2, … varK son las variables con coeficiente aleatorio.
Por defecto se supone que la constante está incluida.
Si se desea que la constante no sea aleatoria
~ var1 + ... + varK - 1 | indiv.
Si sólo la constante es aleatoria
~ 1 | indiv
Para especificar que la matriz \(\Omega\) es diagonal se usa la función pdDiag
list(indiv = pdDiag( ~ var1 + var2 + ... + varK))
Si los individuos estuvieran anidados en clústers aleatorios:
~ var1+..| clusters / indiv
correlation: Para especificar la forma de la matriz de covarianzas de los residuos \(\Sigma_i\). Para más estructuras:
?corClassesResiduos independientes (valor por defecto):
NULLSimetría compuesta:
corCompSymm()AR(1):
corAR1()ARMA(p,q):
corARMA(p,q)\(\phi^{|t_i-t_j|}\):
corCAR1(form = ~ tiempo | indiv)Sin estructura |
corSymm()
Para corCAR1, \(\phi\) es la correlación entre dos medidas a distancia de una unidad de tiempo.
Importante!: para corCompSymm, corAR1, corARMA o corSymm, las medidas tienen que estar ordenadas dentro de cada individuo. Si no, hay que especificar la variable momento,
corAR1(form = ~ tiempo | indiv)weights: Este argumento modeliza la varianza, \(\sigma^2\) según variables. Por defecto,
NULLque supone que la matriz de covarianzas es la misma para todos los individuos. En lugar de una variable, puede ser el valor predicho,varFixed(fitted(.)). Para ver más?varClasses.varPower(): \(\sigma^2(x) = |x|^{2*\theta}\)varFixed(): \(\sigma^2(x) = |x|\)varConstPower()\(\sigma^2(x) = (\theta_1 + |x|^{\theta_2})^2\)
method: Método usado para estimar los parámetros (ML o REML). Para usar el LRT, o calcular los índices AIC o BIC se usa el método ML. La función
anovaque compara dos modelos por LRT, reajusta los modelos automàticamente bajo el método ML si han sido estimados con REML.REML (“REstricted Maximum Likelihood”): método por defecto y que proporciona estimaciones no sesgadas de los parámetros.
ML (“Maximum Likelihood”): proporciona estimaciones de los parámetros sesgados.
4.4 Otros modelos
Existen técnicas para ajustar modelos sin efectos aleatorios pero que permiten correlacion entre las medidas de un mismo individuo.
4.4.1 “Generalized Least Squares”
Para ajustar estos modelos en R se usa la función gls del paquete nlme.
Asumen que la variable respuesta es normal. Los coeficientes y la constante es fija, o sea, que no admiten efectos aleatorios. Sin embargo sí que se puede especificar cómo es la correlación entre las medidas de un mismo individuo, de la misma forma que en la función lme.
Este modelo puede ser útil para contrastar si la constante de un LMM es aleatoria o fija, ya que la función lme no puede ajustar modelos con todos los coeficientes incluida la constante fijos.
library(nlme)
data("Orthodont")
# modelo con la constante aleatoria
modelo1 <- lme(distance ~ age,
data = Orthodont,
random = ~ 1|Subject,
correlation = corAR1(form = ~ age | Subject))
# modelo con la constante y la pendiente fijos
modelo0 <- gls(distance ~ age,
data = Orthodont,
correlation = corAR1(form = ~ age | Subject))
# compara los dos modelos
anova(modelo0, modelo1) Model df AIC BIC logLik Test L.Ratio p-value
modelo0 1 4 517.1695 527.8233 -254.5848
modelo1 2 5 457.0025 470.3197 -223.5013 1 vs 2 62.16698 <.0001lme(distance ~ age,
data = Orthodont,
random = ~ -1|Subject,
correlation = corAR1(form = ~ age | Subject))Error in matrix(unlist(value), nrow = nrow(data), dimnames = list(row.names(data), :
'data' must be of a vector type, was 'NULL'Además, la función gls admiten otras distribuciones a parte de la normal para la variable respuesta.
4.4.2 Modelos GEE
Los modelos GEE (General Estimation Equation), a diferencia de los modelos gls y de los modelos lineales mixtos, no estan basados en la función de verosimilitud y se explicarán en el próximo tema.
-Ventajas: Las estimaciones son robustas aunque la variable respuesta no siga una distribución normal y/o la estructura de correlación de los errores no esté bien especificada.
-Desventajas: Al no estar baso en la verosimilitud no se puede comparar su estimación con otros modelos mediante el LRT ni se puede calcular el AIC ni el BIC. Si la variable respuesta sigue una distribución normal y se especifica bien las correlaciones entre los errores, los modelos gls y los LMM son más potentes.
En R hay diferentes paquetes para ajustar los modelos GEE. El más popular es el gee que contiene la función gee.
library(gee)
?gee
data(warpbreaks)
summary(gee(breaks ~ tension, id=wool, data=warpbreaks, corstr="exchangeable"))
summary(gee(breaks ~ tension, id=wool, data=warpbreaks, corstr="AR-M", Mv=1))
summary(gee(breaks ~ tension, id=wool, data=warpbreaks, corstr="unstructured"))La función gee, y en general los GEE incluyen otras distribuciones (de la familia exponencial) para la variable respuesta a parte de la normal. Ésto lo veremos en el siguiente capítulo.
4.5 Ejemplos
Vamos a ver algunos ejemplos que se analizarán mediante las técnicas que se acaban de describir.
4.5.1 Ejemplo 1
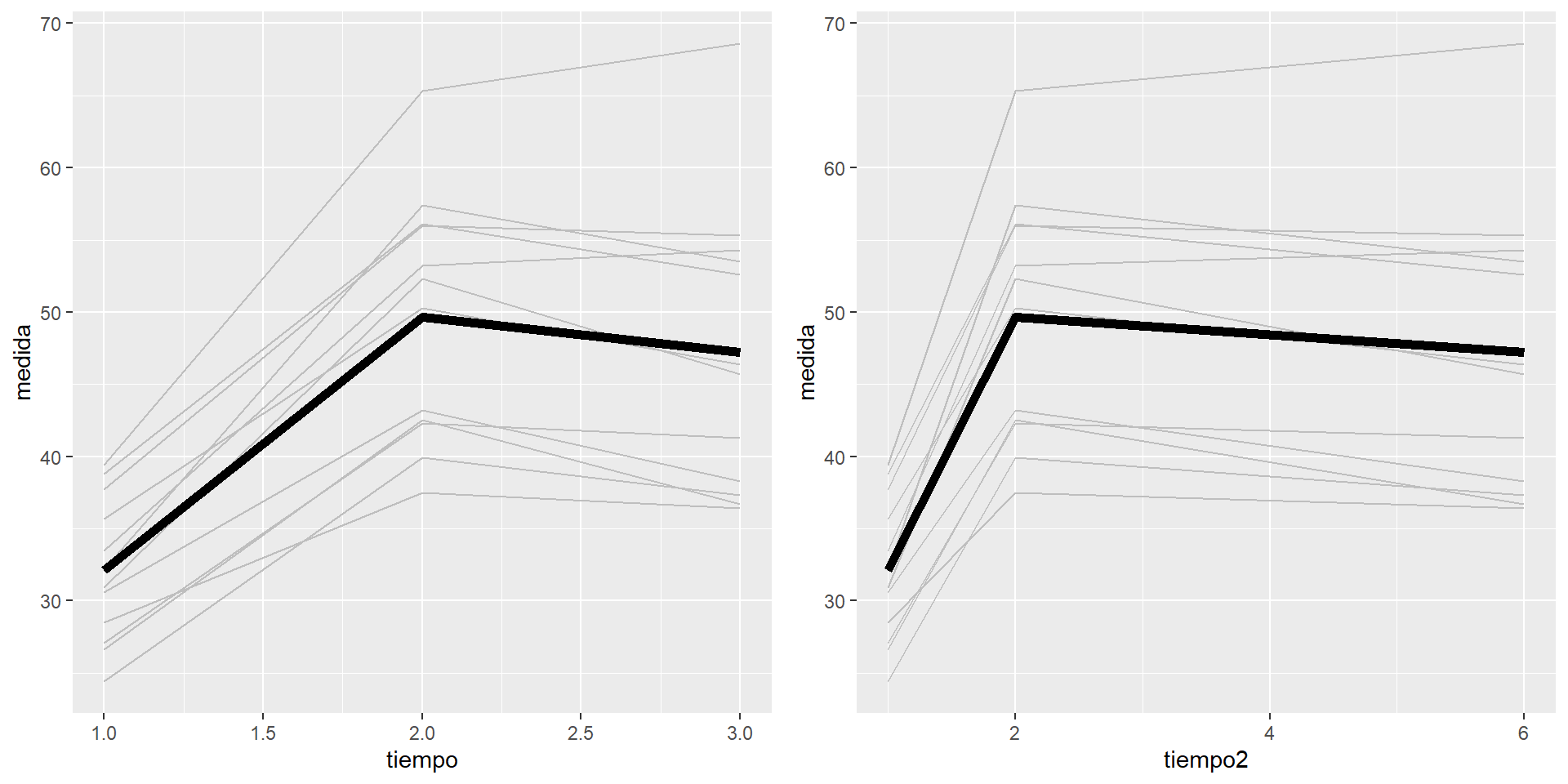
En la base de datos “Ejemplo_1W.csv” se tienen los datos de un diseño con 12 individuos en los que se toman los niveles en sangre de un cierto parámetro lipídico. Para cada invidivuo se miden los niveles a 1, 2 y 3 horas.
Ordenamos por individuo y dentro por tiempo dentro de individuo
4.5.1.1 Exploración de los datos
library(ggplot2)
p <- ggplot(data = datos, aes(x = tiempo, y = medida, group = indiv))
p + geom_line(col="grey") + stat_summary(aes(group = 1),
geom = "line", fun = mean, size=2)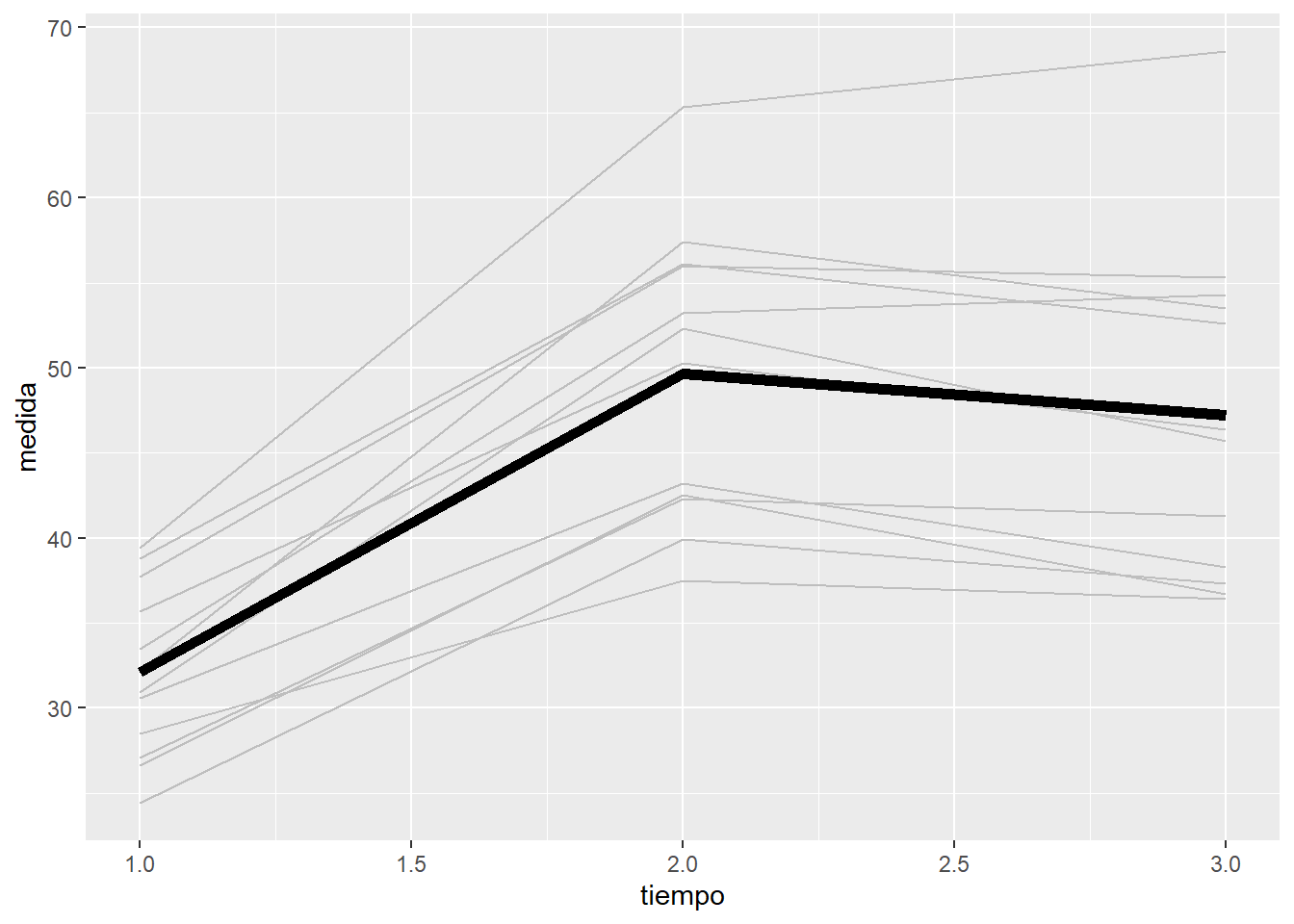
Cada línea representa a un individuo, mientras que la línea más gruesa es el promedio de los 12 individuos.
Vemos como el efecto del tiempo no es del todo lineal. Además las líneas están bastante separadas indicando variabilidad entre los individuos.
4.5.1.2 Modelo de respuesta multivariante
Para analizar los datos mediante el modelo de respuesta multivariante hay que disponer los datos de forma horizontal.
datosh <- reshape(data=datos,
direction="wide",
v.names=c("medida"),
times=1:3,
timevar="tiempo",
idvar="indiv")
datosh indiv medida.1 medida.2 medida.3
1 1 39.4 65.3 68.6
2 2 33.5 53.2 54.3
3 3 27.1 42.3 41.3
4 4 30.9 52.3 45.7
5 5 32.2 57.4 53.5
6 6 26.6 42.5 36.7
7 7 28.5 37.5 36.4
8 8 37.7 56.0 55.3
9 9 35.7 50.3 46.4
10 10 30.6 43.2 38.3
11 11 24.4 39.9 37.3
12 12 38.8 56.1 52.6Para ajustar un modelo de regresión lineal con respuesta multivariante se puede usar la función lm. Y hay que poner la variable respuesta a la izquierda de ~ como una matriz de las tres variables (medida.1, medida.2 y medida.3):
respuesta <- as.matrix(datosh[,c("medida.1","medida.2","medida.3")])
modelo <- lm(respuesta ~ 1, data=datosh)
class(modelo)[1] "mlm" "lm" Response medida.1 :
Call:
lm(formula = medida.1 ~ 1, data = datosh)
Residuals:
Min 1Q Median 3Q Max
-7.7167 -3.9667 -0.5667 4.0833 7.2833
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 32.117 1.445 22.23 1.72e-10 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 5.006 on 11 degrees of freedom
Response medida.2 :
Call:
lm(formula = medida.2 ~ 1, data = datosh)
Residuals:
Min 1Q Median 3Q Max
-12.167 -7.217 1.633 6.358 15.633
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 49.667 2.456 20.22 4.75e-10 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 8.509 on 11 degrees of freedom
Response medida.3 :
Call:
lm(formula = medida.3 ~ 1, data = datosh)
Residuals:
Min 1Q Median 3Q Max
-10.80 -9.15 -1.15 6.50 21.40
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 47.200 2.867 16.47 4.25e-09 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 9.93 on 11 degrees of freedomPara obtener la matriz de covarianzas de los residuos:
medida.1 medida.2 medida.3
medida.1 25.05970 36.58970 42.08727
medida.2 36.58970 72.39879 81.30000
medida.3 42.08727 81.30000 98.60364Y a partir de la matriz de covarianzas, se puede calcular fácilmente la matriz de correlaciones de los residuos:
medida.1 medida.2 medida.3
medida.1 1.0000000 0.8590239 0.8466744
medida.2 0.8590239 1.0000000 0.9622290
medida.3 0.8466744 0.9622290 1.0000000Para obtener los resultados se usa la función anova (?anova.mlm)
Analysis of Variance Table
Contrasts orthogonal to
~1
Df Pillai approx F num Df den Df Pr(>F)
(Intercept) 1 0.945 85.903 2 10 5.035e-07 ***
Residuals 11
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Los estadísticos disponibles (argumento test) son: “Pillai”, “Wilks”, “Hotelling-Lawley”, “Roy” o “Spherical”.
Con la opcion X=~1 se contrasta si \(\mu_1 = \mu_2 = \mu_3\). En cambio la opción por defecto X = ~ 0, contrasta \(\mu_1 = \mu_2 = \mu_3=0\) que no es de interés.
El término (Intercept) corresponde al efecto del tiempo.
Resultado
Hay efecto del tiempo porque el p-valor < 0.05.
4.5.1.3 Suma de cuadrados
Para ajustar este modelo hay que usar los datos en disposición vertical.
Además, hay que convertir las variables tiempo e indiv a factor.
library(ez)
datos.ez <- datos
datos.ez$tiempo <- factor(datos.ez$tiempo)
datos.ez$indiv <- factor(datos.ez$indiv)
ezANOVA(data=datos.ez, dv=medida, wid=indiv, within=tiempo, detailed = TRUE)$ANOVA
Effect DFn DFd SSn SSd F p p<.05
1 (Intercept) 1 11 66546.801 1892.0589 386.88796 6.390053e-10 *
2 tiempo 2 22 2166.376 264.6244 90.05264 2.542699e-11 *
ges
1 0.9686088
2 0.5011210
$`Mauchly's Test for Sphericity`
Effect W p p<.05
2 tiempo 0.4433135 0.01712201 *
$`Sphericity Corrections`
Effect GGe p[GG] p[GG]<.05 HFe p[HF] p[HF]<.05
2 tiempo 0.6423901 5.662497e-08 * 0.6905331 1.998401e-08 *La condición de esfericidad no se cumple dado que el test de Mauchly es significativo. Por lo tanto, hay que corregir los grados de libertad y, en consecuencia, el p-valor del factor tiempo. Después de la corrección, éste sigue siendo significativo.
Observación: Fíjate que los resultados son los mismos que usando la función lm, y anova con la opción test="Spherical":
Analysis of Variance Table
Contrasts orthogonal to
~1
Greenhouse-Geisser epsilon: 0.6424
Huynh-Feldt epsilon: 0.6905
Df F num Df den Df Pr(>F) G-G Pr H-F Pr
(Intercept) 1 90.053 2 22 2.5427e-11 5.6625e-08 1.9984e-08
Residuals 11 4.5.1.4 Modelo lineal mixto
Primero, ajustamos el modelo más complejo con constante y pendiente aleatoria, y añadimos el tiempo al cuadrado ya que vemos por el gráfico que la tendencia no es lineal.
modelo <- lme(fixed = medida ~ poly(tiempo, 2),
data=datos,
random = ~ poly(tiempo, 2) | indiv,
correlation = corAR1(form = ~ tiempo | indiv)
)
modelo$modelStruct$corStructCorrelation structure of class corAR1 representing
Phi
0.0001428372 Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
208.6888 225.1504 -93.34439
Random effects:
Formula: ~poly(tiempo, 2) | indiv
Structure: General positive-definite, Log-Cholesky parametrization
StdDev Corr
(Intercept) 7.526056 (Intr) p(,2)1
poly(tiempo, 2)1 14.559990 0.881
poly(tiempo, 2)2 5.178443 -0.663 -0.729
Residual 1.441564
Correlation Structure: AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.0001428372
Fixed effects: medida ~ poly(tiempo, 2)
Value Std.Error DF t-value p-value
(Intercept) 42.99444 2.185832 22 19.669598 0
poly(tiempo, 2)1 36.94647 4.443447 22 8.314823 0
poly(tiempo, 2)2 -28.30784 2.076632 22 -13.631610 0
Correlation:
(Intr) p(,2)1
poly(tiempo, 2)1 0.828
poly(tiempo, 2)2 -0.475 -0.497
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-1.0450552 -0.4346930 -0.1931843 0.5001057 1.1924184
Number of Observations: 36
Number of Groups: 12 - Valor esperado de la constante y coeficientes, \(\beta_0, \ldots, \beta_K\). También se conoce como los coeficientes fijos. Para obtener la tabla de sus estimaciones y los p-valores:
Value Std.Error DF t-value p-value
(Intercept) 42.99444 2.185832 22 19.669598 1.886788e-15
poly(tiempo, 2)1 36.94647 4.443447 22 8.314823 3.092000e-08
poly(tiempo, 2)2 -28.30784 2.076632 22 -13.631610 3.311649e-12- Estimación de los efectos aleatorios, \(\color{green}{(\beta_{0i}^{*},\beta_{1i}^{*},\beta_{2i}^{*})}\)
(Intercept) poly(tiempo, 2)1 poly(tiempo, 2)2
1 14.730441065 30.8722315 -6.5833764
2 4.054046210 11.0739532 -1.2028619
3 -5.907126225 -5.2717958 2.8619041
4 0.001779692 0.6665792 -3.8718583
5 4.796479128 13.7400443 -6.6013406
6 -7.601895827 -11.9016785 1.3545235
7 -8.829202865 -17.6630563 7.7680601
8 6.499328908 7.4984525 -0.9466624
9 0.924045128 -6.5624802 1.6842059
10 -5.693831230 -15.1669218 3.7578624
11 -8.860625561 -8.7246544 2.6200805
12 5.886561577 1.4393264 -0.8405368Hay una fila para cada individuo.
La función ranef retorna \(\hat{\theta}_{ki}\), donde \(\beta_{ki} = \beta_k + \theta_{ki}\). Así pues, \(\theta_{ki} \sim N\left(0,\sigma_{\beta_{k}}^2\right)\) se pueden interpretar como los “efectos aleatorios centrados” tal y como se ha escrito la ecuación del modelo.
- Matriz de covarianzas de la constante y coeficientes aleatorios, \(\Omega\):
Random effects variance covariance matrix
(Intercept) poly(tiempo, 2)1 poly(tiempo, 2)2
(Intercept) 56.642 96.553 -25.856
poly(tiempo, 2)1 96.553 211.990 -54.998
poly(tiempo, 2)2 -25.856 -54.998 26.816
Standard Deviations: 7.5261 14.56 5.1784 - Matriz de correlaciones de los residuos, \(\Sigma_i\)
Correlation structure of class corAR1 representing
Phi
0.0001428372 Podemos especificar que la correlación entre efectos aleatorios sea cero con la función pdDiag en el argumento random:
modelo2 <- lme(fixed = medida ~ poly(tiempo, 2),
data=datos,
random = list(indiv=pdDiag(~ poly(tiempo, 2))),
correlation = corAR1()
)
summary(modelo2)Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
217.8098 229.7819 -100.9049
Random effects:
Formula: ~poly(tiempo, 2) | indiv
Structure: Diagonal
(Intercept) poly(tiempo, 2)1 poly(tiempo, 2)2 Residual
StdDev: 0.00304907 9.921619 6.785107e-05 7.683502
Correlation Structure: AR(1)
Formula: ~1 | indiv
Parameter estimate(s):
Phi
0.896943
Fixed effects: medida ~ poly(tiempo, 2)
Value Std.Error DF t-value p-value
(Intercept) 42.99444 2.116748 22 20.311555 0
poly(tiempo, 2)1 36.94647 4.443466 22 8.314786 0
poly(tiempo, 2)2 -28.30784 2.065204 22 -13.707045 0
Correlation:
(Intr) p(,2)1
poly(tiempo, 2)1 0.000
poly(tiempo, 2)2 -0.098 0.000
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-1.58347912 -0.87272384 0.04840927 0.77580090 2.40352231
Number of Observations: 36
Number of Groups: 12 Random effects variance covariance matrix
(Intercept) poly(tiempo, 2)1 poly(tiempo, 2)2
(Intercept) 9.2968e-06 0.000 0.0000e+00
poly(tiempo, 2)1 0.0000e+00 98.439 0.0000e+00
poly(tiempo, 2)2 0.0000e+00 0.000 4.6038e-09
Standard Deviations: 0.0030491 9.9216 6.7851e-05 Y para contrastar esta asunción
Model df AIC BIC logLik Test L.Ratio p-value
modelo 1 11 208.6888 225.1504 -93.34439
modelo2 2 8 217.8098 229.7819 -100.90491 1 vs 2 15.12105 0.0017El mejor a escoger es el que contempla que hay correlación entre los efectos aleatorios.
Simplificación del modelo
Miramos primero si los coeficientes son aleatorios o fijos. Para ello comparamos el modelo completo con el modelo sólo con la constante aleatoria.
Model df AIC BIC logLik
modelo 1 11 208.6888 225.1504 -93.34439
update(modelo, random = ~1 | indiv) 2 6 214.6890 223.6680 -101.34449
Test L.Ratio p-value
modelo
update(modelo, random = ~1 | indiv) 1 vs 2 16.00021 0.0068Con la función anova se comparan los dos modelos mediante el LRT, uno con los coeficientes aleatorios y el otro sólo con la constante aleatoria. En este caso, y como se ha dicho, el LRT para constrastar si las varianzas son cero no es del todo adecuado. Existen otros tests basados en remuestreo, pero hasta la fecha no funcionan con lme y no se explicarán en este curso.
Basándonos en el LRT, y también el criterio AIC o BIC, se tiene que el modelo más complejo (el que supone que los coeficientes son aleatorios) es el que se eligirá.
Posteriormente miramos si la correlación entre los efectos aleatorios es cero o no:
Model df
modelo 1 11
update(modelo, random = list(indiv = pdDiag(~poly(tiempo, 2)))) 2 8
AIC
modelo 208.6888
update(modelo, random = list(indiv = pdDiag(~poly(tiempo, 2)))) 217.8098
BIC
modelo 225.1504
update(modelo, random = list(indiv = pdDiag(~poly(tiempo, 2)))) 229.7819
logLik
modelo -93.34439
update(modelo, random = list(indiv = pdDiag(~poly(tiempo, 2)))) -100.90491
Test L.Ratio
modelo
update(modelo, random = list(indiv = pdDiag(~poly(tiempo, 2)))) 1 vs 2 15.12105
p-value
modelo
update(modelo, random = list(indiv = pdDiag(~poly(tiempo, 2)))) 0.0017Nos quedamos con el modelo más complejo, ya que el p-valor del LRT es < 0.005.
Finalmente, miramos si podemos simplificar la matriz de correlación de los residuos. Comparamos mediante el LRT el modelo ajustado con uno que suponga independencia de los residuos:
Model df AIC BIC logLik Test
modelo 1 11 208.6888 225.1504 -93.34439
update(modelo, correlation = NULL) 2 10 206.6888 221.6539 -93.34439 1 vs 2
L.Ratio p-value
modelo
update(modelo, correlation = NULL) 3.668106e-10 1Como el p-valor > 0.05, elegimos el modelo más simple (el de independencia de los residuos). Además, según el criteria AIC, o BIC (cuánto más bajo mejor), también nos decantamos por el modelo de independencia de los residuos.
En el siguiente paso evaluamos la significación de los efectos fijos:
Value Std.Error DF t-value p-value
(Intercept) 42.99444 2.185834 22 19.669582 1.886821e-15
poly(tiempo, 2)1 36.94647 4.443441 22 8.314834 3.091930e-08
poly(tiempo, 2)2 -28.30784 2.076630 22 -13.631623 3.311588e-12Todos los coeficientes son significativos. Por lo tanto no podemos simplificar el modelo.
Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
206.6888 221.6539 -93.34439
Random effects:
Formula: ~poly(tiempo, 2) | indiv
Structure: General positive-definite, Log-Cholesky parametrization
StdDev Corr
(Intercept) 7.526075 (Intr) p(,2)1
poly(tiempo, 2)1 14.560044 0.881
poly(tiempo, 2)2 5.178188 -0.663 -0.729
Residual 1.441501
Fixed effects: medida ~ poly(tiempo, 2)
Value Std.Error DF t-value p-value
(Intercept) 42.99444 2.185834 22 19.669582 0
poly(tiempo, 2)1 36.94647 4.443441 22 8.314834 0
poly(tiempo, 2)2 -28.30784 2.076630 22 -13.631623 0
Correlation:
(Intr) p(,2)1
poly(tiempo, 2)1 0.828
poly(tiempo, 2)2 -0.475 -0.497
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-1.0451512 -0.4348223 -0.1931091 0.5002053 1.1925501
Number of Observations: 36
Number of Groups: 12 Finalmente, validamos el modelo:
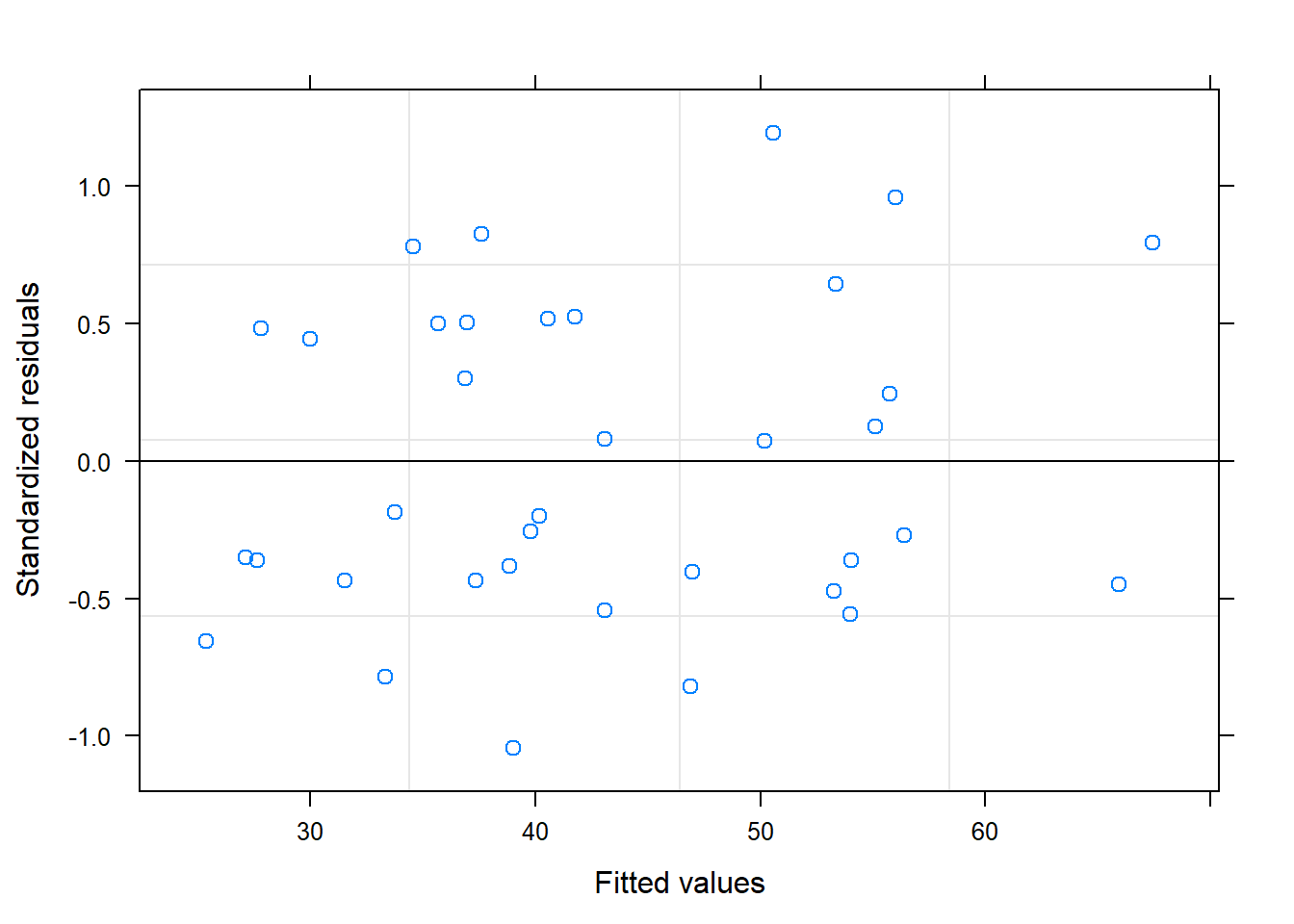
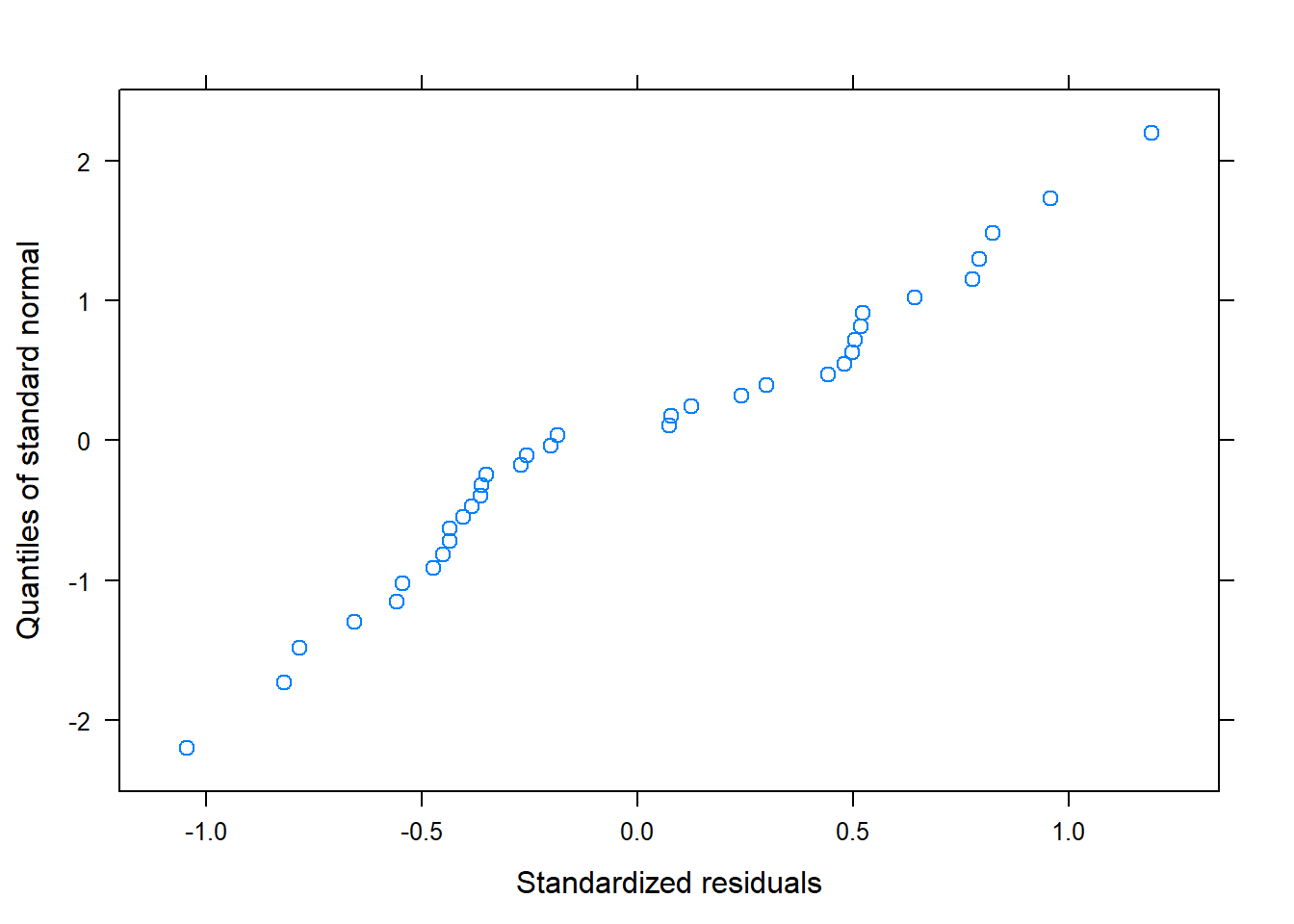
Según estos gráficos, diremos que sí se cumplen las premisas sobre los residuos.
Predicciones:
Para calcular las predicciones nos será útil usar las funciones del paquete ggeffects. Con este paquete se pueden realizar las predicciones de distinto tipo y también graficarlas con el paquete ggplot2.
# Predicted values of medida
# x = tiempo
x | Predicted | 95% CI
------------------------------
1 | 32.12 | [29.28, 34.95]
2 | 49.67 | [44.85, 54.48]
3 | 47.20 | [41.58, 52.82]
Adjusted for:
* indiv = 0 (population-level)
# Predicted values of medida
# x = tiempo
x | Predicted | 95% CI
------------------------------
1 | 32.12 | [28.12, 36.12]
2 | 49.67 | [44.08, 55.25]
3 | 47.20 | [40.91, 53.49]
Adjusted for:
* indiv = 0 (population-level)Con el argumento type="random", el intervalo es más ancho porque no sólo tiene en cuenta el error estándar de las estimaciones de los parámetros sinó también la varianza de los efectos aleatorios.
library(gridExtra)
grid.arrange(
plot(pr.fixed) + ylim(25,60) + ggtitle("CI: fixed"),
plot(pr.random) + ylim(25,60) + ggtitle("CI: random"),
nrow=1, ncol=2)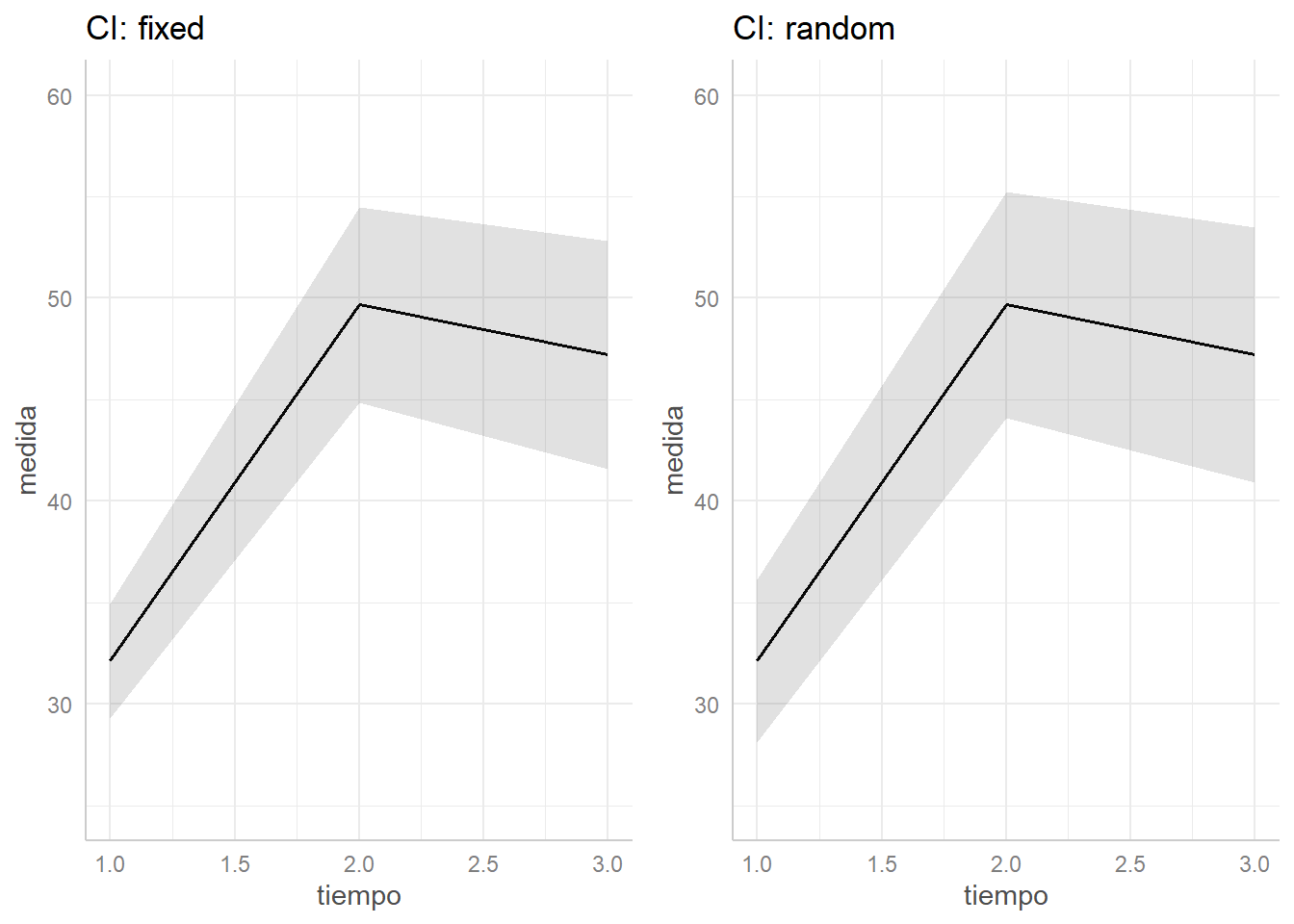
Resultado
Por lo tanto el modelo final contendrá el tiempo, el tiempo al cuadrado, la contaste y los coeficientes aleatorios. Finalmente, los residuos se puede suponer independientes.
Observaciones
- Se pueden incorporar términos splines en la fórmula usando la función
nsdel paquetespline. Es útil cuando se tienen muchas medidas repetidas y/o en distintos momentos para los diferentes individuos. Se usa en las fórmulas (argumentosfixedirandom)
Comparación con las otras técnicas
A fin de poder comparar los resultados de los modelo LMM con los modelos basados en la suma de cuadrados y en la respuesta multivariante, el tiempo se debe tratar como factor. Fíjate en el uso de as.factor para convertir una variable numérica a factor o variable categórica. Para ello, hay que tener las mismas categorías de tiempo para todos los individuos. Además, tanto los modelos de respuesta multivariable como los basados en suma de cuadrados, asumen la pendiente fija o constante, y la correlación sin estructura.
Nota: Es importante notar que las técnicas de respuesta multivariable y de suma de cuadrados al tratar la variable tiempo como factor, no se puede distinguir si tiempos de las medidas son o no equiespaiados. Por ejemplo los resultados obtenidos mediante estas dos técnicas serán los mismos tanto si se recogen las medidas a 1h, 2h y 3h, o si se recoge a 1h, 2h y 6h. En cambio, si se desea estudiar el efecto lineal ambas situaciones son muy distintas.
modelo <- lme(fixed = medida ~ as.factor(tiempo),
data=datos,
random = ~ 1 | indiv,
correlation = corSymm()
)
summary(modelo)Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
218.5233 230.4954 -101.2616
Random effects:
Formula: ~1 | indiv
(Intercept) Residual
StdDev: 7.069756 3.498312
Correlation Structure: General
Formula: ~1 | indiv
Parameter estimate(s):
Correlation:
1 2
2 0.081
3 -0.561 0.628
Fixed effects: medida ~ as.factor(tiempo)
Value Std.Error DF t-value p-value
(Intercept) 32.11667 2.277053 22 14.104488 0
as.factor(tiempo)2 17.55000 1.368874 22 12.820757 0
as.factor(tiempo)3 15.08333 1.784415 22 8.452817 0
Correlation:
(Intr) as.()2
as.factor(tiempo)2 -0.301
as.factor(tiempo)3 -0.392 0.880
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-1.95466422 -0.48588298 -0.07605957 0.47680669 2.08061488
Number of Observations: 36
Number of Groups: 12 Value Std.Error DF t-value p-value
(Intercept) 32.11667 2.277053 22 14.104488 1.683824e-12
as.factor(tiempo)2 17.55000 1.368874 22 12.820757 1.104451e-11
as.factor(tiempo)3 15.08333 1.784415 22 8.452817 2.338498e-08La función anova aplicada a un sólo modelo ajustado es útil para contrastar la significación de un factor de más de una categoría (o posibles interacciones de factores de más de dos categorías).
numDF denDF F-value p-value
(Intercept) 1 22 319.4667 <.0001
as.factor(tiempo) 2 22 99.9590 <.00014.5.2 Ejemplo 2
En la base de datos “Ejemplo_1W1B.csv” se tienen los datos de un estudio en el que participan 24 individuos randomizados en dos grupos de tratamiento (trat). Como en el anterior ejemplo, para cada invidivuo se miden los niveles a 1, 2 y 3 horas.
Como antes, ordenamos por individuo (de 1 a 24) y por tiempo
Fíjate que hay dos variables que codifican al individuo: la variable indiv va de 1 a 12 que son los individuos que hay dentro de cada grupo de tratamiento, mientras que indiv2 va de 1 a 24 que son el total de individuos.
4.5.2.1 Exploración de los datos
datos$trat <- factor(datos$trat, 1:2, c("Control","Tratados"))
library(ggplot2)
p <- ggplot(data = datos, aes(x = tiempo, y = medida, group = indiv2))
p <- p + geom_line(col="grey") + stat_summary(aes(group = 1),
geom = "line", fun = mean, size=2)
p + facet_grid( ~ trat)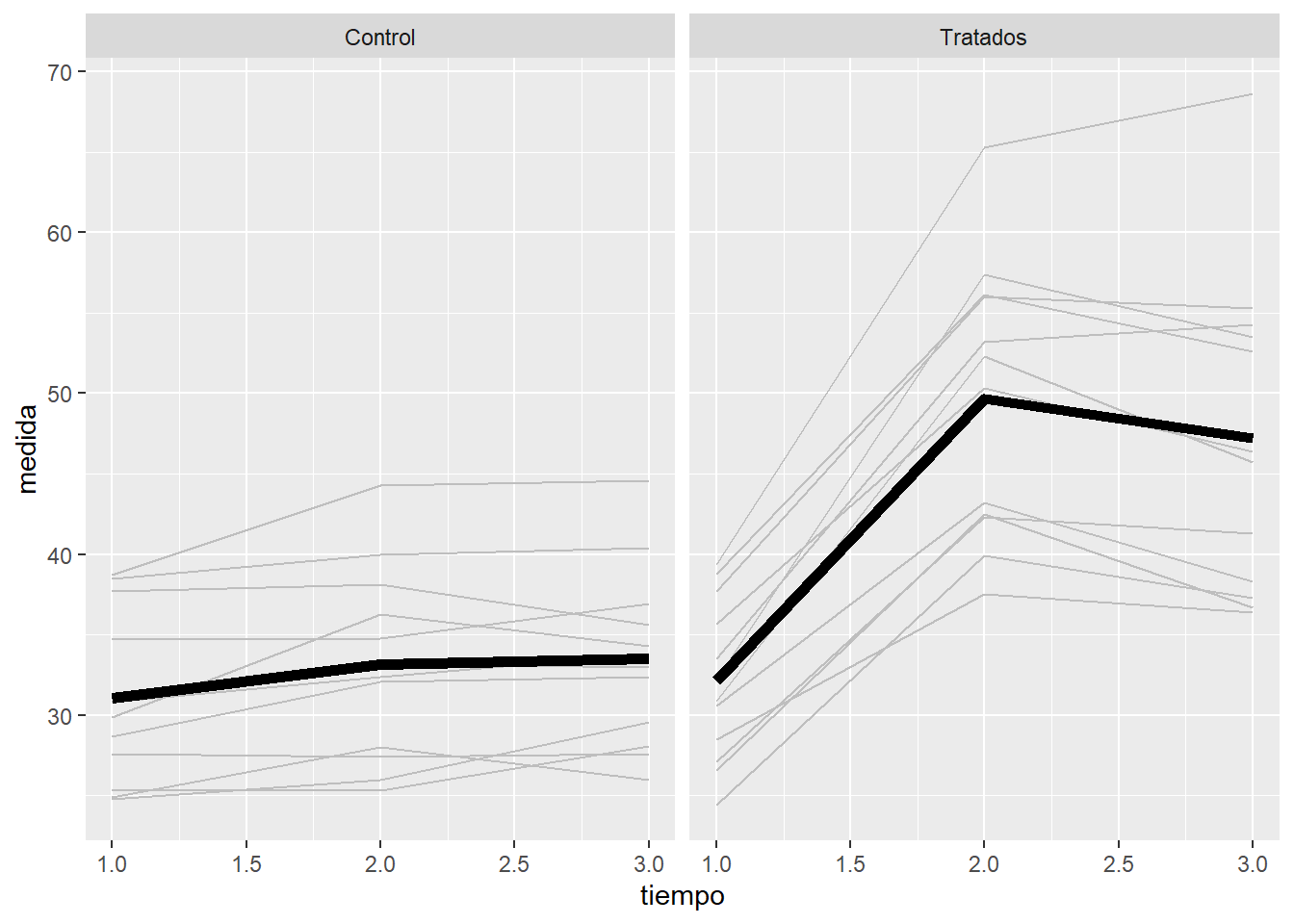
Para trat=1, la medida parece que no sube o sube muy poco. Mientras que para trat=2 sube mucho hasta la segunda medida y se estabiliza en la tercera medida. Por lo tanto, parece que sí hay una interacción entre el tiempo y el grupo de tratamiento.
4.5.2.2 Modelo de respuesta multivariante
Para analizar los datos mediante el modelo de respuesta multivariante, como antes hay que disponer los datos de forma horizontal.
datosh <- reshape(data=datos,
direction="wide",
v.names=c("medida"),
times=1:3,
timevar="tiempo",
idvar="indiv2")
datosh indiv trat indiv2 medida.1 medida.2 medida.3
1 1 Control 1 34.7 34.8 36.9
2 2 Control 2 38.7 44.3 44.6
3 3 Control 3 28.7 32.1 32.4
4 4 Control 4 30.8 32.4 33.8
5 5 Control 5 29.9 36.3 34.3
6 6 Control 6 27.6 27.4 27.6
7 7 Control 7 24.9 28.0 26.0
8 8 Control 8 37.7 38.1 35.6
9 9 Control 9 31.0 33.2 33.0
10 10 Control 10 25.4 25.3 28.1
11 11 Control 11 24.8 26.0 29.6
12 12 Control 12 38.5 40.0 40.4
37 1 Tratados 13 39.4 65.3 68.6
38 2 Tratados 14 33.5 53.2 54.3
39 3 Tratados 15 27.1 42.3 41.3
40 4 Tratados 16 30.9 52.3 45.7
41 5 Tratados 17 32.2 57.4 53.5
42 6 Tratados 18 26.6 42.5 36.7
43 7 Tratados 19 28.5 37.5 36.4
44 8 Tratados 20 37.7 56.0 55.3
45 9 Tratados 21 35.7 50.3 46.4
46 10 Tratados 22 30.6 43.2 38.3
47 11 Tratados 23 24.4 39.9 37.3
48 12 Tratados 24 38.8 56.1 52.6respuesta <- as.matrix(datosh[,c("medida.1","medida.2","medida.3")])
modelo <- lm(respuesta ~ trat, data=datosh)
summary(modelo)Response medida.1 :
Call:
lm(formula = medida.1 ~ trat, data = datosh)
Residuals:
Min 1Q Median 3Q Max
-7.7167 -3.9667 -0.7083 4.1271 7.6417
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 31.058 1.476 21.048 4.57e-16 ***
tratTratados 1.058 2.087 0.507 0.617
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 5.112 on 22 degrees of freedom
Multiple R-squared: 0.01156, Adjusted R-squared: -0.03337
F-statistic: 0.2572 on 1 and 22 DF, p-value: 0.6171
Response medida.2 :
Call:
lm(formula = medida.2 ~ trat, data = datosh)
Residuals:
Min 1Q Median 3Q Max
-12.1667 -6.6396 0.3375 5.2896 15.6333
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 33.158 2.113 15.692 1.98e-13 ***
tratTratados 16.508 2.988 5.524 1.50e-05 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 7.32 on 22 degrees of freedom
Multiple R-squared: 0.5811, Adjusted R-squared: 0.5621
F-statistic: 30.52 on 1 and 22 DF, p-value: 1.495e-05
Response medida.3 :
Call:
lm(formula = medida.3 ~ trat, data = datosh)
Residuals:
Min 1Q Median 3Q Max
-10.8000 -5.9062 -0.6625 5.6250 21.4000
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 33.525 2.310 14.511 9.55e-13 ***
tratTratados 13.675 3.267 4.186 0.000384 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 8.003 on 22 degrees of freedom
Multiple R-squared: 0.4433, Adjusted R-squared: 0.418
F-statistic: 17.52 on 1 and 22 DF, p-value: 0.0003835Analysis of Variance Table
Contrasts orthogonal to
~1
Df Pillai approx F num Df den Df Pr(>F)
(Intercept) 1 0.88537 81.102 2 21 1.326e-10 ***
trat 1 0.83628 53.633 2 21 5.599e-09 ***
Residuals 22
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Con la instrucción summary, contrasta si la media es diferente entre los dos grupos de tratamiento, y esto lo hace para cada momento por separado.
En la tabla ANOVA, el p-valor de término trat contrasta si el efecto del tiempo es el mismo para los dos tratamientos, o sea, la interacción tratamiento y tiempo, que es lo que nos interesa. Mientras que el término (Intercept) corresponde al efecto marginal del tiempo.
Fíjate qué pasa si no se especifica el argumento X:
Analysis of Variance Table
Df Pillai approx F num Df den Df Pr(>F)
(Intercept) 1 0.97752 289.829 3 20 < 2.2e-16 ***
trat 1 0.84439 36.176 3 20 2.854e-08 ***
Residuals 22
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1En este caso, el p-valor del tratamiento contrasta si hay diferencias entre tratamientos en alguno de los momentos (hay 3 grados de libertad). Y este contraste no es la interacción entre tratamiento y tiempo.
4.5.2.3 Suma de cuadrados
Para ajustar este modelo hay que usar los datos en disposición vertical.
Como antes hay que convertir las variables tiempo, indiv2 y trat a factor.
library(ez)
datos.ez <- datos
datos.ez$tiempo <- factor(datos.ez$tiempo)
datos.ez$indiv2 <- factor(datos.ez$indiv2)
datos.ez$trat <- factor(datos.ez$trat)
ezANOVA(data=datos.ez,
dv=medida,
wid=indiv,
within=tiempo,
between=trat,
detailed = TRUE)$ANOVA
Effect DFn DFd SSn SSd F p p<.05
1 (Intercept) 1 22 102808.4513 2849.9219 793.63083 9.363797e-19 *
2 trat 1 22 1952.0835 2849.9219 15.06913 8.040878e-04 *
3 tiempo 2 44 1397.0700 312.7422 98.27755 5.878117e-17 *
4 trat:tiempo 2 44 811.8211 312.7422 57.10794 5.921847e-13 *
ges
1 0.9701554
2 0.3816578
3 0.3063929
4 0.2042582
$`Mauchly's Test for Sphericity`
Effect W p p<.05
3 tiempo 0.5725954 0.002866835 *
4 trat:tiempo 0.5725954 0.002866835 *
$`Sphericity Corrections`
Effect GGe p[GG] p[GG]<.05 HFe p[HF] p[HF]<.05
3 tiempo 0.7005722 1.520842e-12 * 0.7336894 4.932994e-13 *
4 trat:tiempo 0.7005722 1.003472e-09 * 0.7336894 4.400536e-10 *Vemos como se aplican las correcciones sólo en los términos “intra sujeto” que son tiempo y la interacción trat:tiempo, ya que el test de Mauchly es significativo (p-valor < 0.05).
Una vez aplicados las correcciones sobre los grados de libertad, los p-valores cambian aunque las conclusiones son las mismas: tanto el efecto del tiempo como su interacción con el tratamiento son significativos.
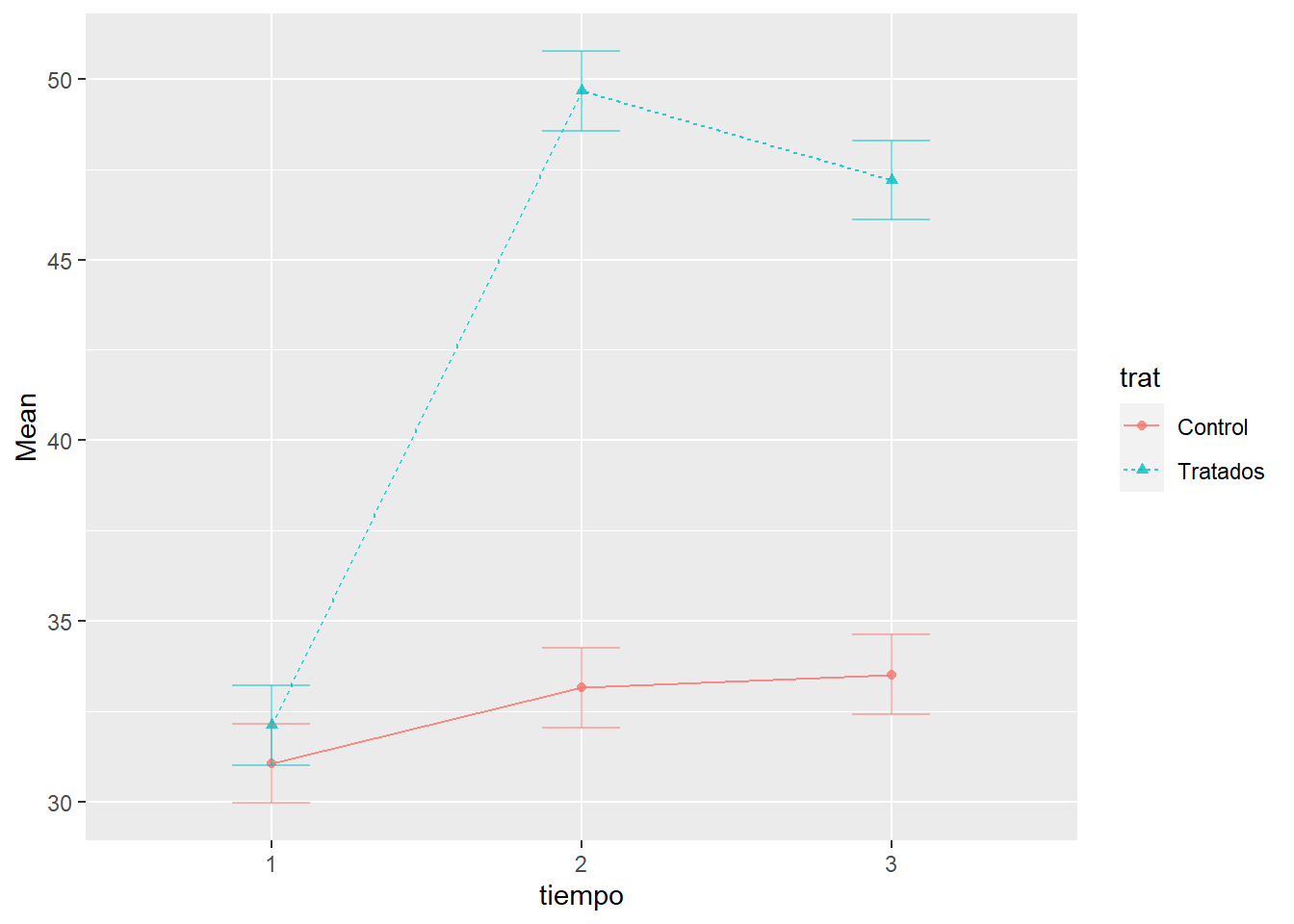
Las conclusiones con la tabla ANOVA corregida (tanto por GG como por HF), se ven claramente en el gráfico de interacción.
Nota
Este resultado es el mismo que se obtiene mediante la función anova del método multivariante, especificando test="Sphericity":
Analysis of Variance Table
Contrasts orthogonal to
~1
Greenhouse-Geisser epsilon: 0.7006
Huynh-Feldt epsilon: 0.7337
Df F num Df den Df Pr(>F) G-G Pr H-F Pr
(Intercept) 1 98.278 2 44 6.0000e-17 1.5200e-12 4.9000e-13
trat 1 57.108 2 44 5.9218e-13 1.0035e-09 4.4005e-10
Residuals 22 4.5.2.4 Modelo lineal mixto
modelo <- lme(fixed = medida ~ poly(tiempo,2)*trat,
data=datos,
random = ~ poly(tiempo,2) | indiv2,
correlation = corAR1()
)
summary(modelo)Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
386.914 417.5692 -179.457
Random effects:
Formula: ~poly(tiempo, 2) | indiv2
Structure: General positive-definite, Log-Cholesky parametrization
StdDev Corr
(Intercept) 6.528817 (Intr) p(,2)1
poly(tiempo, 2)1 15.016142 0.720
poly(tiempo, 2)2 5.983554 -0.637 -0.653
Residual 1.290085
Correlation Structure: AR(1)
Formula: ~1 | indiv2
Parameter estimate(s):
Phi
5.459222e-05
Fixed effects: medida ~ poly(tiempo, 2) * trat
Value Std.Error DF t-value p-value
(Intercept) 32.58056 1.896933 44 17.175384 0.0000
poly(tiempo, 2)1 8.54478 4.703086 44 1.816846 0.0761
poly(tiempo, 2)2 -3.46667 2.512364 44 -1.379842 0.1746
tratTratados 10.41389 2.682669 22 3.881914 0.0008
poly(tiempo, 2)1:tratTratados 43.70542 6.651168 44 6.571089 0.0000
poly(tiempo, 2)2:tratTratados -36.56667 3.553020 44 -10.291715 0.0000
Correlation:
(Intr) pl(,2)1 pl(,2)2 trtTrt p(,2)1:
poly(tiempo, 2)1 0.659
poly(tiempo, 2)2 -0.435 -0.414
tratTratados -0.707 -0.466 0.308
poly(tiempo, 2)1:tratTratados -0.466 -0.707 0.293 0.659
poly(tiempo, 2)2:tratTratados 0.308 0.293 -0.707 -0.435 -0.414
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-1.17636038 -0.41451565 -0.05512437 0.37021865 1.36156338
Number of Observations: 72
Number of Groups: 24 Nota:
Si los individuos estuvieran anidados dentro de clusters, se especificaría en el argumento random = ~ 1 | indiv / clusters, donde “cluster” sería el nombre de la variable que codifica los clusters.
Observación Para que el modelo quede bien definido no es posible poner la interacción del tiempo y el tratamiento como coeficiente aleatorio. De esta manera se especifican como aleatorios la costante y los coeficientes del tiempo (lineal y cuadrático) para el grupo control.
Como en el anterior ejemplo, contrastamos la significación de los coeficientes aleatorios del tiempo:
Model df AIC BIC logLik Test
modelo 1 14 386.9140 417.5692 -179.457
update(modelo, random = ~1 | indiv2) 2 9 393.6099 413.3168 -187.805 1 vs 2
L.Ratio p-value
modelo
update(modelo, random = ~1 | indiv2) 16.69594 0.0051Según el criterio AIC o BIC, el modelo con pendientes aleatorias es mejor.
Luego, contrastamos si se puede simplificar la matriz de correlaciones de los efectos aleatorios:
Model df
modelo 1 14
update(modelo, random = list(indiv2 = pdDiag(~poly(tiempo, 2)))) 2 11
AIC
modelo 386.9140
update(modelo, random = list(indiv2 = pdDiag(~poly(tiempo, 2)))) 397.1845
BIC
modelo 417.5692
update(modelo, random = list(indiv2 = pdDiag(~poly(tiempo, 2)))) 421.2707
logLik
modelo -179.4570
update(modelo, random = list(indiv2 = pdDiag(~poly(tiempo, 2)))) -187.5922
Test
modelo
update(modelo, random = list(indiv2 = pdDiag(~poly(tiempo, 2)))) 1 vs 2
L.Ratio
modelo
update(modelo, random = list(indiv2 = pdDiag(~poly(tiempo, 2)))) 16.27047
p-value
modelo
update(modelo, random = list(indiv2 = pdDiag(~poly(tiempo, 2)))) 0.001El test LRT es significativo (p-valor < 0.05). Por lo tanto nos quedamos con el modelo más complejo que supone que hay correlación entre los efectos aleatorios.
Seguidamente, miramos si se puede simplificar la matriz de correlaciones de los errores.
Model df AIC BIC logLik Test
modelo 1 14 386.914 417.5692 -179.457
update(modelo, correlation = NULL) 2 13 384.914 413.3795 -179.457 1 vs 2
L.Ratio p-value
modelo
update(modelo, correlation = NULL) 1.169383e-08 0.9999Sí que se puede suponer que hay indipendencia entre los residuos.
Por lo tanto el modelo final, que supone independencia entre residuos, tiene la siguiente estimación de los efectos fijos:
Value Std.Error DF t-value p-value
(Intercept) 32.580556 1.896946 44 17.175271 3.966321e-21
poly(tiempo, 2)1 8.544784 4.703101 44 1.816840 7.605690e-02
poly(tiempo, 2)2 -3.466667 2.512343 44 -1.379854 1.746035e-01
tratTratados 10.413889 2.682686 22 3.881888 8.041051e-04
poly(tiempo, 2)1:tratTratados 43.705415 6.651190 44 6.571067 4.875992e-08
poly(tiempo, 2)2:tratTratados -36.566667 3.552990 44 -10.291803 2.730972e-13Vemos como el efecto del tiempo para el grupo control no llega a ser significativo (p-valores >0.05) tanto para su componente lineal como cuadrático. Hay efecto del tratamiento en el momento basal (trat2).
Si quieremos ver el efecto del tiempo para el grupo 2, hay que cambiar su categoría de referencia.
Value Std.Error DF t-value p-value
(Intercept) 42.99444 1.896932 44 22.665259 6.756030e-26
poly(tiempo, 2)1 52.25020 4.703133 44 11.109658 2.358252e-14
poly(tiempo, 2)2 -40.03333 2.512374 44 -15.934465 6.870764e-20
tratControl -10.41389 2.682666 22 -3.881917 8.040486e-04
poly(tiempo, 2)1:tratControl -43.70542 6.651235 44 -6.571023 4.876723e-08
poly(tiempo, 2)2:tratControl 36.56667 3.553033 44 10.291676 2.732023e-13Vemos que para el grupo 2 tanto la componente lineal como la cuadrática del tiempo son significativas.
Con la siguiente matriz de varianzas y covarianzas de los efectos aleatorios:
Random effects variance covariance matrix
(Intercept) poly(tiempo, 2)1 poly(tiempo, 2)2
(Intercept) 42.626 70.565 -24.889
poly(tiempo, 2)1 70.565 225.490 -58.668
poly(tiempo, 2)2 -24.889 -58.668 35.800
Standard Deviations: 6.5289 15.016 5.9833 Y varianza de los residuos
[1] 1.664249Por último, validamos el modelo
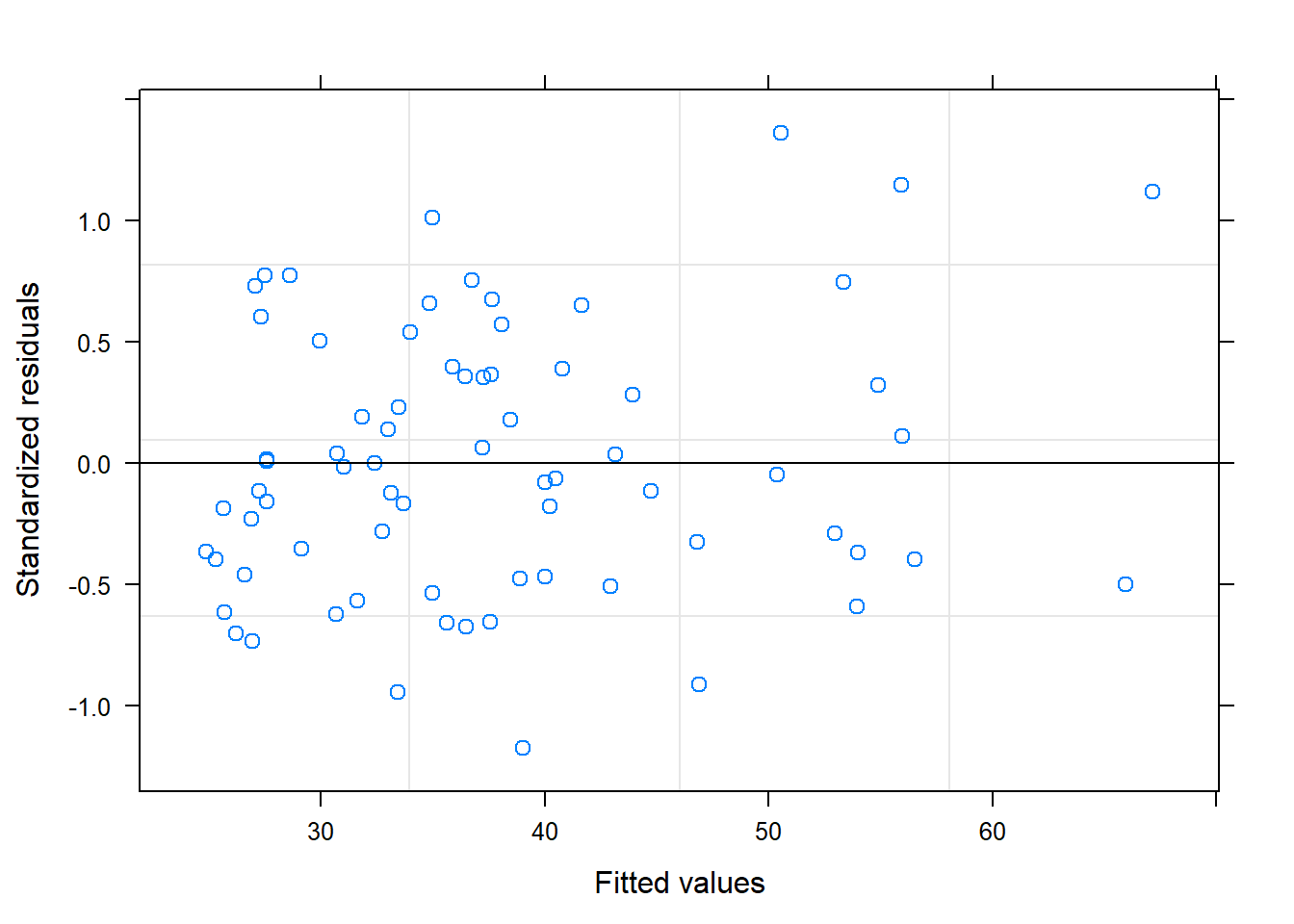
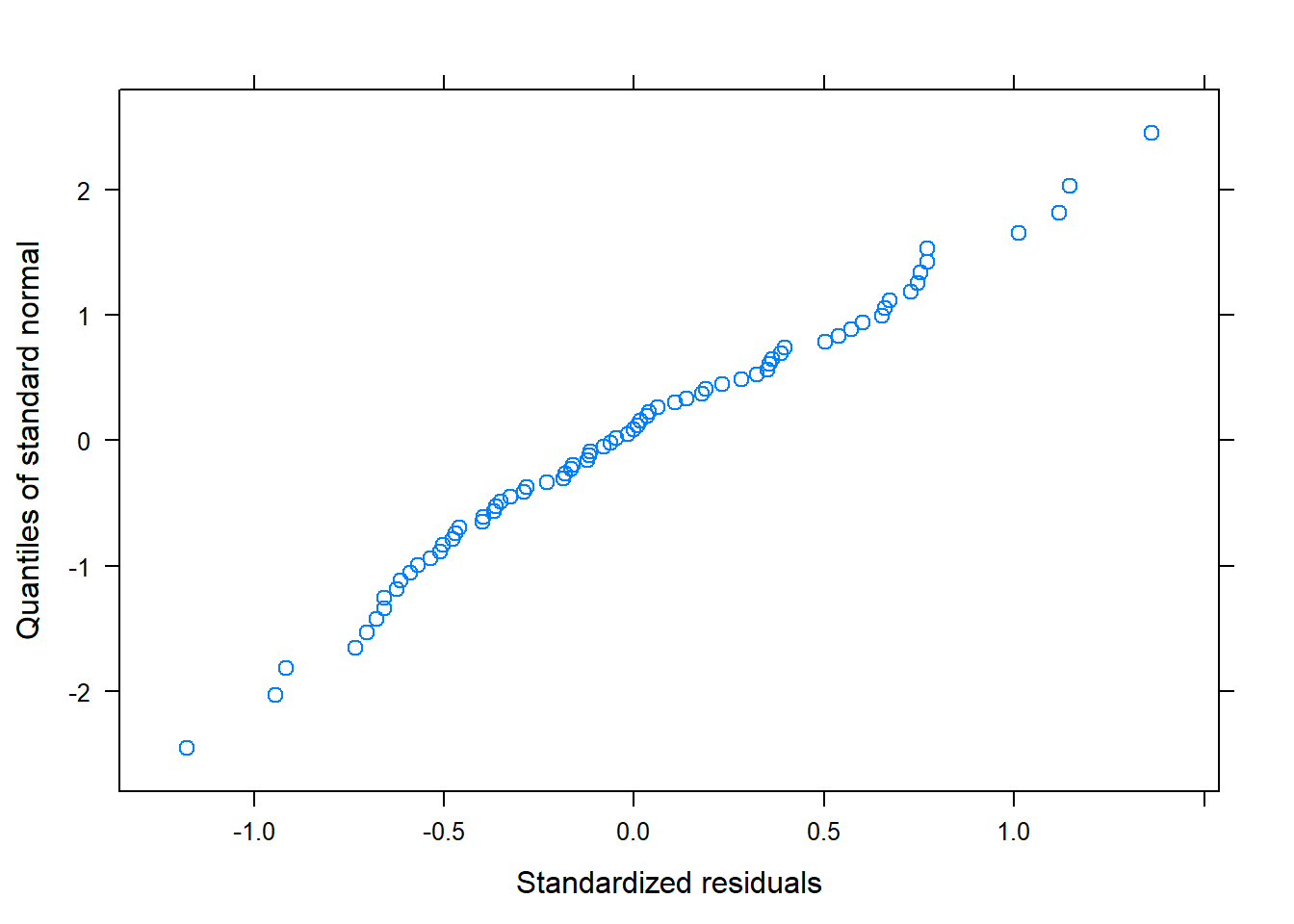
Según los gráficos, parece que sí que se cumplen las premisas sobre los residuos.
Predicciones
# Predicted values of medida
# x = tiempo
# trat = Control
x | Predicted | 95% CI
------------------------------
1 | 31.06 | [28.17, 33.95]
2 | 33.16 | [29.02, 37.30]
3 | 33.52 | [29.00, 38.05]
# trat = Tratados
x | Predicted | 95% CI
------------------------------
1 | 32.12 | [29.22, 35.01]
2 | 49.67 | [45.53, 53.81]
3 | 47.20 | [42.67, 51.73]
Adjusted for:
* indiv2 = 0 (population-level)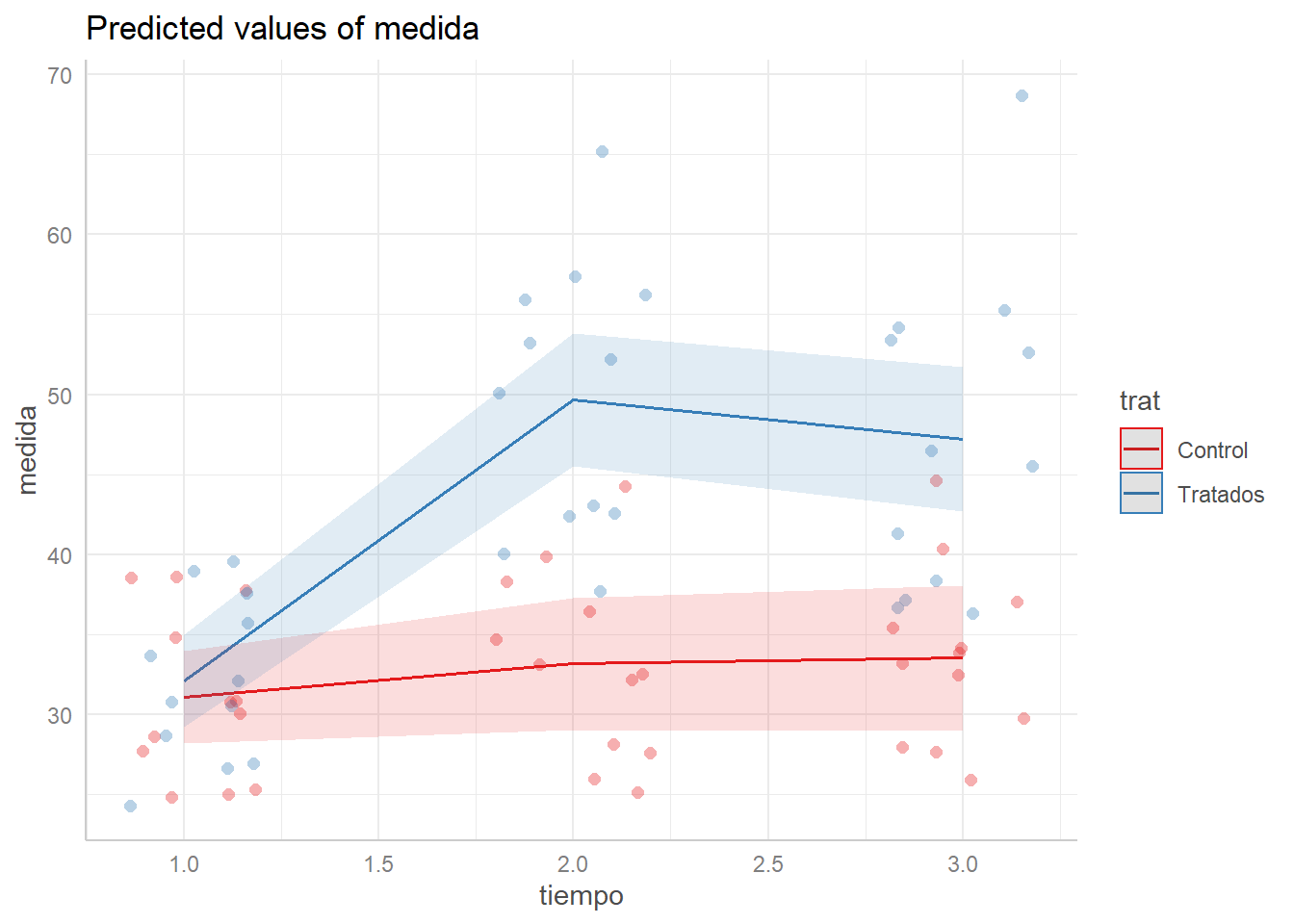
4.5.3 Ejemplo 3
En un estudio se quieren comparar el efecto de régimen de ejercicio sobre el sobrepeso. Para ello se reclutan 100 personas. A la mitad se le asigna el régimen y al resto se le hacen algunas recomendaciones (grupo control). Se mide el índice de masa corporal justo antes de empezar el estudio (momento basal), y al cabo de 1, 2 y 3 semanas. Como la edad es una variable importante para predecir el IMC también se registra.
Los datos los encontrarás en el fichero “imc.csv”
En este ejemplo, vemos como en algunos de los individuos nos falta alguna medida en a partir de la primera semana. Por este motivo usaremos la técnica de los LMM.
Nos aseguramos que los datos estén ordenados por individuo y tiempo
respuesta indiv tiempo edad tx
Min. : 9.80 Min. : 1.00 Min. :0.00 Min. :25.00 Control :200
1st Qu.:27.02 1st Qu.: 25.75 1st Qu.:0.75 1st Qu.:43.00 Tratados:200
Median :30.75 Median : 50.50 Median :1.50 Median :49.00
Mean :30.46 Mean : 50.50 Mean :1.50 Mean :49.03
3rd Qu.:34.60 3rd Qu.: 75.25 3rd Qu.:2.25 3rd Qu.:57.00
Max. :43.70 Max. :100.00 Max. :3.00 Max. :69.00
NA's :50 [1] 42Elimino las observaciones con valores missing
datos <- subset(datos, !is.na(respuesta))
# número de individuos con 2, 3 o 4 medidas válidas.
table(table(datos$indiv))
2 3 4
8 34 58 library(ggplot2)
p <- ggplot(data = datos, aes(x = tiempo, y = respuesta, group = indiv))
p <- p + geom_line(col="grey") + stat_summary(aes(group = 1),
geom = "line", fun = mean, size=2)
p + facet_grid( ~ tx)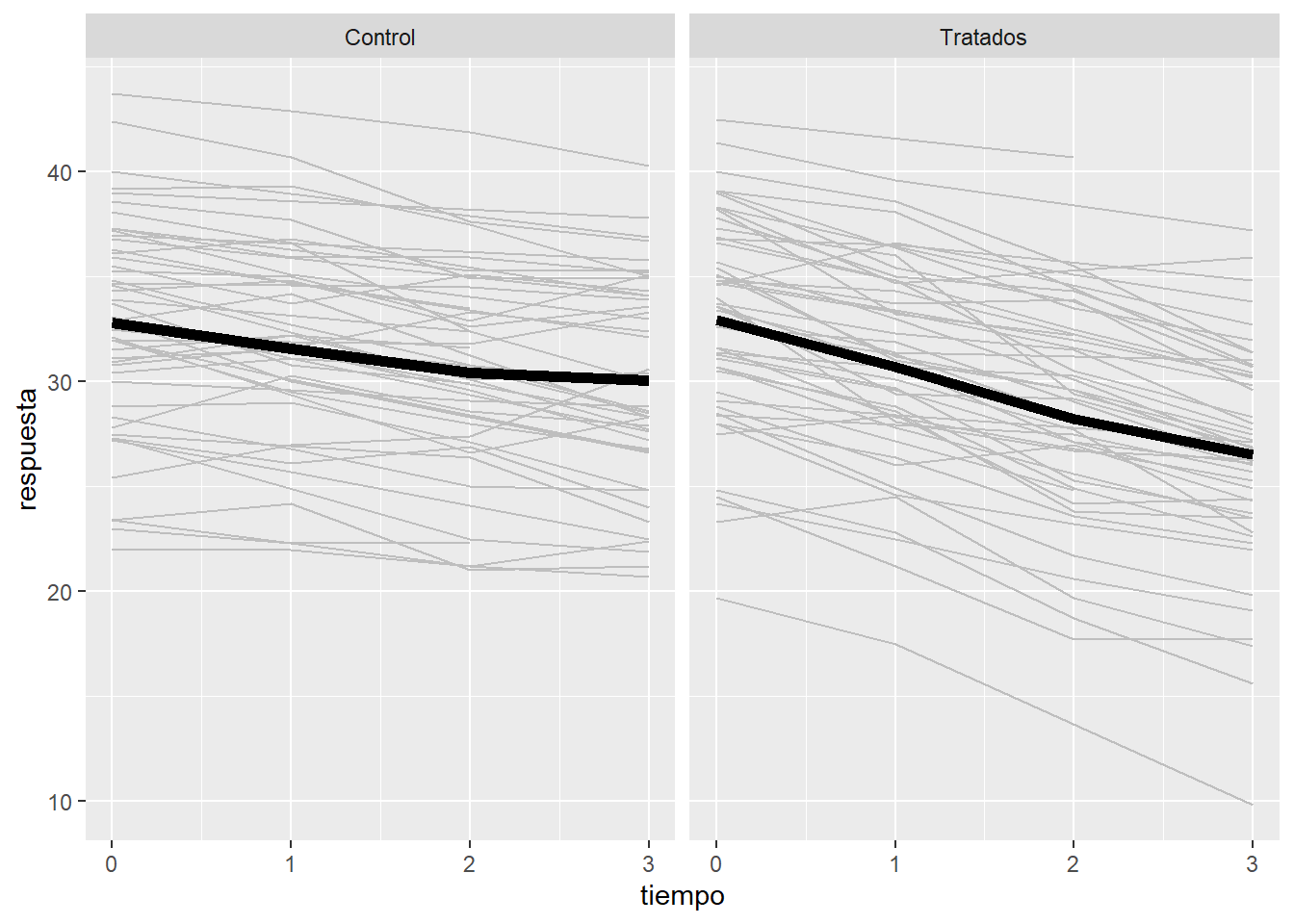
4.5.3.1 Análisis del grupo control
Si analizamos sólo el grupo control, se trata de un diseño 1W con una covariable (edad).
Ajustamos el modelo más completo, con la edad y el tiempo hasta el término cúbico ya que tenemos cuatro medidas.
library(nlme)
modelo <- lme(respuesta ~ poly(tiempo,3) + edad,
random= ~ poly(tiempo,3) | indiv,
data=datos,
#correlation = corSymm(), # sin estructura
correlation=corCAR1(form = ~ tiempo | indiv),
control=lmeControl(opt="optim"))
summary(modelo)Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
672.3888 724.9826 -319.1944
Random effects:
Formula: ~poly(tiempo, 3) | indiv
Structure: General positive-definite, Log-Cholesky parametrization
StdDev Corr
(Intercept) 2.1235727 (Intr) p(,3)1 p(,3)2
poly(tiempo, 3)1 10.8729435 -0.143
poly(tiempo, 3)2 5.3455110 0.317 0.071
poly(tiempo, 3)3 4.2631158 0.057 -0.026 0.193
Residual 0.5431964
Correlation Structure: Continuous AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.1999231
Fixed effects: respuesta ~ poly(tiempo, 3) + edad
Value Std.Error DF t-value p-value
(Intercept) 10.409728 1.4770932 115 7.047442 0.0000
poly(tiempo, 3)1 -15.045596 1.6657189 115 -9.032494 0.0000
poly(tiempo, 3)2 -0.518758 1.0075975 115 -0.514846 0.6076
poly(tiempo, 3)3 1.865008 0.8918255 115 2.091225 0.0387
edad 0.432945 0.0297828 48 14.536718 0.0000
Correlation:
(Intr) p(,3)1 p(,3)2 p(,3)3
poly(tiempo, 3)1 -0.024
poly(tiempo, 3)2 0.081 0.089
poly(tiempo, 3)3 0.012 -0.041 0.149
edad -0.978 -0.001 -0.037 -0.003
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-1.025920132 -0.293663743 -0.008367651 0.290693758 1.093099300
Number of Observations: 168
Number of Groups: 50 El modelo de correlación sin estructura no converge. Es normal ya que tenemos distintas medidas.
Es importante especificar la AR(1) continua corCAR1 ya que tenemos algunos individuos con datos faltantes en algunas de sus medidas. Luego el tiempo que ha pasado entre las medidas disponibles hay que tenerlo en cuenta.
Fíjate en la varianza de los efectos aleatorios, sobretodo en la constante si en el modelo no ponemos la edad,
Random effects variance covariance matrix
(Intercept) poly(tiempo, 3)1 poly(tiempo, 3)2 poly(tiempo, 3)3
(Intercept) 4.5096 -3.2917 3.6035 0.5144
poly(tiempo, 3)1 -3.2917 118.2200 4.1274 -1.1839
poly(tiempo, 3)2 3.6035 4.1274 28.5740 4.4056
poly(tiempo, 3)3 0.5144 -1.1839 4.4056 18.1740
Standard Deviations: 2.1236 10.873 5.3455 4.2631 Random effects variance covariance matrix
(Intercept) poly(tiempo, 3)1 poly(tiempo, 3)2 poly(tiempo, 3)3
(Intercept) 22.65700 -2.56910 0.79669 0.60630
poly(tiempo, 3)1 -2.56910 108.79000 2.88170 0.23627
poly(tiempo, 3)2 0.79669 2.88170 20.71700 3.90920
poly(tiempo, 3)3 0.60630 0.23627 3.90920 13.19500
Standard Deviations: 4.76 10.43 4.5516 3.6326 Es importante poner la edad ya que si no, la varianza de los efectos aleatorios quedan infladas y la inferencia no es válida.
Contraste de los efectos aleatorios
Model df AIC BIC logLik Test
modelo 1 17 672.3888 724.9826 -319.1944
update(modelo, random = ~1 | indiv) 2 8 662.1079 686.8579 -323.0539 1 vs 2
L.Ratio p-value
modelo
update(modelo, random = ~1 | indiv) 7.719062 0.5627Nos quedamos con el modelo con los coeficientes del tiempo fijos:
Observación Fíjate en el valor de \(\phi\) de la matriz de correlaciones de los errores: al considerar el coeficiente del tiempo como fijo, ha pasado de ser cero a un valor alto. Al considerar el coeficiente aleatorio en cierta manera se inducía una estructura de AR entre las observaciones y ya no hacía falta considerar los errores correlacionados. Por esto, el orden en que se simplifica el modelo es importante.
Si queremos contrastar si la constante es aletoria se compara el modelo con todos los efectos fijos. Así que ya no se podrá usar la función lme sino que se usará la función gls en su lugar.
modelo.gls <- gls(respuesta ~ poly(tiempo,3) + edad,
data=datos,
correlation=corCAR1(form = ~ tiempo |indiv))
summary(modelo.gls)Generalized least squares fit by REML
Model: respuesta ~ poly(tiempo, 3) + edad
Data: datos
AIC BIC logLik
660.2088 681.8651 -323.1044
Correlation Structure: Continuous AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.8409838
Coefficients:
Value Std.Error t-value p-value
(Intercept) 10.743564 1.5857081 6.775247 0.0000
poly(tiempo, 3)1 -15.049949 1.6527760 -9.105861 0.0000
poly(tiempo, 3)2 -0.382757 1.0563295 -0.362347 0.7176
poly(tiempo, 3)3 1.978662 0.8789044 2.251283 0.0257
edad 0.425951 0.0319656 13.325311 0.0000
Correlation:
(Intr) p(,3)1 p(,3)2 p(,3)3
poly(tiempo, 3)1 0.001
poly(tiempo, 3)2 -0.022 0.019
poly(tiempo, 3)3 0.003 -0.121 0.024
edad -0.980 -0.002 -0.008 -0.001
Standardized residuals:
Min Q1 Med Q3 Max
-2.39865570 -0.70572628 -0.01464924 0.74989249 2.07647314
Residual standard error: 2.492824
Degrees of freedom: 168 total; 163 residualLinear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
662.1079 686.8579 -323.0539
Random effects:
Formula: ~1 | indiv
(Intercept) Residual
StdDev: 0.8028047 2.378488
Correlation Structure: Continuous AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.8266025
Fixed effects: respuesta ~ poly(tiempo, 3) + edad
Value Std.Error DF t-value p-value
(Intercept) 10.738507 1.6033545 115 6.697525 0.0000
poly(tiempo, 3)1 -15.048689 1.6348726 115 -9.204808 0.0000
poly(tiempo, 3)2 -0.382488 1.0548435 115 -0.362602 0.7176
poly(tiempo, 3)3 1.978466 0.8786767 115 2.251643 0.0262
edad 0.426057 0.0323229 48 13.181289 0.0000
Correlation:
(Intr) p(,3)1 p(,3)2 p(,3)3
poly(tiempo, 3)1 0.001
poly(tiempo, 3)2 -0.020 0.019
poly(tiempo, 3)3 0.003 -0.122 0.024
edad -0.980 -0.002 -0.008 -0.001
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-2.25670886 -0.66026076 0.01991706 0.66013218 1.92440305
Number of Observations: 168
Number of Groups: 50 Model df AIC BIC logLik Test L.Ratio p-value
modelo 1 8 662.1079 686.8579 -323.0539
modelo.gls 2 7 660.2088 681.8651 -323.1044 1 vs 2 0.1009386 0.7507Los dos modelos no están anidados. Así que a parte del p-valor del LRT también miraremos el AIC y el BIC. Bajo los tres criterios nos decantamos por el modelo con la constante aleatoria.
Estructura de correlación de los errores
Comparamos con la matriz de independencia
Model df AIC BIC logLik Test
modelo 1 8 662.1079 686.8579 -323.0539
update(modelo, correlation = NULL) 2 7 677.6130 699.2693 -331.8065 1 vs 2
L.Ratio p-value
modelo
update(modelo, correlation = NULL) 17.50515 <.0001Nos quedamos con la estructura AR1
Contraste de los efectos fijos
Value Std.Error DF t-value p-value
(Intercept) 10.7385066 1.6033545 115 6.697525 8.187045e-10
poly(tiempo, 3)1 -15.0486889 1.6348726 115 -9.204808 1.848306e-15
poly(tiempo, 3)2 -0.3824884 1.0548435 115 -0.362602 7.175678e-01
poly(tiempo, 3)3 1.9784662 0.8786767 115 2.251643 2.624396e-02
edad 0.4260575 0.0323229 48 13.181289 1.441756e-17Vemos como la parte cuadrática no es significativa y la cúbico tampoco. Para contrastar los dos términos (cuadrático y cúbico) a la vez comparamos mediante el LRT el modelo completo con el modelo que supone el efecto del tiempo lineal
anova(
update(modelo, method="ML"),
update(modelo, fixed = . ~ . - poly(tiempo,3) + tiempo, method="ML")
) Model
update(modelo, method = "ML") 1
update(modelo, fixed = . ~ . - poly(tiempo, 3) + tiempo, method = "ML") 2
df
update(modelo, method = "ML") 8
update(modelo, fixed = . ~ . - poly(tiempo, 3) + tiempo, method = "ML") 6
AIC
update(modelo, method = "ML") 662.6555
update(modelo, fixed = . ~ . - poly(tiempo, 3) + tiempo, method = "ML") 663.4848
BIC
update(modelo, method = "ML") 687.6473
update(modelo, fixed = . ~ . - poly(tiempo, 3) + tiempo, method = "ML") 682.2286
logLik
update(modelo, method = "ML") -323.3278
update(modelo, fixed = . ~ . - poly(tiempo, 3) + tiempo, method = "ML") -325.7424
Test
update(modelo, method = "ML")
update(modelo, fixed = . ~ . - poly(tiempo, 3) + tiempo, method = "ML") 1 vs 2
L.Ratio
update(modelo, method = "ML")
update(modelo, fixed = . ~ . - poly(tiempo, 3) + tiempo, method = "ML") 4.82924
p-value
update(modelo, method = "ML")
update(modelo, fixed = . ~ . - poly(tiempo, 3) + tiempo, method = "ML") 0.0894El p-valor > 0.05, por lo tanto nos quedamos con el modelo lineal.
Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
671.7705 690.4062 -329.8853
Random effects:
Formula: ~1 | indiv
(Intercept) Residual
StdDev: 1.61273 1.877586
Correlation Structure: Continuous AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.7045807
Fixed effects: respuesta ~ edad + tiempo
Value Std.Error DF t-value p-value
(Intercept) 12.084507 1.5894665 117 7.602870 0
edad 0.426773 0.0319158 48 13.371839 0
tiempo -0.959328 0.1030230 117 -9.311792 0
Correlation:
(Intr) edad
edad -0.976
tiempo -0.092 -0.002
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-2.13574976 -0.49274745 0.00586512 0.57010471 2.16657427
Number of Observations: 168
Number of Groups: 50 Value Std.Error DF t-value p-value
(Intercept) 12.0845065 1.58946650 117 7.602870 7.939399e-12
edad 0.4267727 0.03191578 48 13.371839 8.365041e-18
tiempo -0.9593285 0.10302297 117 -9.311792 9.105728e-16Validación del modelo
- Errores
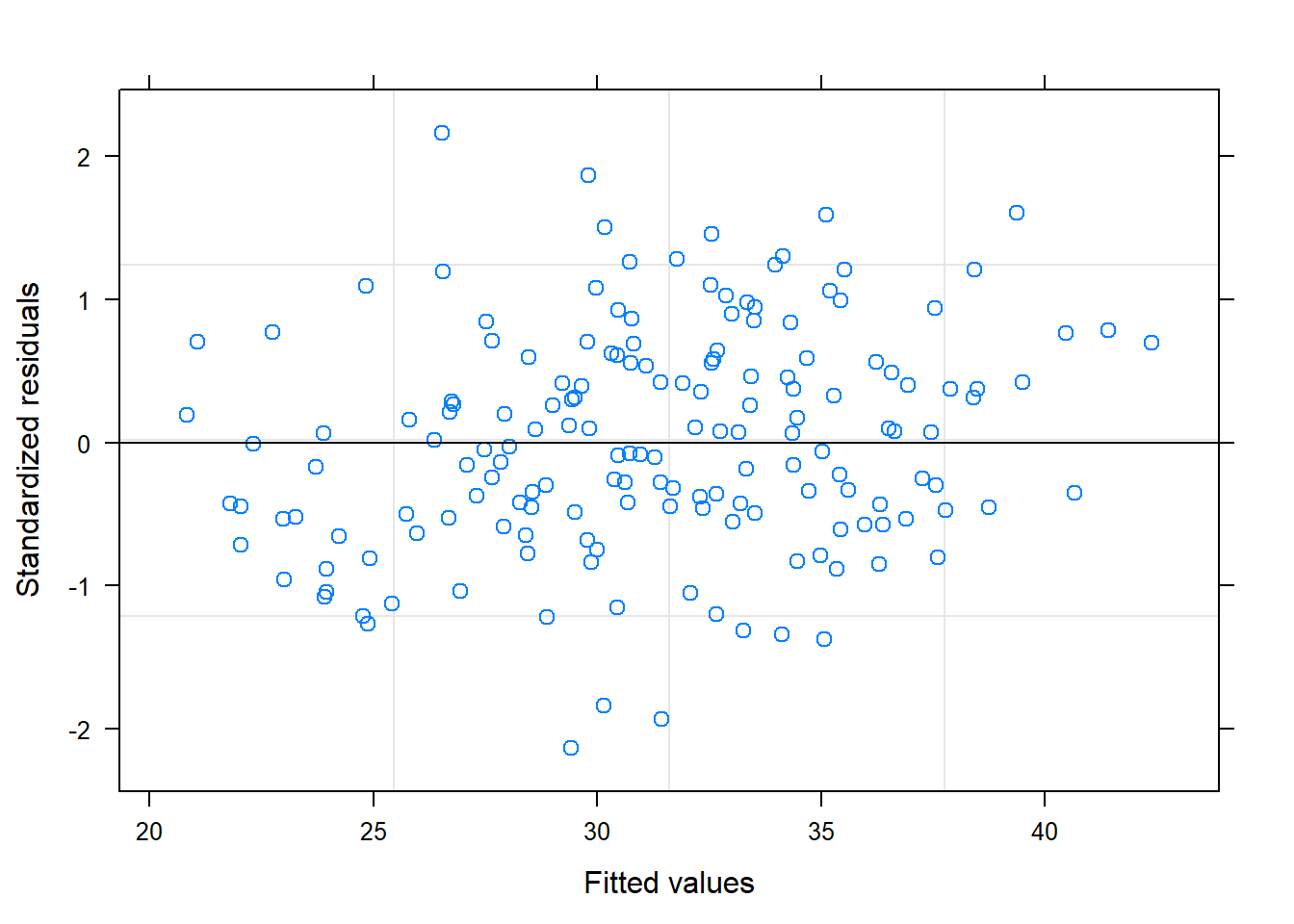
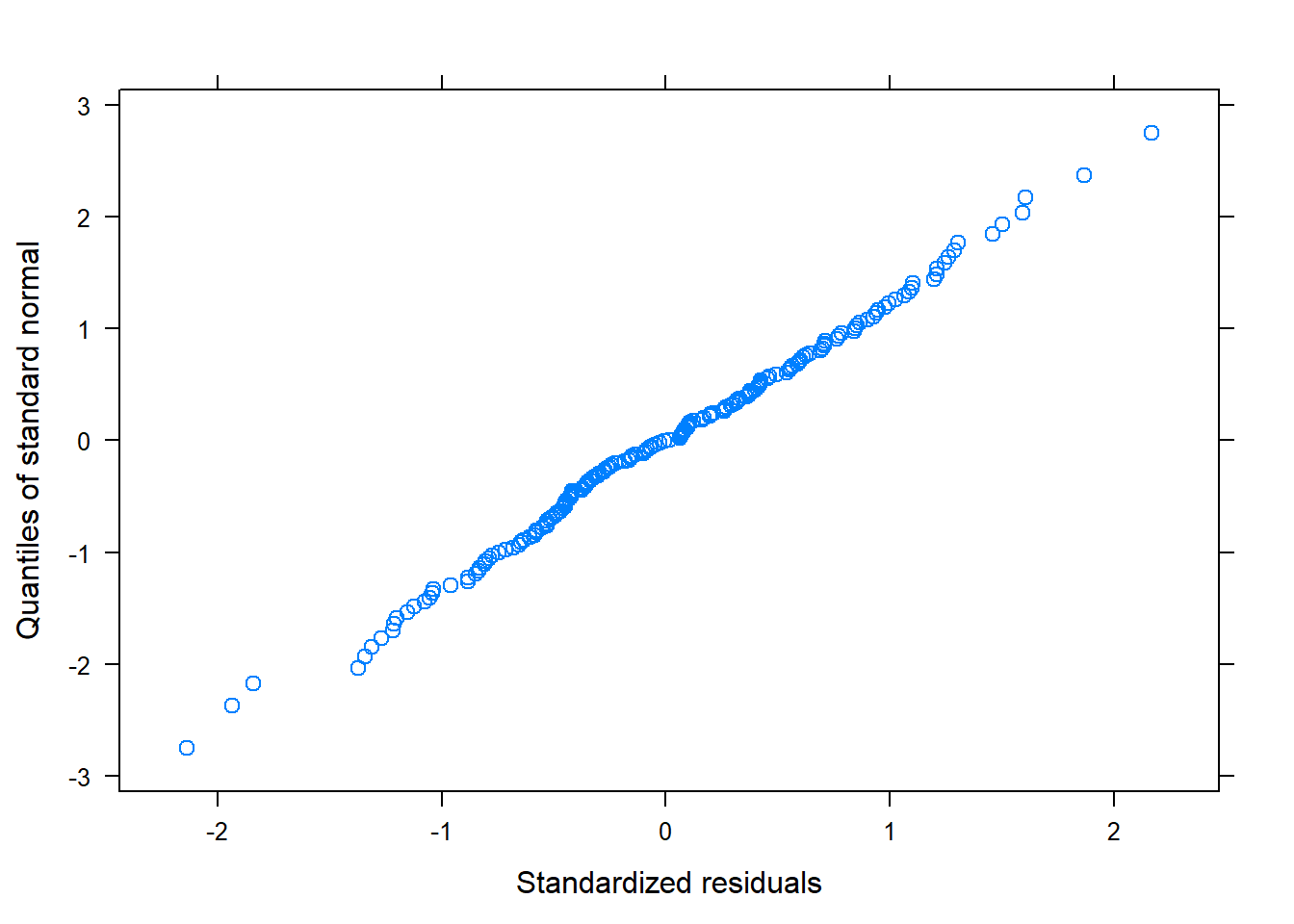
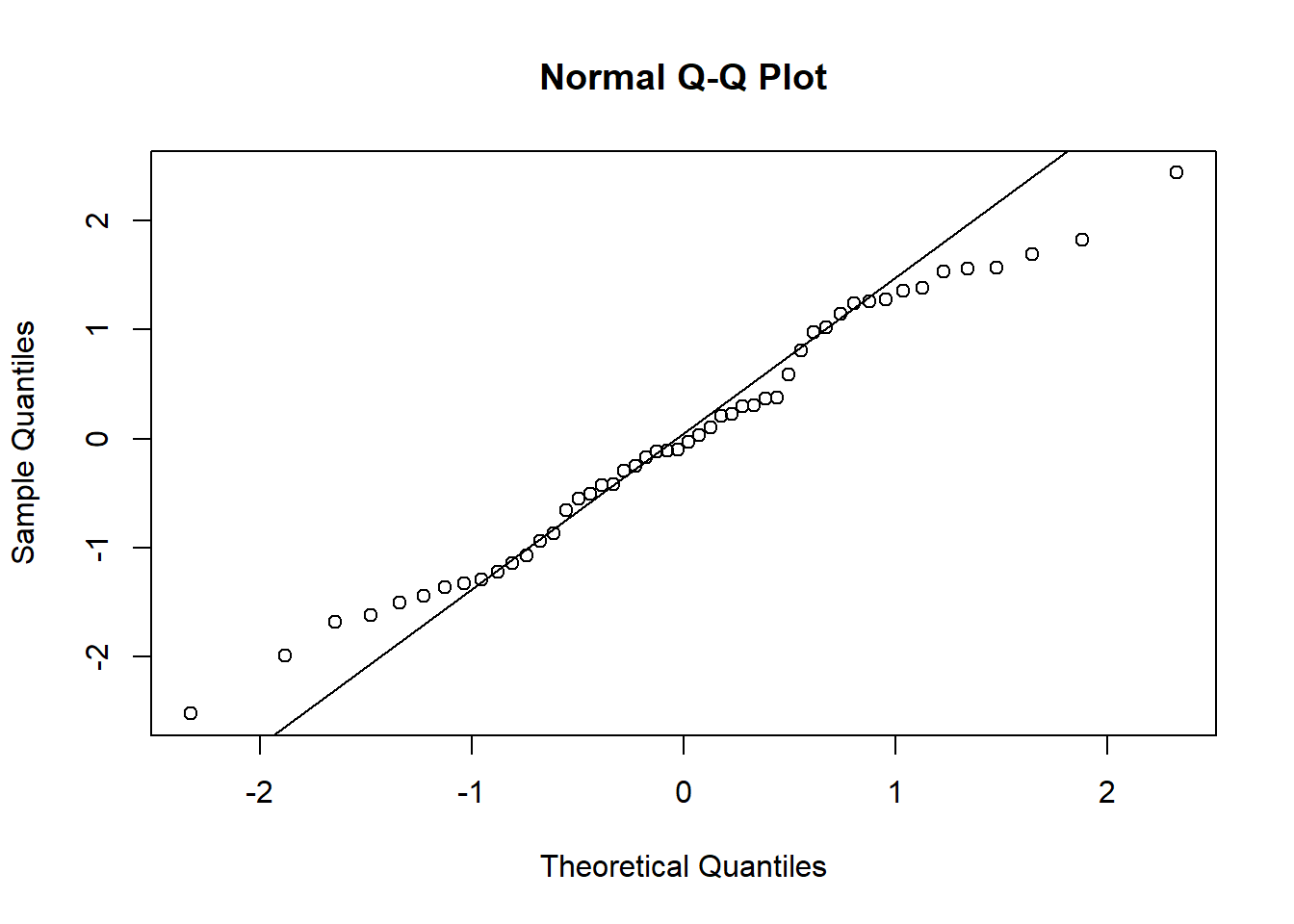
- Efectos aleatorios
edad <- with(datos, tapply(edad, indiv, mean))
# gráfico de los efectos aleatorios vs variables individuo
plot(edad, beta0i)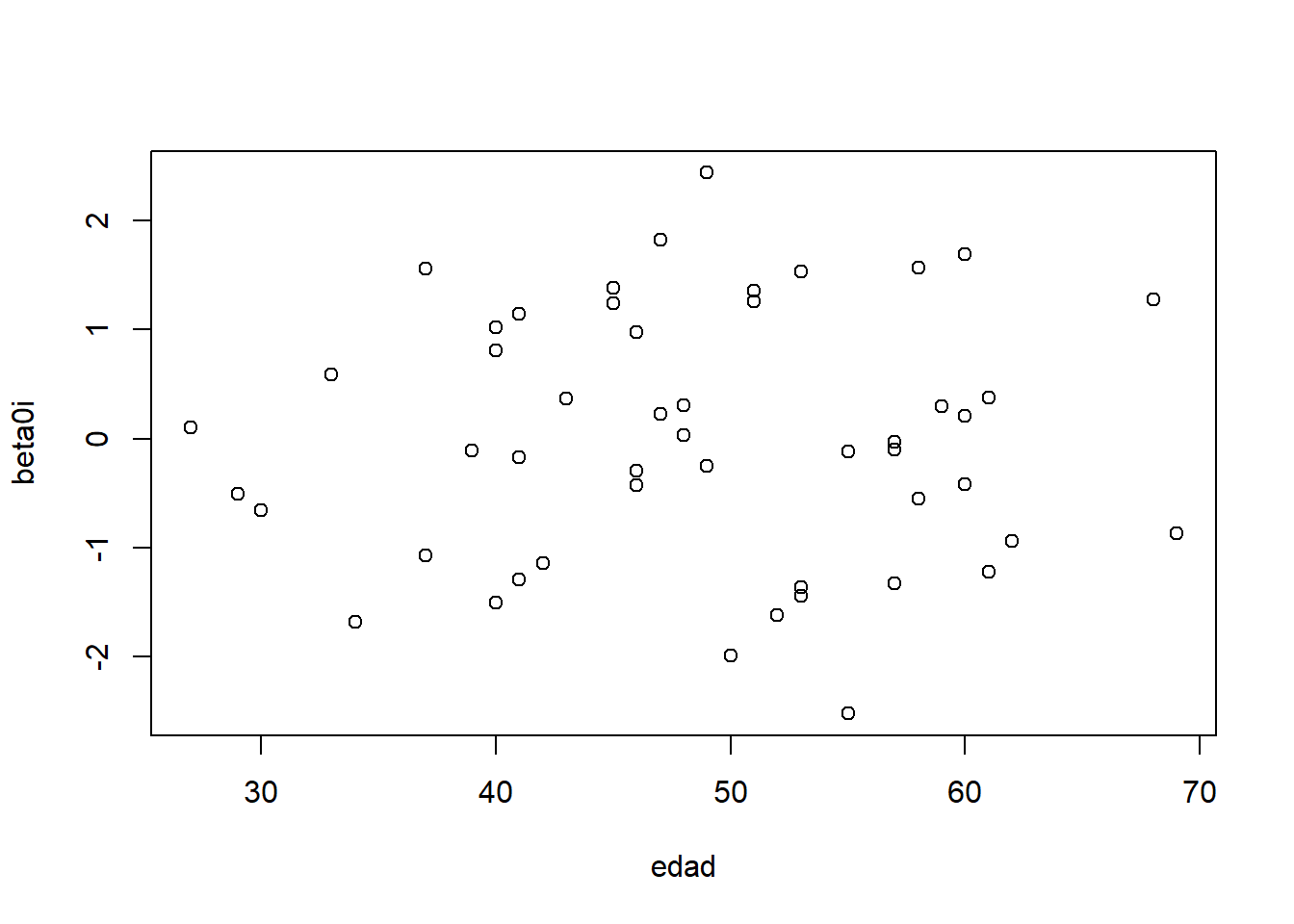
Parece que los efectos aleatorios siguen una distribución normal. Y no están relacionados con la edad.
Predicciones
# Predicted values of respuesta
# x = tiempo
x | Predicted | 95% CI
------------------------------
0 | 32.57 | [31.89, 33.25]
1 | 31.61 | [30.99, 32.24]
2 | 30.65 | [30.02, 31.28]
3 | 29.69 | [29.00, 30.38]
Adjusted for:
* edad = 48.00
* indiv = 26.00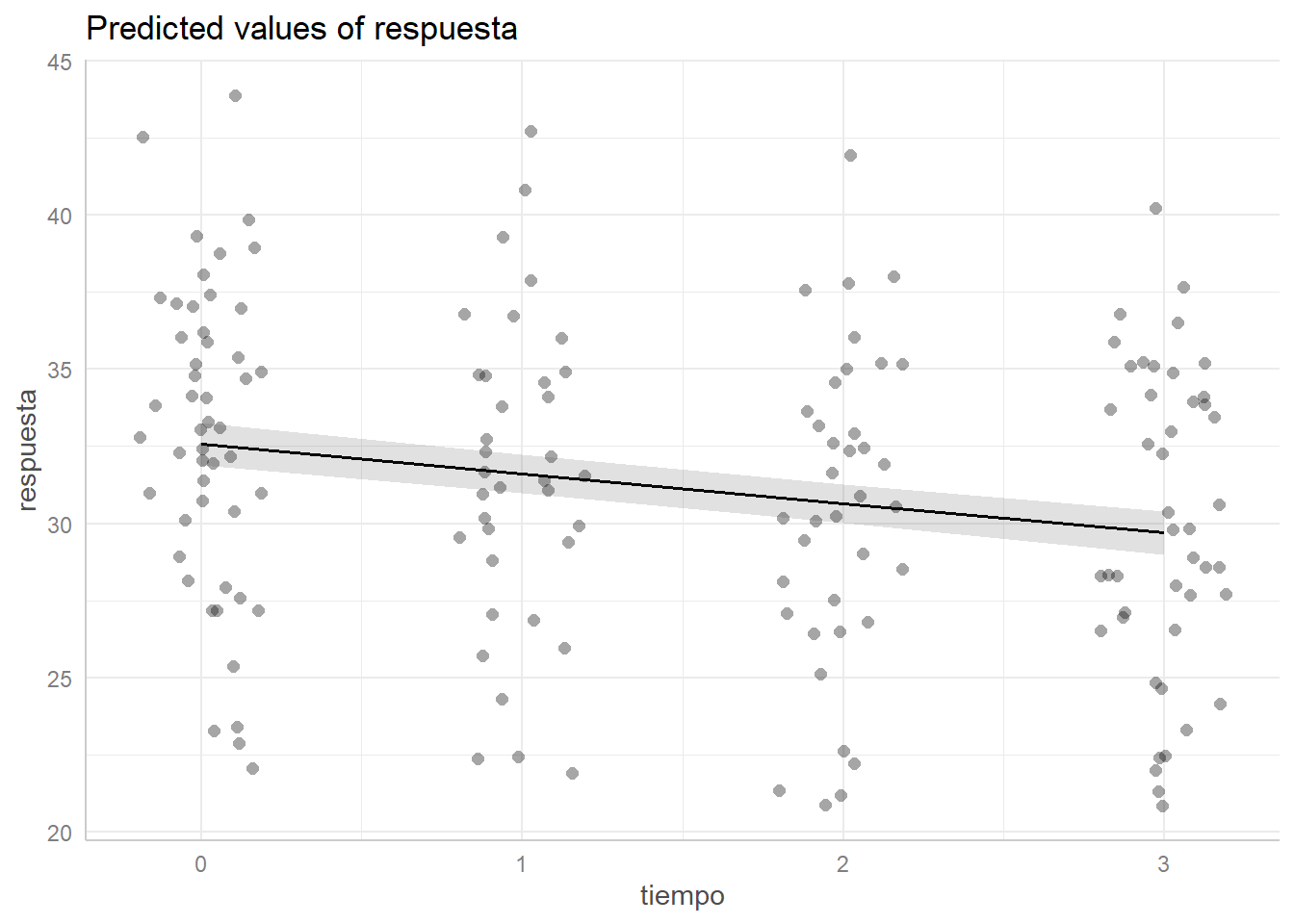
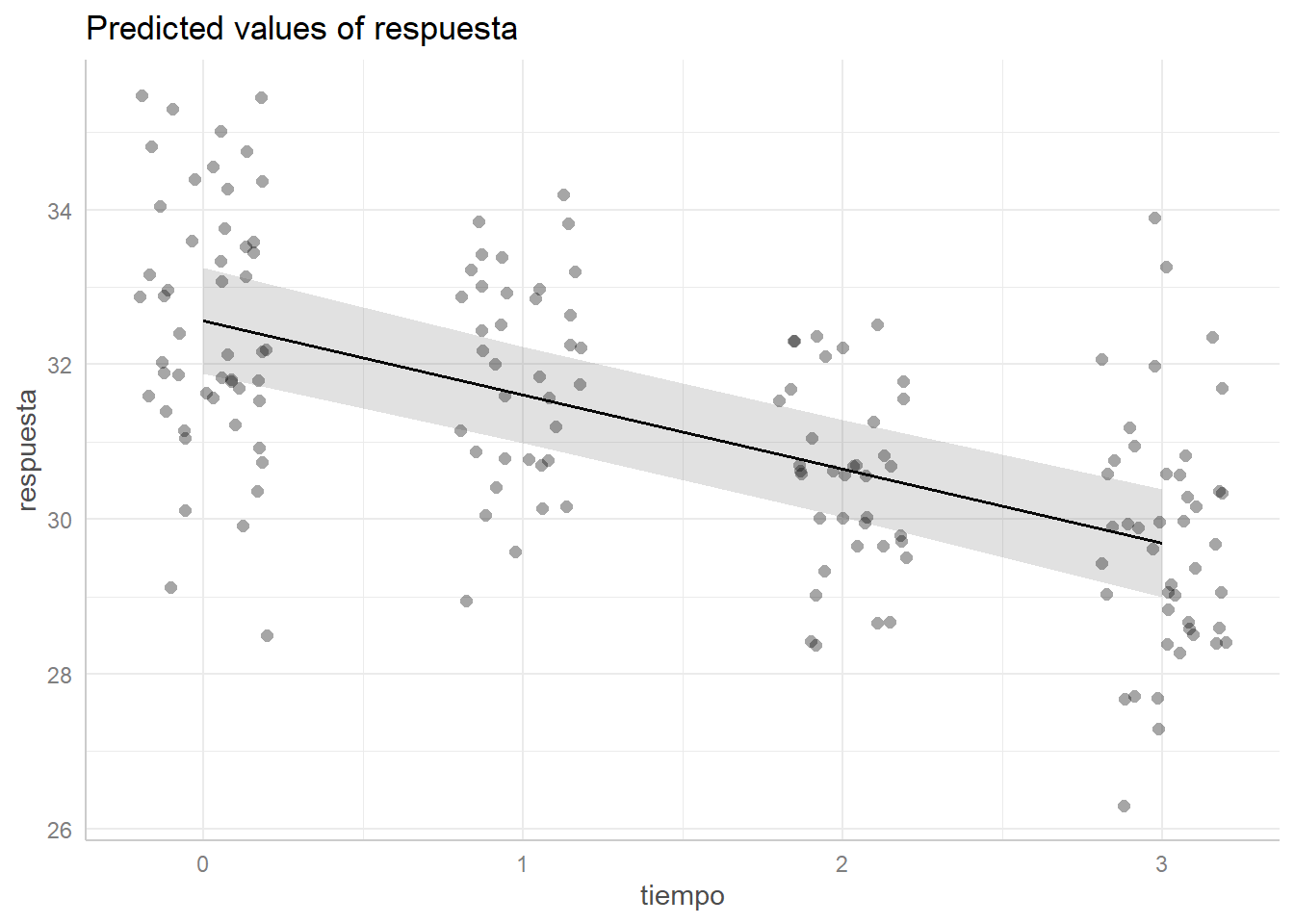
Las predicciones las realiza en la media de las covariables, en este caso la edad. Si queremos que las predicciones las haga para un individuo de 55 años:
# Predicted values of respuesta
# x = tiempo
x | Predicted | 95% CI
------------------------------
0 | 35.56 | [34.76, 36.35]
1 | 34.60 | [33.86, 35.34]
2 | 33.64 | [32.89, 34.38]
3 | 32.68 | [31.88, 33.48]
Adjusted for:
* indiv = 26.00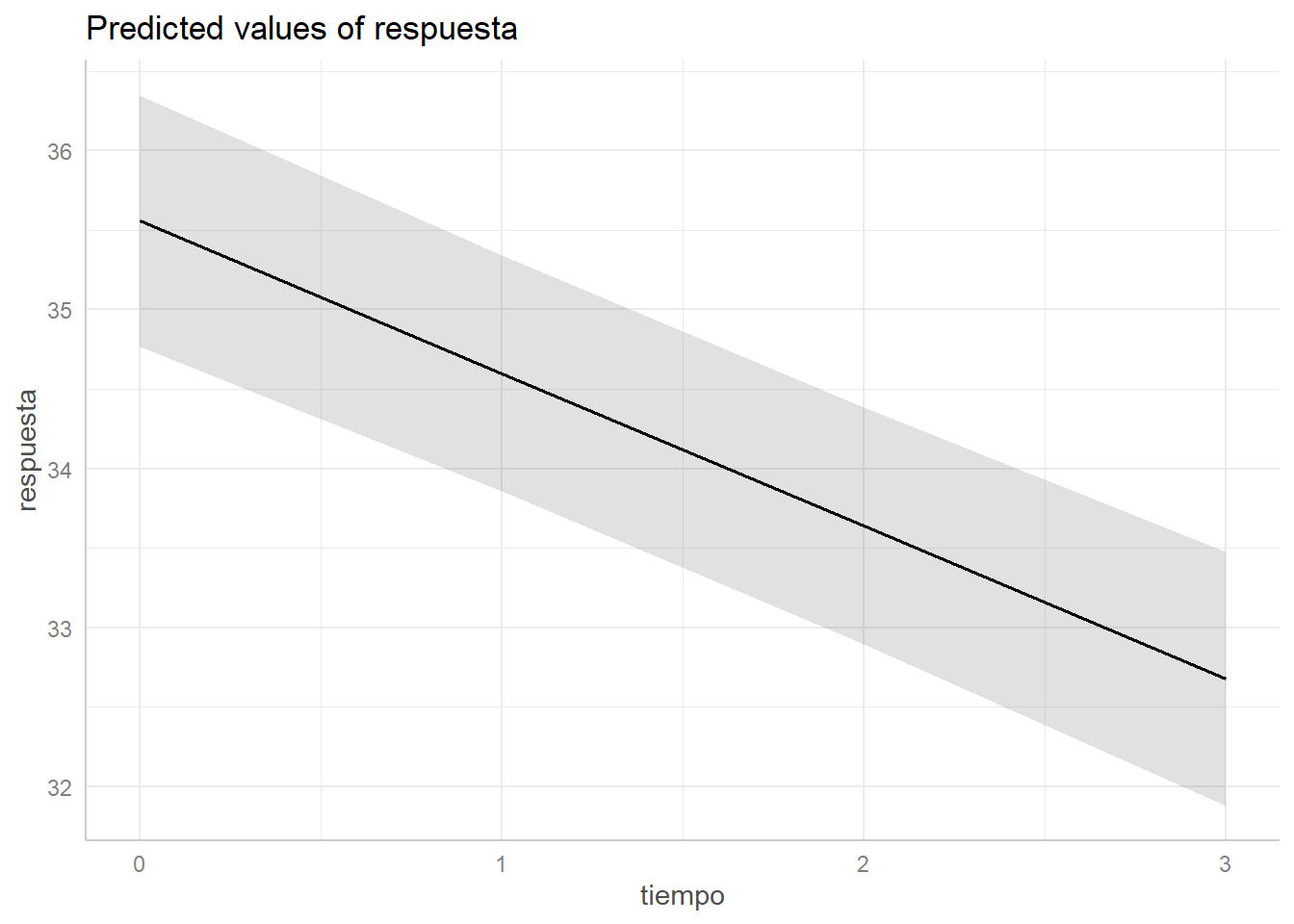
4.5.3.2 Comparación de los dos tratamientos
datos <- read.csv2("./datos/imc.csv")
datos <- datos[order(datos$indiv,datos$tiempo),]
datos <- na.omit(datos)Volvemos al enunciado original trabajando con todos los datos. Ahora el objetivo es comparar la evolución de los dos tratamientos ajustando por la edad.
library(nlme)
modelo <- lme(respuesta ~ poly(tiempo, 3, raw=3) + tx:poly(tiempo, 3, raw=3) + edad,
random= ~ poly(tiempo, 3, raw=3) | indiv,
data=datos,
correlation=corCAR1(form = ~ tiempo|indiv))
summary(modelo)Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
1417.874 1494.57 -688.9369
Random effects:
Formula: ~poly(tiempo, 3, raw = 3) | indiv
Structure: General positive-definite, Log-Cholesky parametrization
StdDev Corr
(Intercept) 2.3069274 (Intr) p(,3,r=3)1 p(,3,r=3)2
poly(tiempo, 3, raw = 3)1 1.9231468 -0.470
poly(tiempo, 3, raw = 3)2 1.7013762 0.354 -0.904
poly(tiempo, 3, raw = 3)3 0.3562430 -0.342 0.852 -0.990
Residual 0.9432126
Correlation Structure: Continuous AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.248855
Fixed effects: respuesta ~ poly(tiempo, 3, raw = 3) + tx:poly(tiempo, 3, raw = 3) + edad
Value Std.Error DF t-value p-value
(Intercept) 11.168714 1.1301845 244 9.882204 0.0000
poly(tiempo, 3, raw = 3)1 2.402332 1.2555058 244 1.913438 0.0569
poly(tiempo, 3, raw = 3)2 -2.075890 1.1602056 244 -1.789243 0.0748
poly(tiempo, 3, raw = 3)3 0.447207 0.2546185 244 1.756379 0.0803
edad 0.442918 0.0224834 98 19.699782 0.0000
poly(tiempo, 3, raw = 3)1:tx -2.107063 0.7636948 244 -2.759037 0.0062
poly(tiempo, 3, raw = 3)2:tx 0.905552 0.7097238 244 1.275922 0.2032
poly(tiempo, 3, raw = 3)3:tx -0.194165 0.1561157 244 -1.243724 0.2148
Correlation:
(Intr) pl(,3,r=3)1 pl(,3,r=3)2 pl(,3,r=3)3 edad
poly(tiempo, 3, raw = 3)1 -0.048
poly(tiempo, 3, raw = 3)2 0.033 -0.949
poly(tiempo, 3, raw = 3)3 -0.028 0.899 -0.989
edad -0.975 0.027 -0.020 0.016
poly(tiempo, 3, raw = 3)1:tx 0.020 -0.948 0.903 -0.856 -0.020
poly(tiempo, 3, raw = 3)2:tx -0.013 0.898 -0.951 0.941 0.013
poly(tiempo, 3, raw = 3)3:tx 0.010 -0.849 0.939 -0.951 -0.010
p(,3,r=3)1: p(,3,r=3)2:
poly(tiempo, 3, raw = 3)1
poly(tiempo, 3, raw = 3)2
poly(tiempo, 3, raw = 3)3
edad
poly(tiempo, 3, raw = 3)1:tx
poly(tiempo, 3, raw = 3)2:tx -0.947
poly(tiempo, 3, raw = 3)3:tx 0.896 -0.988
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-1.65153170 -0.36449401 -0.04684107 0.39784060 1.80027927
Number of Observations: 350
Number of Groups: 100 Nota: poly(tiempo, 3, raw=TRUE) es lo miso que tiempo + I(tiempo^2) + I(tiempo^3).
Fijate cómo se ha escrito la fórmula. De esta manera, cuando tiempo=0 (momento basal) no hay diferencias entre en los tratamientos.
Efectos aleatorios
Model df AIC BIC logLik Test
modelo 1 20 1417.874 1494.57 -688.9369
update(modelo, random = ~1 | indiv) 2 11 1408.597 1450.78 -693.2985 1 vs 2
L.Ratio p-value
modelo
update(modelo, random = ~1 | indiv) 8.7233 0.4632Vemos en este caso, como el p-valor del LRT no coincide con la decisión basada en el AIC o el BIC. Podemos decantarnos con el modelo más simple, o sea, el que supone que los coeficientes del tiempo son fijos.
Estructura de correlación de los errores
Comparamos con la matriz de independencia
Model df AIC BIC logLik Test
modelo 1 11 1408.597 1450.780 -693.2985
update(modelo, correlation = NULL) 2 10 1446.555 1484.903 -713.2774 1 vs 2
L.Ratio p-value
modelo
update(modelo, correlation = NULL) 39.95779 <.0001Nos quedamos con la estructura AR1.
Finalmente, comprovamos los efectos fijos:
Value Std.Error DF t-value p-value
(Intercept) 10.9983674 1.17012354 244 9.399322 4.150278e-18
poly(tiempo, 3, raw = 3)1 2.2104398 1.28087236 244 1.725730 8.566199e-02
poly(tiempo, 3, raw = 3)2 -1.8762381 1.14921815 244 -1.632621 1.038384e-01
poly(tiempo, 3, raw = 3)3 0.4026454 0.25308582 244 1.590944 1.129169e-01
edad 0.4463927 0.02329682 98 19.161101 6.509723e-35
poly(tiempo, 3, raw = 3)1:tx -1.9653182 0.78262641 244 -2.511183 1.268004e-02
poly(tiempo, 3, raw = 3)2:tx 0.7598944 0.70301744 244 1.080904 2.808070e-01
poly(tiempo, 3, raw = 3)3:tx -0.1617992 0.15513365 244 -1.042967 2.979960e-01Como era de esperar, la edad es muy significativa.
Contrastamos el efecto cuadrático y cúbico del tiempo (tanto para el grupo control como para el grupo de tratados):
Value Std.Error DF t-value p-value
(Intercept) 11.0143619 1.16920806 248 9.4203610 3.281588e-18
tiempo 0.1947449 0.21775844 248 0.8943163 3.720198e-01
edad 0.4462810 0.02328366 98 19.1671311 6.352777e-35
tiempo:tx -1.1381679 0.13574697 248 -8.3844810 3.860440e-15 Model df AIC BIC logLik Test L.Ratio p-value
modelo 1 11 1408.597 1450.780 -693.2985
modelo2 2 7 1394.327 1421.252 -690.1637 1 vs 2 6.269749 0.1799Como el p-valor del LRT es >0.05, nos quedamos con el modelo más simple, en que el tiempo tiene un efecto lineal en ambos grupos
Value Std.Error DF t-value p-value
(Intercept) 11.0143619 1.16920806 248 9.4203610 3.281588e-18
tiempo 0.1947449 0.21775844 248 0.8943163 3.720198e-01
edad 0.4462810 0.02328366 98 19.1671311 6.352777e-35
tiempo:tx -1.1381679 0.13574697 248 -8.3844810 3.860440e-15El efecto del tiempo en el grupo control no es significativo.
Para ver el efecto del tiempo en el grupo de tratamiento, cambiamos la categoria de referencia:
Value Std.Error DF t-value p-value
(Intercept) 11.014362 1.16920806 248 9.420361 3.281588e-18
tiempo -2.081591 0.10101027 248 -20.607715 1.166972e-55
edad 0.446281 0.02328366 98 19.167131 6.352777e-35
tiempo:tx1 1.138168 0.13574697 248 8.384481 3.860440e-15Vemos como el efecto del tiempo en el grupo de tratados es significativo y la pendiente es negativa.
Validación
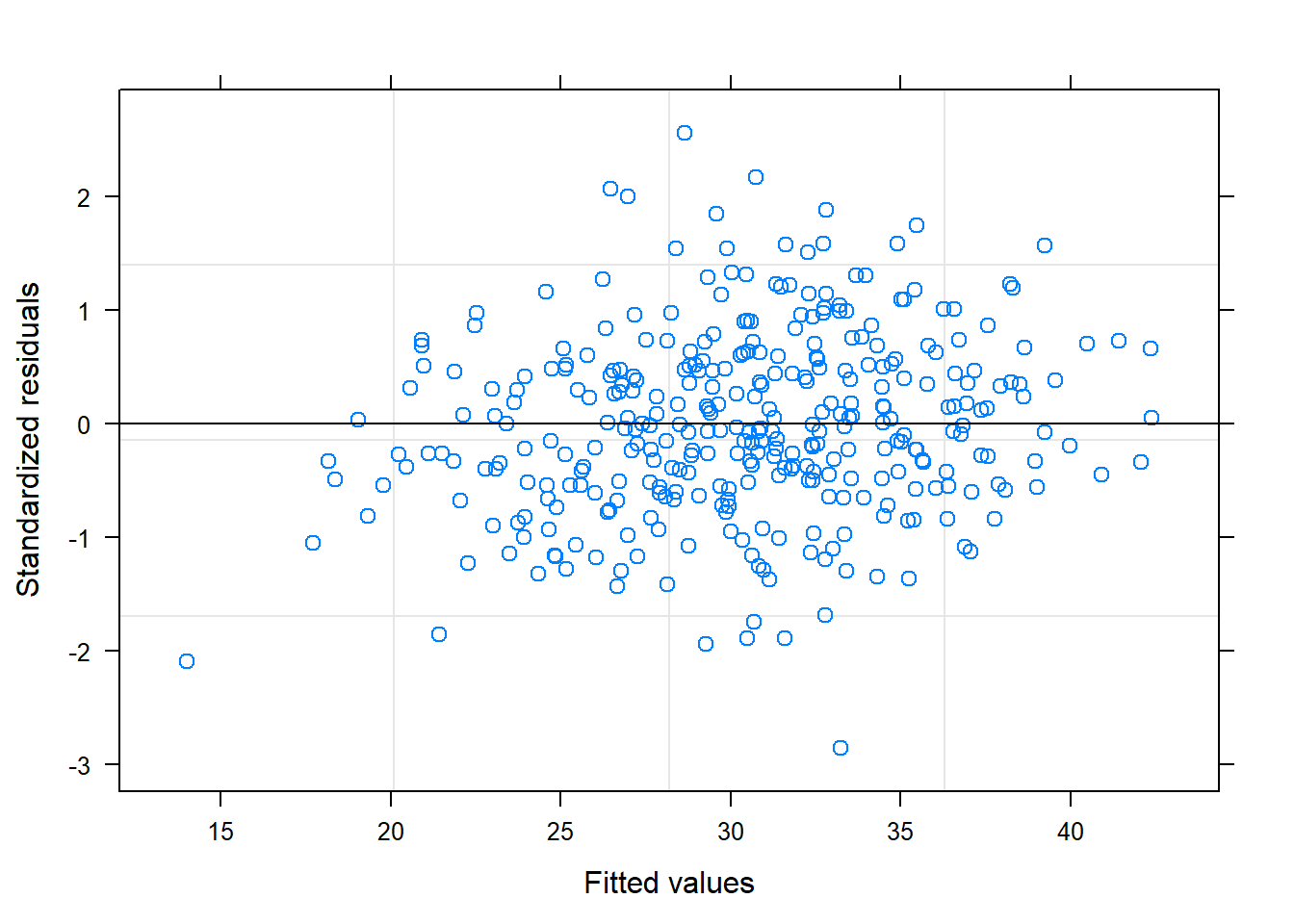
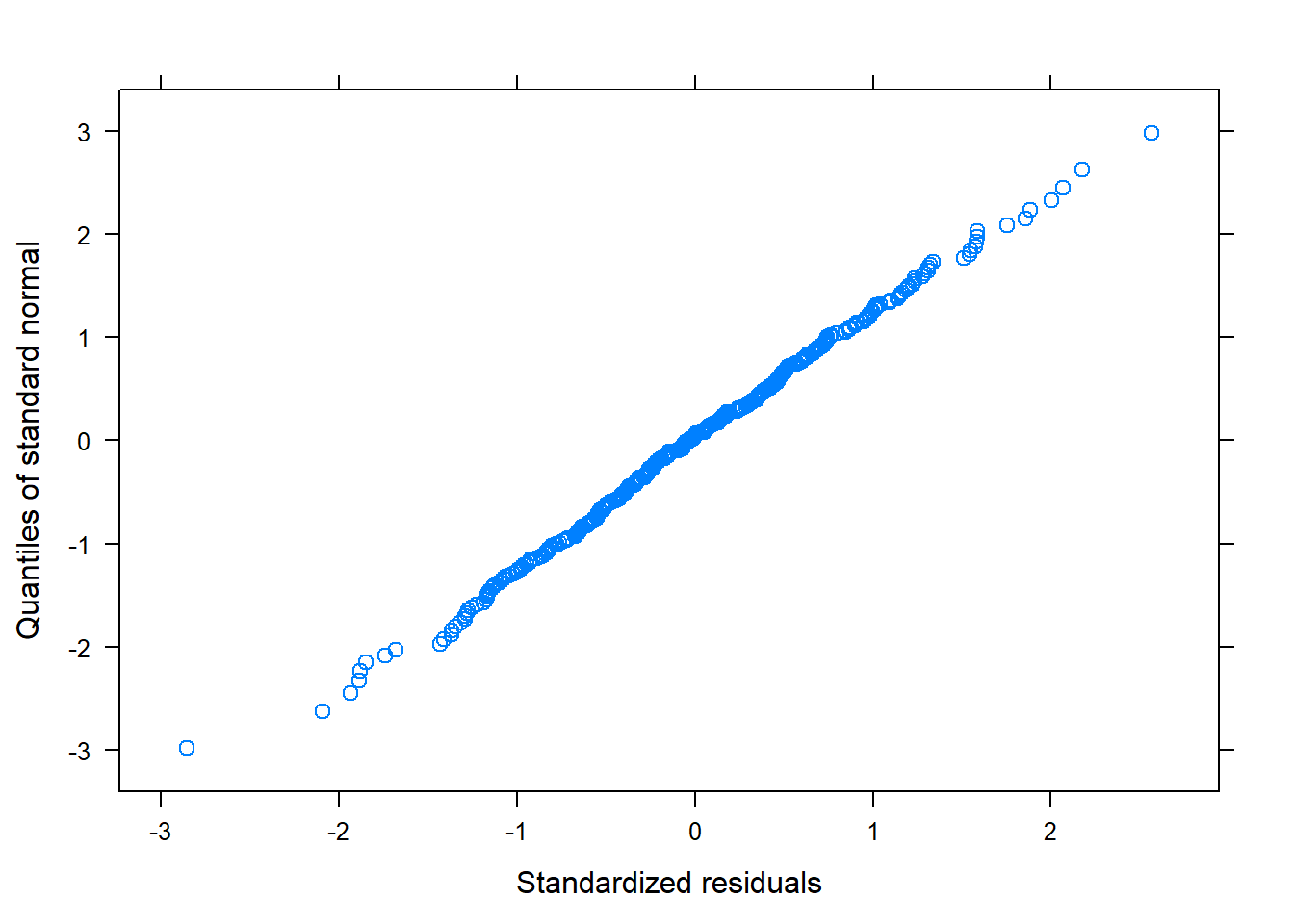
# efectos aleatorios
beta0i <- ranef(modelo)[,1]
grupo <- with(datos, tapply(tx, indiv, head, n=1))
edad <- with(datos, tapply(edad, indiv, mean))
boxplot(beta0i ~ grupo)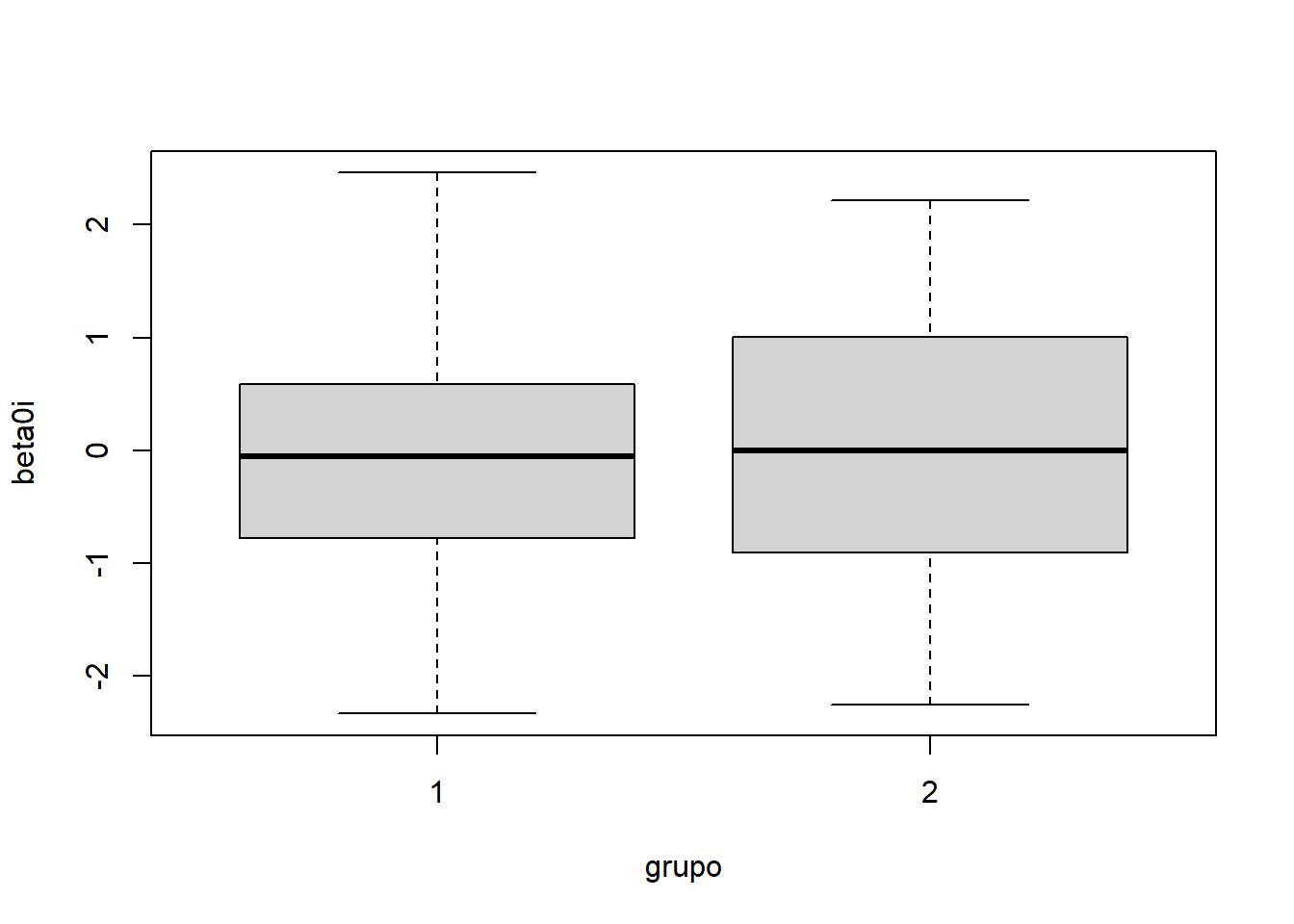

Predicciones
datos$tx <- factor(datos$tx, 1:2)
modelo <- update(modelo)
pr.cont <- ggpredict(modelo, terms = c("tiempo"), condition=c(tx=1), type="fixed")
pr.tx <- ggpredict(modelo, terms = c("tiempo"), condition=c(tx=2), type="fixed")
pr.cont
# Predicted values of respuesta
# x = tiempo
x | Predicted | 95% CI
------------------------------
0 | 32.88 | [32.39, 33.38]
1 | 31.94 | [31.47, 32.41]
2 | 31.00 | [30.47, 31.52]
3 | 30.05 | [29.41, 30.69]
Adjusted for:
* edad = 49.00
* indiv = 52.50
# Predicted values of respuesta
# x = tiempo
x | Predicted | 95% CI
------------------------------
0 | 32.88 | [32.39, 33.38]
1 | 30.80 | [30.33, 31.27]
2 | 28.72 | [28.19, 29.25]
3 | 26.64 | [25.99, 27.28]
Adjusted for:
* edad = 49.00
* indiv = 52.50library(gridExtra)
grid.arrange(
plot(pr.cont) + ylim(24,40) + ggtitle("controles"),
plot(pr.tx) + ylim(24,40) + ggtitle("tratados"),
nrow=1, ncol=2)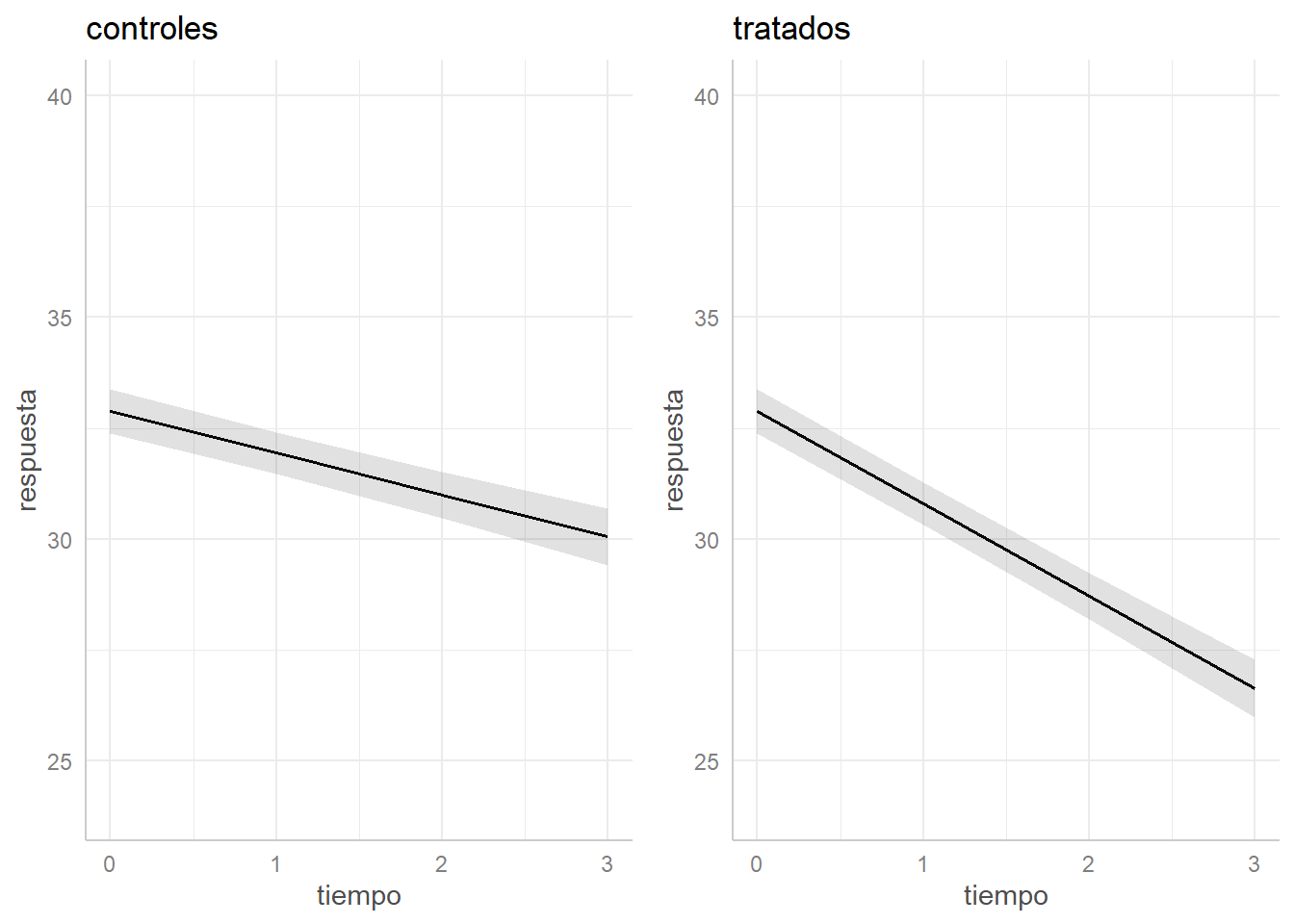
4.5.4 Ejemplo 3b
En el archivo “imc2.csv” hay los datos del mismo diseño que en el ejemplo anterior con 100 individuos a los que se les ha medido el IMC. Ahora los datos están generados de tal manera que la varianza es diferente segun el sexo. Veamos como
respuesta indiv tiempo edad tx
Min. :14.10 Min. : 1.00 Min. :0.00 Min. :25.00 Min. :1.0
1st Qu.:29.30 1st Qu.: 25.75 1st Qu.:0.75 1st Qu.:43.00 1st Qu.:1.0
Median :32.00 Median : 50.50 Median :1.50 Median :49.00 Median :1.5
Mean :31.71 Mean : 50.50 Mean :1.50 Mean :49.03 Mean :1.5
3rd Qu.:34.40 3rd Qu.: 75.25 3rd Qu.:2.25 3rd Qu.:57.00 3rd Qu.:2.0
Max. :41.40 Max. :100.00 Max. :3.00 Max. :69.00 Max. :2.0
NA's :50
sexo
Min. :0.0
1st Qu.:0.0
Median :0.5
Mean :0.5
3rd Qu.:1.0
Max. :1.0
Vemos cómo si ajustamos el mismo modelo que antes, la variabilidad de los residuos es diferente según el sexo (distinguido en este gráfico con distintos colores)
modelo <- lme(respuesta ~ tiempo:tx + tiempo + edad, random= ~ 1 | indiv,
data=datos,
correlation=corCAR1(form = ~ tiempo | indiv),
control=lmeControl(opt="optim"))
plot(modelo, col=datos$sexo+1)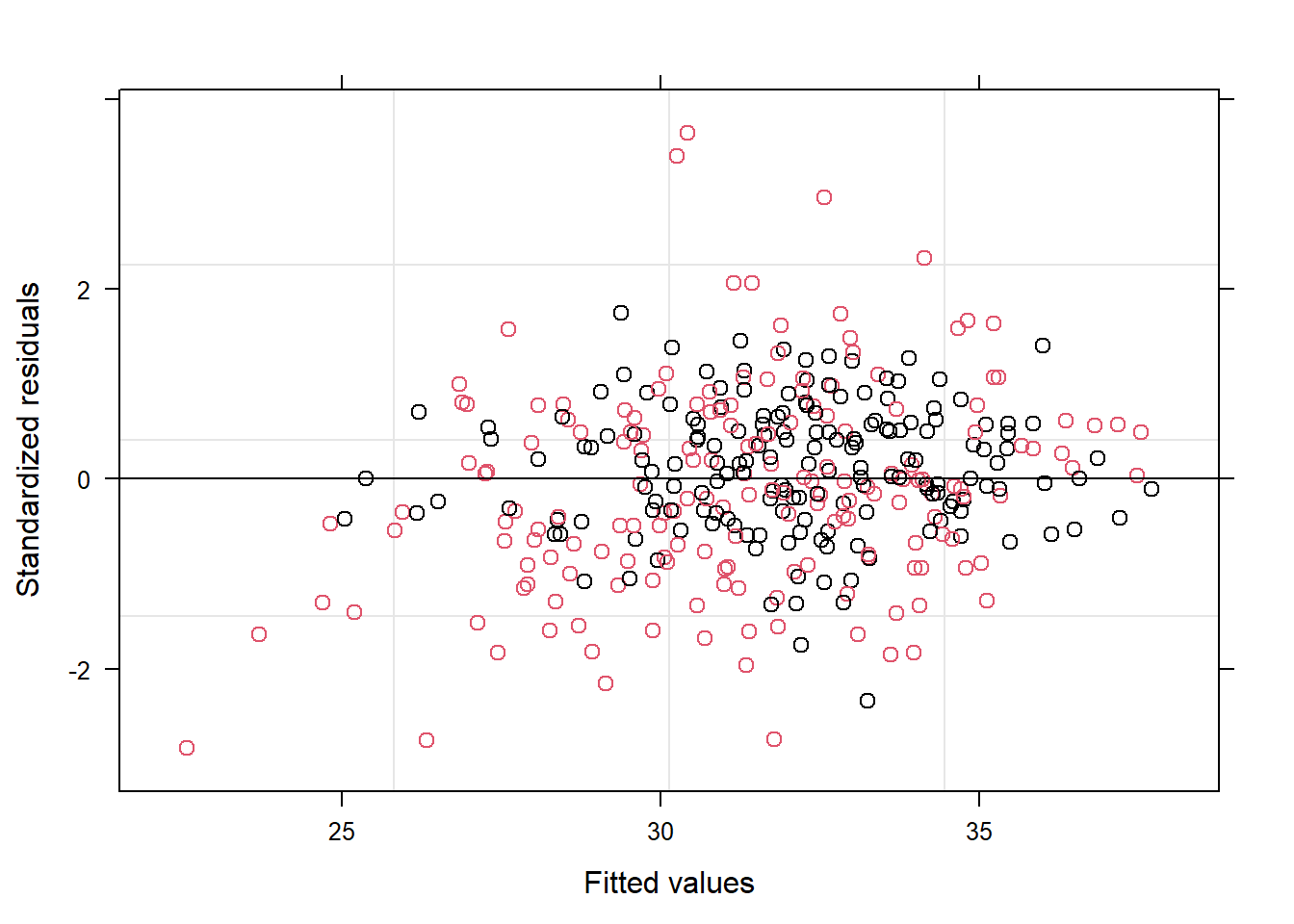
Value Std.Error DF t-value p-value
(Intercept) 21.0501634 1.38589002 248 15.188913 2.784394e-37
tiempo 0.4045266 0.32544983 248 1.242977 2.150502e-01
edad 0.2387408 0.02751099 98 8.678016 8.885782e-14
tiempo:tx -0.7660070 0.20126351 248 -3.805991 1.780062e-04Para espeficar varianzas distintas según el sexo, se usa el argumento weights con la función varIdent.
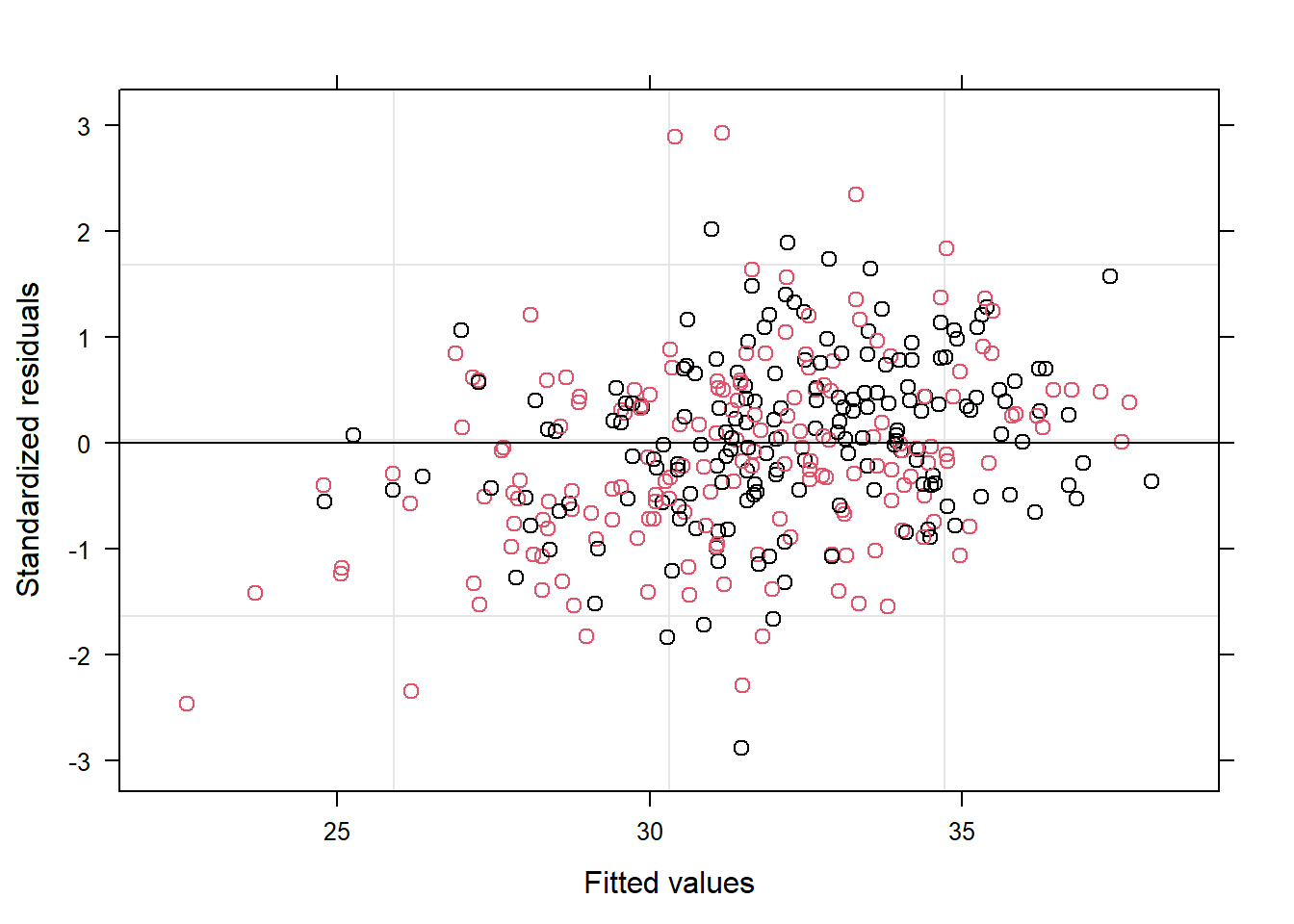
Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
1596.799 1627.57 -790.3995
Random effects:
Formula: ~1 | indiv
(Intercept) Residual
StdDev: 1.651654 1.791477
Correlation Structure: Continuous AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.6320083
Variance function:
Structure: Different standard deviations per stratum
Formula: ~1 | sexo
Parameter estimates:
0 1
1.000000 1.926961
Fixed effects: respuesta ~ tiempo:tx + tiempo + edad
Value Std.Error DF t-value p-value
(Intercept) 21.885078 1.3526322 248 16.179623 0.0000
tiempo 0.167589 0.2659804 248 0.630080 0.5292
edad 0.226018 0.0266881 98 8.468844 0.0000
tiempo:tx -0.632787 0.1665493 248 -3.799399 0.0002
Correlation:
(Intr) tiempo edad
tiempo -0.051
edad -0.977 0.016
tiempo:tx 0.017 -0.935 -0.018
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-2.88328812 -0.54093628 -0.01169392 0.50055963 2.93441998
Number of Observations: 350
Number of Groups: 100 Ahora la variabilidad es la misma en ambos grupos. Fíjate que las estimaciones de los efectos fijos también cambian.
Fíjate en la salida de summary, concretamente en “Variance function:”, como se especifica el factor multiplicador de la varianza para los hombre y para las mujeres.
Por último, si comparamos las estimaciones de los efectos fijos, éstos son ligeramente distintos si se ponera (“weights”) por la variable sexo o no.
Value Std.Error DF t-value p-value
(Intercept) 21.0501634 1.38589002 248 15.188913 2.784394e-37
tiempo 0.4045266 0.32544983 248 1.242977 2.150502e-01
edad 0.2387408 0.02751099 98 8.678016 8.885782e-14
tiempo:tx -0.7660070 0.20126351 248 -3.805991 1.780062e-04 Value Std.Error DF t-value p-value
(Intercept) 21.8850785 1.35263220 248 16.1796226 1.106347e-40
tiempo 0.1675889 0.26598039 248 0.6300798 5.292225e-01
edad 0.2260175 0.02668812 98 8.4688442 2.509049e-13
tiempo:tx -0.6327872 0.16654927 248 -3.7993995 1.825378e-044.5.5 Ejemplo 3c
En el archivo “imc3.csv” hay los datos del mismo diseño pero ahora los datos están generados de tal manera que la varianza es diferente según la inversa de la variable “pesos”.
respuesta indiv tiempo edad tx
Min. :18.30 Min. : 1.00 Min. :0.00 Min. :25.00 Min. :1.0
1st Qu.:29.60 1st Qu.: 25.75 1st Qu.:0.75 1st Qu.:43.00 1st Qu.:1.0
Median :32.10 Median : 50.50 Median :1.50 Median :49.00 Median :1.5
Mean :31.94 Mean : 50.50 Mean :1.50 Mean :49.03 Mean :1.5
3rd Qu.:34.48 3rd Qu.: 75.25 3rd Qu.:2.25 3rd Qu.:57.00 3rd Qu.:2.0
Max. :41.80 Max. :100.00 Max. :3.00 Max. :69.00 Max. :2.0
NA's :50
pesos
Min. :1.022
1st Qu.:3.319
Median :5.767
Mean :5.541
3rd Qu.:7.614
Max. :9.987
Si no tenemos en cuenta los pesos se observa una cierta heterocedesticidad: la dispersión de los residuos aumenta según la variable pesos.
modelo <- lme(respuesta ~ tiempo:tx + tiempo + edad, random= ~ 1 | indiv,
data=datos,
correlation=corCAR1(form = ~ tiempo | indiv),
control=lmeControl(opt="optim"))
plot(modelo, form = resid(., type="p") ~ pesos)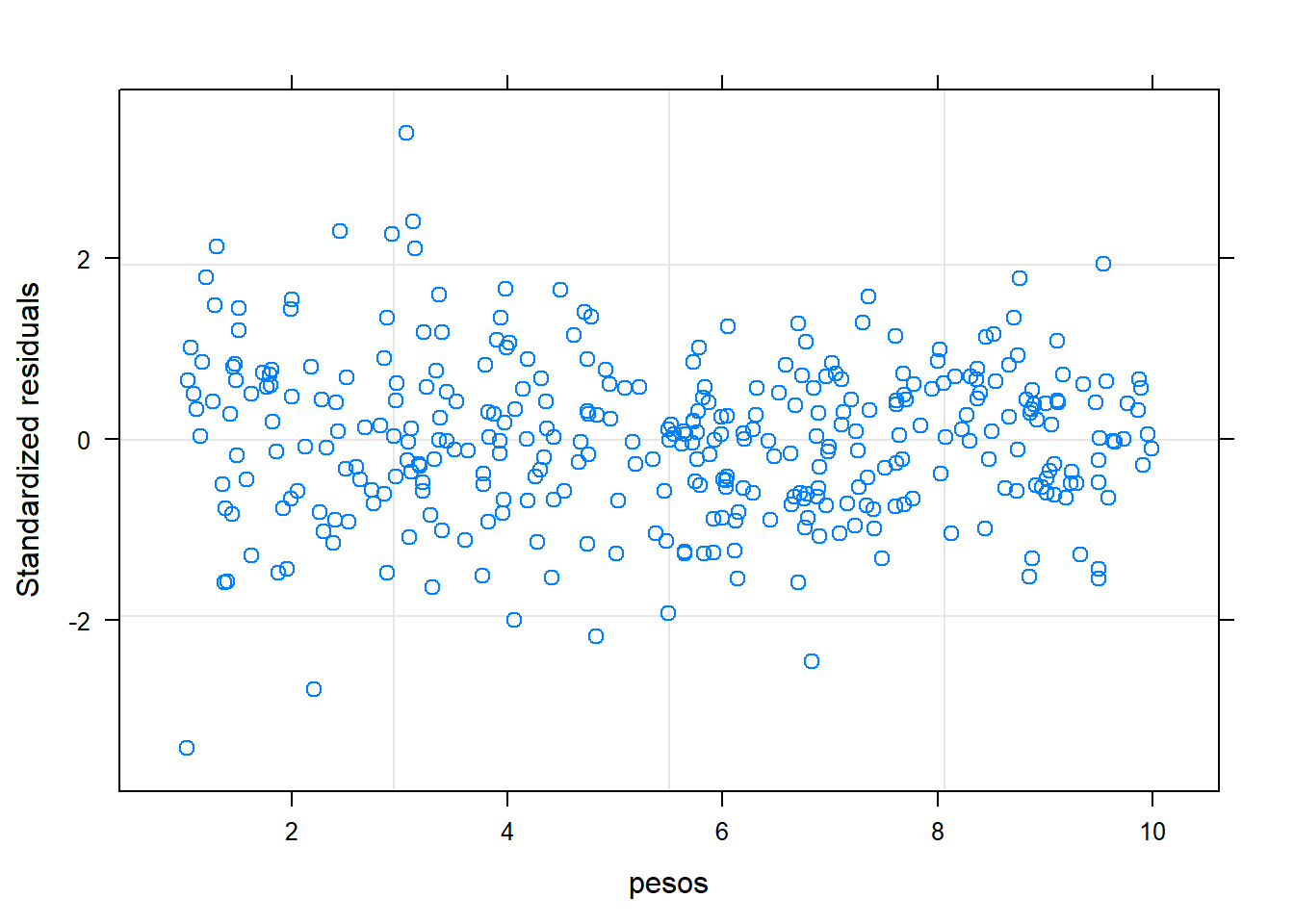
Para espeficar varianzas distintas según la variable pesos, se usa el argumento weights con la función varPower.
modelo2 <- update(modelo, weights = varPower(form=~pesos))
plot(modelo2, form = resid(., type="p") ~ pesos)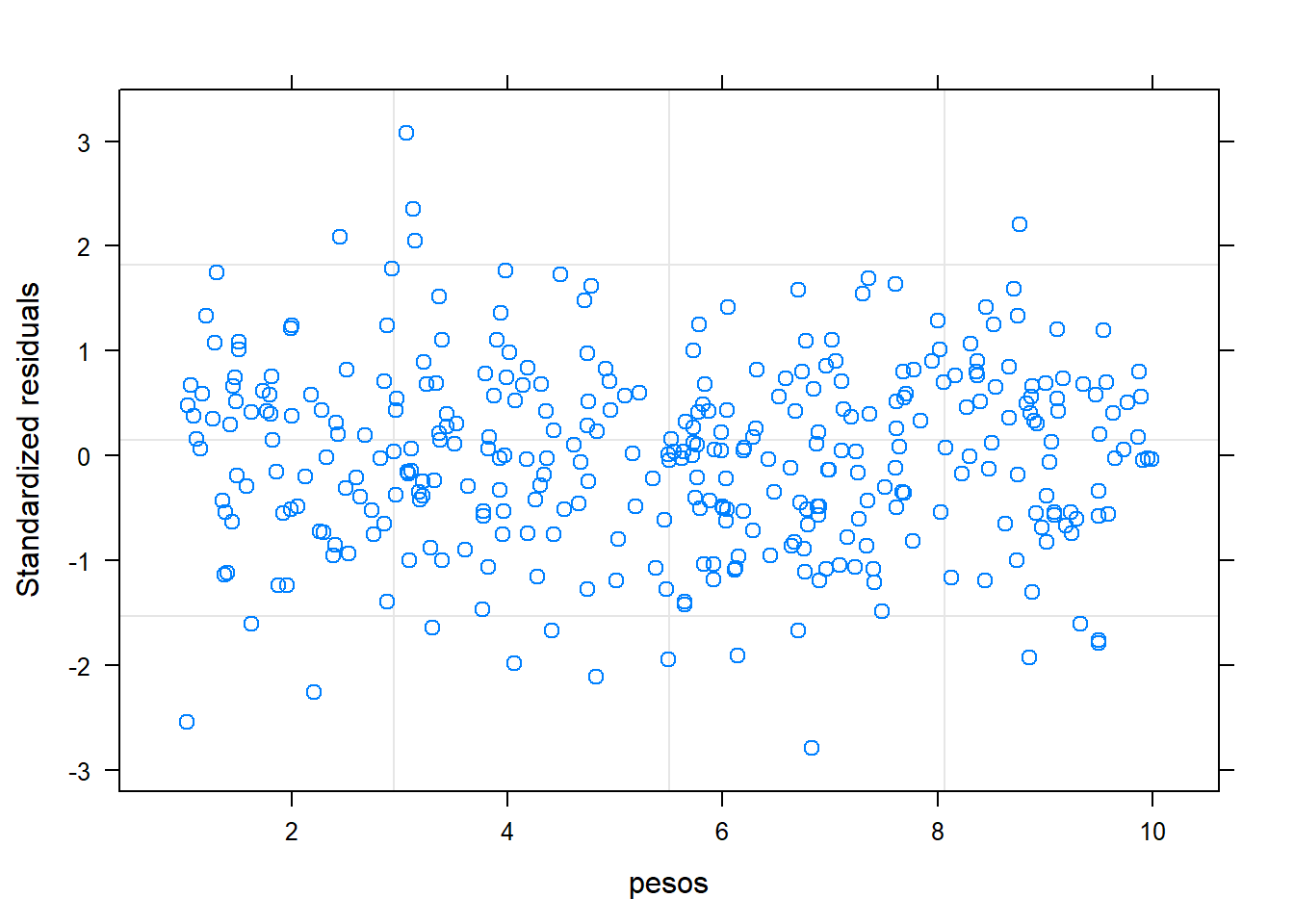
Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
1588.68 1619.451 -786.3398
Random effects:
Formula: ~1 | indiv
(Intercept) Residual
StdDev: 1.874515 3.304256
Correlation Structure: Continuous AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.004226045
Variance function:
Structure: Power of variance covariate
Formula: ~pesos
Parameter estimates:
power
-0.3882534
Fixed effects: respuesta ~ tiempo:tx + tiempo + edad
Value Std.Error DF t-value p-value
(Intercept) 19.653648 1.0755546 248 18.273036 0.0000
tiempo 0.315146 0.2309727 248 1.364430 0.1737
edad 0.269414 0.0213525 98 12.617473 0.0000
tiempo:tx -0.620561 0.1448331 248 -4.284664 0.0000
Correlation:
(Intr) tiempo edad
tiempo -0.058
edad -0.974 0.022
tiempo:tx 0.012 -0.933 -0.017
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-2.79314406 -0.54635193 0.04275529 0.58155772 3.08203173
Number of Observations: 350
Number of Groups: 100 Ahora se observa más homocedesticidad de los residuos.
Fíjate también que estima el valor de la potencia. En este caso la varianza de los residuos es proporcional a pesos^(-0.3807)
Si se quiere forzar a que la varianza sea proporcional a la inversa de los pesos:
Linear mixed-effects model fit by REML
Data: datos
AIC BIC logLik
1588.621 1615.546 -787.3104
Random effects:
Formula: ~1 | indiv
(Intercept) Residual
StdDev: 1.865023 3.932214
Correlation Structure: Continuous AR(1)
Formula: ~tiempo | indiv
Parameter estimate(s):
Phi
0.003969225
Variance function:
Structure: Power of variance covariate
Formula: ~pesos
Parameter estimates:
power
-0.5
Fixed effects: respuesta ~ tiempo:tx + tiempo + edad
Value Std.Error DF t-value p-value
(Intercept) 19.655542 1.0667801 248 18.425111 0.0000
tiempo 0.313887 0.2277158 248 1.378415 0.1693
edad 0.269394 0.0211739 98 12.722882 0.0000
tiempo:tx -0.621442 0.1429088 248 -4.348520 0.0000
Correlation:
(Intr) tiempo edad
tiempo -0.056
edad -0.974 0.021
tiempo:tx 0.009 -0.933 -0.015
Standardized Within-Group Residuals:
Min Q1 Med Q3 Max
-2.88375941 -0.55185483 0.02711027 0.57796918 2.94751228
Number of Observations: 350
Number of Groups: 100 Ahora el valor de power de la “Variance function:” es -0.5, o sea que no se estima.
4.6 Ejercicios
4.6.1 Ejercicio 1
Los datos o2cons, disponibles en el paquete MANOVA.RM, contiene medidas sobre el consumo de oxígeno de los leucocitos (“O2”) de 144 individuos, 72 de ellos asignados al grupo control (“Group=P”) y el resto al grupo de tratamiento con Verum (Group=V). Además, para cada individuo se recoge si los estafilococos (“Staphylococci”) estaban activados o no (0/1). Para cada individuo se tomaron los niveles de oxígeno de los leucocitos después de 6, 12 y 18 minutos.
Haz una pequeña descriptiva de los datos contenidos en esta base de datos
Analiza la evolución del consumo de oxígeno del grupo de tratamiento (“Group=V”).
Compara la evolución del consumo de O2 entre el grupo de intervención con el grupo de tratamiento.
4.6.2 Ejercicio 2
Datos Spruce del paquete nlme.
Los datos “Spruce” disponible en el paquete nlme contiene la información referente al crecimiento de unos árboles.
Describe las variables.
¿Qué matriz de correlaciones de los residuos sería más adecuada para analizar estos datos?
Estima la evolución del crecimiento de los árboles.
¿Cuál es la correlación de dos residuos entre dos medidas consecutivas? ¿Y entre dos medidas separadas por una semana?
Nota: para ajutar el modelo, no tengas en cuenta la variable “plot”.
data(Spruce)
library(ggplot2)
p <- ggplot(data = Spruce, aes(x = days, y = logSize, group = Tree))
p <- p + geom_line(col="grey") + stat_summary(aes(group = 1),
geom = "line", fun = mean, size=2)
p + facet_grid( ~ plot)
p
library(nlme)
library(splines)
modelo <- lme(logSize ~ ns(days,3),
random = ~ ns(days,3) | Tree,
data=Spruce,
correlation = ...,
control=lmeControl(opt="optim",gradHess=FALSE))
# simplifica el modelo y valida...4.6.3 Ejercicio 3
Analiza los datos Remifentanil disponibles en el paquete nlme.
Los datos contienen distintas medidas hechas a lo largo de un tiempo de la concentración de un fármaco (“Remifentanil”) subministrado a 65 pacientes. Entre otras variables se ha recogido el sexo y la edad del paciente.
Estudia la evolución de la concentración del fármaco según el sexo y ajustando por edad y otras variables.
Nota: haz la transformación logarítmica de la concentración.
data(Remifentanil)
Remifentanil <- na.omit(Remifentanil)
Remifentanil <- Remifentanil[order(Remifentanil$Subject,Remifentanil$Time),]
Remifentanil$logconc <- log(Remifentanil$conc)
library(ggplot2)
p <- ggplot(data = Remifentanil, aes(x = Time, y = logconc, group = Subject))
p <- p + geom_line(col="grey") + stat_summary(aes(group = 1),
geom = "line", fun = mean, size=0.5)
p + facet_grid( ~ Sex)
library(nlme)
library(splines)
modelo <- lme(logconc ~ ns(Time,3)*Sex + Age + BSA,
random = ~ns(Time,3) | Subject,
data = Remifentanil,
correlation = corCAR1(form = ~Time | Subject),
control = lmeControl(opt="optim"))4.6.4 Ejercicio 4
Datos sleepstudy del paquete lme4.
Se siguen 18 dindividuos durante 9 días, y se registra el tiempo de reacción.
Modeliza el tiempo de reacción (variable respuesta) a lo largo de los días. Usa tanto polinomios como splines con la función ns.
Puedes usar la función anova con el test LRT para contrastar si es necesario usar términos splines (ns()) o polinomios poly().
4.6.5 Ejercicio 5
Analiza los datos de dietox disponibles en el package geepack.
Haz una descriptiva de las variables (
summary)¿Qué variables son tiempo dependientes y cuáles no?
Fíjate en las siguientes instrucciones:
- ¿qué modelo se está ajustando?
- ¿cuál es la variable respuesta?
- ¿qué variables se consideran con efecto aleatorio
- ¿qué estructura de correlaciones se considera?
Intenta cambiar algunos aspectos (añadir quitar coeficientes aleatorios, estructura de correlación de los errores, simplificar o añadir variables/interacciones,…)
Una vez escogido el modelo final, haz un gráfico de la evolución de la variable respuesta a diferentes niveles de
Start.
library(geepack)
library(nlme)
library(ggeffects)
data(dietox)
summary(dietox)
dietox <- dietox[order(dietox$Pig, dietox$Time),]
dietox <- na.omit(dietox)
mod <- lme(Weight ~ Start*poly(Time,3) + Feed + Evit + Cu,
random = ~ 1 | Litter/Pig,
correlation=corCAR1(form = ~ Time | Litter/Pig),
control=lmeControl(opt="optim"),
data=dietox)
summary(mod)
pr <- ggpredict(mod, c("Time [all]","Start[20,25,30,35]"))
plot(pr)Referencias
Halekoh, Ulrich, and Søren Højsgaard. 2014. “A Kenward-Roger Approximation and Parametric Bootstrap Methods for Tests in Linear Mixed Models – the R Package pbkrtest.” Journal of Statistical Software 59 (9): 1–30. http://www.jstatsoft.org/v59/i09/.
Lawrence, Michael A. 2016. Ez: Easy Analysis and Visualization of Factorial Experiments. http://github.com/mike-lawrence/ez.
Pinheiro, José, Douglas Bates, and R-core. 2020. Nlme: Linear and Nonlinear Mixed Effects Models. https://svn.r-project.org/R-packages/trunk/nlme.