Módulo 4 Fatores e datas
Script da aula do Módulo IV abaixo. Download 04-dates-categorical-variables.R
4.1 O pacote forcats
Em R, os factors são uma classe de dados que permitem categorias ordenadas com um conjunto fixo de valores aceitáveis.
Normalmente, você converteria uma coluna de caractere ou classe numérica em um fator se quiser definir uma ordem intrínseca para os valores (“níveis”) para que eles possam ser exibidos de forma não alfabética em gráficos e tabelas. Outro uso comum de fatores é padronizar as legendas dos gráficos para que não flutuem se certos valores estiverem temporariamente ausentes dos dados.
Vamos demonstrar o uso de funções do pacote forcats (um nome abreviado
para “For categorical variables”) e algumas funções básicas do R.
Isso significa que fatores são úteis. Eles representam uma forma muito prática de lidar com variáveis categorizadas, tanto para fins de modelagem quanto para fins de visualização.
As principais funções do forcats servem para alterar a ordem e modificar
os níveis de um fator. Para exemplificar a utilidade dessas funções,
vamos utilizá-las em situações do dia a dia.
Se você não tem o pacote {forcats} ou {tidyverse}, instalado rode o código abaixo
antes de utilizá-lo:
install.packages("forcats")Nos exemplos a seguir, utilizarmos os seguintes pacotes:
library(forcats)
library(ggplot2)
library(dplyr)4.1.1 O que são fatores?
Fatores são uma classe de objetos no R criada para representar as variáveis categóricas numericamente.
Eles são necessários pois muitas vezes precisamos representar variáveis categóricas como números. Quando estamos fazendo um gráfico, por exemplo, só podemos mapear variáveis numéricas em seus eixos, pois o plano cartesiano é formado por duas retas de números reais.
O que fazemos então quando plotamos uma variável categórica? Nós a transformamos em fatores.
Mas como a manipulação de fatores é diferente da manipulação de números e strings (graças aos famosos levels), tarefas que parecem simples, como ordenar as barras de um gráfico de barras, acabam se tornando grandes desafios quando não sabemos lidar com essa classe.
Nos exemplos a seguir, vamos utilizar a base starwars (do pacote {dplyr}) para aprendermos a fazer as principais operações com fatores utilizando o pacote {forcats}.
4.1.2 Modificando níveis de um fator
Vamos trabalhar primeiro com a coluna sex, que diz qual é o sexo de cada personagem. As possibilidades são:
starwars %>%
pull(sex) %>%
unique()
## [1] "male" "none" "female" "hermaphroditic" NAVeja que se transformarmos a coluna em fator, esses serão os levels da variável, não importa se o sub-conjunto que estivermos observando possua ou não todas as categorias.
starwars %>%
mutate(sex = as.factor(sex)) %>%
pull(sex) %>%
head()
## [1] male none none male female male
## Levels: female hermaphroditic male noneVamos criar um objeto com os 16 primeiros valores da coluna sex já transformados em fator.
fator_sex <- starwars %>%
pull(sex) %>%
as.factor() %>%
head(16)
fator_sex
## [1] male none none male female male female none male male
## [11] male male male male male hermaphroditic
## Levels: female hermaphroditic male nonePara mudar os níveis de um fator, podemos utilizar a função lvls_revalue(). Veja que, ao mudarmos os níveis de um fator, o label de cada valor também muda. Os novos valores precisam ser passados conforme a ordem dos níveis antigos.
lvls_revalue(
fator_sex,
new_levels = c("Fêmea", "Hermafrodita", "Macho", "Nenhum")
)
## [1] Macho Nenhum Nenhum Macho Fêmea Macho Fêmea Nenhum Macho Macho Macho Macho
## [13] Macho Macho Macho Hermafrodita
## Levels: Fêmea Hermafrodita Macho NenhumEssa função é uma boa alternativa para mudar o nome das categorias de uma variável antes de construir um gráfico.
starwars %>%
filter(!is.na(sex)) %>%
count(sex) %>%
mutate(
sex = lvls_revalue(sex, c("Fêmea", "Hermafrodita", "Macho", "Nenhum"))
) %>%
ggplot() +
geom_col(aes(x = sex, y = n)) 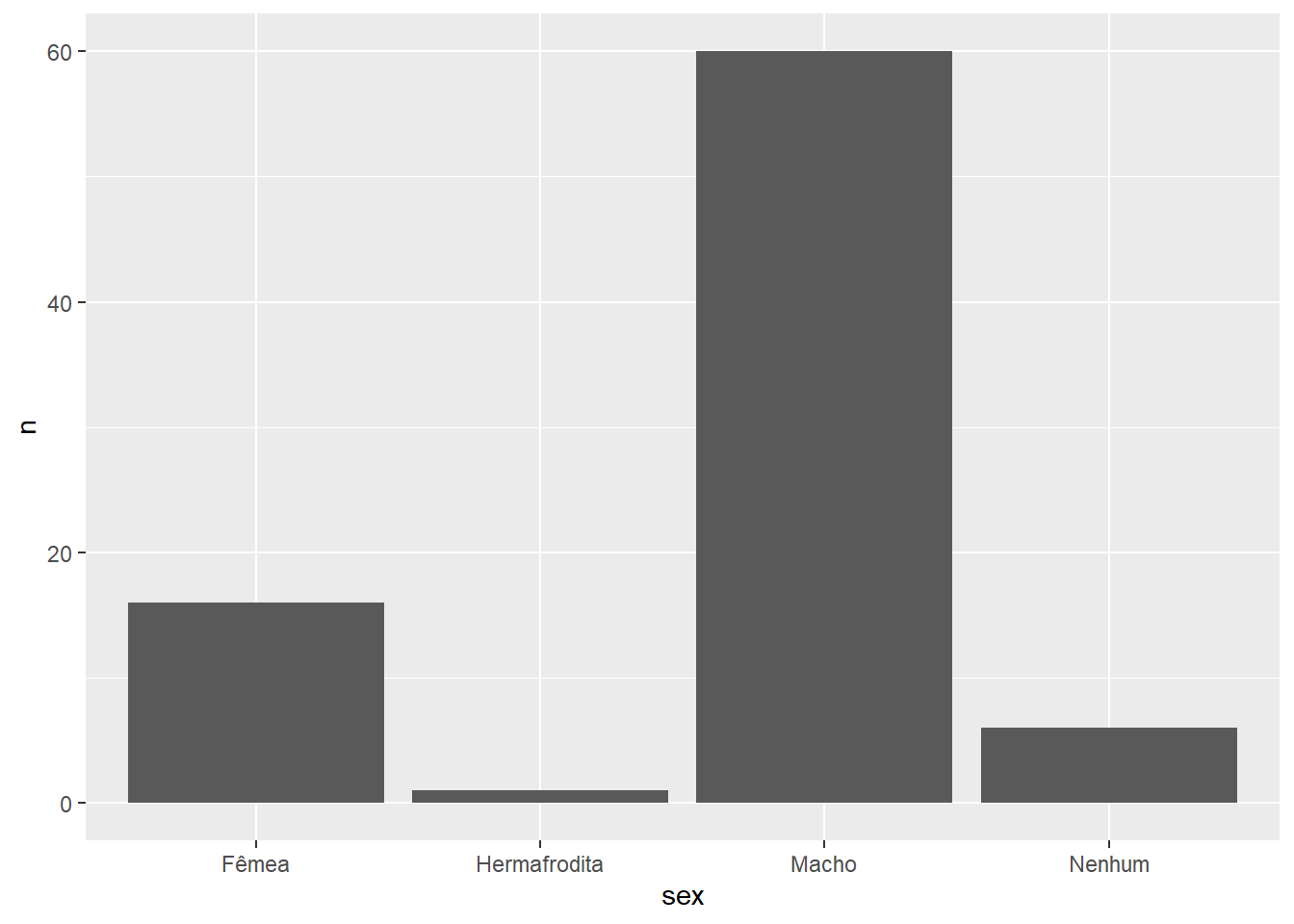
Como as colunas no gráfico respeitam a ordem dos níveis do fator, não importa a ordem que as linhas aparecem na tabela, o gráfico sempre será gerado com as colunas na mesma ordem. Assim, se quiséssemos alterar a ordem das barras do gráfico anterior, precisamos mudar a ordem dos níveis do fator sex.
4.1.3 Mudando a ordem dos níveis de um fator
Para mudar a ordem dos níveis de um fator, podemos utilizar a função lvls_reorder(). Basta passarmos qual a nova ordem dos fatores, com relação à ordem anterior. No exemplo abaixo definimos que, na nova ordem,
a primeira posição terá o nível que estava na terceira posição na ordem antiga;
a segunda posição terá o nível que estava na primeira posição na ordem antiga;
a terceira posição terá o nível que estava na quarta posição na ordem antiga;
a quarta posição terá o nível que estava na segunda posição na ordem antiga.
lvls_reorder(fator_sex, c(3, 1, 4, 2))
## [1] male none none male female male female none male male
## [11] male male male male male hermaphroditic
## Levels: male female none hermaphroditicAssim, poderíamos usar essa nova ordem para ordenar as colunas do nosso gráfico.
starwars %>%
filter(!is.na(sex)) %>%
count(sex) %>%
mutate(
sex = lvls_revalue(sex, c("Fêmea", "Hermafrodita", "Macho", "Nenhum")),
sex = lvls_reorder(sex, c(3, 1, 4, 2))
) %>%
ggplot() +
geom_col(aes(x = sex, y = n)) 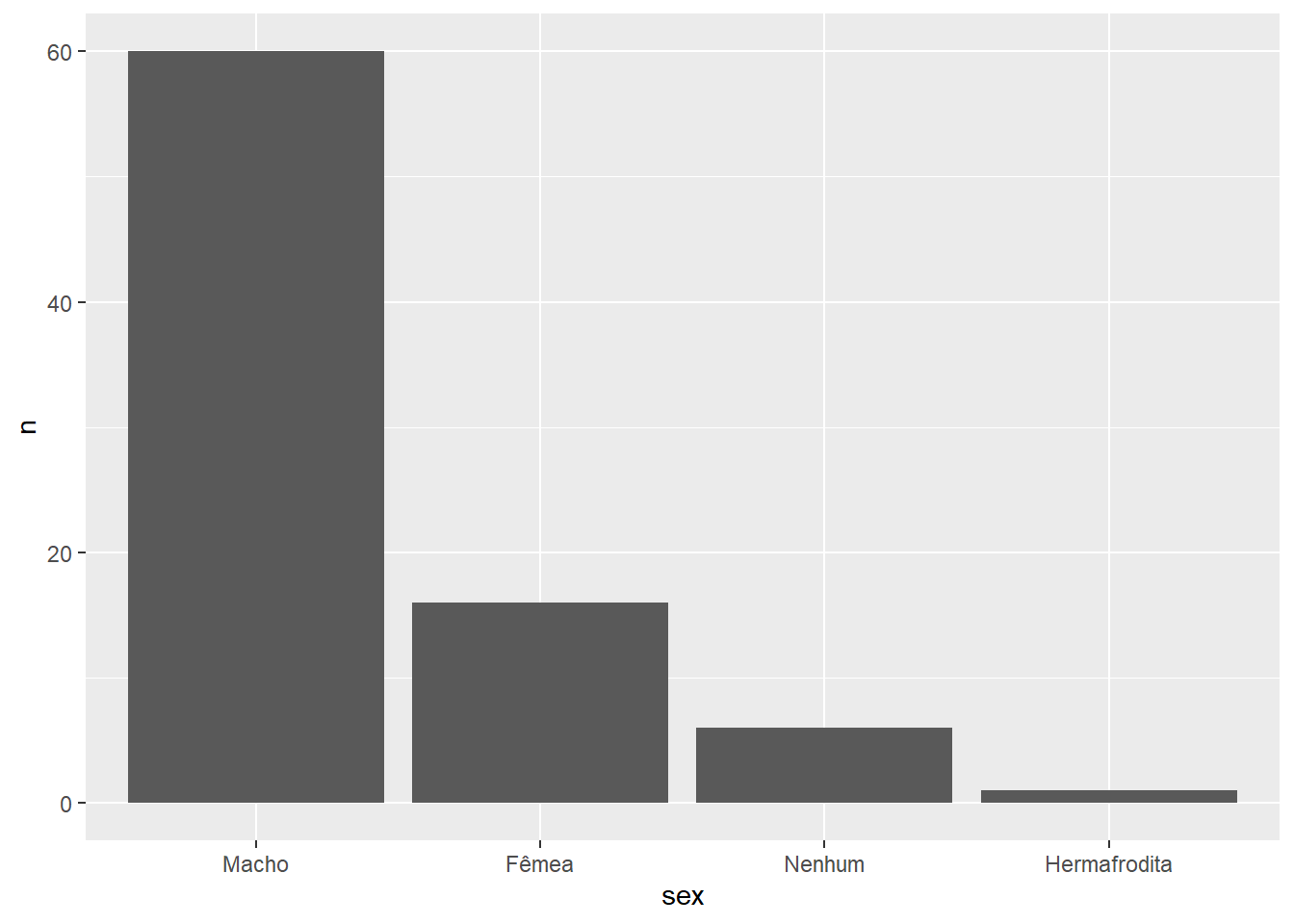
Repare que precisamos passar a nova ordem na mão, o que pode deixar de funcionar se a nossa base mudar (recebemos mais linhas ou fizermos um filtro anteriormente).
Anterior ao mutate() temos a seguinte tabela:
starwars %>%
filter(!is.na(sex)) %>%
count(sex)
## # A tibble: 4 x 2
## sex n
## <chr> <int>
## 1 female 16
## 2 hermaphroditic 1
## 3 male 60
## 4 none 6O que queremos é que os níveis da coluna sex sejam ordenados segundo os valores da coluna n, isto é, quem tiver o maior valor de n deve ser o primeiro nível, o segundo maior valor de n seja o segundo nível e assim por diante.
Podemos melhorar esse código utilizando a função fct_reorder(). Com ela, em vez de definirmos na mão a ordem dos níveis do fator, podemos ordená-lo segundo valores de uma segunda variável.
starwars %>%
filter(!is.na(sex)) %>%
count(sex) %>%
mutate(
sex = lvls_revalue(sex, c("Fêmea", "Hermafrodita", "Macho", "Nenhum")),
sex = fct_reorder(sex, n)
) %>%
ggplot() +
geom_col(aes(x = sex, y = n)) 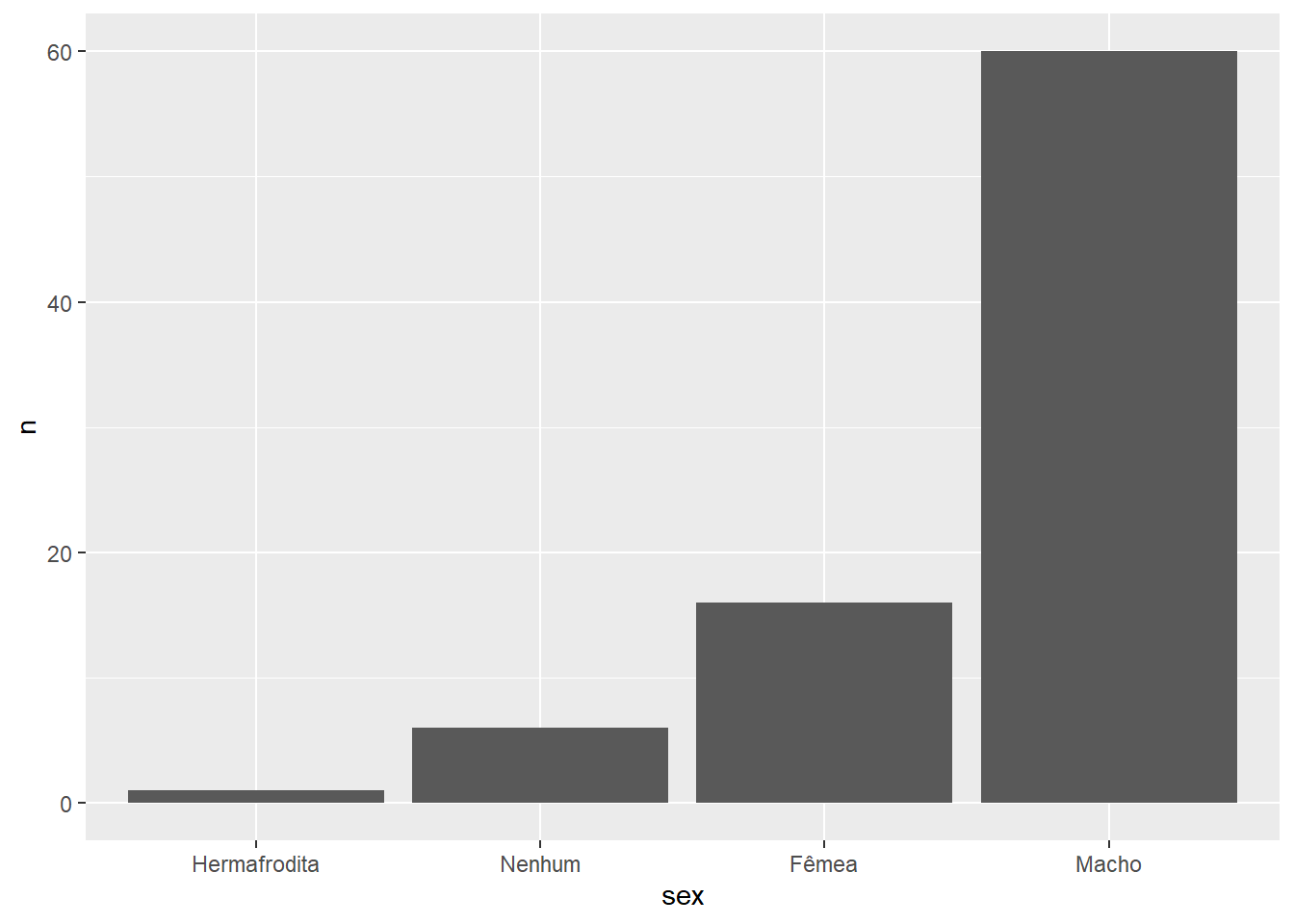
É quase o que queríamos! O problema é que os níveis estão sendo ordenados de forma crescente e gostaríamos de ordenar na ordem decrescente. Para isso, basta utilizarmos o parâmetro .desc.
starwars %>%
filter(!is.na(sex)) %>%
count(sex) %>%
mutate(
sex = lvls_revalue(sex, c("Fêmea", "Hermafrodita", "Macho", "Nenhum")),
sex = fct_reorder(sex, n, .desc = TRUE)
) %>%
ggplot() +
geom_col(aes(x = sex, y = n)) 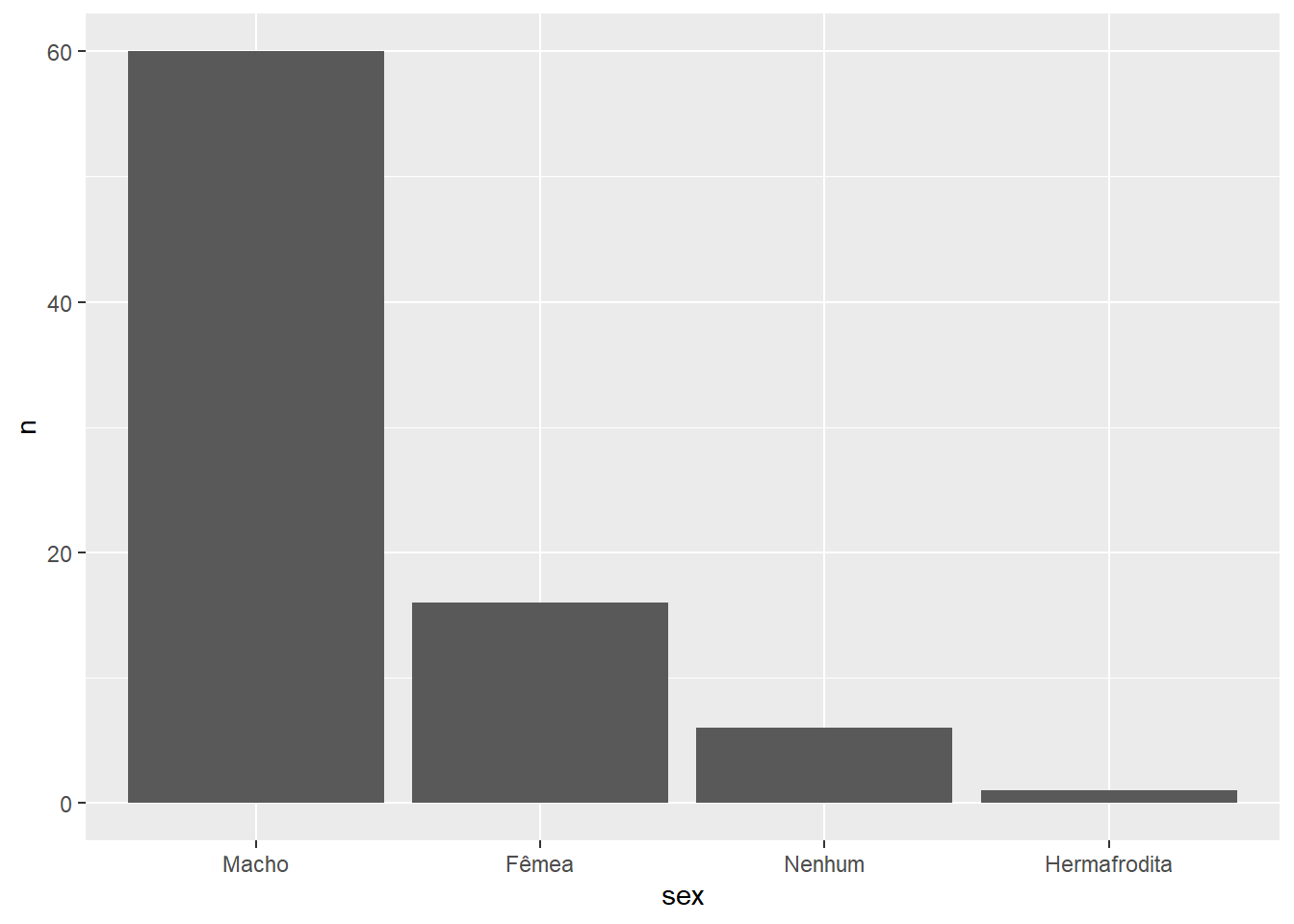
Agora sim! Com esse código, as colunas estarão sendo ordenadas pela frequência, independentemente dos valores de n e sex que chegarem no mutate().
Se olharmos a documentação da função fct_reorder() vamos descobrir que esse exemplo é apenas um caso particular de como podemos utilizá-la. No contexto geral, ela ordena os níveis de um fator segundo uma função dos valores de uma segunda variável.
Se em vez de construirmos um gráfico de barras da frequência da variável sex, construíssemos boxplots da altura para cada sexo diferente, teríamos o gráfico a seguir.
starwars %>%
filter(!is.na(sex)) %>%
ggplot() +
geom_boxplot(aes(x = sex, y = height))
## Warning: Removed 5 rows containing non-finite values (stat_boxplot).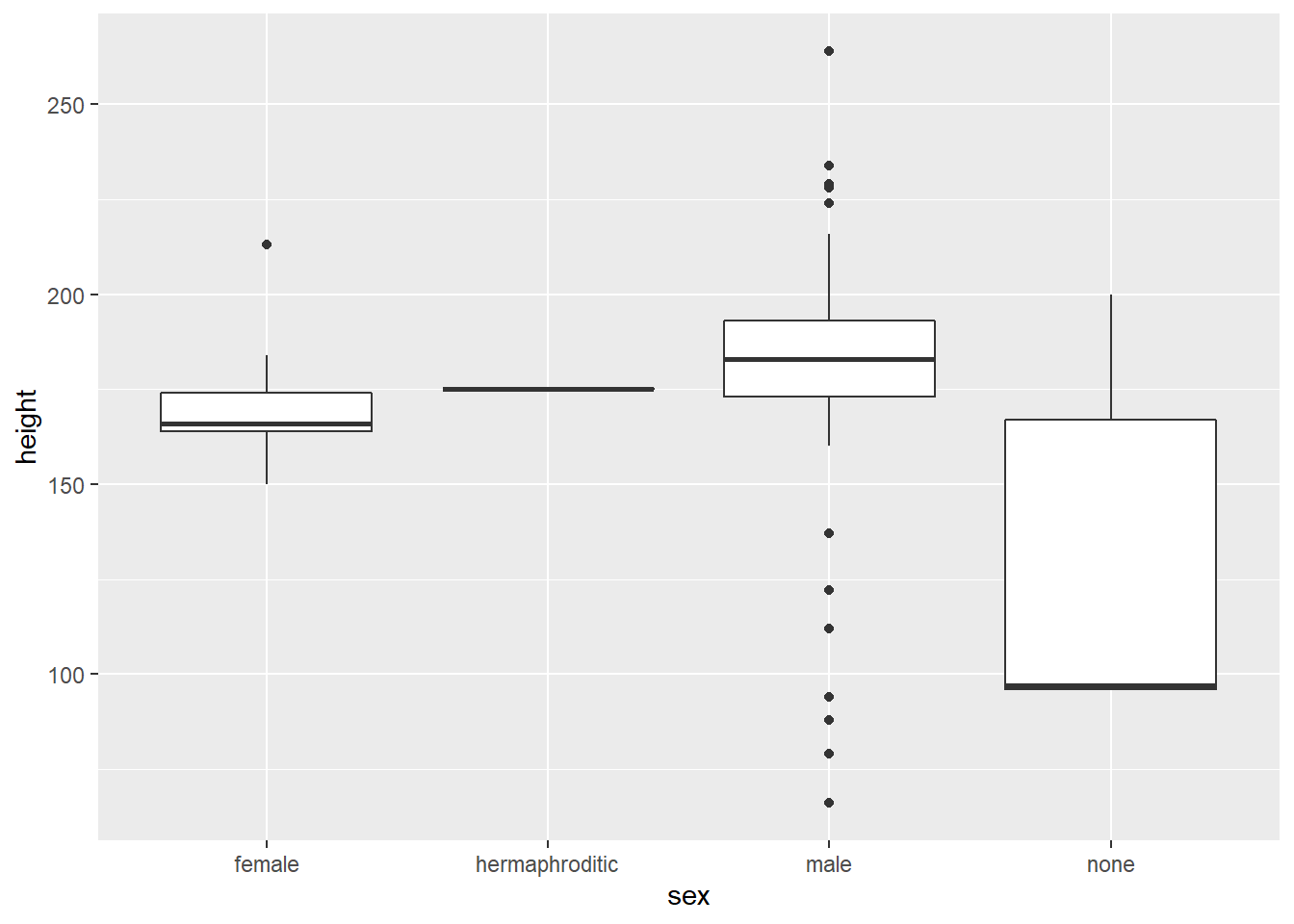 Se quiséssemos ordenar cada boxplot (pela mediana, por exemplo), continuamos podendo usar a função
Se quiséssemos ordenar cada boxplot (pela mediana, por exemplo), continuamos podendo usar a função fct_reorder(). Veja que ela possui o argumento .fun, que indica qual função será utilizada na variável secundária para determinar a ordem dos níveis.
No exemplo abaixo, utilizamos .fun = median, o que significa que, para cada nível da variável sex, vamos calcular a mediana da variável height e ordenaremos os níveis de sex conforme a ordem dessas medianas. Assim, o primeiro nível será o sexo com menor altura mediana, o segundo nível será o sexo com a segunda menor algura media e assim por diante. Se quiséssemos ordenar de forma decrescente, bastaria utilizar o argumento .desc = TRUE.
starwars %>%
filter(!is.na(sex)) %>%
mutate(
sex = lvls_revalue(sex, c("Fêmea", "Hermafrodita", "Macho", "Nenhum")),
sex = fct_reorder(sex, height, .fun = median, na.rm = TRUE)
) %>%
ggplot() +
geom_boxplot(aes(x = sex, y = height))
## Warning: Removed 5 rows containing non-finite values (stat_boxplot).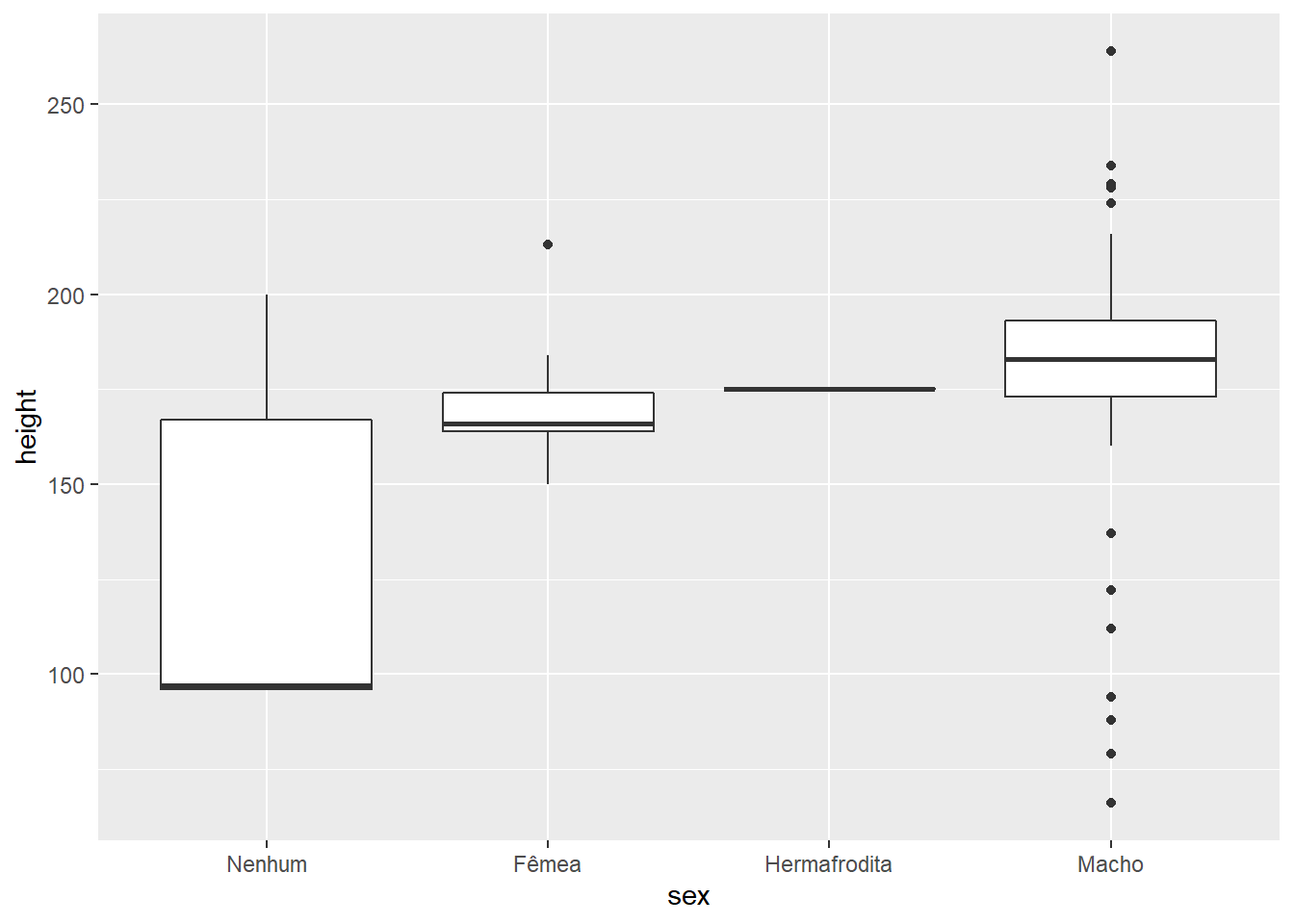 Também poderíamos ordenar pelo máximo, utilizando
Também poderíamos ordenar pelo máximo, utilizando .fun = max. Neste argumento, podemos usar qualquer função sumarizadora: min(), mean(), median(), max(), sd(), var() etc.
starwars %>%
filter(!is.na(sex)) %>%
mutate(
sex = lvls_revalue(sex, c("Fêmea", "Hermafrodita", "Macho", "Nenhum")),
sex = fct_reorder(sex, height, .fun = max, na.rm = TRUE)
) %>%
ggplot() +
geom_boxplot(aes(x = sex, y = height))
## Warning: Removed 5 rows containing non-finite values (stat_boxplot).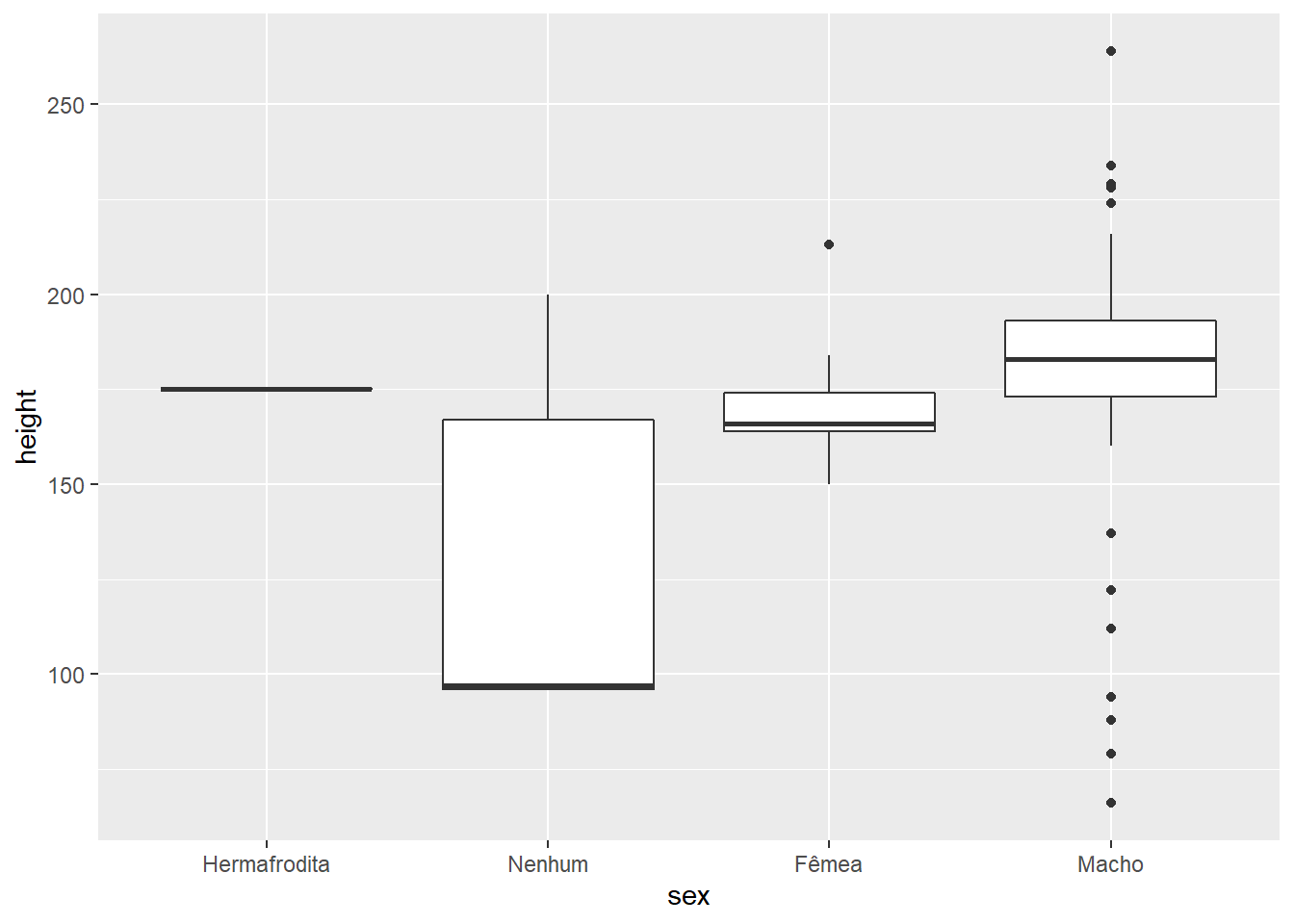
4.1.4 Colapsando níveis de um fator
Imagine que quermos fazer um gráfico de barras com a frequência de personagens por espécie.
starwars %>%
ggplot(aes(x = species)) +
geom_bar()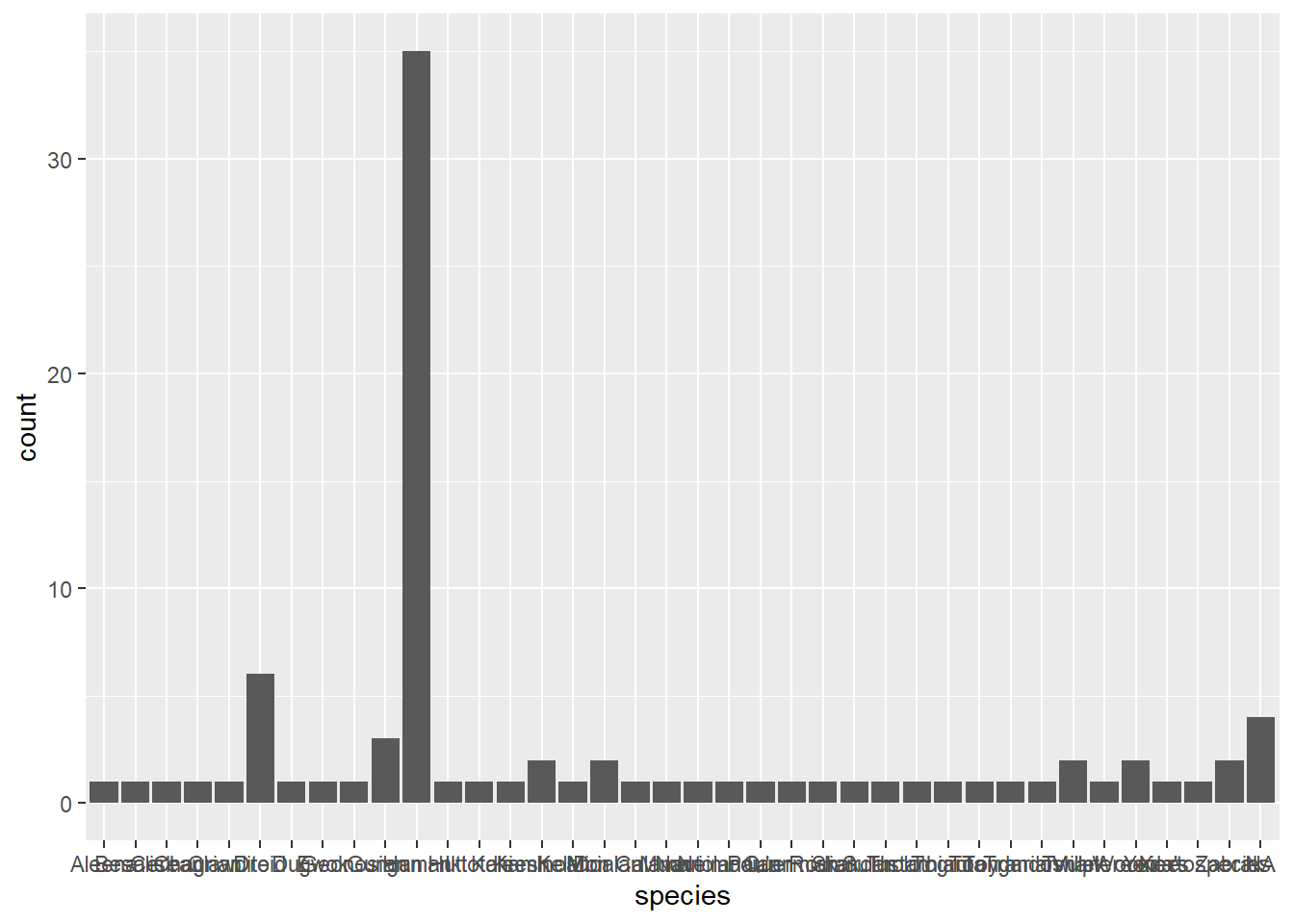
O gráfico resultante é horrível, pois temos muitas espécies diferentes. Uma solução seria agrupar as espécies menos frequentes, criando uma nova categoria (outras, por exemplo).
Para isso, podemos usar a função fct_lump(). Vamos fazer isso primeiro com o vetor de espécies.
fator_especies <- as.factor(starwars$species)
fator_especies
## [1] Human Droid Droid Human Human Human Human Droid Human Human
## [11] Human Human Wookiee Human Rodian Hutt Human Human Yoda's species Human
## [21] Human Droid Trandoshan Human Human Mon Calamari Human Human Ewok Sullustan
## [31] Human Neimodian Human Gungan Gungan Gungan <NA> Toydarian Dug <NA>
## [41] Human Zabrak Twi'lek Twi'lek Vulptereen Xexto Toong Human Cerean Nautolan
## [51] Zabrak Tholothian Iktotchi Quermian Kel Dor Chagrian Human Human Human Geonosian
## [61] Mirialan Mirialan Human Human Human Human Clawdite Besalisk Kaminoan Kaminoan
## [71] Human Aleena Droid Skakoan Muun Togruta Kaleesh Wookiee Human <NA>
## [81] Pau'an Human Human Human Droid <NA> Human
## 37 Levels: Aleena Besalisk Cerean Chagrian Clawdite Droid Dug Ewok Geonosian Gungan Human Hutt Iktotchi Kaleesh Kaminoan Kel Dor Mirialan Mon Calamari ... ZabrakTemos 37 espécies diferentes na base. Podemos deixar apenas as 3 mais frequentes da seguinte forma:
fct_lump(fator_especies, n = 3)
## [1] Human Droid Droid Human Human Human Human Droid Human Human Human Human Other Human Other Other Human Human Other Human Human Droid
## [23] Other Human Human Other Human Human Other Other Human Other Human Gungan Gungan Gungan <NA> Other Other <NA> Human Other Other Other
## [45] Other Other Other Human Other Other Other Other Other Other Other Other Human Human Human Other Other Other Human Human Human Human
## [67] Other Other Other Other Human Other Droid Other Other Other Other Other Human <NA> Other Human Human Human Droid <NA> Human
## Levels: Droid Gungan Human OtherO fator resultante possui 4 níveis: Droid, Gungan, Human e Other. Os 3 primeiros níveis são os mais frequentes, enquanto o nível Other foi atribuído a todos os outros 34 níveis que existiam anteiormente.
Poderíamos definir o nome do nível Others usando o argumento other_level.
fct_lump(fator_especies, n = 3, other_level = "Outras espécies")
## [1] Human Droid Droid Human Human Human Human Droid Human
## [10] Human Human Human Outras espécies Human Outras espécies Outras espécies Human Human
## [19] Outras espécies Human Human Droid Outras espécies Human Human Outras espécies Human
## [28] Human Outras espécies Outras espécies Human Outras espécies Human Gungan Gungan Gungan
## [37] <NA> Outras espécies Outras espécies <NA> Human Outras espécies Outras espécies Outras espécies Outras espécies
## [46] Outras espécies Outras espécies Human Outras espécies Outras espécies Outras espécies Outras espécies Outras espécies Outras espécies
## [55] Outras espécies Outras espécies Human Human Human Outras espécies Outras espécies Outras espécies Human
## [64] Human Human Human Outras espécies Outras espécies Outras espécies Outras espécies Human Outras espécies
## [73] Droid Outras espécies Outras espécies Outras espécies Outras espécies Outras espécies Human <NA> Outras espécies
## [82] Human Human Human Droid <NA> Human
## Levels: Droid Gungan Human Outras espéciesTambém podemos transformar em Outras os níveis cuja frequência relativa é menor que um determinado limite, por exemplo, 2%. No exemplo abaixo, apenas espécies que representam mais de 2% dos personagem na base são mantidas. As demais foram transformadas em Outras.
fct_lump(fator_especies, p = 0.02, other_level = "Outras")
## [1] Human Droid Droid Human Human Human Human Droid Human Human Human Human Wookiee Human Outras Outras Human
## [18] Human Outras Human Human Droid Outras Human Human Outras Human Human Outras Outras Human Outras Human Gungan
## [35] Gungan Gungan <NA> Outras Outras <NA> Human Zabrak Twi'lek Twi'lek Outras Outras Outras Human Outras Outras Zabrak
## [52] Outras Outras Outras Outras Outras Human Human Human Outras Mirialan Mirialan Human Human Human Human Outras Outras
## [69] Kaminoan Kaminoan Human Outras Droid Outras Outras Outras Outras Wookiee Human <NA> Outras Human Human Human Droid
## [86] <NA> Human
## Levels: Droid Gungan Human Kaminoan Mirialan Twi'lek Wookiee Zabrak OutrasCom isso, já conseguimos fazer um gráfico de barras mais agradável.
starwars %>%
filter(!is.na(species)) %>%
mutate(species = fct_lump(species, n = 2)) %>%
ggplot(aes(x = species)) +
geom_bar()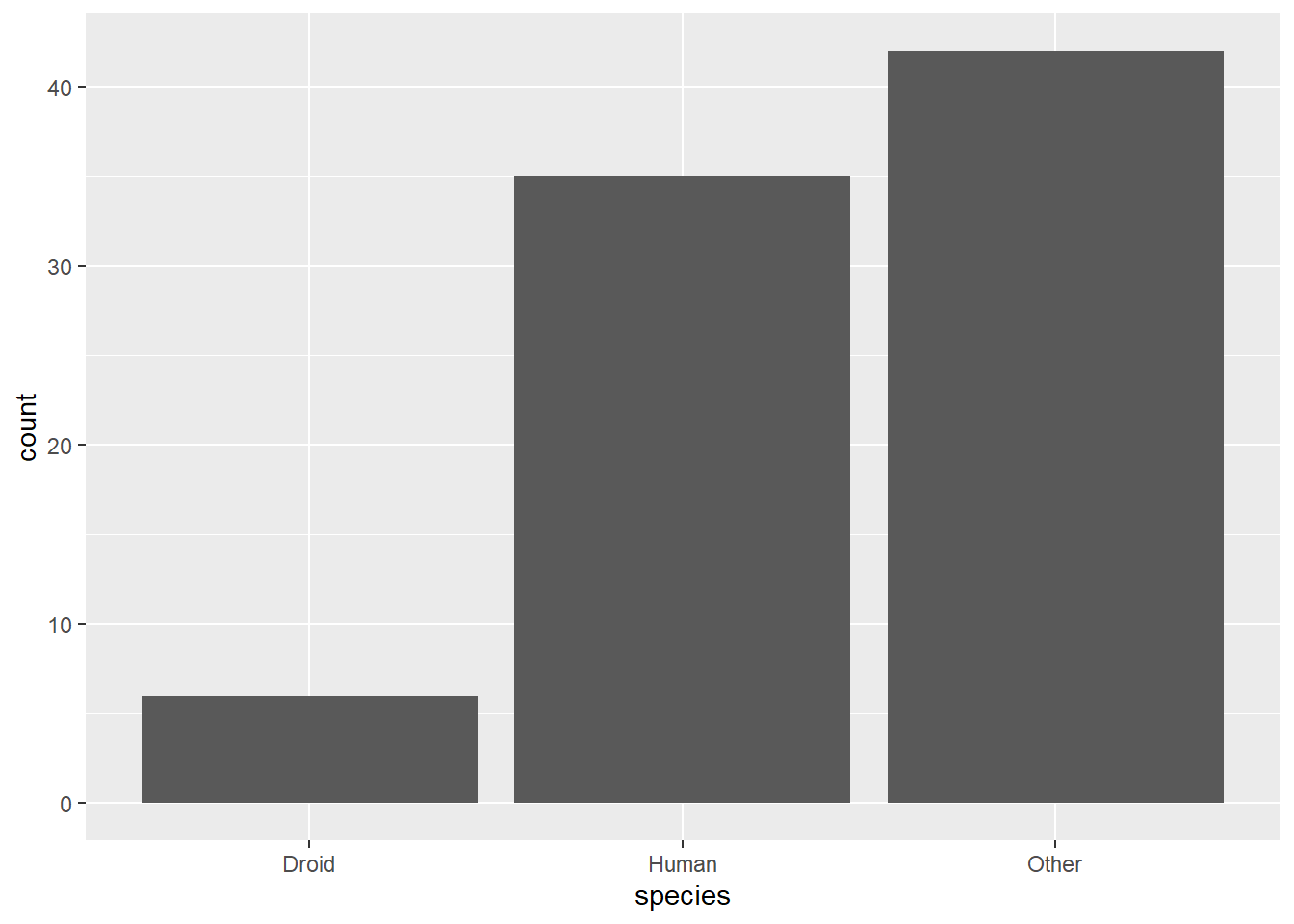
starwars %>%
filter(!is.na(species)) %>%
mutate(
species = fct_lump(species, p = 0.02),
species = fct_infreq(species) # Ordena pela frequência de cada nível
) %>%
ggplot(aes(x = species)) +
geom_bar()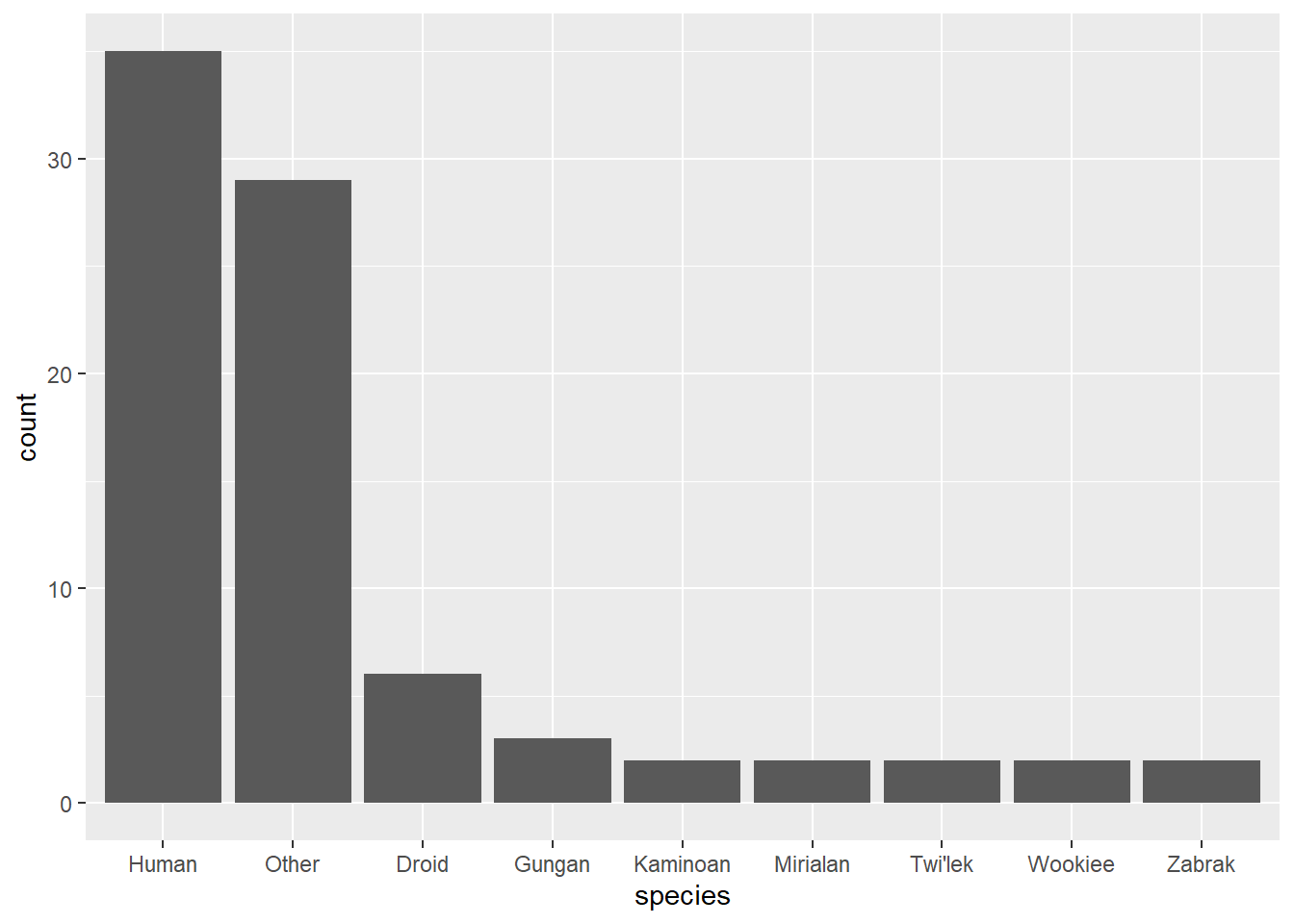
Também é possível colapsar níveis criando manualmente os grupos. Para isso, utilizamos a função fct_collapse(). No exemplo a seguir, reclassificamos os níveis da variável eye_color. Os níveis não listados são reclassificados como "outros".
fator_cor_olhos <- as.factor(starwars$eye_color)
fct_collapse(
fator_cor_olhos,
preto = "black",
castanho = c("brown", "hazel"),
azul_verde = c("blue", "green"),
exotico = c("pink", "red", "white"),
colorido = c("red, blue", "green, yellow"),
other_level = "outros"
)
## Warning: Unknown levels in `f`: green
## [1] azul_verde outros exotico outros castanho azul_verde azul_verde exotico castanho outros azul_verde azul_verde azul_verde castanho
## [15] preto outros castanho azul_verde castanho outros castanho exotico exotico castanho azul_verde outros azul_verde castanho
## [29] castanho preto azul_verde exotico azul_verde outros outros outros azul_verde outros outros castanho castanho outros
## [43] exotico castanho outros preto outros castanho outros preto castanho azul_verde outros outros preto azul_verde
## [57] castanho castanho azul_verde outros azul_verde azul_verde castanho castanho castanho castanho outros outros preto preto
## [71] azul_verde outros colorido outros outros preto colorido azul_verde castanho exotico preto outros castanho castanho
## [85] preto outros castanho
## Levels: preto azul_verde castanho colorido exotico outros4.1.5 Outras funções úteis
A seguir, listamos outras funções úteis do pacote {forcats}, apresentando exemplos simples de como usá-las.
fator <- factor(c("a", "b", "a", "c", "c", "a"))
fator
## [1] a b a c c a
## Levels: a b cfct_c()
Junta dois fatores (e seus níveis).
fator2 <- factor(c("d", "e"))
fct_c(fator, fator2)
## [1] a b a c c a d e
## Levels: a b c d efct_count()
Devolve a frequência dos níveis de um vetor.
fct_count(fator)
## # A tibble: 3 x 2
## f n
## <fct> <int>
## 1 a 3
## 2 b 1
## 3 c 2fct_expand()
Acrescenta níveis a um fator
fct_expand(fator, c("d", "e"))
## [1] a b a c c a
## Levels: a b c d efct_rev()
Inverte os níveis de um fator.
fct_rev(fator)
## [1] a b a c c a
## Levels: c b a4.1.6 Exercícios
A base casas nos exercícios abaixo está no pacote dados:
remotes::install_github("cienciadedatos/dados")1. Qual a diferença nos fatores criados com os códigos abaixo?
fator1 <- as.factor(c("c", "a", "z", "B"))
fator2 <- forcats::as_factor(c("c", "a", "z", "B"))2. Ordene os níveis do fator frutas conforme a sua preferência, isto é, as que você mais gosta primeiro e as que você menos gosta por último.
frutas <- as.factor(c("maçã", "banana", "mamão", "laranja", "melancia"))3. Com base no vetor series, resolva os itens a seguir.
series <- as.factor(c("Game of Thrones", "How I Met your Mother", "Friends", "Lost", "The Office", "Breaking Bad"))a. Ordene os níveis do vetor
seriesconforme a sua preferência, isto é, as que você mais gosta primeiro e as que você menos gosta por último.b. Junte ao vetor
serieso vetornovas_seriesa seguir, reordenando os níveis para manter a sua ordem de preferência.
novas_series <- as.factor(c("The Boys", "Stranger Things", "Queen's Gambit"))- c. Renomeie o níveis do vetor criado no item (b) para os nomes em Português das séries. Mantenha o mesmo nome caso não haja tradução.
4. Ordene as categorias do eixo y do gráfico abaixo para que os pontos no eixo x fique em ordem crescente.
library(dplyr)
library(ggplot2)
mtcars %>%
tibble::rownames_to_column("modelo") %>%
ggplot(aes(x = mpg, y = modelo)) +
geom_point()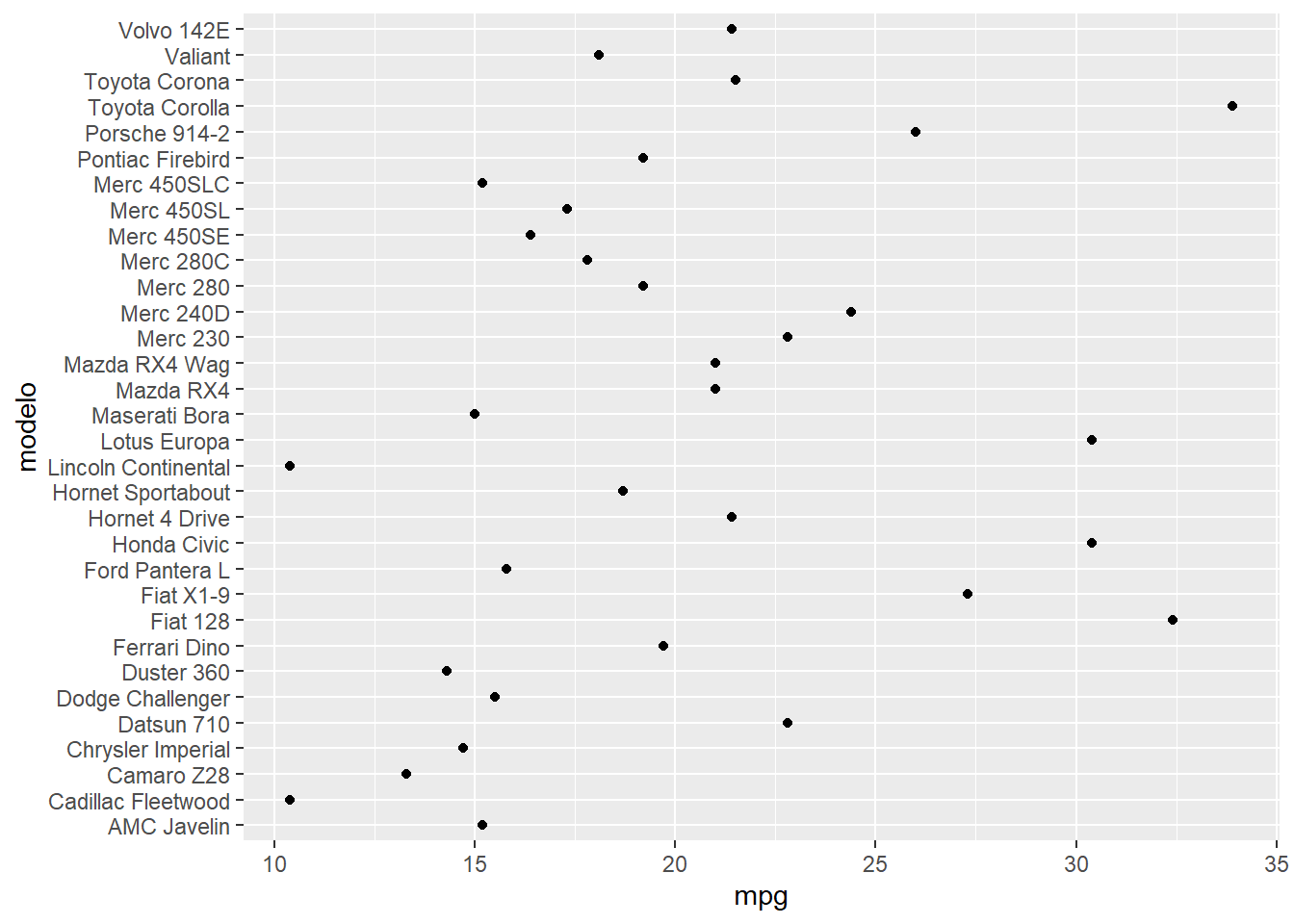
5. Utilize a base dados::casas para fazer um gráfico de barras mostrando as vizinhanças (coluna vizinhanca) com casas mais caras (segundo a coluna venda_valor). O gráfico deve conter as 9 vizinhanças mais frequentes e as demais devem ser agrupadas em uma categoria chamada Outras vizinhanças.
4.2 O pacote lubridate
Trabalhar com datas no R era uma chateação. As funções do R base são, em grande parte, contraintuitivas e podem mudar de acordo com o tipo do objeto que você está usando (data, hora, data/hora). Além disso, características como fusos horários, anos bissextos, horários de verão, entre outras, podem não estar bem especificadas ou nem mesmo sendo levadas em conta.
O pacote {lubridate}, criado por Garrett Grolemund e Hadley Wickham, surgiu para lidar com esses problemas, fazendo o trabalho com datas ser muito mais fácil.
Antes de começar a usar, você precisa instalar e carregar o pacote.
install.packages("lubridate")
library(lubridate)Neste seção, falaremos sobre:
- transformar e extrair datas;
- algumas funções úteis para trabalhar com datas;
- trabalhar com fusos horários; e
- operações aritméticas com datas.
4.2.1 A classe date
Datas no R são tratadas como um tipo especial de objeto, com classe date. Há várias formas de criar objetos dessa classe com o pacote {lubridate}:
data_string <- "21-10-2015"
class(data_string)
## [1] "character"
# data_date <- date(data_string)
# class(data_date)
# data_date
data_as_date <- as_date(data_string)
## Warning: All formats failed to parse. No formats found.
class(data_as_date)
## [1] "Date"
data_as_date
## [1] NA
data_mdy <- dmy(data_string)
class(data_mdy)
## [1] "Date"
data_mdy
## [1] "2015-10-21"Veja que as funções date() e as_date() converteram a string para data, mas não devolveram o valor esperado. A função dmy() resolve esse problema estabelecendo explicitamente a ordem dia-mês-ano. Existem funções para todas as ordens possíveis: dmy(), mdy(), myd(), ymd(), ydm() etc.
As funções date() e as_date() assumem que a ordem segue o padrão da língua inglesa: ano-mês-dia (ymd).
# date("2015-10-21")
as_date("2015-10-21")
## [1] "2015-10-21"Uma grande facilidade que essas funções trazem é poder criar objetos com classe date a partir de números e strings em diversos formatos.
dmy(21102015)
## [1] "2015-10-21"
dmy("21102015")
## [1] "2015-10-21"
dmy("21/10/2015")
## [1] "2015-10-21"
dmy("21.10.2015")
## [1] "2015-10-21"Se além da data você precisar especificar o horário, basta usar as funções do tipo ymd_h(), ymd_hm(), ymd_hms(), sendo que também há uma função para cada formato de dia-mês-ano.
ymd_hms(20151021165411)
## [1] "2015-10-21 16:54:11 UTC"No R base, utilizamos a função as.Date() para transformar uma string em data:
data_base <- as.Date("2015-10-21")
class(data_base)
## [1] "Date"
data_base
## [1] "2015-10-21"Repare que a função date() do R base retorna a data e horário no momento da execução:
base::date()
## [1] "Thu Jul 22 18:29:54 2021"4.2.2 Funções úteis
O lubridate traz diversas funções para extrair os componentes de um objeto da classe date.
second()- extrai os segundos.minute()- extrai os minutos.hour()- extrai a hora.wday()- extrai o dia da semana.mday()- extrai o dia do mês.month()- extrai o mês.year()- extrai o ano.
Os nomes são tão óbvios que a explicação do que cada função faz é praticamente desnecessária.
bday <- ymd_hms("1989-07-29 030142")
bday
## [1] "1989-07-29 03:01:42 UTC"
second(bday)
## [1] 42
day(bday)
## [1] 29
month(bday)
## [1] 7
year(bday)
## [1] 1989
wday(bday)
## [1] 7
wday(bday, label = TRUE)
## [1] sáb
## 7 Levels: dom < seg < ter < qua < qui < ... < sábVocê também pode usar essas funções para atribuir esses componentes a uma data:
data <- dmy("01/04/1991")
data
## [1] "1991-04-01"
hour(data) <- 20
data
## [1] "1991-04-01 20:00:00 UTC"Também existem funções para extrair a data no instante da execução.
# Data e horário do dia em que essa página foi editada pela última vez.
today()
## [1] "2021-07-29"
now()
## [1] "2021-07-29 17:19:26 -03"4.2.3 Fusos horários
Uma característica inerente das datas é o fuso horário. Se você estiver trabalhando com datas de eventos no Brasil e na Escócia, por exemplo, é preciso verificar se os valores seguem o mesmo fuso horário. Além disso, quando a data exata de um evento for relevante, você pode precisar converter horários para outros fusos para comunicar seus resultados em outros países.
Para fazer essas coisas, o lubridate fornece as funções with_tz() e force_tz(). Veja um exemplo de como usá-las.
estreia_GoT <- ymd_hms("2017-07-16 22:00:00", tz = "America/Sao_Paulo")
estreia_GoT
## [1] "2017-07-16 22:00:00 -03"
# Devolve qual seria a data em outro fuso
with_tz(estreia_GoT, tzone = "GMT")
## [1] "2017-07-17 01:00:00 GMT"
with_tz(estreia_GoT, tzone = "US/Alaska")
## [1] "2017-07-16 17:00:00 AKDT"
# Altera o fuso sem mudar a hora
force_tz(estreia_GoT, tzone = "GMT")
## [1] "2017-07-16 22:00:00 GMT"4.2.4 Operações com datas
O pacote lubridate possui ainda funções para calcular intervalos e fazer operações aritméticas com datas.
Intervalos
Intervalos podem ser salvos em objetos com classe interval.
inicio <- dmy("01-04-1991")
evento <- dmy("31-10-1993")
sobrev <- interval(inicio, evento)
sobrev
## [1] 1991-04-01 UTC--1993-10-31 UTC
class(sobrev)
## [1] "Interval"
## attr(,"package")
## [1] "lubridate"Você pode verificar se dois intervalos tem intersecção com a função int_overlaps().
# Outra forma de definir um intervalo: o operador %--%
intervalo_1 <- dmy("01-02-2003") %--% dmy("02-03-2005")
intervalo_2 <- dmy("04-05-2004") %--% dmy("12-03-2012")
int_overlaps(intervalo_1, intervalo_2)
## [1] TRUE4.2.4.1 Aritmética com datas
Veja alguns exemplos de operações aritméticas que você pode fazer utilizando funções do lubridate:
# Somando datas
data <- dmy("31/01/2000")
data + ddays(1)
## [1] "2000-02-01"
data + dyears(1)
## [1] "2001-01-30 06:00:00 UTC"
# A operação abaixo retornaria uma data inválida
# por isso obtemos NA
data + months(1)
## [1] NA
# Criando datas recorrentes
reuniao <- dmy("18-03-2017")
reunioes <- reuniao + weeks(0:10)
reunioes
## [1] "2017-03-18" "2017-03-25" "2017-04-01"
## [4] "2017-04-08" "2017-04-15" "2017-04-22"
## [7] "2017-04-29" "2017-05-06" "2017-05-13"
## [10] "2017-05-20" "2017-05-27"
# Duração de um intervalo
intervalo <-dmy("01-03-2003") %--% dmy("31-03-2003")
intervalo / ddays(1) # Número de dias
## [1] 30
intervalo / dminutes(1) # Número de minutos
## [1] 43200
as.period(intervalo)
## [1] "30d 0H 0M 0S"Para mais informações sobre olubridate, visite o vignette do pacote.
4.2.5 Exercícios
Nos exercícios a seguir, vamos utilizar a base lakers, que contém estatísticas jogo a jogo do Los Angeles Lakers na temporada 2008-2009.
lakers %>% as_tibble()
## # A tibble: 34,624 x 13
## date opponent game_type time period etype
## <int> <chr> <chr> <chr> <int> <chr>
## 1 20081028 POR home 12:00 1 jump ba~
## 2 20081028 POR home 11:39 1 shot
## 3 20081028 POR home 11:37 1 rebound
## 4 20081028 POR home 11:25 1 shot
## 5 20081028 POR home 11:23 1 rebound
## 6 20081028 POR home 11:22 1 shot
## 7 20081028 POR home 11:22 1 foul
## 8 20081028 POR home 11:22 1 free th~
## 9 20081028 POR home 11:00 1 foul
## 10 20081028 POR home 10:53 1 shot
## # ... with 34,614 more rows, and 7 more variables:
## # team <chr>, player <chr>, result <chr>,
## # points <int>, type <chr>, x <int>, y <int>1. Repare que a coluna date no data.frame é um vetor de inteiros. Transforme essa coluna em um vetor de valores com classe date.
2. Crie uma coluna que junte as informações de data e tempo de jogo (colunas date e time) em objetos da classe date.
3. Crie as colunas dia, mês e ano com as respectivas informações sobre a data do jogo.
4. Em média, quanto tempo o Lakers demora para arremessar a primeira bola no primeiro período?
Dica: arremessos são representados pela categoria shot da coluna etype.
5. Em média, quanto tempo demora para sair a primeira cesta de três pontos? Considere toda a base, e cestas de ambos os times.
6. Construa boxplots do tempo entre pontos consecultivos para cada períodos. Considere toda a base de dados e apenas pontos do Lakers.
7. Qual foi o dia e mês do jogo que o Lakers demorou mais tempo para fazer uma cesta? Quanto tempo foi?