Chapter 4 Carga de datos
4.1 Introducción
Ya sabeis que R es un lenguaje de programación orientado al análisis de datos. Lo primero que tenemos que hacer para empezar un análisis con datos en R es, evidentemente, cargar los datos en R.
Vamos a importar y exportar datos en diferentes formatos. Existen mucho tipos de datos, pero nos vamos a centrar en conjuntos de datos que pueden almacenarse en hojas de calculo, ya que creo que es lo que más vais a usar
Este diagrama me parece muy bueno para resumir donde reside el poder de R
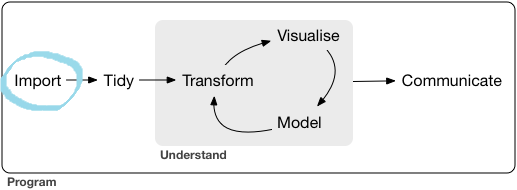
Primer paso: Importar datos (http://r4ds.had.co.nz/)
Cargar datos es una de las primeras frustraciones de alguien que comienza a aprender R. Generalmente piensan: pero si en Excel/SSPSS sólo tengo que pinchar en el fichero!! Como mucho tengo que usar los menús desplegables!!
En R esto también es posible pero lo siento, yo no os voy a mostrar eso. Cuesta un poco más al principio pero una vez lo tienes en tu SCRIPT, es mucho más rápido volver a cargar los datos y hacer todas las operaciones pertinentes para sacar las conclusiones de manera REPRODUCIBLE.
4.2 R base vs nuevos paquetes
R tiene ya unos 20 años. Las funciones de R-base se construyeron pensando en los estadísticos de hace 20 años. Modificar las funciones de R-base haría que código antiguo dejase de funcionar, así que la mayoría de avances y mejoras se producen en los packages.
En el caso de la carga de datos vamos a trabajar con el paquete readr, que es muy parecido a R-base pero en cierto sentido mejora las funciones y las hace más consistentes. Por ejemplo para leer datos CSV la función de R-base es read.csv(); mientas que la función equivalente de readr es read_csv(). Las dos hacen lo mismo, leer datos en formato CSV, pero las nuevas funciones tienen algunas ventajas:
Son más rápidas.
En lugar de generar data.frames, producen tibbles. Las tibbles son en realidad data.frames pero con algunas particularidades.
Las tibbles o “data frames tuneados” tienen unas ciertas ventajas: no convierten por defecto vectores de texto en factores, no usan row names, ni transforman los column names (estás 3 cosas que sí hacen los “data.frames tradicionales” pueden provocar algunas complicaciones, así que mejor tener herramientas que las sorteen).
4.3 Obtener información de una función
Si quisieramos ver todas las funciones (en realidad todos los objetos) que tiene un paquete concreto, por ejemplo ‘readr’, haríamos: ‘ls(“package:readr”, all = TRUE)’
Si queremos ver la documentación de una función concreta; por ejemplo de la función ‘read_csv()’, tenemos que utilizar ‘help()’ o el operador ‘?’. Veámoslo:
#- visualiza la documentación de la función read_csv del package readr.
help(read_csv, package = "readr")
#- si el pkg ya estuviera cargado, funcionaría también así. Si hubiese ambigüedad, RStudio nos avisaría
help(mean)
#- en ese caso tambien funcionaría con el operador ?
?meanAbajo tienes un ejemplo con la función ‘mean()’.

Como leer las ayudas de R
4.3.1 Datos precargados en R
R-base viene con muchos datos precargados; concretamente en el pkg de R-base llamado ‘datasets’. Además muchos packages contienen también conjuntos de datos. Para ver los datos que tenemos precargados y disponibles en R se usa la función ‘data()’:
Si queremos ver los datos que hay en un package concreto usaremos data(package = “nombre_pkg”)
4.3.2 Tipos de bases de datos que veremos
Introduciremos funciones para importar/exportar datos de los siguientes formatos:
- Datos en formato texto (o tabulares)
- CSV: .csv (comma separated values o , en castellano, datos separados por comas)
- otros datos en formato texto
- Formatos de otros programas (software propietario)
- EXCEL: .xls y .xlsx
4.3.3 Datos separados por carácteres
Estamos acostumbrados a visualizar datos en formato tabla. Generalmente las columnas son variables y las filas son observaciones o individuos de esas variables.
Las columnas se separan con un carácter y las filas con un salto de linea. Este carácter puede ser un espacio, un tabulador, una coma, punto y coma etc… El formato más extendido es el CSV, donde las observaciones están separadas por comas.
Estos datos se pueden visualizar en los editores de texto y por eso también se llaman datos en formato texto.
El package ‘readr’ lee datos tabulares con las siguientes funciones:
- si los datos están delimitados por caracteres utiliza: ‘read_delim()’, ‘read_csv()’, ‘read_tsv()’
- si los datos son de anchura fija: ‘read_fwf()’ y ‘read_table()’
Sólo veremos como importar/exportar datos tabulares del primer tipo; es decir, separados por caracteres. Comenzaremos con el formato CSV que es el más utilizado.
4.3.3.1 CSV
CSV significa “comma separated data”. En realidad CSV es un caso particular de “tabular o text data”
Vamos a comenzar exportando el data.frame ‘iris’, presente en R-Base a formato CSV y luego importarlo.
data("iris")
head(iris)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosaPara exportar ‘iris’ a un fichero en formato CSV utilizaremos la función ‘write_csv()’: solo hay que decirle el objeto que queremos exportar (en este caso un data.frame) y el nombre (junto con la ruta) del archivo donde queremos guardarlo.
Podemos especificar la ruta completa. Yo pongo la mia, pero vosotros tendréis que poner la que os corresponda en cada caso. Por ejemplo:
write_csv(iris, path = "C:/Users/ip30/Dropbox/Terreno/cursos/Formadores IT/Curso intro R/datos/iris.csv")Si estamos usando un proyecto (tal y como vimos en el capítulo 2) o establecido nuestro directorio de trabajo, podemos guardar objetos directamente en ese directorio usando:
Recuerda que para saber cual es tu directorio de trabajo puedes usar la función ‘getwd()’ y puedes cambiarlo desde los menús de RStudio o con ‘setwd()’
Otra forma que uso muchas veces es copiar mi directorio de trabajo en un objeto
Y cuando quiero guardar uso la función ‘paste()’ o ‘paste0()’ para crear el path que me interesa
## [1] "C:/Users/ip30/Dropbox/Terreno/cursos/Formadores IT/Curso intro R/Curso R base/iris.csv"4.3.3.1.1 Algunas opciones de ‘read_csv()’ que conviene conocer
A veces los datos tienen ciertos problemas que hay que arreglar; por lo que conviene conocer algunas opciones de read_csv():
col_names: ‘read_csv()’ asume que la primera fila contiene los nombres de las variables. Esto puede cambiarse con col_names = FALSE. Puedes proveer nombres a las variables (o columnas) con col_names = c(“X1”, “X2”)
‘read_csv()’ por defecto importa todas las filas del archivo, pero puedes hacer que comience a importar en la fila que quieras con ‘skip = n’
‘na’: En algunos ficheros con datos tabulares los NAs se especifican con algún carácter. Esto podemos tratarlo al leer los datos con el argumento ‘na = “xxx”’
Por ejemplo, el chunk que ves abajo utiliza read_csv() para cargar el fichero “iris.csv”. Comienza a importar datos desde la quinta columna, trata los valores 0.2 como NAs y provee un vector con los nombres que queremos para las variables (o columnas)
mi_IRIS <- read_csv(paste0(data.link,"iris.csv"), skip = 5, na = c("0.2"),
col_names = c("X1", "X2", "X3", "X4", "X5"))
## Parsed with column specification:
## cols(
## X1 = col_double(),
## X2 = col_double(),
## X3 = col_double(),
## X4 = col_double(),
## X5 = col_character()
## )
mi_IRIS
## # A tibble: 146 x 5
## X1 X2 X3 X4 X5
## <dbl> <dbl> <dbl> <dbl> <chr>
## 1 5 3.6 1.4 NA setosa
## 2 5.4 3.9 1.7 0.4 setosa
## 3 4.6 3.4 1.4 0.3 setosa
## 4 5 3.4 1.5 NA setosa
## 5 4.4 2.9 1.4 NA setosa
## 6 4.9 3.1 1.5 0.1 setosa
## 7 5.4 3.7 1.5 NA setosa
## 8 4.8 3.4 1.6 NA setosa
## 9 4.8 3 1.4 0.1 setosa
## 10 4.3 3 1.1 0.1 setosa
## # ... with 136 more rows4.3.3.2 Otros datos tabulares
En realidad, todos los datos tabulares (separados por caracteres) son muy similares. Solo se diferencian en el carácter que hace de separador.
El package ‘readr’ tiene una función especifica para cada tipo de datos tabulares. Por ejemplo, si el separador es un punto y coma, la función para importar estos datos es ‘read_csv2()’; si el separador es un tabulador, la función es ‘read_tsv()’. Pero también tiene una función genérica que sirve para cualquier tipo de separador: ‘read_delim()’. Yo recomiendo usar esta última función.
Por ejemplo, podemos cargar el fichero “iris.csv” que hemos exportado anteriormente utilizando la función genérica ‘read_delim()’, solo hay que decirle que el separador es una coma. Se lo decimos con la opción ‘delim = “,”’. Veámoslo:
iris <- read_delim("iris.csv", delim = ",")
## Parsed with column specification:
## cols(
## Sepal.Length = col_double(),
## Sepal.Width = col_double(),
## Petal.Length = col_double(),
## Petal.Width = col_double(),
## Species = col_character()
## )Como el formato tabular más extendido es el CSV; en general, no tendremos necesidad de exportar datos tabulares separados por caracteres distintos a la coma, pero si quisiéramos hacerlo, podríamos hacerlo con ‘write_delim()’
Y si quisiéramos importarlos, tendríamos que hacer:
## Parsed with column specification:
## cols(
## Sepal.Length = col_double(),
## Sepal.Width = col_double(),
## Petal.Length = col_double(),
## Petal.Width = col_double(),
## Species = col_character()
## )## # A tibble: 150 x 5
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## <dbl> <dbl> <dbl> <dbl> <chr>
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosa
## 7 4.6 3.4 1.4 0.3 setosa
## 8 5 3.4 1.5 0.2 setosa
## 9 4.4 2.9 1.4 0.2 setosa
## 10 4.9 3.1 1.5 0.1 setosa
## # ... with 140 more rowsPROBAMOS A CARGAR LOS DATOS QUE ME PASASTEIS?
4.3.4 Excel
Creo que todos conocemos Excel…
Vamos a usar los package ‘readxl’ y ‘writexl’ para exportar e imoprtar ficheros excel, respectivamante
4.3.4.1 Exportar a excel
Instalemos el paquete ‘writexl’ y veámoslo:
if (!require("writexl")) install.packages("writexl")
library(writexl)
write_xlsx(iris, paste0(data.link,"iris.xlsx"))La función ‘write_xlsx()’ permite permite crear Excel enormes de manera eficiente mediante la especificación ‘use_zip64 = T’:
La función ‘write_xlsx()’ permite añadir datos varias pestañas a la vez. Para ello:
4.3.4.2 Imortar archivos excel
Instalemos el paquete ‘readxl’ y veámoslo:
if (!require("writexl")) install.packages("writexl")
library(writexl)
iris2 = read_xlsx(paste0(data.link,"iris.xlsx"))
iris2
## # A tibble: 150 x 5
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## <dbl> <dbl> <dbl> <dbl> <chr>
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosa
## 7 4.6 3.4 1.4 0.3 setosa
## 8 5 3.4 1.5 0.2 setosa
## 9 4.4 2.9 1.4 0.2 setosa
## 10 4.9 3.1 1.5 0.1 setosa
## # ... with 140 more rowsPor defecto nos ha cargado la primera pestaña, pero si queremos cargar algún otra, podemos usar la especificación ‘sheet =’:
## # A tibble: 146 x 5
## X1 X2 X3 X4 X5
## <dbl> <dbl> <dbl> <dbl> <chr>
## 1 5 3.6 1.4 NA setosa
## 2 5.4 3.9 1.7 0.4 setosa
## 3 4.6 3.4 1.4 0.3 setosa
## 4 5 3.4 1.5 NA setosa
## 5 4.4 2.9 1.4 NA setosa
## 6 4.9 3.1 1.5 0.1 setosa
## 7 5.4 3.7 1.5 NA setosa
## 8 4.8 3.4 1.6 NA setosa
## 9 4.8 3 1.4 0.1 setosa
## 10 4.3 3 1.1 0.1 setosa
## # ... with 136 more rows## # A tibble: 146 x 5
## X1 X2 X3 X4 X5
## <dbl> <dbl> <dbl> <dbl> <chr>
## 1 5 3.6 1.4 NA setosa
## 2 5.4 3.9 1.7 0.4 setosa
## 3 4.6 3.4 1.4 0.3 setosa
## 4 5 3.4 1.5 NA setosa
## 5 4.4 2.9 1.4 NA setosa
## 6 4.9 3.1 1.5 0.1 setosa
## 7 5.4 3.7 1.5 NA setosa
## 8 4.8 3.4 1.6 NA setosa
## 9 4.8 3 1.4 0.1 setosa
## 10 4.3 3 1.1 0.1 setosa
## # ... with 136 more rowsTambién podemos ver que pestañas tiene el archivo de excell mediante la función ‘excel_sheets()’
## [1] "Iris1" "Iris2"La función ‘read_xlsx()’ tiene más posibilidades; como ejemplo, la opción ‘skip = 40’ permite empezar a importar a partir de la fila 40.
Si queremos importar todos los libros (o sheets) de un archivo Excel, podemos hacerlo así:
file_path = paste0(data.link,"iris.xlsx")
lista_iris <- lapply(excel_sheets(file_path), read_excel, path = file_path)Hemos guardado los 2 sheets del archivo “iris.xlsx” en un objeto R llamado lista_iris Este objeto es una lista con 2 elementos. Cada elemento contiene los datos de cada uno de los 2 sheets. Podemos verlo con ‘str()’:
## List of 2
## $ : tibble [150 x 5] (S3: tbl_df/tbl/data.frame)
## ..$ Sepal.Length: num [1:150] 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## ..$ Sepal.Width : num [1:150] 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## ..$ Petal.Length: num [1:150] 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## ..$ Petal.Width : num [1:150] 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## ..$ Species : chr [1:150] "setosa" "setosa" "setosa" "setosa" ...
## $ : tibble [146 x 5] (S3: tbl_df/tbl/data.frame)
## ..$ X1: num [1:146] 5 5.4 4.6 5 4.4 4.9 5.4 4.8 4.8 4.3 ...
## ..$ X2: num [1:146] 3.6 3.9 3.4 3.4 2.9 3.1 3.7 3.4 3 3 ...
## ..$ X3: num [1:146] 1.4 1.7 1.4 1.5 1.4 1.5 1.5 1.6 1.4 1.1 ...
## ..$ X4: num [1:146] NA 0.4 0.3 NA NA 0.1 NA NA 0.1 0.1 ...
## ..$ X5: chr [1:146] "setosa" "setosa" "setosa" "setosa" ...