2 Transformando nuestros datos (data wrangling)
Al terminar este capítulo ustedes van a poder:
- Entender qué es un paquete de R, cómo se instala y cómo se carga
- Comprender a qué se llama Ciencia de Datos
- Realizar lastransformaciones mas comunes de datos
- Realizar algunos gráficos elementalesEn el primer capítulo de este libro realizamos algunas de las etapas de un típico proyecto de Ciencia de Datos: leímos los datos y realizamos un breve análisis descriptivo. Sin embargo, en la práctica esto no suele suceder de esta manera e inmediatamente después de leer los datos tenemos que realizar una serie de transformaciones que, como regla general, ocupa la mayor parte del tiempo. Este segundo capítulo tiene el objetivo de cubrir las herramientas que tidyverse nos ofrece para realizar estas transformaciones, conocidas de manera coloquial como Data Wrangling.
Vamos a intentar cumplir este objetivo en dos partes. En primer lugar, vamos a pasar lista de las funciones que los van a acompañar de ahora en adelante para hacer las transformaciones de datos necesarias. En segundo lugar, vamos a usarlas para llegar al mismo data frame de precio de los inmuebles que se usó en el primer capítulode los datos tal como son descargados desde Properati
2.1 Instalando nuestro primer paquete en R: tidyverse
De ahora en adelante vamos a ir usando funciones que no vienen instaladas con la instalación de R base que ya hicieron. Este conjunto de funciones, que hacen a R realmente poderoso a partir de la colaboración de miles de personas, se guardan en paquetes (packages, en inglés) que podemos instalar de una manera muy simple: con la función install.packages()
install.packages('tidyverse')¡Perfecto! una vez que lo hayan instalado no lo tienen que hacer más en esa computadora. Ya van a poder usar las funciones que tiene, que nos serán muy importantes. Para usarlas, solo tenemos que llamar cada vez que abramos RStudio - en rigor, cada vez que tengamos una nueva sesión de R - a la función library() o require(), ambas hacen lo mismo.
Pero ¿Qué es tidyverse? Es un conjunto de packages. Según RStudio, Tidyverse es “[…] a coherent system of packages for data manipulation, exploration and visualization that share a common design philosophy”.
En la práctica, tidyverse nos va a permitir articular e implementar diversas facetas del proceso de análisis de datos de una manera unificada. Tiene una curva de aprendizaje, por lo cual no se preocupen si en esta primera sección hay algunas cosas que no terminan de entenderse.
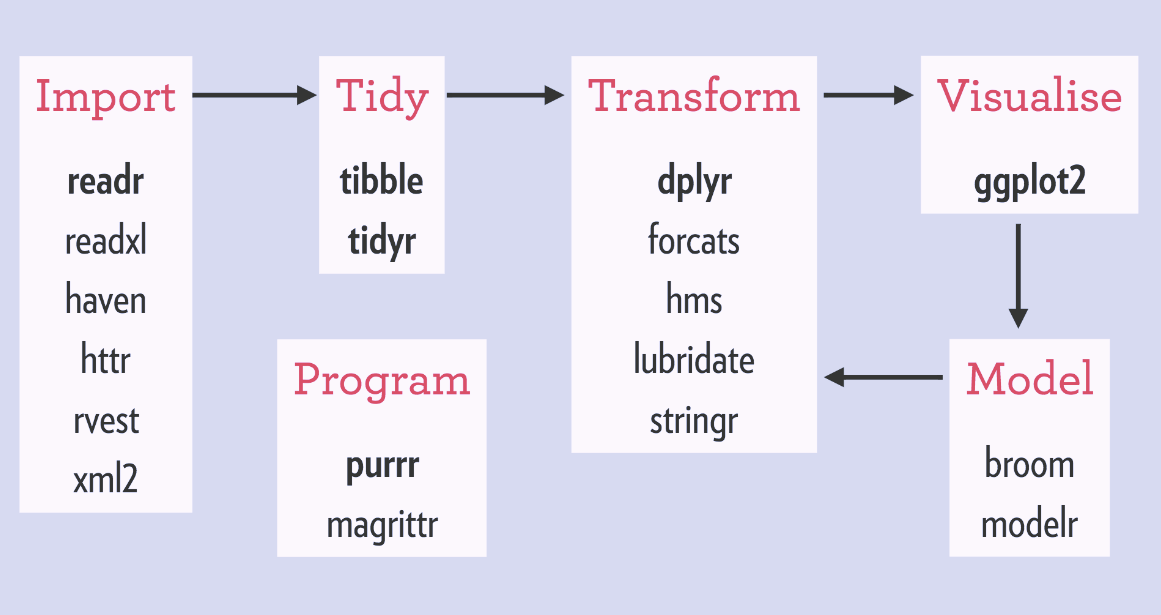
Ecosistema de paquetes que componen tidyverse y su vinculación con cada proceso de un proyecto de Ciencia de Datos
2.2 El dataset gapminder
En 2007 Hans Rosling (1948-2017), un médico sueco, dio una de las charlas Ted más famosas2. Su presentación mostraba la evolución de tres variables en el tiempo: PIB per cápita, expectativa de vida al nacer y población. Uno de los principales mensajes de su charla es que, aunque no lo notemos, el mundo ha mejorado considerablemente - y continúa haciéndolo. Vamos a trabajar con estos optimistas datos en este capítulo.
Podemos acceder a los datos con los que trabajó Rosling con el siguiente código:
gapminder_df <- read.table(file = "https://raw.githubusercontent.com/martintinch0/CienciaDeDatosParaCuriosos/master/data/gapminder.csv",
sep=';',
header = TRUE,
stringsAsFactors = FALSE)Este data frame cuenta con 6 columnas, cuyo nombre podemos obtenerlo de la siguiente manera:
colnames(gapminder_df)## [1] "country" "continent" "year" "lifeExp" "pop" "gdpPercap"E identifican los siguientes datos:
- country: Nombre de país
- continent: Nombre del continente
- year: año de la observación
- lifeExp: expectativa de vida al nacer (en años)
- pop: cantidad de habitantes
- gdpPercap: Producto Interno Bruto (PIB) por habitante
2.3 Transformaciones de los datos
Recordemos que precisamos de las funciones de tidyverse para transformar nuestros datos. Para esto, solo tenemos que aplicar el comando \(`require()`\) o \(`library()`\), los dos cumplen con nuestro objetivo:
require(tidyverse) # Pueden usar library(tidyverse), el resultado debería ser el mismo.2.3.1 Selección de columnas: select()
El comando select() nos permite elegir columnas de nuestros data frames. Solo debemos pasarle los nombres de las variables que deseamos retener. Conservar solo algunas de las variables de un Data Frame es una operación que se realiza muy frecuentemente, así que practiquemos con dos ejemplos. Es importante remarcar el papel que cumple el pipe (%>%): todo lo que está antes es pasado a lo que le sigue para ser procesado.
gapminder_df %>% select(country) # Seleccionamos solo la variable de paísRevisen el panel de “Enviroment” ¿Ven algún objeto nuevo creado? ¿gapminder_df perdió alguna columna (pueden usar str() o colnames() para probarlo?) No, y esto es muy simple de explicar: no asignamos nada a un nuevo objeto. Simplemente le dijimos a R que queríamos solo esa columna, y fue lo que nos devolvió. Para asignar, nunca se olviden de usar <- luego del nombre del objeto.
Vayamos un poco más allá ¿En qué formato devolvió la columna que queríamos seleccionar? Para esto, ahora sí vamos a asignar lo que sea que devuelva select() a una nueva variable. Luego vamos a usar la función class() para ver qué es lo que devolvió.
gapminderCol <- gapminder_df %>% select(country)
class(gapminderCol)## [1] "data.frame"Un data.frame… pero si es solo una columna? no debería ser un vector? La función select() siempre devuelve un data.frame, aun si se selecciona una sola columna. Esto podría no parecer un problema para sus análisis, pero muchas funciones que usarán más adelante necesitarán que estemos pasando un vector, no un vector dentro de una lista (recuerden del capítulo 1 que un data.frame era un conjunto de n listas que tenían un vector de igual tamaño en cada una de ellas)
Si queremos seleccionar una columna, pero que quede guardado en un vector tenemos muchas opciones. Una ya la conocen: es simplemente usar el operador $, que devuelve un vector. Otra alternativa es usar la función unlist() luego de select() o pull(). Verifiquen ustedes mismos usando class() y las herramientas que ya conocen para ver si efectivamente son iguales
gapminderVec1 <- gapminder_df %>% select(country) %>% unlist()
gapminderVec2 <- gapminder_df %>% pull(country)
gapminderVec3 <- gapminder_df$countryPero no siempre queremos seleccionar una sola columna, sea que nos devuelva un data.frame o un vector ¿Cómo podemos seleccionar dos columnas? Muy simple: escribimos otra columna separado por una coma.
gapminder_subset <- gapminder_df %>%
select(country, continent)¿Cuántos países únicos hay en el dataset? ¿Cuantos continentes? En este contexto nos va a sere muy útil la función unique(). Esta función toma un objeto como argumento y devuleve un nuevo objeto que contiene los casos únicos. Usémosla para saber la cantidad de países y continentes:
paises <- unique(gapminder_subset$country)
length(paises) # Length() nos devuelve la cantidad de elementos que tiene un vector## [1] 142length(unique(gapminder_subset$continent)) # Podemos combinar las funciones## [1] 5Existen 142 países y 5 continentes en nuestro dataset.
2.3.2 Selección de casos: filter()
Cuando queramos analizar nuestros datos según ciertas características que tengan nuestras observaciones, es preciso poder seleccionar los casos según los valores que toman en una o más variables. tidyverse (en rigor, uno de sus paquetes: dplyr) nos ofrece el método filter() para realizar esta clase de transformaciones. Por ejemplo: ¿Cuáles son las observaciones que corresponden al año 2002?
gapminder_df %>% filter(year == 2002) # Año 2002Como podemos ver, este comando nos devuelve un tibble (o data frame) con las observaciones correspondientes al año 2002. Recordamos: Las funciones que aplicamos a un objeto (en este caso gapminder) tienen que estar mediadas por lo que se conoce como pipe (%>%). Detengámonos para analizar qué fue lo que hicimos en mayor detalle.
La función filter toma operadores lógicos como argumentos. Estos operadores devuelven TRUE o FALSE dependiendo si una comparación se verifica o no. En nuestro caso particular, el operador lógico utilizado fue ==, que singifica exactamente igual a. Puede parecer raro que usemos doble igual en lugar de un solo igual, pero recuerden que en R el = se encuentra reservado para la asignación, al igual que <-.
Utilizando el mismo operador lógico podemos hacer todavía más cosas. Por ejemplo, podemos filtrar con respecto a otras variables o combinar condiciones de filtrado mediante el mismo método
# Combinando filtros
gapminder_df %>% filter(country == "Argentina", year == 2007)Como podemos ver en el último de los ejemplos, se pueden combinar más de un operador lógico separado por comas. Cada condición se concatena a la anterior como un AND lógico ¿Qué signfica esto? Que la función filter() toma cada uno de los operadores lógicos y busca que todos se cumplan de manera SIMULTÁNEA. En nuestro último caso, filtramos los datos que correspondían tanto a Argentina como al año 2007, lo que devolvió un objeto de una sola fila: los valores correspondientes para Argentina en el año 2007.
La Figura 1 muestra todas las combinaciones booleanas (o lógicas) posibles dados dos conjuntos. La función filter() aplica el operador lógico & a cada una de las condiciones que imponemos. No se preocupen: podemos generar el resto de las condiciones booleanas usando operadores booleanos tales como |, que representa OR, o ! que representa NOT, es decir que lo podemos usar para negar una condición. Si todo esto suena complejo es totalmente razonable. De cualquiera manera, algunos ejemplos (y mucha práctica) va a hacer que todo sea muy intuitivo. Veamos algunos ejemplos
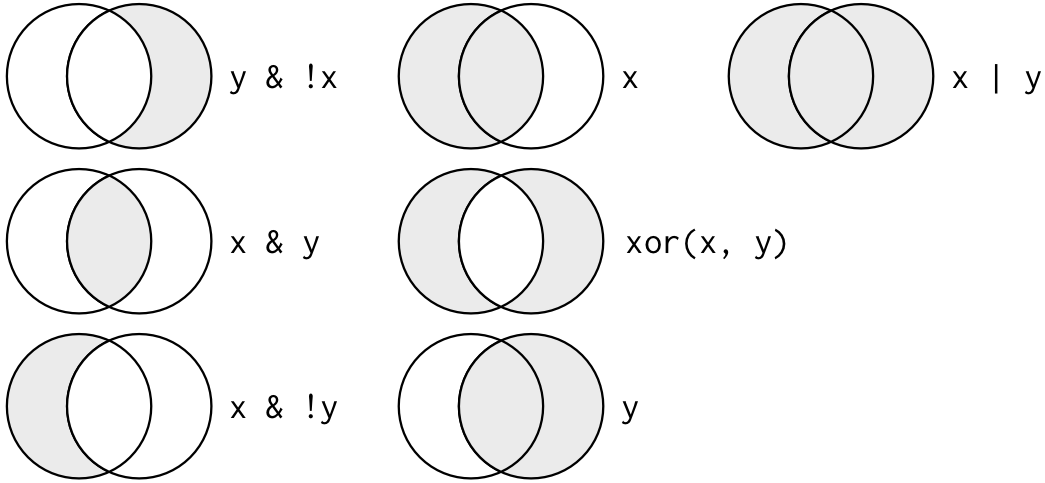
Representación gráfica de todas las operaciones lógicas posibles. Fuente: R for Data Science
# Datos de argentina pero que NO incluyan al año 2007
gapminder_df %>% filter(country == "Argentina", !year == 2007) # ! representa la negación## country continent year lifeExp pop gdpPercap
## 1 Argentina Americas 1952 62.485 17876956 5911.315
## 2 Argentina Americas 1957 64.399 19610538 6856.856
## 3 Argentina Americas 1962 65.142 21283783 7133.166
## 4 Argentina Americas 1967 65.634 22934225 8052.953
## 5 Argentina Americas 1972 67.065 24779799 9443.039
## 6 Argentina Americas 1977 68.481 26983828 10079.027
## 7 Argentina Americas 1982 69.942 29341374 8997.897
## 8 Argentina Americas 1987 70.774 31620918 9139.671
## 9 Argentina Americas 1992 71.868 33958947 9308.419
## 10 Argentina Americas 1997 73.275 36203463 10967.282
## 11 Argentina Americas 2002 74.340 38331121 8797.641# Uno de los inconvenientes cuando queremos filtrar por más de un criterio de una misma variable es que tenemos que hacer lo siguiente
gapminder_df %>% filter(country == "Argentina", year == 2002 | year == 2007)## country continent year lifeExp pop gdpPercap
## 1 Argentina Americas 2002 74.34 38331121 8797.641
## 2 Argentina Americas 2007 75.32 40301927 12779.380Bastante más simple que en la explicación anterior ¿No? Vamos a complejizarlo levemente introduciendo al operador %in%. Este operador %in% es muy útil para matchear múltiples condiciones de manera simple. Lo que hace es devolver TRUE en todos los elementos de un vector que cumplen con alguno de los valores contenidos en el vector de la derecha. En el siguiente caso devuelve las observaciones que corresponden a los años 2002 o 2007.
# Podemos solucionarlo mediante el siguiente método
gapminder_df %>% filter(year %in% c(2002,2007))
# Es una forma de concatenar condiciones de tipo | (OR)
# gapminder_df %>% filter(year == 2002 | year == 2007) # Da el mismo resultadoHagamos algo útil con lo que aprendimos hasta ahora, aun haciendo referencia a una librería que todavía no usamos como ggplot. Este paquete es uno de los más utilizados para realizar gráficos.
gapminder_argentina <- gapminder_df %>% filter(country == "Argentina")# Expectativa de vida al nacer en Argentina 1952-2007
ggplot(gapminder_argentina) + geom_line(aes(x = year, y = lifeExp))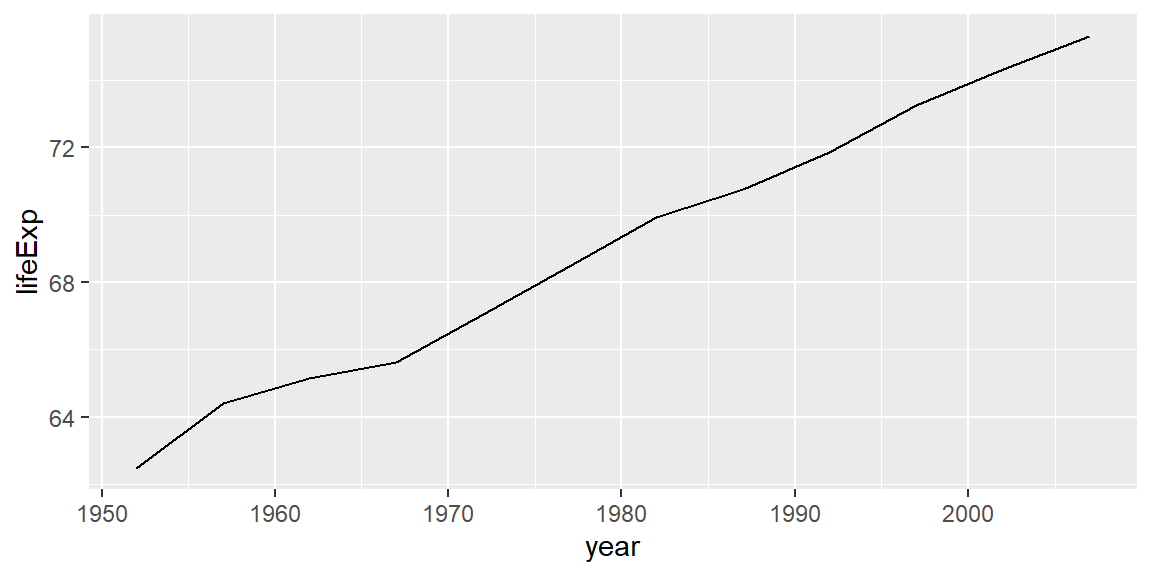
# PIB per cápita en Argentina 1952-2007
ggplot(gapminder_argentina) + geom_line(aes(x = year, y = gdpPercap))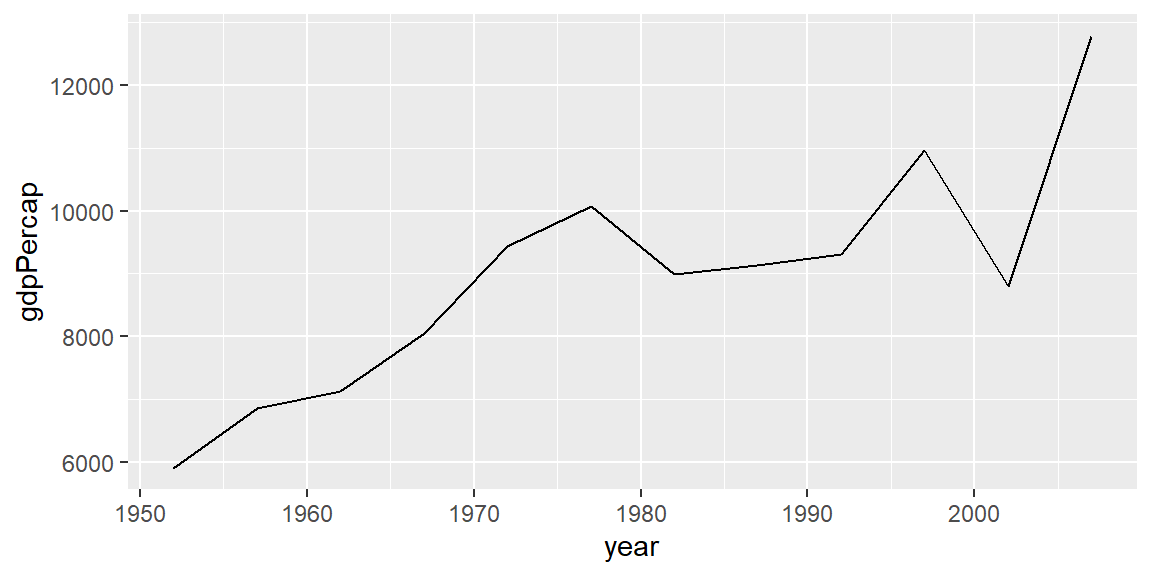
Creo que podemos sacar nuestras primeras conclusiones para Argentina: la expectativa de vida parece haber crecido bastante estable durante todo el período bajo análisis, pero la evolución del PIB per cápita bien puede parecerse a lo que registra un sismógrafo !
2.3.3 Ordenando: la función arrange()
La función arrange nos permite ordenar un dataset en orden ascendente o descendente en base a los valores de las variables. Está parte del proceso suele ser relevante para algunas transformaciones de datos y para la inspección visual de valores extremos.
# En sentido ascendente (del valor más bajo al más alto)
gapminder_df %>% arrange(lifeExp)En sentido descendente (del valor más alto al más
gapminder_df %>% arrange(desc(lifeExp))Veamos una de las principales funcionalidades de tidyverse al combinar algunos de los comandos que aprendimos hasta ahora. Vamos a filtrar el dataset para el continente de América y el año 2007 y luego (recordar: está a la derecha del último pipe) ordenar las observaciones según la expectativa de vida al nacer, de mayor a menor:
gapminder_df %>%
filter(continent == "Americas", year == 2007) %>%
arrange(desc(lifeExp))## country continent year lifeExp pop gdpPercap
## 1 Canada Americas 2007 80.653 33390141 36319.235
## 2 Costa Rica Americas 2007 78.782 4133884 9645.061
## 3 Puerto Rico Americas 2007 78.746 3942491 19328.709
## 4 Chile Americas 2007 78.553 16284741 13171.639
## 5 Cuba Americas 2007 78.273 11416987 8948.103
## 6 United States Americas 2007 78.242 301139947 42951.653
## 7 Uruguay Americas 2007 76.384 3447496 10611.463
## 8 Mexico Americas 2007 76.195 108700891 11977.575
## 9 Panama Americas 2007 75.537 3242173 9809.186
## 10 Argentina Americas 2007 75.320 40301927 12779.380
## 11 Ecuador Americas 2007 74.994 13755680 6873.262
## 12 Venezuela Americas 2007 73.747 26084662 11415.806
## 13 Nicaragua Americas 2007 72.899 5675356 2749.321
## 14 Colombia Americas 2007 72.889 44227550 7006.580
## 15 Jamaica Americas 2007 72.567 2780132 7320.880
## 16 Brazil Americas 2007 72.390 190010647 9065.801
## 17 Dominican Republic Americas 2007 72.235 9319622 6025.375
## 18 El Salvador Americas 2007 71.878 6939688 5728.354
## 19 Paraguay Americas 2007 71.752 6667147 4172.838
## 20 Peru Americas 2007 71.421 28674757 7408.906
## 21 Guatemala Americas 2007 70.259 12572928 5186.050
## 22 Honduras Americas 2007 70.198 7483763 3548.331
## 23 Trinidad and Tobago Americas 2007 69.819 1056608 18008.509
## 24 Bolivia Americas 2007 65.554 9119152 3822.137
## 25 Haiti Americas 2007 60.916 8502814 1201.6372.3.4 Creando y modificando variables: mutate()
Crear nuevas variables en base a los valores de otras variables que ya existen suele ser una parte necesaria para enriquecer el análisis. En el marco de tidyverse la forma de lograrlo es a través del verb (verb es tan solo otra forma de llamar a las funciones en el contexto de tidyverse) mutate(). Imagemos, por ejemplo, que queremos la población medida en millones de personas para hacer más fácil su lectura:
new_gapminder <- gapminder_df %>% mutate(pop = pop / 1000000)
head(new_gapminder, n = 3) # Head nos permite ver solo una determinada cantidad de filas## country continent year lifeExp pop gdpPercap
## 1 Afghanistan Asia 1952 28.801 8.425333 779.4453
## 2 Afghanistan Asia 1957 30.332 9.240934 820.8530
## 3 Afghanistan Asia 1962 31.997 10.267083 853.1007La sintaxis es simple: del lado izquierdo de la igualdad escribimos el nombre de la variable y del lado derecho definimos su valor (en este caso, el valor de pop dividido por un millón). Al utilizar el nombre de una variable que anteriormente ya existía no creamos una nueva, sino que reemplazamos a pop. Si escribimos el nombre de una variable que no existe R agrega esa variable al dataset:
# Calcuando el PIB
gapminder_df %>%
mutate(gdp = gdpPercap * pop)2.3.5 Resumiendo y transformando datos en base a grupos
Muchas veces es necesario resumir diversas variables de un dataset en base a grupos. En nuestro ejemplo, preguntas que precisarían de agrupar datos serían algunas como las siguientes:
- ¿Cuál es la expectativa de vida al nacer promedio por continente para cada uno de los años?
- ¿Cuál es el país más pobre y más rico, medido por PIB per cápita, de cada continente para cada uno de los años?
- ¿Cuál es la diferencia en la expectativa de vida al nacer para cada país con respecto a la media del continente para cada año?
Para responder estas preguntas, tidyverse brinda dos funciones: group_by(), para agrupar observaciones según categorías de una variable, y summarise(), para aplicar alguna transformación sobre cada conjunto de datos. Veamos cómo podríamos resolver estas tres preguntas con estas funciones y otras que ya hemos aprendido.
# Primera pregunta
gapminder_df %>%
group_by(year, continent) %>%
summarise(mean_lifeExp = mean(lifeExp))## # A tibble: 60 x 3
## # Groups: year [12]
## year continent mean_lifeExp
## <int> <chr> <dbl>
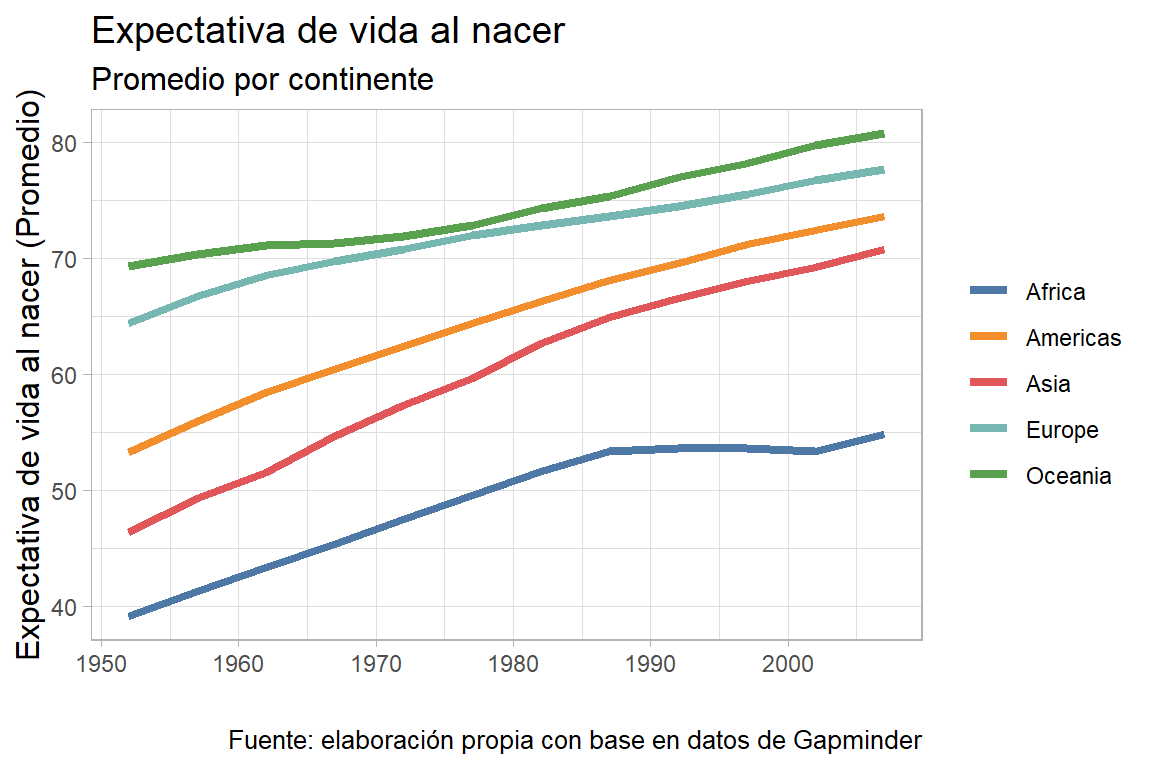
## 1 1952 Africa 39.1
## 2 1952 Americas 53.3
## 3 1952 Asia 46.3
## 4 1952 Europe 64.4
## 5 1952 Oceania 69.3
## 6 1957 Africa 41.3
## 7 1957 Americas 56.0
## 8 1957 Asia 49.3
## 9 1957 Europe 66.7
## 10 1957 Oceania 70.3
## # ... with 50 more rows# Segunda pregunta
gapminder_df %>% group_by(year, continent) %>%
summarise(poor_country = min(gdpPercap),
rich_country = max(gdpPercap),
poor_country_nom = country[gdpPercap == poor_country],
rich_country_nom = country[gdpPercap == rich_country])## # A tibble: 60 x 6
## # Groups: year [12]
## year continent poor_country rich_country poor_country_nom rich_country_nom
## <int> <chr> <dbl> <dbl> <chr> <chr>
## 1 1952 Africa 299. 4725. Lesotho South Africa
## 2 1952 Americas 1398. 13990. Dominican Republic United States
## 3 1952 Asia 331 108382. Myanmar Kuwait
## 4 1952 Europe 974. 14734. Bosnia and Herzegovina Switzerland
## 5 1952 Oceania 10040. 10557. Australia New Zealand
## 6 1957 Africa 336. 5487. Lesotho South Africa
## 7 1957 Americas 1544. 14847. Dominican Republic United States
## 8 1957 Asia 350 113523. Myanmar Kuwait
## 9 1957 Europe 1354. 17909. Bosnia and Herzegovina Switzerland
## 10 1957 Oceania 10950. 12247. Australia New Zealand
## # ... with 50 more rows# Tercera pregunta
gapminder_df %>% group_by(year, continent) %>%
mutate(dif_lifeExp = lifeExp - mean(lifeExp))## # A tibble: 1,704 x 7
## # Groups: year, continent [60]
## country continent year lifeExp pop gdpPercap dif_lifeExp
## <chr> <chr> <int> <dbl> <int> <dbl> <dbl>
## 1 Afghanistan Asia 1952 28.8 8425333 779. -17.5
## 2 Afghanistan Asia 1957 30.3 9240934 821. -19.0
## 3 Afghanistan Asia 1962 32.0 10267083 853. -19.6
## 4 Afghanistan Asia 1967 34.0 11537966 836. -20.6
## 5 Afghanistan Asia 1972 36.1 13079460 740. -21.2
## 6 Afghanistan Asia 1977 38.4 14880372 786. -21.2
## 7 Afghanistan Asia 1982 39.9 12881816 978. -22.8
## 8 Afghanistan Asia 1987 40.8 13867957 852. -24.0
## 9 Afghanistan Asia 1992 41.7 16317921 649. -24.9
## 10 Afghanistan Asia 1997 41.8 22227415 635. -26.3
## # ... with 1,694 more rowsLas tres respuestas comienzan de la misma manera, es decir con la función group_by(). Entre los paréntesis hay que colocar los nombres de las variables por las cuáles se quiere agrupar, en nuestro caso las variables year y continent. De esta manera todo lo que siga después de la próxima pipe se aplicará sobre cada uno de los grupos generados por las combinaciones año y continente.
En el caso de la primera respuesta, usamos la función summarise(), que devuelve un Data Frame (en rigor, tibble) con resúmenes para cada uno de los grupos. Debido a que existen 12 “fotos” de las variables (una cada cinco años) y se definen 5 continentes (África, América, Asia, Europa y Oceanía), el data frame que devuelve tiene 60 filas (12 países multiplicado por 5 continentes da 60 grupos).
Dentro de los paréntesis de summarise() podemos crear tantas variables como queramos. En el primer caso, creamos una variable que se llama mean_lifeExp que recibe el resultado de aplicar mean() a la variable lifeExp. Esta función es aplicada a cada uno de los grupos que definimos anteriormente.
En el segundo caso hacemos algo similar a lo anterior, pero definimos cuatro variables. poor_country busca el valor mínimo del PIB per cápita para cada grupo a través de min(), rich_country hace lo opuesto a través de la función max(), poor_country_nom busca el nombre que corresponde al PIB per cápita más bajo y rich_country_nom busca el nombre que corresponde al PIB per cápita más alto. Estas últimas dos variables se generan a través de filtrar el vector country y buscar el valor que corresponde al PIB per cápita más bajo o alto, según corresponda, mediante el código country[gdpPercap == poor_country].
Finalmente, la tercera parte del código combina las funciones group_by() y mutate(), que ya vimos anteriormente. La novedad es que ahora podemos utilizarla para crear nuevas variables en el dataset basadas en agregaciones de otras variables. En este ejemplo, creamos la variable dif_lifeExp, que toma la diferencia entre la expectativa de vida al nacer para cada observación y la media de cada grupo (en nuestro caso, año y continente).
El siguiente gráfico muestra una visualización basada en este dataset que acabamos de generar, usando ggplot
2.4 Transformando la presentación de los datos: pivot_wider y pivot_longer
Los datos pueden venir presentados en dos formatos: largo o ancho. Los datasets con formato largo tienen pocas columnas y muchas filas, mientras los de formato ancho poseen muchas columnas y pocas filas. Sin embargo, en ambas representaciones los datos son exactamente los mismos. 
En diversas situaciones es preferible tener una de las dos representaciones. Por ejemplo, para graficar con la librería ggplot2 muchas veces es conveniente contar con representaciones en formato largo de los datos. Tidyverse ofrece dos métodos que sirven para este propósito: pivot_wider() y pivot_longer() (estas funciones reemplazaron a gather() y a spread(), respectivamente).
- pivot_wider(): toma un conjunto de variables (vectores/columnas) y las colapsa en una sola columna con valores que resumen los datos de ese conjunto de variables. Hace que el data frame sea más largo
- pivot_longer(): toma dos variables y las descompone entre múltiples variables (hace que el data frame sea más ancho)
Como pueden ver, no existe una medida absoluta de largo o ancho. Simplemente un dataset puede tener una representación más ancha o más larga.
Vamos a exportar una tabla desde R para que lo puedan usar en otro software. En general, cuando los datos ya han sido procesados, las salidas se muestran en tablas más bien anchas. Con los datos que contamos vamos a crear un archivo .csv que tenga en las filas a los paises y en las columnas a los años (como valor, a la pboblación en millones).
# Nos quedamos con tres coolumnas: country, year y pop.
# Además, con mutate hacemos que la población esté representada por millones
gapminder_sub <- gapminder_df %>%
select(country,year,pop) %>%
mutate(pop = round(pop / 1000000,1))
head(gapminder_sub,n = 5) # Muestra las primeras cinco filas ## country year pop
## 1 Afghanistan 1952 8.4
## 2 Afghanistan 1957 9.2
## 3 Afghanistan 1962 10.3
## 4 Afghanistan 1967 11.5
## 5 Afghanistan 1972 13.1Ya estamos en condiciones de usar pivot_wider
masAncho <- gapminder_sub %>%
pivot_wider(names_from = year,values_from = pop)
head(masAncho,6)## # A tibble: 6 x 13
## country `1952` `1957` `1962` `1967` `1972` `1977` `1982` `1987` `1992` `1997` `2002` `2007`
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Afghanistan 8.4 9.2 10.3 11.5 13.1 14.9 12.9 13.9 16.3 22.2 25.3 31.9
## 2 Albania 1.3 1.5 1.7 2 2.3 2.5 2.8 3.1 3.3 3.4 3.5 3.6
## 3 Algeria 9.3 10.3 11 12.8 14.8 17.2 20 23.3 26.3 29.1 31.3 33.3
## 4 Angola 4.2 4.6 4.8 5.2 5.9 6.2 7 7.9 8.7 9.9 10.9 12.4
## 5 Argentina 17.9 19.6 21.3 22.9 24.8 27 29.3 31.6 34 36.2 38.3 40.3
## 6 Australia 8.7 9.7 10.8 11.9 13.2 14.1 15.2 16.3 17.5 18.6 19.5 20.4La función pivot_wider() necesita solo dos parámetros. En primer lugar, debemos decirle de cuál columna hay que tomar los nuevos nombres de columnas en el parámetro names_from. Sn segundo lugar, solo tenemos que decirle de qué columna tiene que tomar los valores, en values_from.
¿Qué tenemos que hacer para guardar esta salida? usamos la función write.table(). Vamos a usar cuatro parámetros. En x solo tenemos que pasarle el objeto a escribir, en file un nombre de archivo (vean que es .csv), en sep usamos un caracter que queremos que use para separar a las columnas (recomiendo usar ;). Finalmente, en el parámetro row.names usamos FALSE con el objetivo que no incluya el número de filas en la salida
write.table(x = masAncho,
file = 'PaisPob.csv',
sep = ';',
row.names=FALSE)Ahora que exportamos nuestro primer data frame, podemos ponernos del otro lado del mostrador. Imaginemos que nos pasan un .csv con estos datos, pero nosotros queremos procesarlos todavía un poco más. En ese caso, muchas de las herramientas que conocemos nos piden que los datos estén un poco más “largos”. Acá entra en juego pivot_longer(), pero antes tenemos que leer los datos que acabamos de exportar
# El parámetro header sirve para avisar que los datos tienen una primera
# fila que es el nombre de las columnas y check.names es otro parámetro
# que es neceario cuando los nombres de las columnas son números
masAncho <- read.table(file = 'PaisPob.csv',
sep = ';',
header=TRUE,
check.names = FALSE,
stringsAsFactors = FALSE)
head(masAncho)## country 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 2002 2007
## 1 Afghanistan 8.4 9.2 10.3 11.5 13.1 14.9 12.9 13.9 16.3 22.2 25.3 31.9
## 2 Albania 1.3 1.5 1.7 2.0 2.3 2.5 2.8 3.1 3.3 3.4 3.5 3.6
## 3 Algeria 9.3 10.3 11.0 12.8 14.8 17.2 20.0 23.3 26.3 29.1 31.3 33.3
## 4 Angola 4.2 4.6 4.8 5.2 5.9 6.2 7.0 7.9 8.7 9.9 10.9 12.4
## 5 Argentina 17.9 19.6 21.3 22.9 24.8 27.0 29.3 31.6 34.0 36.2 38.3 40.3
## 6 Australia 8.7 9.7 10.8 11.9 13.2 14.1 15.2 16.3 17.5 18.6 19.5 20.4Ahora ya podemos recrear nuestra versión anterior, más larga
reconstruccion <- masAncho %>%
pivot_longer(cols = 2:13,
names_to = 'year',
values_to = 'pop') %>%
arrange(country,year)
head(reconstruccion, n = 5) # Muestra las primeras cinco filas## # A tibble: 5 x 3
## country year pop
## <chr> <chr> <dbl>
## 1 Afghanistan 1952 8.4
## 2 Afghanistan 1957 9.2
## 3 Afghanistan 1962 10.3
## 4 Afghanistan 1967 11.5
## 5 Afghanistan 1972 13.1La función pivot_longer() requiere un poco más de atención. A diferencia de pivot_wider, tenemos que 1) definir el nombre de la nueva variable que tendrá como categoría los nombres de otras columnas (parámetro names_to), 2) el nombre de la variable que tendrá los valores que estaban en variables que ahora se resumirán en la nueva columna (parámetro values_to y 3) las columnas que quieren colapsarse dentro de las dos nuevas variables (van en cols). Los primeros dos parámetros no tienen gran complejidad, pero la selección de las variables puede realizarse de varias maneras. En el caso anterior, lo hicimos indicando la posición de las columnas en el Data Frame. En el siguiente código exhibimos tres métodos que logran lo mismo.
reconstruccion2 <- masAncho %>%
pivot_longer(cols = -country,
names_to = 'year',
values_to = 'pop') %>%
arrange(country,year) # Todas las columnas menos country
reconstruccion3 <- masAncho %>%
pivot_longer(cols = -1,
names_to = 'year',
values_to = 'pop') %>%
arrange(country,year) # Todas las columnas menos la primera
reconstruccion4 <- masAncho %>%
pivot_longer(cols = c('1952','1957','1962','1967','1972','1977', '1982',
'1987','1992','1997','2002','2007'),
names_to = 'year',
values_to = 'pop') %>%
arrange(country,year) # Todas las columnas con esos nombres
# Esta línea nos devuelve TRUE si los 4 objetos sin iguales entre sí o FALSE si hay
# al menos uno que no lo es
all(sapply(list(reconstruccion2, reconstruccion3, reconstruccion4), FUN = identical, reconstruccion))## [1] TRUEAlgunas leves sutilezas ¿Es igual nuestro dataset reconstruido al original? Veamoslo
identical(gapminder_sub,reconstruccion)## [1] FALSER nos dice que no, que no son iguales ¿Cómo puede ser? tienen exactamente los mismos valores, mismas filas y mismas columnas… Indagemos un poco más sobre el tipo de datos de las columnas
class(gapminder_sub$year)## [1] "integer"class(reconstruccion$year)## [1] "character"El problema es que la reconstrucción tiene a la variable de años guardada como tipo character, mientras que en el original era númerica ¿Qué fue lo que pasó?. Lo que sucede es normal: siempre que usen pivot_longer() y los nombres de las variables sean números, va a asumir que es texto, ya que eso es lo que se espera de los nombres de las columnas. No se preocupen, podemos usar as.integer() para realizar la conversión
reconstruccion <- reconstruccion %>%
mutate(year=as.integer(year))
# Además hay otra razón (menor) por la cual no son iguales, y es que
# gapminder_sub es solo un data frame y reconstruccion es un
# tibble, un data.frame modificado para tidyverse.
gapminder_sub <- gapminder_sub %>%
as_tibble(gapminder_sub)
# Son iguales !
identical(reconstruccion, gapminder_sub)## [1] TRUE2.5 Uniendo datos de distintas fuentes: left_join
Otra de las transformaciones más comunes que hacemos sobre los datos es agregar nueva información en base a una (o más) columnas en las que coinciden. Por ejemplo, sabiendo que un dato corresponde a Argentina, podríamos agregar variables específicas para ese país sobre otra dimensión, como podría ser la tasa de mortalidad infantil. Haremos exactamente esto, con la ayuda de otro dataset:
mortalidadInfantil <- read.table(file="https://raw.githubusercontent.com/martintinch0/CienciaDeDatosParaCuriosos/master/data/MortalidadInfantilLong.csv",
sep = ",",
header = TRUE,
stringsAsFactors = FALSE)
gapminder_argentina <- gapminder_df %>%
filter(country == "Argentina")
gapminder_argentina1952 <- gapminder_argentina %>%
filter(year==1952)
mortalidadInfantilArgentina1952 <- mortalidadInfantil %>%
filter(Year==1952 & country=="Argentina")
gapminder_argentina1952 <- left_join(gapminder_argentina1952,
mortalidadInfantilArgentina1952,
by="country")
gapminder_argentina1952## country continent year lifeExp pop gdpPercap Year Mortalidad
## 1 Argentina Americas 1952 62.485 17876956 5911.315 1952 88.8La función clave es left_join(). Lo que hace es tomar dos data frames y los une por una o más variables que tienen en común, que en este caso es la variable country. Por cada fila del primer dataset que le pasamos, busca el valor que corresponde en la variable que tiene een comun en el segundo data frame que le pasamos, y agrega como columna la información que existe en el segundo data frame. De esta manera, agregamos la mortalidad infantil para Argentina en 1952, que es de 88.8 cada 1000 niños de entre 0 y 5 años.
En rigor, con este dataset podemos agregar más que la información de la mortalidad infantil para Argentina en 1952, sino que lo podemos hacer para todas las observaciones de la siguiente manera:
gapminder_full <- left_join(gapminder_df,
mortalidadInfantil,
by=c("country","year"="Year"))
gapminder_fullSi prestaron atención, hicimos dos cosas distintas esta vez: elegimos dos variables para hacer el join: country e year ¿Por qué? Porque de esa manera podemos completar la información sobre la mortalidad infantil para cada combinación de país y año. Pero por otro lado, al nombre de la segunda columna le aclaramos que “year” en el primer data frame en realidad se llama “Year” en el segundo. Prueben que pasa si solo dejan “year” en esa segunda parte.
2.6 La mise en place: preparando el dataset de inmuebles
A esta altura vimos una gran cantidad de comandos para transformar datos con un interesante dataset como es gapminder. Sin embargo, probablemente hayan sido demasiados para procesarlos de una sola vez. Lo que vamos a hacer ahora es aplicarlo en nuestro objetivo inicial: transformar los datasetas tal como se descargan desde Properati hasta el dataset que trabajamos en el capítulo anterior. Vamos a usar varios de los comandos que ya vimos.
Vamos a trabajar con una versión levemente modificada de los datos descargados de Properati porque vamos a procesar una muestra estratificada por barrio y año. Esto quiere decir que tomamos aleatoriamente 30 observaciones (siempre que las hayan) de cada combinación barrio y año. Esto lo hacemos para reducir la cantidad de filas, que eran más de 400mil en el dataset original y logramos reducirlas sensiblemente, sin perder generalidad en nuestra explicación.
barriosOriginal <- read.table(file="https://github.com/datalab-UTDT/datasets/raw/master/barriosSample.csv",
sep = ";",
header = TRUE,
stringsAsFactors = FALSE)
dim(barriosOriginal) # Número de filas y de columnas## [1] 4780 31colnames(barriosOriginal) # Nombres de las columnas## [1] "BARRIOS" "COMUNA" "NUM_DE_BAR"
## [4] "created_on" "operation" "property_type"
## [7] "place_name" "place_with_parent_names" "geonames_id"
## [10] "lat.lon" "price" "currency"
## [13] "price_aprox_local_currency" "price_aprox_usd" "surface_in_m2"
## [16] "price_usd_per_m2" "floor" "rooms"
## [19] "expenses" "properati_url" "image_thumbnail"
## [22] "description" "title" "extra"
## [25] "surface_total_in_m2" "surface_covered_in_m2" "price_per_m2"
## [28] "id" "country_name" "state_name"
## [31] "year"Recuerden que el dataset con el que trabajamos en el capítulo anterior tenia tan solo 6 variables, que contenían 1) los nombres de los barrios y 2) el valor de los precios en dólares para cada año (2013-2017).
Para empezar, vamos a quedarnos solo con las variables que son relevantes: barrios, precios en dolares y años
barriosOriginal <- barriosOriginal %>%
select(BARRIOS, price_usd_per_m2, year)
str(barriosOriginal)## 'data.frame': 4780 obs. of 3 variables:
## $ BARRIOS : chr "AGRONOMIA" "AGRONOMIA" "AGRONOMIA" "AGRONOMIA" ...
## $ price_usd_per_m2: num 1304 1642 1333 2800 1467 ...
## $ year : int 2013 2013 2013 2013 2013 2013 2013 2013 2013 2013 ...Ahora, necesitamos calcular el precio PROMEDIO por barrio y año. Por suerte, ya conocemos las funciones group_by() y summarise(). Es todo lo que necesitamos para esta transformación:
barriosOriginal <- barriosOriginal %>%
group_by(BARRIOS, year) %>%
summarise(precioPromedio = mean(price_usd_per_m2))Ya estamos bastante cerca de nuestro objetivo: contamos con un Data Frame donde se registra el precio promedio en USD del metro cuadrado de los inmuebles por barrio para el período 2013-2017. La diferencia con el dataset de la clase pasada es que este se presenta en formato largo y el anterior en formato ancho. La función pivot_wider, que ya vimos anteriormente, va a hacer lo que necesitamos
barriosOriginal <- barriosOriginal %>%
pivot_wider(names_from = year,
values_from = precioPromedio)
head(barriosOriginal, n = 3)## # A tibble: 3 x 6
## # Groups: BARRIOS [3]
## BARRIOS `2013` `2014` `2015` `2016` `2017`
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 AGRONOMIA 1682. 1254. 1588. 2051. 1781.
## 2 ALMAGRO 2024. 1922. 1989. 1891. 2270.
## 3 BALVANERA 1857. 1791. 1908. 1642. 1668.Ya casi estamos ! Lo único que nos falta es cambiar el nombre de las columnas. Para eso vamos a utilizar la función paste():
colnames(barriosOriginal)[2:6] <- paste('USDm2_',colnames(barriosOriginal)[2:6], sep="")
head(barriosOriginal, n =3)## # A tibble: 3 x 6
## # Groups: BARRIOS [3]
## BARRIOS USDm2_2013 USDm2_2014 USDm2_2015 USDm2_2016 USDm2_2017
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 AGRONOMIA 1682. 1254. 1588. 2051. 1781.
## 2 ALMAGRO 2024. 1922. 1989. 1891. 2270.
## 3 BALVANERA 1857. 1791. 1908. 1642. 1668.Lo que hace esta función es tomar vectores y concatenarlos entre sí. En este caso le pasamos dos vectores: uno de tipo character con un solo elemento (“USDm2_”) y otro que contiene la posición 2 a 6 del vector character colnames(barriosOriginal), es decir los nombres de las variables 2 a 6 del dataset barriosOriginal. En estos simples pasos ya construimos el dataset con el que trabajamos la clase anterior desde el formato que tenían cuando se descargaron desde el portal de datos.
2.7 Ejercicios
Vuelvan a cargar el data frame de gapminder y respondan las siguientes preguntas:
- ¿Cuál es la observación con mayor expectativa al necer de todo el dataset? ¿A qué país corresponde y en qué año?
- ¿Cuál es la expectativa de vida a nacer promedio por continente en 1952? ¿Y en 2007?
- ¿Cuánto aumento la expectativa de vida al nacer por continente entre 2007 y 1952?
- ¿Cuál fue el país, por continente, que más aumentó su expectativa de vida al nacer en términos absolutos?
- Entre 1952 y 2007 ¿Cuál fue el país que más aumento su PIB per cápita? ¿Y por continente?
- ¿Cuánto aumento el PIB per cápita de Argentina entre 1952 y2007? ¿Y entre 1977 y 2002?
2.8 Extensiones
2.8.1 R Cheatsheets
RStudio elabora y publica de manera periódica distintas “cheatsheets” donde tienen resumidas todas las funciones de algunos paquetes, para qué sirven y ejemplo sencillos. Suelen ser muy útiles como para tener presente cuando trabajan con sus datasets y para que conozcan más funciones que las introducidas en el libro. Además, los paquetes van cambiand con el tiempo y algunas funciones pueden quedar desactualizadas.
En https://rstudio.com/resources/cheatsheets/ van a encontrar todas las disponibles. La relevante para este capítulo es la del paquete dyplr https://github.com/rstudio/cheatsheets/raw/master/data-transformation.pdf
Este video resume en unos pocos minutos gran parte de lo que vamos a ver acá y es recomendable↩