datos <- read.csv("https://raw.githubusercontent.com/Ed-Perez-G/Taller_ML_DGH/refs/heads/main/bd_CM.csv",
header = TRUE, sep = ";", stringsAsFactors = T, na="")EDA
Análisis Exploratorio de Datos
El Análisis Exploratorio de Datos (EDA) es la etapa inicial en el análisis de datos, la cual consiste en explorar y resumir un conjunto nuestros datos para comprender su estructura, detectar patrones y anomalías, y formular hipótesis para estudios más profundos o modelos predictivos.
El EDA se utiliza principalmente para:
- Comprender la estructura del conjunto de datos: Verificar el tamaño, las variables y el tipo de datos que se van a analizar.
- Identificar la calidad de los datos: Detectar datos faltantes, valores atípicos (outliers) y duplicados, que pueden afectar los análisis posteriores.
- Explorar la distribución de las variables: Verificar si los datos siguen una distribución específica, como la normal, y conocer la variabilidad y los rangos de las variables.
- Encontrar relaciones entre variables: Identificar correlaciones o patrones entre variables numéricas y categóricas, lo que puede ayudar en la creación de modelos y análisis de causalidad.
- Formular hipótesis iniciales: Observar patrones y comportamientos para desarrollar hipótesis que se puedan validar en etapas posteriores del análisis.
Este documento, está diseñado para copie y pegue el código en la consola de R y lo ejecute. Puede descargar la versión de este documento y después abrirlo en Rstudio aquí: o aquí
Importar base datos a R
Para importar la base de datos a R utilice la función read.csv de la siguiente manera:
Asegúrese de que la base fue leída correctamente utilizando la función head y View de la siguiente manera:
head(datos) country overall_survival_time_months age_at_diagnosis_years
1 mexico 84.63333 43.19
2 usa 163.70000 48.87
3 canada 164.93333 47.68
4 mexic 41.36667 76.97
5 USA 7.80000 78.77
6 Caada 164.33333 56.45
lymph_nodes_examined_positive cellularity estrogen_receptors_ihc
1 0 High Positve
2 1 High Positve
3 3 Moderate Positve
4 8 High Positve
5 0 Moderate Positve
6 1 Moderate Positve
menopausal_state claudin_subtype vital_status
1 Pre LumA Living
2 Pre LumB Died of Disease
3 Pre LumB Living
4 Post LumB Died of Disease
5 Post LumB Died of Disease
6 Post LumB Living
cancer_type_detailed her2_status
1 Breast Invasive Ductal Carcinoma Negative
2 Breast Invasive Ductal Carcinoma Negative
3 Breast Mixed Ductal and Lobular Carcinoma Negative
4 Breast Mixed Ductal and Lobular Carcinoma Negative
5 Breast Invasive Ductal Carcinoma Negative
6 Breast Invasive Ductal Carcinoma Negative
progesterone_receptors_status tumor_size_mm CT_responde
1 Positive 10 0
2 Positive 15 0
3 Positive 25 0
4 Positive 40 0
5 Positive 31 0
6 Positive 10 0El siguiente código le permitirá visualizar su base completa en una nueva ventana de Rstudio:
View(datos)Visaluzación general de la base de datos
Para visualizar la estructura de la base de datos utilice la función str de la siguiente manera:
str(datos)'data.frame': 1830 obs. of 14 variables:
$ country : Factor w/ 7 levels "Caada","canada",..: 5 6 2 4 7 1 3 5 6 2 ...
$ overall_survival_time_months : num 84.6 163.7 164.9 41.4 7.8 ...
$ age_at_diagnosis_years : num 43.2 48.9 47.7 77 78.8 ...
$ lymph_nodes_examined_positive: int 0 1 3 8 0 1 1 1 0 0 ...
$ cellularity : Factor w/ 3 levels "High","Low","Moderate": 1 1 3 1 3 3 3 3 1 3 ...
$ estrogen_receptors_ihc : Factor w/ 2 levels "Negative","Positve": 2 2 2 2 2 2 2 2 1 2 ...
$ menopausal_state : Factor w/ 2 levels "Post","Pre": 2 2 2 1 1 1 1 1 1 1 ...
$ claudin_subtype : Factor w/ 7 levels "Basal","claudin-low",..: 4 5 5 5 5 5 2 5 3 4 ...
$ vital_status : Factor w/ 3 levels "Died of Disease",..: 3 1 3 1 1 3 2 2 1 1 ...
$ cancer_type_detailed : Factor w/ 7 levels "Breast","Breast Invasive Ductal Carcinoma",..: 2 2 5 5 2 2 5 2 3 2 ...
$ her2_status : Factor w/ 2 levels "Negative","Positive": 1 1 1 1 1 1 1 1 NA 1 ...
$ progesterone_receptors_status: Factor w/ 2 levels "Negative","Positive": 2 2 2 2 2 2 1 1 1 2 ...
$ tumor_size_mm : num 10 15 25 40 31 10 29 16 28 22 ...
$ CT_responde : int 0 0 0 0 0 0 0 0 1 0 ...Una manera más eficiente es utilizar la función vistdat que permite visualmente identificar los tipos de variables y datos faltantes:
visdat::vis_dat(datos)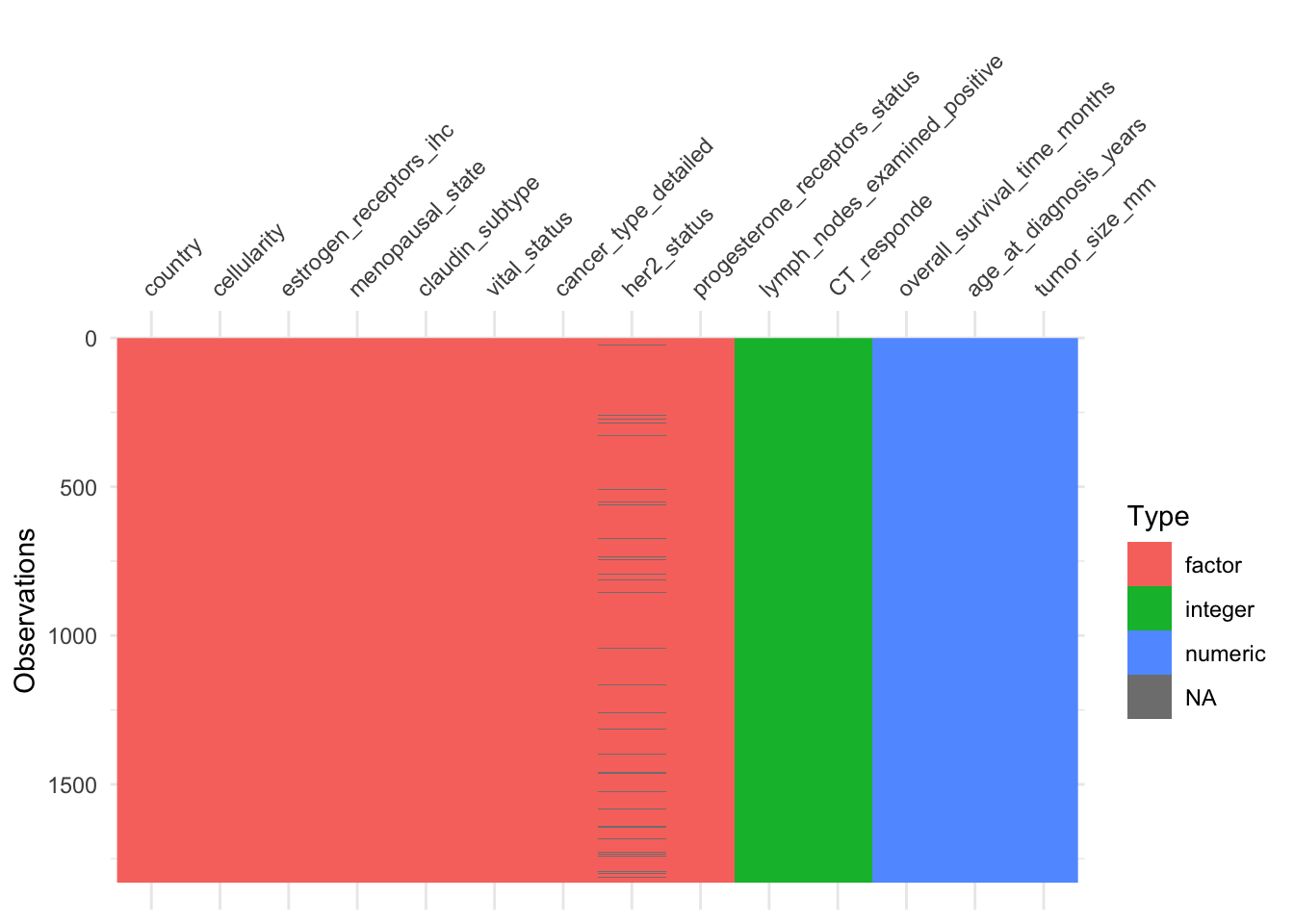
# Recuerde que debió instalar la librería visdatEDA utilizando DataExplorer
La librería DataExplorer es una herramienta que permite realizar un análisis exploratorio de datos de manera rápida y eficiente.
DataExplorer::introduce(datos) # Introducción al conjunto de datos rows columns discrete_columns continuous_columns all_missing_columns
1 1830 14 9 5 0
total_missing_values complete_rows total_observations memory_usage
1 90 1740 25620 134496DataExplorer::plot_intro(datos) # Resumen rápido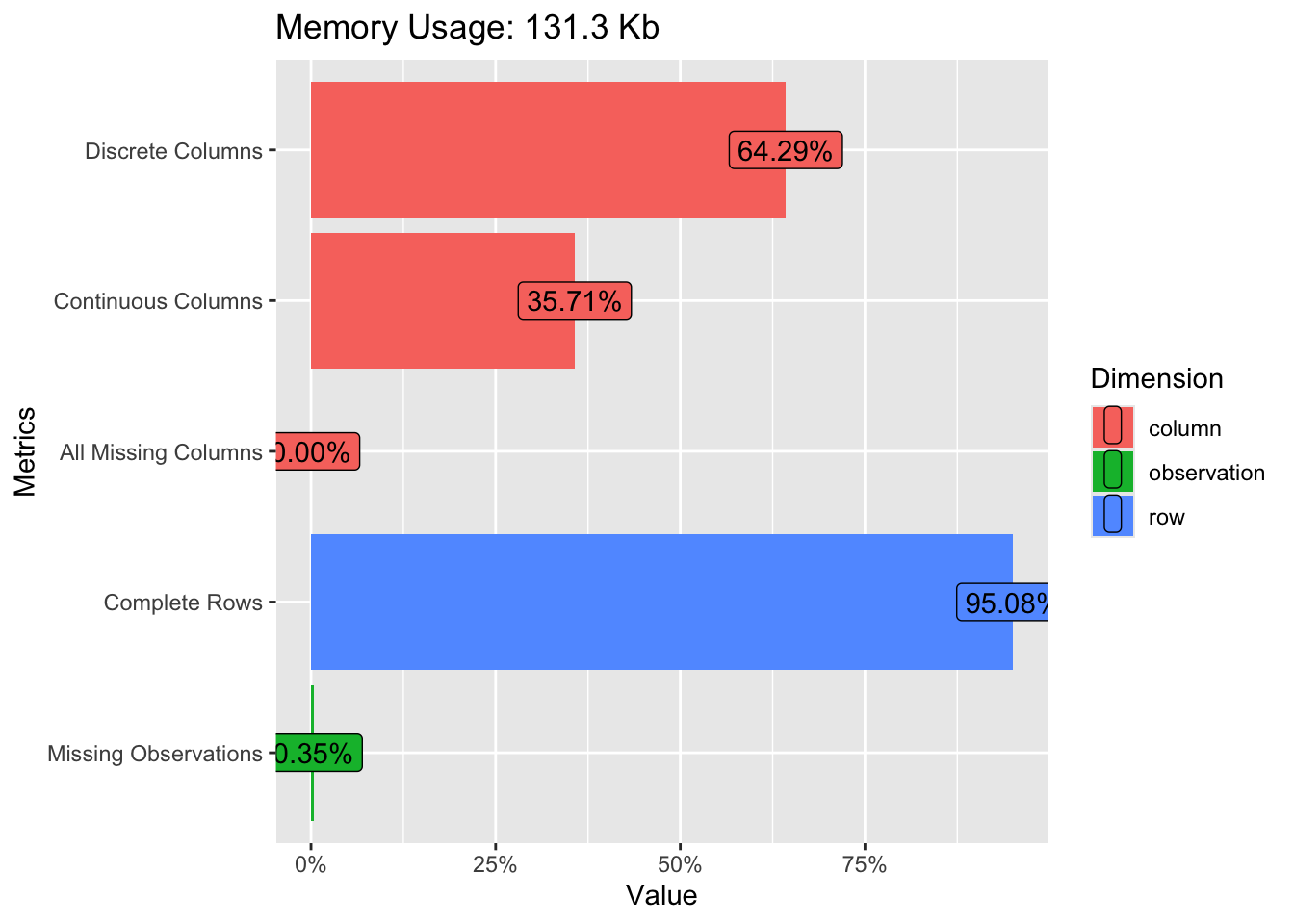
DataExplorer::plot_histogram(datos) # Histogramas de variables numéricas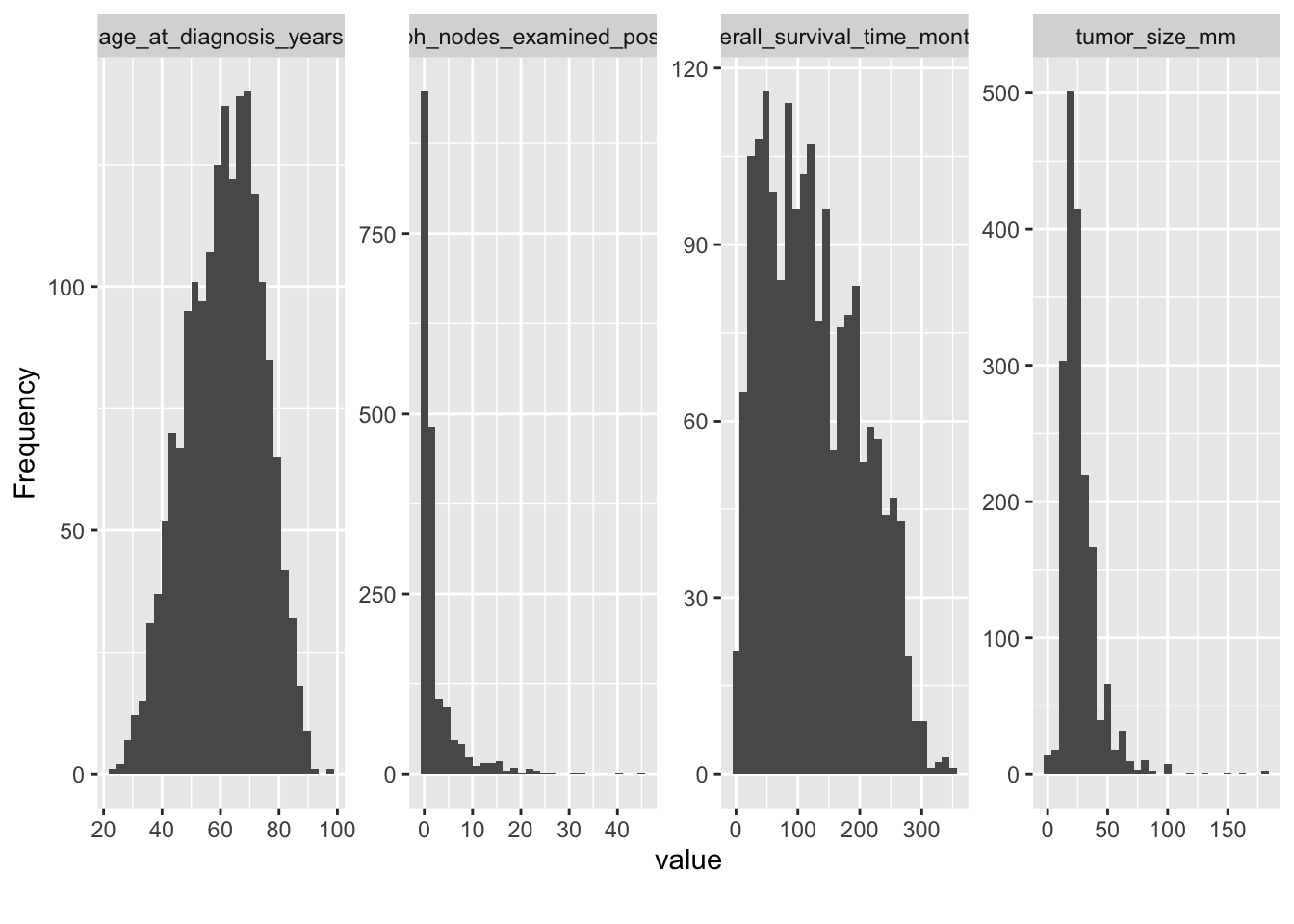
DataExplorer::plot_bar(datos) # Frecuencia de variables categóricas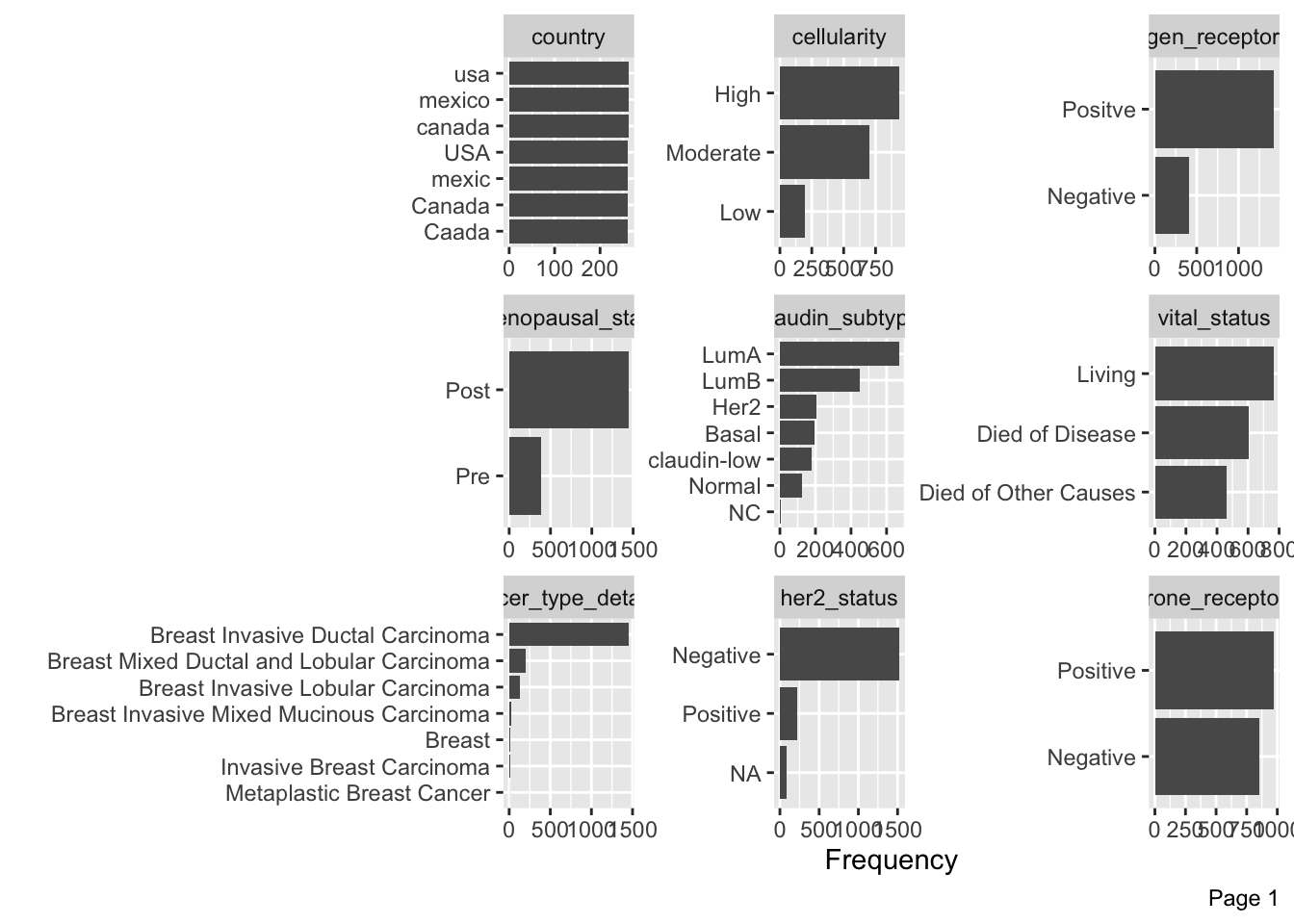
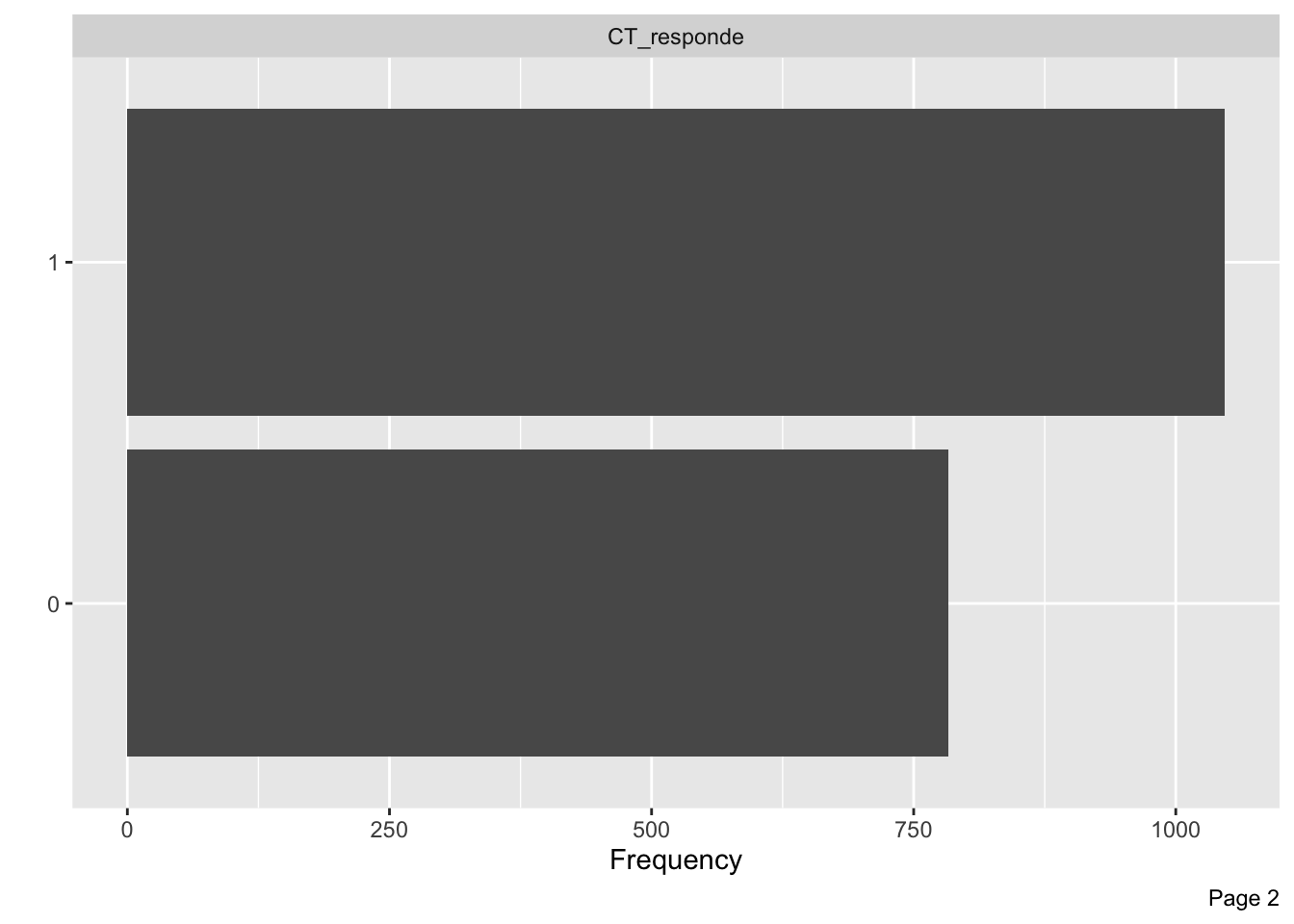
library(tidyverse)── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
✔ dplyr 1.1.4 ✔ readr 2.1.5
✔ forcats 1.0.0 ✔ stringr 1.5.1
✔ ggplot2 3.5.1 ✔ tibble 3.2.1
✔ lubridate 1.9.3 ✔ tidyr 1.3.1
✔ purrr 1.0.2
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errorsdatos %>%
select(where(is.numeric)) %>%
# Selecciona solo las columnas numéricas
DataExplorer::plot_correlation() # Correlación entre variables numéricas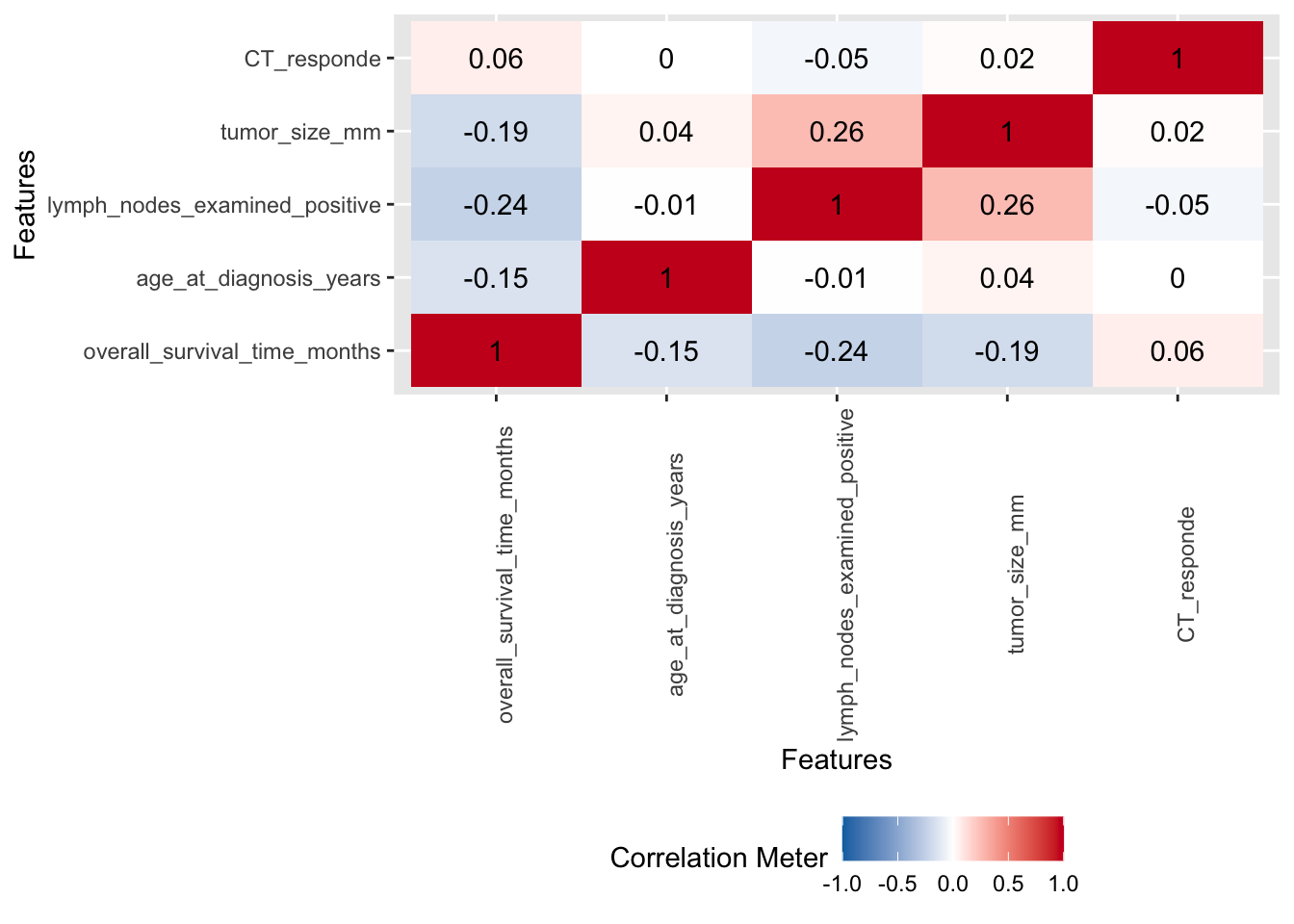
Finalmente, puede crear un reporte completa utilizando la función create_report:
DataExplorer::create_report(datos)Estadística descriptiva
También puede utilizar la función summary para obtener un resumen de las variables de la base de datos:
summary(datos) country overall_survival_time_months age_at_diagnosis_years
Caada :261 Min. : 0.10 Min. :21.93
canada:262 1st Qu.: 59.83 1st Qu.:51.52
Canada:261 Median :115.82 Median :61.88
mexic :261 Mean :124.64 Mean :61.21
mexico:262 3rd Qu.:184.33 3rd Qu.:70.69
usa :262 Max. :351.00 Max. :96.29
USA :261
lymph_nodes_examined_positive cellularity estrogen_receptors_ihc
Min. : 0.000 High :934 Negative: 406
1st Qu.: 0.000 Low :195 Positve :1424
Median : 0.000 Moderate:701
Mean : 2.034
3rd Qu.: 2.000
Max. :45.000
menopausal_state claudin_subtype vital_status
Post:1441 Basal :193 Died of Disease :602
Pre : 389 claudin-low:180 Died of Other Causes:461
Her2 :206 Living :767
LumA :670
LumB :449
NC : 6
Normal :126
cancer_type_detailed her2_status
Breast : 16 Negative:1518
Breast Invasive Ductal Carcinoma :1448 Positive: 222
Breast Invasive Lobular Carcinoma : 135 NA's : 90
Breast Invasive Mixed Mucinous Carcinoma : 20
Breast Mixed Ductal and Lobular Carcinoma: 196
Invasive Breast Carcinoma : 14
Metaplastic Breast Cancer : 1
progesterone_receptors_status tumor_size_mm CT_responde
Negative:857 Min. : 1.00 Min. :0.0000
Positive:973 1st Qu.: 18.00 1st Qu.:0.0000
Median : 23.00 Median :1.0000
Mean : 26.43 Mean :0.5721
3rd Qu.: 30.00 3rd Qu.:1.0000
Max. :182.00 Max. :1.0000
Además, en R puede utilizar la función summarytools para obtener un resumen más detallado de las variables numéricas y categóricas:
datos %>%
select(overall_survival_time_months, age_at_diagnosis_years, tumor_size_mm) %>%
descr() %>%
stview()El código anterior generará un resumen de las variables de la base de datos. Este resumen se visualizará en la ventana de Wiewer de Rstudio.´
datos %>%
freq() %>%
stview()Comparación entre los pacientes con respuesta y sin respuesta
Variables numéricas
datos %>%
ggplot(aes(x = as.factor(CT_responde), y = tumor_size_mm, fill=as.factor(CT_responde))) +
geom_boxplot(alpha = 0.5) +
theme_minimal() +
labs(title = "Comparación de tumor_size_mm por respuesta",
x = "CT_responde",
y = "Tamaño en mm")+
theme(legend.position = "none")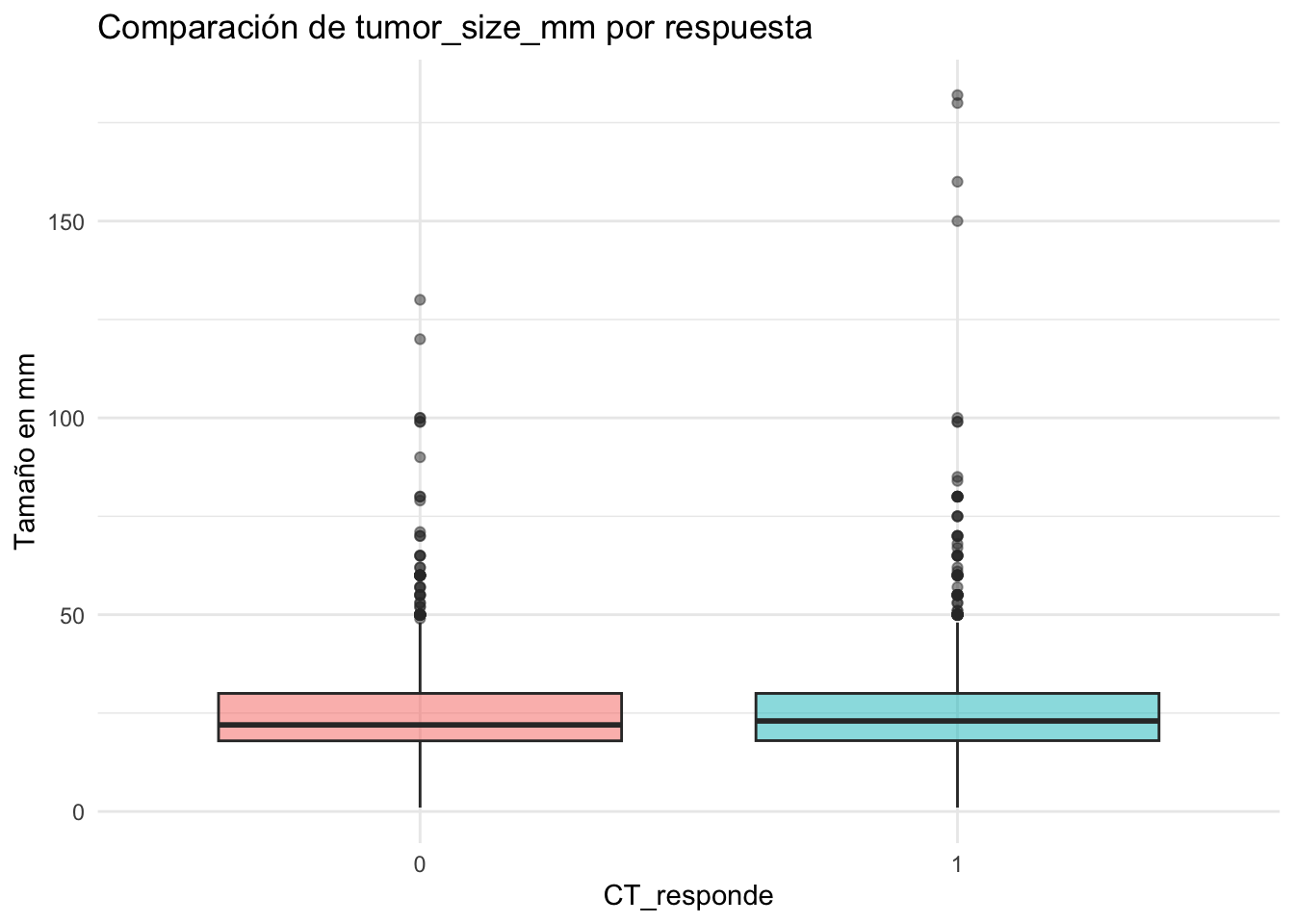
datos %>%
ggplot(aes(x = as.factor(CT_responde), y = age_at_diagnosis_years, fill=as.factor(CT_responde))) +
geom_boxplot(alpha = 0.5) +
theme_minimal() +
labs(title = "Comparación de la edad de diagnóstico por respuesta",
x = "CT_responde",
y = "Edad (años)")+
theme(legend.position = "none")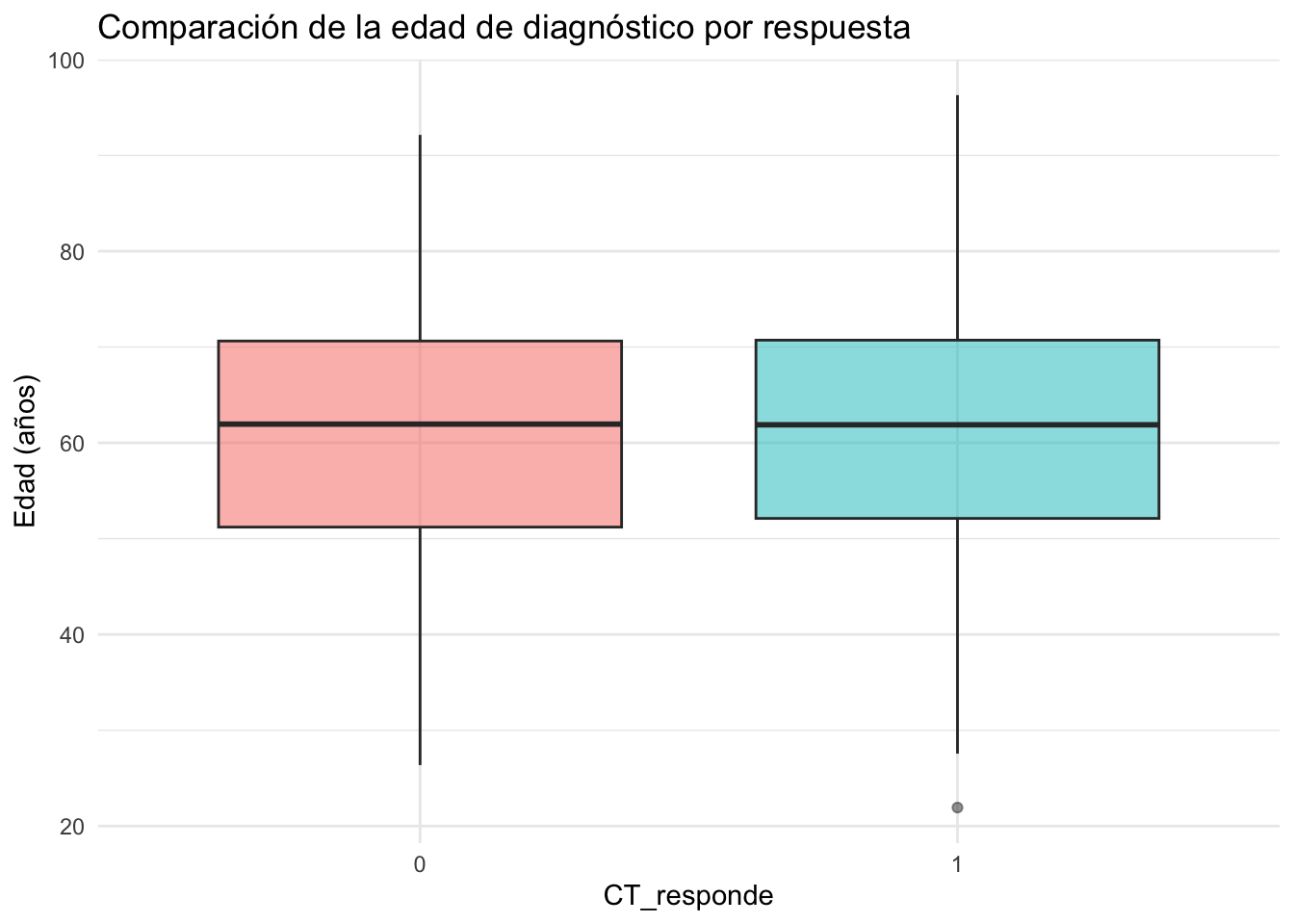
datos %>%
ggplot(aes(x = as.factor(CT_responde), y = overall_survival_time_months, fill=as.factor(CT_responde))) +
geom_boxplot(alpha = 0.5) +
theme_minimal() +
labs(title = "Comparación de la edad de diagnóstico por respuesta",
x = "CT_responde",
y = "Meses")+
theme(legend.position = "none")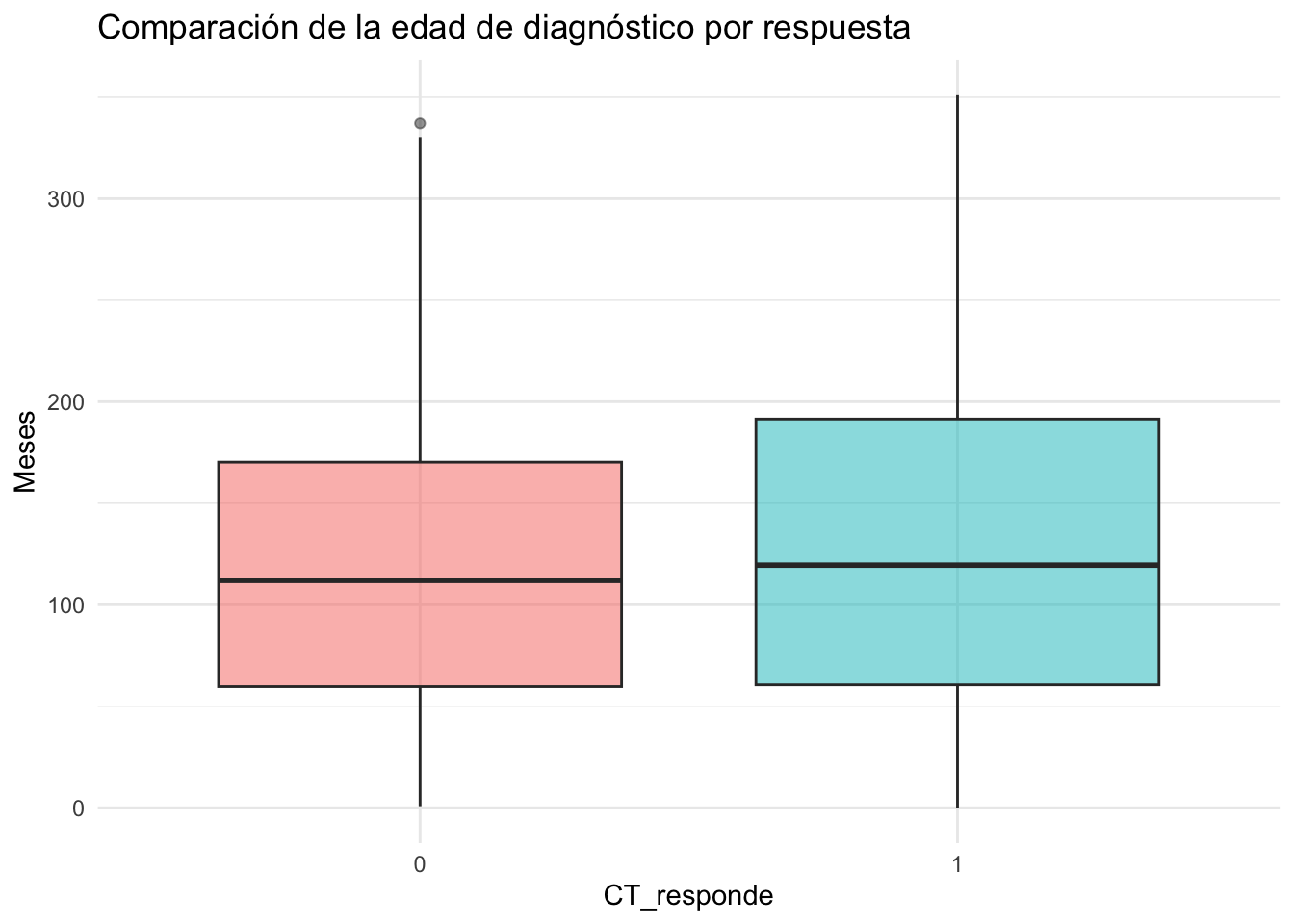
Variables categóricas
# Gráfico de barras para la relación entre `country` y `CT_responde`
datos %>%
ggplot( aes(x = country, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "country vs CT_responde", x = "country", y = "Count", fill = "CT_responde") +
theme_minimal()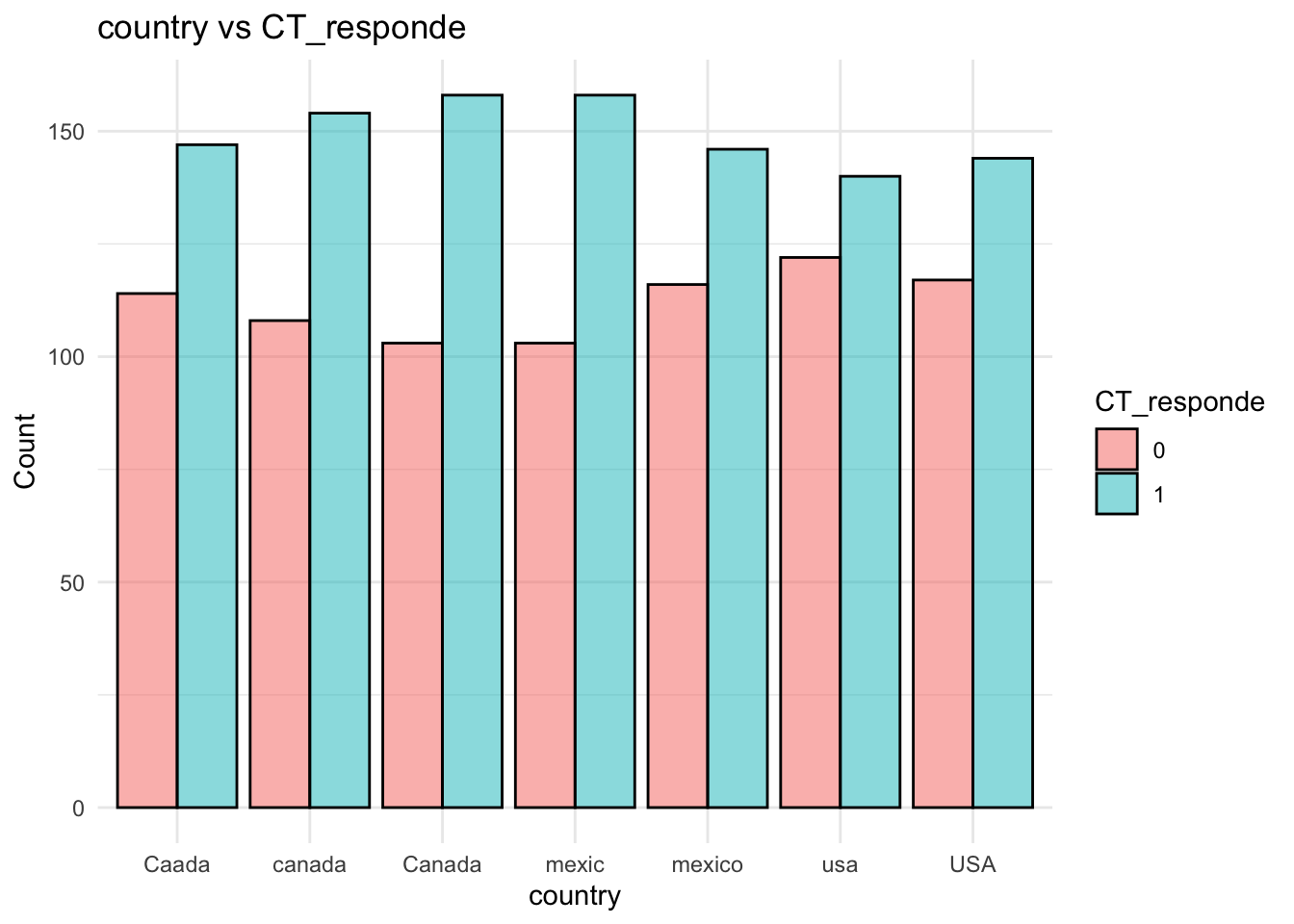
# Gráfico de barras para la relación entre `cellularity` y `CT_responde`
datos %>%
ggplot( aes(x = cellularity, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "Cellularity vs CT_responde", x = "Cellularity", y = "Count", fill = "CT_responde") +
theme_minimal()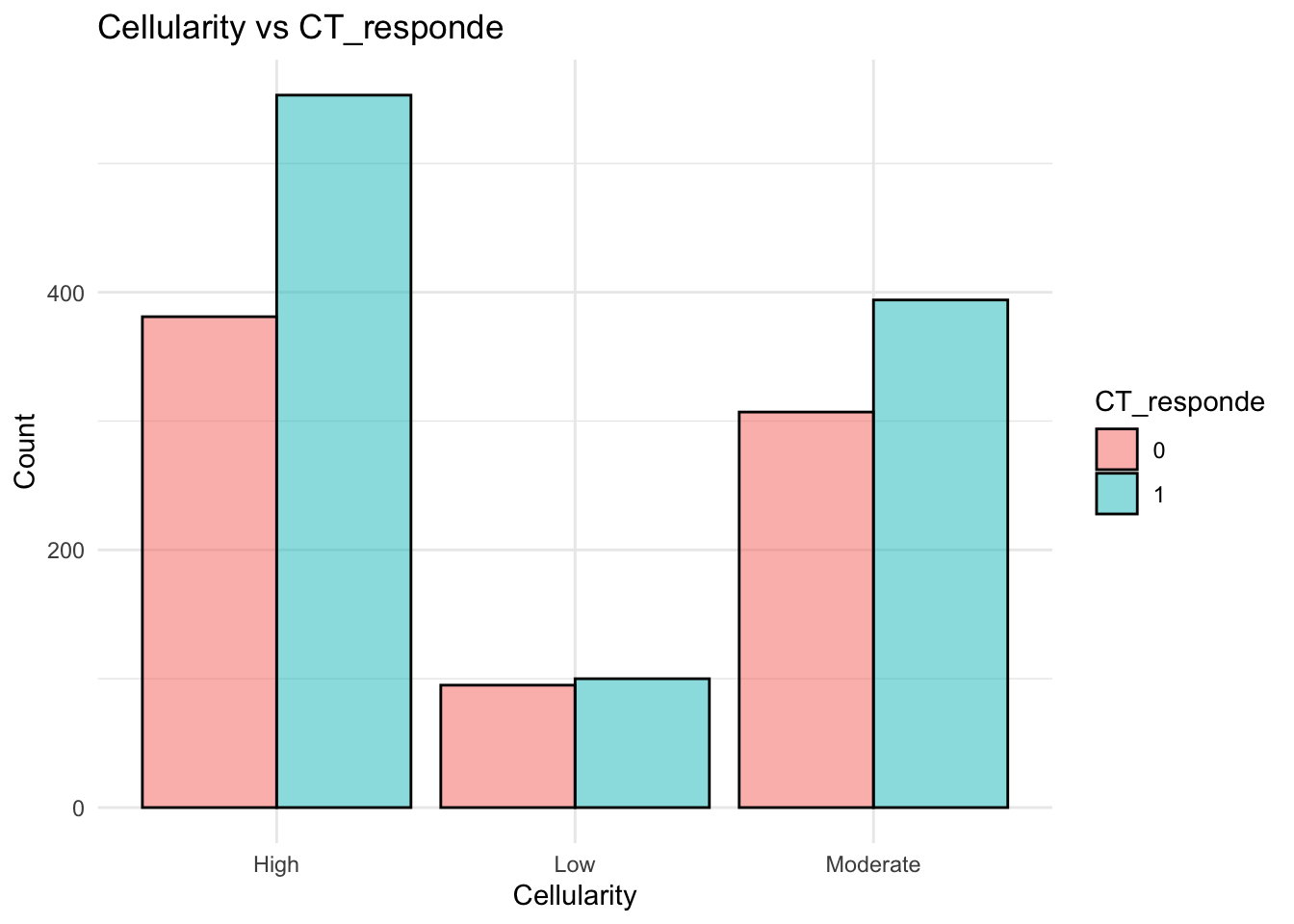
# Gráfico de barras para la relación entre `estrogen_receptors_ihc` y `CT_responde`
datos %>%
ggplot( aes(x = estrogen_receptors_ihc, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "estrogen_receptors_ihc vs CT_responde", x = "estrogen_receptors_ihc", y = "Count", fill = "CT_responde") +
theme_minimal()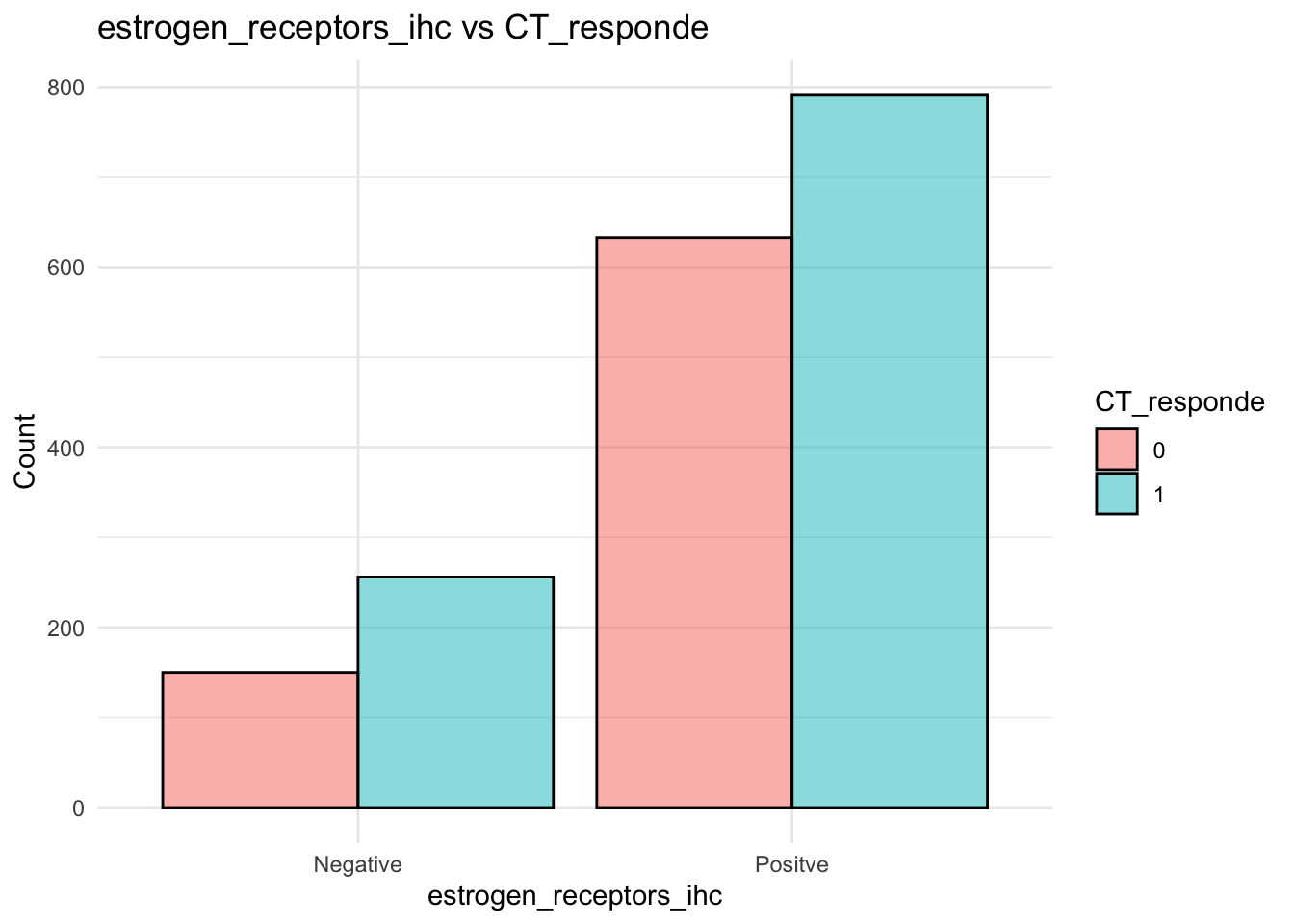
# Gráfico de barras para la relación entre `menopausal_state` y `CT_responde`
datos %>%
ggplot( aes(x = menopausal_state, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "menopausal_state vs CT_responde", x = "menopausal_state", y = "Count", fill = "CT_responde") +
theme_minimal()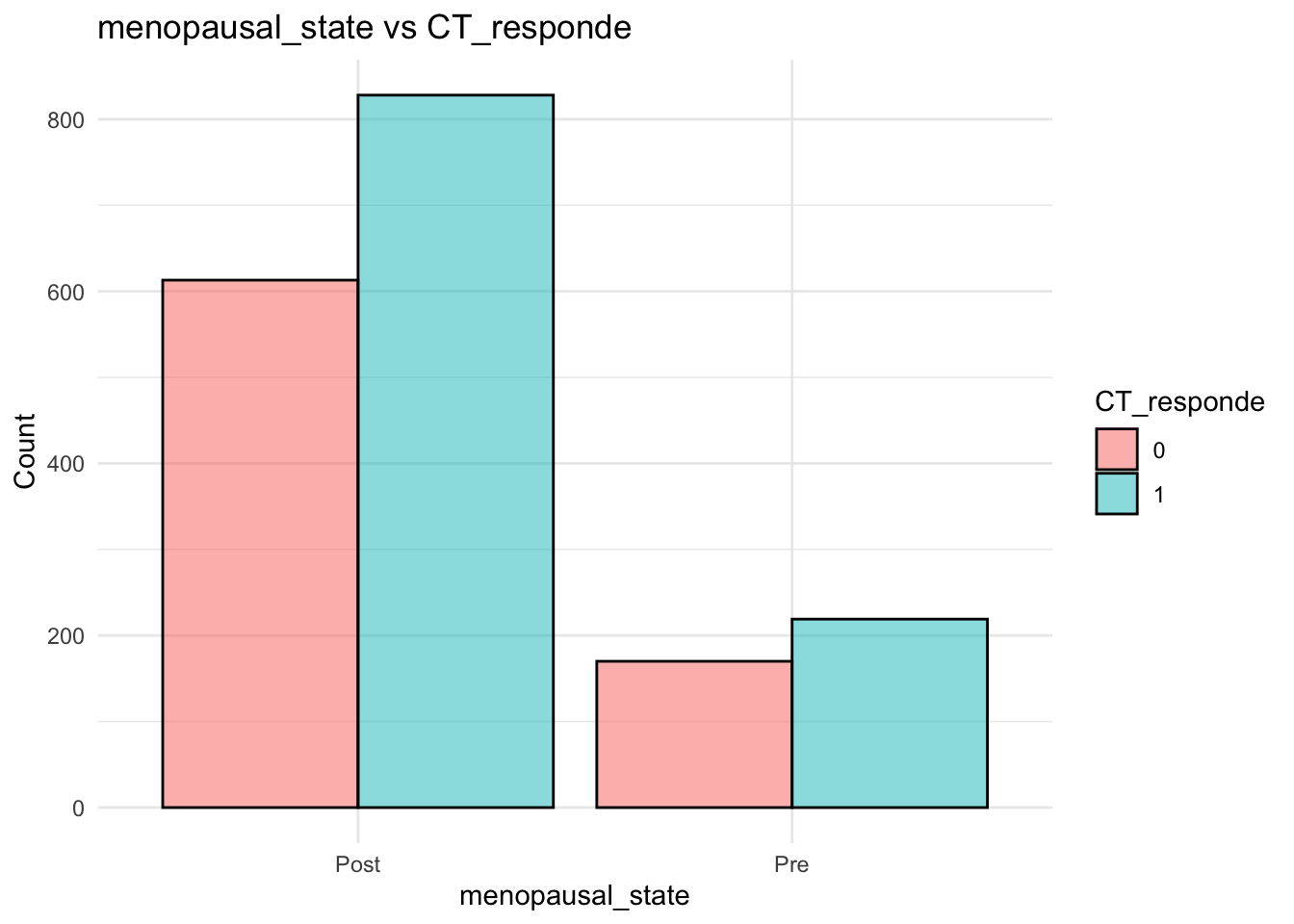
# Gráfico de barras para la relación entre `claudin_subtype` y `CT_responde`
datos %>%
ggplot( aes(x = claudin_subtype, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "claudin_subtype vs CT_responde", x = "claudin_subtype", y = "Count", fill = "CT_responde") +
theme_minimal()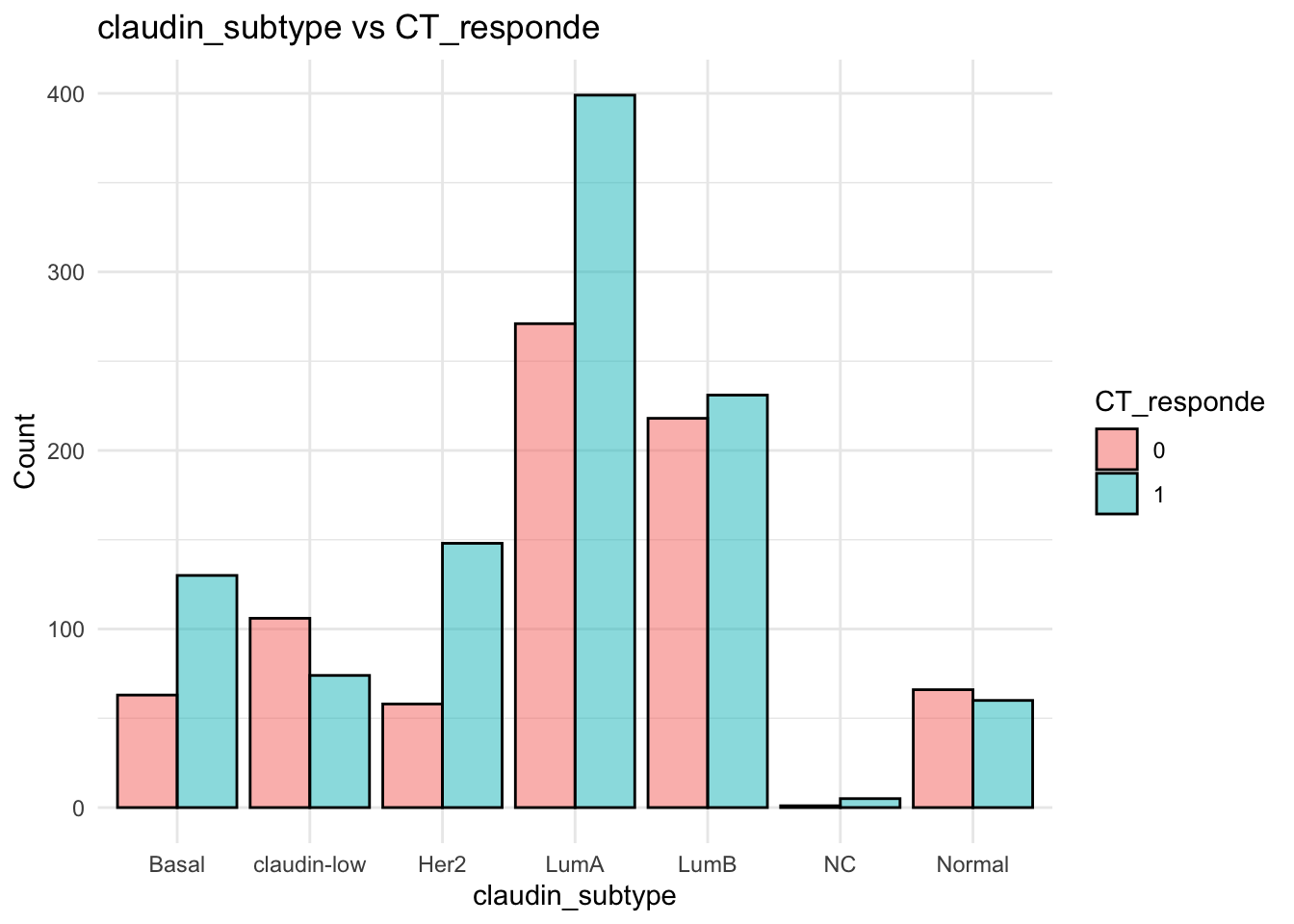
# Gráfico de barras para la relación entre `vital_status` y `CT_responde`
datos %>%
ggplot( aes(x = vital_status, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "vital_status vs CT_responde", x = "vital_status", y = "Count", fill = "CT_responde") +
theme_minimal()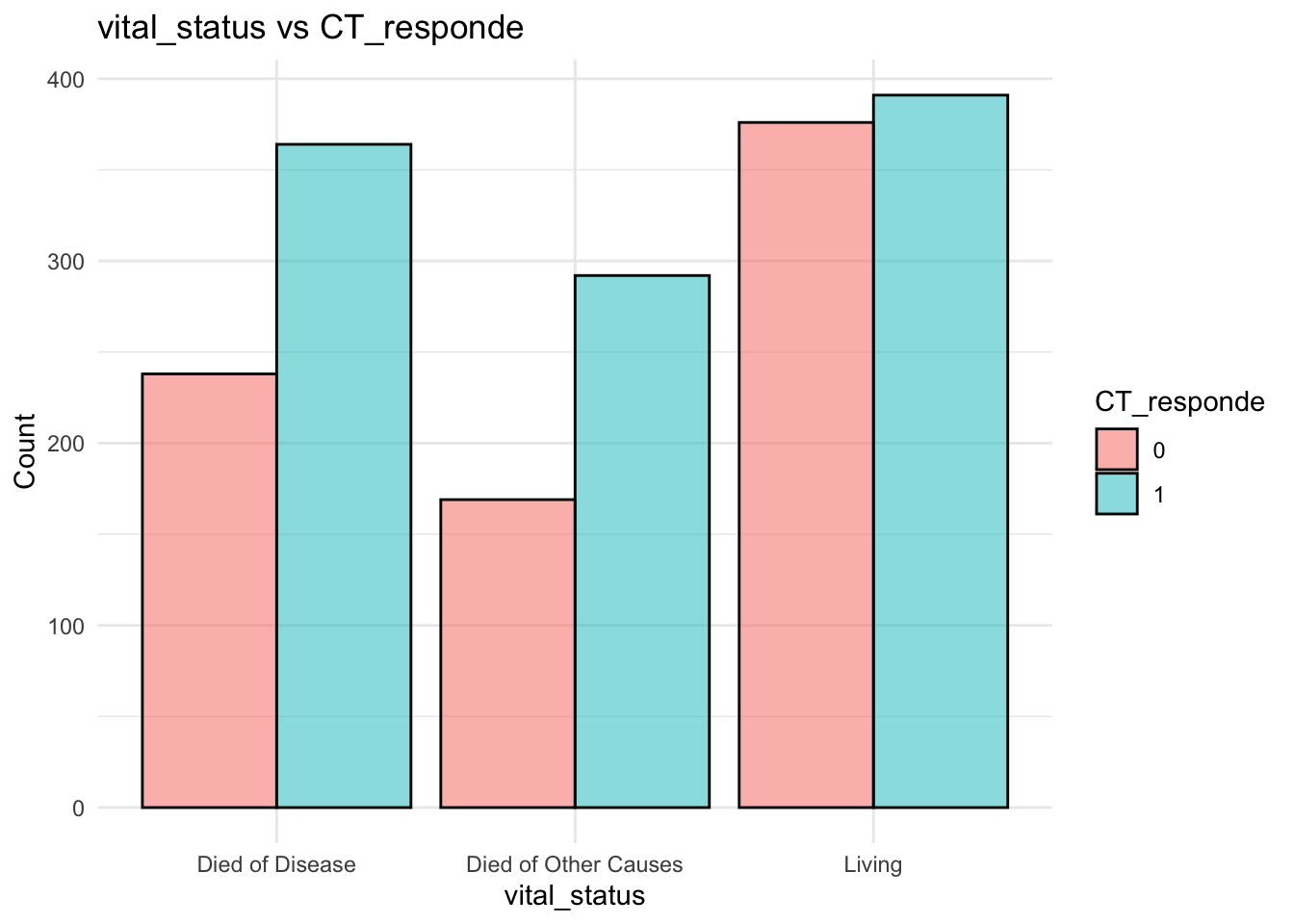
# Gráfico de barras para la relación entre `cancer_type_detailed` y `CT_responde`
datos %>%
ggplot( aes(x = cancer_type_detailed, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "cancer_type_detailed vs CT_responde", x = "cancer_type_detailed", y = "Count", fill = "CT_responde") +
theme_minimal()+
theme(axis.text.x = element_text(angle = 45, hjust = 1))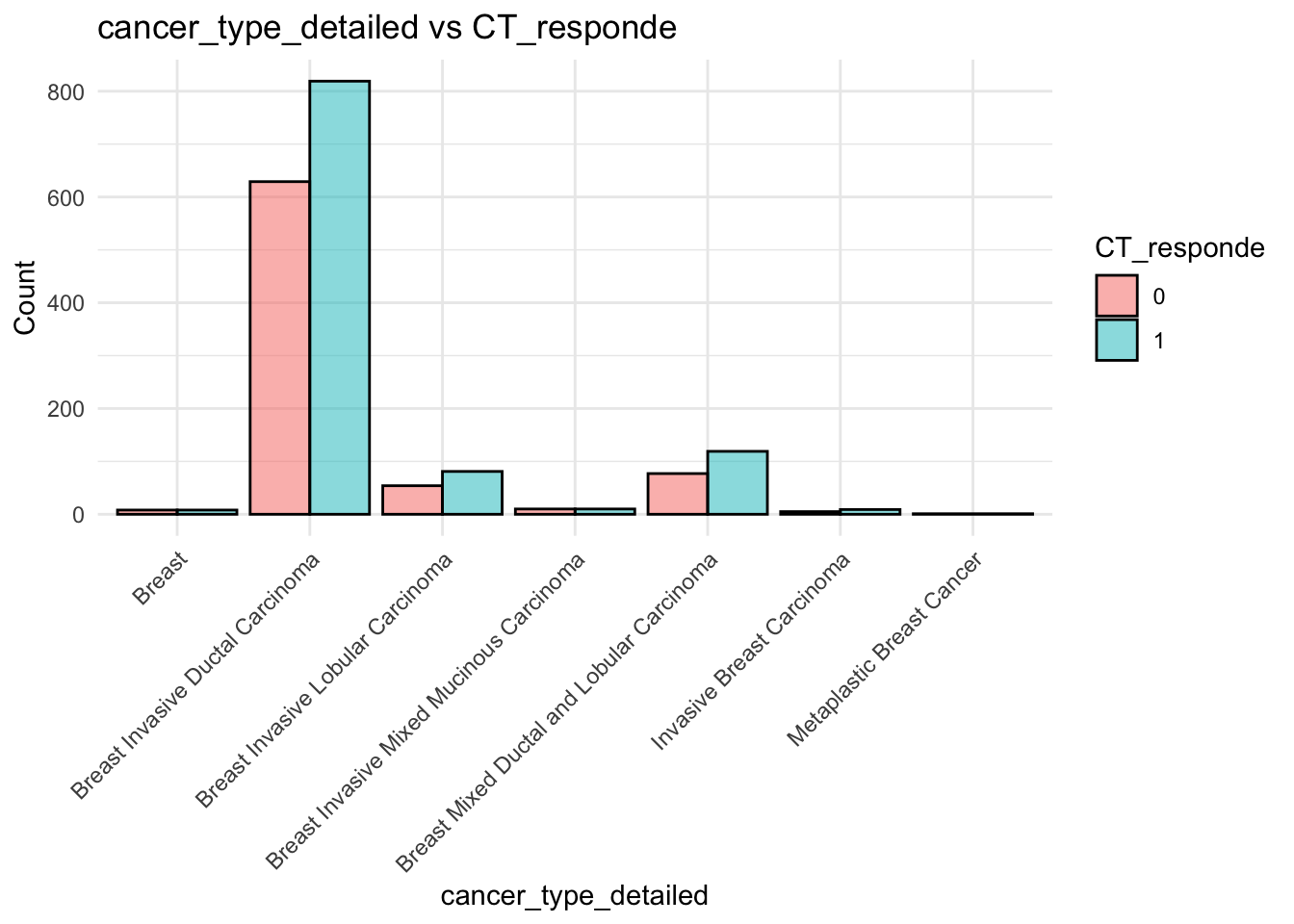
# Gráfico de barras para la relación entre `her2_status` y `CT_responde`
datos %>%
ggplot( aes(x = her2_status, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "her2_status vs CT_responde", x = "her2_status", y = "Count", fill = "CT_responde") +
theme_minimal()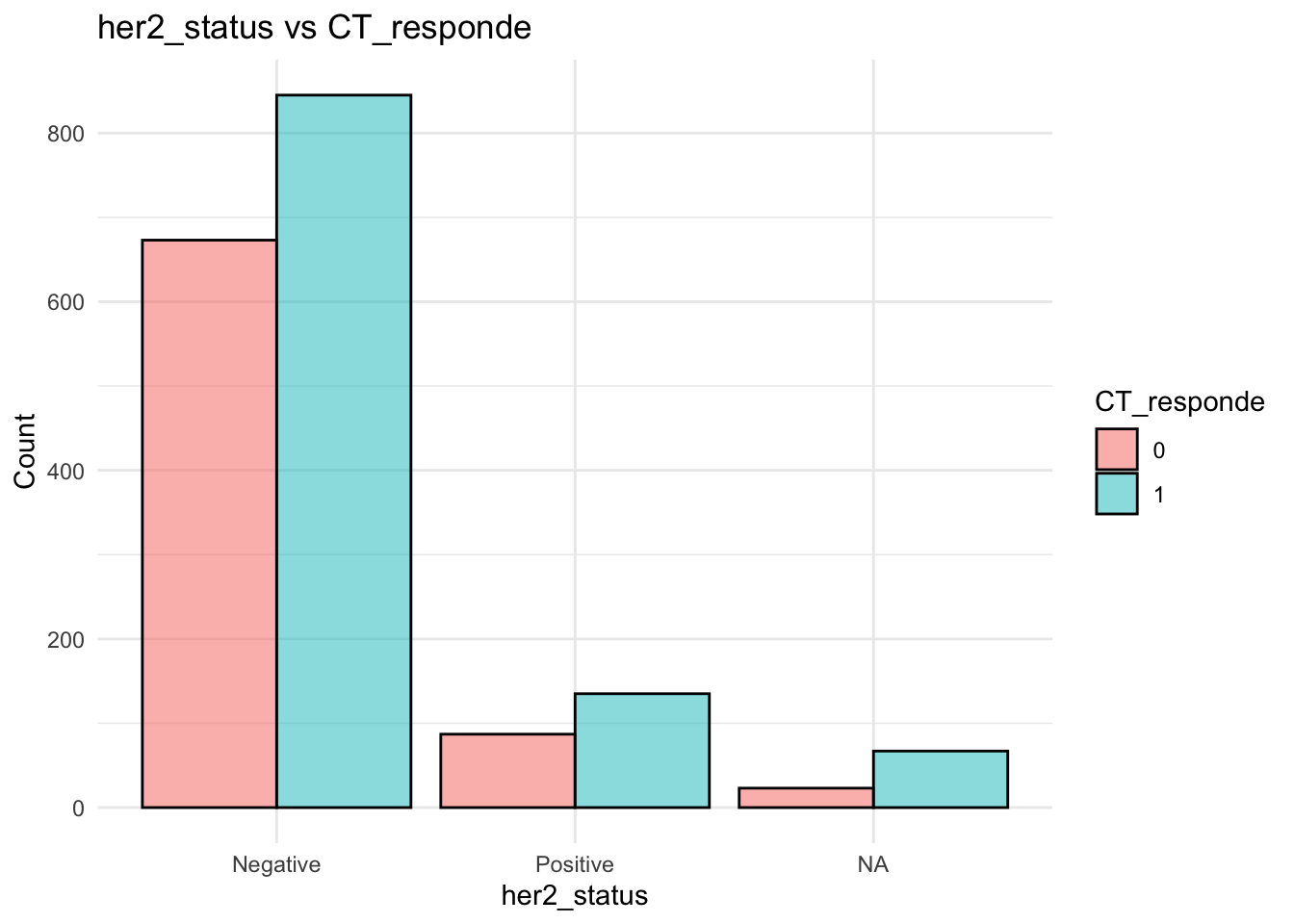
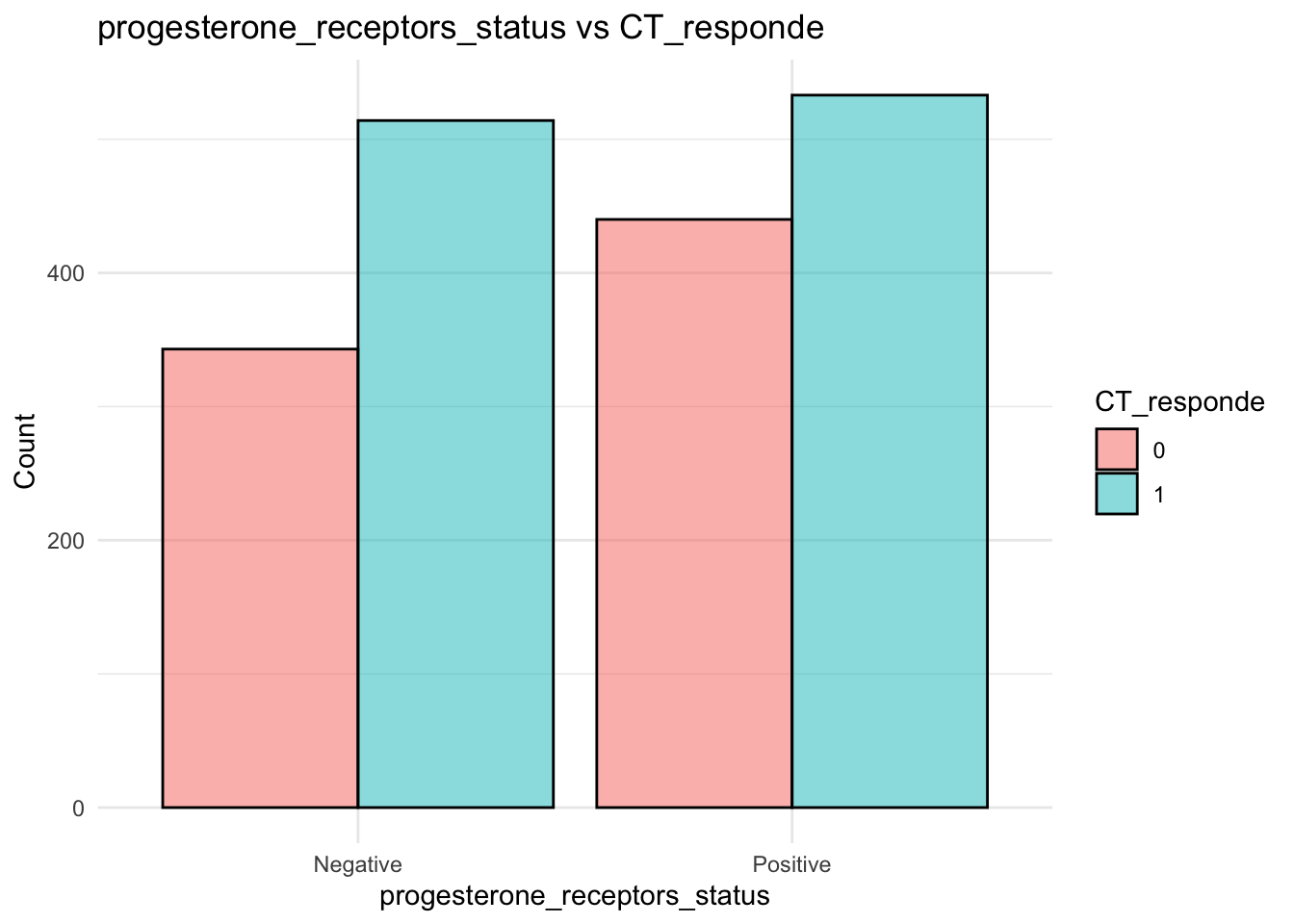
# Gráfico de barras para la relación entre `progesterone_receptors_status` y `CT_responde`
datos %>%
ggplot( aes(x = progesterone_receptors_status, fill = factor(CT_responde))) +
geom_bar(position = "dodge", col="black", alpha=0.5) +
labs(title = "progesterone_receptors_status vs CT_responde", x = "progesterone_receptors_status", y = "Count", fill = "CT_responde") +
theme_minimal()