4 Лабораторная работа № 2
Ссылка из Лабораторной работы № 1 - (“CRISP-Dm” 2012).
library("plyr")
library(GGally)
# # загружаем данные из файла
# df = pd.read_csv('dat.csv')
df <- read.csv(file = 'data/dat.csv')
# print(df.shape) # смотрим размер таблицы. Столбщы - значащие данные (признаки, ответы),
# # строки - прецеденты (объекты, отсчёты и т.д.)
dim(df)## [1] 300 3# print(df.columns) # смотрим названия столбцов таблицы
names(df) ## [1] "response" "rost" "ves"# print(df.info()) # можно посмотреть сводную информацию о таблице
str(df) ## 'data.frame': 300 obs. of 3 variables:
## $ response: int 0 0 0 0 0 0 0 0 0 0 ...
## $ rost : num 144 154 150 137 146 ...
## $ ves : num 62.5 46.6 62.2 48.1 65.5 ...# df['response'] = df['response'].astype('bool') # изменение типа данных в столбце таблицы
df$response <- factor(df$response, levels = c(0,1))
levels(df$response) <- c("women","men")
# df.describe() # основная статистика по числовым столбцам таблицы
summary(df) # по всем## response rost ves
## women:150 Min. :128.9 Min. :41.09
## men :150 1st Qu.:149.1 1st Qu.:59.07
## Median :157.9 Median :71.38
## Mean :160.2 Mean :69.65
## 3rd Qu.:170.8 3rd Qu.:79.51
## Max. :208.0 Max. :93.76# df.describe(include=['object', 'bool']) # статистика по нечисловым столбцам таблицы (их тип нужно явно указывать)
summary(df$response) ## women men
## 150 150# df.rost.describe() # статистика по отдельному столбцу, если его название без пробелов
summary(df$rost) ## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 128.9 149.1 157.9 160.2 170.8 208.0############################################### Дополнительно - начало вставки 1
library(Hmisc)
describe(df)## df
##
## 3 Variables 300 Observations
## ---------------------------------------------------------------------------
## response
## n missing distinct
## 300 0 2
##
## Value women men
## Frequency 150 150
## Proportion 0.5 0.5
## ---------------------------------------------------------------------------
## rost
## n missing distinct Info Mean Gmd .05 .10
## 300 0 300 1 160.2 16.58 138.7 143.9
## .25 .50 .75 .90 .95
## 149.1 157.9 170.8 180.9 185.7
##
## lowest : 128.8930 130.4864 130.8788 133.2490 134.8811
## highest: 197.6742 198.1256 198.3857 202.2963 207.9950
## ---------------------------------------------------------------------------
## ves
## n missing distinct Info Mean Gmd .05 .10
## 300 0 300 1 69.65 13.56 51.12 53.44
## .25 .50 .75 .90 .95
## 59.07 71.38 79.51 84.14 86.18
##
## lowest : 41.08715 43.05476 44.09614 46.45545 46.60425
## highest: 88.63190 90.70080 90.78764 93.40361 93.75670
## ---------------------------------------------------------------------------library(pastecs)
stat.desc(df)## response rost ves
## nbr.val NA 3.000000e+02 3.000000e+02
## nbr.null NA 0.000000e+00 0.000000e+00
## nbr.na NA 0.000000e+00 0.000000e+00
## min NA 1.288930e+02 4.108715e+01
## max NA 2.079950e+02 9.375670e+01
## range NA 7.910207e+01 5.266955e+01
## sum NA 4.804969e+04 2.089501e+04
## median NA 1.579315e+02 7.138486e+01
## mean NA 1.601656e+02 6.965003e+01
## SE.mean NA 8.498706e-01 6.817182e-01
## CI.mean NA 1.672485e+00 1.341573e+00
## var NA 2.166840e+02 1.394219e+02
## std.dev NA 1.472019e+01 1.180771e+01
## coef.var NA 9.190605e-02 1.695291e-01library(psych)
describe(df)## df
##
## 3 Variables 300 Observations
## ---------------------------------------------------------------------------
## response
## n missing distinct
## 300 0 2
##
## Value women men
## Frequency 150 150
## Proportion 0.5 0.5
## ---------------------------------------------------------------------------
## rost
## n missing distinct Info Mean Gmd .05 .10
## 300 0 300 1 160.2 16.58 138.7 143.9
## .25 .50 .75 .90 .95
## 149.1 157.9 170.8 180.9 185.7
##
## lowest : 128.8930 130.4864 130.8788 133.2490 134.8811
## highest: 197.6742 198.1256 198.3857 202.2963 207.9950
## ---------------------------------------------------------------------------
## ves
## n missing distinct Info Mean Gmd .05 .10
## 300 0 300 1 69.65 13.56 51.12 53.44
## .25 .50 .75 .90 .95
## 59.07 71.38 79.51 84.14 86.18
##
## lowest : 41.08715 43.05476 44.09614 46.45545 46.60425
## highest: 88.63190 90.70080 90.78764 93.40361 93.75670
## ---------------------------------------------------------------------------############################################### Дополнительно - конец вставки 1
# df['ves'].describe() # статистика по отдельному столбцу, если его название с пробелами и т.п.
#
# df['response'].value_counts() # статистика по значениям (частота)
#
# df['rost'].value_counts() # статистика по значениям (частота)
#
# df['rost'].value_counts(normalize=True) # относительные частоты
#
# df[df['rost']>180]['rost'].value_counts() # можно выбирать данные по некоторому условию
table(which(df$rost>180))##
## 153 156 158 163 166 168 170 183 187 189 193 207 211 212 215 217 218 219
## 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## 225 227 228 244 245 246 260 265 276 287 288 289 290 292 295
## 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1# или
as.data.frame(table(which(df$rost>180)))## Var1 Freq
## 1 153 1
## 2 156 1
## 3 158 1
## 4 163 1
## 5 166 1
## 6 168 1
## 7 170 1
## 8 183 1
## 9 187 1
## 10 189 1
## 11 193 1
## 12 207 1
## 13 211 1
## 14 212 1
## 15 215 1
## 16 217 1
## 17 218 1
## 18 219 1
## 19 225 1
## 20 227 1
## 21 228 1
## 22 244 1
## 23 245 1
## 24 246 1
## 25 260 1
## 26 265 1
## 27 276 1
## 28 287 1
## 29 288 1
## 30 289 1
## 31 290 1
## 32 292 1
## 33 295 1# или
df[df$rost>180,]## response rost ves
## 153 men 181.1116 82.42039
## 156 men 180.2999 79.80749
## 158 men 187.8765 85.43642
## 163 men 185.6610 74.99964
## 166 men 185.0639 79.57333
## 168 men 181.3835 73.06895
## 170 men 195.1993 76.25309
## 183 men 202.2963 73.76578
## 187 men 184.7543 74.34362
## 189 men 180.9998 77.61756
## 193 men 182.7181 79.27003
## 207 men 188.8581 80.51690
## 211 men 182.5276 75.32260
## 212 men 185.1184 79.23645
## 215 men 184.7683 84.94426
## 217 men 186.7997 81.95425
## 218 men 184.5979 73.24000
## 219 men 198.1256 81.09497
## 225 men 187.0668 80.35839
## 227 men 181.5270 76.79385
## 228 men 189.6189 86.64277
## 244 men 180.9408 82.60832
## 245 men 197.6742 78.44300
## 246 men 180.1717 84.13064
## 260 men 193.4545 85.39594
## 265 men 198.3857 75.16473
## 276 men 188.8862 76.39678
## 287 men 190.9366 72.28251
## 288 men 191.8145 82.85528
## 289 men 182.7396 79.23039
## 290 men 183.0078 82.76664
## 292 men 185.3093 76.80294
## 295 men 207.9950 80.20416# или
which(df$rost>180)## [1] 153 156 158 163 166 168 170 183 187 189 193 207 211 212 215 217 218
## [18] 219 225 227 228 244 245 246 260 265 276 287 288 289 290 292 295# df['rost'].plot() # построим график одного из признаков
plot(df$rost)
Figure 4.1: Название рисунка
plot(df$rost,type = "l")
Figure 4.2: Название рисунка
# df[['rost','ves']].plot() # или нескольких
#####################################################
par(mfrow=c(2,1))
plot(df$rost,type = "l",col="blue")
plot(df$ves,type = "l",col="orange") 
Figure 4.3: Название рисунка
# или
coplot(df$rost~df$ves|df$response) 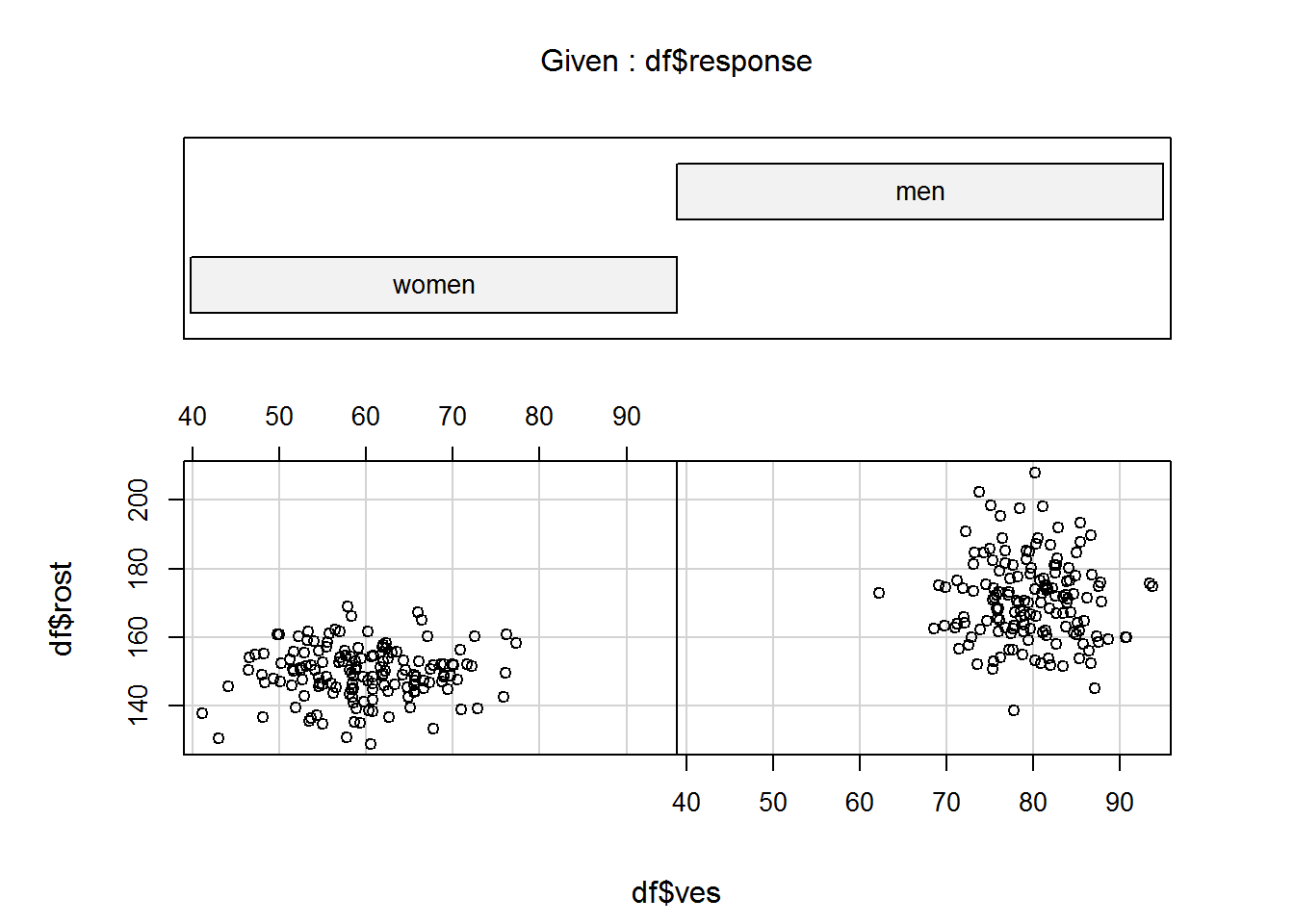
Figure 4.4: Название рисунка
# или
library(ggplot2)
g <- ggplot(df, aes(1:nrow(df))) + # basic graphical object
geom_line(aes(y=rost), colour="blue") + # first layer
geom_line(aes(y=ves), colour="orange") # second layer
print(g)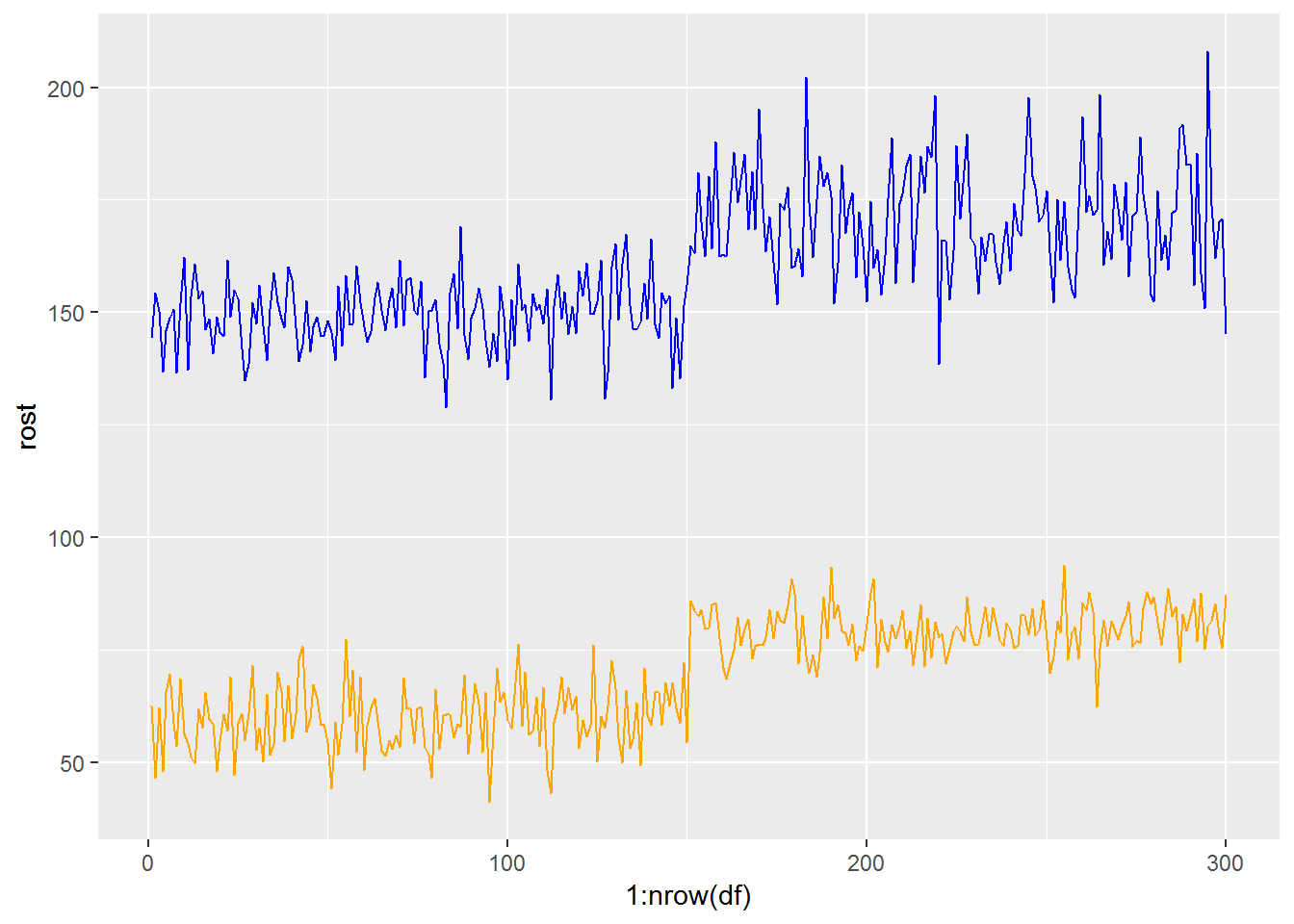
Figure 4.5: Название рисунка
######################################
# scatter_matrix(df[['rost','ves']], alpha=0.5, figsize=(10, 10)) # можно строить диаграммы рассеяния
pairs(df[,c('rost','ves')],col=df$response)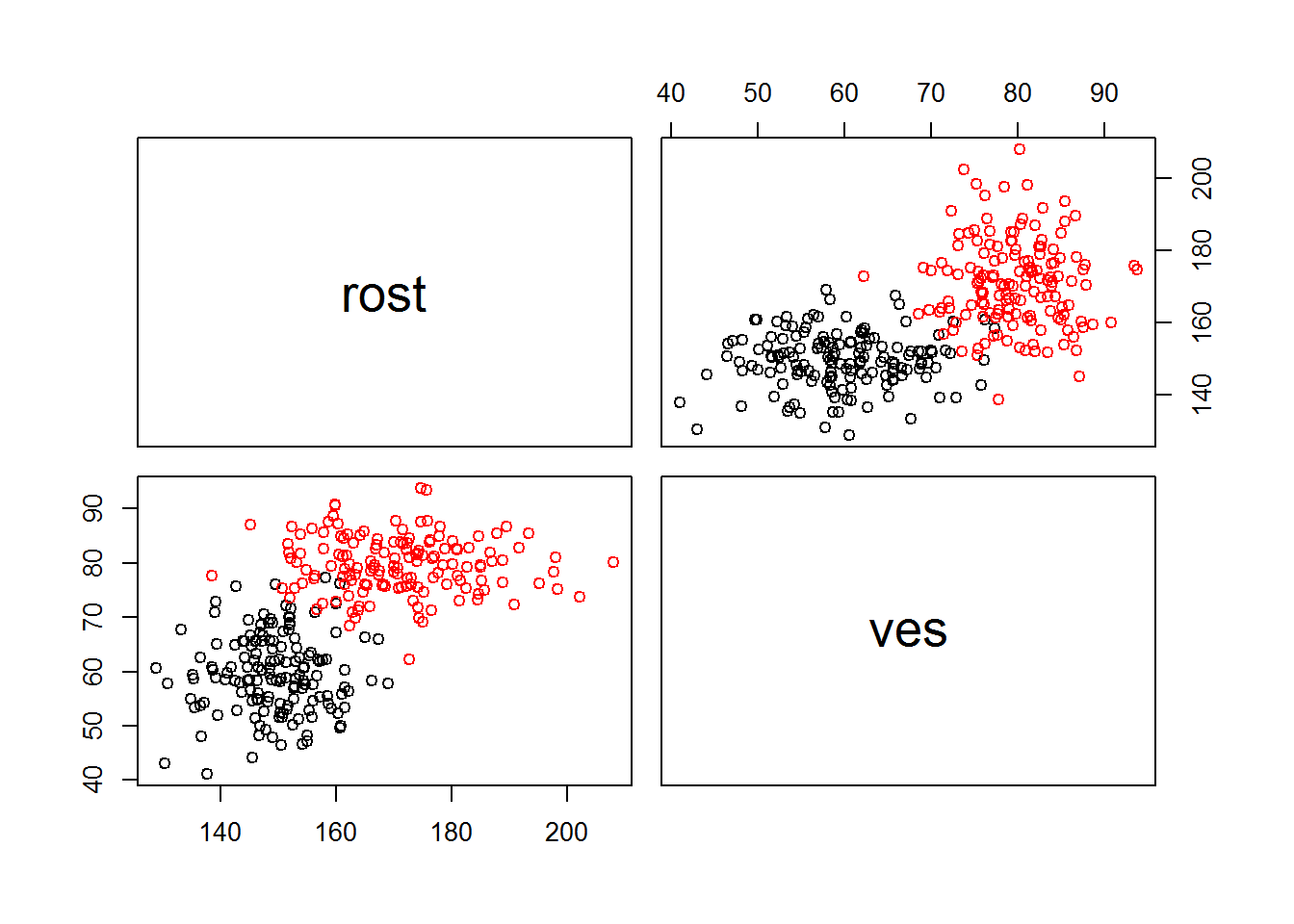
Figure 4.6: Название рисунка
# или
######################################
panel.cor <- function(x, y, digits = 2, prefix = "", cex.cor, ...)
{
usr <- par("usr"); on.exit(par(usr))
par(usr = c(0, 1, 0, 1))
r <- abs(cor(x, y))
txt <- format(c(r, 0.123456789), digits = digits)[1]
txt <- paste0(prefix, txt)
if(missing(cex.cor)) cex.cor <- 0.8/strwidth(txt)
text(0.5, 0.5, txt, cex = cex.cor * r)
}
panel.hist <- function(x, ...)
{
usr <- par("usr"); on.exit(par(usr))
par(usr = c(usr[1:2], 0, 1.5) )
h <- hist(x, plot = FALSE)
breaks <- h$breaks; nB <- length(breaks)
y <- h$counts; y <- y/max(y)
rect(breaks[-nB], 0, breaks[-1], y, col = "cyan", ...)
}
pairs(df[,c('rost','ves')],upper.panel=panel.cor,diag.panel=panel.hist)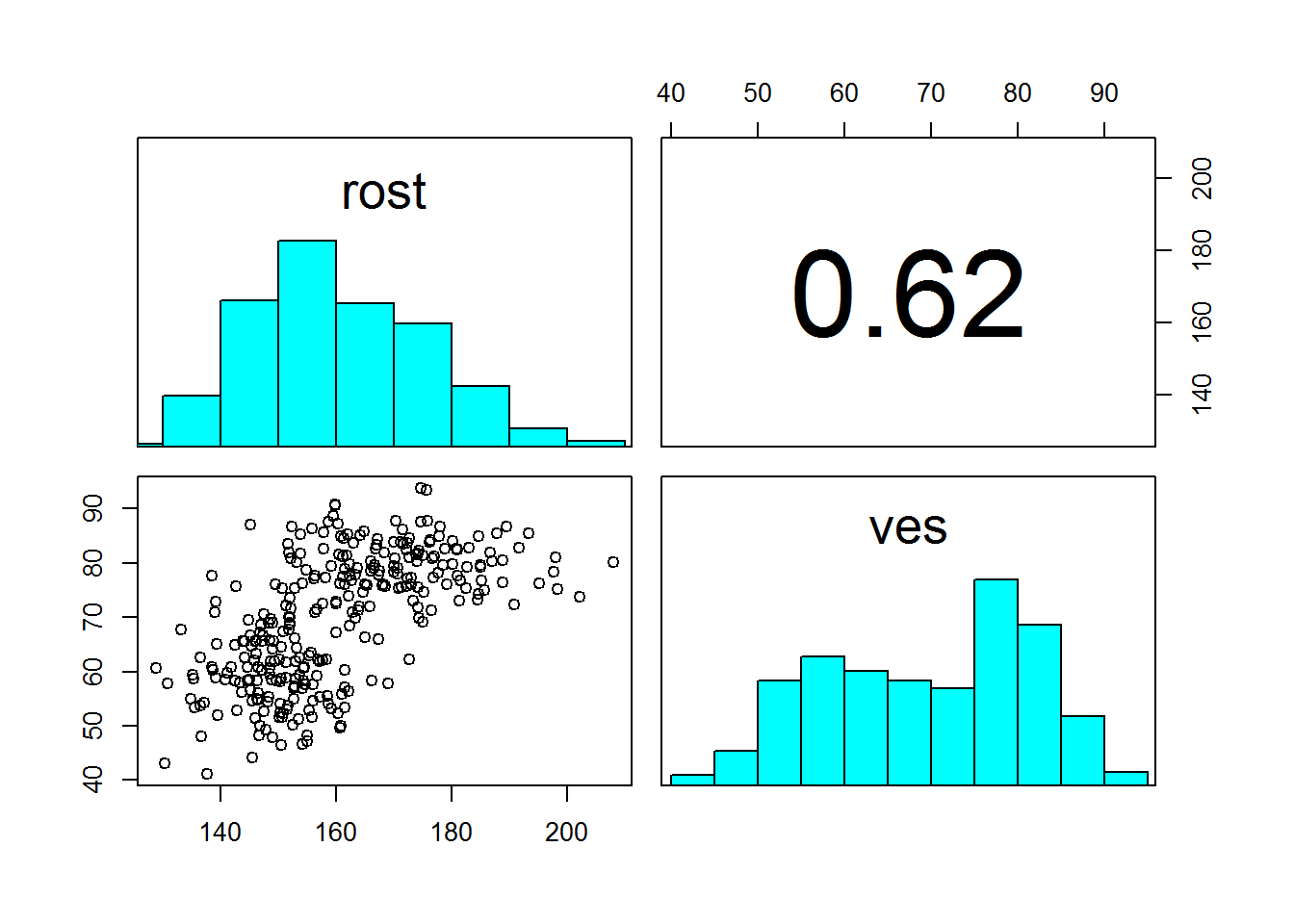
Figure 4.7: Название рисунка
######################################
# или
library(psych)
with(df,scatter.hist(rost,ves))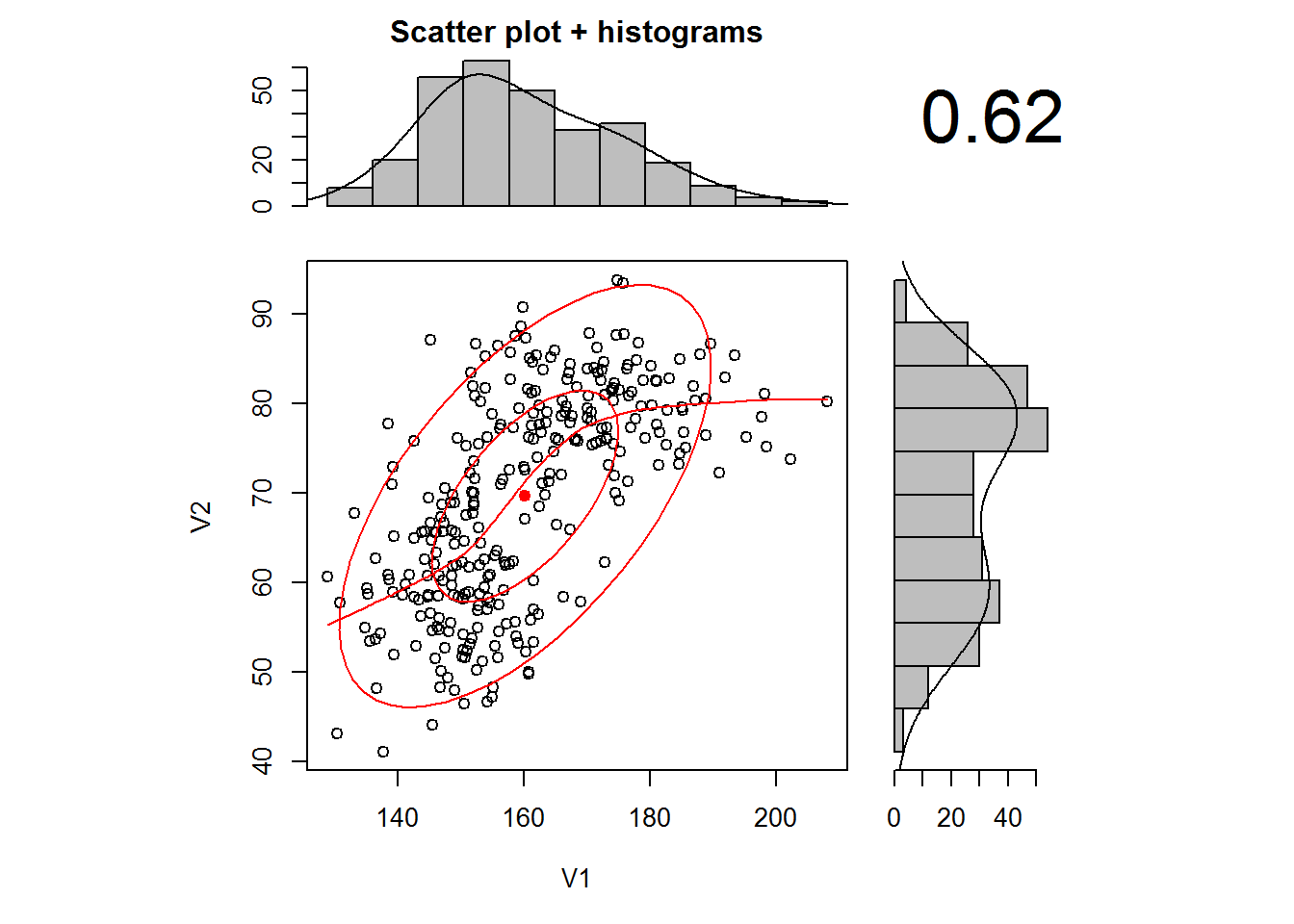
Figure 4.8: Название рисунка
scatter.hist(df[,2:3],col=c("blue","red")[df$response],grid=TRUE)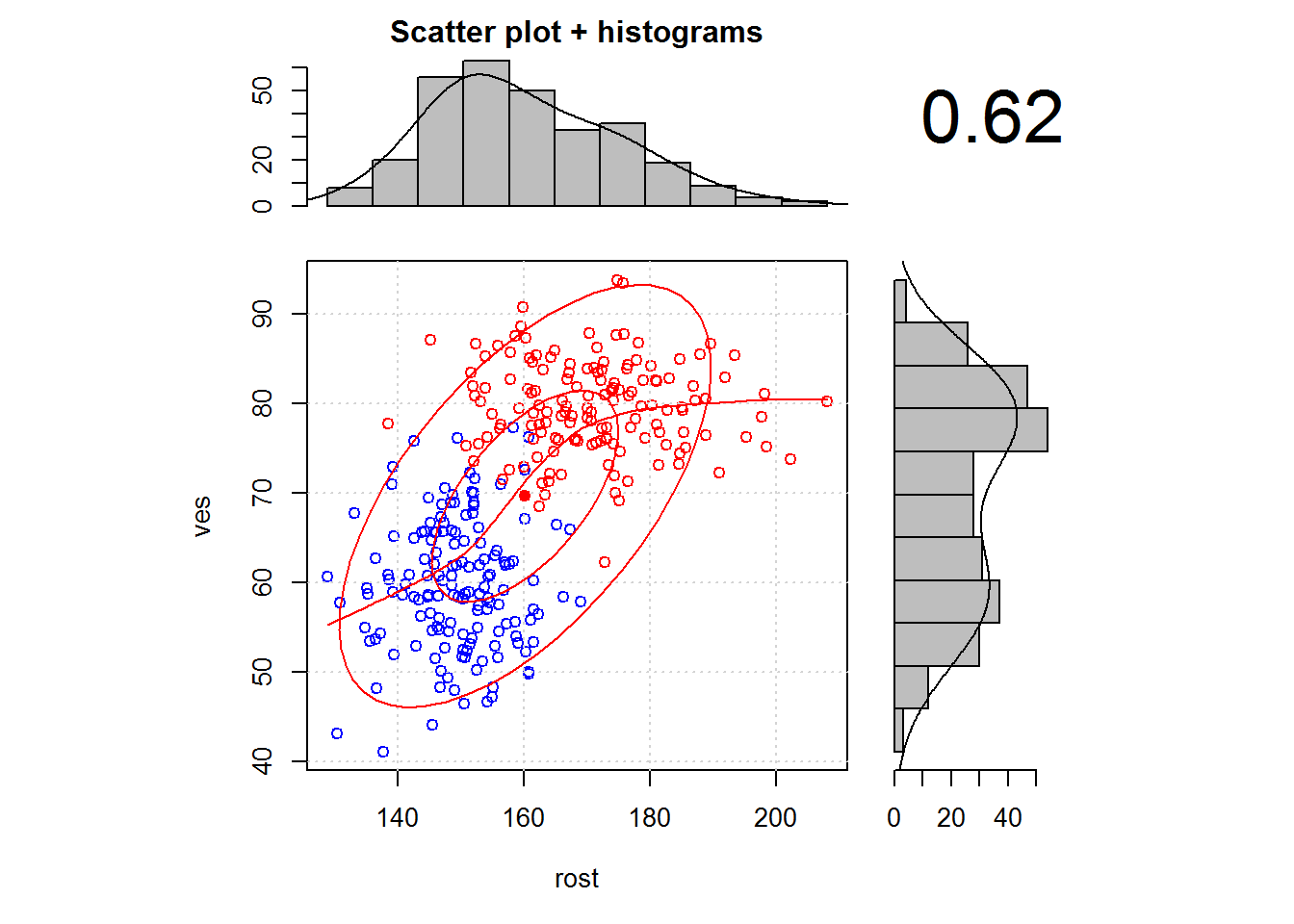
Figure 4.9: Название рисунка
# или
library(GGally)
g <- ggpairs(df, aes(colour = response, alpha = .4))
print(g)## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.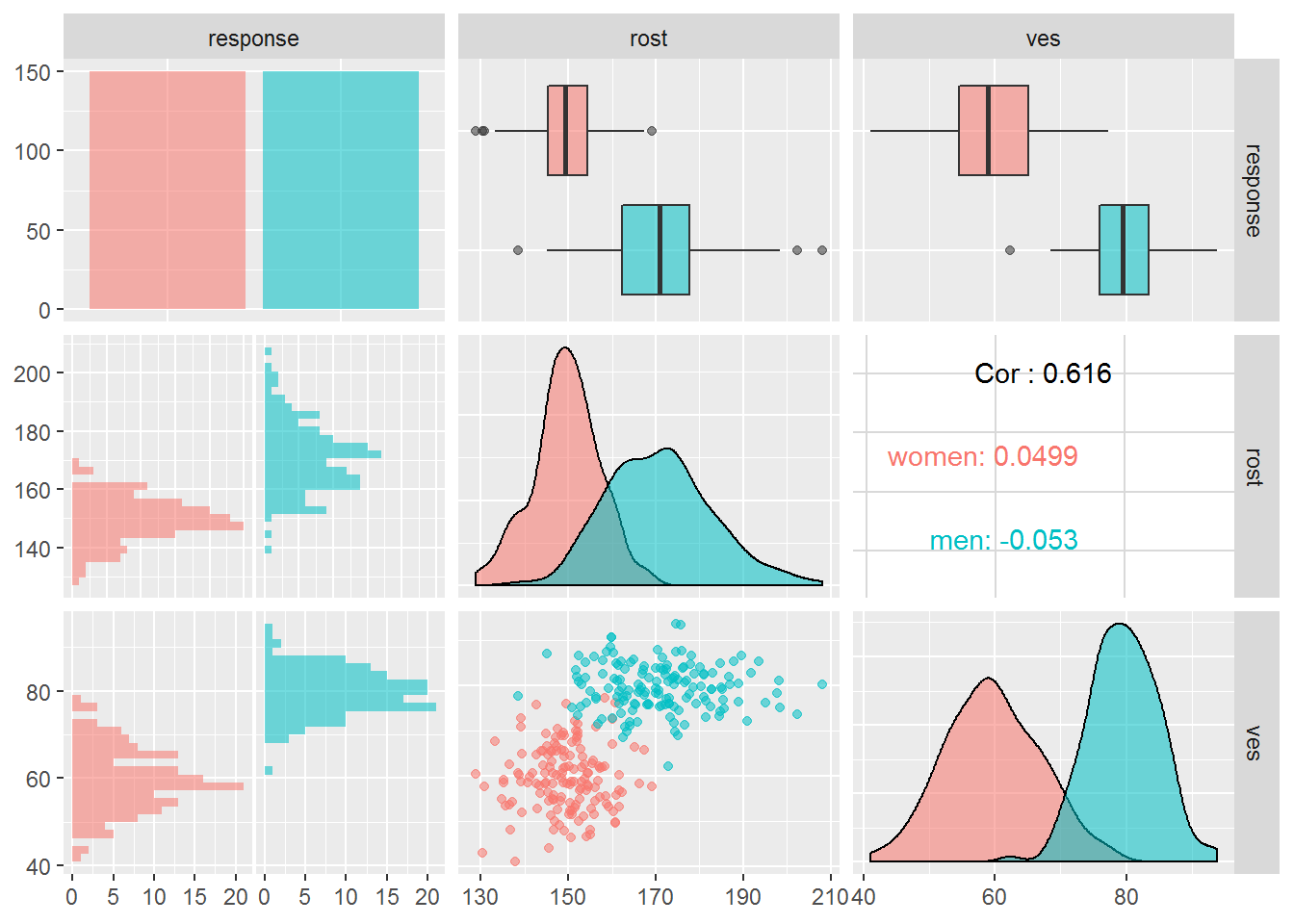
Figure 4.10: Название рисунка
# df['rost'].hist(bins=10) # или отдельно гистограмму
par(mfrow=c(1,1))
hist(df$rost, breaks = 10,col = "cyan")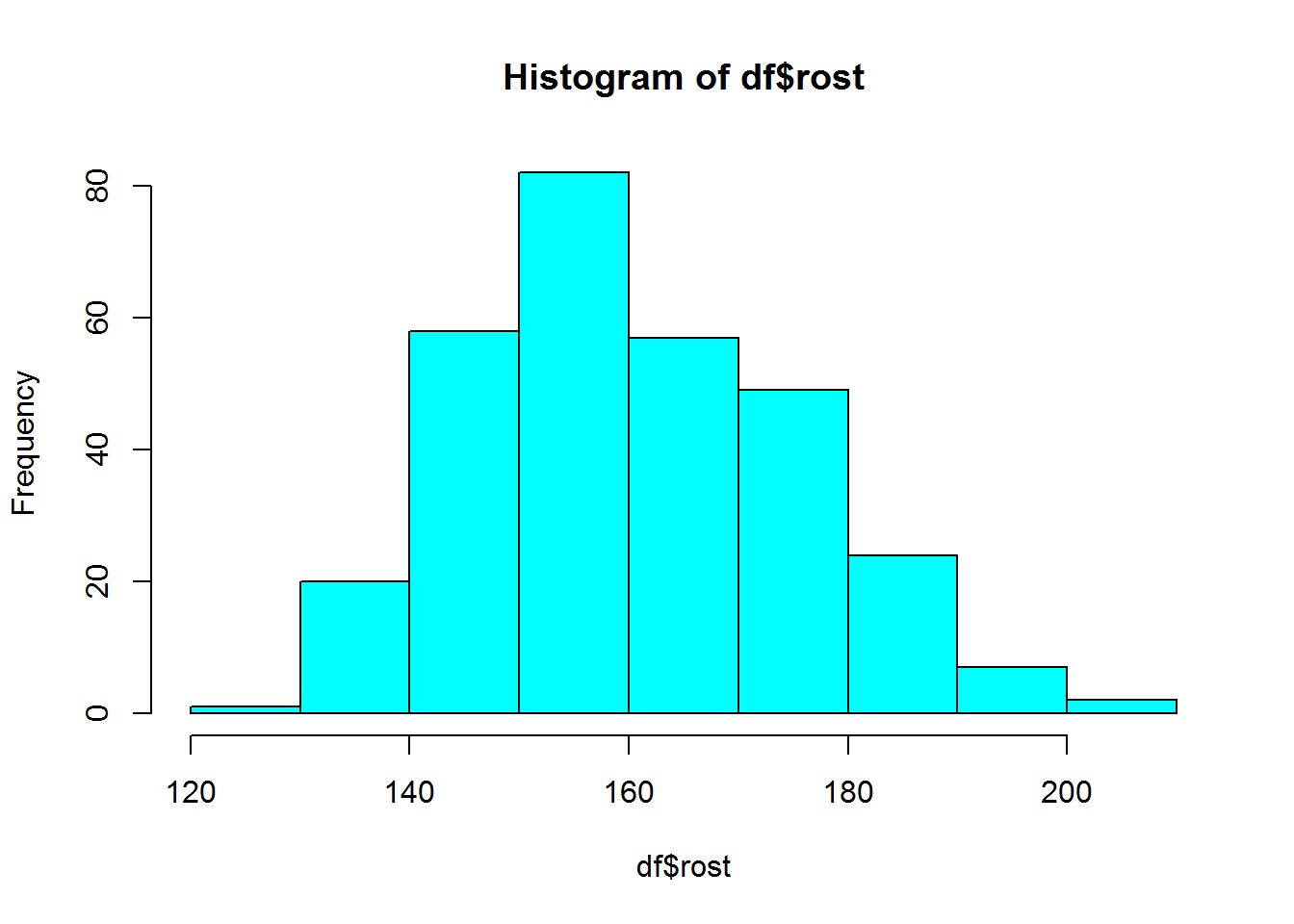
Figure 4.11: Название рисунка
#
# # Метод ближайших соседей
#
# k = 3 # Количество соседей
k = 3
# X = np.array(df[['rost','ves']])
# y = np.array(df['response'])
# y = y.astype('int') # изменение типа данных на числовой
X <- df[,-1]
y <- df[,1]
y <- as.integer(y)
# yc=np.array(['women' for i in range(len(y))])
# yc[y == 1] = 'men'
yc <- factor(y)
levels(yc) <- c("women","men")
# scatter_matrix(df[['rost','ves']], alpha=0.8, figsize=(10, 10),c=y, cmap=ListedColormap(['#0000FF','#FF0000'])) # можно строить диаграммы рассеяния c разными цветами классов
pairs(df[,c('rost','ves')],col=df$response) 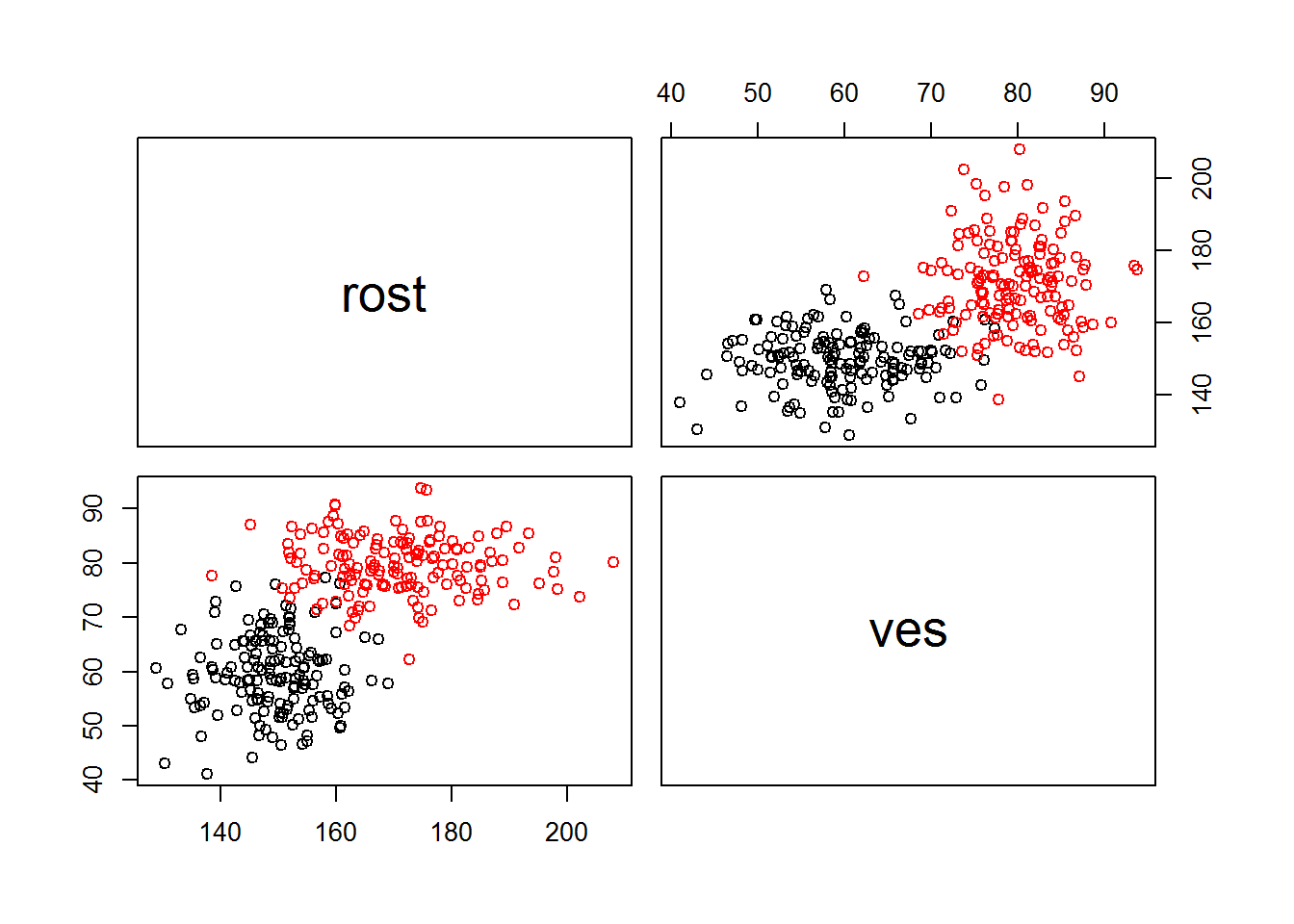
Figure 4.12: Название рисунка
#
# # Цветовые карты
# cmap_light = ListedColormap(['#FFAAAA', '#AAFFAA'])
# cmap_bold = ListedColormap(['#FF0000', '#00FF00'])
#
# distances = []
# targets = []
# ctarget = []
# x_test=np.array([ 158, 43])
distances <- data.frame()
targets <- vector()
ctarget <- vector()
x_test=c(158,43)
#
# for i in range(len(X)):
# # вычисляем расстояние от текущего до всех
# temp_distance = np.sqrt(np.sum(np.square(x_test - X[i, :])))
# distances.append([temp_distance, i]) # сохраняем в массив с запоминанаием номера
for (i in 1:nrow(X))
{
temp_distance = sqrt(sum((x_test-X[i,])^2))
#print(temp_distance)
distances <- rbind(distances,c(temp_distance,i))
#distances <- append(distances,temp_distance)
}
#
# distances = sorted(distances) # сортируем
distances <- distances[order(distances[,1]),]
# # Выбираем классы соседей
# for i in range(k):
# ind = distances[i][1]
# ctarget.append(yc[ind])
# targets.append(y[ind])
#
for (i in 1:k) {
ind = distances[i,2]
print(ind)
targets <- append(targets,y[ind])
print(yc[ind])
ctarget <- append(ctarget,as.character(yc[1]))
}## [1] 24
## [1] women
## Levels: women men
## [1] 2
## [1] women
## Levels: women men
## [1] 111
## [1] women
## Levels: women men#
# Counter(targets).most_common(1)[0][0] # определяем номер класса по частоте вхождения
#
# Counter(ctarget).most_common(1)[0][0] # определяем название класса по частоте вхождения
sort(unique(targets))[which.max(table(targets))] ## [1] 1sort(unique(ctarget))[which.max(table(ctarget))] ## [1] "women"#
# # построим область решения
# h = .5 # Шаг сетки
# x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
# y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
# xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
# np.arange(y_min, y_max, h))
#
# # Возьмём классификатор из библиотеки sklearn
# clf = neighbors.KNeighborsClassifier(k, weights='uniform')
# clf.fit(X, y) # обучаем модель на наших данных
#
# Z = clf.predict(np.c_[xx.ravel(), yy.ravel()]) # смотрим ответы на сетке
# Z = Z.reshape(xx.shape)
#
# # Строим область решения
# plt.figure()
# plt.pcolormesh(xx, yy, Z, cmap=cmap_light)
#
# # добавляем обучающие данные
# plt.scatter(X[:, 0], X[:, 1], c=y, cmap=cmap_bold)
# plt.xlim(xx.min(), xx.max())
# plt.ylim(yy.min(), yy.max())
# plt.title("2-Class classification (k = %i)" % (k))
# plt.show()
#
# # Смотрим вероятность правильного ответа
# np.mean(clf.predict(X)==y)
library(class)
Z <- knn(train = X, test = X, cl = y, k = k)
Z ## [1] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [36] 1 1 1 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [71] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 1 1
## [106] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 1 1 1 1 2 1 1 1 1 1 1 1 1 2 1 1
## [141] 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [176] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [211] 2 2 2 2 2 2 2 2 2 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [246] 2 2 2 2 2 2 1 2 2 2 2 2 2 2 2 2 2 2 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [281] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## Levels: 1 2y## [1] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [36] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [71] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [106] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [141] 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [176] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [211] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [246] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [281] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2library("gmodels")
CrossTable(x = y, y = Z, prop.chisq=FALSE)##
##
## Cell Contents
## |-------------------------|
## | N |
## | N / Row Total |
## | N / Col Total |
## | N / Table Total |
## |-------------------------|
##
##
## Total Observations in Table: 300
##
##
## | Z
## y | 1 | 2 | Row Total |
## -------------|-----------|-----------|-----------|
## 1 | 144 | 6 | 150 |
## | 0.960 | 0.040 | 0.500 |
## | 0.980 | 0.039 | |
## | 0.480 | 0.020 | |
## -------------|-----------|-----------|-----------|
## 2 | 3 | 147 | 150 |
## | 0.020 | 0.980 | 0.500 |
## | 0.020 | 0.961 | |
## | 0.010 | 0.490 | |
## -------------|-----------|-----------|-----------|
## Column Total | 147 | 153 | 300 |
## | 0.490 | 0.510 | |
## -------------|-----------|-----------|-----------|
##
## # Create a dataframe to simplify charting
plot.df = data.frame(X, predicted = Z)
# Use ggplot
# First use Convex hull to determine boundary points of each cluster
plot.df1 = data.frame(x = plot.df$rost,
y = plot.df$ves,
predicted = plot.df$predicted)
find_hull = function(df) df[chull(df$x, df$y), ]
boundary = ddply(plot.df1, .variables = "predicted", .fun = find_hull)
g <- ggplot(plot.df, aes(rost, ves, color = predicted, fill = predicted)) +
geom_point(size = 5) +
geom_polygon(data = boundary, aes(x,y), alpha = 0.5)
print(g)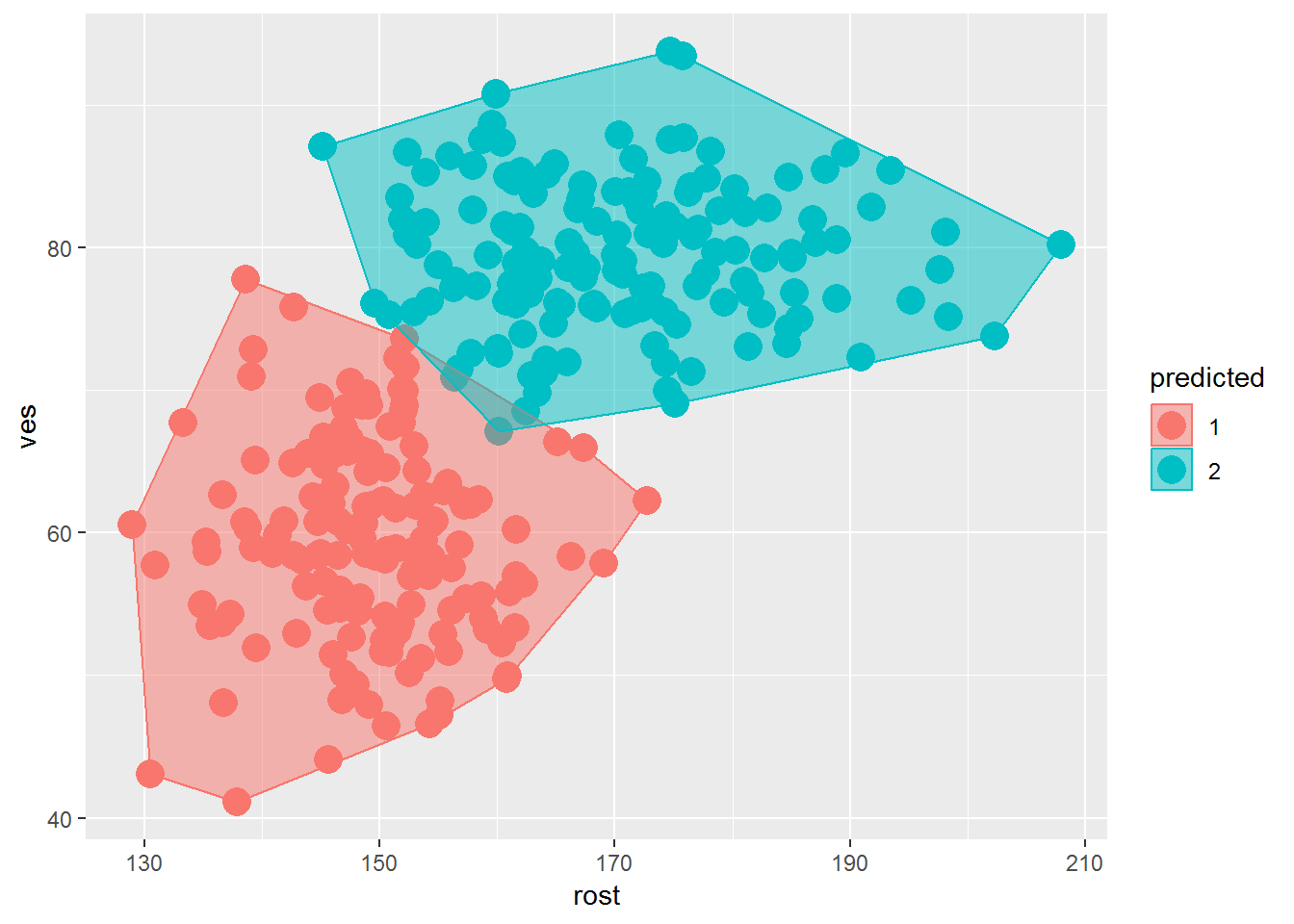
Figure 4.13: Название рисунка
Список использованных источников
“CRISP-Dm.” 2012. December 2012. http://www.machinelearning.ru/wiki/index.php?title=CRISP-DM\&oldid=25814.