3 Paralelización con R en el Cluster (parallel)
3.1 Conexión Remota al Clúster (SSH )
3.1.1 Windows
- PuttyGen.exe (generación clave pública y privada)
- Putty.exe (conexión ssh al cluster)
El paquete completo de Putty puede descargarse de aquí https://www.chiark.greenend.org.uk/~sgtatham/putty/latest.html
3.1.2 Linux-Mac OS.
Para equipos linux y Mac OS la comunicación a través de SSH se puede efectuar directamente desde la terminal
ssh usuario@login.hpc.cedia.org.ecTanto para Windows como para Linux-Mac OS la conexión al clúster CEDIA se establecerá de esta manera:
así la comunicación se efectúa por línea de comandos (terminal). Ahora vamos a generar nuestra contraseña, la cual usaremos para acceder a RStudio en el cluster:
$passwd3.2 Rstudio Server como interfaz en HPC
La siguiente tabla ha sido adaptada de https://support.rstudio.com/hc/en-us/articles/236226087-Scaling-R-and-RStudio
| Caso de uso | Problema | Solución | Tecnología |
|---|---|---|---|
| Escalar varios usuarios R | Flujos de trabajo regulares para un equipo. Cargar datos desde archivos o warehouses | Plataforma para soporte a varias sesiones interactivas R | RStudio Server, RStudio Server Pro + Load Balancer |
| Escalar para HPC | Tareas de paralelización: bootstrapping, cross validation, scoring, ajustes de modelos en sets independientes | Desarrollo de código en sesiones R interactivas en RStudio. Someter jobs en procesos esclavos R. R debe estar instalado en todos los nodos. | Local: parallel, Rmpi, snow, Rcpp parallel; Cluster: LSF, SLURM, Torque, Docker Swarm, batchtools |
| Escalar para Big Data | Big data, rutinas caja negra que requieren ajustar un modelo en un dominio completo. Los datos no caben en un solo computador. | El trabajo pesado se deja a un motor de cómputo diferente. La sintaxis de R se usa para construir pipelines, y analizar resultados. | Hadoop, Spark, Tensorflow, Oracle BDA, Microsoft R Server, Aster, H20.ai |
3.3 Conexión remota a RStudio (Interfaz gráfica)
En sesiones pasadas ya se ha efectuado la generación de claves públicas y privadas desde nuestros equipos locales para posibilitar la conexión al cluster de CEDIA.
La conexión a través de interfaz gráfica a RStudio se realiza ingresando a la dirección https://rstudio.cedia.edu.ec donde se solicitan las credenciales de usuario.
3.3.0.1 Sobre los repositorios CRAN
La lista completa de repositios CRAN se encuentra en https://cran.r-project.org/mirrors.html
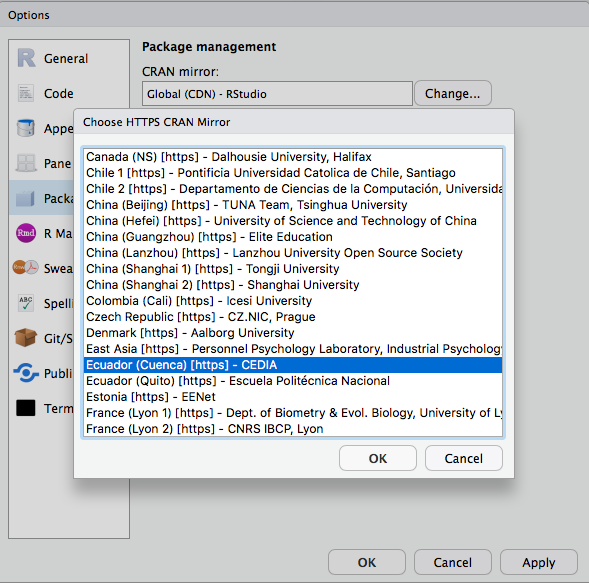
CEDIA cuenta con un respositorio CRAN en uno de los nodos del cluster. Agregaremos su repositorio en la configuración de Rstudio de manera que la descarga de paquetes sea más rápida.
En el menú de Rstudio -> Tools -> Global Options -> Packages , crear un nuevo enlace a repositorio:
3.3.1 Transfrencia de archivos a través de RStudio
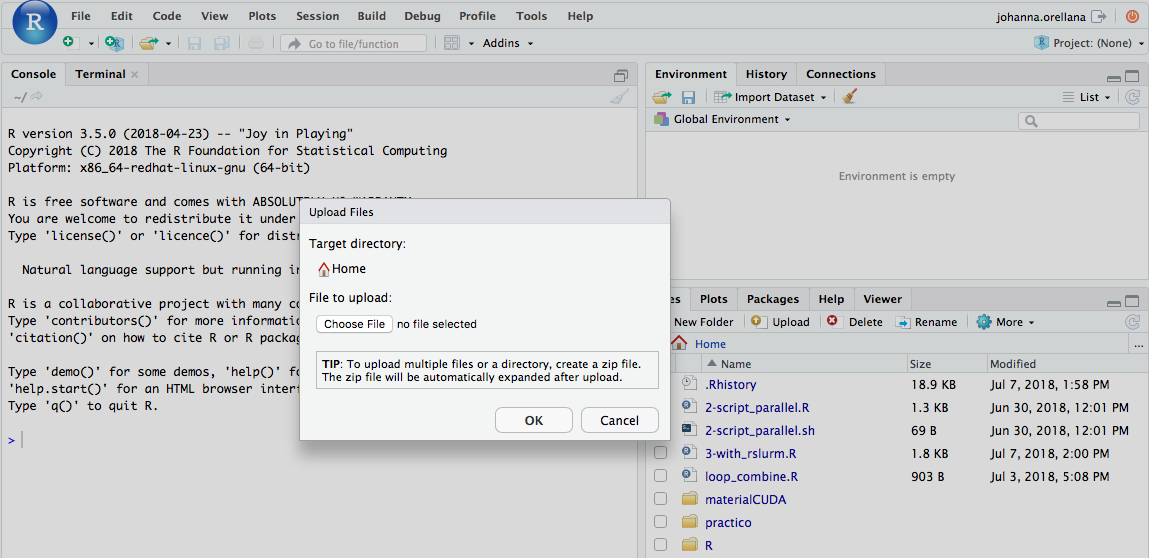
Para subir un archivo desde su computador local a su carpeta /home/usuario en el clúster utilice la opción upload del menú “Files” en el panel inferior derecho de RStudio.
Para descargar un archivo desde su carpeta /home/usuario en el clúster hacia su computador local utilice la opción exportar del menú “Files” en el panel inferior derecho de RStudio. Es importante notar que esta opción transfiere el o los archivos originales sin crear una copia adicional (equivalente a mover un archivo)
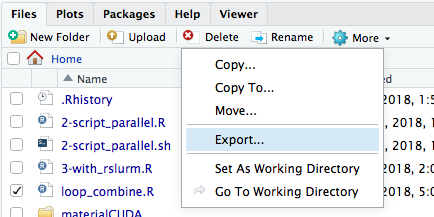
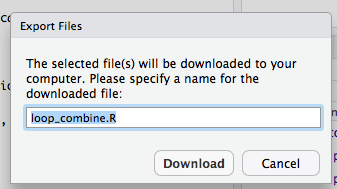
3.3.2 Transfrencia de archivos a través de Interfaz Gráfica
3.3.2.1 Windows
Se recomienda el uso de WinSCP el cual incorpora el protocolo SCP (Linux y Mac OS lo tienen por defecto para uso por terminal directamente).
Este es el enlace para descarga: https://winscp.net/eng/download.php
Y aquí un tutorial paso a paso para la instalación y configuración de nuevo sitio. Revisar la sección “Transfer files from Windows”: https://research.computing.yale.edu/support/hpc/user-guide/transfer-files-or-cluster
Una vez instalado el programa, estos son los pasos a seguir para configurar nuestra cuenta.
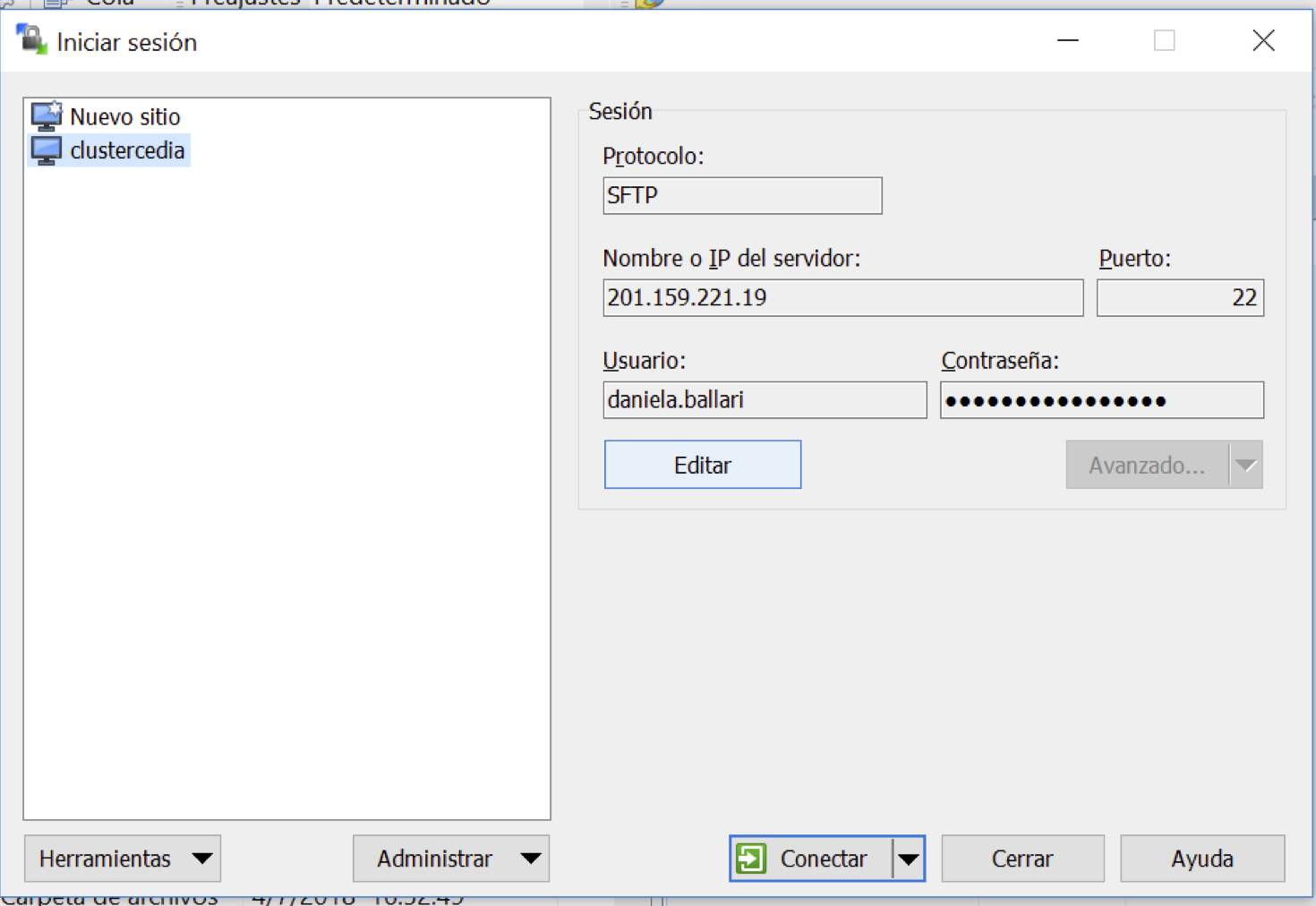
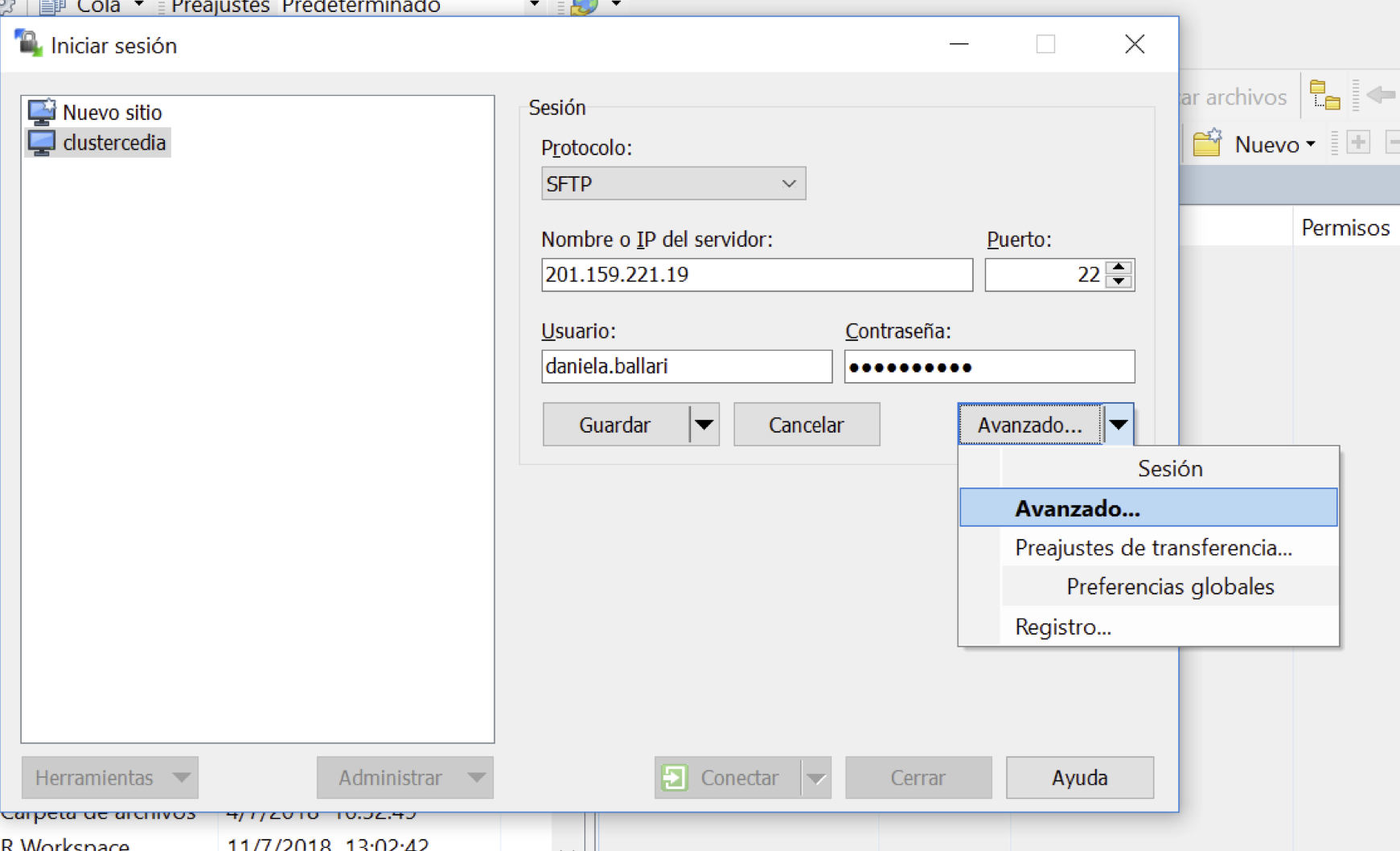
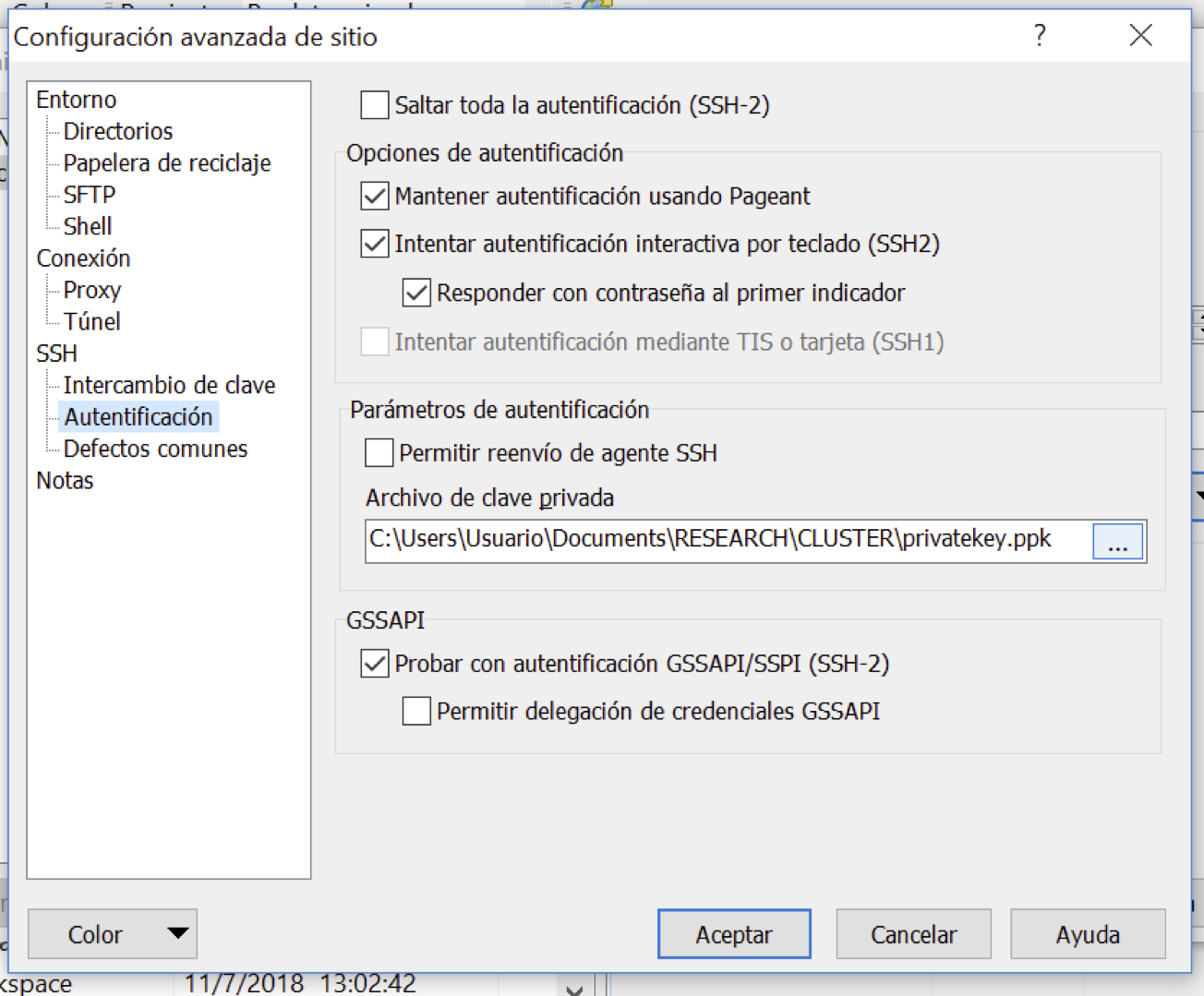
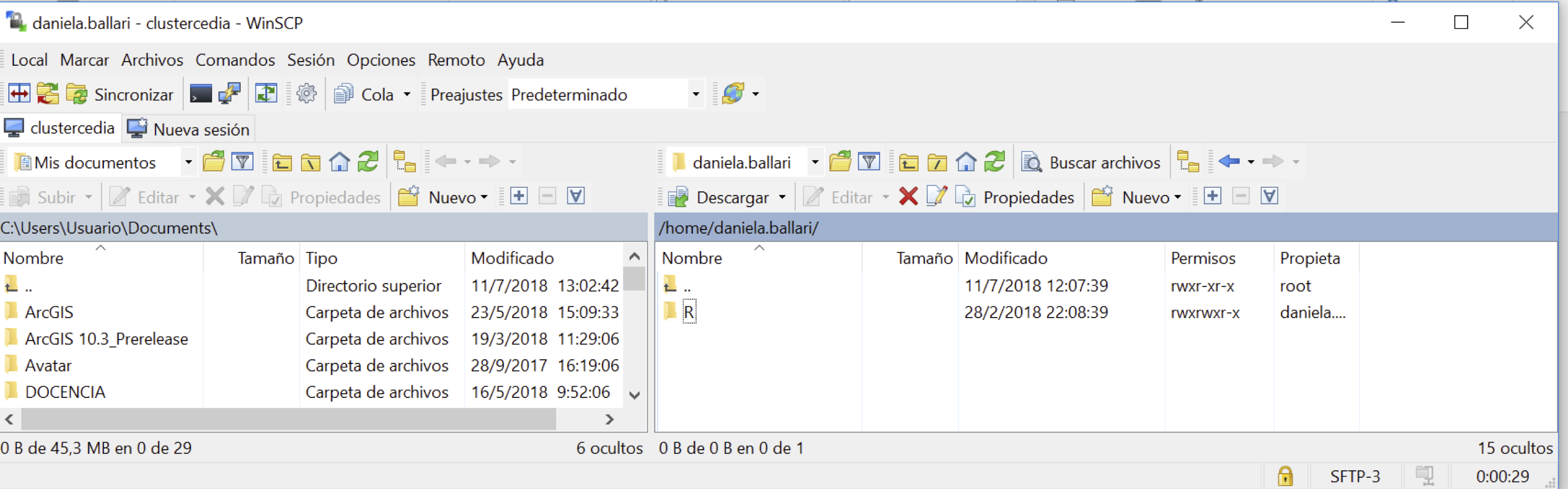
3.3.2.2 Linux-Mac OS
La herammienta Cyberduck es de libre distribución, permite para nuestro caso efectuar transferencia de archivos desde y hacia el clúster de manera segura usando el puerto SFTP. Se puede descargar aquí https://cyberduck.io
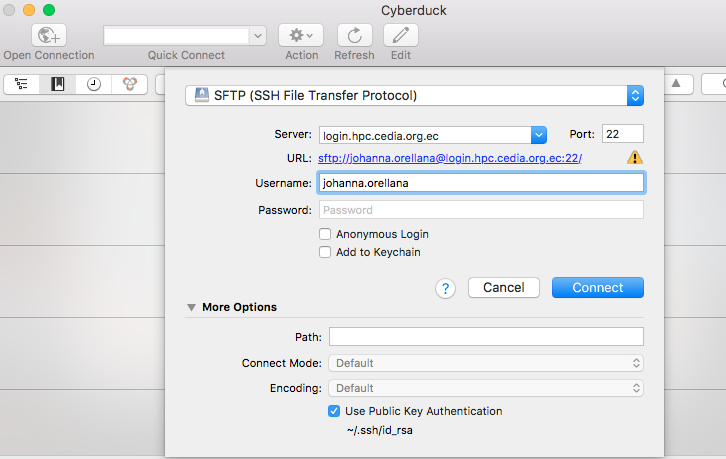
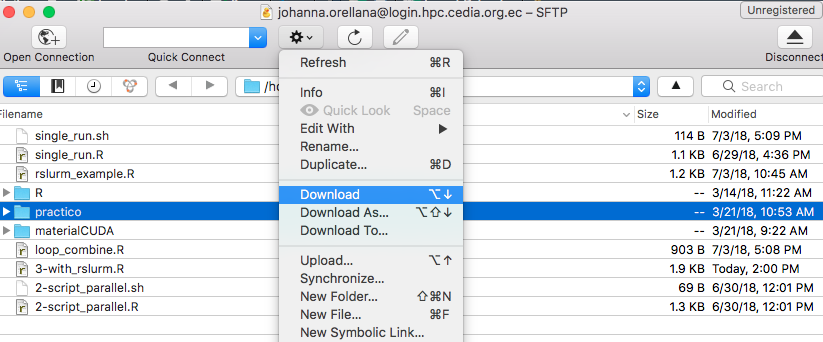
3.3.2.3 Transferencia de archivos (Terminal Linux-Mac OS)
A través de la interfaz de terminal es posible usar scp para trasferencia de archivos. La sintaxis general es:
scp source destinationUn ejemplo para transferir archivos del computador local al clúster:
scp archivo.png nombre.apellido@login.hpc.cedia.org.ec:/home/nombre.apellidoy desde el clúster al computador local:
scp nombre.apellido@login.hpc.cedia.org.ec:/home/nombre.apellido/archivo.png /Users/miUsuario/DesktopPara transferir directorios completos use la opción scp -r.
3.4 mclapply
Ahora que todos estamos trabajando con Linux como sistema operativo base podemos hacer uso de esta función.
- Fácil de implementar; clona el enviroment automáticamente
- Rápido
- Puede usar solamente los procesadores de un computador
- Usa fork y por lo tanto no funciona en MS Windows
#Default options
mclapply(X, FUN, ...,
mc.preschedule = TRUE, mc.set.seed = TRUE,
mc.silent = FALSE, mc.cores = getOption("mc.cores", 2L),
mc.cleanup = TRUE, mc.allow.recursive = TRUE)3.4.1 mclapply vs. lapply
Para los ejemplos a continuación usaremos el archivo dataset.csv. Este archivo contiene 250000 muestras. En este enlace puede descargar tanto el archivo .csv como el archivo .R a partir del cual fue generado.
Ejemplo usando lapply
###### using lapply
time_lapply_kmeans<-system.time({
data <- read.csv('dataset.csv')
parallel.function <- function(i) {
kmeans( data, centers=4, nstart=i )
}
results <- lapply( c(25, 25, 25, 25), FUN=parallel.function )
temp.vector <- sapply( results, function(result) { result$tot.withinss } )
result <- results[[which.min(temp.vector)]]
print(result$tot.withinss)
})## [1] 133747.2Ejemplo usando mclapply (2 cores)
### using mclapply
library(parallel)
time_mclapply_kmeans<-system.time({
data <- read.csv('dataset.csv')
parallel.function <- function(i) {
kmeans( data, centers=4, nstart=i )
}
results <- mclapply( c(25, 25, 25, 25), FUN=parallel.function, mc.cores = 2L )
temp.vector <- sapply( results, function(result) { result$tot.withinss } )
result <- results[[which.min(temp.vector)]]
print(result$tot.withinss)
})## [1] 133747.2Tiempos de respuesta
time_lapply_kmeans## user system elapsed
## 28.564 0.383 29.041time_mclapply_kmeans## user system elapsed
## 16.313 0.527 17.3583.4.2 Usando multicore en mclapply
El argumento mc.cores permite modificar el número de cores (procesadores) que se utilizan en el proceso de paralelización. Exploremos el rendimiento de nuestro modelo kmeans con diferentes números de procesadores (2,4,8,16). El código abajo indica el argumento mc.cores = 2L, ejecutemos el script modificando a las otras posibilidades 4, 8 y 16.
Usemos la sentencia rm(list= ls()[! ls() %in% c("mc_2","mc_4",..)]) al inicio de cada corrida para mantener los valores resultado de system.time previos y al final visualizarlos juntos mandando a imprimir las variables mc_2, mc_4, etc.
rm(list=ls(all=TRUE))
gc()## used (Mb) gc trigger (Mb) max used (Mb)
## Ncells 1561134 83.4 3205452 171.2 3205452 171.2
## Vcells 3015920 23.1 11874215 90.6 14756876 112.6require(parallel)
time_2cores<-system.time({
data <- read.csv('dataset.csv')
parallel.function <- function(i) {
kmeans( data, centers=4, nstart=i )
}
RNGkind("L'Ecuyer-CMRG")
set.seed(1231231)
mc.reset.stream()
results <- mclapply( rep(5,8), FUN=parallel.function, mc.cores = 2L)
temp.vector <- sapply( results, function(result) { result$tot.withinss } )
result <- results[[which.min(temp.vector)]]
print(result$tot.withinss)
})## [1] 133747.2Los resultados ejecutados en el cluster usando mclapply para kmeans:
| Cores | user | system | elapsed |
|---|---|---|---|
| 2 | 17.694 | 0.802 | 9.644 |
| 4 | 18.819 | 1.695 | 5.757 |
| 8 | 20.604 | 2.489 | 3.623 |
| 16 | 20.948 | 2.679 | 3.818 |
Conclusión :para este ejemplo, únicamente 8 cores es el óptimo pues nuestro vector tiene 8 elementos (usar más cores puede incluso afectar el tiempo de cómputo por la comunicación).
3.4.3 Comparando parLapply (fork y socket)
Vamos a plantear el problema ahora usando las funciones de make cluster y parLapply tanto usando PSOCK como FORK. PSOCK es una versión mejorada de SOCK. El código abajo está elaborado para 8 procesadores. Genera nuevamente este código con 2 procesadores para cuantificar la diferencia de tiempos.
rm(list=ls(all=TRUE))
gc()## used (Mb) gc trigger (Mb) max used (Mb)
## Ncells 1561449 83.4 3205452 171.2 3205452 171.2
## Vcells 3017848 23.1 9499372 72.5 14756876 112.6require(parallel)
makeFORKCluster8<-system.time({
data <- read.csv( 'dataset.csv' )
parallel.function <- function(i) {
kmeans( data, centers=4, nstart=i )
}
cl <- makeForkCluster(8L) # Equivalente makeCluster(4L, type= "FORK")
clusterSetRNGStream(cl, 123)
#clusterExport(cl, c('data')) -> Fork o requiere exportación de datos
results <- parLapply( cl, rep(5,8), fun=parallel.function )
temp.vector <- sapply( results, function(result) { result$tot.withinss } )
result <- results[[which.min(temp.vector)]]
print(result$tot.withinss)
stopCluster(cl)
})## [1] 133747.2######## USANDO PSOCK
rm(list= ls()[! ls() %in% c("makeFORKCluster8")])
gc()## used (Mb) gc trigger (Mb) max used (Mb)
## Ncells 1563441 83.5 3205452 171.2 3205452 171.2
## Vcells 3021600 23.1 9499372 72.5 14756876 112.6require(parallel)
makePSOCKcluster8<-system.time({
data <- read.csv( 'dataset.csv' )
parallel.function <- function(i) {
kmeans( data, centers=4, nstart=i )
}
cl <- makePSOCKcluster(8L) # Equivalent makeCluster(4L, type= "FORK")
clusterSetRNGStream(cl, 123)
clusterExport(cl, c('data'))
results <- parLapply( cl, rep(5,8), fun=parallel.function )
temp.vector <- sapply( results, function(result) { result$tot.withinss } )
result <- results[[which.min(temp.vector)]]
print(result$tot.withinss)
stopCluster(cl)
})## [1] 133747.2makeFORKCluster8## user system elapsed
## 0.995 0.047 18.745makePSOCKcluster8## user system elapsed
## 1.062 0.050 18.889Nota: Seguramente los resultados que acabas de obtener son mucho mejores.
Incorporamos en la tabla los resultados de mclapply obtenidos previamente para propósitos de comparación:
| Función | Cores | user | system | elapsed |
|---|---|---|---|---|
| makePSOCKcluster | 2 | 0.773 | 0.072 | 9.997 |
| makeFORKCluster | 2 | 0.787 | 0.060 | 9.954 |
| mclapply | 2 | 17.694 | 0.802 | 9.644 |
| makePSOCKcluster | 8 | 0.864 | 0.194 | 6.066 |
| makeFORKCluster | 8 | 10.781 | 0.094 | 3.899 |
| mclapply | 8 | 20.604 | 2.489 | 3.623 |
Algunas cosas a recordar: + mclapply (usa FORK) (clona la sesión R a los procesos hijos) + parlapply requiere creación de cluster (clusterPSOCK - clusterFORK…) + parlapply + clusterFORK es equivalente funcionalmente a mclapply + parlapply paraleliza en múltiples computadas, crea un cluster de procesos que se comunican via TCP/IP.
3.4.4 Números Aleatorios y Experimentos reproducibles
Cuando iniciamos una nueva sesión en R o incluso dentro de la misma sesión el dato referencial para generar valores aleatorios cambia. Es decir, no sería posible replicar experimentos que dependen de valores aleatorios. Por ejemplo:
Cuando se usa mclapply la designación del método para generación de valores aleatorios se hace mediante:
RNGkind("L'Ecuyer-CMRG")Por defecto dentro de la función mclapply, el parámetro mc.set.seed = TRUE. Sin embargo para garantizar la generación de valores aleatorios diferentes para cada core (nodo) se debe seleccionar RNGkind("L'Ecuyer-CMRG").
RNGkind("L'Ecuyer-CMRG")
set.seed(7777442)
mc.reset.stream()
unlist(mclapply(1:2, function(i) rnorm(1)))## [1] -2.0043112 0.9315424Que sucede si volvemos a ejecutar la sentencia unlist(mclapply(1:2, function(i) rnorm(1))) -> la función mclapply no modifica la secuencia del valor aleatorio inicial (set.seed) sobre la segunda llamada mclapply.
3.5 Balance de Carga
3.5.1 clusterApplyLB
clusterApplyLB es una versión de clusterApply que maneja balance de carga.
Cuando la longitud n de x no es mayor que el número de nodos p, entonces el job se envía a n nodos. Caso contratio, los primeros p jobs se envían a los p nodos. Cuando el primer job se ha completado, el próximo job se envía al nodo que acaba de ser liberado y así hasta que se completan todos los jobs.
Usar clusterApplyLB puede generar una mejor utilización del cluster que clusterApply, no obstante la necesidad de comunicación puede reducir el rendimiento.
Por lo tanto el nodo que ejecuta un job particular no es siempre el mismo, entonces las simulaciones que asignan RNG streams a los nodos no serán reproducibles.
Un ejemplo sencillo para entender como se distribuye el tiempo usando balance de carga:
cl <- parallel::makeCluster(2)
system.time(
parallel::parLapply(cl, 1:4, function(y) { # Sin balance de carga
if (y == 1) {
Sys.sleep(3)
} else {
Sys.sleep(0.5)
}}))## user system elapsed
## 0.001 0.000 3.504system.time(
parallel::parLapplyLB(cl, 1:4, function(y) { # Con balance de carga parLapply
if (y == 1) {
Sys.sleep(3)
} else {
Sys.sleep(0.5)
}}))## user system elapsed
## 0.001 0.000 3.509system.time(
parallel::clusterApplyLB(cl, 1:4, function(y) { #Con balance de carga clusterApplyLB
if (y == 1) {
Sys.sleep(3)
} else {
Sys.sleep(0.5)
}}))## user system elapsed
## 0.002 0.001 3.002parallel::stopCluster(cl)3.5.2 Cuándo usar balance de carga?
Cuando aplicar la función definida a diferentes elementos de X toma un tiempo de procesamiento bastante variable y, en tanto la función sea determinística o no se requieran resultados reproducibles.
Analicemos la influence de balance de carga en el siguiente ejemplo.
- parLApply : asigna estátimente los chunks de cómputo (procesador no se comunica con master hasta terminar la tarea)
- clusterApply: asigna un chunk y espera por la comunicación de los procesadores para enviar el siguiente.
- clusterApplyLB: recicla los nodos, es decir cada vez que un nodo está libre le asigna una nueva tarea. La asignación de los chunks de cómputo es dinámica.
require(snow)## Loading required package: snow##
## Attaching package: 'snow'## The following objects are masked from 'package:parallel':
##
## clusterApply, clusterApplyLB, clusterCall, clusterEvalQ,
## clusterExport, clusterMap, clusterSplit, makeCluster,
## parApply, parCapply, parLapply, parRapply, parSapply,
## splitIndices, stopClusterset.seed(1237)
sleep = sample(1:10,10)
spec = 4
cl = makeCluster(spec, type="SOCK")
#Los chunks
sleep## [1] 8 6 2 10 7 9 4 3 5 1clusterSplit(cl, sleep)## [[1]]
## [1] 8 6 2
##
## [[2]]
## [1] 10 7
##
## [[3]]
## [1] 9 4
##
## [[4]]
## [1] 3 5 1st = snow.time(clusterApply(cl, sleep, Sys.sleep))
stLB = snow.time(clusterApplyLB(cl, sleep, Sys.sleep))
stPL = snow.time(parLapply(cl, sleep, Sys.sleep))
stPL = snow.time(parLapply(cl, sleep, Sys.sleep))
stopCluster(cl)
par(mfrow=c(3,1),mar=rep(2,4))
plot(st, title="clusterApply")
plot(stLB, title="clusterApplyLB")
plot(stPL, title="parLapply")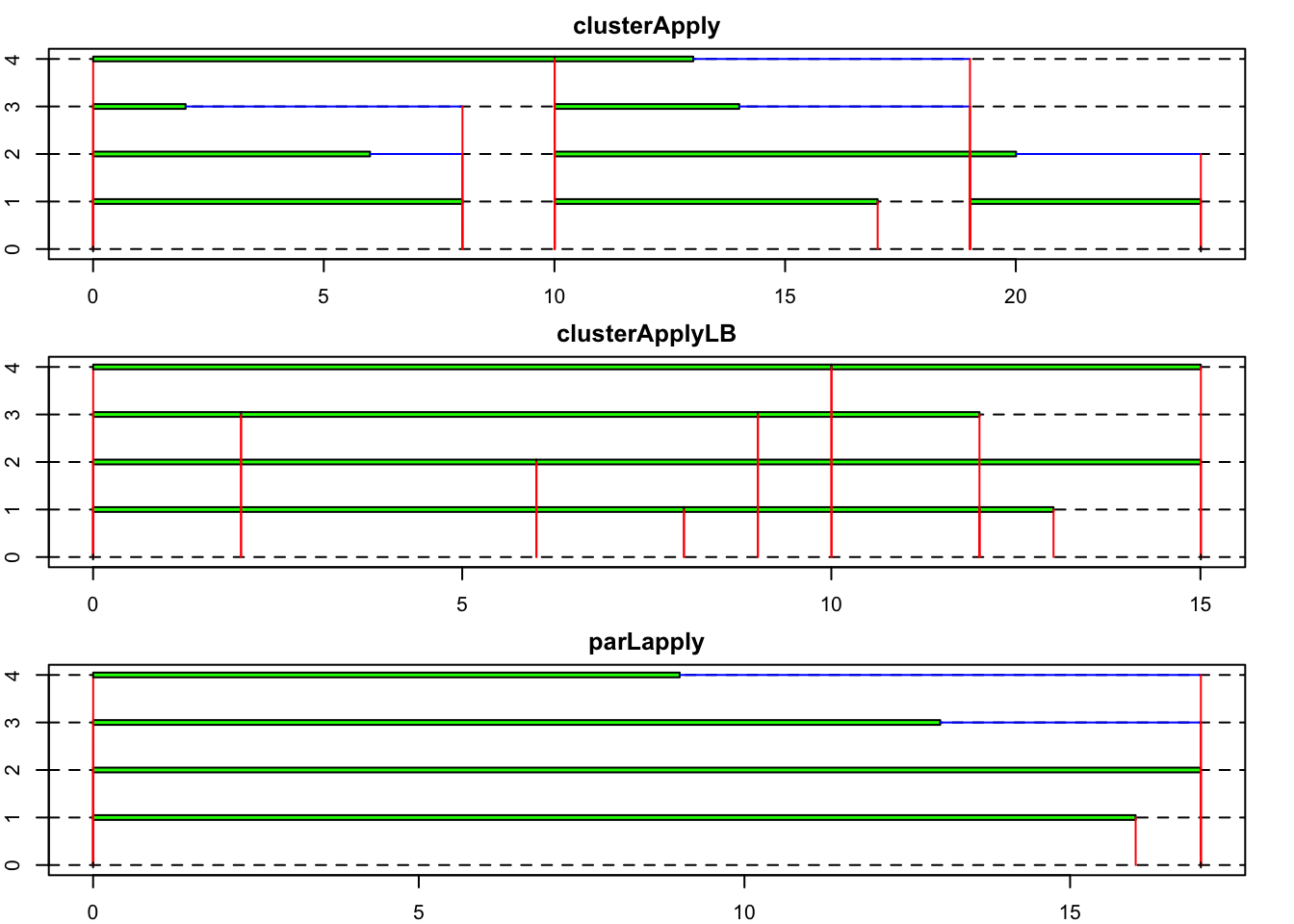
Un ejemplo para probar el balance de carga. ¿En cuánto hemos reducido el tiempo de cómputo?.
rm(list=ls(all=TRUE))
gc()
require(parallel)
data <- read.csv( 'dataset.csv' )
parallel.function <- function(i) {
kmeans( data, centers=4, nstart=i )
}
cl <- parallel::makeForkCluster(4L) # Equivalente makeCluster(4L, type= "FORK")
clusterSetRNGStream(cl, 123)
time_parLapply<-system.time({
results <- parallel::parLapply( cl, c(1,1,5,5,2,2,1,1), fun=parallel.function )
temp.vector <- sapply( results, function(result) { result$tot.withinss } )
result <- results[[which.min(temp.vector)]]
print(result$tot.withinss)
})
time_parLapplyLB<-system.time({
results <- parLapplyLB( cl, c(1,1,5,5,2,2,1,1), fun=parallel.function )
temp.vector <- sapply( results, function(result) { result$tot.withinss } )
result <- results[[which.min(temp.vector)]]
print(result$tot.withinss)
})
stopCluster(cl)
time_parLapply
time_parLapplyLB