Chapter 2 Módulo II
De acordo com o dicionário Oxford languages, script é um “conjunto de instruções para que uma função seja executada em determinado aplicativo”. Ou seja, trabalhar com scripts é uma forma de facilitar a execução das atividades propostas no R.
Script da aula do Módulo II abaixo. Download Aula2.R
2.1 Guardando seus códigos em um script
No RStudio, você pode acrescentar um pequeno painel, que facilitará ainda mais o seu trabalho. Trata-se do source pane, um painel onde você pode criar scripts para armazenar várias linhas de código, de modo a excutá-las de uma única vez ou linha-a-linha. Caso queira guardar as contas e comparações lógicas que fez anteriormente em um arquivo de modo a poder executá-las posteriormente, você poderá fazê-lo por meio de um script. Na janela de script, você consegue manter uma trilha de todos os comandos utilizados.
Isto é importante, pois otimiza o uso do R ao permitir rodar os mesmos códigos para diferentes arquivos e diferentes ocasiões, em vez de termos que refazer toda a programação a cada vez que mudarem os dados de entrada. Você pode abrir várias janelas de script, uma ao lado da outra e navegar entre elas, como se faz com as abas de um navegador. Neste curso, todas as aulas e exercícios utilizarão os arquivos de script.
Para acrescentar a janela de script (source pane), basta criar um novo arquivo, clicando em ![]() , logo abaixo de File, e em seguida em
, logo abaixo de File, e em seguida em  . Na janela do RStudio deve ficar conforme a figura a seguir.
. Na janela do RStudio deve ficar conforme a figura a seguir.

Vamos acrescentar todas as linhas de códigos que executamos anteriormente ao nosso script recém criado.

DICA: Você pode inserir comentários em seus scripts como uma forma de documentar os passos de seu trabalho para refrência futura. No R, os comentários são incluídos com o caractere #.
Para salvar o arquivo de script, basta executar Ctrl+S ou clicar no disquete único na barra de menus. Esse arquivo será salvo com extensão .R, indicando que se trata de um arquivo com códigos da linguagem R.
DICA: Sugerimos que você crie e salve um ou mais scripts a cada aula do curso. Isso o ajudará a organizar seu material e recuperar conceitos e exemplos importantes durante e após o curso.
Você pode executar todos as linhas de um script ou optar executar apenas algumas poucas linhas ou trechos específicos desse código. Selecione o trecho que deseja executar e pressione Ctrl+Enter. Você verá, no console (janela abaixo do script) seu código sendo executado. Uma alternativa ao Ctrl+Enter é selecionar o trecho ou linha(s) e clicar em ![]() .
.
Na maioria das vezes, nosso código pode levar um tempo considerável para ser executado completamente. Para saber se o interpretador ainda está ocupado executando o seu código, basta olhar para o canto direito do painel Console. Se você ver o símbolo ![]() , o interpretador ainda está ocupado executando a última sequência de comandos. O símbolo
, o interpretador ainda está ocupado executando a última sequência de comandos. O símbolo > também não aprecerá livre no console. Você pode tentar parar a execução clicando no ícone de stop. No entanto, nem sempre será possível interrompê-la.
Tente gerar uma distribuição normal com 100 milhões de entradas:
rnorm(1:100000000)DICA: Pode acontecer ainda de você olhar para o console e não ver o símbolo >, sem que tenha aparecido o ícone de stop no canto direito do painel. Isso geralmente acontece quando esquecemos de fechar algum parenteses, chave ou colchete aberto no início de uma linha de código. Faça um teste:
((1007+1)/8^2O console ficará esperando você completar o código. Você tem duas opções, acrescentar o parênteses faltante (caso seja possível), ou pressionar Esc, consertar o código e executá-lo novamente.
2.1.0.1 Aba Environment
Todos os objetos que forem criados na sua sessão do R/RStudio serão automaticamente armazenados no que chamamos de Global Environment, representado pela aba Environment no RStudio.
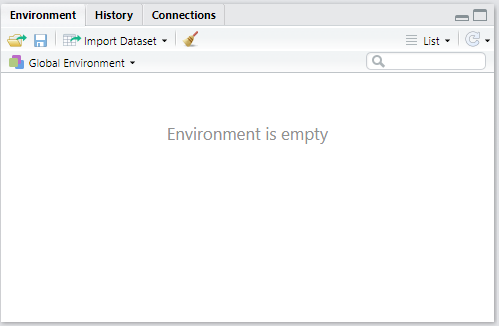
Aba Environment
Note que até o momento, o Environment encontra-se vazio. Isso porque nenhum objeto ou resultados das operações realizadas até o momento foi armazenada.
2.1.0.1.1 Atribuição de objetos no R
Para criar um objeto no R, seja para armazenar um valor específico ou o resultado de uma operação, é necessário utilizar o sinal de atribuição característico do R <-, i.e. a <- b (lê-se a recebe o valor de b). Pode-se utilizar ainda o sentido contrário: b -> a (a recebe o valor de b), ou ainda o sinal de igual =. Essas alternativas são menos convencionais. O consenso é a utilização de a <- b para que não se confunda a criação de objetos com a passagem de parâmetros em argumentos de funções mais a frente.
Para criar um objeto/variável x que contém o valor 2, execute:
# x recebe 2
x <- 2
# para ver o valor de x:
x## [1] 2DICA: O atalho para o operador de atribuição <- é Alt+-.
Note agora que a aba Environment não está mais vazia:

Agora, o exercício é executar algumas operações aritméticas e de comparações lógicas para salvar os objetos:
out1 <- 789/34.5
out2 <- 3^2
out3 <- out1 > out2Observem como ficou o Environment com os objetos criados:

O R é bastante liberal no que se refere a sua política de nomes de objetos. As proibições são apenas as seguintes:
- um nome não pode iniciar com algarismo, ex:
1out <- 2; - um nome não pode conter apenas algarismos, ex:
01 <- 2; - um nome não pode conter caracteres especiais, a não ser
.ou_, ex:out#01 <- 2 - um nome não pode ser idêntico a uma palavra utilizada como token da linguagem R, ex:
TRUE <- 2;for <- 2, etc.
DICA: A linguagem R é case sensitive, ou seja, ela faz distinção entre letras maiúsculas e minúsculas de um nome. Portanto Nome_do_Objeto != nome_do_objeto!
2.1.0.2 Salvando os objetos do Environment
E se você quiser salvar esses objetos criados na sua sessão de R, para continuar trabalhando neles posteriormente? É possível fazê-lo e é simples.
Para salvar todos os objetos do seu Environment, clique em Session e Save Workspace As... no RStudio. Será salvo um arquivo com extensão .RData com o nome e caminho que você especificar.
Uma alternativa também seria:
save.image("C:\\caminho_para_pasta_de_preferencia\\meu_workspace.RData")
# ou
save.image("C:/caminho_para_pasta_de_preferencia/meu_workspace.RData")DICA: A barra invertida \ tem uma função especial no R, ela funciona como um caractere de escape, o quê veremos mais a fundo em sessões futuras. Portanto, para que uma barra invertida deixe de ter sua função especial, precisamos “escapá-la” com outra barra invertida. Por isso, usamos duas barras em endereços do Windows. Uma forma de contornar isso é usar barras normais, como no Linux. Mesmo no Windows, o R saberá que você está especificando um caminho Windows.
Para carregar o arquivo salvo em sessões futuras, você tem novamente duas alternativas. A primeira é clicar em Session e Load Workspace... no RStudio. A segunda é:
load("C:\\caminho_para_pasta_de_preferencia\\meu_workspace.RData")Se você quiser salvar elementos específicos e não todo o environment, você pode fazê-lo da seguinte forma:
save(out1, out2, file="C:\\caminho_para_pasta_de_preferencia\\meu_workspace.RData")Para carregar esses objetos, você também usará a função load().
DICA: Para salvar e carregar um único elemento, como por exemplo um dataset (tabela) que foi trabalhado, mas que deverá ainda ser carregado em uma nova sessão, você pode usar as funções saveRDS() e readRDS().
2.1.1 Estrutura da linguagem R
R pode ser considerado uma linguagem de programação funcional, uma vez que a maioria dos procedimentos e rotinas são realizadas por meio de funções que recebem alguns argumentos como input, executam algumas ações sobre esses argumentos e retornam um output. De modo geral, o uso de funções se dá da seguinte forma:
nome_da_funcao(argumento1 = valor1, argumento2 = valor2, ...)Embora os operadores aritiméticos e lógicos vistos anteriormente não se enquadrem na estrutura funcional descrita acima, acabam por operar internamente como funções.
Como você verá mais à frente, qualquer usuário pode criar uma função no R e não somente utilizar as disponibilizadas pela distribuição default da linguagem.
DICA: Não precisamos utilizar sempre o formato nome_argumento=valor dentro das funções, pois o R é inteligente o suficiente para fazer o matching dos argumentos pela posição em que são passados (nome_da_funcao(valor1, valor2)) ou mesmo pelas letras iniciais do argumento informado.
Nesta etapa serão apresentados alguns exemplos de funções que executam operações matemáticas e que já estão implementadas no base R (distribuição básica do R):
DICA: O RStudio possui a funcionalidade de autocompletar as palvras que você digita com os objetos criados durante sua sessão R ou já existentes na memória. Quando for digitar as funções abaixo, faça um teste digitando as primeiras duas letras de cada função e pressiona TAB.
# raiz quadrada
sqrt(81)## [1] 9# juntando com priorização de operações
sqrt((3*3)^2)## [1] 9## produtório
prod(2,2) # 2x2## [1] 4prod(2,3,5,7,8) # 2x3x5x7x8## [1] 1680## logaritmo
# log de 3 na base e
log(3) # log natural## [1] 1.098612# log de 3 na base 10
log(3,10)## [1] 0.4771213# log3 na base 10
log10(3)## [1] 0.4771213# abs = modulo, |3 - 9|
abs(3-9)## [1] 6# fatorial
# 4 fatorial
factorial(4)## [1] 242.2 Pacotes no R
Um dos motivos do grande sucesso da linguagem R deve-se ao fato de que qualquer usuário pode desenvolver diversas funções, para executar uma ou várias tarefas, e agrupar em determinado ambiente (este procedimento é conhecido como empacotamento ou suíte de funções). Esse conjunto de funções/procedimentos pode ser disponibilizado na forma de um pacote, onde outros usuários poderão instalar e utilizar as funcionalidades disponíveis nele. Após cumprir uma série de requisitos, esses pacotes geralmente são disponibilizados no Comprehensive R Archive Network (CRAN).
O CRAN possui uma política bastante séria de revisão de pacotes. Para que um pacote R possa compor o repositório do CRAN, deve atender a uma série de exigências e ser aprovado em diversos testes focados essencialmente nos seguintes fatores: segurança para o usuário; funcionamento sem erros em pelo menos dois sistemas operacionais; documentação densa (inclusive com citações bibliográficas) sobre as funcionalidades do pacote.
Tudo isso faz com que os pacotes disponibilizados no CRAN sejam bastante confiáveis, transformando-se assim na fonte oficial de distribuição de pacotes da linguagem.
Se você quiser saber o número de pacotes disponíveis no CRAN hoje, execute:
dim(available.packages(contrib.url(getOption("repos")), filters=list()))[1]Considerando os quantitativos de junho de 2021, existiam quase 18 mil pacotes no CRAN para as mais diversas finalidades. Segue uma pequena lista para dar uma ideia dos temas abordados em pacotes: Econometria, Análise Econômica Regional, Estatística, Clusterização, Ensaios Clínicos, Séries Temporais, Otimização, Tratamento de Dados, Aprendizagem de Máquina e muitos outros. Para saber os tipos de pacotes existentes no repositório, recomenda-se uma visita ao Task Views DO cran
2.2.1 Instalação de pacotes
Para instalar pacotes disponíveis no CRAN, deve-se utilizar a função install.packages("nome_do_pacote"), passando-se o nome do pacote desejado entre aspas como argumento da função.
Para exercitar este procedimento, será instalado o pacote janitor, que disponibiliza algumas funções para manipulação de bases de dados (arrumar nome de variáveis, remoção de linhas e/ou colunas vazias, retirar informações duplicadas, etc.), uma biblioteca que provavelmente será bastante utilizada no dia a dia profissional:
install.packages("janitor")Se o pacote foi corretamente instalado, você deve ver algo semelhante a seguinte mensagem no console:
package ‘janitor’ successfully unpacked and MD5 sums checked
The downloaded binary packages are in
C:\Users\...\...\...\...\...\downloaded_packages
>Não se assuste caso outros pacotes também tenham sido instalados. Isso é muito comum, uma vez que alguns pacotes podem recorrer a funções presentes em outras bibliotecas.
Para carregar ou anexar o pacote à sua sessão no R, de modo que você possa utilizar as funções disponíveis nele, empregamos a função library(nome_do_pacote) ou require(nome_do_pacote). Neste caso, o nome do pacote pode vir com ou sem aspas.
library(janitor)Se você não quiser carregar um pacote completamente em sua sessão do R, porque vai apenas utilizar uma função específica, ao invés de library(), você pode usar o formato nome_do_pacote::nome_dafunção(parâmetro).
2.2.2 Outras fontes de pacotes
Cabe destacar que a versão básica instalada do R, que chamamos de base R, já vem com alguns pacotes instalados, como por exemplo os pacotes stats, MASS, foreign, graphics, o próprio base, dentre outros. Para listar todos os pacotes instalados em sua máquina, execute:
installed.packages()Embora o CRAN seja o repositório oficial de pacotes R, é importante mencionar a existência de outras fontes também importantes para obtenção de pacotes.
A primeira das fontes alternativas é o Bioconductor, que é um projeto open source de desenvolvimento de softwares relacionados à análise e compreensão de dados genômicos gerados em experimentos de laboratório relacionados a biologia molecular. É, portanto, um importante repositório de pacotes para quem trabalha com Bioinformatics ou Biostatistics.
A segunda fonte seria o Github que é um sistema para gerenciamento e versionamento de código. Qualquer pessoa pode criar uma conta no github e começar a compartilhar seus códigos, os quais poderão ser acessados e modificados/melhorados (com o consenso do autor original). No caso do R, os pacotes normalmente possuem uma versão estável no CRAN e uma versão de desenvolvimento no Github, onde os autores e demais colaboradores estão trabalhando no melhoramento e resolução de bugs dos pacotes. Uma vez que a versão do GitHub esteja estável e pronta para lançamento, o autor pode enviá-la ao CRAN.
2.3 Como obter ajuda
Essa talvez seja a parte mais importante de todo o material. Saber como e onde buscar ajuda pode significar gastar apenas alguns minutos ao invés de horas ou mesmo dias na resolução de um problema envolvendo análise de dados e programação. Ao longo deste curso e ao empregar R em uma atividade de trabalho, vocês vão se deparar com diversas dúvidas envolvendo lógica de programação, análise de dados, ou mesmo buscando saber se existe algum pacote já implementado que realize a tarefa que vocês precisam.
2.3.1 O Help do R
O primeiro local para se buscar ajuda sobre algo relacionado ao R, normalmente está dentro do próprio R ou RStudio. Conforme mencionado na seção @ref(#pkg), a política do CRAN exige que os pacotes sejam muito bem documentados. E isso ajuda muito os usuários e desenvolvedores. Além da documentação do base R e dos demais pacotes que já acompanham a instalação padrão, ao instalar um novo pacote, a documentação desta nova biblioteca passa a compor o help do R instalado em sua máquina. Então, quando precisar entender uma função, ou seja, conhecer seus parâmetros de entrada, o que ela faz e o que retorna, recomenda-se utilizar o help do próprio R.
No painel direito inferior, você encontra uma série de abas, sendo que a 3ª delas é aba que exibe o material de ajuda. Você pode fazer as buscas utilizando o campo de pesquisa da própria aba, ou por meio de comandos inseridos no console.
Painel de ajuda do R/Rstudio
Como ilustração, imagine que você gostaria de saber se o R possui alguma função que calcula o logaritmo (base \(e\)) de um número. Para isso, existe a função help.search(), que recebe como parâmetro (“entre aspas”) o tópico sobre o qual você deseja pesquisar. Toda a ajuda do R está em inglês; por isso, se você quer encontrar algo relacionado à logaritmo, deve executar o comando help.search("logarithm") ou ??logrithm.
Vejamos:
# alternativa 1
help.search("logarithm")
# ou
# alternativa 2
??logarithmNa aba Help do RStudio aparecerá o resultado dos pacotes e as respectivas funções que contém as palavras que você buscou. Nesse caso, portanto, caso, temos a função log() do pacote base para calcular logaritmos. Veja:
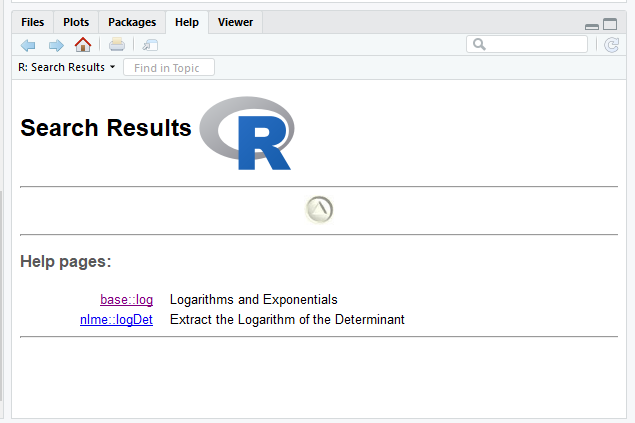
Resultados de busca
Se você já sabe exatamente o nome da função ou objeto do R sobre o qual deseja obter ajuda, pode utilizar também help("palavra") ou ?palavra.
# alternativa 1
help("log")
# ou
# alternativa 2
??log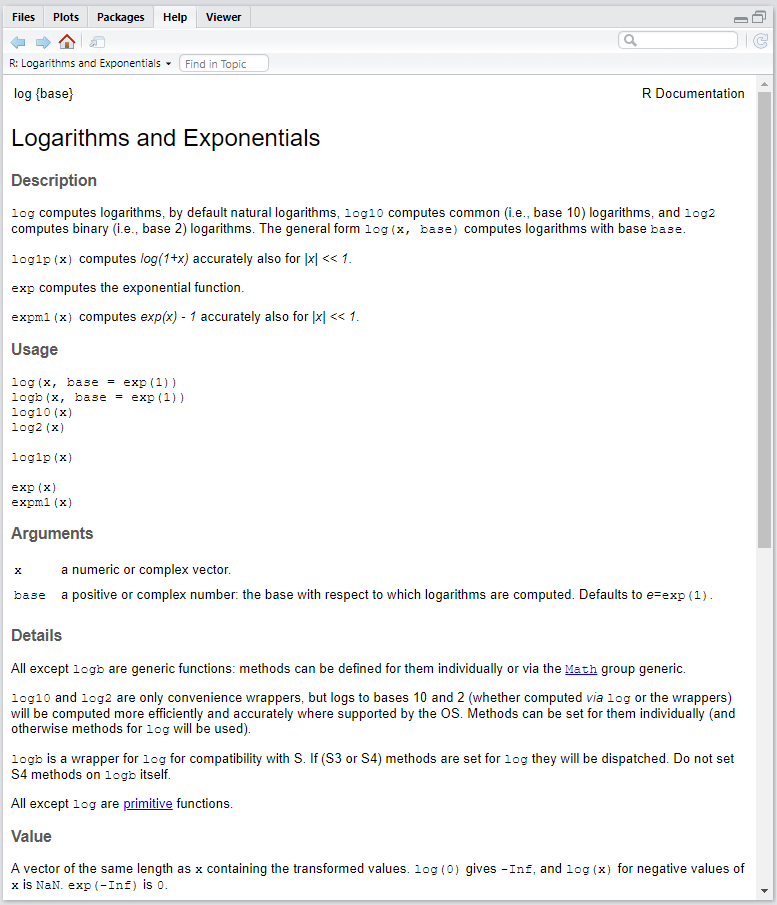
Documentação da função log()
Geralmente, os tópicos mais importantes dos arquivos de ajuda são Description, Usage, Arguments e Examples. O primeiro mostra o que a função faz, o segundo como a função deve ser usada, o terceiro quais argumentos ela recebe e, no quarto, você encontra exemplos de como ela funciona. Value também pode ser interessante, pois informa qual tipo de objeto a função retorna.
2.3.1.1 Vignettes
Mais a frente, será necessário instalar novos pacotes no R, com o objetivo de permitir ao usuário realizar mais operações e atividades na linguagem. Muitos dessas bibliotecas trazem consigo, além da documentação básica, verdadeiros tutoriais sobre como utilizar o pacote em si ou funções específicas dele. São muito mais detalhados que a simples documentação do Help do pacote ou de uma função. Enquanto alguns não fornecem nenhuma documentação (seja ela detalhada, vignette, ou não), outros podem trazer mais de uma vignette. Caso você queira verificar, basta utilizar:
vignette("nome_do_pacote")2.3.1.2 Ajuda via Internet
R é bastante conhecido por sua comunidade extremamente ativa. Muitas vezes, ao analisar dados, surgem problemas que não são passíveis de solução apenas com a documentação interna da linguagem. Nesses casos, é necessário recorrer à experiência de outros usuários que tenham vivenciado o problema e que possam dar dicas de como solucioná-lo. Depois de mais de 20 anos do R, vai ser difícil não encontrar alguém que vivenciou o mesmo problema que você. Nesse ponto a internet é a melhor amiga de todos os usuários e não é vergonha nenhuma procurar ajuda lá. Dos mais inexperientes usuários da linguagem aos mais talentosos desenvolvedores de R, todos, sem exceção, acabam recorrendo à internet como uma valiosa fonte de informação e ajuda. Existem livros gratuitos, blogs com excelentes tutoriais, fóruns e sites de perguntas e respostas (Q&A).
Neste último caso, não há como não mencionar o StackOverflow, que é um site de perguntas e respostas especializado em programação em geral. A comunidade de R no StackOverflow é extremamente ativa e uma das maiores do site. Lá você encontrará usuários iniciantes, intermediários, avançados, desenvolvedores de pacotes e até mesmo gente que atua na atualização da linguagem e do RStudio. Há uma versão em português e uma em inglês. Recorra à versão em inglês do site, por ser obviamente muito mais ativa.
Antes de postar uma pergunta lá faça uma busca com os termos que deseja, por exemplo: “How do a scatter plot in R?”. Se digitar isso na busca do site ou mesmo no Google, nos primeiros resultados você já terá as respostas necessárias do StackOverflow para fazer um scatterplot no R, pelo menos de 3 formas diferentes, usando pacotes diferentes.
Veja um exemplo de pergunta e resposta no StackOverflow:
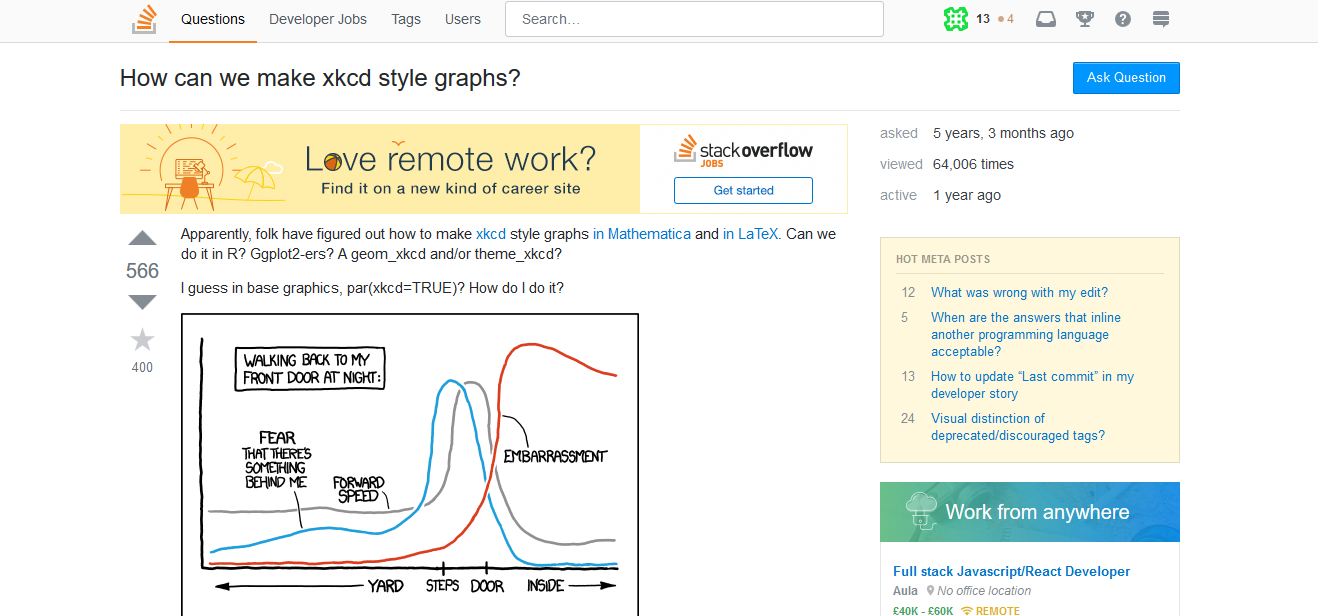
Pergunta
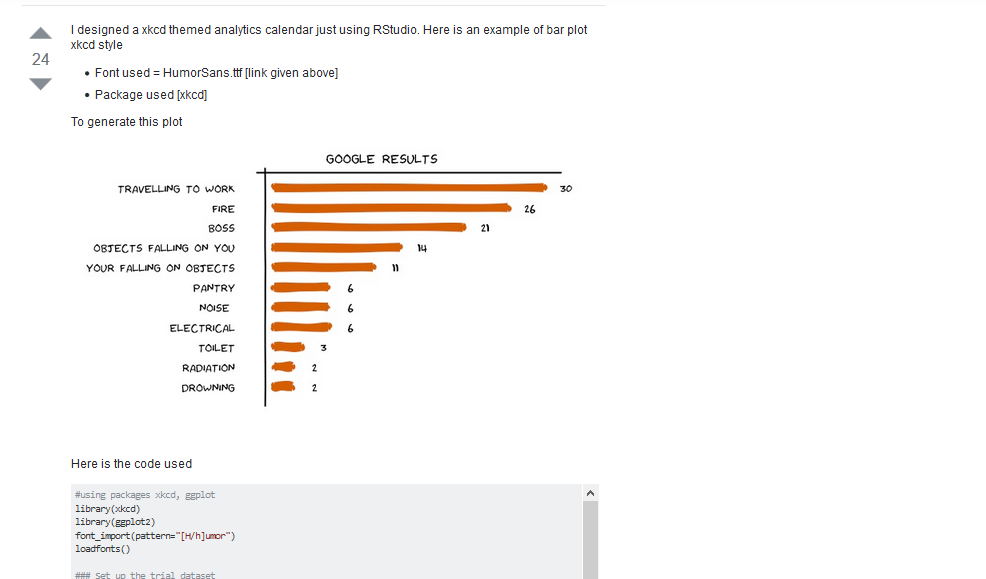
Resposta
2.4 Estrutura de Objetos da Linguagem R
Entender a estrutura de criação e manipulação de objetos no R será essencial para impulsionar sua velocidade de aprendizado nas demais sessões do curso. O bom aproveitamento dos tópicos mais práticos dependem muito de um entendimento sólido sobre os objetos da linguagem R.
Tudo (ou quase tudo) no R são objetos. Os mais importantes são:
- Vetores: são uma espécie de array unidimensional. Consistem em uma sequência de valores que podem ser: numéricos, caracteres ou expressões lógicas (como TRUE ou FALSE). Ressalta-se que as entradas de um vetor podem ser somente de um único tipo.
Exemplo:
## [1] 5.1 4.9 4.7 4.6 5.0 5.4- Matrizes: são arrays multidimensionais com coleção de vetores em linhas ou colunas, sendo que todos os vetores da coleção possuem o mesmo tipo e tamanho.
Exemplo:
## [,1] [,2]
## [1,] "R" "IPEA"
## [2,] "IPEA" "R"
## [3,] "R" "IPEA"
## [4,] "IPEA" "R"
## [5,] "R" "IPEA"- Dataframes: são praticamente idênticos às matrizes, mas com a possibilidade de se ter uma coleção de vetores (colunas) de diferentes tipos (ex: um vetor numérico e outro vetor de caracteres). Por essa característica, é o principal objeto utilizado para armazenar tabelas de dados no R.
Exemplo:
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosa
## 7 4.6 3.4 1.4 0.3 setosa
## 8 5.0 3.4 1.5 0.2 setosa
## 9 4.4 2.9 1.4 0.2 setosa
## 10 4.9 3.1 1.5 0.1 setosa- Listas: é o tipo mais amplo de objeto, que pode reunir coleções de dataframes, vetores e/ou matrizes, ou ainda de todos eles. Uma característica da lista é que, uma vez que os objetos dentro dela não precisam ser do mesmo tipo, também não há a necessidade de que sejam do mesmo tamanho. Isso, muitas vezes, é de grande auxílio na manipulação de dados.
Exemplo:
## [[1]]
## [1] "R" "CEPERJ" "2020"
##
## [[2]]
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
##
## [[3]]
## [,1] [,2]
## [1,] 0.9028944 0.1026966
## [2,] 0.6823824 0.6325349- Funções: são um conjunto de procedimentos que recebem zero, um ou mais parâmetros como input, realizam um cálculo ou procedimento e retornam um resultado para o usuário. Como você pode perceber até funções são consideradas objetos no R.
Exemplo:
## function (x, base = exp(1)) .Primitive("log")Nesta seção, a criação e manipulação de vetores e Dataframes será estudada mais a fundo, por serem os objetos mais importantes para este curso. A criação de funções será objeto de estudo do Módulo 3.
2.4.1 Vetores
2.4.1.1 Como criar um vetor
No R há 3 tipos de vetores: númericos (numeric), de caracteres ou strings (character) e vetores lógicos (logic). Todos eles são criados por meio da função c(), sendo o “c” de concatenate. Esta função faz a concatenação de elementos de um mesmo tipo, produzindo, assim um vetor. Os parâmetros a serem passados são os elementos que comporão o vetor e devem ser separados por vírgulas.
Vetor numérico:
v1 <- c(1, 0.2, 0.3, 2, 2.8); v1## [1] 1.0 0.2 0.3 2.0 2.8DICA: Ao usar ;, você indica ao R que está separando a execução do código, embora haja duas operações na mesma linha - uma que cria o vetor e outra que imprime o resultado no console ao chamar o nome do objeto.
DICA: Os vetores numéricos podem ser de dois tipos: integer, para números inteiros ou double, para ponto flutuante (números decimais). Para saber o tipo de um vetor, use a função typeof(nome_vetor)
Vetor de caracteres ou strings:
v2 <- c("R", "CEPERJ", "2021", "R", "CEPERJ", "2021"); v2## [1] "R" "CEPERJ" "2021" "R" "CEPERJ" "2021"DICA: Note que as strings devem ser passadas "entre aspas".
Vetor lógico:
v3 <- c(TRUE, FALSE, FALSE, TRUE, TRUE, FALSE, FALSE, TRUE); v3## [1] TRUE FALSE FALSE TRUE TRUE FALSE FALSE TRUEExperimente misturar dois vetores. Como os vetores só podem ser de um único tipo, o R automaticamente forçará o resultado final para um único tipo.
Exemplo:
v4 <- c(v1, v2); v4## [1] "1" "0.2" "0.3" "2" "2.8" "R" "CEPERJ" "2021" "R" "CEPERJ" "2021"v5 <- c(v1, v3); v5## [1] 1.0 0.2 0.3 2.0 2.8 1.0 0.0 0.0 1.0 1.0 0.0 0.0 1.0v6 <- c(v2, v3); v6## [1] "R" "CEPERJ" "2021" "R" "CEPERJ" "2021" "TRUE" "FALSE" "FALSE" "TRUE" "TRUE"
## [12] "FALSE" "FALSE" "TRUE"seq(-1, -10)## [1] -1 -2 -3 -4 -5 -6 -7 -8 -9 -10A função typeof(nome_do_vetor) pode ser usada para confirmar os tipos dos vetores que você criou.
2.4.1.1.1 Outras formas de criar um vetor
Há outras funções interessantes que possibilitam criar vetores:
:, exemplo:1:100, cria um vetor numérico composto pela sequência de 1 a 100;seq(), exemplo:seq(-10, -1), cria um vetor numérico composto pela sequência de -10 a -1;rep(), exemplo:rep("CEPERJ", 10), cria um vetor composto pela string"CEPERJ"repetida 10 vezes.
2.4.1.2 Como extrair elementos de um vetor
Para extrair elementos de um vetor (e de qualquer objeto no R), utilizamos a indexação por meio de colchetes nome_do_vetor[posição]. Você pode informar uma única posição, um conjunto de posições ou mesmo um intervalo de posições a extrair:
Extraindo um único elemento:
# 3º elemento do vetor v1
v1[3]## [1] 0.3# 1º elemento do vetor v2
v2[1]## [1] "R"# 5º elemento do vetor v3
v3[5]## [1] TRUEExtraindo mais de um elemento:
Aqui você usará um vetor dentro de outro vetor. A ideia é que o vetor dentro dos colchetes [] contenha as posições (índices) a serem extraídas do vetor original. Lembre-se que o vetor contendo as posições deverá ser criado e, para criar um vetor, usamos a função c().
# 1º e 3º elementos do vetor v1
v1[c(1,3)]## [1] 1.0 0.3# ou
pos1 <- c(1,3)
v1[pos1]## [1] 1.0 0.3# 2º e 4º elemento do vetor v2
v2[c(2,4)]## [1] "CEPERJ" "R"# ou
pos2 <- c(1,3)
v2[pos2]## [1] "R" "2021"# 1º, 2º e 5º elemento do vetor v3
v3[c(1,2,5)]## [1] TRUE FALSE TRUE# ou
pos3 <- c(1,2,5)
v3[pos3]## [1] TRUE FALSE TRUEExtraindo elementos em um intervalo:
Mais uma vez será utilizado um vetor dentro de outro vetor. A diferença é que será criado um vetor utilizando a função :, a qual cria uma sequência de valores ou intervalo. Também é possível utilizar a função seq(a,b)
# do 1º ao 3º elementos do vetor v1
v1[1:3]## [1] 1.0 0.2 0.3# ou
v1[seq(1,3)]## [1] 1.0 0.2 0.3# do 2º ao 5º elemento do vetor v2
v2[2:5]## [1] "CEPERJ" "2021" "R" "CEPERJ"# ou
v2[seq(2,5)]## [1] "CEPERJ" "2021" "R" "CEPERJ"# do 3º ao 6º elemento do vetor v3
v3[2:6]## [1] FALSE FALSE TRUE TRUE FALSE# ou
v3[seq(2,6)]## [1] FALSE FALSE TRUE TRUE FALSE2.4.1.3 Como calcular o tamanho de um vetor?
DICA: Para calcular o tamanho de um vetor, use a função length(nome_vetor):
length(v6)## [1] 142.4.1.4 Como alterar elementos de um vetor
Uma vez que você entendeu o processo de indexação de vetores. Você pode alterar ou substituir um ou mais elementos de um vetor, usando indexação e atribuição.
v1[2] <- 450.78
v2[3] <- 2021
v3[c(3,5)] <- c(TRUE, FALSE) # a substituição tem que ser do mesmo tamanho do resultado da indexação2.4.2 Dataframes
Os Dataframes são as “tabelas” do R. Provavelmente será um dos objetos que você mais utilizará para fazer análise de dados. O Dataframe possui linhas e colunas. Pense nas colunas como vetores, onde cada uma das posições desse vetor indica uma linha. Desta forma, pode-se pensar nos Dataframes, como uma coleção de vetores, que podem inclusive ser de tipos diferentes, mas necessariamente devem possuir o mesmo tamanho. Isso faz todo sentido, pois numa tabela pode existir variáveis numéricas, nomes, e outros tipos que não serão tratados neste curso, como datas, por exemplo.
2.4.2.1 Como criar um Dataframe
Para criar um dataframe, se utiliza a função data.frame(). No entanto, o mais comum é que o dataframe seja criado a partir da leitura de alguma base de dados, por meio das funções de leitura que serão apresentadas principalmente na Seção 2.7.
Para praticar, o objetivo é criar um dataframe de 3 colunas. Os argumentos principais são os vetores que compõem as colunas.
# OBS: todos os vetores precisam ter o mesmo tamanho
v6 <- 11:15
v7 <- seq(0.3, 0.7, by=0.1)
v8 <- rep("CEPERJ", 5)
v9 <- rep(c(TRUE, FALSE), 5)
df1 <- data.frame(v6, v7, v8, v9)
df1## v6 v7 v8 v9
## 1 11 0.3 CEPERJ TRUE
## 2 12 0.4 CEPERJ FALSE
## 3 13 0.5 CEPERJ TRUE
## 4 14 0.6 CEPERJ FALSE
## 5 15 0.7 CEPERJ TRUE
## 6 11 0.3 CEPERJ FALSE
## 7 12 0.4 CEPERJ TRUE
## 8 13 0.5 CEPERJ FALSE
## 9 14 0.6 CEPERJ TRUE
## 10 15 0.7 CEPERJ FALSEDICA: Há vários outros argumentos que também podem ser usados. Vejamos a estrutura da função data.frame(). Vale a pena consultar o help da função data.frame também (help(data.frame)).
data.frame
help("data.frame")Durante a criação de um dataframe, também é possível escolher outros nomes para as colunas:
df1 <- data.frame(col1 = v6, col2 = v7, col3 = v8, col4 = v9)
# ou
# df1 <- data.frame("col1" = v6, "col2" = v7, "col3" = v8, "col4" = v9)
df1## col1 col2 col3 col4
## 1 11 0.3 CEPERJ TRUE
## 2 12 0.4 CEPERJ FALSE
## 3 13 0.5 CEPERJ TRUE
## 4 14 0.6 CEPERJ FALSE
## 5 15 0.7 CEPERJ TRUE
## 6 11 0.3 CEPERJ FALSE
## 7 12 0.4 CEPERJ TRUE
## 8 13 0.5 CEPERJ FALSE
## 9 14 0.6 CEPERJ TRUE
## 10 15 0.7 CEPERJ FALSEVocê deve ter notado que há sempre um coluna à esquerda que contem a numeração ou nomes das linhas do seu dataframe. É o que chamamos de rownames.
DICA: Se você quiser confirmar a estrutura de seu dataframe, ou seja, saber o tipo de suas colunas, use a função str(nome_data_frame)
str(df1)## 'data.frame': 10 obs. of 4 variables:
## $ col1: int 11 12 13 14 15 11 12 13 14 15
## $ col2: num 0.3 0.4 0.5 0.6 0.7 0.3 0.4 0.5 0.6 0.7
## $ col3: chr "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ" ...
## $ col4: logi TRUE FALSE TRUE FALSE TRUE FALSE ...Note que a coluna 2 col2 foi tratada como numeric e não como character, como esperado.
df1 <- data.frame(col1 = v6, col2 = v7, col3 = v8, col4 = v9, stringsAsFactors = FALSE)
str(df1)## 'data.frame': 10 obs. of 4 variables:
## $ col1: int 11 12 13 14 15 11 12 13 14 15
## $ col2: num 0.3 0.4 0.5 0.6 0.7 0.3 0.4 0.5 0.6 0.7
## $ col3: chr "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ" ...
## $ col4: logi TRUE FALSE TRUE FALSE TRUE FALSE ...DICA: A partir da versão 4.0.0, não é mais necessário declarar stringsAsFactors = FALSE na função data.frame. Este passou a ser o comportamento padrão da função: não transformar colunas de strings em fatores.
Note que, diferentemente dos vetores que eram unidimensionais, os dataframes são bidimensionais. Temos uma dimensão representada pelas linhas e outra dimensão representada pelas colunas. Para calcular as dimensões de seu dataframe você pode usar utilizar as seguintes funções:
# linhas vs colunas
dim(df1)## [1] 10 4# nro de linhas
nrow(df1)## [1] 10# nro de colunas
ncol(df1)## [1] 42.4.2.2 Como extrair elementos de um Dataframe
Para extrair os elementos de um Dataframe, basta utilizar a mesma técnica de indexação dos vetores, usando colchetes []. A diferença é que, como o Dataframe possui duas dimensões, será necessário trabalhar com ambas, separando os índices de cada dimensão com um vírgula dentro dos colchetes nome_df[índice_linhas, indice_colunas].
Agora, os valores que são passados dentro dos colchetes referem-se às linhas e/ou as colunas de um Dataframe.
DICA: Se você quiser extrair apenas linha(s) inteira(s), deixe a dimensão da coluna em branco, ex: nome_df[linha_X, ]. Se você quiser extrair apenas coluna(s), deixe a dimensão das linhas em branco, ex: nome_df[, coluna_Y].
Extraindo uma ou mais linhas:
# única linha
df1[3, ]## col1 col2 col3 col4
## 3 13 0.5 CEPERJ TRUE# algumas linhas
# note o posicionamento das vírgulas
df1[c(1,2,5), ]## col1 col2 col3 col4
## 1 11 0.3 CEPERJ TRUE
## 2 12 0.4 CEPERJ FALSE
## 5 15 0.7 CEPERJ TRUE# uma sequência de linhas
df1[3:5, ]## col1 col2 col3 col4
## 3 13 0.5 CEPERJ TRUE
## 4 14 0.6 CEPERJ FALSE
## 5 15 0.7 CEPERJ TRUEExtraindo uma ou mais colunas:
# única coluna
df1[ ,2]## [1] 0.3 0.4 0.5 0.6 0.7 0.3 0.4 0.5 0.6 0.7# algumas colunas
# note o posicionamento das vírgulas
df1[, c(2,3)]## col2 col3
## 1 0.3 CEPERJ
## 2 0.4 CEPERJ
## 3 0.5 CEPERJ
## 4 0.6 CEPERJ
## 5 0.7 CEPERJ
## 6 0.3 CEPERJ
## 7 0.4 CEPERJ
## 8 0.5 CEPERJ
## 9 0.6 CEPERJ
## 10 0.7 CEPERJ# uma sequência de colunas
df1[, 2:4]## col2 col3 col4
## 1 0.3 CEPERJ TRUE
## 2 0.4 CEPERJ FALSE
## 3 0.5 CEPERJ TRUE
## 4 0.6 CEPERJ FALSE
## 5 0.7 CEPERJ TRUE
## 6 0.3 CEPERJ FALSE
## 7 0.4 CEPERJ TRUE
## 8 0.5 CEPERJ FALSE
## 9 0.6 CEPERJ TRUE
## 10 0.7 CEPERJ FALSEExtraindo elementos específicos, cruzando linhas e colunas
Você pode misturar índices de ambas dimensões, para extrair subconjuntos específicos do seu Dataframe. É como jogar batalha naval:
# elemento único
# elemento no cruzamento da 2ºlinha e 3º coluna
df1[2, 3]## [1] "CEPERJ"# subconjuntos
# elementos no cruzamento da 2ª e 5ª linha vs 1ª e 4ª coluna
df1[c(2,5), c(1,4)]## col1 col4
## 2 12 FALSE
## 5 15 TRUE# subconjuntos
# sequência da 2ª a 4ª linha vs sequência da 3ª a 5ª linha
df1[1:3, 2:4]## col2 col3 col4
## 1 0.3 CEPERJ TRUE
## 2 0.4 CEPERJ FALSE
## 3 0.5 CEPERJ TRUEDICA: Você também pode misturar os exemplos anteriores, por exemplo: df1[2, 2:4].
2.4.2.3 Outras formas de indexar as colunas de um Dataframe
Há mais duas formas de indexar as colunas de um Dataframe. Ambas utilizam os nomes das colunas e não os índices.
Primeira forma alternativa:
df1[, "col2"]## [1] 0.3 0.4 0.5 0.6 0.7 0.3 0.4 0.5 0.6 0.7Segunda forma alternativa:
df1$col3## [1] "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ" "CEPERJ"Note que o output é impresso na horizontal Isso ocorre, porque, quando apenas uma coluna é extraída, está trabalhando com um vetor. E a forma de output de um vetor é na horizontal, como visto anteriormente.
2.5 Leitura de Dados no R
Até o momento, só foram manipulados objetos a partir de dados informados via console ou script. No entanto, o mais comum quando se faz análise de dados, é fazer o carregamento dos mesmos a partir de fontes externas, como um arquivo .txt ou .csv, um banco de dados relacional (SQL) no servidor da empresa, ou ainda, a partir de páginas da internet. Nesta seção, será abordado como ler bases de dados armazenadas localmente no computador.
Habitualmente, trabalhando com programação é possível realizar uma mesma tarefa de mais de uma forma diferente. No caso da leitura de dados no R/RSudio, isso não é diferente, uma vez que o R possui diversos pacotes para realizar a leitura dos dados; uns focam na rapidez, outros na praticidade e outros, ainda, são feitos especificamente para manusear bases de dados massivas. Quanto a esse último aspecto, há de se reconhecer que o R não tem um gerenciamento de memória que permita ao usuário carregar bancos de dados muito grandes (na casa das 10 milhões de linhas mais ou menos). Isso é, de certa forma, proposital, uma vez que o R foi criado para tornar mais fácil a vida do usuário que lida com análise dados. Mas é necessário ressaltar que, devido a sua comunidade extremamente ativa, vários pacotes foram e estão sendo desenvolvidos com o objetivo de melhorar a forma como o R lida com o armazenamento de objetos na memória.
Neste curso, focará na utilização do pacote readr, por haver um consenso que ele alia praticidade e rapidez, considerando as alternativas do base R e da função fread() do pacote data.table.
2.6 O Tidyverse
O readr compõe um conjunto de pacotes chamado de Tidyverse.
“The tidyverse is a set of packages that work with harmony because they share common data representtions and API design. The tidyverse package is designed to make it easy to install and load core packages from the tidyverse in a single command.” * Hadley Wickham *

Hexsticker do Tidyverse
Considerando a afirmação de Hadley Wickham, é possível definir o Tidyverse como um meta-package que congrega uma coleção de diversos outros pacotes R voltados para importação, exploração, manipulação e visualização de dados. Vão desde pacotes para manipulação de strings, expressões regulares, datas, passando por pacotes de leitura e importação, manipulação e visualização de dados, até a geração de relatórios e criação de páginas web, dentre outras coisas.
Esses pacotes buscam propiciar uma maior padronização e facilidade na forma de lidar com dados no R.
2.7 Leitura e Exportação de Dados usando readr
O objetivo do pacote readr é tornar mais fácil e amigável a leitura de bases de dados retangulares/tabulares (csv, txt e fwf) no R. Comparado às alternativas do base R, o readr é mais inteligente no sentido de tentar advinhar os formatos das colunas dos dados, ainda permitindo, se necessário, a especificação de padrões pelo usuário.
As funções de leitura mais importantes no pacote são:
read_csv(): lê arquivos.csvou.txtno formato americano, onde o separador de colunas é a vírgula;read_csv2(): lê arquivos.csvou.txtno formato pt-br, onde o separador de colunas é o ponto e vírgula;read_tsv(): lê arquivos.csvou.txt, onde o separador de colunas é o tab\t;read_table(): lê arquivos.txt, onde o separador de colunas é o espaço em branco;read_delim(): permite ler arquivos de diversas extensões, onde o usuário pode especificar o delimitador por meio do parâmetro delim. A atividade prática proposta é utilizar os dados disponibilizados no repositório Git deste livro. A primeira base de dados que será utilizada étaxa_mortalidade, que contém o quantitativo de nascimentos óbitos infantis nos municípios de Pernambuco considerando o período entre 1999 e 2012. Estas e outras informações estão disponíveis no site do DATASUS. Como o intuito nesta etapa é promover a experiência de leitura/carregamento dos dados, a etapa de manipulação dessas informações já foi realizada. Uma vez na página, você pode salvá-la em seu computador clicando com o botão direito do mouse e depois em Salvar como. Note que os dados estão em um arquivo.csvno padrão norte-americano (colunas separadas por vírgulas ,). Será necessário realizar a leitura do arquivo que foi salvo no seu computador. DICA: No seu computador, o caminho para o arquivo será outro.
library(readr)
tb_tx_mort_infantil <- read_csv(file="C:\\Users\\...\\taxa_mortalidade.csv")
# ou utilizando a função mais genérica
tb_tx_mort_infantil <- read_delim(file="C:\\Users\\...\\taxa_mortalidade.csv",
delim = ",")Se você não quiser salvar a base em seu computador, lendo o arquivo direto do site, você também pode. É só copiar o endereço do arquivo apresentado no browser e passar para o argumento file. Caminhos passados para o argumento file começados em http://, https://, ftp://, ou ftps:// resultam no download e leitura automática do arquivo.
##
## -- Column specification ---------------------------------------------------------------------------------------
## cols(
## Cod_mun = col_double(),
## `C<U+663C><U+3E33>digo do Item Geogr<U+653C><U+3E31>fico` = col_double(),
## `Munic<U+653C><U+3E64>pio` = col_character(),
## `Ordem Geogr<U+653C><U+3E31>fica` = col_double(),
## Ano = col_double(),
## Totobit = col_character(),
## ObitN = col_double(),
## ObitP = col_double(),
## `Popula<U+653C><U+3E37><U+653C><U+3E33>o` = col_double()
## )Note que assim que o arquivo é lido, as funções imprimem no console os nomes de cada coluna e as formas como elas foram lidas. Isto permite ao usuário checar se tudo ocorreu bem durante a leitura. Caso alguma coluna não tenha sido lida no formato em que você esperava, é só copiar a saída e rodar a função novamente, mudando a especificação daquela coluna em particular.
tb_tx_mort_infantil <- read_csv(file="https://raw.githubusercontent.com/brunolucian/cursoBasicoR/71de395a83f40736e5ea4f908209f960305c8173/datasets/taxa_mortalidade.csv"
)##
## -- Column specification ---------------------------------------------------------------------------------------
## cols(
## Cod_mun = col_double(),
## `C<U+663C><U+3E33>digo do Item Geogr<U+653C><U+3E31>fico` = col_double(),
## `Munic<U+653C><U+3E64>pio` = col_character(),
## `Ordem Geogr<U+653C><U+3E31>fica` = col_double(),
## Ano = col_double(),
## Totobit = col_character(),
## ObitN = col_double(),
## ObitP = col_double(),
## `Popula<U+653C><U+3E37><U+653C><U+3E33>o` = col_double()
## )Uma vez lido o arquivo, é necessário observar chamando o objeto criado, ou clicando no nome do objeto em nosso environment.
Porém antes de fazer isso iremos limpar o nomes das variáveis usando a função clean_names do pacote janitor.
tb_tx_mort_infantil <- janitor::clean_names(tb_tx_mort_infantil)
tb_tx_mort_infantil## # A tibble: 2,576 x 9
## cod_mun c_digo_do_item_geogr_u_f~ munic_pio ordem_geogr_u_fffd~ ano totobit obit_n obit_p popula_o
## <dbl> <dbl> <chr> <dbl> <dbl> <chr> <dbl> <dbl> <dbl>
## 1 260005 2600054 "Abreu e Lima" 2 1999 33 24 9 82849
## 2 260010 2600104 "Afogados da Ing~ 3 1999 24 15 9 33874
## 3 260020 2600203 "Afr\xe2nio" 4 1999 15 8 7 14146
## 4 260030 2600302 "Agrestina" 5 1999 26 10 16 18605
## 5 260040 2600401 "\xc1gua Preta" 6 1999 53 21 32 25442
## 6 260050 2600500 "\xc1guas Belas" 7 1999 16 7 9 34791
## 7 260060 2600609 "Alagoinha" 8 1999 4 1 3 11728
## 8 260070 2600708 "Alian\xe7a" 9 1999 42 11 31 36579
## 9 260080 2600807 "Altinho" 10 1999 25 8 17 20777
## 10 260090 2600906 "Amaraji" 11 1999 15 11 4 20768
## # ... with 2,566 more rowsObserve que este arquivo não se parece muito com o formato clássico de Dataframe que visto no início deste módulo. Ele na verdade é um novo tipo de objeto, que segue a filosofia do Tydeverse, comentada na seção anterior. Aqui se temi um tibble, que nada mais é que um Dataframe mais amigável. Em termos gerais, a diferença de um Dataframe clássico para um tibble está principalmente nas informações que são apresentadas quando o conteúdo do objeto é impresso no console. Diferentemente do Dataframe, um tibble apresenta apenas as 10 primeiras linhas da tabela, os tipos das colunas, e apenas o conteúdo das colunas que cabem em na tela.
Uma outra diferença é no resultado ao executar uma identificação. Ainda é possível utilizar [indice_linhas, indice_colunas] ou nome_dataframe$nome_coluna. A questão é que indexações com chaves, retornaram um tibble, mesmo que a operação resulte em uma única coluna. Já a indexação via $, retornará um vetor. Resumindo, a primeira não altera o tipo de objeto - permanece sendo um tibble. A segunda altera o tipo de objeto.
Teste isso na prática:
tb_tx_mort_infantil[,2]
tb_tx_mort_infantil$`Município`Outras vantagens do tibble são:
- não precisarmos mais nos preocupar com
stringsAsFactors=FALSE; - ele funciona de forma muito mais fluida com os demais pacotes do tidyverse que utilizaremos mais a frente no curso;
2.7.0.1 Alternativas
Quando se trata de leitura de dados, há muitas alternativas no R. Embora não pertença ao escopo deste curso, seria interessante que você estudasse o funcionamento das funções da família read.table() do base R. Essas funções são mais lentas que as funções do pacote readr, mas apresentam maior flexibilidade em relação à especificação de parâmetros.
Já para em relação aos bancos de dados com muitas de observações (da ordem de milhões), a função fread() do pacote data.table é mais adequada. Mas essa velocidade vem com o preço de se ter que aprender um paradigma um pouco diferente de programação, mesmo sendo dentro do R.
Muitas pessoas também têm interesse em ler dados que estão em planilhas ou abas de planilhas Excel em formatos .xls e .xlsx. Embora não seja a forma mais adequada de armazenar dados, é válido saber que existe uma alternativa para a leitura de dados do Excel. Também é interessante a leitura sobre o pacote readxl e a função read_excel().
2.7.1 Escrita e exportação de dados
É importante destacar que, apesar do nome, o pacote readr também possui funções para escrita de dados. Para exportar o resultado das análises no R para uma tabela de extensão .txt ou .csv, por exemplo, se utiliza funções bastante parecidas com as de leitura, alterando apenas os prefixos de read para write. Os sufixos, novamente, dependerão do tipo de arquivo que se deseja gravar.
As funções mais utilizadas são: write.csv(); ẁrite_csv2; write_table(); e write.delim().
Agora a proposta é alterar o arquivo tb_tx_mort_infantil para, então, exportar a nova versão como um novo arquivo .csv no padrão pt-br. Utilizando o conhecimento sobre indexação de objetos deste módulo, você terá que eliminar a primeira coluna, que era uma coluna de índices.
1º) mudando o status
tb_tx_mort_infantil[1:50000, "STATUS_GF"] <- rep("NÃO VERIFICADO", 50000)
# ou
tb_tx_mort_infantil[1:50000, 3] <- rep("NÃO VERIFICADO", 50000)
# ou
tb_tx_mort_infantil$STATUS_GF[1:50000] <- rep("NÃO VERIFICADO", 50000)2º) excluindo a primeira coluna
No R, assim como em outras linguagens de programação, existe uma palavra reservada pela linguagem que transforma os objetos ou elementos de um objeto em vazios. É a palavra NULL.
tb_tx_mort_infantil[, "Totobit "] <- NULL# ou
tb_tx_mort_infantil[, 1] <- NULL
# ou
tb_tx_mort_infantil_modif <- tb_tx_mort_infantil[, 2:ncol(tb_tx_mort_infantil)]
# substituimos a versão completa do tibble por uma versão que traz...
#...apenas os dados da coluna 2 em diante3º) salvando o novo tibble em um arquivo
write_csv2(x=tb_tx_mort_infantil_modif,
path="C://caminho//...//tabela_tx_mort_infantil_alterada.csv")
# ou
write_delim(x=tb_tx_mort_infantil_modif,
path="C://caminho//...//tabela_tx_mort_infantil_alterada.csv",
delim=";")DICA: É muito comum termos dúvidas sobre quais são os prâmetros de uma função e como ela trabalha. Não se esqueça de sempre consultar o Help do R, ou de digitar o nome_da_função do Console, para ver todos os seus parâmetros.
2.7.2 Leitura de dados oriundos do Stata, SAS e SPSS
Ao longo dos anos, o R cresceu consideravelmente. Durante esse tempo, diversos pesquisadores e profissionais das mais diversas áreas migraram para o R, oriundos principalmente de Stata, SAS e SPSS. Ao encarar o custo da mudança, muitas pessoas se deparam com o desafio de “traduzir” seus scripts de uma linguagem para outra, bem como de lerem dados escritos ou armazenados em extensões usadas por aquelas linguagens.
A instalação padrão do R já oferece uma biblioteca para ler bases de dados oriundas de outras linguagens. O nome do pacote faz todo o sentido: foreign. Ele traz diversas funções que tornam simples a importação. Em complemento, recomenda-se a instalação e utilização do pacote HMisc também.
A seguir, serão apresentadas apenas algumas ideias sobre como lidar com datasets “estrangeiros” no R:
- SPSS:
# salvando o dataset no SPSS no formato transport (XPORT)
get file='C:/meus_dados.sav'.
export outfile='C:/meus_dados.por'.# no R
# install.packages("Hmisc") # caso necessário
library(Hmisc)
df <- spss.get("C:/meus_dados_SPSS.por", use.value.labels=TRUE)
# use.value.labels=TRUE converte os valores dos rótulos em factors- SAS:
# salva o dataset SAS no transport format (XPORT)
libname out xport 'C:/meus_dados_SAS.xpt';
data out.mydata;
set sasuser.mydata;
run;# in R
library(Hmisc)
mydata <- sasxport.get("C:/meus_dados_SAS.xpt")
# as variaveis do tipo character serão convertidas para factors- Stata:
library(foreign)
mydata <- read.dta("C:/meus_dados_stata.dta")2.8 Referências do Módulo
Quick R website. (2020). URL https://www.statmethods.net/.
Wickham, H.; Hester, J.; François R. (2018). readr: Read Rectangular Text Data. R package version 1.3.1. URL https://CRAN.R-project.org/package=readr.
____. (2020). readr official website. URL https://readr.tidyverse.org/index.html.
Wickham, H.; Grolemund, G. (2016). R for Data Science: Import, Tidy, Transform, Visualize, and Model Data. O’Reilly Media. december 2016. 522 pages. Disponível em: https://www.r4ds.co.nz.
Estatística Computacional 2 (2015). Notas de aula. Curso de Estatística, UnB, 1º semestre, 2015.
Nyffenegger, R. (2020). R: a computer language for statistical data analysis. URL https://renenyffenegger.ch/notes/development/languages/R/.
Wickham, H. (2014). Advanced R. September 25, 2014. Chapman and Hall/CRC. 476 Pages. Disponível em: https://adv-r.hadley.nz/.
R Core Team (2020). R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. URL http://www.R-project.org/.
2.9 Exercícios
1) Vetores
- Crie dois vetores numéricos (A e B) de tamanho 10 com números aleatórios escolhidos por você.
- Multiplique todos os itens do vetor A por \(\times3\) e os itens do vetor B por \(\times4\).
- Crie um novo vetor (C) resultante da operação de \(\frac{log(B)}{|A| + (A+B)}\), onde \(|A|\) é o tamanho do vetor A.
- Crie um novo vetor lógico (D), verificando quais valores de A são maiores que os respectivos valores de B.
- Acrescente 3 nomes escolhidos por você ao final de cada vetor. Os vetores mudam de tipo?
2) Dataframes
- Crie um Dataframe de 10 linhas e 5 colunas, com pelo menos 3 colunas de tipos diferentes, sendo pelo menos duas númericas.
- Crie uma 6ª coluna resultante da soma entre duas colunas numéricas do Dataframe.
- Esolha duas colunas numéricas A e B, e crie uma 7ª coluna resultante da operação de \(\frac{log(B)}{|A| + (A+B)}\), onde \(|A|\) é o tamanho da coluna A.