2 Breve recapitulación de R
2.1 Entorno de RStudio y Ayuda
Navegaremos sobre el entorno de RStudio para familiarizarnos con la distribución de ventanas y opciones de los menus.
Utilizaremos la pestaña de ‘Help’ (ayuda) de RStudio para identificar como utilizar comandos/funciones que no son familiares o para obtener más detalles de un método específico. También es posible usar el comando
?, por ejemplo ?sum
2.2 Asignación de Variables
a <- 30
b = 10
a## [1] 30c = a + b
a + b## [1] 402.3 Directorios, Scripts y Librerías
La ubicación del espacio de trabajo actual
getwd()## [1] "/Users/johannaorellana/Documents/JOHANNA/CURSOS/Curso_iDRHiCA_Random_Forest/Book_markdown_curso_RF"Listar los archivos que se encuentran en el espacio de trabajo actual
list.files()## [1] "01-Introduccion_R.Rmd" "02-Arboles_parteI.Rmd"
## [3] "03-Arboles_parteII.Rmd" "03-Arboles_parteII_files"
## [5] "04-RandomForest_parteI.Rmd" "05-RandomForest_parteII.Rmd"
## [7] "06-references.Rmd" "DESCRIPTION"
## [9] "LICENSE" "README.md"
## [11] "_book" "_bookdown.yml"
## [13] "_bookdown_files" "_build.sh"
## [15] "_deploy.sh" "_output.yml"
## [17] "book.bib" "bookdown-demo.Rmd"
## [19] "bookdown-demo.Rproj" "bookdown-demo_files"
## [21] "breve-recapitulaion-de-r.html" "data"
## [23] "images" "index.Rmd"
## [25] "introduccion.html" "mi_script.R"
## [27] "packages.bib" "preamble.tex"
## [29] "style.css" "toc.css"Para crear un directorio
dir.create("temporal")Para cambiar el espacio de trabajo actual
setwd("/directorio_inicial/temporal")Para crear un archivo en el directorio actual de trabajo
file.create("mi_script.R")
list.files()Para instalar una nueva librería use el comando install.packages (una sola vez). Luego para habilitar su uso, debe usar el comando library
install.packages("dplyr")
library(dplyr)2.4 Tipos de Datos Básicos
La interacción final con el computador se efectúa a través de letras y números, lo que denominamos como caracteres alfanuméricos.
Los tipos atómicos de datos en R son: + Caracteres + Enteros + Reales + Complejos + Boleanos
var_caracter = "Un texto"
var_entero = 50
var_real = 50.6
var_complejo = 5i
var_booleano = TRUE2.5 Tipos de Datos Compuestos
2.5.0.1 Atomic Vectors
Este es simplemente un vector de datos y se puede crear usando el comando c(). Este comando permite agrupar ciertos datos en un vector.
estaciones<-c('Toreadora','La Virgen','Chirimachay','Tres Cruces','Balzay','Sayausí')
estaciones## [1] "Toreadora" "La Virgen" "Chirimachay" "Tres Cruces"
## [5] "Balzay" "Sayaus<U+00ED>"length(estaciones)## [1] 6coordsX <-c(697618.73,701110.74,705703.88,695540.08,718267.1,714620)
coordsX## [1] 697618.7 701110.7 705703.9 695540.1 718267.1 714620.0length(coordsX)## [1] 6Existen seis tipos básicos de vectores: dobles, enteros, caracteres, lógicos, complejos, y crudos. Estos son algunos ejemplos de creación.
typeof(estaciones)## [1] "character"typeof(coordsX)## [1] "double"text <- c("Hello", "World") #character
comp<-c(1+1i,1+2i,1+3i)
raw(3)## [1] 00 00 003>4 # FALSE## [1] FALSElogic <- c(TRUE, FALSE, TRUE)
a <- c(-1L, 2L, 4L)
b <- c(-1, 2, 4)Coerción
Sobre los valores lógicos TRUE y FALSE, estos se interpretan también como valores enteros 1 y 0. Por lo tanto es posible contabilizar el número de elementos verdaderos en un vector con una sentencia simple… a este proceso se le denomina coerción.
sum(c(TRUE, TRUE, FALSE, FALSE))## [1] 22.5.1 Matrices
Las matrices almacenan información en múltiples dimensiones. Ahora crearemos una matriz a partir del vector estaciones que contiene nombres de estaciones
m <- matrix(estaciones, nrow = 2)
m## [,1] [,2] [,3]
## [1,] "Toreadora" "Chirimachay" "Balzay"
## [2,] "La Virgen" "Tres Cruces" "Sayaus<U+00ED>"m <- matrix(estaciones, nrow = 2, byrow = TRUE)
m## [,1] [,2] [,3]
## [1,] "Toreadora" "La Virgen" "Chirimachay"
## [2,] "Tres Cruces" "Balzay" "Sayaus<U+00ED>"Es posible aplicar ciertas operaciones y funciones a una variable según su tipo. Para ello lo primero es identificar la clase de la variable.
class(m)## [1] "matrix"class(text)## [1] "character"2.5.2 Factores
Este tipo de datos se utiliza frecuentemente para denotar algún tipo de clasificación o disgregación. Tienen asociada una descripción a un valor numérico. Esto permite reducir el espacio en disco para el almacenamiento.
gender <- factor(c("male", "female", "female", "male"))
typeof(gender)## [1] "integer"attributes(gender)## $levels
## [1] "female" "male"
##
## $class
## [1] "factor"Para visualizar como R almacena la información de la variable tipo factor se usa el comando unclass().
unclass(gender)## [1] 2 1 1 2
## attr(,"levels")
## [1] "female" "male"gender## [1] male female female male
## Levels: female maleas.character(gender)## [1] "male" "female" "female" "male"2.5.3 Listas
Similares a los vectores por cuanto agrupan datos en un conjunto unidimensional, sin embargo las listas no agrupan valores individuales sino objetos R. Cada uno de los elementos de la lista puede a su vez contener varios valores individuales. Para crear este tipo de objeto se usa la función list() separando cada elmento con una coma.
list1 <- list(100:130, "R", list(TRUE, FALSE))
list1## [[1]]
## [1] 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116
## [18] 117 118 119 120 121 122 123 124 125 126 127 128 129 130
##
## [[2]]
## [1] "R"
##
## [[3]]
## [[3]][[1]]
## [1] TRUE
##
## [[3]][[2]]
## [1] FALSELos resultados de funciones más complejas de R, usualmenre devuelven un objeto tipo lista.
2.5.4 Data Frames
El tipo de objeto data frame es una versión de dos dimensiones de una lista. Esta estructura es la más utilizada para el almacenamiento de datos. Esta estructura es equivalente a una hoja de cálculo en excel. Los data frames agrupan vectores, donde cada uno de ellos representa una columna del data frame, así pues cada columna puede ser de un tipo diferente.
df <- data.frame(descripcion = c("Toreadora", "Chirimachay", "La Virgen"),
cuenca = c("Quinoas", "Quinoas", "Quinoas"), id= c(1, 2, 3))
df## descripcion cuenca id
## 1 Toreadora Quinoas 1
## 2 Chirimachay Quinoas 2
## 3 La Virgen Quinoas 3typeof(df) ## [1] "list"class(df)## [1] "data.frame"str(df)## 'data.frame': 3 obs. of 3 variables:
## $ descripcion: Factor w/ 3 levels "Chirimachay",..: 3 1 2
## $ cuenca : Factor w/ 1 level "Quinoas": 1 1 1
## $ id : num 1 2 3El tipo factor es uno de los “preferidos” de R, por lo tanto, cualquier dato que corresponda inicialmente a texto podría ser interpretado por R como un factor. Sin embargo, esto no siempre es lo que se desea y para evitar la creación (conversión) a este tipo en las columnas del data frame podemos usar el argumento stringsAsFactors = FALSE
df <- data.frame(descripcion = c("Toreadora", "Chirimachay", "La Virgen"),
cuenca = c("Quinoas", "Quinoas", "Quinoas"), id= c(1, 2, 3), stringsAsFactors = FALSE)
df## descripcion cuenca id
## 1 Toreadora Quinoas 1
## 2 Chirimachay Quinoas 2
## 3 La Virgen Quinoas 3str(df)## 'data.frame': 3 obs. of 3 variables:
## $ descripcion: chr "Toreadora" "Chirimachay" "La Virgen"
## $ cuenca : chr "Quinoas" "Quinoas" "Quinoas"
## $ id : num 1 2 3¿Cómo acceder a las columnas de mi data frame?
df$descripcion## [1] "Toreadora" "Chirimachay" "La Virgen"df$id## [1] 1 2 3desc <- df$descripcion
desc## [1] "Toreadora" "Chirimachay" "La Virgen"class(desc)## [1] "character"typeof(desc)## [1] "character"Crear un data frame manualmente puede ser muy extenso y además ser sensible a errores de escritura. La mayor parte de aplicaciones trabajan con archivos de texto que pueden ser importados o exportados.
df.data <- iris
nrow(df.data)## [1] 150head(df.data)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosatail(df.data)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 145 6.7 3.3 5.7 2.5 virginica
## 146 6.7 3.0 5.2 2.3 virginica
## 147 6.3 2.5 5.0 1.9 virginica
## 148 6.5 3.0 5.2 2.0 virginica
## 149 6.2 3.4 5.4 2.3 virginica
## 150 5.9 3.0 5.1 1.8 virginicanames(df.data)## [1] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width"
## [5] "Species"Ahora vamos a extraer una sola columna del data frame y contabilizar el número de elementos.
longitudP <- df.data$Petal.Length
nrow(longitudP)## NULLlength(longitudP)## [1] 1502.6 Lectura y escritura de archivos de texto
Recapitulemos el uso de las funciones getwd() y setwd(). El primero obtiene la ruta del directorio actual de trabajo, en tanto que el segundo especifica o cambia la ruta del directorio sobre el cual se desea trabajar. Ahora vamos a ubicarnos en el directorio que contiene el archivo de texto que deseamos abrir en R (cargar en la memoria).
df.observaciones <- read.table("data/observaciones_2003.csv", header = TRUE)
head(df.observaciones)## FECHA VALOR STATUS ESTACION year day month
## 7671 01/01/2003 0 M012 2003 1 1
## 7672 02/01/2003 0 M012 2003 2 1
## 7673 03/01/2003 0 M012 2003 3 1
## 7674 04/01/2003 0 M012 2003 4 1
## 7675 05/01/2003 0 M012 2003 5 1
## 7676 06/01/2003 0 M012 2003 6 1¿Qué sucede con los valores de la columna STATUS?
De ser necesario es posible indicarle a R como se desea manejar los valores nulos (donde no existen datos). En ocasiones inclusive nuestro archivo de texto podría usar un caracter especial de su fuente inicial para representar estos datos (e.g la palabra NULL). En este caso se incrementa un argumento a la función read.table.
df.observaciones <- read.table("data/observaciones_2003.csv", header = TRUE, na.strings = "", stringsAsFactors = FALSE)
df.observaciones <- read.table("data/observaciones_2003.csv", header = TRUE, stringsAsFactors = FALSE)
head(df.observaciones)## FECHA VALOR STATUS ESTACION year day month
## 7671 01/01/2003 0 M012 2003 1 1
## 7672 02/01/2003 0 M012 2003 2 1
## 7673 03/01/2003 0 M012 2003 3 1
## 7674 04/01/2003 0 M012 2003 4 1
## 7675 05/01/2003 0 M012 2003 5 1
## 7676 06/01/2003 0 M012 2003 6 1class(df.observaciones)## [1] "data.frame"nrow(df.observaciones)## [1] 8030Existen otros argumentos para leer un archivo y también otras funciones.
df.observaciones.sep <- read.table("data/observaciones_2003.csv", sep = ",", header = TRUE, skip = 3, nrow = 5)
df.observaciones.csv <- read.csv("data/observaciones_2003.csv")Así como es posible leer un archivo y cargarlo en R como un data frame, es posible hacer lo opuesto; almacenando un data frame en un archivo de texto.
write.csv(df.observaciones, "data/df.observaciones.csv", row.names = FALSE)
write.table(df.observaciones, "data/dataFrame_observaciones.txt", row.names = FALSE)2.7 Indexación
2.7.1 Método básico – Cómo acceder a ciertos datos de los objetos de R
Para acceder a uno o varios elementos de un data frame, se colocan los índices de fila(s) y columna(s) deseados entre corchetes, así [filas,columnas]. El primer índice se utilizará para recuperar las filas del data frame y el segundo para recuperar las columnas.
Existen 5 maneras diferentes (las más utilizadas) de escribir los índices en R.
- Enteros Positivos
- Enteros Negativos
- Espacios en blanco
- Valores lógicos
- Nombres
2.7.1.1 Enteros Positivos
La analogía es la notación algebraica de i,j: df.observaciones[i,j].
head(df.observaciones)## FECHA VALOR STATUS ESTACION year day month
## 7671 01/01/2003 0 M012 2003 1 1
## 7672 02/01/2003 0 M012 2003 2 1
## 7673 03/01/2003 0 M012 2003 3 1
## 7674 04/01/2003 0 M012 2003 4 1
## 7675 05/01/2003 0 M012 2003 5 1
## 7676 06/01/2003 0 M012 2003 6 1df.observaciones[1, 1]## [1] "01/01/2003"Para extraer más de un valor, se debe usar un vector de enteros positivos. Por ejemplo para recuperar la primera fila de nuestro data frame de observaciones.
df.observaciones[1,c(1,2,3,4,5,6,7)]## FECHA VALOR STATUS ESTACION year day month
## 7671 01/01/2003 0 M012 2003 1 1df.observaciones[1,1:7]## FECHA VALOR STATUS ESTACION year day month
## 7671 01/01/2003 0 M012 2003 1 1Si el objetivo es trabajar posteriormente con este subconjunto de datos, al asignar a una nueva variable en realidad obtenemos una copia de la primera fila del data frame.
new <- df.observaciones[1, 1:7]
new## FECHA VALOR STATUS ESTACION year day month
## 7671 01/01/2003 0 M012 2003 1 1La misma sintaxis es válida para cualquier objeto R en tanto que se utilice la dimensión adecuada. Por ejemplo un vector posee una sola dimensión por lo tanto requiere un sólo índice. La numeración de índices en R empieza en 1
vec<-c(6,1,3,6,10,5)
vec[1:3]## [1] 6 1 3¿Qué pasa al seleccionar dos o más columnas de un data frame o cuándo seleccionamos solo una columna?
En caso de que se requiera como salida un data frame para el segundo caso basta con agregar el argumento drop
df.observaciones[1:2, 1, drop = FALSE]## FECHA
## 7671 01/01/2003
## 7672 02/01/20032.7.1.2 Enteros Negativos
Es el caso opuesto de la indexación con enteros positivos. R retornará todos los elementos a excepción de los elementos que pertenecen al índice negativo. Esta es una forma más eficiente de obtener subconjuntos de datos para el caso de que se desee conservar la mayor cantidad de filas o columnas.
df.observaciones[-(2:52), 1:3]2.7.1.3 Espacios en blanco
Se puede utilizar un espacio en blanco para indicarle a R que debe extraer todos los valores en una dimensión. Esto es útil para recuperar/extraer todas las columnas o todas las filas de un data frame.
df.observaciones[1, ]## FECHA VALOR STATUS ESTACION year day month
## 7671 01/01/2003 0 M012 2003 1 12.7.1.4 Valores lógicos
Si se provee un vector de valores TRUEs y FALSEs como índices, R asociará cada valor TRUE o FALSE a la fila (o columna) correspondiente del data frame. Todas las filas o columnas que estén asociadas al valor TRUE se extraerán. Para que este método funcione, el vector booleano debe tener la misma longitud que la dimensión que se desea obtener el subconjunto de datos.
df.observaciones[1, c(TRUE, TRUE, FALSE,FALSE,TRUE,FALSE,TRUE)]## FECHA VALOR year month
## 7671 01/01/2003 0 2003 1Hacer el ejercicio con las columnas es posible pero…. y con las filas?. Debería asegurar la creación de un vector booleano de la misma longitud que el total de filas del data frame. Entonces la pregunta es…cómo obtener un vector booleano del tamaño de filas del data frame? La sección de subconjuntos lógicos aborda este tema
2.7.1.5 Nombres
Finalmente es posible extraer/recuperar un subconjunto de datos a partir del nombre de las columas.
df.observaciones[, c("FECHA","ESTACION")]2.7.1.6 Otro método de indexación:Signos de dólares
Se puede extraer toda la columna de un data frame utilizando el signo de dólares como el separador entre el nombre del data frame y el nombre de la columna(variable) de interés. R retornará un vector. Esta acción es útil cuando queremos por ejemplo evaluar la media de esta variable. Tomemos en cuenta que la función mean(x), espera que x sea un VECTOR.
df.observaciones$VALOR
mean(df.observaciones$VALOR)2.7.2 Subconjuntos lógicos
Previamente identificamos la posibilidad de obtener un subconjunto de datos a partir de un índice de vector booleano.
vec## [1] 6 1 3 6 10 5vec[c(FALSE, FALSE, FALSE, FALSE, TRUE, FALSE, FALSE)]## [1] 10No es necesario que nosotros creemos el vector booleano, podemos efectuar un test lógico para obtener esta estructura. Existen 7 operadores lógicos para realizar comparaciones.
- a>b
- a>=b
- a<b
- a<=b
- a==b
- a!=b
- a %in% c(a, b, c)
1>2## [1] FALSE1>c(0,1,2)## [1] TRUE FALSE FALSEc(1,2,3)==c(3,2,1)## [1] FALSE TRUE FALSEEl único caso particular que no efectúa comparación elemento a elemento es %in%.
1 %in% c(3, 4, 5)## [1] FALSEc(1, 2) %in% c(3, 4, 5)## [1] FALSE FALSEc(1, 2, 3) %in% c(3, 4, 5)## [1] FALSE FALSE TRUEc(1, 2, 3, 4) %in% c(3, 4, 5)## [1] FALSE FALSE TRUE TRUEModificar valores a partir de una expresión de vector booleano.
df.observaciones$ESTACION == "M012"
df.observaciones$VALOR[df.observaciones$ESTACION == "M012"]
df.observaciones$VALOR[df.observaciones$ESTACION == "M012"] <- 102.8 Subconjuntos
El paquete dplyr de R es parte del framework tidyverse de R. Este contiene un conjunto de herramientas (funciones) sumamente intuitivas y poderosas para una rápida y fácil manipulación y procesamiento de los datos. Las posibilidades de dplyr son muy extensas, pero abordaremos las más importantes aquí.
Si no tiene instalada la librería dplyr o tidyverse, hágalo ahora.
The diamonds dataset
En esta sección usaremos el dataset diamonds, el cual es parte del paquete ggplot2. Si no ha trabajado con estos datos anteriormente, se puede revisar su estructura usando ?diamonds.
Nota: diamonds no es un data.frame estándar de R (solía serlo), pero es actualmente un objeto de la clase tbl_df.
library(ggplot2)
class(diamonds)## [1] "tbl_df" "tbl" "data.frame"La ventaja de este tipo de objeto en R es que su función print() sintentiza varias funciones como head(), tail(), o str().
diamonds## # A tibble: 53,940 x 10
## carat cut color clarity depth table price x y z
## <dbl> <ord> <ord> <ord> <dbl> <dbl> <int> <dbl> <dbl> <dbl>
## 1 0.23 Ideal E SI2 61.5 55 326 3.95 3.98 2.43
## 2 0.21 Premium E SI1 59.8 61 326 3.89 3.84 2.31
## 3 0.23 Good E VS1 56.9 65 327 4.05 4.07 2.31
## 4 0.29 Premium I VS2 62.4 58 334 4.20 4.23 2.63
## 5 0.31 Good J SI2 63.3 58 335 4.34 4.35 2.75
## 6 0.24 Very Good J VVS2 62.8 57 336 3.94 3.96 2.48
## 7 0.24 Very Good I VVS1 62.3 57 336 3.95 3.98 2.47
## 8 0.26 Very Good H SI1 61.9 55 337 4.07 4.11 2.53
## 9 0.22 Fair E VS2 65.1 61 337 3.87 3.78 2.49
## 10 0.23 Very Good H VS1 59.4 61 338 4.00 4.05 2.39
## # ... with 53,930 more rows2.8.1 Summarizing data
Las funciones que cubriremos son:
filter(),select(),arrange(),mutate(),summarise(), ygroup_by(),
estas serán comparadas con las versiones equivalentes en R estándar.
Obteniendo subconjunto de datos usando filter()
Así como las opciones básicas de R para obtener subconjuntos de datos a través de expresiones condicionales como el uso de corchetees doblesdf[,], filter() crear un subconjunto de datos basado en algún criterio definido por el usuario.
Supongamos que se desea crear un subconjunto de diamantes diamonds manteniendo todos aquellos de color D,E o F con un corte de calidad (cut quality) Premium’ or ‘Ideal’ y un peso de más de 3 carat. Una forma estándar muy básica sería:
Usando filter() notemos que no se requiere repetir el nombre del data frame:
library(dplyr)##
## Attaching package: 'dplyr'## The following objects are masked from 'package:stats':
##
## filter, lag## The following objects are masked from 'package:base':
##
## intersect, setdiff, setequal, unionfilter(diamonds, carat > 3 &
cut %in% c("Premium", "Ideal") &
color %in% c("D", "E", "F"))## # A tibble: 2 x 10
## carat cut color clarity depth table price x y z
## <dbl> <ord> <ord> <ord> <dbl> <dbl> <int> <dbl> <dbl> <dbl>
## 1 3.01 Premium F I1 62.2 56 9925 9.24 9.13 5.73
## 2 3.05 Premium E I1 60.9 58 10453 9.26 9.25 5.66
Seleccinando columnas usando select()
dplyr::select(diamonds, carat, cut, color, clarity)## # A tibble: 53,940 x 4
## carat cut color clarity
## <dbl> <ord> <ord> <ord>
## 1 0.23 Ideal E SI2
## 2 0.21 Premium E SI1
## 3 0.23 Good E VS1
## 4 0.29 Premium I VS2
## 5 0.31 Good J SI2
## 6 0.24 Very Good J VVS2
## 7 0.24 Very Good I VVS1
## 8 0.26 Very Good H SI1
## 9 0.22 Fair E VS2
## 10 0.23 Very Good H VS1
## # ... with 53,930 more rowsNótese que tanto el uso de c() para combinar los nombres de varias columnas como el uso de corchetes dobles se vuelve obsoleto. Además es posible omitir el criterio de conteo de columnas (2:5) y expresar la selección de columnas contiguas a través de los nombres.
dplyr::select(diamonds, carat:clarity, price)## # A tibble: 53,940 x 5
## carat cut color clarity price
## <dbl> <ord> <ord> <ord> <int>
## 1 0.23 Ideal E SI2 326
## 2 0.21 Premium E SI1 326
## 3 0.23 Good E VS1 327
## 4 0.29 Premium I VS2 334
## 5 0.31 Good J SI2 335
## 6 0.24 Very Good J VVS2 336
## 7 0.24 Very Good I VVS1 336
## 8 0.26 Very Good H SI1 337
## 9 0.22 Fair E VS2 337
## 10 0.23 Very Good H VS1 338
## # ... with 53,930 more rowsNote: otras funciones útiles para definir el criterio de selección: starts_with(),
ends_with(), matches() and contains().
Pipelining
Ahora, supongamos que se desea seleccionar algunas columnas a partir del subconjunto de datos creado anteriormente. Normalmente usaríamos dos pasos con resultados intermedios innecesarios:
## first, create subset
diamonds_sub <- filter(diamonds, carat > 3 &
cut %in% c("Premium", "Ideal") &
color %in% c("D", "E", "F"))
## second, select columns
select(diamonds_sub, carat:clarity, price)o quizas funciones anidadas que usualmnete son difíciles de interpretar:
## all-in-one nested solution
select(
filter(diamonds, carat > 3 &
cut %in% c("Premium", "Ideal") &
color %in% c("D", "E", "F")),
carat:clarity, price
)dplyr introduce el operador %>% el cual creado para interconectar los pasos de procesamiento en los datos eliminando el uso de variables intermedias o funciones anidadas como se ilustró anteriormente. Podemos pensar en %>% como una traducción de “entonces” o “luego” que conecta dos partes de una oración:
diamonds %>%
filter(carat > 3 &
cut %in% c("Premium", "Ideal") &
color %in% c("D", "E", "F")) %>%
dplyr::select(carat:clarity, price)## # A tibble: 2 x 5
## carat cut color clarity price
## <dbl> <ord> <ord> <ord> <int>
## 1 3.01 Premium F I1 9925
## 2 3.05 Premium E I1 10453Este encadenamiento se vuelve aún más útil caundo se efectúan múltiples operaciones a la vez, reduciendo el código y haciéndolo fácil de leer e interpretar. La analogía es la lectura de la página de un libro, que se efectúa de arriba-abajo e izquierda-derecha.
Reordenar filas usando arrange()
arrange() facilita la rapidez en el reordenamiento de filas basada en criterios multi-columna.
Asumamos un ejemplo con nuestro data set diamonds. Se quiere obtener un listado del subconjunto de datos referentes a los atributos de color, precio y carat (color, price, carat) ordenados en base a los siguientes criterios.
- De acuerdo al color con el mejor al inicio (color D)
- Sobre el orden anterior, es decir sobre diamantes de igual color ordenarlos con el menor precio al inicio
- Finalmente de cada conjunto de color y precio iguales, se desea ordenar sus pesos de manera descendente.
La forma trivial:
Usando la librería dplyr:
diamonds %>%
dplyr::select(color, price, carat) %>%
arrange(color, price, desc(carat))## # A tibble: 53,940 x 3
## color price carat
## <ord> <int> <dbl>
## 1 D 357 0.23
## 2 D 357 0.23
## 3 D 361 0.32
## 4 D 362 0.23
## 5 D 367 0.24
## 6 D 367 0.20
## 7 D 367 0.20
## 8 D 367 0.20
## 9 D 373 0.24
## 10 D 373 0.23
## # ... with 53,930 more rows
Añadir nuevas columnas usando mutate()
mutate() permite añadir nuevas variables a un data frame existente. Además tiene la ventaja de poder utilizar las columnas (variables) recién creadas, así:
diamonds %>%
dplyr::select(color, carat, price) %>%
mutate(ppc = price / carat,
ppc_rnd = round(ppc, 2))## # A tibble: 53,940 x 5
## color carat price ppc ppc_rnd
## <ord> <dbl> <int> <dbl> <dbl>
## 1 E 0.23 326 1417.391 1417.39
## 2 E 0.21 326 1552.381 1552.38
## 3 E 0.23 327 1421.739 1421.74
## 4 I 0.29 334 1151.724 1151.72
## 5 J 0.31 335 1080.645 1080.65
## 6 J 0.24 336 1400.000 1400.00
## 7 I 0.24 336 1400.000 1400.00
## 8 H 0.26 337 1296.154 1296.15
## 9 E 0.22 337 1531.818 1531.82
## 10 H 0.23 338 1469.565 1469.57
## # ... with 53,930 more rows
Resumir valores usando summarise()
Asumamos que se desea calcular el mínimo, media y máximo precio por color de diamante. Usando la versión muy conocida de R estándar:
Ahora, usando la solución de la función group_by, la cual es sumamente útil para trabajar sobre subconjuntos de datos:
diamonds %>%
group_by(color) %>%
summarise(MIN = min(price), MEAN = mean(price), MAX = max(price))## # A tibble: 7 x 4
## color MIN MEAN MAX
## <ord> <dbl> <dbl> <dbl>
## 1 D 357 3169.954 18693
## 2 E 326 3076.752 18731
## 3 F 342 3724.886 18791
## 4 G 354 3999.136 18818
## 5 H 337 4486.669 18803
## 6 I 334 5091.875 18823
## 7 J 335 5323.818 187102.9 Funciones
2.9.1 Funciones implementadas en R
Existen en R funciones muy comunes como mean, min o max. Por ejemplo la función round sirve para redondear un número, la función factorial para calcular el factorial, etc. En general se hace uso de una función escribiendo (invocando) el nombre de la función y entre paréntesis () el dato o datos sobre los cuales se desea aplicar la función. Ejm:
round(3.1415)## [1] 3factorial(3)## [1] 6El valor o valores que se “pasan” dentro de la función se denomina argumento de la función. El argumento puede ser cualquier objeto R o incluso el resultado de otra función R. En el último caso la evaluación de las funciones se hace secuencialmente desde la más interna hasta la llamada más externa. Así en el ejemplo siguiente, R evalúa inicialmente la creación del vector c(1,2,3,4), luego evalúa la media a través de mean(c(1,2,3,4)) y finalmente redondea el resultado que se ha obtenido de esta última operación.
round(mean(c(1,2,3,4)))## [1] 2Una función puede recibir tantos argumentos como se desee, siempre y cuando estos se encuentren separados con una coma ,. Normalmente no se conoce a priori cuales son los argumentos que acepta una función en particular, sin embargo es posible obtener esta información a través del comando args(), donde el nombre de la función de interés se coloca entre paréntesis, así: args(min). Nótese, que no todos los argumentos se visualizan, pero sí los más frecuentes. En tanto que para las funciones básicas como args(round) o args(rnorm) presentan todos sus argumentos.
args(round)## function (x, digits = 0)
## NULLargs(rnorm)## function (n, mean = 0, sd = 1)
## NULLargs(min)## function (..., na.rm = FALSE)
## NULLEn el caso de la función de redondeo round(3.5), el argumento digits no se ha “seteado”, es decir no se ha incluido en la llamada de la función. En este caso, R mantiene el valor por defecto que se incluye en la función, es decir redondeo a 0 dígitos decimales. A estos argumentos se les conoce como opcionales ya que tienen un valor predefinido.
Importante: es siempre recomendable incluir en la llamada de la función,el nombre del argumento por dos razones principales. Primero, a medida que se incrementa el número de argumentos es más difícil recordar (o interpretar en el código de alguien más) a que hace referencia cada valor. Segundo, el orden en el que colocan los argumentos es definido y R interpretará los valores asignados al argumento en dicho orden. ¿Cuál de los dos ejemplos es más fácil de interpretar?
rnorm(5, 3, 1)## [1] 3.295735 1.606609 3.500572 2.014032 2.472407rnorm(5, mean = 3, sd = 1) ## [1] 3.488812 1.558758 3.808533 3.982415 4.5350962.9.2 Funciones creadas por el usuario
¿Para qué crear una función?
- Para reutilizar código fuente en R, es decir cuando vamos a necesitar ejecutar la misma función en distintos programas o en diversos lugares de un mismo programa.
- Para organizar el código fuente. Es más sencillo inspeccionar una sección de código y asegurarse que el funcionamiento es el correcto en comparación con inspeccionar todo mi código fuente.
Primero, vamos a crear una función para simular el proceso de lanzar dos dados y obtener el resultado de la suma resultante. Vamos a guardar la codificación de esta función en un archivo llamado funcion_roll.R.
roll <- function() {
die <- 1:6
dice <- sample(die, size = 2, replace = TRUE)
sum(dice)
}
roll()## [1] 8roll## function() {
## die <- 1:6
## dice <- sample(die, size = 2, replace = TRUE)
## sum(dice)
## }Observe que la función definida roll no tiene argumentos de entrada, ahora modificaremos el código para incluir un argumento con valor por “default” que simula el número de caras del dado; a esta función la llamaremos roll2. Se omite la línea die <- 1:6, pero ahora el número de caras del dado está dado por el argumento bones.
roll2 <- function(bones = 1:6) {
dice <- sample(bones, size = 2, replace = TRUE)
sum(dice)
}
roll2()## [1] 6roll2## function(bones = 1:6) {
## dice <- sample(bones, size = 2, replace = TRUE)
## sum(dice)
## }###Componentes de una función
Generalizando la estructura para la creación de una función (ilustración tomada del libro “Hands on Programming with R”).
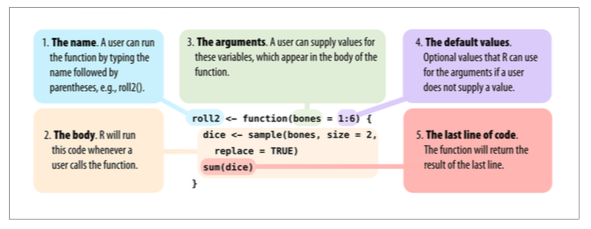
Esquema de una función
Ya que sabemos como definir una función, vamos a crear nuestra propia función. Considerando que la función scan(n=1) se utiliza para leer un (1) valor por teclado, vamos a escribir el código en R para una función que, ingresado un valor por teclado, indique al usuario a través de un mensaje si este número es mayor, menor o igual a 0.
valor <- scan(n=1)En los ejemplos ilustrados hasta ahora hemos creado la función y la hemos utilizado en un mismo script (programa) R. Sin embargo, es posible que las funciones creadas por un usuario se encuentren en distintos archivos. En este caso, al igual que la invocación de una librería library(rgdal), es necesario cargar en memoria de R el código de creación y ejecución de estas funciones. Para ello, basta con usar el comando source("nombre_archivo_funcion.R"). Según el ejemplo anterior de la función roll(), la llamada se efectúa como source("funcion_roll.R").
2.10 Ejercicios
Crear un script que permita cargar el dataset iris (df.plantas <- iris)
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosay calcular, para el data.frame df.plantas, lo siguiente:
- Media de la anchura de sépalo agrupada por especie.
- Media de la anchura de sépalo agrupada por especie cuando la anchura del pétalo es superior a 2.6.
- Media de la longitud del sépalo cuando la anchura del sépalo es inferior a 3.5.
- Media de la longitud del sépalo cuando la anchura del sépalo es inferior a 3.5 y la anchura del pétalo es superior a 0.2.