7 Les data frames
Idéalement, pour un data scientist, chaque jeu de données (en anglais dataset ou data set) à analyser doit être sous forme d’un tableau où chaque ligne correspondant à une observation (individu) et chaque colonne à une caractéristique (variable).
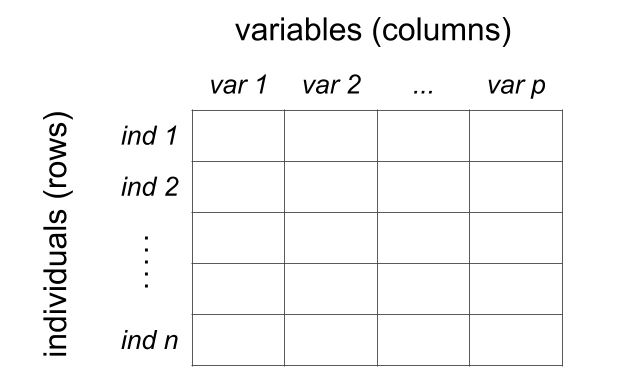
En R, les data frames sont les structures qui permettent de stoker de tels jeux de données. Ce sont les objets les plus courants et les plus importants. Les colonnes/variables d’un data frame peuvent être de différents types ce qui offre plus de flexibilité que les matrices.
Pour créer un data frame, nous allons commencer par créer les vecteurs qui constitueront les colonnes de notre future data frame.
taille <- c(167, 192, 173, 174, 172, 167, 171, 185, 163, 170) # mesures en cm
poids <- c(86, 74, 83, 50, 78, 66, 66, 51, 50, 55) # mesures en Kg
prog <- c("Bac+2", "Bac", "Master", "Bac", "Bac", "DEA", "Doctorat", NA, "Certificat", "DES") # programme d'étude
sexe <- c("H", "H", "F", "H", "H", "H", "F", "H", "H", "H")Maintenant il suffit d’utiliser la fonction data.frame() pour construire notre data frame.
height weight prog sexe X11.20
1 167 86 Bac+2 H 11
2 192 74 Bac H 12
3 173 83 Master F 13
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 DEA H 16
7 171 66 Doctorat F 17
8 185 51 <NA> H 18
9 163 50 Certificat H 19
10 170 55 DES H 20Les colonnes d’un data frame sont toujours nommées. Ils héritent automatiquement les noms des vecteurs fournis (voir la 3e et la 4e colonne de mydata). Au souhait, l’utilisateur peut attribuer d’autres noms (voir la 1re et la 2e colonne). Dans le cas où un vecteur n’a pas de nom, R attribue lui même un nom (voir la 5e colonne). Il est conseillé de ne pas laisser R faire, car cela résulte souvent en des noms non pertinents.
Les lignes quant à elles sont automatiquement nommées/numérotées par ordre (1 = première ligne, 2 = deuxième ligne,…). En pratique, on a rarement besoin de modifier ces noms.
Il faut savoir qu’un data frame se pressente sous forme d’une matrice, mais qu’en réalité (en interne) il s’agit d’une liste particulière dont les éléments ne peuvent être que des vecteurs de même longueur. Nous pouvons voir cela, en partie, en examinant la structure et les caractéristiques de notre objet mydata.
[1] 5
[1] 10 5
[1] "list"'data.frame': 10 obs. of 5 variables:
$ height: num 167 192 173 174 172 167 171 185 163 170
$ weight: num 86 74 83 50 78 66 66 51 50 55
$ prog : chr "Bac+2" "Bac" "Master" "Bac" ...
$ sexe : chr "H" "H" "F" "H" ...
$ X11.20: int 11 12 13 14 15 16 17 18 19 20$names
[1] "height" "weight" "prog" "sexe" "X11.20"
$class
[1] "data.frame"
$row.names
[1] 1 2 3 4 5 6 7 8 9 10Dans RStudio, on peut visionner un data frame de façon interactive. Pour cela, il suffit de taper
ou de cliquer sur la petite icône en forme de tableau située à droite de la ligne d’un tableau de données dans l’onglet Environment du quadrant supérieur droit.
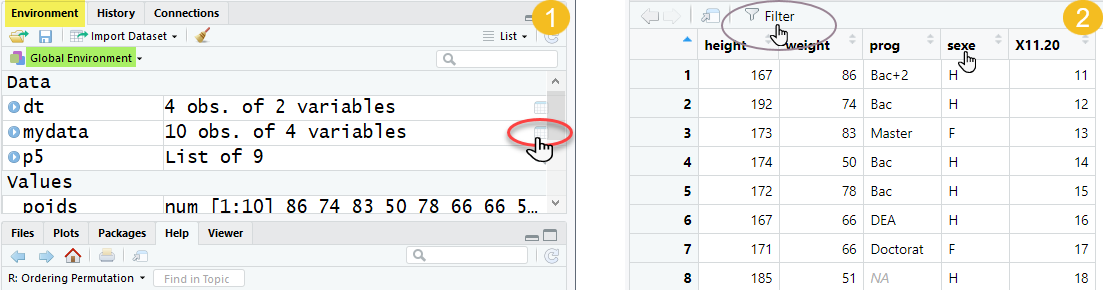
Il est possible de trier les données selon une variable en cliquant sur le nom de cette dernière. Il y a également un champs de recherche et un bouton “Filter” donnant accès à des options de filtrage.
Avant de contunuez, sachez que de très nombreuses opérations, vues dans les chapitres précédents, pouvant être effectuées sur les matrices ou les listes peuvent aussi être effectuées sur les data frames. Pour éviter les redondances, nous allons nous focaliser par la suite sur les aspects les plus importants.
7.1 Accéder aux éléments d’un data frame
En plus des techniques vues précédemment (voir les deux derniers chapitres), nous pouvons utiliser ici une nouvelle fonction très pratique appelée subset(); voire le Help de cette fonction.
Sélectionner certaines colonnes
+ Par numéro/position
mydata[c(2, 3)] # colonnes 2 et 3
mydata[, c(2, 3)] # idem
mydata |> subset(select = c(2, 3)) # idem weight prog
1 86 Bac+2
2 74 Bac
3 83 Master
4 50 Bac
5 78 Bac
6 66 DEA
7 66 Doctorat
8 51 <NA>
9 50 Certificat
10 55 DESmydata[-c(2, 3)] # toutes les colonnes sauf 2 et 3
mydata[, -c(2, 3)] # idem
mydata |> subset(select = -c(2, 3)) # idem height sexe X11.20
1 167 H 11
2 192 H 12
3 173 F 13
4 174 H 14
5 172 H 15
6 167 H 16
7 171 F 17
8 185 H 18
9 163 H 19
10 170 H 20+ Par nom
mydata$weight # colonne "weight" (resultat = vecteur)
mydata["weight"] # colonne "weight" (resultat = data.frame)
mydata |> subset(select = "weight") # idem
mydata |> subset(select = weight) # idem weight
1 86
2 74
3 83
4 50
5 78
6 66
7 66
8 51
9 50
10 55 height sexe X11.20
1 167 H 11
2 192 H 12
3 173 F 13
4 174 H 14
5 172 H 15
6 167 H 16
7 171 F 17
8 185 H 18
9 163 H 19
10 170 H 20Sélectionner certaines lignes
+ Par numéro/position
height weight prog sexe X11.20
2 192 74 Bac H 12
3 173 83 Master F 13 height weight prog sexe X11.20
1 167 86 Bac+2 H 11
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 DEA H 16
7 171 66 Doctorat F 17
8 185 51 <NA> H 18
9 163 50 Certificat H 19
10 170 55 DES H 20+ Par condition logique
En pratique, il arrive très souvent que l’on souhaite soustraire un sous-ensemble d’observations (lignes) qui remplissent une certaine condition. Voici quelques exemples.
mydata[mydata$sexe == "H", ] # sélectionner uniquement les hommes
mydata |> subset(subset = sexe == "H") # idem
mydata |> subset(sexe == "H") # idem height weight prog sexe X11.20
1 167 86 Bac+2 H 11
2 192 74 Bac H 12
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 DEA H 16
8 185 51 <NA> H 18
9 163 50 Certificat H 19
10 170 55 DES H 20# les hommes dont le poids est inférieur au poids moyen
mydata |> subset(sexe == "H" & weight < mean(weight)) height weight prog sexe X11.20
4 174 50 Bac H 14
8 185 51 <NA> H 18
9 163 50 Certificat H 19
10 170 55 DES H 20# idem, mais en plus nous conservons uniquement les colonnes height et prog
mydata |> subset(sexe == "H" & weight < mean(weight), select = c(height, prog)) height prog
4 174 Bac
8 185 <NA>
9 163 Certificat
10 170 DES7.2 Ajouter/transformer des colonnes
En plus des techniques vues précédemment (voir les deux derniers chapitres), nous pouvons utiliser ici une nouvelle fonction très pratique appelée transform(); voire le Help de cette fonction.
Ajouter de nouvelles variables
Pour commencer, imaginez que nous aimerions ajouter les deux variables suivantes à notre data frame mydata .
nom <- c("Benjamin", "Hugo", "Emma", "Alex", "Tom", "Axel", "Alice", "Martin", "Robin", "Enzo")
grade <- c("A", "A", "C", "B", "B", "B", "C", "A", "A", "A")Pour cela, nous pouvons utiliser l’une des commandes suivantes
mydata$prenom <- nom ; mydata$grade <- grade # méthode 1
mydata <- cbind(mydata, prenom = nom, grade = grade) # méthode 2
mydata <- mydata |> transform(prenom = nom, grade = grade) # méthode 3 height weight prog sexe X11.20 prenom grade
1 167 86 Bac+2 H 11 Benjamin A
2 192 74 Bac H 12 Hugo A
3 173 83 Master F 13 Emma C
4 174 50 Bac H 14 Alex B
5 172 78 Bac H 15 Tom B
6 167 66 DEA H 16 Axel B
7 171 66 Doctorat F 17 Alice C
8 185 51 <NA> H 18 Martin A
9 163 50 Certificat H 19 Robin A
10 170 55 DES H 20 Enzo ATransformer des variables existantes
Supposons maintenant que nous voulons transformer les variables sexe et grade en facteur. Cela peut se faire comme ceci
mydata$sexe <- factor(mydata$sexe); mydata$grade <- factor(mydata$grade)
mydata <- mydata |> transform(sexe = factor(sexe), grade = factor(grade)) # idemNous pouvons vérifier que les colonens/varaibles sexe et grade sont effectivement des facteurs en examinant la structure des mydata.
'data.frame': 10 obs. of 7 variables:
$ height: num 167 192 173 174 172 167 171 185 163 170
$ weight: num 86 74 83 50 78 66 66 51 50 55
$ prog : chr "Bac+2" "Bac" "Master" "Bac" ...
$ sexe : Factor w/ 2 levels "F","H": 2 2 1 2 2 2 1 2 2 2
$ X11.20: int 11 12 13 14 15 16 17 18 19 20
$ prenom: chr "Benjamin" "Hugo" "Emma" "Alex" ...
$ grade : Factor w/ 3 levels "A","B","C": 1 1 3 2 2 2 3 1 1 1Cela est important, car, comme expliquées précédemment, certaines fonctions réservent un traitement particulier aux facteurs. Par exemple, voici ce que la fonction générique summary() donne à présent (regardez la colonne “sexe” et “grade” ci-dessous).
height weight prog sexe X11.20
Min. :163 Min. :50.0 Length:10 F:2 Min. :11.0
1st Qu.:168 1st Qu.:52.0 Class :character H:8 1st Qu.:13.2
Median :172 Median :66.0 Mode :character Median :15.5
Mean :173 Mean :65.9 Mean :15.5
3rd Qu.:174 3rd Qu.:77.0 3rd Qu.:17.8
Max. :192 Max. :86.0 Max. :20.0
prenom grade
Length:10 A:5
Class :character B:3
Mode :character C:2
Le plus souvent, la fonction transform() est utilisée pour ajouter de nouvelles variables à partir de ceux qui existent déjà. Comme exemple, considérons le cas où nous voulons (1) créer la variable height.m = la taille (height) en m, (2) transformer le poids (weight) en g, (3) supprimer la variable height, et (4) créer la variable \(imc=taille/poids^2\) (indice de masse corporelle) habituellement exprimée en Kg/m^2.
# méthode 1
mydata |> transform(height.m = 0.01 * height, weight = weight * 1000,
height = NULL, imc = weight / (height / 100)^2)
# méthode 2
mydata |> transform(height.m = 0.01 * height, weight = weight * 1000, height = NULL) |>
transform(imc = (weight / 1000) / height.m^2) weight prog sexe X11.20 prenom grade height.m imc
1 86000 Bac+2 H 11 Benjamin A 1.67 30.8
2 74000 Bac H 12 Hugo A 1.92 20.1
3 83000 Master F 13 Emma C 1.73 27.7
4 50000 Bac H 14 Alex B 1.74 16.5
5 78000 Bac H 15 Tom B 1.72 26.4
6 66000 DEA H 16 Axel B 1.67 23.7
7 66000 Doctorat F 17 Alice C 1.71 22.6
8 51000 <NA> H 18 Martin A 1.85 14.9
9 50000 Certificat H 19 Robin A 1.63 18.8
10 55000 DES H 20 Enzo A 1.70 19.0Pour comprendre le syntaxe ci-dessous, il faut savoir qu’une variable nouvellement créée, via la fonction transform(), ne peut pas être immédiatement utilisée. En effet, la fonction transform() ne reconnait que les variables/colonnes originales (et leur valeurs) telles que stockées dans le data frame original/fourni. Ainsi le code suivant est faux et renvoie une erreur.
mydata |> transform(height.m = 0.01 * height, weight = weight * 1000, height = NULL, imc = (weight / 1000) / height.m^2)Error in eval(substitute(list(...)), `_data`, parent.frame()): object 'height.m' not found7.3 Quelques fonctions/manipulations utiles
+ Obtenir un aperçu/résumé
height weight prog sexe X11.20 prenom grade
1 167 86 Bac+2 H 11 Benjamin A
2 192 74 Bac H 12 Hugo A
3 173 83 Master F 13 Emma C
4 174 50 Bac H 14 Alex B
5 172 78 Bac H 15 Tom B
6 167 66 DEA H 16 Axel B height weight prog sexe X11.20 prenom grade
5 172 78 Bac H 15 Tom B
6 167 66 DEA H 16 Axel B
7 171 66 Doctorat F 17 Alice C
8 185 51 <NA> H 18 Martin A
9 163 50 Certificat H 19 Robin A
10 170 55 DES H 20 Enzo A height weight prog sexe X11.20
Min. :163 Min. :50.0 Length:10 F:2 Min. :11.0
1st Qu.:168 1st Qu.:52.0 Class :character H:8 1st Qu.:13.2
Median :172 Median :66.0 Mode :character Median :15.5
Mean :173 Mean :65.9 Mean :15.5
3rd Qu.:174 3rd Qu.:77.0 3rd Qu.:17.8
Max. :192 Max. :86.0 Max. :20.0
prenom grade
Length:10 A:5
Class :character B:3
Mode :character C:2
+ Statistiques calculées par groupe
Il arrive souvent qu’on veuille calculer des statistiques descriptives tels que le minimum, la moyenne, la médiane, d’une variable pour un sous-ensemble (regroupement ) d’individus d’un jeu de données. Cela est possible garce à la fonction aggregate(). Nous allons parler ici de la version “formula” dont le syntaxe (de base) est donné par
où formula est une formule de type y ~ x, où y est la variable d’intérêt et x le facteur de découpage, data est le jeu de données auquel appartient x et y et Fun le nom de la fonction à utiliser pour les calculs. L’argument ... indique qu’on peut passer des arguments supplémentaires à Fun. Il est fort conseillé, dans ce cas, de nommer explicitement ces arguments. Voici quelques exemples.
sexe height
1 F 172
2 H 174# quantiles de la taille par sexe et grade
aggregate(height ~ sexe + grade, data = mydata, FUN = quantile) sexe grade height.0% height.25% height.50% height.75% height.100%
1 H A 163 167 170 185 192
2 H B 167 170 172 173 174
3 F C 171 172 172 172 173# médiane de la taille par sexe
aggregate(height ~ sexe, data = mydata, FUN = quantile, probs = 0.5) sexe height
1 F 172
2 H 171# médiane de la taille et le poids par sexe et grade
aggregate(cbind(height, weight) ~ sexe + grade, data = mydata, FUN = quantile, probs = 0.5) sexe grade height weight
1 H A 170 55.0
2 H B 172 66.0
3 F C 172 74.5+ Dénombrer les individus
On a déjà vu la fonction table() qu’on peut utiliser pour faire du comptage.
F H
2 8 Le même résultat est produit avec la fonction xtabs() dont les arguments principaux son formula et data.
sexe
F H
2 8 Cette fonction s’avère plus utile lorsqu’il s’agit de construire des tableaux de comptages croisés à partir de plusieurs variables. Voici un exemple.
sexe
grade F H
A 0 5
B 0 3
C 2 0+ Renommer des ligens/colonnes
height weight program sexe id prenom grade
1th 167 86 Bac+2 H 11 Benjamin A
2th 192 74 Bac H 12 Hugo A
3th 173 83 Master F 13 Emma C
4th 174 50 Bac H 14 Alex B
5th 172 78 Bac H 15 Tom B
6th 167 66 DEA H 16 Axel B
7th 171 66 Doctorat F 17 Alice C
8th 185 51 <NA> H 18 Martin A
9th 163 50 Certificat H 19 Robin A
10th 170 55 DES H 20 Enzo A+ Ordonner les lignes
height weight program sexe id prenom grade
9th 163 50 Certificat H 19 Robin A
1th 167 86 Bac+2 H 11 Benjamin A
6th 167 66 DEA H 16 Axel B
10th 170 55 DES H 20 Enzo A
7th 171 66 Doctorat F 17 Alice C
5th 172 78 Bac H 15 Tom B
3th 173 83 Master F 13 Emma C
4th 174 50 Bac H 14 Alex B
8th 185 51 <NA> H 18 Martin A
2th 192 74 Bac H 12 Hugo A height weight program sexe id prenom grade
2th 192 74 Bac H 12 Hugo A
8th 185 51 <NA> H 18 Martin A
4th 174 50 Bac H 14 Alex B
3th 173 83 Master F 13 Emma C
5th 172 78 Bac H 15 Tom B
7th 171 66 Doctorat F 17 Alice C
10th 170 55 DES H 20 Enzo A
1th 167 86 Bac+2 H 11 Benjamin A
6th 167 66 DEA H 16 Axel B
9th 163 50 Certificat H 19 Robin A height weight program sexe id prenom grade
9th 163 50 Certificat H 19 Robin A
6th 167 66 DEA H 16 Axel B
1th 167 86 Bac+2 H 11 Benjamin A
10th 170 55 DES H 20 Enzo A
7th 171 66 Doctorat F 17 Alice C
5th 172 78 Bac H 15 Tom B
3th 173 83 Master F 13 Emma C
4th 174 50 Bac H 14 Alex B
8th 185 51 <NA> H 18 Martin A
2th 192 74 Bac H 12 Hugo A+ Empiler des collones
Devoir empiler les colonnes d’un data frame arrive fréquemment en R, principalement pour préparer les données avant de les fournir à d’autres fonctions (graphiques). Voici un exemples.
dt <- data.frame(
groupe = c("A", "A", "B", "B", "C", "C"),
essaye = c(1, 2, 1, 2, 1, 2),
traitement_1 = c(4, 2, 7, 3, 5, 6),
traitement_2 = c(8, 5, 0, 7, 7, 8),
traitement_3 = c(0, 5, 6, 1, 3, 4))
stack(dt) values ind
1 A groupe
2 A groupe
3 B groupe
4 B groupe
5 C groupe
6 C groupe
7 1 essaye
8 2 essaye
9 1 essaye
10 2 essaye
11 1 essaye
12 2 essaye
13 4 traitement_1
14 2 traitement_1
15 7 traitement_1
16 3 traitement_1
17 5 traitement_1
18 6 traitement_1
19 8 traitement_2
20 5 traitement_2
21 0 traitement_2
22 7 traitement_2
23 7 traitement_2
24 8 traitement_2
25 0 traitement_3
26 5 traitement_3
27 6 traitement_3
28 1 traitement_3
29 3 traitement_3
30 4 traitement_3 values ind
1 4 traitement_1
2 2 traitement_1
3 7 traitement_1
4 3 traitement_1
5 5 traitement_1
6 6 traitement_1
7 8 traitement_2
8 5 traitement_2
9 0 traitement_2
10 7 traitement_2
11 7 traitement_2
12 8 traitement_2
13 0 traitement_3
14 5 traitement_3
15 6 traitement_3
16 1 traitement_3
17 3 traitement_3
18 4 traitement_3 values ind
1 4 traitement_1
2 2 traitement_1
3 7 traitement_1
4 3 traitement_1
5 5 traitement_1
6 6 traitement_1
7 8 traitement_2
8 5 traitement_2
9 0 traitement_2
10 7 traitement_2
11 7 traitement_2
12 8 traitement_2
13 0 traitement_3
14 5 traitement_3
15 6 traitement_3
16 1 traitement_3
17 3 traitement_3
18 4 traitement_3 groupe essaye values ind
1 A 1 4 traitement_1
2 A 2 2 traitement_1
3 B 1 7 traitement_1
4 B 2 3 traitement_1
5 C 1 5 traitement_1
6 C 2 6 traitement_1
7 A 1 8 traitement_2
8 A 2 5 traitement_2
9 B 1 0 traitement_2
10 B 2 7 traitement_2
11 C 1 7 traitement_2
12 C 2 8 traitement_2
13 A 1 0 traitement_3
14 A 2 5 traitement_3
15 B 1 6 traitement_3
16 B 2 1 traitement_3
17 C 1 3 traitement_3
18 C 2 4 traitement_37.4 Variantes de data frames
Au cours de temps, ont vu se développer des variantes “plus moderne” de data frames. Parmi les structures les plus connues/utilisées, on trouve les data tables et les tibbles, chacune a ses avantages et ses inconvénients….
Les tibbles font partie du packages tibble lui-même faisant partie de la très fameuse collection de packages tidyverse. Pour pouvoir créer ou manipuler des tibbles, vous devez installer tibble (ou tidyverse).
La création d’un tibble est similaire à celle d’un data frame.
taille <- c(167, 192, 173, 174, 172, 167, 171, 185, 163, 170)
poids <- c(86, 74, 83, 50, 78, 66, 66, 51, 50, 55)
prog <- c("Bac+2", "Bac", "Master", "Bac", "Bac", "DEA", "Doctorat", NA, "Certificat", "DES")
sexe <- c("H", "H", "F", "H", "H", "H", "F", "H", "H", "H")# A tibble: 10 × 4
height weight prog sexe
<dbl> <dbl> <chr> <chr>
1 167 86 Bac+2 H
2 192 74 Bac H
3 173 83 Master F
4 174 50 Bac H
5 172 78 Bac H
6 167 66 DEA H
7 171 66 Doctorat F
8 185 51 <NA> H
9 163 50 Certificat H
10 170 55 DES H Remarquez que lorsque l’on affiche un tibble en évaluant son nom dans la console :
- R affiche un commentaire indiquant la nature de l’objet (tibble) et ces dimensions (ici 10 x 4, pur 10 lignes et 4 colonnes)
- R n’affiche que les 10-20 premières lignes et l’ensemble des colonnes qui peuvent s’afficher (adéquatement) sur l’écran ;
- Le type de chaque variable/colonne visible est décrit, en abrégé, juste après les noms de colonnes.
De plus, les lignes ne sont pas, proprement dit, nommées, elles sont simplement numérotées. Et il est possible de créer un tible ligne par ligne à l’aide de la fonction tribble().
tribble(
~height, ~weight, ~prog, ~sexe,
67, 86, "Bac+2", "H",
192, 74, "Bac", "H",
173, 83, "Master", "F"
)# A tibble: 3 × 4
height weight prog sexe
<dbl> <dbl> <chr> <chr>
1 67 86 Bac+2 H
2 192 74 Bac H
3 173 83 Master F Mais malgré ces différences, un tibble et un data frame se manipulent exactement de la même manière. On peut donc appliquer sur un tibble tout ce que l’on a appris avec les data frames. Aussi, il facile de convertir un tible en data frame et vice versa.
'data.frame': 10 obs. of 4 variables:
$ height: num 167 192 173 174 172 167 171 185 163 170
$ weight: num 86 74 83 50 78 66 66 51 50 55
$ prog : chr "Bac+2" "Bac" "Master" "Bac" ...
$ sexe : chr "H" "H" "F" "H" ...
tibble [10 × 4] (S3: tbl_df/tbl/data.frame)
$ height: num [1:10] 167 192 173 174 172 167 171 185 163 170
$ weight: num [1:10] 86 74 83 50 78 66 66 51 50 55
$ prog : chr [1:10] "Bac+2" "Bac" "Master" "Bac" ...
$ sexe : chr [1:10] "H" "H" "F" "H" ...7.5 Sources de données
Une analyse de données débute typiquement par l’acquisition de données qui peut se faire de différentes manières et sous différentes formes. Nous allons survoler (très brièvement) quelques façons de réaliser cette première étape.
Packages R
De très nombreuses packages viennent avec plusieurs jeux de données accessibles une fois le package chargé. C’est le cas, par exemple, du package de base datasets qui est automatiquement installé et chargé par R.
Pour avoir une liste complète de tous les données disponibles dans les packages chargés dans la session R en cours, tapez :
La sortie de cette commande varie en fonction des packages chargés. Par exemple, après avoir charger le package MASS, voici la sortie qu’on obtient
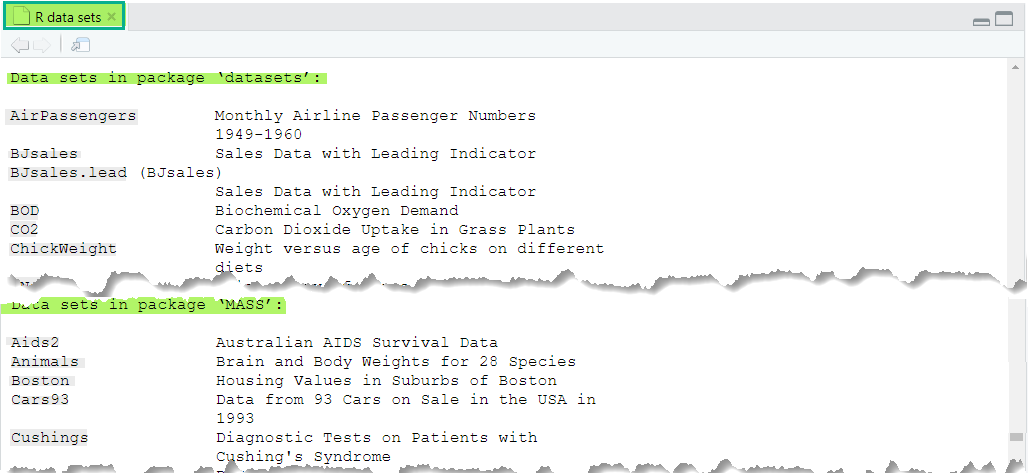
Il s’agit des noms suivis par une brève description pour chaque jeu de données. Si vous voulez voir uniquement les données disponible dans MASS, tapez
Parmi les noms affichés il y a, par exemple,Animals. Si vous tapez ce nom dans la console, vous verrez le contenu de ce data frame. Pour obtenir une description de ces données, tapez
Fichiers externes
Très souvent les données à analyser sont disponibles dans un fichier externe (local ou sur le web) sous divers formats : texte délimités (dont .txt, .csv, .dsv, etc.), JSON, EXCEL, etc. Nous aborderons ici uniqument le format texte qui est le plus rencontré en pratique.
Pour la suite de la démonstration, nous allons utiliser le fichier Med.csv qu’on vous invite à télécharger et sauvegarder sur votre machine. Voici un aperçu du contenu de ce fichier.
subject sex condition before after change
1 F placebo 10.1 6.9 -3.2
2 F placebo 6.3 4.2 -2.1
3 M aspirin 12.4 6.3 -6.1
4 F placebo 8.1 6.1 -2.0
5 M aspirin 15.2 9.9 -5.3
6 F aspirin 10.9 7.0 -3.9Pour importer ces données, nous allons utiliser la fonction read.csv(). Cette fonction accepte beaucoup d’arguments qui permettent de s’adapter à la nature de fichier à importer; voir le Help de cette fonction pour plus de détails. Parmi ces arguments il y a : (1) header : valeur logique (TRUE ou FALSE) pour la présence ou non d’un en-tête avec les noms des variables (par défaut TRUE), (2) sep : la maniéré dont les champs (variables) sont séparés (par défaut une virgule), et (3) dec: le séparateur décimal (par défaut un point).
Le seul argument obligatoire est le chemin d’accès au fichier à lire. Mais, comme déjà expliqué, il n’est pas nécessaire de spécifier le chemin complet si ce le fichier à lire se trouve dans votre Working directory. Si c’est le ce cas, pour charger “Med.csv” dans R, il suffit de taper
De là, on peut utiliser l’objet Med comme tout autre data frame en R.
RStudio fournit une interface graphique simple pour faciliter l’import d’un fichier. Pour cela, il suffit d’aller dans le menu File > Import Dataset > From Text (base), ou via l’onglet Environment, et indiquer l’emplacement du fichier et ces caractéristiques.
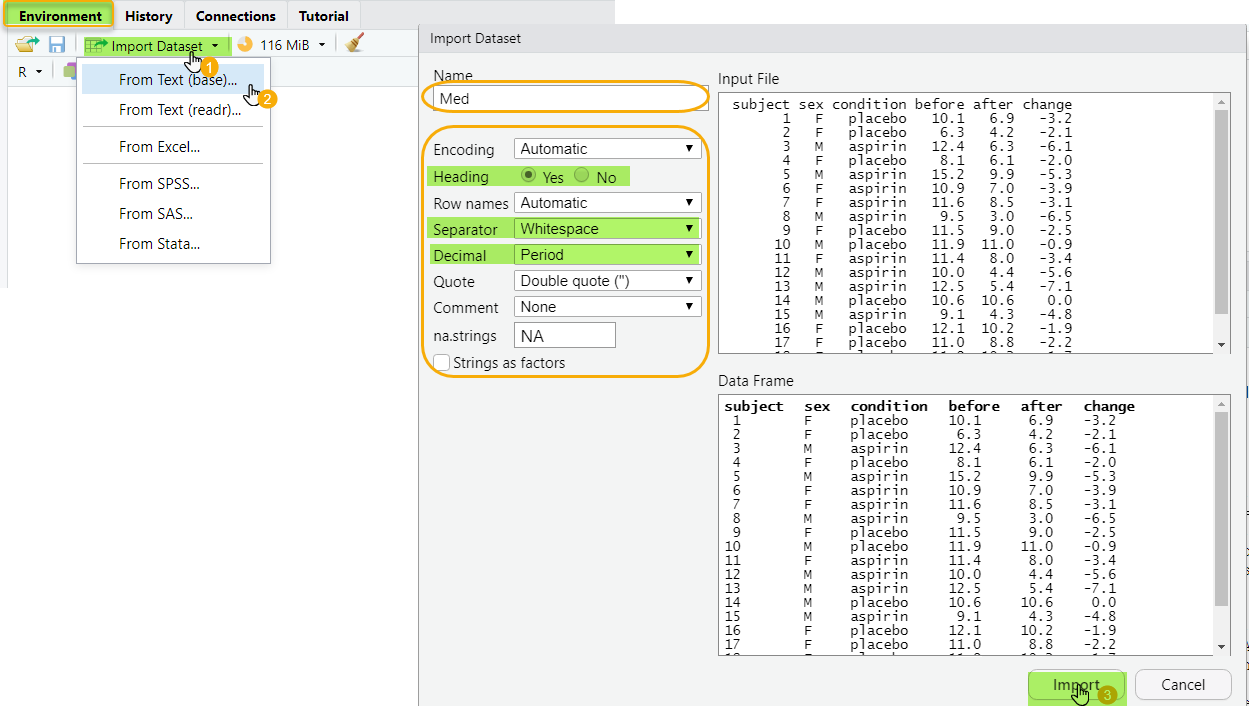
Le code généré et les données importées sont automatiquement affichés.
On peut aussi exporter des données à partir de R vers un fichier texte. Pour cela, il suffit d’utiliser la fonction write.csv().