Chapter 1 衛星データ解析基礎準備
1.1 APIでSentinel-2の画像を取得する①
必要となるパッケージをインストールします。#で始まる行はコメント文です。
install.packages("mapview")
install.packages("mapedit")
install.packages("sen2r")
install.packages("sf")1.2 APIでSentinel-2の画像を取得する②
まず利用するパッケージを呼び出します。
library(mapview)
library(mapedit)
library(sen2r)
library(sf)取得したい画像の場所を決定するために、座標情報を得る必要があります。今回はmapeditパッケージのeditMap()を利用して、その座標情報を取得しましょう。
m <- editMap()
sf::st_write(m, dsn = "m.geojson") # 取得した座標情報をgeojsonとして保存取得した領域が合っているか、mapviewパッケージを用いて確認しましょう。
mapview(m)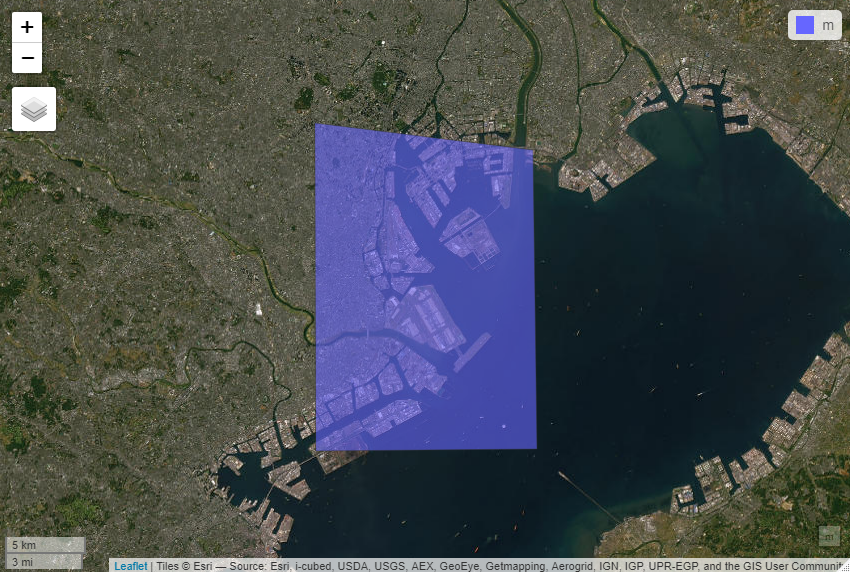
指定した領域であることを確認できたら、衛星データを取得します。RのSentinel-2のAPIであるsen2rパッケージを利用します。sen2rを利用するには事前の登録が必要となります。まずは、ユーザー情報を入力します。
username <- "*********"
password <- "*********"
write_scihub_login(username, password)
# APIの規約上、ESA登録後すぐには利用できません(1日~数日後には利用可能)。衛星データを取得します。「場所」「時間」「データの処理レベル」「被雲率」を指定しています。
products <- s2_list(spatial_extent = m,
time_interval = as.Date(c("2021-11-10", "2021-11-20")),
time_period = "full",
level = "L2A",
max_cloud = 100)取得した衛星データ数を確認します。今回の例では、2シーンあることがわかります。
length(products)雲がない画像を取得するため、被雲率が低い衛星データの行を確認しましょう。
products_gdf <- as.data.frame(products)
products_gdf_sorted <- products_gdf[order(products_gdf$clouds, decreasing = FALSE), ]
products_gdf_sorted # 並び替え結果を確認し、取得したい衛星データの行を確認被雲率が低い衛星データの行を確認できたら、取得したい衛星データをダウンロードしましょう。
s2_download(products[1], order_lta = TRUE) # s2_listの結果を格納した変数を利用1.3 対象領域の画像を表示する
terraパッケージを用いて、取得したSentinel-2のデータを確認します。今回は、Sentinel-2のBand4,3,2を合成したトゥルーカラー画像を作成しましょう。 まずは、先ほどダウンロードした衛星データのパスを用いてラスターデータとして読み込みます。格納先のパスに適宜変更してください。
b4 <- rast("..\\S2A_MSIL2A_20211113T012911_N0301_R074_T54SUE_20211113T040103.SAFE\\GRANULE\\L2A_T54SUE_A033390_20211113T012913\\IMG_DATA\\R10m\\T54SUE_20211113T012911_B04_10m.jp2")
b3 <- rast("..\\S2A_MSIL2A_20211113T012911_N0301_R074_T54SUE_20211113T040103.SAFE\\GRANULE\\L2A_T54SUE_A033390_20211113T012913\\IMG_DATA\\R10m\\T54SUE_20211113T012911_B03_10m.jp2")
b2 <- rast("..\\S2A_MSIL2A_20211113T012911_N0301_R074_T54SUE_20211113T040103.SAFE\\GRANULE\\L2A_T54SUE_A033390_20211113T012913\\IMG_DATA\\R10m\\T54SUE_20211113T012911_B02_10m.jp2")次に、取得した衛星データを一つの変数に統合します。その前に取得した衛星データのnameを変更しておきましょう。
#取得した衛星データのnameを変更
names(b4) <- "b4"
names(b3) <- "b3"
names(b2) <- "b2"
# 取得した衛星データを一つの変数に統合
rgb <- c(b4, b3, b2)取得した衛星データは関心領域以外も含んだ範囲のデータとなっていますので、関心領域のみを切り取る必要があります。その際、取得した衛星データのcrsはEPSG:32654となっていますので、EPSG:4326へと変更しておきます。
rgb_reproject <- project(rgb, "EPSG:4326") # rgbのcrsを一般的なEPSG:4326へと変更
rgb_crop <- crop(x = rgb_reproject, y = m)取得した衛星データをヒストグラムで表示してみます。右に裾の長い分布であることが分かります。
par(mfrow = c(1,3), mar = c(5, 5, 5, 1)) # 複数のヒストグラムの表示、各ヒストグラムの余白設定
hist(rgb_crop[,,1], col = "red", main = "Red")
hist(rgb_crop[,,2], col = "green", main = "Green")
hist(rgb_crop[,,3], col = "blue", main = "Blue")
dev.off() # ヒストグラムの設定を解除最後に、取得した衛星データをRGBで表示してみましょう。
plotRGB(rgb_crop, axes = TRUE, stretch = "lin")
1.4 STACを利用した衛星データの取得
STACを利用して衛星データを取得できるようになると、同じ手法で異なった衛星データを取得することが可能となり、解析する衛星データとして様々なものを取り扱えるようになります。詳しくは、「Pythonで学ぶ衛星データ解析基礎」等をご覧ください。
まず、STACの利用に必要なrstacパッケージをインストールします。
install.packages("rstac")rstacパッケージを呼び出します。
library(rstac)STACにはEarth SearchやPlanetary Computer等のAPIがカタログ化されており、そうしたAPIを利用することで同じ手法で異なった衛星データを取得することが可能となります。 今回は、Earth SearchのAPIを利用します。
search <- stac("https://earth-search.aws.element84.com/v0")指定の条件下でSTAC経由でSentinel-2のデータを取得します。関心領域のデータを取得するにはboundig boxの入力が必要なため、bounding boxを予め確認しておく必要があります。
m # 関心領域のbounding boxを確認
result <- search %>%
stac_search(collections = "sentinel-s2-l2a-cogs",
bbox = c(139.7049, 35.47745, 139.8573, 35.66065),
datetime = "2021-11-10/2021-11-20") %>%
post_request() %>%
items_filter(`eo:cloud_cover` < 100)取得した衛星データ数を確認します。今回の例では、先と同様なので2シーンあることがわかります。
result取得した衛星データの被雲率を確認します。コードが少し特殊ですが、以下のようになります。
result$features[[1]]$properties$`eo:cloud_cover`
result$features[[2]]$properties$`eo:cloud_cover`被雲率が低い方のデータを取得します。今回のデータでは被雲率は99%と0.02%でしたので、0.02%のデータを取得できるよう、10%でフィルターをかけました(0.02%のデータを取得できるのであれば何%でも構いません)。
result_designated <- result %>%
items_filter(`eo:cloud_cover` < 10)被雲率が低いデータを取得できたので、バンド4,3,2をそれぞれダウンロードします。
download_B04 <- result_designated %>%
assets_download(asset_names = "B04", overwrite = TRUE)
download_B03 <- result_designated %>%
assets_download(asset_names = "B03", overwrite = TRUE)
download_B02 <- result_designated %>%
assets_download(asset_names = "B02", overwrite = TRUE)ここから先は先と同様の手順でRGB画像を表示します。
# 取得した衛星データ(バンド2, 3, 4)を読み込み
stac_b04 <- rast("B04.tif")
stac_b03 <- rast("B03.tif")
stac_b02 <- rast("B02.tif")
# 取得した衛星データのnameを変更
names(stac_b4) <- "b4"
names(stac_b3) <- "b3"
names(stac_b2) <- "b2"
# 取得した衛星データを一つの変数に統合
stac_rgb <- c(stac_b04, stac_b03, stac_b02)
# 取得した衛星データを対象地域のみに切り取り
stac_rgb_reproject <- project(stac_rgb, "EPSG:4326")
stac_rgb_crop <- crop(x = stac_rgb_reproject, y = m)
# 取得した衛星データをRGB表示
plotRGB(stac_rgb_crop, axes = TRUE, stretch = "lin")
1.5 Landsat8の表示
まずは、画像を読み込みます。今回、読み込む画像のファイル名が長く、また、ファイルの種類も同じため、使用予定のファイルを一括で読み込み、その後で、バンドごとにデータを読み込みます。
images <- list.files("フォルダ名", pattern = ".TIF") # 指定フォルダに存在する".TIF"データを一括読込
b2_image <- rast(images[1])
b3_image <- rast(images[2])
b4_image <- rast(images[3])読み込んだデータの内容を確認します。まずは、ヒストグラムでデータのばらつきを確認します。
par(mfrow = c(3,1)) # 複数のヒストグラムの表示、各ヒストグラムの余白設定
hist(b2_image, breaks = 300, main = "BlueBand", xlim = c(1, 65545))
hist(b3_image, breaks = 300, main = "GreenBand", xlim = c(1, 65545))
hist(b4_image, breaks = 300, main = "RedBand", xlim = c(1, 65545))
dev.off() # ヒストグラムの設定を解除次に、画像を表示します。
par(mfrow = c(1,3)) # 複数の画像の表示
plot(b2_image, range = c(8000, 15000), col = topo.colors(45), main = "BlueBand", legend = FALSE)
plot(b3_image, range = c(7000, 13000), col = terrain.colors(45), main = "GreenBand", legend = FALSE)
plot(b4_image, range = c(6000, 12000), col = heat.colors(45), main = "RedBand", legend = FALSE)
dev.off() # 画像表示の設定を解除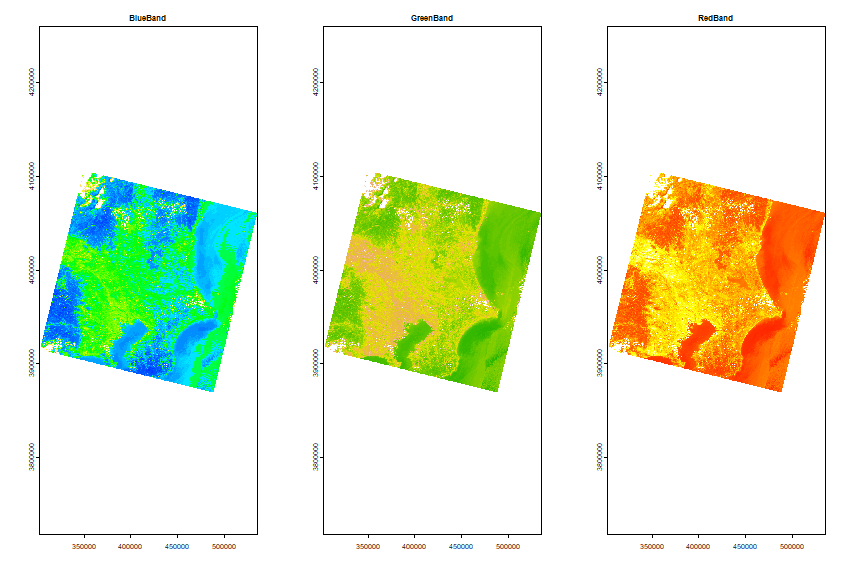
1.5.1 4隅の緯度経度に基づくポリゴンの作成
地図を見ながらのポリゴン作成はeditMap()を活用して行いましたが、今回は4隅の緯度経度からポリゴンを作成します。少しややこしいですが、以下のように作成します。
aoi <- as(raster::extent(139.7101, 139.7201, 35.6721, 35.6841), "SpatialPolygons")
aoi <- st_as_sf(aoi, crs = 4612)
st_crs(aoi) <- "+proj=longlat +ellps=WGS84 +no_defs"作成できたら、データを確認します。
mapview(aoi)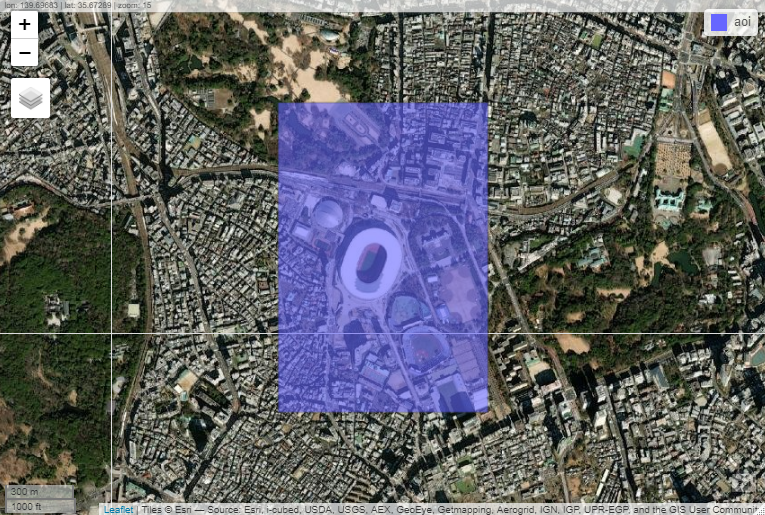
1.5.2 バンド4,8の画像を作成したポリゴンで切り取りから表示
画像の読み込みから、関心領域での切り取り、画像の表示は、上述した通りですので、割愛しますが、コードとしては以下となります。
# バンド4,8の読み込み
images <- list.files("フォルダ名", pattern = ".TIF")
b4_image <- rast(images[4])
b8_image <- rast(images[5])
# ファイルのnameを変更
names(b4_image) <- "b4"
names(b8_image) <- "b8"
# ファイルの座標変更
b4_image_reproject <- project(b4_image, "EPSG:4326")
b8_image_reproject <- project(b8_image, "EPSG:4326")
# 画像の切り出し
b4_image_crop <- crop(b4_image_reproject, aoi)
b8_image_crop <- crop(b8_image_reproject, aoi)
# 画像の表示
par(mfrow = c(1,2))
plot(b4_image_crop, col = gray.colors(100), legend = FALSE)
plot(b8_image_crop, col = gray.colors(100), legend = FALSE)
dev.off()
1.5.3 カラー合成
Landsat8のデータを用いてトゥルーカラー、フォルスカラー、ナチュラルカラー画像を表示します。手順は上述した通りですので割愛します。コードは、以下となります。
# 関心領域の生成
m2 <- editMap()
st_write(m2, "m2.geojson")
# バンド2,3,4,5の読み込み
images <- list.files("フォルダ名", pattern = ".TIF")
b2_image <- rast(images[1])
b3_image <- rast(images[2])
b4_image <- rast(images[3])
b5_image <- rast(images[4])
# ファイルのnameを変更
names(b2_image) <- "b2"
names(b3_image) <- "b3"
names(b4_image) <- "b4"
names(b5_image) <- "b5"
# ファイルの座標変更
b2_image_reproject <- project(b2_image, "EPSG:4326")
b3_image_reproject <- project(b3_image, "EPSG:4326")
b4_image_reproject <- project(b4_image, "EPSG:4326")
b5_image_reproject <- project(b5_image, "EPSG:4326")
# 画像の切り出し
b2_image_crop <- crop(b2_image_reproject, m2)
b3_image_crop <- crop(b3_image_reproject, m2)
b4_image_crop <- crop(b4_image_reproject, m2)
b5_image_crop <- crop(b5_image_reproject, m2)
# 各画像の合成
allImages <- c(b2_image_crop, b3_image_crop, b4_image_crop, b5_image_crop)
# 画像の表示
par(mfrow = c(1,3))
plotRGB(allImages, r = 3, g = 2, b = 1, stretch = "lin") # トゥルーカラー
plotRGB(allImages, r = 4, g = 3, b = 2, stretch = "lin") # フォルスカラー
plotRGB(allImages, r = 3, g = 4, b = 2, stretch = "lin") # ナチュラルカラー
dev.off()
1.5.4 パンシャープン画像の生成
Landsat8のデータでは、解像度15mのバンド8(パンクロマティック)を活用することで、解像度30mの他のバンドの解像度を15mにまで引き上げることができます。その処理のことを、パンシャープン処理と言います。
まずは、パンシャープン処理に必要なRStoolboxパッケージをインストールし、読み込みます。
install.packages("RStoolbox")
library(RStoolbox)次に、パンシャープン用のバンド8を読み込みます。パンシャープン処理を受ける他のバンドについては先ほど作成した変数allImagesを用います。また、パンシャープン処理を行うには、パンシャープン処理の基になるデータ(ここではバンド8)とパンシャープン処理を受けるデータ(ここではallImages)の情報(crsなど)を同じにする必要がありますので、その加工もしておきます。
# バンド8の読み込み
b8_image <- rast(images[5])
# ファイルのnameを変更
names(b8_image) <- "b8"
# ファイルの座標変更
b8_image_reproject <- project(b8_image, "EPSG:4326")
# 画像の切り出し
b8_image_crop <- crop(b8_image_reproject, m2)いよいよ、パンシャープン処理を行います。パンシャープン処理は、RStoolboxパッケージのpanSharpenを用いることでできます。コードとしては次のようになります。 RStoolbox::panSharpen(パンシャープン処理を受けるデータ, パンシャープン処理の基になるデータ, RGBの設定, パンシャープン処理の方法)
allImages_pan <- RStoolbox::panSharpen(allImages, b8_image_crop, r = 3, g = 2, b = 1, method = "brovey")パンシャープン画像を生成できましたので、元画像と比較します。
par(mfrow = c(1,2))
plotRGB(allImages, r = 3, g = 2, b = 1, stretch = "lin")
plotRGB(allImages_pan, r = 3, g = 2, b = 1, stretch = "lin")
dev.off()